É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Linearização do Ensaio de Proteína Bradford
Neste Artigo
Resumo
A precisão e sensibilidade da determinação de proteína pelo ensaio Bradford rápida e conveniente é comprometida pela não-linearidade intrínseca. Nós mostramos um procedimento de linearização simples que aumenta a precisão, melhora a sensibilidade do ensaio cerca de 10 vezes, e reduz significativamente a interferência por detergentes.
Resumo
Determinação das quantidades microgramas de proteína no ensaio Bradford Coomassie azul brilhante é realizado pela medição da absorbância a 590 nm. Este ensaio mais comum permite a quantificação de proteínas rápidas e simples em lisados celulares, frações celulares, ou amostras de proteínas recombinantes, com a finalidade de normalização das dosagens bioquímicas. No entanto, uma não-linearidade intrínseca compromete a sensibilidade e precisão deste método. É mostrado que, sob condições de ensaio padrão, a relação entre as medidas de absorbância a 590 nm e 450 nm é estritamente linear com a concentração de proteína. Este simples procedimento aumenta a precisão e melhora a sensibilidade do ensaio cerca de 10 vezes quantificação, permitindo até 50 ng de albumina bovina. Além disso, a interferência comumente introduzidas por detergentes que são utilizados para criar a lisados celulares é muito reduzida pelo novo protocolo. Uma equação linear desenvolvido com base em ação de massa e lei de Beer se encaixa perfeitamente os dados experimentais.
Protocolo
Linearização da curva de calibração Protein Bradford:
O ensaio de proteína Coomassie azul brilhante, vulgarmente conhecido como o ensaio de Bradford 1, é amplamente utilizado devido ao seu protocolo rápida e conveniente, bem como a sua sensibilidade relativa. Infelizmente, não há um grande grau de curvatura sobre uma ampla gama de concentrações de proteína (Fig. 1). Portanto, somente uma estreita faixa de concentrações de proteína relativamente alto, 2-10 mg / ml BSA, é usado para o gráfico de calibração, que então se encaixa melhor regressão linear (Fig. 1, verde). No entanto, a não-linearidade exige concentração de proteína das amostras desconhecidas para cair dentro da faixa limitada do gráfico de calibração, a fim de evitar um grande erro, e também reduz a precisão dentro da faixa limitada. A não-linearidade apresenta um sério problema em particular quando as quantias microgramas de proteínas não estão disponíveis, e que muitas vezes requer várias diluições das amostras desconhecidas.
Como observado no papel Bradford original, "a fonte da não-linearidade é o reagente em si uma vez que há uma sobreposição no espectro das duas formas de cores diferentes do corante." 1. Na verdade, três formas de o corante Coomassie azul brilhante estão em equilíbrio ácido-base no pH ácido usual do ensaio 2. Os formulários de vermelho, azul e verde têm absorbância maxima de 470, 590 e 650 nm, respectivamente (Fig. 2). O azul é a forma que se liga a proteínas, formando um complexo que absorve a luz intensa em 594 nm 3, 4 (Fig. 2). Bradford também observou que "o valor de fundo para o reagente está continuamente diminuindo à medida que mais corante é ligada à proteína" 1 (Fig. 3). Portanto, buscou-se calcular a redução do fundo nm 590 como quantidades de proteína crescente são adicionados, medindo a variação de absorvância a 450 nm, onde o complexo proteína-corante não absorve. Nós descobrimos que o fundo diminuindo parcialmente, mas não totalmente, as contas para a não-linearidade (Fig. 4).
Em seguida, a hipótese de que a diminuição da concentração de corante livre produz outra distorção da resposta linear, porque como proteínas dye-binding está em equilíbrio 5, a formação do complexo não depende apenas da concentração da proteína livre, mas também na da tintura livre (Fig. 5). Tendo em conta as questões relacionadas com a concentração variável do corante livre, nós desenvolvemos uma equação matemática que descreve uma relação linear entre a concentração de proteína ea relação de medidas de absorbância, 590 nm mais de 450 nm (Fig. 6). Uma descrição detalhada do estudo teórico e experimental podem ser encontrados em nossa publicação de 1996, em Analytical Biochemistry 6. A equação matemática foi experimentalmente testado e está a produzir uma curva de calibração linear em toda a faixa concentrações de proteína (Fig. 7). Além disso, a equação foi validada também por uma determinação independente do valor do pH-dependente correta da interceptação do eixo Y 6.
Protocolo detalhado para o ensaio Melhor Proteína Bradford, utilizando um leitor de Absorbância Microplaca:
- Prepare uma solução estoque 0,1 mg / ml do padrão, albumina de soro bovino. Qualquer outra norma pode ser escolhido, mas note que o mesmo padrão deve ser utilizado em todos os experimentos.
- Diluir as amostras desconhecidas em água deionizada. Objetivo de 50-50 mcg / ml. No entanto, as concentrações de proteína maior ou menor são aceitáveis, já que não há limite aparente para o intervalo linear do ensaio. No entanto, a medição deve estar dentro da faixa linear do leitor absorbância.
- Diluir o reagente de Bradford (Bio-Rad) de 2,5 vezes em água deionizada.
- Adicionar 0 e 10-50 mL de solução estoque de BSA poços triplicado (criação de uma curva de calibração 0-5 mg BSA). Complementar com água deionizada para alcançar 100 l / poço.
- Em poços diferentes, adicione 100 ml da amostra desconhecida em triplicatas. Várias concentrações da amostra desconhecida pode ser usado para aumentar a precisão.
- Adicionar 100 ul do reagente de Bradford diluído para todos os poços. Volume total é de 200 mL / poço.
- Espere pelo menos 5 min, mas não mais de 60 min para o desenvolvimento de cor.
- O branco deve ser de 200 mL de água deionizada (e não o corante de proteínas de zero também).
- Medir a absorvância a 590 nm e 450 nm.
- Prepare uma curva de calibração, dividindo os valores de absorbância líquida a 590 nm e 450 nm. Note-se que a proteína zero (corante apenas) o valor deve ser incluído como um ponto de dados (Fig. 8).
- Calcular a concentração da amostra desconhecida com base na equação linear da curva de calibração (Fig. 9).
Resultados representativos:
Ao contrário da absorvância a um único comprimento de onda de 590 nm, a proporção de valores de absorbância, 590 nm mais de 450 nm, é linear wconcentração de proteína i (Fig. 10).
A concentração de proteína da amostra desconhecida pode ser simplesmente calculado usando a equação linear da curva de calibração (Fig. 10, a equação).
No entanto, uma maior precisão é obtida através da medição várias diluições de amostras desconhecidas. Para este efeito, prepare dois gráficos. O primeiro é um gráfico de calibração para o padrão com proteína mg no eixo X (Fig. 11, direita). O segundo gráfico é para a amostra desconhecida, com mL da amostra original não diluído no eixo X (Fig. 11, à esquerda). O valor da tintura só devem ser incluídos em ambos os gráficos (Fig. 11). A concentração de proteína da amostra desconhecida é derivado dividindo as pistas da amostra desconhecida e do padrão (Fig. 11, a equação).
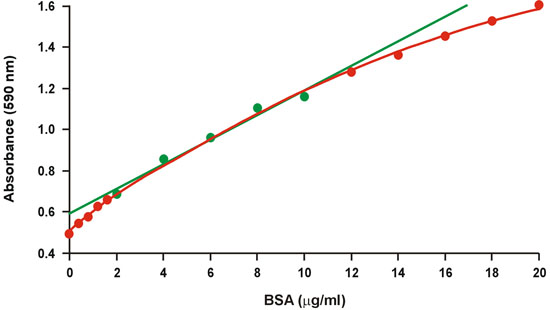
Figura 1. Gráfico de calibração convencional Bradford. A faixa linear é representado por símbolos verdes.

Figura 2. Spectra do complexo proteína-corante e do corante sozinho.

Figura 3. Protein equilíbrio dye.
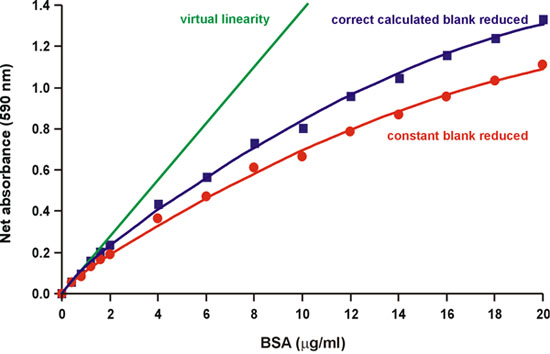
Figura 4. Redução do fundo correta calculada diminui parcialmente não-linearidade.
Figura 5. Protein dye-equilíbrio.

Figura 6. Base matemática para a linearização do ensaio de proteína Bradford.
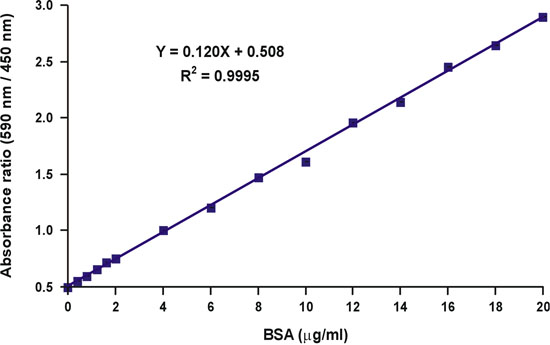
Figura 7. Linearização da curva de calibração Bradford.

Figura 8. Cálculo dos valores de absorbância relação.

Figura 9. A curva de calibração linear Bradford.
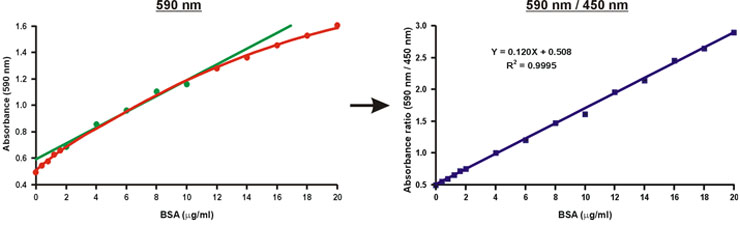
Figura 10. Linearização da curva de calibração Bradford.
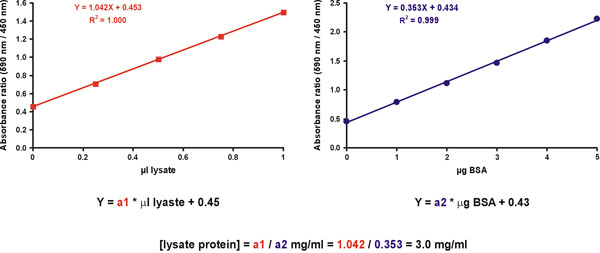
Figura 11. Cálculo da amostra concentração Desconhecido.
Discussão
O ensaio de proteína Bradford é popular devido à sua facilidade de desempenho e de sensibilidade relativa. A linearização sobre as concentrações de proteína toda a gama obtidos pelo protocolo aqui apresentado simplifica ainda mais o ensaio, como amostras desconhecidas não precisa estar dentro do intervalo da curva de calibração.
Importante, o protocolo prevê ainda melhorou as seguintes vantagens sobre o original Bradford protocolo:
- Maior precisão.
- A sensib...
Agradecimentos
Este artigo é dedicado à memória do falecido Dr. Zvi Selinger, que sediou a pesquisa original descreveu aqui.
Materiais
| Name | Company | Catalog Number | Comments |
| Bradford reagent | Bio-Rad | 500-0006 | |
| Bovine Serum Albumin | Amersco | 0332 | |
| Multiplate absorbance reader | BioTek | Synergy2 |
Referências
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem. 72, 248-254 (1976).
- Chial, H. J., Thompson, H. B., Splittgerber, A. G. A spectral study of the charge forms of Coomassie blue G. Anal Biochem. 209, 258-266 (1993).
- Chial, H. J., Splittgerber, A. G. A comparison of the binding of Coomassie brilliant blue to proteins at low and neutral pH. Anal Biochem. 213, 362-369 (1993).
- Compton, S. J., Jones, C. G. Mechanism of dye response and interference in the Bradford protein assay. Anal Biochem. 151, 369-374 (1985).
- Congdon, R. W., Muth, G. W., Splittgerber, A. G. The binding interaction of Coomassie blue with proteins. Anal Biochem. 213, 407-413 (1993).
- Zor, T., Selinger, Z. Linearization of the Bradford protein assay increases its sensitivity: theoretical and experimental studies. Anal Biochem. 236, 302-308 (1996).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados