Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Linéarisation du dosage des protéines Bradford
Dans cet article
Résumé
La précision et la sensibilité du dosage des protéines par le test rapide et pratique Bradford est compromise par la non-linéarité intrinsèque. Nous montrons une procédure de linéarisation simple qui augmente considérablement la précision, améliore la sensibilité du dosage d'environ 10 fois, et réduit considérablement les interférences par les détergents.
Résumé
Détermination des quantités de microgrammes de protéine de Bradford dans le bleu de Coomassie brillant essai est réalisé par la mesure de l'absorbance à 590 nm. Ce test permet de quantifier la plus courante de protéines rapides et simples dans les lysats cellulaires, les fractions cellulaires, ou des échantillons de protéines recombinantes, dans le but de la normalisation des mesures biochimiques. Toutefois, une non-linéarité intrinsèque compromis la sensibilité et la précision de cette méthode. Il est montré que dans des conditions test standard, le ratio des mesures d'absorbance à 590 nm et 450 nm est strictement linéaire avec la concentration de protéines. Cette procédure simple augmente la précision et améliore la sensibilité du dosage d'environ 10 fois, la quantification permettant à 50 ng d'albumine sérique bovine. Par ailleurs, le brouillage couramment introduites par les détergents qui sont utilisés pour créer les lysats cellulaires est fortement réduite par le nouveau protocole. Une équation linéaire développé sur la base d'une action de masse et la loi de Beer s'adapte parfaitement aux données expérimentales.
Protocole
Linéarisation du graphique d'étalonnage Bradford Protéines:
Le dosage de Coomassie brillant protéines bleu, communément connu comme le dosage de Bradford 1, est largement utilisé en raison de son protocole rapide et pratique ainsi que sa sensibilité relative. Malheureusement, il ya un grand degré de courbure sur une large gamme de concentrations de protéines (Fig. 1). Par conséquent, seule une gamme étroite de concentrations en protéines relativement élevée, 2-10 mg / ml de BSA, est utilisé pour la courbe d'étalonnage, ce qui correspond le mieux, puis de régression linéaire (Fig. 1, en vert). Cependant, la non-linéarité exige de la concentration en protéines des échantillons inconnus à tomber dans la fourchette limitée de la courbe d'étalonnage afin d'éviter une erreur importante, et il réduit aussi la précision dans la gamme limitée. La non-linéarité présente un problème sérieux, en particulier lorsque des quantités microgrammes de protéine ne sont pas disponibles, et il faut souvent plusieurs dilutions des échantillons inconnus.
Comme indiqué dans le document original Bradford, «la source de la non-linéarité est dans le réactif lui-même car il ya un chevauchement dans le spectre des deux formes différentes couleurs de la teinture." 1. En fait, trois formes de la teinture bleu de Coomassie sont en équilibre acido-basique au pH acide de l'habitude de dosage 2. Les formes rouges, bleues et vertes ont absorbance maxima à 470, 590 et 650 nm, respectivement (Fig. 2). Le bleu est la forme qui lie la protéine, formant un complexe qui absorbe intensément la lumière à 594 nm 3, 4 (fig. 2). Bradford a également noté que "la valeur du fond pour le réactif ne cesse de diminuer comme plus colorant est liée à la protéine" 1 (Fig. 3). Par conséquent, nous avons tenté de calculer la réduction de l'arrière-plan 590 nm comme les quantités de protéines augmente sont ajoutés, en mesurant la variation d'absorbance à 450 nm, où le complexe protéine-colorant n'absorbe pas. Nous avons constaté que le fond diminue partiellement, mais pas complètement, les comptes pour la non-linéarité (Fig. 4).
Nous avons alors émis l'hypothèse que la diminution de la concentration du colorant libre produit une autre distorsion de la réponse linéaire, parce que la protéine-colorant liant est en équilibre 5, la formation de complexes ne dépend pas seulement de la concentration de la protéine libre, mais aussi sur celle du colorant libre (fig. 5). Prenant en compte à la fois les questions liées à la concentration variable du colorant libre, nous avons développé une équation mathématique qui décrit une relation linéaire entre la concentration de protéines et le ratio des mesures d'absorbance, 590 nm sur 450 nm (Fig. 6). Une description détaillée de l'étude théorique et expérimentale peut être trouvé dans notre publication de 1996 dans Analytical Biochemistry 6. L'équation mathématique a été testé et trouvé expérimentalement pour obtenir une courbe d'étalonnage linéaire sur la protéine concentrations varient (Fig. 7) tout entier. Par ailleurs, l'équation a été validé également par une détermination indépendante de la bonne valeur de pH-dépendante de l'interception de l'axe Y 6.
Protocole détaillé pour le dosage de Bradford protéine améliorée, en utilisant un lecteur d'absorbance pour microplaques:
- Préparer une solution à 0,1 mg / ml de la norme, l'albumine sérique bovine. Toute autre norme peut être choisi, mais il faut noter que la même norme doit être utilisée dans toutes les expériences.
- Diluer les échantillons inconnus dans de l'eau déminéralisée. Viser à 50-50 pg / ml. Pourtant, les concentrations en protéines supérieures ou inférieures sont acceptables, car il n'ya pas de limite apparente de la gamme linéaire du dosage. Toutefois, la mesure doit être comprise dans la gamme linéaire du lecteur d'absorbance.
- Diluer le réactif de Bradford (Bio-Rad) de 2,5 fois dans de l'eau déminéralisée.
- Ajouter 0 et 10-50 ul de la solution stock de BSA à trois puits (création d'une courbe de calibration 0-5 mg BSA). Complément à l'eau déminéralisée pour atteindre 100 l / puits.
- Dans les différents puits, ajouter 100 ul d'échantillon inconnu en triple. Plusieurs concentrations de l'échantillon inconnu peut être utilisé pour augmenter la précision.
- Ajouter 100 ul de réactif dilué Bradford à tous les puits. Le volume total est de 200 l / puits.
- Attendez au moins 5 minutes, mais pas plus de 60 min pour le développement de couleur.
- Le blanc doit être de 200 l d'eau déminéralisée (et pas le colorant de protéines de zéro bien).
- Mesurer l'absorbance à 590 nm et à 450 nm.
- Préparer une courbe d'étalonnage en divisant les valeurs d'absorbance nette à 590 nm et à 450 nm. Notez que la protéine zéro (colorant uniquement) valeur doit être incluse comme un point de données (fig. 8).
- Calculer la concentration de l'échantillon inconnu basé sur l'équation linéaire de la courbe de calibration (fig. 9).
Les résultats représentatifs:
Contrairement à l'absorbance à une seule longueur d'onde de 590 nm, le rapport des valeurs d'absorbance, 590 nm sur 450 nm, est linéaire wvec la concentration de protéines (Fig. 10).
La concentration en protéines de l'échantillon inconnu peut être simplement calculé selon l'équation linéaire de la courbe de calibration (fig. 10, l'équation).
Toutefois, une précision accrue est obtenue en mesurant plusieurs dilutions des échantillons inconnus. À cette fin, préparer deux graphiques. Le premier est un graphique d'étalonnage pour la norme avec ug de protéines sur l'axe X (fig. 11, à droite). Le deuxième graphique est pour l'échantillon inconnu, avec pl de l'échantillon non dilué d'origine sur l'axe X (Fig. 11, gauche). La valeur de teinture ne devrait être inclus dans les deux graphiques (fig. 11). La concentration en protéines de l'échantillon inconnu est obtenu en divisant les pentes de l'échantillon inconnu et la norme (fig. 11, l'équation).
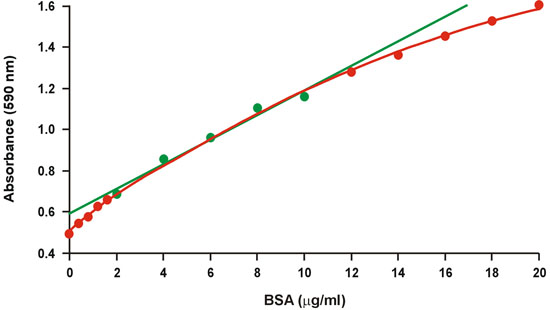
Figure 1. Conventionnel graphique d'étalonnage Bradford. La gamme linéaire est représenté par des symboles verts.

Figure 2. Spectres du complexe protéine-colorant et de la teinture seulement.

Figure 3. Protéine-colorant équilibre.
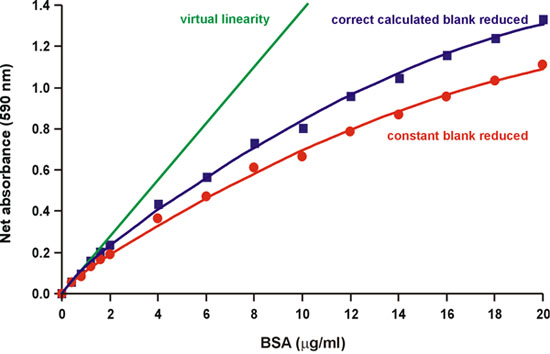
Figure 4. Réduction de l'arrière-plan correcte calculée diminue partiellement non-linéarité.
Figure 5. Protéine-colorant équilibre.

Figure 6. Base mathématique pour la linéarisation de l'analyse des protéines de Bradford.
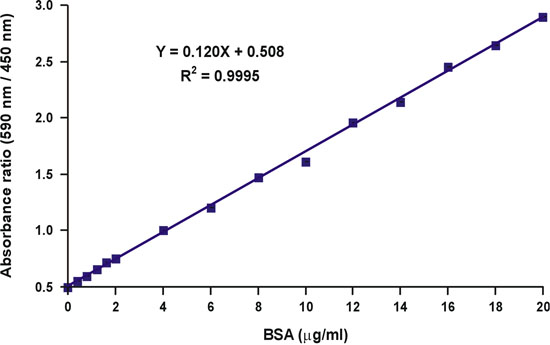
Figure 7. Linéarisation de la courbe d'étalonnage de Bradford.

Figure 8. Calcul des valeurs de rapport d'absorbance.

Figure 9. Un graphique linéaire de calibration Bradford.
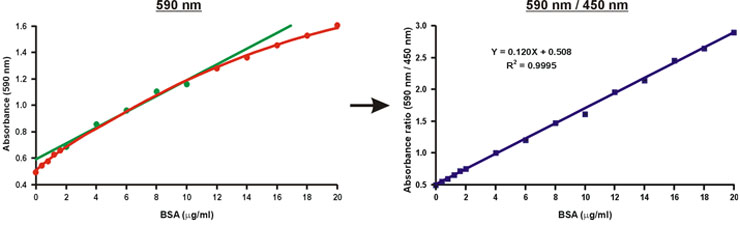
Figure 10. Linéarisation de la courbe d'étalonnage de Bradford.
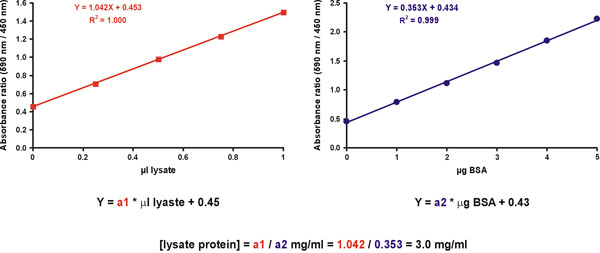
Figure 11. Inconnue échantillon de concentration de calcul.
Discussion
Le dosage des protéines de Bradford est populaire en raison de sa facilité d'exécution et de la sensibilité relative. La linéarisation sur les concentrations de protéine ensemble de la gamme obtenue par le protocole présenté ici simplifie encore le dosage, comme des échantillons inconnus n'ont pas besoin de tomber dans la plage de la courbe d'étalonnage.
Fait important, le protocole prévoit en outre améliorée les avantages suivants sur l'original du protocole de ...
Remerciements
Cet article est dédié à la mémoire du regretté Dr Zvi Selinger, qui a accueilli la recherche originale décrite ici.
matériels
| Name | Company | Catalog Number | Comments |
| Bradford reagent | Bio-Rad | 500-0006 | |
| Bovine Serum Albumin | Amersco | 0332 | |
| Multiplate absorbance reader | BioTek | Synergy2 |
Références
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem. 72, 248-254 (1976).
- Chial, H. J., Thompson, H. B., Splittgerber, A. G. A spectral study of the charge forms of Coomassie blue G. Anal Biochem. 209, 258-266 (1993).
- Chial, H. J., Splittgerber, A. G. A comparison of the binding of Coomassie brilliant blue to proteins at low and neutral pH. Anal Biochem. 213, 362-369 (1993).
- Compton, S. J., Jones, C. G. Mechanism of dye response and interference in the Bradford protein assay. Anal Biochem. 151, 369-374 (1985).
- Congdon, R. W., Muth, G. W., Splittgerber, A. G. The binding interaction of Coomassie blue with proteins. Anal Biochem. 213, 407-413 (1993).
- Zor, T., Selinger, Z. Linearization of the Bradford protein assay increases its sensitivity: theoretical and experimental studies. Anal Biochem. 236, 302-308 (1996).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon