È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Linearizzazione del saggio proteine Bradford
In questo articolo
Riepilogo
La precisione e la sensibilità di determinazione delle proteine da parte del test rapido e conveniente Bradford è compromessa dalla non linearità intrinseca. Mostriamo una semplice procedura di linearizzazione che aumenta notevolmente la precisione, migliora la sensibilità del test di circa 10 volte, e riduce significativamente le interferenze da detergenti.
Abstract
Determinazione delle quantità di microgrammi di proteina nel saggio Bradford brillante blu Coomassie si ottiene la misura di assorbanza a 590 nm. Questo test permette di quantificare più comuni proteine rapido e semplice in lisati di cellule, frazioni cellulari, o campioni di proteine ricombinanti, ai fini della normalizzazione delle misure biochimiche. Tuttavia, un non-linearità intrinseca compromette la sensibilità e la precisione di questo metodo. E 'dimostrato che in condizioni di test standard, il rapporto tra le misure di assorbanza a 590 nm e 450 nm è strettamente lineare con la concentrazione di proteine. Questa semplice procedura aumenta la precisione e migliora la sensibilità del dosaggio circa 10 volte, permettendo la quantificazione fino al 50 ng di sieroalbumina bovina. Inoltre, l'interferenza introdotta da detergenti comunemente utilizzati per creare i lisati cellulari è notevolmente ridotta dal nuovo protocollo. Una equazione lineare sviluppato sulla base di azione di massa e la legge Beer si adatta perfettamente ai dati sperimentali.
Protocollo
Linearizzazione della curva di taratura Proteine Bradford:
Il brillante saggio di proteine Coomassie blu, comunemente noto come il saggio Bradford 1, è ampiamente utilizzato per il suo protocollo rapidi e convenienti così come la sua sensibilità relativa. Purtroppo, c'è un ampio grado di curvatura su una vasta gamma di concentrazioni di proteine (Fig. 1). Pertanto, solo una gamma ristretta di relativamente alte concentrazioni di proteine, 2-10 mg / ml BSA, viene utilizzato per la curva di taratura, che poi si adatta meglio di regressione lineare (Fig. 1, verde). Tuttavia, la non linearità richiede una concentrazione proteica dei campioni sconosciuti rientrano nella gamma limitata di curva di taratura allo scopo di evitare un errore grande, e riduce anche la precisione nel range limitato. La non linearità presenta un serio problema in particolare quando quantità microgrammi di proteina non sono disponibili, e richiede spesso più diluizioni dei campioni sconosciuti.
Come sottolineato nel documento originale Bradford, "la fonte della non linearità è nel reagente stesso poiché non vi è una sovrapposizione nello spettro delle due forme colore diverso del colorante." 1. Infatti, tre forme del colorante Coomassie blu brillante in equilibrio acido-base al pH acido del solito saggio 2. Le forme rosso, blu e verde sono assorbanza massimi a 470, 590 e 650 nm, rispettivamente (Fig. 2). Il blu è la forma che lega la proteina, formando un complesso che assorbe intensamente la luce a 594 nm 3, 4 (Fig. 2). Bradford ha anche osservato che "il valore di fondo per il reagente è in costante riduzione come più colorante si lega alle proteine" 1 (Fig. 3). Pertanto, abbiamo cercato di calcolare la riduzione dello sfondo 590 nm come quantità proteico sono aggiunti, misurando la variazione di assorbanza a 450 nm, in cui la proteina-colorante non assorbe. Abbiamo trovato che lo sfondo diminuendo in parte, ma non completamente, spiega la non-linearità (Fig. 4).
Abbiamo quindi ipotizzato che la diminuzione della concentrazione di colorante libero produce un'altra distorsione della risposta lineare, perché, come proteina-colorante legame è in equilibrio 5, formazione di complessi dipende non solo dalla concentrazione della proteina libero, ma anche su quello del colorante libero (Fig. 5). Tenendo conto sia questioni legate alla concentrazione variabile del colorante libero, abbiamo sviluppato una equazione matematica che descrive una relazione lineare tra la concentrazione di proteine e il rapporto delle misure di assorbanza, 590 nm oltre 450 nm (Fig. 6). Una descrizione dettagliata dello studio teorico e sperimentale si possono trovare nel nostro 1996 pubblicazione nella biochimica analitica 6. L'equazione matematica è stata sperimentalmente testato ed è risultato fino ad ottenere una curva di calibrazione lineare su tutta la gamma concentrazioni di proteine (Fig. 7). Inoltre, l'equazione è stata convalidata anche da una determinazione indipendente dal corretto pH-dipendente valore della Y intercettare 6.
Protocollo dettagliato per la migliore analisi delle proteine Bradford, utilizzando un lettore di micropiastre Assorbanza:
- Preparare un 0,1 mg / ml soluzione madre dello standard, albumina sierica bovina. Tutte le norme possono essere scelti altri, ma si noti che lo stesso standard deve essere usato in tutti gli esperimenti.
- Diluire i campioni sconosciuti in acqua deionizzata. Puntate a 5-50 mg / ml. Tuttavia, le concentrazioni di proteine superiori o inferiori sono accettabili, poiché non vi è alcun limite apparente per la gamma lineare del test. Tuttavia, la misura deve essere all'interno della gamma lineare del lettore assorbanza.
- Diluire il reagente di Bradford (Bio-Rad) 2,5 volte in acqua deionizzata.
- Aggiungi 0 e 10-50 ml di soluzione madre di BSA pozzi triplice copia (la creazione di una curva di calibrazione mcg 0-5 BSA). Complemento con acqua deionizzata per raggiungere 100 l / pozzetto.
- In diversi pozzetti, aggiungere 100 ml di campione sconosciuto in triplicato. Diverse concentrazioni del campione sconosciuto può essere utilizzato per aumentare la precisione.
- Aggiungere 100 l di reagente diluito Bradford a tutti i pozzetti. Volume totale è di 200 microlitri / bene.
- Attendere almeno 5 minuti, ma non più di 60 minuti per lo sviluppo del colore.
- Il vuoto deve essere di 200 ml di acqua deionizzata (e non il colorante proteico pari a zero bene).
- Misurare l'assorbanza a 590 nm e 450 nm.
- Preparare una curva di taratura dividendo i valori di assorbanza netta a 590 nm e 450 nm. Si noti che la proteina zero (colorante solo) valore deve essere inclusa come un punto di dati (Fig. 8).
- Calcolare la concentrazione del campione sconosciuto in base all'equazione lineare della curva di calibrazione (Fig. 9).
Rappresentante dei risultati:
A differenza l'assorbanza ad una singola lunghezza d'onda di 590 nm, il rapporto tra i valori di assorbanza, più di 590 nm a 450 nm, è lineare wconcentrazione di proteine-esimo (fig. 10).
La concentrazione proteica del campione sconosciuto può essere semplicemente calcolato utilizzando l'equazione lineare della curva di calibrazione (Fig. 10, l'equazione).
Tuttavia, una maggiore precisione si ottiene misurando diverse diluizioni del campione sconosciuto. A tal fine, preparare due grafici. La prima è una curva di taratura per lo standard con proteine mcg sull'asse X (Fig. 11, a destra). Il secondo grafico è per il campione incognito, con l di campione originale non diluito sull'asse X (Fig. 11, a sinistra). Il valore di colorante solo dovrebbero essere incluse in entrambi i grafici (Fig. 11). La concentrazione proteica del campione sconosciuto si ottiene dividendo le pendici del campione sconosciuto e lo standard (Fig. 11, l'equazione).
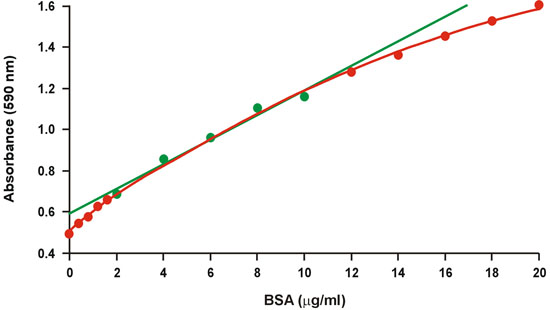
Figura 1. Convenzionale grafico di calibrazione Bradford. Il range lineare è rappresentata da simboli verdi.

Figura 2. Spectra della proteina-colorante e del colorante da solo.

Figura 3. Proteina-colorante equilibrio.
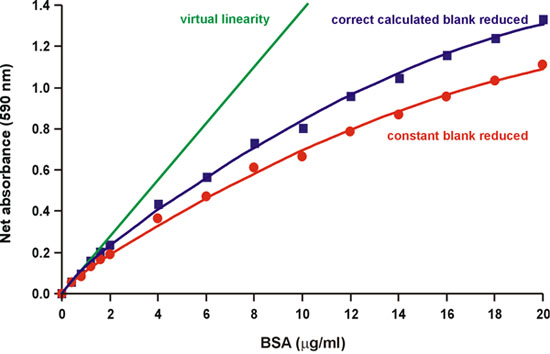
Figura 4. Riduzione del fondo corretta calcolato diminuisce parzialmente non-linearità.
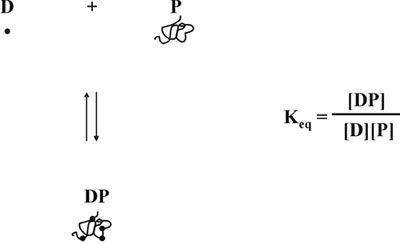
Figura 5. Proteina-colorante equilibrio.

Figura 6. Fondamento matematico per la linearizzazione del dosaggio proteina Bradford.
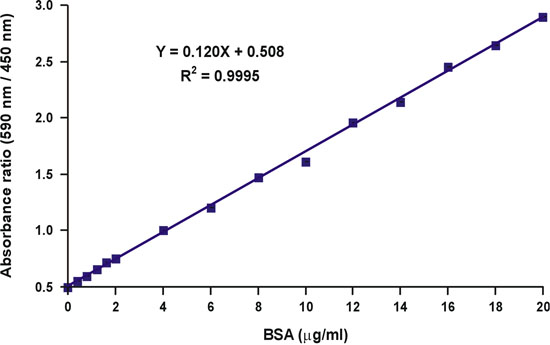
Figura 7. Linearizzazione della curva di taratura Bradford.

Figura 8. Calcolo dei valori di rapporto tra l'assorbanza.

Figura 9. Una calibrazione lineare Bradford grafico.
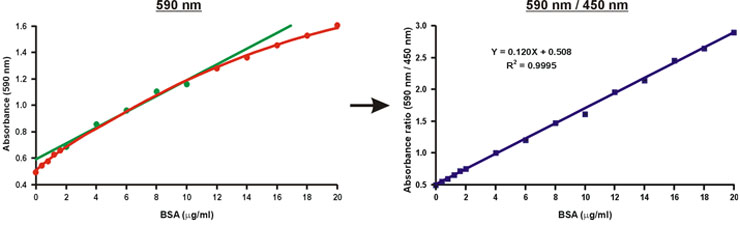
Figura 10. Linearizzazione della curva di taratura Bradford.
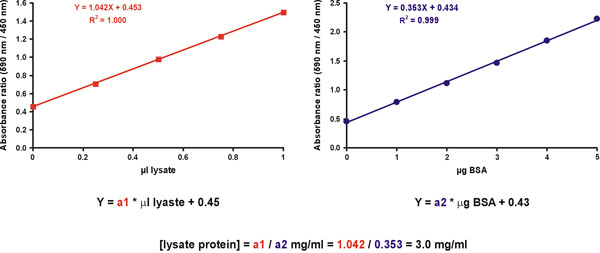
Figura 11. Sconosciuto campione concentrazione calcolo.
Discussione
Il dosaggio di proteine Bradford è popolare grazie alla sua facilità di prestazioni e di sensibilità relativa. La linearizzazione sulle concentrazioni proteina intera gamma ottenuto dal protocollo presentato qui semplifica ulteriormente l'analisi, come campioni sconosciuti non hanno bisogno di rientrare nel campo della curva di calibrazione.
È importante sottolineare che il protocollo migliorata Provvede inoltre i seguenti vantaggi rispetto l'originale Bradford protocollo: ...
Riconoscimenti
Questo saggio è dedicato alla memoria del compianto Dr. Zvi Selinger, che ha ospitato la ricerca originale descritta qui.
Materiali
| Name | Company | Catalog Number | Comments |
| Bradford reagent | Bio-Rad | 500-0006 | |
| Bovine Serum Albumin | Amersco | 0332 | |
| Multiplate absorbance reader | BioTek | Synergy2 |
Riferimenti
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem. 72, 248-254 (1976).
- Chial, H. J., Thompson, H. B., Splittgerber, A. G. A spectral study of the charge forms of Coomassie blue G. Anal Biochem. 209, 258-266 (1993).
- Chial, H. J., Splittgerber, A. G. A comparison of the binding of Coomassie brilliant blue to proteins at low and neutral pH. Anal Biochem. 213, 362-369 (1993).
- Compton, S. J., Jones, C. G. Mechanism of dye response and interference in the Bradford protein assay. Anal Biochem. 151, 369-374 (1985).
- Congdon, R. W., Muth, G. W., Splittgerber, A. G. The binding interaction of Coomassie blue with proteins. Anal Biochem. 213, 407-413 (1993).
- Zor, T., Selinger, Z. Linearization of the Bradford protein assay increases its sensitivity: theoretical and experimental studies. Anal Biochem. 236, 302-308 (1996).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon