Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Fluorescencia de Complementación bimolecular
En este artículo
Resumen
La localización subcelular de las proteínas es importante para determinar la regulación espacio-temporal de la señalización celular. Aquí se describe la complementación bimolecular de fluorescencia (BiFC) como un método sencillo para el control de la interacción espacial de las proteínas en la célula.
Resumen
La definición de la distribución subcelular de los complejos de señalización es imprescindible para entender el resultado de ese complejo. Los métodos convencionales, tales como la inmunoprecipitación no proporcionan información sobre la localización espacial de los complejos. Por el contrario, BiFC supervisa la interacción y la compartimentación subcelular de los complejos de proteínas. En este método, una proteína fluororescent se divide en amino-y carboxi-terminal no fluorescente fragmentos que se fusionan dos proteínas de interés. La interacción de los resultados de las proteínas en la reconstitución del fluoróforo (Figura 1) 1,2. Una limitación de BiFC es que una vez que el fluoróforo fragmentada se reconstituye el complejo es irreversible 3. Esta limitación es una ventaja en la detección de interacciones transitorias o débil, pero se opone a un análisis cinético de la compleja dinámica. Una advertencia adicional es que el flourophore reconstituido requiere 30 minutos para madurar y fluorescentes, una vez más se opone a la observación de las interacciones en tiempo real 4. BiFC es un ejemplo específico de la proteína de ensayo de complementación de fragmentos (PCA), que emplea las proteínas reportero como el verde variantes de la proteína fluorescente (BiFC), la dihidrofolato reductasa, b-lactamasa, y luciferasa para medir las proteínas: las interacciones proteína 5,6. Métodos alternativos para el estudio de proteínas: las interacciones de proteínas en las células incluyen fluorescencia co-localización y transferencia de energía por resonancia de Förster (FRET) 7. De co-localización, dos proteínas son etiquetados individualmente, ya sea directamente con un fluoróforo o por inmunofluorescencia indirecta. Sin embargo, este enfoque lleva a un segundo plano alto de la no interacción de proteínas por lo que es difícil de interpretar co-localización de los datos. Además, debido a los límites de la resolución de la microscopía confocal, dos proteínas pueden aparecer co-localizados sin necesidad de interactuar. Con BiFC, la fluorescencia sólo se observa cuando las dos proteínas de interés interactúan. FRET es otro método excelente para el estudio de las proteínas: las interacciones proteína, pero puede ser técnicamente difícil. FRET experimentos requieren el donante y el receptor a ser un brillo similar y la estequiometría de la célula. Además, uno debe tener en cuenta para sangrar a través de los donantes en el canal aceptor y viceversa. A diferencia de FRET, BiFC tiene pocos antecedentes de procesamiento de fluorescencia, poco después de los datos de imagen, no requiere de la sobreexpresión de alta, y puede detectar interacciones débiles o transitoria. Bioluminiscencia la transferencia de energía de resonancia (BRET) es un método similar al FRET salvo que el donante es una enzima (luciferasa, por ejemplo) que cataliza un sustrato para ser bioluminiscentes lo emocionante aceptador. BRET carece de los problemas técnicos de la sangre a través de la fluorescencia de fondo y alto, pero carece de la capacidad de proporcionar la información espacial, debido a la falta de localización del sustrato a compartimentos específicos 8. En general, BiFC es un excelente método para visualizar la localización subcelular de los complejos de proteínas para profundizar en la señalización en compartimientos.
Protocolo
A. BiFC calibración
- Elegir un fluoróforo. Hay varios fluoróforos, como YFP y Venus, que funcionan bien como BiFC socios de la fusión (Tabla 1). Extremos amino y carboxi-terminal de Venus son capaces de formar un complejo a 37 ° C, mientras que los fragmentos YFP BiFC requieren un pre-incubación a 30 ° C con el fin de facilitar la formación del fluoróforo 2. Esta incubación a una temperatura baja puede alterar algunos procesos celulares y se debe tomar en cuenta al elegir los fragmentos. Los vectores de la fusión de Venus con el extremo carboxilo de las proteínas están disponibles en Addgene ( http://www.addgene.org/pgvec1 ; seepBiFC-VN173 y pBiFC VC155-), junto con construcciones adicionales para su uso como controles, por ejemplo, pBiFC-bJunVN173 y pBiFC bFosVC155-2. Vectores adicionales, incluyendo amino-terminal de vectores de Venus (PFLAG-VN173 y PHA-VC155), están disponibles en la siguiente dirección: http://people.pnhs.purdue.edu/ hu1 ~ / .
- Etiqueta de la proteína de interés. Los fragmentos BiFC se funden en los extremos amino o carboxi-terminal de las proteínas candidato. Algunas proteínas no puede permitir que para el etiquetado en los extremos debido a la interrupción de la función de la proteína. Por ejemplo, muchos miembros de la superfamilia Ras de GTPasas son de lípidos modificados en el archivo adjunto carboxi impidiendo así de los fragmentos BiFC en ese extremo. Por lo tanto, es importante tener una idea de cómo la unión de los fragmentos BiFC puede afectar la función de las proteínas de interés. Si no está claro cómo el etiquetado de una proteína que afecta a su función de múltiples combinaciones deben ser probados. Además de los fragmentos BiFC, un péptido se pueden incluir para aumentar la flexibilidad entre el fluoróforo fragmentado y las proteínas candidato. Mientras que los sitios de clonación múltiple (MCS) en los vectores BiFC codificar corta tramos de aminoácidos que pueden proporcionar la suficiente flexibilidad, la RSIAT, KQKVMNH, y enlazadores RPACKIPNDLKQKVMNH han utilizado con éxito en experimentos BiFC 3,9
- Determinar las condiciones de transfección. Antes de probar varios mutantes, un control de los experimentos se deben realizar unos cuantos. Las dos primeras combinaciones BiFC que debería ser juzgado dos proteínas de tipo salvaje que se sabe que interactúan y un tipo salvaje y mutante que no interactúan. El uso de estas dos combinaciones, diferentes cantidades de ADN y los tiempos de la transfección debe ser probado para determinar las condiciones óptimas para la detección de una señal BiFC de las proteínas candidato. Le sugerimos probar 0.25ug, 0.5ug, 1.0ug de cada construcción BiFC de un solo pozo de una placa de 6 pocillos. El día después de la transfección las células seguimiento por microscopía de fluorescencia para determinar el tiempo óptimo para el desarrollo de la señal. El pBiFC-bJunVN173 y pBiFC-bFosVC155 construye dos son útiles los controles positivos en BiFC y están disponibles en Addgene (ver arriba). Además, el análisis de Western blot se debe realizar para confirmar la expresión de la igualdad de las construcciones. Las condiciones deben ser escogidos de tal manera que una señal fluorescente que se observa entre las dos proteínas de tipo salvaje, pero muy poca o ninguna señal de que se observa entre el tipo salvaje y las proteínas mutantes. Por último, lo mejor es mantener la expresión de proteínas lo más baja posible para evitar cualquier interacción no específica.
- Determinar si la adición de los fragmentos BiFC altera la localización de las proteínas de interés. Cada construcción BiFC contiene ya sea un HA o etiqueta de epítopo FLAG. Inmunotinción transefected células, tanto para el HA o etiqueta de epítopo FLAG, así como también la proteína endógena (si es posible) para determinar si la etiqueta BiFC afecta a la localización de las proteínas de interés.
B. Revestimiento y transfección de células
- Las células COS (1.3x10 5) se colocan cada uno en un fondo de la placa de vidrio Matek y dos pocillos de una placa de 6 pocillos por muestra. Permiten a las células reposar toda la noche a 37 ° C. Otros tipos de células que son más relevantes para las proteínas candidatas de interés también puede ser utilizado.
- Prepare las autoridades nacionales designadas para la transfección. Por lo general utilizan Lipofectamine (Invitrogen) para transfecciones COS. Sin embargo, otros reactivos pueden ser más apropiados para la línea celular de interés. Dado que la mezcla de transfección se dividirá entre un plato con fondo de cristal y dos pocillos de una placa de 6 pocillos, use la cantidad apropiada de ADN para dar cuenta de esta división. Diluir el ADN en 250uL de suero libre (SF) DMEM. Añadir PPC a 1 / 5 la cantidad total de ADN BiFC como control de transfección. Para la cuantificación BiFC, sólo las células que son positivas para el PPC será analizada para detectar la presencia de una señal de BiFC. Tenga en cuenta que el espectro de la PPC se solapará con algunos de los pares BiFC en la Tabla 1, por lo tanto un control de transfección alternativotal vez sea necesario. Diluir Lipofectamine en 250uL SF DMEM (10 ul Lipofectamine/1ug de ADN). Mezcla el ADN y las diluciones Lipofectamine. Incubar a temperatura ambiente durante 20 minutos.
Nota: El Lipofectamine: relación de ADN puede variar dependiendo del tipo de célula. Utilice el método apropiado para la transfección de la línea celular de interés. - Enjuague con agua tibia células 2x DMEM SF. Añadir 2 ml de SF DMEM a cada plato con fondo de cristal o bien de un plato de 6 pocillos.
- Dividir cada mezcla de transfección en partes iguales entre un plato con fondo de cristal y dos pozos de una placa de 6 pocillos.
- Se incuban las células a 37 ° C durante 5h.
- Retire el medio de transfección y reemplazar con los medios de comunicación completo (DMEM +10% FBS).
- Las células se incuban durante la noche a 37 ° C. La longitud de la transfección después de incubación puede variar en función de los niveles de expresión de las proteínas de interés. Incubación prolongado puede resultar en la no interacción específica con lo que este paso tendrá que ser determinada empíricamente.
C. Preparación de las células de la Imagen
- Las células son inicialmente examinados con un microscopio de epifluorescencia para asegurar que el control positivo es fluorescente. Si no, puede ser necesario para dar tiempo a otras células a 37 ° C hasta que la señal que se observa.
- Enjuague las células 3 veces con PBS (pH 7,4). A las células en el plato con fondo de cristal añadir paraformaldehído al 2% (pH 7,4). Fijar las células durante 10 minutos en hielo. Enjuague las células 3 veces con PBS (pH 7,4). Las células se almacenan a 4 ° C cubierto con 1 ml de PBS (pH 7,4). Las células no tienen que ser fijos para las imágenes, pero una vez que los fragmentos de la reforma BiFC un fluoróforo intacta es irreversible lo que impide el análisis de las interacciones dinámicas 3. Además, tenga en cuenta que las células no fijadas continuará desarrollando la señal. Lisis de las células en la placa de 6 pocillos y se preparan lisados de Western blot. Es importante que todas las células están dispuestas en el mismo tiempo para que las células lisadas son representativos de las células de imágenes.
D. Las células de imagen
- La intensidad de la fluorescencia se calculará por celda. Asegúrese de que las células individuales de la imagen.
- PPC se incluyó como un control tranfection y sólo las células positivas PPC son seleccionados para el análisis de las señales de BiFC. Hacemos la suposición de que si la célula es transfectada con la PPC, también es transfectadas con las construcciones BiFC. Este enfoque asegura que las células de imágenes que carecen de una señal de BiFC son negativos debido a la falta de una interacción entre las proteínas de interés, y no debido a la ausencia de uno o dos construcciones de expresión BiFC en esa celda.
- Utilizamos un Zeiss LSM 510 microscopio confocal de imágenes de células. Cuando se usa este microscopio es importante para mantener el zoom, diafragma, ganancia del detector, el amplificador de compensación, tamaño, velocidad de exploración, el promedio de exploración, y la potencia del láser coherente. Al utilizar cualquier sistema de imagen es importante para mantener la configuración constante, de modo que la fluorescencia es comparable entre las muestras. Además, la hora de cuantificar la fluorescencia es importante que los píxeles no están saturados.
E. Cuantificación de fluorescencia
- Fluorescencia puede cuantificarse utilizando cualquier software de imágenes. Utilizamos ImageJ que está disponible gratuitamente desde el NIH (http://rsb.info.nih.gov/ij/). Abrir archivos de imagen en ImageJ. Ir a mediciones AnalyzeSet. Marque las casillas para el área y el valor medio gris en el cuadro de medidas.
- Utilizando la herramienta de "selección de manos libres", dibujar un contorno alrededor del borde de la célula entera en el canal de la PPC.
- Salir de este esquema en su lugar, el cambio en el canal de YFP. Ir al AnalyzeMeasure.
El valor de gris medio es la suma de los valores de gris de todos los píxeles de la selección, dividido por el número de píxeles (es decir, la intensidad media de fluorescencia por área de la célula). - Para cada imagen dibujar un círculo en el canal de YFP en un área que no contiene una célula. Tomar una medida para esta zona como fondo. Resta el fondo de cada imagen.
- La media del valor medio gris menos los antecedentes de todas las células de imágenes en una muestra. Esta será la intensidad media de fluorescencia para una población de células. Le sugerimos unos 60 células en tres experimentos se cuantificó.
F. Representante Resultados:
Nuestro laboratorio se centra en el andamio de proteínas con varios dominios, Intersectin (ITSN) que interactúa con numerosas proteínas que regulan múltiples procesos bioquímicos y de señalización 10,11,12,13,14. ITSN contiene dos Eps15 homología (EH) dominios, una región en espiral-espiral y cinco homología Src 3 (SH3) dominios. La isoforma más larga de ITSN también contiene homología Dbl (DH) y pleckstrin homología (PH) dominios que actúan en conjunto como un factor de intercambio de nucleótidos guanina de Cdc42 15. Esta estructura modular promueve la proteína: proteína y hace ITSN un candidato ideal para experimentos BiFC. La localización subcelular de ITSN puede alterar sus socios vinculante y por lo tanto alterar las vías reguladas por ITSN (unpublished de datos). Recientemente, nuestro laboratorio ha demostrado que ITSN regula la supervivencia neuronal a través de la regulación de una novela de PI3K de clase II, PI3K-C2β 11. El amino-terminal pro-rico dominio de la PI3K-C2β contiene dos sitios de unión para SH3 ITSN sus dominios. El uso de co-inmunoprecipitación con ITSN y PI3K-C2β mutantes de truncamiento se demostró que SH3A ITSN y dominios SH3C interactuar con la región amino-terminal de la PI3K-C2β. A continuación, se utiliza para visualizar BiFC la localización subcelular de este complejo. ITSN se fusionó con la amino-terminal de Venus (PFLAG-VN173) y construye PI3K-C2β fusionado con el carboxilo terminal de Venus (PHA-VC155). Como otro control negativo, un péptido no específicos se fusionó con PHA-VC155. VN-ITSN y VC-PI3K C2β formado un complejo BiFC con una distribución de puntuar (Figura 2A, paneles superiores). Las mutaciones en el dominio de Pro-ricos de la PI3K-C2β que interrumpir la co-precipitación de ITSN y PI3K C2β-disminución de la señal BiFC (Figura 2A, paneles inferiores) 11. Esta diferencia en la señal de BiFC entre ITSN y las dos proteínas C2β PI3K-no se debió a diferencias en la expresión de la proteína (Figura 2B).
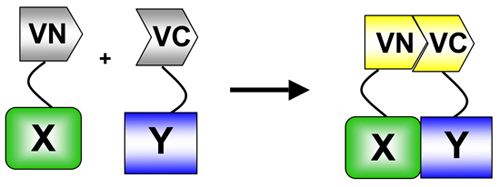
. Figura 1 En BiFC, un fluoróforo (en este caso Venus) se divide en aminoácidos (VN) - y carboxi (VC) termina-terminal. Estos extremos se fusionan dos proteínas de interés. Cuando las dos proteínas interactúan, los fragmentos de VN y VC volver a asociar resultantes de una reconstitución del fluoróforo y la fluorescencia en los sitios de interacción. BiFC es un ejemplo específico de la proteína de ensayo de complementación de fragmentos (PCA) que se utiliza para medir la proteína: las interacciones proteína 5.
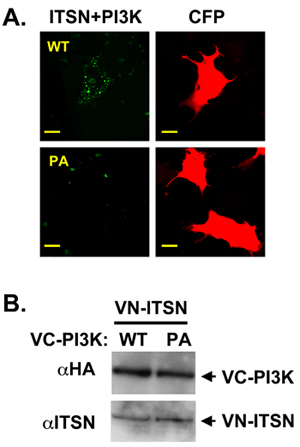
Figura 2. ITSN y PI3K-C2β formar un complejo BiFC. A. VN-etiquetados ITSN fue co-transfectadas con el VC-etiquetados PI3K-C3β WT o un dominio rico en prolina mutante (PI3K-C2β-PA). ITSN y forma PESO PI3K-C2β un complejo (verde). PPC (rojo) se utilizó como control de transfección B. se llevó a cabo un Western blot para demostrar la expresión de la igualdad de las construcciones. El VC-etiquetados construcciones se HA etiquetados.
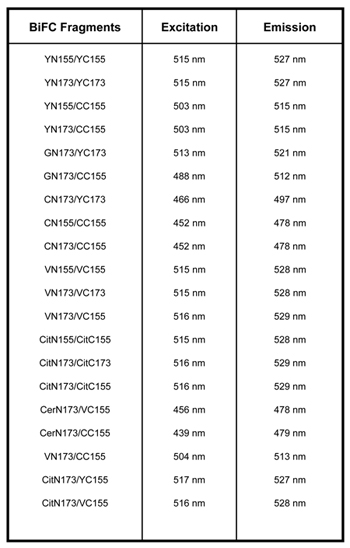
Tabla 1. Existen varios vectores que son compatibles con BiFC. YN155: 1-155aa de YFP, YC155: 155 238aa de YFP, YN173: 1-172aa de YFP, YC173, 173-238aa de YFP, VN155: 1-154aa de Venus; VC155: 155-238 de Venus; VN173: 1-172aa de Venus; VC173: 173-238aa de Venus; CN155 1-154aa de la PPC; CC155 155 238aa de la PPC, GN173: 1-172aa de la GFP, CitN155: 1-155aa de citrino, CitC155: 155 238aa de citrino, CitN173: 1-172aa de citrino, CitC173: 173-238aa de citrino, CerN173: 1-172aa de Cerulean 2,3,9.
Discusión
BiFC es un excelente método para la visualización de proteínas: las interacciones de proteínas en células enteras y la determinación de la localización subcelular de estos complejos. Las ventajas de BiFC es que sólo las proteínas que interactúan son fluorescentes, las interacciones transitorias se han estabilizado, y post-tratamiento de los datos de imagen es mínima. Dos desventajas de este método son el tiempo de maduración para el fluoróforo y la irreversibilidad de la compleja fluoróforo. En algunas ap...
Divulgaciones
Agradecimientos
El ITSN, PI3K C2β-, y el control de vectores utilizados en este protocolo están disponibles a partir de los autores que lo soliciten para fines no comerciales solamente. Los autores desean agradecer al Dr. Chang-Deng, Hu por la amabilidad de prestar asesoramiento y los reactivos utilizados en el establecimiento del protocolo BiFC en el laboratorio O'Bryan. KAW fue apoyado por fondos de la Fundación Jerome Lejeune. Trabajo en el laboratorio O'Bryan es apoyado por subvenciones del NIH (HL090651), DOD (PR080428), la Fundación San Baldrick, y la Fundación Jerome Lejeune.
Materiales
| Name | Company | Catalog Number | Comments |
| DMEM | Cellgro | 10-013 | |
| Fetal bovine serum | Cellgro | 35-011-CV | |
| Glass Bottom Microwell dishes | Matek | P35G-1.5-14C | |
| 6-well dishes | Falcon BD | 35-3846 | |
| Lipofectamine | Invitrogen | 18324020 | |
| PBS | Cellgro | 21-031-CV | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Confocal Microscope | Carl Zeiss, Inc. | LSM510 META |
Referencias
- Kerppola, T. K. Visualization of molecular interactions by fluorescence complementation. Nat Rev Mol Cell Biol. 7, 449-456 (2006).
- Shyu, Y. J., Liu, H., Deng, X., Hu, C. D. Identification of new fluorescent protein fragments for bimolecular fluorescence complementation analysis under physiological conditions. Biotechniques. 40, 61-66 (2006).
- Hu, C. D., Chinenov, Y., Kerppola, T. K. Visualization of interactions among bZIP and Rel family proteins in living cells using bimolecular fluorescence complementation. Mol Cell. 9, 789-798 (2002).
- Tsien, R. Y. The green fluorescent protein. Annu Rev Biochem. 67, 509-544 (1998).
- Michnick, S. W., Remy, I., Campbell-Valois, F. X., Vallee-Belisle, A., Pelletier, J. N. Detection of protein-protein interactions by protein fragment complementation strategies. Methods Enzymol. S328, 208-230 (2000).
- Michnick, S. W., MacDonald, M. L., Westwick, J. K. Chemical genetic strategies to delineate MAP kinase signaling pathways using protein-fragment complementation assays (PCA). Methods. 40, 287-293 (2006).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends Biochem Sci. 32, 407-414 (2007).
- Ciruela, F. Fluorescence-based methods in the study of protein-protein interactions in living cells. Curr Opin Biotechnol. 19, 338-343 (2008).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21, 539-545 (2003).
- Martin, N. P. Intersectin regulates epidermal growth factor receptor endocytosis, ubiquitylation, and signaling. Mol Pharmacol. 70, 1643-1653 (2006).
- Das, M. Regulation of neuron survival through an intersectin-phosphoinositide 3'-kinase C2beta-AKT pathway. Mol Cell Biol. 27, 7906-7917 (2007).
- Mohney, R. P. Intersectin activates Ras but stimulates transcription through an independent pathway involving. JNK. J Biol Chem. 278, 47038-47045 (2003).
- Tong, X. K., Hussain, N. K., Adams, A. G., O'Bryan, J. P., McPherson, P. S. Intersectin can regulate the Ras/MAP kinase pathway independent of its role in endocytosis. J Biol Chem. 275, 29894-29899 (2000).
- Adams, A., Thorn, J. M., Yamabhai, M., Kay, B. K., O'Bryan, J. P. Intersectin an adaptor protein involved in clathrin-mediated endocytosis, activates mitogenic signaling pathways. J Biol Chem. 275, 27414-27420 (2000).
- O'Bryan, J. P., Mohney, R. P., Oldham, C. E. Mitogenesis and endocytosis: What's at the INTERSECTIoN?. Oncogene. 20, 6300-6308 (2001).
- Shyu, Y. J., Suarez, C. D., Hu, C. D. Visualization of AP-1 NF-kappaB ternary complexes in living cells by using a BiFC-based FRET. Proc Natl Acad Sci U S A. 105, 151-156 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados