このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
二分子蛍光相補
要約
タンパク質の細胞内局在は、細胞シグナル伝達の時空間制御を決定する上で重要です。ここで、我々は、細胞内のタンパク質の空間的な相互作用を監視するための簡単な方法として、二分子蛍光相補(BiFC)を記述する。
要約
シグナル伝達複合体の細胞内分布を定義すると、その複合体からの出力を理解する上で不可欠です。 このような免疫沈降などの従来の方法は、複合体の空間的局在に関する情報を提供していません。対照的に、BiFCはタンパク質複合体の相互作用と細胞内局在を監視します。この方法では、fluororescentタンパク質は、関心のある2つのタンパク質に融合されているアミノおよびカルボキシ末端に非蛍光フラグメントに分割されます。蛍光体の再構成におけるタンパク質の結果の相互作用(図1)1,2。 BiFCの制限は、断片化された蛍光体が再構成されると、複雑な3不可逆であるということです。この制限は、一時的または弱い相互作用を検出するのに有利であるが、複雑なダイナミクスの動態解析を排除する。追加の注意点は、再構成されたflourophoreが再びリアルタイムの相互作用4の観察を排除、成熟し、蛍光を発するために30分必要があるということです。タンパク質相互作用5,6:BiFCはそのような緑色蛍光タンパク質変異などのレポータータンパク質(BiFC)、ジヒドロ葉酸還元酵素、β-ラクタマーゼ、および蛋白質を測定するルシフェラーゼを採用した蛋白質フラグメントの相補アッセイ(PCA)の具体例です。蛋白質研究するために代替方法:細胞内のタンパク質の相互作用は、蛍光共局在し、フェルスター共鳴エネルギー移動(FRET)7が含まれています。共局在のために、二つのタンパク質は、個別に直接蛍光体でまたは間接蛍光抗体のいずれかによってタグ付けされます。しかし、このアプローチは、それが困難な共局在のデータを解釈すること、非相互作用するタンパク質の高いバックグラウンドにつながります。さらに、共焦点顕微鏡の分解能の限界のため、2つのタンパク質は必ずしも相互に作用することなく、共局在表示されることがあります。興味のある2つのタンパク質が相互作用するときBiFCで、蛍光のみが観察される。タンパク質相互作用が、技術的に挑戦することができます:FRETはタンパク質を研究するための別の優れた方法です。 FRETの実験では、ドナーとアクセプターは、細胞内で同様の明るさと化学量論であることが必要です。さらに、一つはアクセプターチャネルとその逆にドナーの通過ブリードを考慮する必要があります。 FRETのとは異なり、BiFCは、画像データの少ないバックグラウンド蛍光、少し後処理を有し、高い過剰発現を必要とせず、弱または一時的な相互作用を検出することができます。生物発光共鳴エネルギー移動(BRET)はドナーがそれによって刺激的なアクセプター発光になるために基質を触媒する酵素(例えばルシフェラーゼ)であることを除いてFRETを同様の方法です。 BRETは、スルーブリードの技術的な問題や高いバックグラウンドの蛍光を欠いていることもありましたが、特定のコンパートメント8〜基板の局在の欠如に空間的な情報を提供する能力を欠いている。全体的に、BiFCは区分シグナリングへの洞察を得るためにタンパク質複合体の細胞内局在を可視化するための優れた方法です。
プロトコル
A. BiFCキャリブレーション
- 蛍光体を選択してください。BiFC融合パートナー(表1)としてうまく機能などYFPや金星など、複数の蛍光体が、、ある。 YFP BiFCフラグメントは蛍光体の形成2を容易にするために30℃でプレインキュベーションを必要としながら金星のアミノおよびカルボキシ末端の末端は、37℃での複合体を形成することが可能です。低い温度でこのインキュベーションは、いくつかの細胞プロセスを変更することがありますし、フラグメントを選択する際に考慮する必要があります。タンパク質のカルボキシ末端に融合させる金星のためのベクターは、Addgene(から入手できますhttp://www.addgene.org/pgvec1 、コントロールとして使用するための構造体が追加、などと一緒に、seepBiFC - VN173とpBiFC - VC155) pBiFC - bJunVN173とpBiFC - bFosVC155 2。アミノ末端金星のベクトル(pFLAG - VN173およびPHA - VC155)を含む追加のベクトルは、、以下のサイトから入手できます。 http://people.pnhs.purdue.edu/〜HU1 / 。
- 目的のタンパク質にタグを付ける。BiFC断片が候補タンパク質のアミノまたはカルボキシ末端の末端に融合されています。いくつかのタンパク質は、タンパク質の機能の破壊が原因でどちらかの端にタグ付けを可能にしない場合があります。例えば、GTPアーゼのRasスーパーファミリーの多くのメンバーは、その端にBiFCフラグメントの添付ファイルを排除ので、カルボキシ末端に脂質を変更されます。従って、BiFCフラグメントの添付ファイルは目的のタンパク質の機能に影響を与える可能性のある方法のいくつかのアイデアを持っていることが重要です。それは蛋白質がどう影響するかタギング不明な点などがその機能を複数の組み合わせをテストする必要があります。 BiFC断片に加えて、ペプチドリンカーは、断片化された蛍光団との候補タンパク質の間に柔軟性を高めるために含めることができます。十分な柔軟性を提供することができる短いアミノ酸のストレッチをエンコードBiFCベクトルのマルチクローニングサイト(MCS)一方で、RSIAT、KQKVMNH、およびRPACKIPNDLKQKVMNHリンカーはBiFC実験3,9で正常に使用されている
- トランスフェクション条件を決定します。複数の変異をテストする前に、いくつかの制御実験を行う必要があります。試行すべき最初の二つBiFCの組み合わせは、対話と相互作用しない1つの野生型と変異することが知られている2つの野生型タンパク質である。これらの2つの組み合わせを使用して、DNAとトランスフェクション時間の異なる量は、候補タンパク質のBiFC信号を検出するための最適条件を決定するためにテストする必要があります。我々は、6ウェルディッシュのほか、単一のために各BiFC構造の0.25ug、0.5ug、1.0ugをテスト示唆している。信号の開発のための最適な時間を決定するために蛍光顕微鏡法によるトランスフェクションモニター細胞の翌日。 pBiFC - bJunVN173とpBiFC - bFosVC155 2の構造はBiFCのための有用なポジティブコントロールであるとAddgene(上記参照)から入手できます。さらに、ウエスタンブロット分析は、構造の同じ表現を確認するために実行する必要があります。条件は、蛍光シグナルは、2つの野生型タンパク質の間に観察されているが、無信号にはほとんどが野生型と変異体タンパク質との間で観察されるように選択しなければなりません。最後に、これは非特異的な相互作用を防ぐために、可能な限り低くタンパク質の発現を維持するのが最善です。
- BiFCフラグメントの添加は、目的タンパク質の局在を変化させるかどうかを調べます。各BiFC構造はどちらHAまたはFLAGエピトープタグを含む。免疫染色では、HAまたはFLAGエピトープタグと同様にBiFCタグが目的タンパク質の局在に影響を与えるかどうかを判断するためにも内因性タンパク質(可能な場合)の両方の細胞をtransefected。
セルのB.播種とトランスフェクション
- COS細胞(1.3 × 5)1つのグラスの底Matekプレートおよびサンプルあたり6ウェルプレートの2ウェル上、それぞれのメッキが施されています。細胞は一晩37℃を解決することができます関心の候補タンパク質に関連性の高い代替細胞型を使用してもよい。
- トランスフェクション用のDNAを準備します。我々は通常、COSのトランスフェクションのためにリポフェクトアミン(Invitrogen)を利用する。しかし、他の試薬は、関心の細胞株に対してより適切かもしれない。トランスフェクション混合物を1つのグラスの底皿と6ウェルプレートの2ウェルの間で分割されるので、この区分を考慮してDNAの適当な量を使用してください。無血清(SF)DMEMの250uLでDNAを希釈する。トランスフェクションのコントロールとして合計BiFCのDNAの1 / 5量でCFPを追加。 BiFC定量では、CFPのために陽性の細胞のみがBiFC信号の存在について分析されます。したがって、代替のトランスフェクションのコントロールを、CFPのスペクトルは、表1のBiFCペアの一部と重なることに注意してください多分必要。 250uL SF DMEM(DNAの10uL Lipofectamine/1ug)にリポフェクタミンを希釈する。 DNAとリポフェクトアミンの希釈液を混ぜる。 20分室温でインキュベートする。
注:リポフェクタミン:DNA比は細胞の種類によって異なりますができます。興味の細胞株に適したトランスフェクション法を使用してください。 - 暖かいSF DMEMで2倍の細胞をすすいでください。各グラスの底板または同様の6ウェルディッシュにSF DMEMの2mLのを追加。
- のガラスボトムディッシュ、6ウェルディッシュの二つの井戸間で均等にそれぞれのトランスフェクション混合物を分割する。
- 5時間、37℃で細胞をインキュベートする。
- トランスフェクションのメディアを取り出して、(DMEM +10%FBS)の完全なメディアと交換してください。
- 37℃で一晩細胞をインキュベート℃をインキュベーション以下のトランスフェクションの長さは、目的のタンパク質の発現レベルによって異なります。このステップは経験的に決定する必要があるので、長時間のインキュベーションでは、非特異的な相互作用が発生する可能性があります。
イメージングのための細胞のC.の準備
- 細胞は、最初に陽性コントロールが蛍光であることを保証する落射蛍光顕微鏡で検査されています。そうでない場合、それはシグナルが観察されるまで37℃で細胞をさらに時間を許可する必要があるかもしれません。
- PBS(pH7.4)で3倍の細胞をすすいでください。ガラスボトムディッシュ中の細胞に2%のパラホルムアルデヒド(pH7.4)を加える。氷上で10分間細胞を固定する。 PBS(pH7.4)で3倍の細胞をすすいでください。 4℃店舗細胞° C 1mLのPBS(pH7.4)で覆われた。細胞は、イメージングのために固定する必要はありませんが、一度BiFCの断片をそのまま蛍光団を改革し、それはこのようにダイナミックな相互作用3の解析を防止する不可逆的です。また、未固定の細胞が信号を開発し続けることに留意してください。 6ウェルディッシュに細胞を溶解し、ウェスタンブロット解析のためにライセートを準備。溶解した細胞は、撮像された細胞の代表になるように、すべてのセルが同時に準備されていることが重要です。
D.イメージング細胞
- 蛍光強度は細胞ごとに計算されます。画像個々のセルにしてください。
- CFPは、トランスフェクションのコントロールとして含まれていたとだけCFP陽性細胞がBiFC信号の分析のために選択されています。我々は、セルがCFPをトランスフェクトされている場合、それはまたBiFC構築物でトランスフェクトされていることを前提にしてください。このアプローチは、BiFC信号を欠いている画像化された細胞は、目的のタンパク質ではなく、一つの欠如に起因またはそのセルの両方BiFC発現コンストラクトとの間の相互作用の欠如に起因する負であることを保証します。
- 我々は、細胞イメージングのためのツァイスLSM 510共焦点顕微鏡を使用してください。この顕微鏡を使用するときにはズーム、ピンホール、検出器のゲイン、アンプのオフセット、フレームサイズ、走査速度、スキャンの平均、およびレーザパワーの一貫性を保つことが重要です。あらゆるイメージングシステムを使用する場合には、蛍光がサンプル間で比較可能となるように設定を一定に保つことが重要です。また、蛍光を定量するとき、それは、ピクセルが飽和していないことが重要です。
蛍光を定量化するE.
- 蛍光は、任意の画像処理ソフトウェアを用いて定量することができます。私たちは、NIH(http://rsb.info.nih.gov/ij/)から自由に利用可能であるImageJを利用する。 ImageJのにイメージファイルを開きます。 AnalyzeSet測定に進みます。エリアのチェックボックスをオンにし、測定ボックス内のグレー値を平均。
- "フリーハンド選択"ツールを使用して、CFPのチャネルの全体のセルのエッジの周りにアウトラインを描きます。
- 場所にこのアウトラインを残し、YFPチャンネルにシフト。 AnalyzeMeasureに移動します。
平均グレー値は、ピクセルの数(すなわち、セルの面積あたりの平均蛍光強度)で割って選択範囲のすべてのピクセルのグレー値の合計です。 - イメージごとにセルが含まれていない地域においてYFPチャンネルで円を描画します。背景として、この領域の測定を行う。それぞれの画像から背景を引きます。
- 平均的な一つのサンプルで結像されるすべてのセルの背景マイナス平均グレー値。これは、細胞の集団の平均蛍光強度になります。我々は3つの実験上の約60の細胞を定量することが示唆している。
F.代表の結果:
私たちの研究室では、複数の生化学的およびシグナル伝達経路10,11,12,13,14を規制する多くのタンパク質と相互作用する複数のドメイン足場タンパク質、intersectin(ITSN)に焦点を当てています。 ITSNは2つのEps15相同性(EH)ドメイン、コイルドコイル状の地域、および5つのSrcホモロジー3(SH3)ドメインが含まれています。 ITSNの長いアイソフォームもDBLホモロジー(DH)およびCdc42 15グアニンヌクレオチド交換因子としてコンサートのプレクストリン相同(PH)ドメインをその行為が含まれています。このモジュラー構造は、タンパク質促進:タンパク質間相互作用をしBiFC実験のための理想的な候補をITSNです。 ITSNの細胞内局在は、その結合パートナーを変えるため、ITSN(unpublisによって規制経路を変更することができますHEDデータ)。最近、私たちの研究室では、ITSNはPI3K -C2β11、小説クラスII PI3Kの調節を介してニューロンの生存を調節することが実証されています。 PI3K -C2βのアミノ末端プロリッチドメインは、ITSNのSH3ドメインの2つの結合部位が含まれています。 ITSNとPI3K -C2β切断型変異体との共免疫沈降法を用いて、我々は、ITSNのSH3AとSH3Cドメインは、PI3K -C2βのアミノ末端領域と相互作用することを示した。次に、我々はこの複合体の細胞内局在を可視化するBiFC使用。 ITSNは金星(PHA - VC155)のカルボキシ末端に融合金星(pFLAG - VN173)とPI3K -C2β構造のアミノ末端に融合されました。別のネガティブコントロールとして、非特異的なペプチドは、PHA - VC155に融合した。 VN - ITSNとVC - PI3K -C2βは、句読点の分布(図2A、上部のパネル)でBiFC複合体を形成。 ITSNとPI3K -C2βの共沈を混乱させるPI3K -C2βのプロが豊富なドメインの変異はBiFC信号(図2A、下のパネル)11に減少。 ITSN二PI3K -C2βタンパク質間のBiFC信号の差は、蛋白質の発現の違い(図2B)によるものではなかった。
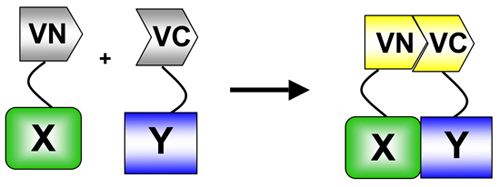
図1 BiFCでは、蛍光体(この場合は金星は)(VN)アミノ酸に分割されます-とカルボキシ(VC)端子が終了。これらの目的は、関心のある2つのタンパク質に融合されています。 2つのタンパク質が相互作用する場合、VNとVCの断片が再アソシエートの相互作用部位と蛍光団と蛍光の再構成をもたらす。タンパク質相互作用5:BiFCは蛋白質を測定するために使用される蛋白質の断片の相補アッセイ(PCA)の具体例です。
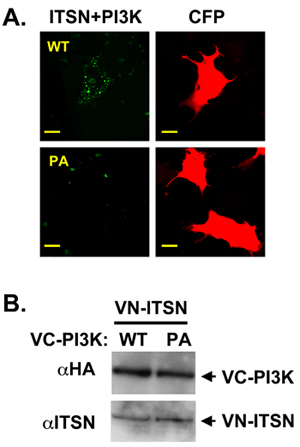
図2。ITSNとPI3K -C2βフォームBiFC複雑。 A. VN -タグ付きITSNは、VC -タグ付きPI3K -C3βWTまたはプロリンリッチドメイン変異体(PI3K -C2β- PA)と共同tranfectedした。 ITSNとWT PI3K -C2βフォーム複合体(緑)。 CFP(赤)ウエスタンブロット法は構造の等しい発現を実証するために実行されたトランスフェクションコントロールB.として使用されていました。 VC -タグ付きのコンストラクトは、HAタグ付けされています。
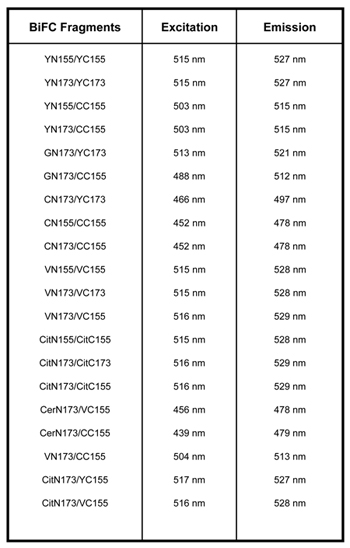
表1。BiFCと互換性のある複数のベクトルがあります。 YN155:YFPの1 - 155aa、YC155:YFPの155 - 238aa、YN173:YFPの1 - 172aa、YFPのYC173、173 - 238aa、VN155:金星の1 - 154aa、VC155:金星の155から238、VN173:金星の1 - 172aa、VC173:金星の173 - 238aa、CN155 CFPの1 - 154aa、CC155 CFPの155 - 238aa、GN173:GFPの1 - 172aa、CitN155:シトリンの1 - 155aa、CitC155:155 - 238aaシトリン、CitN173:シトリン、CitC173の1 - 172aa:シトリン、CerN173の173 - 238aa:セルリアン2,3,9の1 - 172aa。
Access restricted. Please log in or start a trial to view this content.
ディスカッション
細胞全体のタンパク質の相互作用およびこれらの複合体の細胞内局在を決定する:BiFCは、タンパク質を可視化するための優れた方法です。 BiFCの利点は唯一相互作用するタンパク質は、蛍光、過渡的な相互作用が安定しているか、および画像データの後処理が最小である。ということですこのメソッドの2つの欠点は、蛍光団と蛍光団複合体の不可逆性のための成熟の時間です。一部のアプリ...
Access restricted. Please log in or start a trial to view this content.
開示事項
謝辞
ITSN、このプロトコルで使用されるPI3K -C2β、およびコントロールベクターは、非商用目的のみのため、ご要望に応じて、著者から入手できます。著者は、親切にアドバイスとオブライアンの実験室でBiFCプロトコルの確立に使用される試薬を提供するために博士はチャントウ胡を認識したい。カーは、財団ジェロームルジューンからの資金によってサポートされていました。オブライアンの実験室での作業は、NIH(HL090651)、DOD(PR080428)、聖剣帯の財団、及び財団ジェロームルジューンからの補助金によってサポートされています。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| DMEM | Cellgro | 10-013 | |
| Fetal bovine serum | Cellgro | 35-011-CV | |
| Glass Bottom Microwell dishes | Matek | P35G-1.5-14C | |
| 6-well dishes | Falcon BD | 35-3846 | |
| Lipofectamine | Invitrogen | 18324020 | |
| PBS | Cellgro | 21-031-CV | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Confocal Microscope | Carl Zeiss, Inc. | LSM510 META |
参考文献
- Kerppola, T. K. Visualization of molecular interactions by fluorescence complementation. Nat Rev Mol Cell Biol. 7, 449-456 (2006).
- Shyu, Y. J., Liu, H., Deng, X., Hu, C. D. Identification of new fluorescent protein fragments for bimolecular fluorescence complementation analysis under physiological conditions. Biotechniques. 40, 61-66 (2006).
- Hu, C. D., Chinenov, Y., Kerppola, T. K. Visualization of interactions among bZIP and Rel family proteins in living cells using bimolecular fluorescence complementation. Mol Cell. 9, 789-798 (2002).
- Tsien, R. Y. The green fluorescent protein. Annu Rev Biochem. 67, 509-544 (1998).
- Michnick, S. W., Remy, I., Campbell-Valois, F. X., Vallee-Belisle, A., Pelletier, J. N. Detection of protein-protein interactions by protein fragment complementation strategies. Methods Enzymol. S328, 208-230 (2000).
- Michnick, S. W., MacDonald, M. L., Westwick, J. K. Chemical genetic strategies to delineate MAP kinase signaling pathways using protein-fragment complementation assays (PCA). Methods. 40, 287-293 (2006).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends Biochem Sci. 32, 407-414 (2007).
- Ciruela, F. Fluorescence-based methods in the study of protein-protein interactions in living cells. Curr Opin Biotechnol. 19, 338-343 (2008).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21, 539-545 (2003).
- Martin, N. P. Intersectin regulates epidermal growth factor receptor endocytosis, ubiquitylation, and signaling. Mol Pharmacol. 70, 1643-1653 (2006).
- Das, M. Regulation of neuron survival through an intersectin-phosphoinositide 3'-kinase C2beta-AKT pathway. Mol Cell Biol. 27, 7906-7917 (2007).
- Mohney, R. P. Intersectin activates Ras but stimulates transcription through an independent pathway involving. JNK. J Biol Chem. 278, 47038-47045 (2003).
- Tong, X. K., Hussain, N. K., Adams, A. G., O'Bryan, J. P., McPherson, P. S. Intersectin can regulate the Ras/MAP kinase pathway independent of its role in endocytosis. J Biol Chem. 275, 29894-29899 (2000).
- Adams, A., Thorn, J. M., Yamabhai, M., Kay, B. K., O'Bryan, J. P. Intersectin an adaptor protein involved in clathrin-mediated endocytosis, activates mitogenic signaling pathways. J Biol Chem. 275, 27414-27420 (2000).
- O'Bryan, J. P., Mohney, R. P., Oldham, C. E. Mitogenesis and endocytosis: What's at the INTERSECTIoN? Oncogene. 20, 6300-6308 (2001).
- Shyu, Y. J., Suarez, C. D., Hu, C. D. Visualization of AP-1 NF-kappaB ternary complexes in living cells by using a BiFC-based FRET. Proc Natl Acad Sci U S A. 105, 151-156 (2008).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved