Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Бимолекулярные флуоресценции комплементации
В этой статье
Резюме
Субклеточные локализации белков играет важную роль в определении пространственно-временной регуляции клеточной сигнализации. Здесь мы описываем бимолекулярной флуоресценции дополнения (BiFC) как прямой метод мониторинга пространственных взаимодействий белков в клетке.
Аннотация
Defining the subcellular distribution of signaling complexes is imperative to understanding the output from that complex. Conventional methods such as immunoprecipitation do not provide information on the spatial localization of complexes. In contrast, BiFC monitors the interaction and subcellular compartmentalization of protein complexes. In this method, a fluororescent protein is split into amino- and carboxy-terminal non-fluorescent fragments which are then fused to two proteins of interest. Interaction of the proteins results in reconstitution of the fluorophore (Figure 1)1,2. A limitation of BiFC is that once the fragmented fluorophore is reconstituted the complex is irreversible3. This limitation is advantageous in detecting transient or weak interactions, but precludes a kinetic analysis of complex dynamics. An additional caveat is that the reconstituted flourophore requires 30min to mature and fluoresce, again precluding the observation of real time interactions4. BiFC is a specific example of the protein fragment complementation assay (PCA) which employs reporter proteins such as green fluorescent protein variants (BiFC), dihydrofolate reductase, b-lactamase, and luciferase to measure protein:protein interactions5,6. Alternative methods to study protein:protein interactions in cells include fluorescence co-localization and Förster resonance energy transfer (FRET)7. For co-localization, two proteins are individually tagged either directly with a fluorophore or by indirect immunofluorescence. However, this approach leads to high background of non-interacting proteins making it difficult to interpret co-localization data. In addition, due to the limits of resolution of confocal microscopy, two proteins may appear co-localized without necessarily interacting. With BiFC, fluorescence is only observed when the two proteins of interest interact. FRET is another excellent method for studying protein:protein interactions, but can be technically challenging. FRET experiments require the donor and acceptor to be of similar brightness and stoichiometry in the cell. In addition, one must account for bleed through of the donor into the acceptor channel and vice versa. Unlike FRET, BiFC has little background fluorescence, little post processing of image data, does not require high overexpression, and can detect weak or transient interactions. Bioluminescence resonance energy transfer (BRET) is a method similar to FRET except the donor is an enzyme (e.g. luciferase) that catalyzes a substrate to become bioluminescent thereby exciting an acceptor. BRET lacks the technical problems of bleed through and high background fluorescence but lacks the ability to provide spatial information due to the lack of substrate localization to specific compartments8. Overall, BiFC is an excellent method for visualizing subcellular localization of protein complexes to gain insight into compartmentalized signaling.
протокол
А. BiFC калибровки
- Выберите флуорофор. Есть несколько флуорофоров, таких как YFP и Венеры, которые хорошо работают в качестве партнеров BiFC синтеза (табл. 1). Амино-и карбокси-концевые концы Венера способны образовывать комплекс при 37 ° С, а YFP BiFC фрагменты требуют предварительной инкубации при температуре 30 ° C в целях содействия образованию флуорофора 2. Это инкубации при низкой температуре может привести к изменению некоторых клеточных процессов и должны быть приняты во внимание при выборе фрагментов. Векторы для плавки Венеры к карбокси-конца белка можно получить Addgene ( http://www.addgene.org/pgvec1 ; seepBiFC-VN173 и pBiFC-VC155), а также дополнительные конструкции для использования в качестве элементов управления, например, pBiFC-bJunVN173 и pBiFC-bFosVC155 2. Дополнительные векторы, в том числе аминоконцевого Венеры векторов (PFLAG-VN173 и PHA-VC155), можно ознакомиться на следующем сайте: http://people.pnhs.purdue.edu/ ~ hu1 / .
- Тег белок. BiFC фрагменты, слитый с амино-и карбокси-концевые концы кандидата белков. Некоторые белки могут не позволить пометки на концах из-за нарушения функции белка. Например, многие члены суперсемейства РАН ГТФаз это липидные изменения в карбокси-конца что не позволяет крепление BiFC фрагментов в этом направлении. Таким образом, важно иметь некоторое представление о том, как прикрепление BiFC фрагментов может повлиять на функции белков, представляющих интерес. Если непонятно, как пометки белка будет влиять на его функцию несколько комбинаций должна быть проверена. В дополнение к BiFC фрагмент, пептидный линкер может быть включено для повышения гибкости между фрагментированным флуорофора и кандидат белков. В то время как множество сайтов клонирования (MCS) в BiFC векторов кодировать короткие участки кислот аминокислоты, которые могут обеспечить достаточную гибкость, RSIAT, KQKVMNH и RPACKIPNDLKQKVMNH линкеров были успешно использованы в экспериментах 3,9 BiFC
- Определите трансфекции условиях. Перед тестированием нескольких мутантов, несколько контрольных опытов должны быть выполнены. Первые два BiFC комбинаций, которые должны предстать перед судом два дикие белки типа, который, как известно, взаимодействуют и одного дикого типа и мутантов, которые не взаимодействуют. С помощью этих двух комбинаций, разное количество ДНК и трансфекции раз должна быть проверена, чтобы определить оптимальные условия для обнаружения BiFC сигнал для кандидата белков. Мы предлагаем тестирование 0.25ug, 0.5ug, 1.0ug каждого BiFC построить для одной скважины из 6 хорошо блюдо. На следующий день после трансфекции клеток монитор флуоресцентной микроскопии, чтобы определить оптимальное время для сигнала развития. PBiFC-bJunVN173 и pBiFC-bFosVC155 2 конструкции полезны положительного контроля для BiFC и доступны у Addgene (см. выше). Кроме того, Вестерн-блот анализа должны быть выполнены, чтобы подтвердить равные выражения конструкций. Условия должны быть выбраны так, что флуоресцентный сигнал наблюдается между двумя дикие белки типа, но немного, чтобы сигнал не наблюдается между дикого типа и мутантных белков. Наконец, лучше держать экспрессии белка как можно ниже, чтобы предотвратить любые неспецифические взаимодействия.
- Определите, если добавление BiFC фрагменты изменяет локализации белков, представляющих интерес. Каждая построить BiFC содержит либо HA или тега ФЛАГ эпитоп. Immunostain transefected клетки для HA или тега ФЛАГ эпитопа, а также эндогенный белок (если это возможно), чтобы определить, BiFC теги влияет локализация белков, представляющих интерес.
Б. Покрытие и трансфекции клеток
- COS клетки (1.3x10 5) высевают каждый на один со стеклянным дном Матек пластину и две скважины 6-луночный планшет на образец. Разрешить клетки урегулировать в течение ночи при температуре 37 ° C. Альтернативные типы клеток, которые являются более актуальными для кандидата белки проценты могут также быть использованы.
- Подготовка ДНК для трансфекции. Мы обычно используют Lipofectamine (Invitrogen) для трансфекции COS. Однако другие реагенты могут быть более подходящим для клеточной линии интересов. После трансфекции смесь будет разделена между одним стеклянную посуду дно и две скважины 6-луночного планшета, используйте соответствующее количество ДНК с учетом этого деления. Развести ДНК в 250uL из сыворотки (SF) DMEM. Добавить CFP на 1 / 5 общего количества BiFC ДНК как трансфекции контроля. Для количественного BiFC, только клетки, которые являются положительными для CFP будут проанализированы на предмет наличия BiFC сигнала. Обратите внимание, что спектры СФП будет пересекаться с некоторыми из BiFC пар в таблице 1, поэтому альтернативные контроля трансфекцииможет быть необходимо. Развести Lipofectamine в 250uL SF DMEM (10uL Lipofectamine/1ug ДНК). Смешайте ДНК и Lipofectamine разведений. Инкубируйте при комнатной температуре в течение 20 минут.
Примечание: Lipofectamine: ДНК соотношение может меняться в зависимости от типа клеток. Использование соответствующего метода трансфекции для клеточной линии интересов. - Промойте клетки 2x теплой SF DMEM. Добавить 2 мл SF DMEM на каждой пластине со стеклянным дном или хорошо из 6-и блюдо.
- Сплит каждой трансфекции смесь равномерно между одним стеклянную посуду дно и две скважины из 6-и блюдо.
- Инкубируйте клетки при 37 ° С в течение 5 часов.
- Удалить трансфекции материал и замените с полной среде (DMEM +10% FBS).
- Инкубируйте клетки ночи при 37 ° C. Длина инкубации трансфекции следующий будет варьироваться в зависимости от уровня экспрессии белков, представляющих интерес. Длительный инкубационный может привести к неспецифическим взаимодействием так что этот шаг необходимо будет определить эмпирически.
С. Подготовка Клетки для работы с изображениями
- Клетки изначально рассматривается в соответствии с epifluorescent микроскопом, чтобы обеспечить положительный управления флуоресцентные. Если нет, то это может быть необходимо, чтобы клетки дополнительное время при температуре 37 ° С, пока сигнал не наблюдается.
- Промойте клетки 3x с PBS (рН 7,4). Для клеток в блюдо стеклянным дном добавить 2% параформальдегида (рН 7,4). Fix клетки в течение 10 минут на льду. Промойте клетки 3x с PBS (рН 7,4). Магазин клетки при 4 ° С покрытой 1 мл PBS (рН 7,4). Клетки не должны быть установлены для работы с изображениями, но как только BiFC фрагменты реформы нетронутыми флуорофора это таким образом предотвращая необратимые анализ динамических взаимодействий 3. Кроме того, имейте в виду, что нефиксированные клетки будут продолжать развиваться сигнала. Lyse клеток в 6-а блюдо и подготовить лизатов для анализа блот западной. Важно, что все клетки готовятся в то же время так, чтобы лизировали клетки представитель отображаемого клеток.
Д. изображений Клетки
- Интенсивности флуоресценции будет рассчитана на ячейку. Убедитесь, что изображение отдельных ячеек.
- CFP был включен в качестве tranfection контроль и только CFP положительных клеток отбираются для анализа BiFC сигналов. Мы делаем предположение, что если ячейка трансфицированных CFP, это также трансфицированных BiFC конструкций. Такой подход гарантирует, что отображаемого клеток, отсутствие BiFC сигнала являются отрицательными из-за отсутствия взаимодействия между белками интерес, и не из-за отсутствия одного или обоих BiFC конструкций выражение в этой ячейке.
- Мы используем Zeiss LSM 510 конфокальной микроскопии для сотовых изображений. При использовании этого микроскопа важно сохранить зум, отверстие, детектор усиления, усилитель смещение, размер кадра, скорость сканирования, сканирования среднего, а мощность лазера последовательным. При использовании любых изображений системы важно сохранить настройки постоянными, так что флуоресценцию сопоставима между образцами. Кроме того, при количественной флуоресценции важно, что пиксели не насыщен.
Е. Количественная флуоресценции
- Флуоресценции может быть определена количественно с помощью любого обработки изображений. Мы используем ImageJ которых находится в свободном доступе из NIH (http://rsb.info.nih.gov/ij/). Открытые файлы изображений в ImageJ. К AnalyzeSet измерений. Установите флажки для района и среднего серого значение в поле измерения.
- Используя инструмент "свободного выбора руки», нарисуйте контур вокруг краю всей клетки в канале СФП.
- Оставив этот контур в месте, переход на канале YFP. К AnalyzeMeasure.
Средние Серые Значение суммы серый значения всех пикселей в выборе делится на число пикселей (т.е. средняя интенсивность флуоресценции в области клетки). - Для каждого изображения нарисуйте круг в канале YFP в области, которая не содержит клетки. Возьмите измерений в этой области в качестве фона. Вычтите фон от каждого изображения.
- Среднее Среднее значение Серый минус фон для всех ячеек отображается в одном образце. Это будет средняя интенсивность флуоресценции для популяции клеток. Мы хотели бы предложить около 60 клеток в течение трех экспериментов иметь количественное выражение.
Ф. представитель Результаты:
Наши лаборатории фокусируется на нескольких доменов леса белка, intersectin (ITSN), которая взаимодействует со многими белками регулировать нескольких биохимических и сигнальных путей 10,11,12,13,14. ITSN содержит два Eps15 гомологии (EH) доменов, гибких спиральный региона, а также пять Src гомологии 3 (SH3) доменами. Больше изоформы ITSN также содержит Dbl гомологии (DH) и pleckstrin гомологии (PH) доменов, действовать согласованно, как гуанин нуклеотидных фактор обмен на Cdc42 15. Эта модульная структура способствует белок: белковых взаимодействий и делает ITSN идеальным кандидатом для BiFC экспериментов. Субклеточные локализации ITSN может изменить его связывания партнерами и, следовательно, изменяют пути регулируются ITSN (unpublisHed данных). В последнее время в нашей лаборатории показали, что ITSN регулирует нейронов выживания путем регулирования нового класса II PI3K, PI3K-C2β 11. Аминоконцевого Pro богатой областью PI3K-C2β состоит из двух сайтов связывания SH3 домена ITSN в. Использование совместно с иммунопреципитации ITSN и PI3K-C2β мутантов усечения мы показали, что SH3A ITSN и SH3C области взаимодействуют с амино-концевой области PI3K-C2β. Далее, мы использовали BiFC визуализировать субклеточные локализации этого комплекса. ITSN был слит с амино-конца Венеры (PFLAG-VN173) и PI3K-C2β конструкций, слитый с карбокси-конца Венеры (PHA-VC155). В качестве другого негативного контроля, неспецифические пептид был слит с PHA-VC155. VN-ITSN и VC-PI3K-C2β формируется BiFC комплекс с перемежать распределения (рис. 2А, верхней панели). Мутации в Pro-богатые области PI3K-C2β, которые нарушают соосаждения ITSN и PI3K-C2β снизилась BiFC сигнала (рис. 2А, нижние панели) 11. Эта разница в BiFC сигнала между ITSN и два PI3K-C2β белков не было связано с различиями в экспрессии белка (рис. 2В).
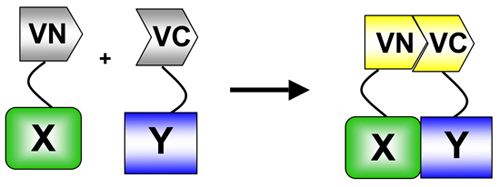
. Рисунок 1 В BiFC, флуорофор (в данном случае Венеры) расщепляется на аминокислоты (VN) - и карбокси (VC)-терминал концов. Эти концы сливаются в двух белков, представляющих интерес. Когда два белка взаимодействуют, В. Н. и VC фрагменты повторно связать в результате чего восстановление флуорофора и флуоресценции в местах взаимодействия. BiFC является конкретным примером дополнения белка фрагмент анализа (РСА), используемые для измерения белка: белковых взаимодействий 5.
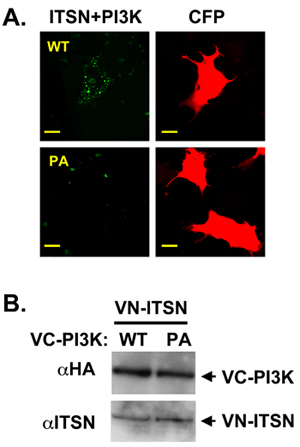
Рисунок 2. ITSN и PI3K-C2β форме BiFC комплекса. А. Н. с метками ITSN был одним из tranfected с VC-меткой PI3K-C3β WT или пролин-богатого домена мутант (PI3K-C2β-PA). ITSN и WT PI3K-C2β форме комплекса (зеленый). CFP (красный) был использован в качестве контроля трансфекции Б. пятно Западной проводилось, чтобы продемонстрировать равные выражения конструкций. VC-меткой конструкции HA помечены.
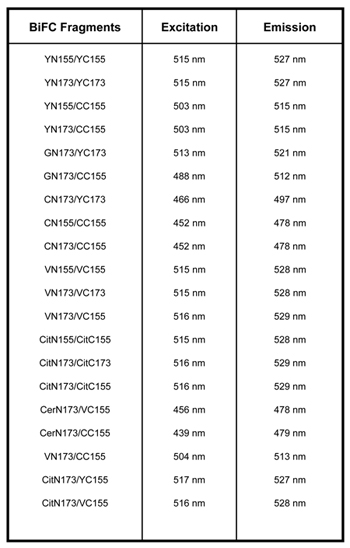
Таблица 1. Есть несколько векторов, которые совместимы с BiFC. YN155: 1-155aa из YFP; YC155: 155-238aa из YFP; YN173: 1-172aa из YFP; YC173, 173-238aa из YFP; VN155: 1-154aa Венеры; VC155: 155-238 Венеры; VN173: 1-172aa Венеры; VC173: 173-238aa Венеры; CN155 1-154aa из CFP; CC155 155-238aa из CFP, GN173: 1-172aa из GFP, CitN155: 1-155aa из цитрин, CitC155: 155-238aa из цитрин, CitN173: 1-172aa из цитрин, CitC173: 173-238aa из цитрин, CerN173: 1-172aa из Cerulean 2,3,9.
Обсуждение
BiFC является превосходным методом для визуализации белка: белковых взаимодействий в целых клеток и субклеточных определения локализации этих комплексов. Преимущества BiFC в том, что только взаимодействующие флуоресцентные белки, преходящие взаимодействия стабилизировалась, и пост-обр...
Раскрытие информации
Благодарности
ITSN, PI3K-C2β и управляющие векторы, используемые в настоящем протоколе можно получить у авторов по запросу, в некоммерческих целях. Авторы хотели бы выразить признательность д-р Чан-Дэн Ху за любезное предоставление консультаций и реагенты, используемые в создании BiFC протокол в лаборатории О'Брайен. KAW было поддержано финансирования из Фонда Джерома Лежен. Работа в О'Брайен лаборатории поддержана грантами NIH (HL090651), DOD (PR080428), Фонд святого Baldrick, и Фонд Иероним Лежен.
Материалы
| Name | Company | Catalog Number | Comments |
| DMEM | Cellgro | 10-013 | |
| Fetal bovine serum | Cellgro | 35-011-CV | |
| Glass Bottom Microwell dishes | Matek | P35G-1.5-14C | |
| 6-well dishes | Falcon BD | 35-3846 | |
| Lipofectamine | Invitrogen | 18324020 | |
| PBS | Cellgro | 21-031-CV | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Confocal Microscope | Carl Zeiss, Inc. | LSM510 META |
Ссылки
- Kerppola, T. K. Visualization of molecular interactions by fluorescence complementation. Nat Rev Mol Cell Biol. 7, 449-456 (2006).
- Shyu, Y. J., Liu, H., Deng, X., Hu, C. D. Identification of new fluorescent protein fragments for bimolecular fluorescence complementation analysis under physiological conditions. Biotechniques. 40, 61-66 (2006).
- Hu, C. D., Chinenov, Y., Kerppola, T. K. Visualization of interactions among bZIP and Rel family proteins in living cells using bimolecular fluorescence complementation. Mol Cell. 9, 789-798 (2002).
- Tsien, R. Y. The green fluorescent protein. Annu Rev Biochem. 67, 509-544 (1998).
- Michnick, S. W., Remy, I., Campbell-Valois, F. X., Vallee-Belisle, A., Pelletier, J. N. Detection of protein-protein interactions by protein fragment complementation strategies. Methods Enzymol. S328, 208-230 (2000).
- Michnick, S. W., MacDonald, M. L., Westwick, J. K. Chemical genetic strategies to delineate MAP kinase signaling pathways using protein-fragment complementation assays (PCA). Methods. 40, 287-293 (2006).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends Biochem Sci. 32, 407-414 (2007).
- Ciruela, F. Fluorescence-based methods in the study of protein-protein interactions in living cells. Curr Opin Biotechnol. 19, 338-343 (2008).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21, 539-545 (2003).
- Martin, N. P. Intersectin regulates epidermal growth factor receptor endocytosis, ubiquitylation, and signaling. Mol Pharmacol. 70, 1643-1653 (2006).
- Das, M. Regulation of neuron survival through an intersectin-phosphoinositide 3'-kinase C2beta-AKT pathway. Mol Cell Biol. 27, 7906-7917 (2007).
- Mohney, R. P. Intersectin activates Ras but stimulates transcription through an independent pathway involving. JNK. J Biol Chem. 278, 47038-47045 (2003).
- Tong, X. K., Hussain, N. K., Adams, A. G., O'Bryan, J. P., McPherson, P. S. Intersectin can regulate the Ras/MAP kinase pathway independent of its role in endocytosis. J Biol Chem. 275, 29894-29899 (2000).
- Adams, A., Thorn, J. M., Yamabhai, M., Kay, B. K., O'Bryan, J. P. Intersectin an adaptor protein involved in clathrin-mediated endocytosis, activates mitogenic signaling pathways. J Biol Chem. 275, 27414-27420 (2000).
- O'Bryan, J. P., Mohney, R. P., Oldham, C. E. Mitogenesis and endocytosis: What's at the INTERSECTIoN?. Oncogene. 20, 6300-6308 (2001).
- Shyu, Y. J., Suarez, C. D., Hu, C. D. Visualization of AP-1 NF-kappaB ternary complexes in living cells by using a BiFC-based FRET. Proc Natl Acad Sci U S A. 105, 151-156 (2008).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены