È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Bimolecolari complementazione fluorescenza
In questo articolo
Riepilogo
La localizzazione subcellulare delle proteine è importante nel determinare la regolazione spazio-temporale di segnalazione cellulare. Qui, descriviamo biomolecolare complementazione fluorescenza (BiFC) come un metodo semplice per il monitoraggio delle interazioni spaziali delle proteine all'interno della cellula.
Abstract
Definire la distribuzione subcellulare di complessi di segnalazione è fondamentale per comprendere l'output di quel complesso. Metodi convenzionali come immunoprecipitazione non forniscono informazioni sulla localizzazione spaziale dei complessi. Al contrario, BiFC controlla l'interazione e la compartimentazione subcellulare di complessi proteici. In questo metodo, una proteina fluororescent è suddivisa in amino-e carbossi-terminale non fluorescente frammenti che vengono poi fuse per due proteine di interesse. Interazione dei risultati proteine ricostituzione del fluoroforo (Figura 1) 1,2. Una limitazione di BiFC è che una volta che il fluoroforo frammentato è ricostituito il complesso è irreversibile 3. Questa limitazione è vantaggioso nel rilevare interazioni transitoria o deboli, ma esclude l'analisi cinetica di dinamiche complesse. Un ulteriore avvertimento è che il flourophore ricostituito richiede 30 minuti di maturare e di fluorescenza, di nuovo precludendo l'osservazione di interazioni in tempo reale 4. BiFC è un esempio specifico del test di complementazione proteica frammento (PCA), che impiega proteine giornalista come varianti proteina fluorescente verde (BiFC), la diidrofolato reduttasi, b-lattamasi, e luciferasi per misurare le proteine: interazioni proteina 5,6. Metodi alternativi per lo studio delle proteine: interazioni proteina nelle cellule includono fluorescenza co-localizzazione e Förster trasferimento di energia di risonanza (FRET) 7. Per la co-localizzazione, due proteine sono contrassegnati individualmente o direttamente con un fluoroforo o mediante immunofluorescenza indiretta. Tuttavia, questo approccio porta a di fondo di non interagenti proteine che lo rende difficile da interpretare co-localizzazione dei dati. Inoltre, a causa dei limiti di risoluzione della microscopia confocale, due proteine possono apparire co-localizzati senza necessariamente interagire. Con BiFC, fluorescenza è osservato solo quando le due proteine di interesse interagire. FRET è un altro ottimo metodo per lo studio delle proteine: interazioni delle proteine, ma possono essere tecnicamente difficile. FRET esperimenti richiedono il donatore e accettore di essere di luminosità simili e stechiometria nella cella. Inoltre, si deve tenere conto di sanguinare tramite del donatore nel canale accettore e viceversa. A differenza di FRET, BiFC ha poco fluorescenza di fondo, poco dopo l'elaborazione dei dati di immagine, non richiede l'iperespressione alto, e in grado di rilevare le interazioni deboli o transitorie. Risonanza trasferimento bioluminescenza energia (BRET) è un metodo simile a FRET tranne il donatore è un enzima (luciferasi ad esempio) che catalizza un substrato per diventare bioluminescenti così emozionante un accettore. BRET mancano i problemi tecnici di sanguinare attraverso e fluorescenza di fondo alto, ma manca la capacità di fornire informazioni spaziali a causa della mancanza di localizzazione del substrato ai compartimenti specifici 8. Nel complesso, BiFC è un metodo eccellente per la visualizzazione di localizzazione subcellulare di complessi proteici al fine di conoscere segnalazione compartimenti stagni.
Protocollo
A. BiFC calibrazione
- Scegli un fluoroforo. Fluorofori Ci sono molteplici, come ad esempio YFP e Venere, che funzionano bene come BiFC partner di fusione (Tabella 1). Estremità amino-e carbossi-terminale di Venere sono in grado di formare un complesso a 37 ° C, mentre i frammenti BiFC YFP richiedono una pre-incubazione a 30 ° C al fine di facilitare la formazione di fluoroforo 2. Questo incubazione a bassa temperatura può alterare alcuni processi cellulari e dovrebbero essere presi in considerazione quando si sceglie frammenti. Vettori per la fusione Venere al carbossi-terminale di proteine sono disponibili presso Addgene ( http://www.addgene.org/pgvec1 ; seepBiFC-VN173 e pBiFC-VC155) insieme a costrutti aggiuntivi per l'uso come controlli, ad esempio, pBiFC-bJunVN173 e pBiFC-bFosVC155 2. Ulteriori vettori, tra cui amino-terminale vettori di Venere (pFLAG-VN173 e Pha-VC155), sono disponibili sul seguente sito: http://people.pnhs.purdue.edu/ hu1 ~ / .
- Etichetta la proteina di interesse. BiFC I frammenti sono fusi alle estremità amino-e carbossi-terminale delle proteine candidato. Alcune proteine non possono consentire tagging alle estremità a causa di distorsioni della funzione della proteina. Per esempio, molti membri della superfamiglia delle GTPasi Ras sono lipidi modificati al carbossi-terminale precludendo così l'attaccamento dei frammenti BiFC a tal fine. Quindi, è importante avere un'idea di come l'attaccamento dei frammenti BiFC possono influenzare la funzione delle proteine di interesse. Se non è chiaro come tagging una proteina influenza la sua funzione di molteplici combinazioni devono essere testati. Oltre al frammento BiFC, un linker peptide può essere incluso per aumentare la flessibilità tra il fluoroforo frammentato e le proteine candidato. Mentre i siti clonazione multipla (MCS) nei vettori BiFC codificare brevi tratti di aminoacidi che possono fornire una sufficiente flessibilità, la RSIAT, KQKVMNH, e linker RPACKIPNDLKQKVMNH sono stati utilizzati con successo in esperimenti BiFC 3,9
- Determinare le condizioni di trasfezione. Prima di testare mutanti multipli, un paio di esperimenti di controllo deve essere eseguito. Le prime due combinazioni BiFC che dovrebbero essere processati sono due proteine wild-type che sono noti per interagire e una wild-type e mutanti che non interagiscono. L'utilizzo di questi due combinazioni, diverse quantità di DNA e tempi di transfezione dovrebbero essere testati per determinare le condizioni ottimali per la rilevazione di un segnale BiFC per le proteine candidato. Vi consigliamo di test 0.25ug, 0.5ug, 1.0ug di ciascun costrutto BiFC per un singolo bene di un piatto ben 6. Il giorno dopo la trasfezione le cellule monitorare al microscopio a fluorescenza per determinare il tempo ottimale per lo sviluppo del segnale. Il pBiFC-bJunVN173 e pBiFC-bFosVC155 2 costrutti sono utili controlli positivi per BiFC e sono disponibili da Addgene (vedi sopra). Inoltre, l'analisi Western Blot deve essere eseguita per confermare l'espressione pari dei costrutti. Condizioni devono essere scelto in modo che un segnale di fluorescenza si osserva tra le due proteine wild-type, ma poco o nessun segnale si osserva tra il tipo selvaggio e le proteine mutanti. Infine, è meglio tenere espressione della proteina più basso possibile per evitare interazioni non specifiche.
- Determinare se l'aggiunta dei frammenti BiFC altera la localizzazione delle proteine di interesse. Ogni costruire BiFC contiene sia un epitopo HA o tag BANDIERA. Immunostain transefected cellule sia per l'epitopo HA o tag FLAG così come anche la proteina endogena (se possibile) per determinare se il tag BiFC riguarda la localizzazione delle proteine di interesse.
B. Placcatura e Transfection delle cellule
- Cellule COS (1.3x10 5) sono placcati ciascuno su una piastra inferiore di vetro Matek e due pozzi di un 6-pozzetti per campione. Consentono alle cellule di riposare una notte a 37 ° C. Tipi di cellule alternative più rilevanti per le proteine candidato di interesse possono anche essere utilizzati.
- Preparare DNA per trasfezione. Noi di solito utilizzare Lipofectamine (Invitrogen) per trasfezioni COS. Tuttavia, altri reagenti può essere più appropriato per la linea cellulare di interesse. Dal momento che la miscela di trasfezione sarà diviso tra un piatto fondo di vetro e due pozzi di un 6-pozzetti, utilizzare la giusta quantità di DNA per tenere conto di questa divisione. Diluire DNA in 250uL di siero libero (SF) DMEM. Aggiungi PCP a 1 / 5 del numero totale di DNA BiFC come controllo trasfezione. Per la quantificazione BiFC, solo le cellule che sono positivi per PCP saranno analizzati per la presenza di un segnale BiFC. Da notare che gli spettri PCP sovrapposizione con alcune delle coppie BiFC nella tabella 1, quindi un controllo alternativo trasfezioneforse necessario. Diluire Lipofectamine in 250uL SF DMEM (10uL Lipofectamine/1ug del DNA). Mescolare il DNA e le diluizioni Lipofectamine. Incubare a temperatura ambiente per 20 min.
Nota: Il Lipofectamine: rapporto di DNA può variare a seconda del tipo di cellula. Utilizzare il metodo appropriato per la trasfezione della linea cellulare di interesse. - Lavare le cellule 2x con caldi SF DMEM. Aggiungere 2 ml di SF DMEM per ogni piatto fondo di vetro o anche di un 6-ben piatto.
- Dividere ogni miscela di trasfezione in modo uniforme tra un piatto fondo di vetro e due pozzi di un 6-ben piatto.
- Incubare le cellule a 37 ° C per 5 ore.
- Rimuovere i supporti di trasfezione e sostituirli con i media completo (DMEM +10% FBS).
- Cellule incubare una notte a 37 ° C. La lunghezza della transfezione fase di incubazione varia a seconda dei livelli di espressione delle proteine di interesse. Incubazione prolungata può risultare non in modo specifico l'interazione questa fase dovranno essere determinati empiricamente.
C. La preparazione di cellule per l'imaging
- Le cellule sono inizialmente esaminati al microscopio epifluorescente per garantire che il controllo positivo è fluorescente. In caso contrario, può essere necessario per dare il tempo supplementare le cellule a 37 ° C fino a quando il segnale viene osservato.
- Lavare le cellule 3x con PBS (pH 7,4). Alle cellule nel piatto fondo di vetro aggiungere 2% paraformaldeide (pH 7,4). Fissare le cellule per 10 minuti sul ghiaccio. Lavare le cellule 3x con PBS (pH 7,4). Conservare le cellule a 4 ° C coperto con 1 ml di PBS (pH 7,4). Cellule non devono essere fissati per l'imaging, ma una volta che i frammenti BiFC riforma uno fluoroforo intatto è irreversibile analisi impedendo così di interazioni dinamiche 3. Inoltre, tenete a mente che le cellule non fissate continuerà a sviluppare segnale. Lisare le cellule del 6 ben piatto e preparare lisati per l'analisi Western Blot. E 'importante che tutte le cellule sono preparati allo stesso tempo in modo che le cellule lisate sono rappresentativi delle cellule immagine.
D. Imaging Cellule
- L'intensità della fluorescenza sarà calcolata per cella. Assicurati di cellule singole immagini.
- PCP è stato incluso come controllo tranfection e soltanto le cellule positive PCP vengono selezionati per l'analisi dei segnali BiFC. Facciamo l'ipotesi che se la cella è transfettate con PCP, è anche trasfettate con i costrutti BiFC. Questo approccio garantisce che le cellule immaginato che la mancanza di un segnale BiFC sono negativi a causa della mancanza di una interazione tra le proteine di interesse e non a causa di mancanza di uno o entrambi i costrutti di espressione BiFC in quella cella.
- Usiamo un Zeiss LSM 510 microscopio confocale per l'imaging cellulare. Quando si utilizza questo microscopio è importante mantenere lo zoom, foro stenopeico, il guadagno del rivelatore, amplificatore offset, dimensione del frame, velocità di scansione, media scansione e potenza del laser coerente. Quando si utilizza un qualsiasi sistema di imaging è importante mantenere le impostazioni costante in modo che la fluorescenza è comparabile tra i campioni. Inoltre, nel quantificare la fluorescenza è importante che i pixel non sono saturi.
E. Quantificare fluorescenza
- Fluorescenza può essere quantificato con qualsiasi software di imaging. Utilizziamo ImageJ, che è liberamente disponibile presso il NIH (http://rsb.info.nih.gov/ij/). Aprire file di immagine in ImageJ. Vai a misure AnalyzeSet. Selezionare le caselle per l'Area e il valore medio grigio nella finestra di misure.
- Utilizzando lo strumento 'selezione a mano libera', tracciare un contorno intorno al bordo della cella intera nel canale PCP.
- Lasciando questo schema in atto, spostamento verso il canale YFP. Vai a AnalyzeMeasure.
Il valore medio Gray è la somma dei valori di grigio di tutti i pixel della selezione diviso per il numero di pixel (cioè, l'intensità media di fluorescenza per area della cellula). - Per ogni immagine disegnare un cerchio nel canale YFP in una zona che non contiene una cella. Effettuare una misurazione per questa area come sfondo. Sottrarre lo sfondo di ogni immagine.
- Media il valore medio grigio meno lo sfondo per tutte le celle ripreso in un campione. Questo sarà l'intensità media di fluorescenza per una popolazione di cellule. Vi suggeriamo di circa 60 cellule in tre esperimenti devono essere quantificati.
F. Rappresentante dei risultati:
Il nostro laboratorio si concentra sul multi-dominio proteico ponteggi, intersectin (ITSN) che interagisce con numerose proteine di regolare molteplici vie biochimiche e segnalazione 10,11,12,13,14. ITSN contiene due Eps15 omologia (EH) i domini, una regione avvolto a spirale, e cinque omologia Src 3 (SH3) domini. L'isoforma più di ITSN contiene anche omologia Dbl (DH) e Pleckstrin omologia (PH) i domini che agiscono in concerto come un fattore di scambio del nucleotide guanina per Cdc42 15. Questa struttura modulare promuove la proteina: interazioni proteina e ITSN rende un candidato ideale per esperimenti di BiFC. La localizzazione subcellulare di ITSN possono alterare i suoi partner vincolante e pertanto alterare le vie regolato da ITSN (unpublished dati). Recentemente, il nostro laboratorio ha dimostrato che ITSN regola la sopravvivenza neuronale attraverso la regolamentazione di un romanzo di classe II PI3K, PI3K-C2β 11. L'amino-terminale pro-dominio ricco di PI3K-C2β contiene due siti di legame per ITSN di SH3 domini. Mediante co-immunoprecipitazione con ITSN e PI3K-C2β mutanti troncamento abbiamo dimostrato che è ITSN SH3A e domini SH3C interagire con la regione amino-terminale di PI3K-C2β. Successivamente, abbiamo usato BiFC per visualizzare la localizzazione subcellulare di questo complesso. ITSN era fuso con la amino-terminale di Venere (pFLAG-VN173) e PI3K-C2β costruisce fuso al carbossi-terminale di Venere (PHA-VC155). Come ulteriore controllo negativo, un non-specifico peptide è stata fusa a Pha-VC155. VN-ITSN e VC-PI3K-C2β formato un complesso BiFC con una distribuzione punteggiano (Figura 2A, pannelli superiori). Le mutazioni nel dominio Pro-ricco di PI3K-C2β che distruggono co-precipitazione di ITSN e PI3K-C2β diminuito il segnale BiFC (Figura 2A, pannelli inferiori) 11. Questa differenza di segnale tra BiFC ITSN e le due PI3K-C2β proteine non è dovuto a differenze di espressione della proteina (Figura 2B).
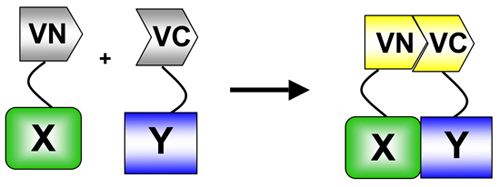
. Figura 1 In BiFC, un fluoroforo (in questo caso Venere) è diviso in aminoacidi (VN) - e carbossi (VC)-terminale finisce. Questi fini si fondono per due proteine di interesse. Quando le due proteine interagiscono, i frammenti VN e VC riassociare conseguente ricostituzione del fluoroforo e la fluorescenza nei siti di interazione. BiFC è un esempio specifico del test di complementazione proteica frammento (APC) utilizzato per misurare la proteina: interazioni delle proteine 5.
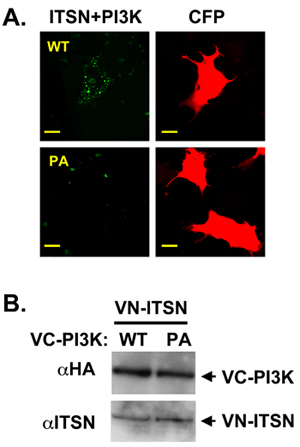
Figura 2. ITSN e PI3K-C2β formano un complesso BiFC. A. VN-tagged ITSN è stato co-tranfected con VC-tagged PI3K-C3β WT o prolina ricco di dominio mutante (PI3K-C2β-PA). ITSN e WT-PI3K C2β formano un complesso (verde). PCP (rosso) è stato utilizzato come controllo di transfezione B. è stato effettuato un Western Blot per dimostrare espressione pari dei costrutti. Il VC-tagged costrutti HA tag.
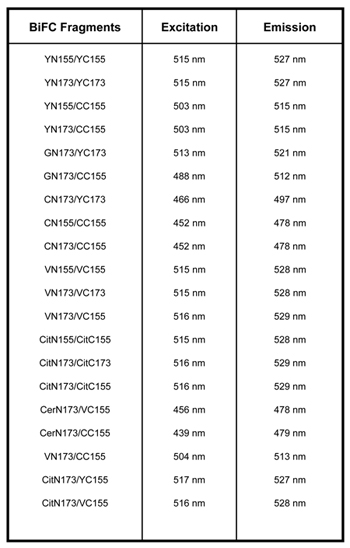
Tabella 1. Ci sono diversi vettori che sono compatibili con BiFC. YN155: 1-155aa di YFP; YC155: 155-238aa di YFP; YN173: 1-172aa di YFP; YC173, 173-238aa di YFP; VN155: 1-154aa di Venere; VC155: 155-238 di Venere; VN173: 1-172aa di Venere; VC173: 173-238aa di Venere; CN155 1-154aa della PCP; CC155 155-238aa della PCP, GN173: 1-172aa di GFP, CitN155: 1-155aa di Citrino, CitC155: 155-238aa di citrino, CitN173: 1-172aa di Citrino, CitC173: 173-238aa di Citrino, CerN173: 1-172aa di Cerulean 2,3,9.
Discussione
BiFC è un metodo eccellente per la visualizzazione di proteine: interazioni proteina nelle cellule intere e determinare la localizzazione subcellulare di questi complessi. I vantaggi di BiFC sono solo le proteine che interagiscono sono fluorescenti, le interazioni transitorie sono stabilizzate, e post-processing dei dati di imaging è minimo. Due gli svantaggi di questo metodo sono il tempo di maturazione per l'fluoroforo e l'irreversibilità del complesso fluoroforo. In alcune applicazioni di questa irre...
Divulgazioni
Riconoscimenti
Il ITSN, PI3K-C2β, e vettori di controllo utilizzati in questo protocollo sono disponibili presso gli autori, su richiesta, per scopi non commerciali solo. Gli autori desiderano ringraziare il Dr. Chang-Deng Hu per la gentile consulenza e dei reagenti utilizzati per stabilire il protocollo BiFC in laboratorio O'Bryan. KAW è stata sostenuta da un finanziamento della Fondazione Jerome Lejeune. Lavoro in laboratorio O'Bryan è sostenuto da finanziamenti della (HL090651) NIH, DOD (PR080428), la Fondazione San Baldrick, e la Fondazione Jerome Lejeune.
Materiali
| Name | Company | Catalog Number | Comments |
| DMEM | Cellgro | 10-013 | |
| Fetal bovine serum | Cellgro | 35-011-CV | |
| Glass Bottom Microwell dishes | Matek | P35G-1.5-14C | |
| 6-well dishes | Falcon BD | 35-3846 | |
| Lipofectamine | Invitrogen | 18324020 | |
| PBS | Cellgro | 21-031-CV | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Confocal Microscope | Carl Zeiss, Inc. | LSM510 META |
Riferimenti
- Kerppola, T. K. Visualization of molecular interactions by fluorescence complementation. Nat Rev Mol Cell Biol. 7, 449-456 (2006).
- Shyu, Y. J., Liu, H., Deng, X., Hu, C. D. Identification of new fluorescent protein fragments for bimolecular fluorescence complementation analysis under physiological conditions. Biotechniques. 40, 61-66 (2006).
- Hu, C. D., Chinenov, Y., Kerppola, T. K. Visualization of interactions among bZIP and Rel family proteins in living cells using bimolecular fluorescence complementation. Mol Cell. 9, 789-798 (2002).
- Tsien, R. Y. The green fluorescent protein. Annu Rev Biochem. 67, 509-544 (1998).
- Michnick, S. W., Remy, I., Campbell-Valois, F. X., Vallee-Belisle, A., Pelletier, J. N. Detection of protein-protein interactions by protein fragment complementation strategies. Methods Enzymol. S328, 208-230 (2000).
- Michnick, S. W., MacDonald, M. L., Westwick, J. K. Chemical genetic strategies to delineate MAP kinase signaling pathways using protein-fragment complementation assays (PCA). Methods. 40, 287-293 (2006).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends Biochem Sci. 32, 407-414 (2007).
- Ciruela, F. Fluorescence-based methods in the study of protein-protein interactions in living cells. Curr Opin Biotechnol. 19, 338-343 (2008).
- Hu, C. D., Kerppola, T. K. Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nat Biotechnol. 21, 539-545 (2003).
- Martin, N. P. Intersectin regulates epidermal growth factor receptor endocytosis, ubiquitylation, and signaling. Mol Pharmacol. 70, 1643-1653 (2006).
- Das, M. Regulation of neuron survival through an intersectin-phosphoinositide 3'-kinase C2beta-AKT pathway. Mol Cell Biol. 27, 7906-7917 (2007).
- Mohney, R. P. Intersectin activates Ras but stimulates transcription through an independent pathway involving. JNK. J Biol Chem. 278, 47038-47045 (2003).
- Tong, X. K., Hussain, N. K., Adams, A. G., O'Bryan, J. P., McPherson, P. S. Intersectin can regulate the Ras/MAP kinase pathway independent of its role in endocytosis. J Biol Chem. 275, 29894-29899 (2000).
- Adams, A., Thorn, J. M., Yamabhai, M., Kay, B. K., O'Bryan, J. P. Intersectin an adaptor protein involved in clathrin-mediated endocytosis, activates mitogenic signaling pathways. J Biol Chem. 275, 27414-27420 (2000).
- O'Bryan, J. P., Mohney, R. P., Oldham, C. E. Mitogenesis and endocytosis: What's at the INTERSECTIoN?. Oncogene. 20, 6300-6308 (2001).
- Shyu, Y. J., Suarez, C. D., Hu, C. D. Visualization of AP-1 NF-kappaB ternary complexes in living cells by using a BiFC-based FRET. Proc Natl Acad Sci U S A. 105, 151-156 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon