Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Mutagénesis y análisis de mutaciones genéticas en la secuencia del receptor GC-ricos KISS1 identificado en el hombre con Trastornos del aparato reproductor
En este artículo
Resumen
Las mutaciones en el receptor kisspeptina (KISS1R) se asocian con trastornos de la reproducción en los pacientes. A continuación se describe la forma de introducir las mutaciones de interés en la secuencia rica en GC de KISS1R, así como el uso de construcciones KISS1R para caracterizar la vía de degradación del receptor por inmunoprecipitación y Western Blot
Resumen
El receptor de kisspeptina (KISS1R) es una proteína G-receptor acoplado reconocido como el desencadenante de la pubertad y un regulador de la competencia reproductiva en la edad adulta 1,2,3. Inactivación de las mutaciones en KISS1R identificados en los pacientes han sido asociados con el hipogonadismo hipogonadotrópico iodiopathic (IHH) 1,2 y la pubertad precoz 4. Estudios funcionales de estos mutantes son cruciales para nuestra comprensión de los mecanismos que subyacen a la regulación de la reproducción de este receptor, así como los que dan forma los resultados de la enfermedad, que resultan de la señalización KISS1R anormal y la función. Sin embargo, la secuencia altamente rica en GC del gen KISS1R hace que sea bastante difícil de introducir mutaciones o amplificar el gen que codifica este receptor por PCR.
Aquí se describe un método para introducir mutaciones de interés en esta secuencia muy rica en GC que ha sido utilizado con éxito para generar más de una docena de mutantes KISS1R en nuestro laboratorio. Hemos optimizado las condiciones de PCR para facilitar la ampliación de una serie de mutantes KISS1R que incluyen sustituciones, eliminaciones o inserciones en la secuencia KISS1R. La adición de una solución de PCR potenciador, así como de un pequeño porcentaje de DMSO fueron especialmente útiles para mejorar la amplificación. Este procedimiento optimizado puede ser útil para otros GC-ricos y plantillas.
El vector de expresión que codifica la KISS1R se ha utilizado para caracterizar la señalización y la función de este receptor con el fin de entender cómo las mutaciones pueden cambiar KISS1R función y llevar a los fenotipos asociados reproductiva. En consecuencia, las posibles aplicaciones de los mutantes KISS1R generado por mutagénesis dirigida se puede ilustrar con muchos estudios 1,4,5,6,7,8. A modo de ejemplo, la mutación de ganancia de función en el KISS1R (Arg386Pro), que está asociada con la pubertad precoz, se ha demostrado que prolonga la capacidad de respuesta del receptor a la estimulación ligando 4, así como para alterar la velocidad de degradación de KISS1R 9 . Curiosamente, nuestros estudios indican que KISS1R se degrada por el proteasoma, a diferencia de la degradación lisosomal clásicos descritos para la mayoría de G receptores acoplados a proteínas 9. En el ejemplo que aquí se presenta, la degradación de la KISS1R se investiga en células de riñón embrionario humano (HEK-293) que expresan transitoriamente Myc-etiquetados KISS1R (MycKISS1R) y tratados con inhibidores del proteasoma o lisosomas. Lisados celulares se immunoprecipitated agarosa usando un conjugado de anticuerpos anti-myc seguido por análisis de Western blot. Detección y cuantificación de MycKISS1R en transferencias se realiza mediante el sistema Odyssey LI-COR infrarrojos. Este enfoque puede ser útil en el estudio de la degradación de otras proteínas de interés.
Protocolo
1. Mutagénesis dirigida de secuencia altamente rica en GC gen KISS1R
- Plantilla: secuencia completa de ADNc de la KISS1R humanos con Myc-tag fusionada a su extremo N-terminal. Esta secuencia se clonó en el vector de expresión pCS2 +, que es compatible con las líneas de células de mamíferos utilizan posteriormente para la transfección. Este vector de expresión se hace referencia en este documento como pCS2 KISS1R + Myc.
- Diseño de la cartilla: primers están diseñados para llevar a mutaciones deseadas, de acuerdo a las instrucciones del kit QuikChange mutagénesis dirigida (Stratagene). En resumen:
- Los dos primers (adelante y atrás) debe contener la mutación deseada y recocido a la misma secuencia en cadenas opuestas del plásmido (por ejemplo, hacia adelante y atrás primers son complementarias entre sí)
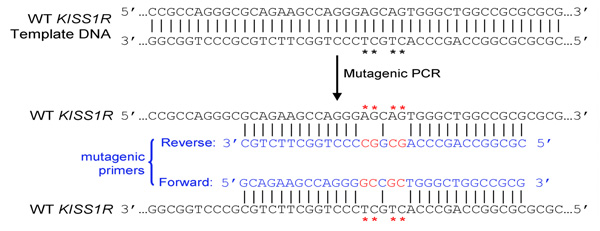
- Cebadores debe ser de 25 a 45 bases de largo y al final en una o más C o bases G
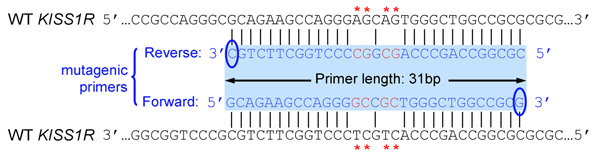

- Mutación introducida (s) deben estar en el medio de imprimación y flanqueado por ~ 10-15 bases de la secuencia correcta en ambos lados
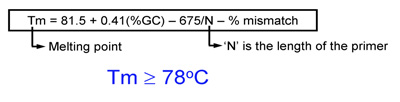
- Temperatura de fusión (Tm) de los cebadores debe ser igual o superior a 78 ° C. Utilice las siguientes fórmulas para calcular Tm:
- Cuando la introducción de mutaciones: Tm = 81.5 + 0.41 (% GC) - 675 / N - desajuste% (N es la longitud de imprimación en las bases)
- En la introducción de inserciones o deleciones: Tm = 81.5 + 0.41 (% GC) - 675 / N (N no incluye las bases que se insertan o se eliminan)
- Use cebos desalado (sin purificación adicional necesaria).
- Los dos primers (adelante y atrás) debe contener la mutación deseada y recocido a la misma secuencia en cadenas opuestas del plásmido (por ejemplo, hacia adelante y atrás primers son complementarias entre sí)
- Las mejores condiciones de amplificación incluyen la adición de PCRx reforzador solución (Invitrogen) y DMSO. Por lo menos dos concentraciones de DMSO, tales como el 4% y 8%, debe ser probado. Las condiciones de PCR se muestran en la Tabla I y en la Figura 1, y los resultados de un representante de la amplificación por PCR de pCS2 + MycKISS1R se muestra en la Figura 2.
- Eliminar el ADN de los padres en los productos de amplificación por la digestión de ADN metilado con DpnI de 1h 30min a 37 ° C: mezclar en un tubo de centrífuga 17.5μl de producto de PCR con 2μl de NEBuffer 10x 4 y 0.5μl de DpnI (10U, New England Biolabs).
- Transformar DpnI tratados con productos PCR: 2μl mezcla de DpnI tratados con ADN con 45μl de XL10-Gold Ultracompetent E. coli (Stratagene) en pre-enfriado tubo de 15 ml de cultivo de células. Añadir 2μl de β-mercaptoetanol y seguir las instrucciones de Stratagene. Placa 100-400μl de bacterias transformadas en placas de agar LB que contienen ampicilina 100μg/ml e incubar a 37 ° C.
- Miniprep 2-4 colonias individuales para aislar el ADN del plásmido. Confirmar la introducción exitosa de mutaciones deseadas mediante la secuenciación y el análisis de ADN aislado.
2. Transfección transitoria de MycKISS1R en células HEK-293
- Los siguientes experimentos se llevan a cabo en células humanas de riñón embrionario (HEK-293) cultivadas en una incubadora de CO 2 (5% CO 2) a 37 ° C en DMEM complementado con un feto de 10% de suero bovino (SFB) y 1% de penicilina / estreptomicina.
- Semillas de células HEK-293 a 2.5x10 5 células / ml en placas de 6 pocillos y dejar que crezcan durante la noche a 37 ° C antes de la transfección. NOTA: (i) el uso de pozos por triplicado para cada condición experimental, (ii) la confluencia celular ideal en el momento de la transfección es de 30% -50%.
- Transfectar células HEK-293 usando el reactivo de transfección GenePorter (Genlantis), de acuerdo con las instrucciones del fabricante: la mitad de la mezcla de la DMEM libre de suero con 0.5μg pCS2 + MycKISS1R más control 0.5μg (vacío) vector para totalizar 1 g de ADN / pozo. Mezclar la otra mitad con el reactivo de transfección 10μl por ug de ADN transfectadas. NOTA: La concentración Ideal ADN plásmido puede variar.
3. Tratamiento de células y la lisis
- 24 horas después de la transfección, reemplace medio celular con 1 ml DMEM conteniendo FBS 2,5% (para disminuir el metabolismo de las células). NOTA: Esta disminución en el suero puede facilitar y / o ampliar la detección de los resultados.
- Añadir inhibidor lisosoma (100 microgramos / pocillo de leupeptina) directamente en cada pozo de un total de 6 pocillos. Se incuba a 37 ° C durante 6 o 16 h (o en otras ocasiones se desea)
- Añadir recién preparados inhibidores del proteasoma (10μM / pocillo de MG132) directamente en todos los pozos de dos entero placas de 6 pocillos. Se incuba a 37 ° C durante 2, 4, 6 o 16 horas (o momento que se desee). Añadir vehículos a todos los pocillos de la cuarta placa de 6 pocillos (0 punto del tiempo) y se incuba a 37 ° C durante 16 horas
- Cuando la incubación es más, se mueven las placas de hielo y llevar a cabo este procedimiento de lisis completa en hielo para evitar la degradación de proteínas:
- Para aumentar la producción de proteínas, se combinan los triplicados en placas de 6 pocillos en un centr solaifuge tubo
- Aspirar las células medianas y lavar una vez con 1 ml de helado de buffer fosfato salino (PBS)
- Agregar 100μl de helado de buffer de lisis (20 mM HEPES, pH 7,4, 1% NP-40, 150 mM NaCl, 1 mM EDTA, 0,25% de desoxicolato de sodio) que contienen inhibidores de la proteasa (1x cóctel que contiene 100 mM AEBSF-HCl, 80μM aprotinina, bestatin 5mM, 1,5 mM E-64, 0,5 M EDTA, 2 mM y 1 mM leupeptina pepstatina A, 2 mM PMSF más) a cada pocillo
- Eliminar las células con un rascador de células y de transferencia de células lisados a tubos de centrífuga
- Pasar las células ~ 10 veces a través de una aguja de calibre 20. NOTA: No sonicar muestras para la detección de Western blot de proteínas de membrana. Sonicación conduce a la agregación de proteínas de membrana, que no se migran correctamente durante la electroforesis
- Incubar lisados celulares durante 1 hora a 4 ° C sobre una plataforma oscilante
- Lisados centrifugar células a 4 ° C durante 10 minutos a 10.000 rpm y el sobrenadante x transferencia a nuevos tubos. NOTA: No molestar pellets durante esta etapa
- Determinar la concentración de proteínas en 10μl de sobrenadantes utilizando el método BCA (Pierce)
- Diluir lisados de 1mg/ml con tampón de lisis que contiene inhibidores de la proteasa.
4. Inmunoprecipitación y la detección de Western blot de MycKISS1R
- Realice los pasos siguientes inmunoprecipitación en el hielo (o menos 4 ° C):
- Lave la cantidad adecuada de agarosa conjugada con anticuerpos anti-myc (2.5μg / muestra) dos veces con helado de PBS y añadir esto a 400μg de proteína lisado.
- Inmunoprecipitar la MycKISS1R en lisados de la noche a 4 ° C en una plataforma de balanceo con una suave agitación.
- Decantar perlas de agarosa por centrifugación pulso a 4 ° C (hasta 10.000 rpm x)
- Aspirar y desechar sobrenadantes (sin molestar a los pellets)
- Lávese las cuentas de una vez con helado de buffer de lisis y dos veces con helado de PBS. Invertir suavemente los tubos antes de hacer girar
- Cuentas Ressuspend contiene anticuerpo-limite MycKISS1R en tampón 2x carga de la muestra que contiene 10% de β-mercaptoetanol.
- Western blot de inmunocomplejos MycKISS1R:
- Muestras de calor durante 30 minutos a 37 ° C. Nota: No hierva muestras para la detección de Western blot de proteínas de membrana. Como ultrasonidos, hirviendo también conduce a la agregación de estas proteínas
- Mover los tubos de inmediato en hielo durante 5 min
- Proteínas separadas por SDS-PAGE en un gel de gradiente de 4-15%.
- Traslado al Immobilon-FL membrana de PVDF (por infrarrojos de detección) a 25V durante 30 minutos en tampón de transferencia (Tris base 48 mm, 39 mm de glicina, 1,2 SDS, 20% de metanol, pH 9,2) utilizando el Bio-Rad semi-seco de transferencia aparato
- Lave las membranas durante 5 minutos a temperatura ambiente con Tris-Buffered Saline (TBS) y el bloqueo durante 1 hora a temperatura ambiente con Odyssey Licor de bloqueo en una plataforma oscilante (alternativamente, 5% de leche en TBS puede ser utilizada para bloquear la unión no específica).
- Las membranas se incuban durante la noche a 4 ° C con conejo anti-myc anticuerpos (1:500) en solución de bloqueo conteniendo 0,1% Tween-20
- Eliminar el anticuerpo primario y lavar las membranas 3 veces de 5 minutos cada uno con TBS que contiene 0,1% de Tween-20 (TBST)
- Incube las membranas durante 1 hora a temperatura ambiente con la cabra Infra-800CW RedDye ® marcado anti-IgG de conejo (1:10.000) en tampón de bloqueo que contiene 0,1% de Tween-20 y 0,01% de SDS
- Eliminar el anticuerpo secundario, lavar las membranas 3 veces de 5 minutos cada uno con TBST y una última vez con TBS sólo (para eliminar el resto de Tween-20)
- Imágenes y la cuantificación de MycKISS1R con el LI-COR Odyssey Infra-Red Imager:
- El MycKISS1R en las membranas se creará una imagen utilizando el LI-COR Odyssey Infra-Red Imager. Para comenzar, coloque la membrana en la esquina inferior izquierda del escáner Odyssey, alineándola con la red. Cubrir con la alfombrilla de goma, suavizar las burbujas con el rodillo y cierre la tapa
- Crear un nuevo archivo de proyecto en el equipo. El nombre del archivo, haga clic en "hacer", y luego entrar en la sesión de escaneado en la "exploración". El tamaño de la caja de la consola escáner para adaptarse a su membrana, y luego elegir una resolución de 169μm y calidad de imagen medio
- Elija los valores de intensidad de 700 (rojo) y 800 (verde) de canales de acuerdo con la fuerza esperada de cada señal. Esto es para fines de visualización de la señal y no va a influir en la cuantificación. Haga clic en "Start Scan"
- Nombre y guardar el escaneo, haga clic en "OK" para abrirlo en una nueva ventana para la cuantificación. Monómeros MycKISS1R deben ser visibles en aproximadamente 43kD
- Usando la "caja" de la herramienta (en la barra lateral izquierda), dibuje un cuadro alrededor de la primera banda. Arrastre el cuadro de alrededor para asegurarse de que todas las bandas de encajar, entonces "copiar" y "pegar" la caja sobre todas las bandas
- Seleccione todas las casillas con "Ctrl + A", y luego seleccione la opción de restar el fondo mediana. Haga clic en "informe" en el menú superior y una hoja de cálculo con los valores de cuantificación aparecerá. Los resultados representativos se muestran aquí son represented como doble-aumento en las células no tratadas (tiempo cero)
5. Los resultados representativos:
- Mutagénesis dirigida de secuencia altamente rica en GC gen KISS1R:
Tabla 1. Muestra la combinación de reactivos para mejorar la eficiencia de la amplificación KISS1R. Esta combinación se ha utilizado con éxito para introducir varias mutaciones dirigidas en contra de las distintas regiones de la secuencia de cDNA KISS1R, así como para la amplificación de este gen altamente rica en GC. La figura 1 muestra las condiciones del ciclo y la amplificación de la mutagénesis del KISS1R. Estas condiciones se han modificado desde el QuikChange kit de mutagénesis dirigida (Stratagene).
Figura 2. Muestra un resultado representativo de usar este protocolo optimizado. La adición de DMSO al 4% o el 8% combinado con el reforzador PCRx mejora significativamente el rendimiento de la amplificación de la GC-ricos KISS1R cDNA que contienen el plásmido. En este experimento representativo, el 4% DMSO siempre un poco mejor las condiciones de amplificación en comparación con el 8% de DMSO. Transformación de los productos de amplificación DpnI tratados con ultracompetent E. coli produce normalmente de 100 a más de 1.000 colonias, y la tasa de introducción con éxito de mutaciones deseadas es del 80-90% según lo determinado por la secuenciación del ADN. - El uso de las construcciones MycKISS1R para estudiar la fisiología de los receptores:
En este experimento representativo, el tipo salvaje pCS2 + MycKISS1R amplificado de acuerdo con el protocolo optimizado se describe aquí se utiliza para estudiar la degradación in vivo del receptor de KISS1 en una línea celular correspondiente (HEK-293) que expresan transitoriamente MycKISS1R. Después de tratar el transfectaron células HEK-293 con el lisosoma (leupeptina) o del proteasoma (MG132) inhibidores de acuerdo con el protocolo descrito en los métodos, las células son lisadas y procesadas para Western blot. El panel superior de la Figura 3 se muestra la exploración de los monómeros MycKISS1R mientras que el panel de abajo muestra la cuantificación de las bandas se muestra en el panel superior. La cuantificación de las bandas indica que ninguno de 6h ni 16h de tratamiento leupeptina afectado los niveles de proteína MycKISS1R. Por el contrario, el tratamiento con MG132 como resultado un incremento en función del tiempo en proteínas MycKISS1R en estas células, que culmina con una acumulación de 45 veces del receptor después de 16 horas de incubación con MG132. Estas observaciones indican que, a diferencia de la mayoría de los G receptores acoplados a proteínas, KISS1R se degrada por el proteasoma (en lugar de los lisosomas).
| DMSO | |||
| Reactivos | 0 | 4% | 8% |
| De agua | 35 | 33 | 31 |
| 10x Pfu ultra buffer Taq | 5 | 5 | 5 |
| Mezcla de dNTP (10 mM) | 1 | 1 | 1 |
| Primer sentido (25pmol/μl) | 1 | 1 | 1 |
| Primer antisentido (25pmol/μl) | 1 | 1 | 1 |
| 10x PCRx Reforzador Solución | 5 | 5 | 5 |
| DMSO | 0 | 2 | 4 |
| Ufp ultra Taq polimerasa (2.5U/μl) | 1 | 1 | 1 |
| ADN plásmido (20ng/μl) | 1 | 1 | 1 |
Tabla 1. Combinación de reactivos utilizado con éxito para mutar y amplificar la KISS1R rica en GC
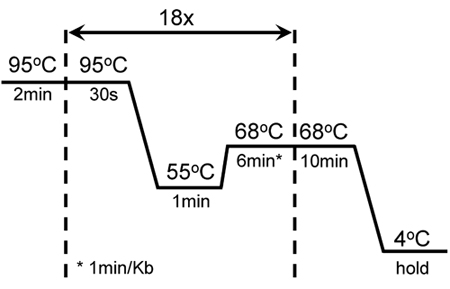
Figura 1. Las condiciones del ciclo de mutagénesis éxito y la amplificación de GC-ricos KISS1R: Un arranque en caliente 2 minutos fue seguido por 18 ciclos de 30 segundos de fusión a 95 ° C, 1 min recocido a 55 ° C min de extensión y 6 a 68 ° C. Una extensión adicional de 10 min a 68 ° C se añadió al final del último ciclo. Estos valores se ajustaron al protocolo original de la mutagénesis QuikChange II kit XL-mutagénesis dirigida (Stratagene).
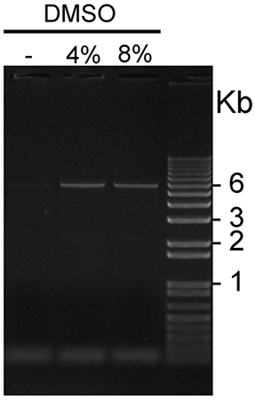
Figura 2. Visualización del GC-ricos pCS2 + Myc KISS1R amplificado en la presencia o ausencia de DMSO: cinco alícuotas de pCS2 + KISS1R Myc amplificado en la presencia de 0, 4 u 8% de DMSO fueron cargados en este representante de 1% en gel de agarosa teñido con etidio bromuro y se visualiza la luz UV usando. Las bandas de plásmido de 6Kb son visibles en ambos carriles cargados con productos de amplificación de PCR en presencia de un 4% y 8% de DMSO, pero no en la primera calle, que estaba cargado con una PCRproducto amplificado en la ausencia de DMSO.
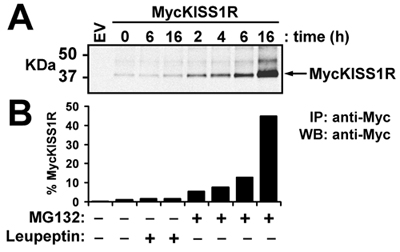
Figura 3. Efecto de la leupeptina o MG132 en los niveles de proteína Myc KISS1R en células HEK-293: HEK-293 células que expresan Myc KISS1R fueron tratados con 100μg/ml leupeptina o 10μM MG132 a 37 ° C durante las horas designadas. Myc KISS1R de 400μg de lisado celular se immunoprecipitated con 2.5μg de agarosa conjugada con anticuerpos anti-myc y analizada por Western blot. (A) LI-COR Odyssey detección de KISS1R Myc después de la incubación de immunoblots con conejo anti-Myc-tag anticuerpos seguido de incubación con IRDye 800CW marcado anti-conejo (B) Cuantificación de las bandas KISS1R Myc se muestra en (A) mediante la LI-COR Odyssey software de cuantificación. Los resultados se representan como doble-aumento en las células no tratadas (tiempo 0).
Discusión
Mutagénesis dirigida se ha utilizado para estudiar la función de proteínas mediante la introducción de cambios de nucleótidos en la secuencia de codificación de los genes blanco de más de tres décadas. La técnica original fue descrita en 1978 por el químico británico-canadiense y ganador del Premio Nobel Michael Smith 10. Michael Smith compartió el 1993 Premio Nobel de Química con Kary Mullis, el bioquímico estadounidense que inventó la Reacción en Cadena de la Polimerasa (PCR) 11. ...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Este trabajo fue financiado parcialmente por el Poder Reproductiva del Instituto Nacional de Salud Infantil y Desarrollo Humano (NICHD - R21 HD059015) y el Premio Charles H. Hood Foundation Niño Pequeño Investigador Investigación en Salud (Boston, MA).
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | |
| 10x PCRx Reforzador Solución | Invitrogen | 52391 | |
| PfuUltra de alta fidelidad de la polimerasa de ADN Alternativa detergente | Stratagene | 600385 | |
| DPN-I | New England Biolabs | R0176 | |
| XL10-Gold Ultracompetent E. células de E. coli | Stratagene | 200314 | |
| DMEM | Cellgro | 10 a 013 CV | |
| De suero fetal bovino | Atlanta Productos Biológicos | S11550 | |
| Geneporter Transfection Reagent | Genlantis | T201007 | |
| Leupeptina | Calbiochem | 108975 | |
| MG132 | Calbiochem | 47491 | |
| 10xPBS | Ambion | AM9625 | |
| Cóctel de inhibidores de la proteasa y PMSF | Santa Cruz de Biotecnología | Sc-24948 | |
| Pierce BCA Protein Kit de ensayo | Thermo Scientific | 23225 | |
| anti-Myc etiqueta (clon 4A6) de agarosa conjugada | Millipore | 16-219 | |
| 2x tampón de carga | BioRad | 161-0737 | |
| Criterio de Tris-HCl prefabricados gel, 4.15% de gradiente | BioRad | 345-0028 | |
| Immobilon-FL membrana de PVDF | Millipore | IPFL00010 | |
| Odyssey tampón de bloqueo | LI-COR Biosciences | 927-40000 | |
| 10xTBS | BioRad | 170-6435 | |
| Conejo anti-myc anticuerpos | Señalización celular | 2272 | |
| Cabra anti-conejo IRDye 800CW | LI-COR Biosciences | 926-32211 | |
| Imagen infrarroja del sistema Odyssey | LI-COR Biosciences | ||
| Proteasa Inhibidor de detener Cocktail (100 veces) | Thermo Scientific | 78430 |
Referencias
- Seminara, S. B., Messager, S., Chatzidaki, E. E. The GPR54 Gene as a Regulator of Puberty. N Engl J Med. 349, 1614-1627 (2003).
- de Roux, N., Genin, E., Carel, J. C. Hypogonadotropic Hypogonadism Due to Loss of Function of the KiSS1-Derived Peptide Receptor GPR54. Proc Natl Acad Sci U S A. 100, 10972-10976 (2003).
- Messager, S., Chatzidaki, E. E., Ma, D. Kisspeptin Directly Stimulates Gonadotropin-Releasing Hormone Release via G Protein-Coupled Receptor 54. Proc Natl Acad Sci U S A. 102, 1761-1766 (2005).
- Teles, M. G., Bianco, S. D., Brito, V. N. A GPR54-Activating Mutation in a Patient with Central Precocious Puberty. N Engl J Med. 358, 709-715 (2008).
- Tenenbaum-Rakover, Y., Commenges-Ducos, M., Iovane, A. Neuroendocrine Phenotype Analysis in Five Patients with Isolated Hypogonadotropic Hypogonadism due to a L102P Inactivating Mutation of GPR54. J Clin Endocrinol Metab. 92, 1137-1144 (2007).
- Semple, R. K., Achermann, J. C., Ellery, J. Two Novel Missense Mutations in G Protein-Coupled Receptor 54 in a Patient with Hypogonadotropic Hypogonadism. J Clin Endocrinol Metab. 90, 1849-1855 (2005).
- Wacker, J. L., Feller, D. B., Tang, X. B. Disease-Causing Mutation in GPR54 Reveals the Importance of the Second Intracellular Loop for Class A G-Protein-Coupled Receptor Function. J Biol Chem. 283, 31068-31078 (2008).
- Szereszewski, J. M., Pampillo, M., Ahow, M. R. GPR54 regulates ERK1/2 activity and hypothalamic gene expression in a Galpha(q/11) and beta-arrestin-dependent manner. PLoS One. 5, e12964-e12964 (2010).
- Bianco, S. D. C., Vandepas, L., Correa-Medina, M., Gereben, B., Mukherjee, A., Kuohung, W., Carroll, R., Teles, M. G., Latronico, A. C., Kaiser, U. B. KISS1R Intracellular Trafficking and Degradation: Effect of the Arg386Pro Disease-Associated Mutation. Endocrinology. , (2011).
- Hutchison, C. A., Phillips, S., Edgell, M. H. Mutagenesis at a Specific Position in a DNA Sequence. J Biol Chem. 253, 6551-6560 (1978).
- Mullis, K. B. The Unusual Origin of the Polymerase Chain Reaction. Sci Am. 262, 56-61 (1990).
- Henke, W., Herdel, K., Jung, K., Schnorr, D., Loening, S. A. Betaine improves the PCR amplification of GC-rich DNA sequences. Nucleic Acids Res. 25, 3957-3958 (1997).
- Sahdev, S., Saini, S., Tiwari, P., Saxena, S., Singh Saini, K. Amplification of GC-rich genes by following a combination strategy of primer design, enhancers and modified PCR cycle conditions. Mol Cell Probes. 21, 303-307 (2007).
- Kong, K. C., Poyner, D. R., Wheatly, M. Chapter 10. MSTA. in G Protein-Coupled Receptors: Essential Methods. , 197-204 (2010).
- Hall, R. A., George, S. R., O'Dowd, B. F. Chapter 9. G protein-Coupled Receptor-Protein Interactions. , 170-171 (2005).
- Chaturvedi, K., Bandari, P., Chinen, N., Howells, R. D. Proteasome Involvement in Agonist-Induced Down-Regulation of Mu and Delta Opioid Receptors. J Biol Chem. 276, 12345-12355 (2001).
- Shenoy, S. K., McDonald, P. H., Kohout, T. A., Lefkowitz, R. J. Regulation of Receptor Fate by Ubiquitination of Activated Beta 2-Adrenergic Receptor and Beta-Arrestin. Science. 294, 1307-1313 (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados