É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Mutagênese e análise de mutações genéticas nos receptores de Seqüência GC-rica KISS1 identificados em humanos com distúrbios reprodutivos
Neste Artigo
Resumo
Mutações no receptor kisspeptina (KISS1R) estão associados com distúrbios reprodutivos em pacientes. Aqui nós descrevemos como introduzir mutações de interesse na seqüência GC-rica de KISS1R, bem como o uso de construções KISS1R para caracterizar a via de degradação do receptor por imunoprecipitação e western blot
Resumo
O receptor kisspeptina (KISS1R) é um receptor acoplado à proteína G reconhecido como o gatilho da puberdade e um regulador de competência reprodutiva na vida adulta 1,2,3. Mutações inativadoras no KISS1R identificados em pacientes têm sido associados com hipogonadismo hipogonadotrófico iodiopathic 1,2 (IHH) e puberdade precoce 4. Estudos funcionais destes mutantes são cruciais para nossa compreensão dos mecanismos subjacentes à regulação da reprodução por este receptor, bem como aqueles moldar a evolução das doenças, que resultam de sinalização KISS1R anormal e função. No entanto, a seqüência altamente GC-rica do gene KISS1R torna bastante difícil a introdução de mutações ou amplificar o gene que codifica este receptor pela PCR.
Aqui nós descrevemos um método para introduzir mutações de interesse para esta seqüência altamente GC-rica que tem sido utilizado com sucesso para gerar mais de uma dúzia de mutantes KISS1R em nosso laboratório. Nós otimizamos as condições de PCR para facilitar a ampliação de uma série de mutantes KISS1R que incluem substituições, supressões ou inserções na seqüência KISS1R. A adição de uma solução enhancer PCR, bem como de uma pequena porcentagem de DMSO foram especialmente úteis para melhorar a amplificação. Este procedimento otimizado pode ser útil para outras GC-rica modelos também.
O vetor de expressão que codifica a KISS1R está sendo usado para caracterizar a sinalização e função desse receptor, a fim de entender como as mutações podem mudar KISS1R função e levar a fenótipos associados reprodutiva. Assim, as aplicações potenciais de mutantes KISS1R gerados por mutagênese sítio-dirigida pode ser ilustrado por muitos estudos 1,4,5,6,7,8. Como exemplo, a mutação de ganho de função-nos KISS1R (Arg386Pro), que está associada com puberdade precoce, foi mostrado para prolongar a capacidade de resposta do receptor à estimulação ligante 4, bem como de alterar a taxa de degradação da KISS1R 9 . Curiosamente, nossos estudos indicam que KISS1R é degradada pelo proteassoma, em oposição à degradação lisossomal clássico descrito para a maioria das proteínas G-coupled receptors 9. No exemplo aqui apresentado, a degradação do KISS1R é investigada em células embrionárias do rim (HEK-293) transitoriamente expressando-Myc tag KISS1R (MycKISS1R) e tratados com inibidores de proteassoma ou lisossomos. Lisados celulares são immunoprecipitated agarose usando um conjugado anticorpo anti-myc seguido de western blot. Detecção e quantificação de MycKISS1R em blots é realizada utilizando o Sistema Odyssey LI-COR Infrared. Esta abordagem pode ser útil no estudo da degradação de outras proteínas de interesse também.
Protocolo
1. Sítio-dirigida mutagênese da seqüência altamente GC-rica gene KISS1R
- Modelo: seqüência de cDNA completo do KISS1R humano com uma tag-Myc fundida ao seu N-terminal. Esta seqüência é clonado no vetor de expressão pCS2 +, que é compatível com as linhas de células de mamíferos posteriormente utilizado para transfections. Este vetor de expressão é aqui referida como pCS2 + Myc KISS1R.
- Desenho de primers: primers são projetados para transportar mutações desejadas, de acordo com instruções do Quikchange Site-Directed Mutagênese kit (Stratagene). Em resumo:
- Ambos os primers (frente e verso) deve conter a mutação desejada e recozer a mesma seqüência de fios oposto do plasmídeo (por exemplo, frente e verso primers são complementares uns aos outros)
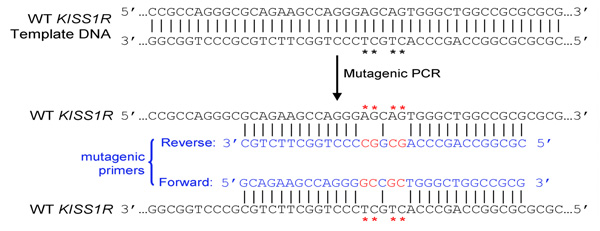
- Primers devem ser 25-45 bases longas e terminam em uma ou mais bases C ou G
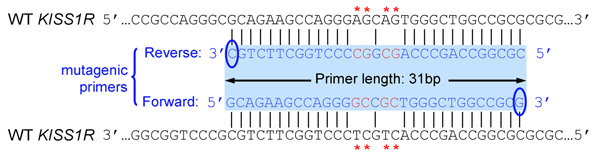

- Mutação introduzido (s) devem estar no meio de primer e ladeado por ~ 10-15 bases da seqüência correta em ambos os lados
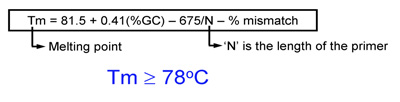
- Temperatura de fusão (Tm) dos primers deve ser igual ou superior a 78 ° C. Use as seguintes fórmulas para estimar a Tm:
- Ao introduzir mutações: Tm = 81,5 + 0,41 (% GC) - 675 / N - incompatibilidade% (N é o comprimento de primer em bases)
- Ao introduzir inserções ou exclusões: Tm = 81,5 + 0,41 (% GC) - 675 / N (N não inclui as bases que estão sendo inseridas ou excluídas)
- Use primers já dessalgado (sem purificações adicionais necessárias).
- Ambos os primers (frente e verso) deve conter a mutação desejada e recozer a mesma seqüência de fios oposto do plasmídeo (por exemplo, frente e verso primers são complementares uns aos outros)
- As melhores condições de amplificação incluem a adição de solução PCRx Enhancer (Invitrogen) e DMSO. Pelo menos, 2 concentrações de DMSO, como 4% e 8%, deve ser testado. Condições de PCR estão apresentados na Tabela I e na Figura 1, e resultados de um representante da amplificação por PCR pCS2 + MycKISS1R é mostrado na Figura 2.
- Eliminar o ADN dos pais sobre os produtos de amplificação por digestão do DNA metilado com DpnI de 1h 30min a 37 ° C: misture em um tubo de centrífuga 17.5μl do produto da PCR com 2μl de NEBuffer 10x 4 e 0.5μl de DpnI (10U; New England Biolabs).
- Transformar DpnI tratados com PCR produto: 2μl mistura de DpnI tratados com DNA com 45μl de XL10-Gold E. Ultracompetent coli (Stratagene) em pré-refrigerados 15 ml tubo de cultura de células. Adicionar 2μl de β-mercaptoetanol e siga as instruções Stratagene. Placa 100-400μl de bactérias transformadas em LB-ágar placas contendo 100μg/ml ampicilina e incubar a 37 ° C.
- Miniprep 2-4 colônias individuais para isolar DNA plasmidial. Confirmar a introdução bem sucedida de mutações desejado por seqüenciamento e análise de DNA isolado.
2. Transfecção transitória da MycKISS1R em células HEK-293
- Os seguintes experimentos são realizados em células embrionárias do rim (HEK-293) cultivadas em uma incubadora de CO 2 (5% CO 2) a 37 ° C em DMEM suplementado com 10% soro fetal bovino (FBS) e 1% de penicilina / estreptomicina.
- Semente HEK-293 células em 2.5x10 5 células / ml em 6 bem-pratos e deixá-los crescer durante a noite a 37 ° C antes de transfecção. NOTA: (i) utilização de poços em triplicado para cada condição experimental, (ii) confluência celular ideal no momento da transfecção é de 30% -50%.
- Transfecção de células HEK-293 utilizando o reagente Transfection GenePorter (Genlantis), de acordo com as instruções do fabricante: metade Mix do DMEM sem soro com 0.5μg pCS2 + MycKISS1R mais 0.5μg controle (vazio) vetor para totalizar 1μg de DNA / poço. Misture a outra metade com o reagente de transfecção 10μl por mg de DNA transfectadas. NOTA: concentração de DNA Ideal plasmídeo pode variar.
3. Tratamento com células e lise
- 24h após a transfecção, substitua médio de células com 1ml DMEM contendo FBS 2,5% (para diminuir o metabolismo celular). NOTA: Esta diminuição no soro podem facilitar e / ou ampliar a detecção de resultados.
- Adicionar inibidor lisossoma (100μg / poço de Leupeptin) diretamente em cada poço de uma placa de 6 poços inteiro. Incubar a 37 ° C por 6 ou 16 h (ou outras vezes desejado)
- Adicionar recém-preparado inibidor de proteassoma (10μM / poço de MG132) diretamente em todos os poços de dois inteiro 6-lamelas. Incubar a 37 ° C por 2, 4, 6 ou 16h (ou vezes desejado). Adicionar veículo para todos os poços da placa 6-bem quarta (0 ponto do tempo) e incubar a 37 ° C por 16h
- Quando a incubação é longo, mover as placas de gelo e realizar o procedimento de lise inteira no gelo para evitar a degradação de proteínas:
- Para aumentar a produção de proteína, combinar os três exemplares em 6 bem-placas em um único centrtubo ifuge
- Aspirar células médio e lave uma vez com 1ml de gelada de tampão fosfato salina (PBS)
- Adicionar 100μl de gelada tampão de lise (20mM HEPES, pH 7.4, 1% NP-40, 150mm NaCl, 1mM EDTA, desoxicolato de sódio 0,25%) contendo inibidores da protease (1x cocktail contendo 100mM AEBSF-HCl, 80μM aprotinina, bestatin 5mM, 1,5 mM E-64, EDTA 0,5 M, 2mM leupeptin e 1mM pepstatin A, além de 2mM PMSF) a cada poço
- Remover as células com um raspador de célula e célula de transferência lisados para tubos de centrífuga
- Passe células ~ 10 vezes através de uma agulha de calibre 20. NOTA: Não sonicate amostras destinadas para a detecção de western blot de proteínas da membrana. Sonicação leva à agregação de proteínas da membrana, que não serão migrados corretamente durante a eletroforese
- Incubar lisados celulares por 1h a 4 ° C em uma plataforma de balanço
- Lisados de células centrífuga a 4 ° C por 10 min a 10.000 rpm x sobrenadantes e transferência para novos tubos. NOTA: Não perturbe pellets durante esta etapa
- Determinar a concentração de proteína em 10μl do sobrenadante usando o método BCA (Pierce)
- Lisados para diluir 1mg/ml com tampão de lise contendo inibidores da protease.
4. Imunoprecipitação e Western blot detecção de MycKISS1R
- Execute as etapas seguintes imunoprecipitação no gelo (ou a 4 ° C):
- Lave a quantidade adequada de agarose-conjugados de anticorpos anti-myc (2.5μg amostra /) duas vezes com PBS gelado e adicionar este 400μg de proteína lisado.
- Immunoprecipitate o MycKISS1R em lisados durante a noite a 4 ° C em uma plataforma de balanço com leve agitação.
- Spin down contas agarose por centrifugação de pulso a 4 ° C (até 10.000 rpm x)
- Aspirar e descartar sobrenadante (sem perturbar a pellets)
- Lavar uma vez com contas gelada tampão de lise e duas vezes com PBS gelado. Inverter os tubos suavemente antes de spinning
- Contas Ressuspend contendo anticorpo ligado MycKISS1R em tampão de amostra 2x carregamento contendo 10% de β-mercaptoetanol.
- Western blot de imunocomplexos MycKISS1R:
- Amostras de calor por 30 min a 37 ° C. Observação: Não ferver a colheita de amostras destinadas para a detecção de western blot de proteínas da membrana. Como sonicação, fervendo também leva a agregação dessas proteínas
- Mover tubos imediatamente em gelo por 5 min
- Proteínas separadas por SDS-PAGE em gel gradiente de 4-15%.
- Transferência para Immobilon-FL membrana PVDF (por infra-vermelho de detecção) de 25V por 30 min em tampão de transferência (48mm Tris base, 39mm glicina, 1,2 mm SDS, metanol 20%, pH 9,2), utilizando o Bio-Rad Transferência Semi-Dry aparelho
- Lavar as membranas por 5 min à temperatura ambiente com Tris-Buffered Saline (TBS) e bloquear por 1h em temperatura ambiente com a Odyssey Licor de bloqueio em uma plataforma de balanço (alternativamente, 5% de leite em TBS pode ser usado para bloquear ligações não específicas).
- Membranas incubar overnight a 4 ° C com coelho anticorpo anti-myc (1:500) em solução de bloqueio contendo 0,1% Tween-20
- Remover o anticorpo primário e lavar as membranas 3 vezes de 5 min cada um com TBS contendo 0,1% Tween-20 (TBST)
- Incubar as membranas por 1h em temperatura ambiente com a cabra Infra-RedDye ® 800CW marcado anti-IgG de coelho (1:10.000) em tampão de bloqueio contendo 0,1% Tween-20 e 0,01% SDS
- Remover anticorpo secundário, lavar as membranas 3 vezes de 5 min cada um com TBST e uma última vez com TBS apenas (para remover restantes Tween-20)
- Imagem e quantificação de MycKISS1R usando o LI-COR Odyssey Imager Infra-Vermelho:
- O MycKISS1R nas membranas será fotografada usando o LI-COR Odyssey Imager Infra-Vermelho. Para começar, coloque membrana no canto esquerdo inferior do scanner Odyssey, alinhando-a com a grade. Cubra com o tapete de borracha, suavizar bolhas com rolo e feche a tampa
- Criar um novo arquivo de projeto no computador. O nome do arquivo, clique em "done", e digite o login scanner em "scan". Tamanho da caixa do console scanner para atender às suas membranas, e depois escolher a resolução e qualidade de imagem 169μm médio
- Escolha as configurações de intensidade para 700 (vermelho) e 800 (verde) canais de acordo com a força esperada de cada sinal. Isto é para fins de visualização de sinal apenas e não deve influenciar a quantificação. Clique em "start scan"
- Nome e salvar a digitalização, clique em "OK" para abri-lo em uma nova janela para a quantificação. Monômeros MycKISS1R deve ser visível em aproximadamente 43kD
- Usando a "caixa" de ferramentas (na barra lateral esquerda), desenhe uma caixa ao redor da primeira banda. Arraste a caixa de volta para se certificar de todas as bandas de se ajustar, em seguida, "copy" e "colar" a caixa sobre todas as bandas
- Selecione todas as caixas usando "Ctrl + A", e selecione a opção para subtrair fundo mediana. Clique em "relatório" no menu superior e uma planilha com os valores de quantificação irá aparecer. Resultados aqui apresentados são representativos represented como dobra-aumento de mais de células não tratadas (tempo zero)
5. Resultados representativos:
- Sítio-dirigida mutagênese da seqüência altamente GC-rica KISS1R gene:
Tabela 1. Mostra a combinação de reagentes para melhorar a eficiência de amplificação KISS1R. Esta combinação é usada com sucesso para introduzir várias mutações dirigidos contra regiões distintas da seqüência de cDNA KISS1R, bem como para a amplificação deste gene altamente GC-rica. A Figura 1 mostra ciclismo e condições para a amplificação de mutagênese KISS1R. Estas condições foram modificados a partir da QuikChange Site-Directed Mutagênese kit (Stratagene).
Figura 2. Mostra um resultado representativo usando este protocolo otimizado. A adição de 4% ou 8% DMSO combinado com o Enhancer PCRx melhora significativamente o rendimento de amplificação da GC-rica KISS1R cDNA contendo plasmídeo. Neste experimento representativo, DMSO 4% desde condições de amplificação ligeiramente melhor em comparação com DMSO 8%. Transformação de produtos DpnI tratados com amplificação com E. ultracompetent coli normalmente produz de 100 a mais de 1.000 colônias, ea taxa de introdução bem sucedida de mutações desejado é de 80-90%, conforme determinado pelo seqüenciamento de DNA. - Uso de construções MycKISS1R para estudo da fisiologia dos receptores:
Neste experimento representante, o tipo selvagem pCS2 + MycKISS1R amplificado de acordo com o protocolo otimizado aqui descrito é usado para estudar in vivo da degradação receptor KISS1 em uma linhagem de células relevantes (HEK-293) transitoriamente expressar MycKISS1R. Depois de tratar da HEK-293 transfectadas células com lisossoma (leupeptin) ou proteassoma (MG132) inibidores de acordo com o protocolo descrito nos métodos, as células são lisadas e processados para western blot. O painel superior da Figura 3 mostra a análise de monômeros MycKISS1R enquanto o painel inferior mostra a quantificação das bandas mostrado no painel superior. Quantificação de bandas indica que nem 6h nem 16h de tratamento leupeptin afetados os níveis de proteína MycKISS1R. Por outro lado, o tratamento com MG132 resultou em um aumento tempo-dependente em proteínas MycKISS1R nessas células, que culmina com um acúmulo de 45 vezes do receptor após 16h de incubação com MG132. Estas observações indicam que, ao contrário da maioria G receptores acoplados à proteína, KISS1R é degradada pelo proteassoma (em vez do lisossoma).
| DMSO | |||
| Reagentes | 0 | 4% | 8% |
| Água | 35 | 33 | 31 |
| 10x Pfu Ultra tampão Taq | 5 | 5 | 5 |
| dNTP mix (10mm) | 1 | 1 | 1 |
| Sentido Primer (25pmol/μl) | 1 | 1 | 1 |
| Antisense Primer (25pmol/μl) | 1 | 1 | 1 |
| 10x PCRx Solução Enhancer | 5 | 5 | 5 |
| DMSO | 0 | 2 | 4 |
| Pfu Ultra polimerase Taq (2.5U/μl) | 1 | 1 | 1 |
| DNA plasmídeo (20ng/μl) | 1 | 1 | 1 |
Tabela 1. Combinação de reagentes usados com sucesso para se transformar e ampliar a KISS1R GC-rica
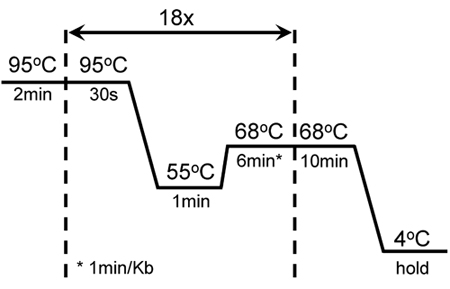
Figura 1. Condições de ciclismo de mutagênese sucesso e ampliação da GC-rica KISS1R: Um começo quente 2min foi seguido por 18 ciclos de 30 seg de fusão a 95 ° C, 1 min de anelamento a 55 ° C min de extensão e 6 a 68 ° C. Uma extensão adicional de 10 min a 68 ° C foi adicionado no fim do último ciclo. Estas definições foram ajustados a partir do protocolo de mutagênese original do II QuikChange XL-Site-Directed Mutagênese kit (Stratagene).
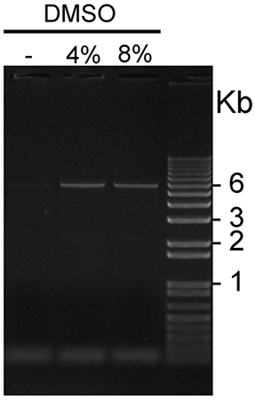
Figura 2. Visualização de GC-rica pCS2 + Myc KISS1R amplificado na presença ou ausência de DMSO: Cinco alíquotas mL de pCS2 + KISS1R Myc amplificado na presença de 0, 4 ou 8% DMSO foram carregados este representante de agarose 1% corados com gel de etídio brometo e luz UV visualizado usando. As bandas de plasmídeo de 6kb são visíveis em ambas as pistas carregado com produtos de PCR amplificados na presença de 4% e DMSO 8%, mas não na primeira pista, que estava carregado com uma PCRproduto amplificado na ausência de DMSO.
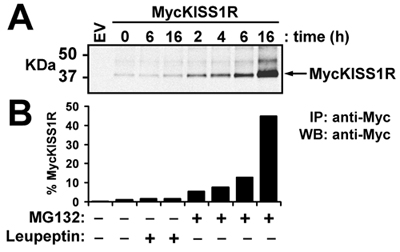
Figura 3. Efeito da leupeptin ou MG132 sobre os níveis de proteína Myc KISS1R em células HEK-293: HEK-293 células que expressam Myc KISS1R foram tratadas com 100μg/ml leupeptin ou 10μM MG132 a 37 ° C para os tempos designados. Myc KISS1R em 400μg de lisado de células foi immunoprecipitated com 2.5μg de agarose-conjugados de anticorpos anti-myc e analisadas por western blot. (A) de detecção de Odyssey LI-COR de KISS1R Myc após incubação de Immunoblots com coelho de anticorpos anti-Myc tag seguido por incubação com IRDye 800CW marcado coelho anti; (B) Quantificação de bandas KISS1R Myc mostrado em (A) usando o LI-COR software quantificação Odyssey. Resultados são representados como dobra-aumento de mais de células não tratadas (tempo 0).
Discussão
Sítio-dirigida mutagênese tem sido utilizado para estudar a função de proteínas através da introdução de mudanças de nucleotídeos na seqüência de codificação de genes alvo por mais de três décadas. A técnica original foi descrita em 1978 pelo químico britânico-canadense e Prêmio Nobel Michael Smith 10. Michael Smith dividiram o Prêmio Nobel de 1993 em Química com Kary Mullis, o bioquímico americano que inventou o Reação em Cadeia da Polimerase (PCR) 11. O método original d...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho foi parcialmente financiado pelo Poder Reprodutiva do Instituto Nacional de Saúde Infantil e Desenvolvimento Humano (NICHD - R21 HD059015) e pela capa Charles H. Young Foundation Investigator Award Criança Pesquisa em Saúde (Boston, MA).
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
| 10x PCRx Solução Enhancer | Invitrogen | 52391 | |
| PfuUltra alta fidelidade DNA polimerase Detergente Alternativa | Stratagene | 600385 | |
| DPN-I | New England Biolabs | R0176 | |
| XL10-Gold E. Ultracompetent células de Escherichia | Stratagene | 200314 | |
| DMEM | Cellgro | 10-013-CV | |
| Soro fetal bovino | Atlanta Biologicals | S11550 | |
| Geneporter Reagente Transfection | Genlantis | T201007 | |
| Leupeptin | Calbiochem | 108975 | |
| MG132 | Calbiochem | 47491 | |
| 10xPBS | Ambion | AM9625 | |
| Inibidor de protease PMSF e cocktail | Santa Cruz Biotechnology | Sc-24948 | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| anti-Myc tag (clone 4A6) agarose conjugado | Millipore | 16-219 | |
| 2x de tampão de carregamento | BioRad | 161-0737 | |
| Critério de Tris-HCl precast gel, gradiente de 4-15% | BioRad | 345-0028 | |
| Immobilon-FL membrana PVDF | Millipore | IPFL00010 | |
| Tampão de bloqueio Odyssey | LI-COR Biosciences | 927-40000 | |
| 10xTBS | BioRad | 170-6435 | |
| Anticorpos anti-myc coelho | Sinalização celular | 2272 | |
| Cabra anti-coelho IRDye 800CW | LI-COR Biosciences | 926-32211 | |
| Odyssey Infrared Imaging System | LI-COR Biosciences | ||
| Halt Cocktail inibidor da protease (100x) | Thermo Scientific | 78430 |
Referências
- Seminara, S. B., Messager, S., Chatzidaki, E. E. The GPR54 Gene as a Regulator of Puberty. N Engl J Med. 349, 1614-1627 (2003).
- de Roux, N., Genin, E., Carel, J. C. Hypogonadotropic Hypogonadism Due to Loss of Function of the KiSS1-Derived Peptide Receptor GPR54. Proc Natl Acad Sci U S A. 100, 10972-10976 (2003).
- Messager, S., Chatzidaki, E. E., Ma, D. Kisspeptin Directly Stimulates Gonadotropin-Releasing Hormone Release via G Protein-Coupled Receptor 54. Proc Natl Acad Sci U S A. 102, 1761-1766 (2005).
- Teles, M. G., Bianco, S. D., Brito, V. N. A GPR54-Activating Mutation in a Patient with Central Precocious Puberty. N Engl J Med. 358, 709-715 (2008).
- Tenenbaum-Rakover, Y., Commenges-Ducos, M., Iovane, A. Neuroendocrine Phenotype Analysis in Five Patients with Isolated Hypogonadotropic Hypogonadism due to a L102P Inactivating Mutation of GPR54. J Clin Endocrinol Metab. 92, 1137-1144 (2007).
- Semple, R. K., Achermann, J. C., Ellery, J. Two Novel Missense Mutations in G Protein-Coupled Receptor 54 in a Patient with Hypogonadotropic Hypogonadism. J Clin Endocrinol Metab. 90, 1849-1855 (2005).
- Wacker, J. L., Feller, D. B., Tang, X. B. Disease-Causing Mutation in GPR54 Reveals the Importance of the Second Intracellular Loop for Class A G-Protein-Coupled Receptor Function. J Biol Chem. 283, 31068-31078 (2008).
- Szereszewski, J. M., Pampillo, M., Ahow, M. R. GPR54 regulates ERK1/2 activity and hypothalamic gene expression in a Galpha(q/11) and beta-arrestin-dependent manner. PLoS One. 5, e12964-e12964 (2010).
- Bianco, S. D. C., Vandepas, L., Correa-Medina, M., Gereben, B., Mukherjee, A., Kuohung, W., Carroll, R., Teles, M. G., Latronico, A. C., Kaiser, U. B. KISS1R Intracellular Trafficking and Degradation: Effect of the Arg386Pro Disease-Associated Mutation. Endocrinology. , (2011).
- Hutchison, C. A., Phillips, S., Edgell, M. H. Mutagenesis at a Specific Position in a DNA Sequence. J Biol Chem. 253, 6551-6560 (1978).
- Mullis, K. B. The Unusual Origin of the Polymerase Chain Reaction. Sci Am. 262, 56-61 (1990).
- Henke, W., Herdel, K., Jung, K., Schnorr, D., Loening, S. A. Betaine improves the PCR amplification of GC-rich DNA sequences. Nucleic Acids Res. 25, 3957-3958 (1997).
- Sahdev, S., Saini, S., Tiwari, P., Saxena, S., Singh Saini, K. Amplification of GC-rich genes by following a combination strategy of primer design, enhancers and modified PCR cycle conditions. Mol Cell Probes. 21, 303-307 (2007).
- Kong, K. C., Poyner, D. R., Wheatly, M. Chapter 10. MSTA. in G Protein-Coupled Receptors: Essential Methods. , 197-204 (2010).
- Hall, R. A., George, S. R., O'Dowd, B. F. Chapter 9. G protein-Coupled Receptor-Protein Interactions. , 170-171 (2005).
- Chaturvedi, K., Bandari, P., Chinen, N., Howells, R. D. Proteasome Involvement in Agonist-Induced Down-Regulation of Mu and Delta Opioid Receptors. J Biol Chem. 276, 12345-12355 (2001).
- Shenoy, S. K., McDonald, P. H., Kohout, T. A., Lefkowitz, R. J. Regulation of Receptor Fate by Ubiquitination of Activated Beta 2-Adrenergic Receptor and Beta-Arrestin. Science. 294, 1307-1313 (2001).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados