È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Mutagenesi e analisi delle mutazioni genetiche in GC-ricchi Sequenza Receptor KISS1 identificati nell'uomo con disturbi dell'apparato riproduttivo
In questo articolo
Riepilogo
Le mutazioni nel recettore kisspeptina (KISS1R) sono associati a disordini riproduttivi nei pazienti. Qui si descrive come introdurre mutazioni di interesse per il GC-ricca sequenza di KISS1R così come l'uso di costrutti KISS1R per caratterizzare il percorso di degradazione del recettore di immunoprecipitazione e western blot
Abstract
Il recettore kisspeptina (KISS1R) è una proteina G-recettore accoppiato riconosciuto come il grilletto della pubertà e un regolatore di competenza riproduttiva in età adulta 1,2,3. Mutazioni inattivanti in KISS1R identificate nei pazienti sono stati associati con iodiopathic ipogonadismo ipogonadotropo (IHH) 1,2 e la pubertà precoce 4. Studi funzionali di questi mutanti sono cruciali per la nostra comprensione dei meccanismi alla base della regolazione della riproduzione di questo recettore così come quelli plasmare gli esiti della malattia, che derivano da segnalazione KISS1R anormale e funzione. Tuttavia, la sequenza altamente GC-ricchi del gene KISS1R rende piuttosto difficile introdurre mutazioni o amplificare il gene che codifica per il recettore mediante PCR.
Qui si descrive un metodo per introdurre mutazioni di interesse in questa sequenza altamente GC-ricco che è stato usato con successo per generare più di un dozzina di mutanti KISS1R nel nostro laboratorio. Abbiamo ottimizzato le condizioni di PCR per facilitare l'amplificazione di una serie di mutanti KISS1R che includono sostituzioni, cancellazioni o inserimenti nella sequenza KISS1R. L'aggiunta di una soluzione di PCR enhancer, oltre che di una piccola percentuale di DMSO sono stati particolarmente utili per migliorare l'amplificazione. Questa procedura ottimizzata può essere utile per altri modelli GC ricchi pure.
Il vettore di espressione che codifica per il KISS1R è stato utilizzato per caratterizzare la segnalazione e la funzione di questo recettore al fine di comprendere come le mutazioni possono cambiare KISS1R funzione e portano alla fenotipi associati riproduttivo. Di conseguenza, le potenziali applicazioni di mutanti KISS1R generato dalla mutagenesi sito-specifica può essere illustrato da molti studi 1,4,5,6,7,8. A titolo di esempio, il guadagno di funzione mutazione nel KISS1R (Arg386Pro), che è associato con pubertà precoce, ha dimostrato di prolungare la risposta del recettore per la stimolazione ligando 4, nonché di modificare la velocità di degradazione di KISS1R 9 . È interessante notare che i nostri studi indicano che KISS1R è degradato dal proteasoma, in contrapposizione al degrado classico lisosomiale descritto per la maggior parte G recettori accoppiati alla proteina 9. Nell'esempio qui presentato, il degrado del KISS1R è studiato in cellule renali embrionali umane (HEK-293) transitoriamente esprimere Myc-tag KISS1R (MycKISS1R) e trattati con inibitori del proteosoma o lisosomi. Lisati cellulari sono immunoprecipitati utilizzando un agarosio coniugata con anticorpi anti-myc seguita da analisi Western Blot. Individuare e quantificare le MycKISS1R su macchine viene eseguita utilizzando il LI-COR sistema a infrarossi Odyssey. Questo approccio può essere utile nello studio della degradazione di altre proteine di interesse pure.
Protocollo
1. Mutagenesi sito-diretta di GC altamente ricca di sequenza genetica KISS1R
- Template: completa sequenza cDNA della KISS1R umano con un Myc-tag fuso al suo N-terminale. Questa sequenza è clonato nel vettore di espressione pCS2 +, che è compatibile con le linee cellulari di mammiferi successivamente utilizzati per trasfezioni. Questo vettore di espressione è qui denominati pCS2 + Myc KISS1R.
- Primer design: primer sono destinati a trasportare le mutazioni si desidera, secondo le istruzioni del QuikChange mutagenesi sito-diretta kit (Stratagene). In sintesi:
- Entrambi (avanti e indietro) primer deve contenere la mutazione desiderata e annealing per la stessa sequenza di filamenti opposti del plasmide (ad esempio, in avanti e reverse primer sono complementari gli uni agli altri)
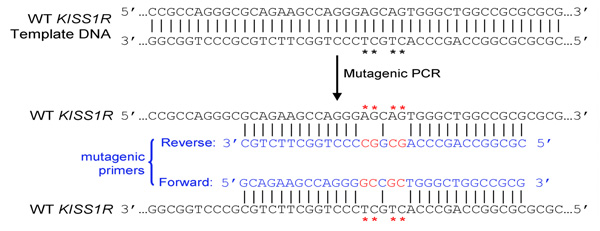
- Primers dovrebbero essere 25-45 basi lungo e alla fine in uno o più C o basi G
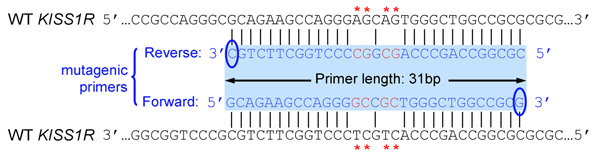

- Mutazione introdotta (s) deve essere al centro di primer e affiancato da ~ 10-15 basi della sequenza corretta da entrambe le parti
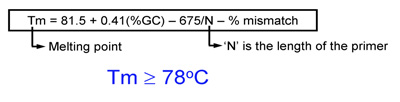
- Temperatura di fusione (Tm) dei primer deve essere uguale o maggiore di 78 ° C. Utilizzare le seguenti formule per stimare Tm:
- Quando l'introduzione di mutazioni: Tm = 81,5 + 0,41 (% GC) - 675 / N - mancata corrispondenza% (N è la lunghezza in fondo basi)
- Quando l'introduzione di inserzioni o delezioni: Tm = 81,5 + 0,41 (% GC) - 675 / N (N non include le basi che vengono inseriti o eliminati)
- Usa primer dissalati (senza ulteriori purificazioni necessario).
- Entrambi (avanti e indietro) primer deve contenere la mutazione desiderata e annealing per la stessa sequenza di filamenti opposti del plasmide (ad esempio, in avanti e reverse primer sono complementari gli uni agli altri)
- Le migliori condizioni di amplificazione includono l'aggiunta di PCRx Enhancer soluzione (Invitrogen) e DMSO. Almeno 2 concentrazioni di DMSO, come ad esempio il 4% e l'8%, devono essere testati. Condizioni di PCR sono riportati nella tabella I e nella Figura 1, è mostrato e risultati di un rappresentante amplificazione PCR di pCS2 + MycKISS1R nella Figura 2.
- Eliminare il DNA dei genitori sui prodotti di amplificazione dalla digestione del DNA metilato con DpnI per 1h 30min a 37 ° C: mescolare in una centrifuga 17.5μl tubo di prodotto PCR con 2μl di NEBuffer 10x 4 e 0.5μl di DpnI (10U; New England Biolabs).
- Trasforma DpnI trattati con prodotti di PCR: 2μl mix di DpnI trattati DNA con 45μl di XL10-Gold Ultracompetent E. coli (Stratagene) nel pre-raffreddata tubo 15 ml di coltura cellulare. Aggiungi 2μl di β-mercaptoetanolo e seguire le istruzioni Stratagene. Piastra 100-400μl di batteri trasformati in LB-agar in piastre contenenti ampicillina 100μg/ml e incubare a 37 ° C.
- Miniprep 2-4 singole colonie per isolare il DNA plasmidico. Confermare il successo delle mutazioni voluto dal sequenziamento e l'analisi del DNA isolata.
2. Transitoria di MycKISS1R in cellule HEK-293
- Gli esperimenti che seguono sono eseguiti in cellule di rene embrionale (HEK-293) coltivate in un incubatore CO 2 (5% CO 2) a 37 ° C in DMEM supplementato con 10% siero fetale bovino (FBS) e 1% di penicillina / streptomicina.
- Seme HEK-293 cellule a 2.5x10 5 cellule / ml in 6 pozzetti e farli crescere overnight a 37 ° C prima di trasfezione. NOTA: (i) utilizzare i pozzi tripla per ogni condizione sperimentale, (ii) confluenza delle cellule ideale al momento della transfezione è del 30% -50%.
- Trasfezione delle cellule HEK-293 utilizzando il reagente Transfection GenePorter (Genlantis), secondo le istruzioni del produttore: la metà Mix del siero senza DMEM con 0.5μg pCS2 + MycKISS1R più controllo 0.5μg (vuoto) vettore di totalizzare 1μg di DNA / bene. Mescolare l'altra metà con reagenti di trasfezione 10μl per mg di DNA transfettate. NOTA: Ideale concentrazione di DNA plasmidico può variare.
3. Trattamento con le cellule e lisi
- 24h dopo la transfezione, sostituire medio delle cellule con 1 ml di DMEM contenente 2,5% FBS (per diminuire il metabolismo cellulare). NOTA: Questa diminuzione nel siero possono facilitare e / o amplificare la rilevazione dei risultati.
- Aggiungi inibitore lisosomi (100μg / pozzetto di leupeptina) direttamente in ciascun pozzetto di una intera 6-pozzetti. Incubare a 37 ° C per 6 o 16 ore (o di altri tempi desiderati)
- Aggiungi fresco inibitore del proteasoma preparato (10μM / pozzetto di MG132) direttamente in tutti i pozzetti di due interi a 6 pozzetti. Incubare a 37 ° C per 2, 4, 6 o 16h (o momenti desiderati). Aggiungi veicolo a tutti i pozzetti del quarto a 6 pozzetti (0 time-point) e incubare a 37 ° C per 16h
- Quando l'incubazione è finita, spostare le piastre di ghiaccio e di eseguire questa procedura intera lisi sul ghiaccio per evitare la degradazione delle proteine:
- Per aumentare la resa di proteine, combinare la triplica il 6-pozzetti in un centr singoloifuge tubo
- Aspirare celle medie e lavare una volta con 1 ml di ghiacciata tampone fosfato (PBS)
- Aggiungere 100μl di ghiaccio-freddo buffer di lisi (HEPES 20 mM, pH 7.4, 1% NP-40, 150mm NaCl, 1mM EDTA, sodio desossicolato 0,25%) contenenti gli inibitori della proteasi (1x cocktail contenente 100mM AEBSF-HCl, 80μM aprotinina, bestatin 5mm, 1,5 E-64, 0.5M EDTA, 2mM leupeptina e 1mM pepstatina A, più 2mM PMSF) in ciascun pozzetto
- Rimuovere le cellule con un raschietto cellula e cellula trasferimento lisati di centrifugare tubi
- Passo cellule ~ 10 volte attraverso una 20-gauge. NOTA: Non sonicare campioni destinati per il rilevamento western blot di proteine di membrana. Sonicazione porta all'aggregazione delle proteine di membrana, che non migrano correttamente durante l'elettroforesi
- Incubare lisati cellulari per 1h a 4 ° C su una piattaforma oscillante
- Lisati cellulari centrifuga a 4 ° C per 10 min a 10.000 giri x surnatanti e trasferimento in nuovi tubi. NOTA: Non disturbare pellet durante questa fase
- Determinare la concentrazione di proteine in 10μl di sovranatante con il metodo BCA (Pierce)
- Diluire lisati di 1mg/ml con buffer di lisi contenente inibitori della proteasi.
4. Immunoprecipitazione e macchia occidentale di rilevamento MycKISS1R
- Eseguire la procedura seguente immunoprecipitazione su ghiaccio (oppure a 4 ° C):
- Lavare la giusta quantità di agarosio coniugata con anticorpi anti-myc (2.5μg / campione), due volte con PBS freddo ghiaccio e aggiungere a questo 400μg di lisato proteico.
- Immunoprecipitato il MycKISS1R su lisati notte a 4 ° C in un oscillante e scuotendo con cautela.
- Spin down perle agarosio per centrifugazione impulso a 4 ° C (fino a 10.000 x rpm)
- Aspirare e scartare surnatanti (senza disturbare il pellet)
- Lavare una volta con perline gelida tampone di lisi e due volte con PBS freddo ghiaccio. Invertire i tubi delicatamente prima filatura
- Perline Ressuspend contenenti anticorpi legati MycKISS1R nel buffer 2x caricamento dei campioni contenenti 10% di β-mercaptoetanolo.
- Western Blot di immunocomplessi MycKISS1R:
- Campioni di calore per 30 minuti a 37 ° C. NOTA: non bollire campioni destinati per il rilevamento western blot di proteine di membrana. Come sonicazione, bollente porta anche ad aggregazione di queste proteine
- Spostare i tubi immediatamente in ghiaccio per 5 minuti
- Proteine separate mediante SDS-PAGE in un gel di pendenza 4-15%.
- Trasferimento a Immobilon-FL membrana PVDF (per infrarossi di rilevamento) a 25V per 30 min in tampone di trasferimento (Tris 48 mm di base, glicina 39mm, 1,2 mm SDS, 20% metanolo, pH 9.2) con il Bio-Rad Semi-Dry trasferimento apparato
- Lavare le membrane per 5 minuti a temperatura ambiente con Tris-Buffered Saline (TBS) e blocco per 1 ora a temperatura ambiente con Odissea Licor blocco su una piattaforma oscillante (in alternativa, 5% latte in TBS può essere utilizzato per bloccare legame non specifico).
- Le membrane incubare una notte a 4 ° C con coniglio anti-myc anticorpi (1:500) in soluzione di blocco contenente 0,1% Tween-20
- Rimuovere l'anticorpo primario e lavare le membrane 3 volte su 5 minuti ciascuno con TBS contenente 0,1% di Tween-20 (TBST)
- Incubare membrane per 1h a temperatura ambiente con caprino Infra-RedDye ® 800CW marcato anti-IgG di coniglio (1:10.000) in tampone bloccante contenente 0,1% di Tween-20 e 0,01% SDS
- Rimuovere l'anticorpo secondario, lavare le membrane 3 volte su 5 minuti ciascuno con TBST e l'ultima volta con TBS solo (per rimuovere rimanenti Tween-20)
- Imaging e quantificazione dei MycKISS1R utilizzando il LI-COR Odyssey Infra-Red Imager:
- Il MycKISS1R sulle membrane sarà ripreso con il LI-COR Odyssey Infra-Red Imager. Per iniziare, posto sulla membrana in basso a sinistra dello scanner Odyssey, allineandolo con la rete. Coprire con il tappetino in gomma, appianare le bolle con rullo e chiudere il coperchio
- Creare un nuovo file di progetto sul computer. Nome del file, fare clic su "fatto", quindi inserire il login scanner su "scan". Dimensioni della finestra di console di scanner per soddisfare le vostre membrana, e poi scegliere la risoluzione 169μm e qualità d'immagine media
- Scegliere le impostazioni di intensità per 700 (rosso) e 800 (verde) i canali in base alla forza atteso di ciascun segnale. Sia a scopo solo la visualizzazione del segnale e non influenza quantificazione. Fare clic su "inizio scansione"
- Nome e salvare la scansione, quindi fare clic su "OK" per aprirlo in una nuova finestra per la quantificazione. Monomeri MycKISS1R dovrebbe essere visibile a circa 43kD
- Usando lo strumento "scatola" (sulla barra laterale sinistra), disegnare un riquadro attorno alla prima band. Trascinare la casella intorno per assicurarsi che tutte le bande di adattarsi, poi "copia" e "incolla" la scatola tutte le bande
- Selezionare tutte le caselle con "Ctrl + A", e quindi selezionare l'opzione per sottrarre sfondo mediana. Fare clic su "report" nel menu in alto e un foglio di calcolo con i valori quantificazione apparirà. Risultati rappresentativi riportati sono represented come fold-aumento rispetto cellule non trattate (tempo zero)
5. Rappresentante dei risultati:
- Mutagenesi sito-diretta di sequenze altamente KISS1R GC ricchi gene:
Tabella 1. Mostra la combinazione di reagenti per migliorare l'efficienza di amplificazione KISS1R. Questa combinazione è stata utilizzata con successo per introdurre mutazioni diverse diretto contro le regioni distinte della sequenza KISS1R cDNA, così come per l'amplificazione di questo gene altamente GC-ricchi. La figura 1 mostra le condizioni di ciclismo e di amplificazione per la mutagenesi di KISS1R. Queste condizioni sono state modificate dal QuikChange mutagenesi sito-diretta kit (Stratagene).
Figura 2. Evidenzia un risultato rappresentativo utilizzando questo protocollo ottimizzato. L'aggiunta del 4% o 8% DMSO combinata con la Enhancer PCRx migliora in modo significativo resa di amplificazione del GC ricchi KISS1R cDNA contenenti plasmide. In questo esperimento di rappresentanza, 4% DMSO fornito le condizioni di amplificazione leggermente migliore rispetto a 8% DMSO. Trasformazione di DpnI trattati con prodotti di amplificazione ultracompetent E. coli produce tipicamente da 100 a oltre 1.000 colonie, e il tasso di successo dell'introduzione mutazioni desiderato 80-90% come determinato dal sequenziamento del DNA. - L'uso di costrutti MycKISS1R a studiare fisiologia del recettore:
In questo esperimento rappresentativo, di tipo selvatico pCS2 + MycKISS1R amplificato secondo il protocollo ottimizzato qui descritto è usato per studiare in vivo KISS1 degradazione del recettore in una linea cellulare rilevante (HEK-293) transitoriamente esprimere MycKISS1R. Dopo aver trattato la trasfezione delle cellule HEK-293 con lisosomi (leupeptina) o proteasoma (MG132 inibitori) secondo il protocollo descritto nei metodi, le cellule sono lisate e trattati per western blot. Il pannello superiore della figura 3 mostra la scansione di monomeri MycKISS1R mentre il pannello in basso mostra la quantificazione delle bande visualizzato sul pannello superiore. Quantificazione delle bande indica che né né 6h 16h di trattamento leupeptina colpite livelli di proteine MycKISS1R. Al contrario, il trattamento con MG132 portato ad un tempo-dipendente aumento della proteina MycKISS1R in queste cellule, che culmina a 45 volte l'accumulo del recettore dopo 16h di incubazione con MG132. Queste osservazioni indicano che, a differenza della maggior parte dei G recettori accoppiati alla proteina, KISS1R è degradato dal proteasoma (piuttosto che i lisosomi).
| DMSO | |||
| Reagenti | 0 | 4% | 8% |
| Acqua | 35 | 33 | 31 |
| Pfu 10x Ultra Taq buffer di | 5 | 5 | 5 |
| dNTP mix (10mm) | 1 | 1 | 1 |
| Primer senso (25pmol/μl) | 1 | 1 | 1 |
| Primer antisenso (25pmol/μl) | 1 | 1 | 1 |
| 10x PCRx Enhancer Solution | 5 | 5 | 5 |
| DMSO | 0 | 2 | 4 |
| Pfu Ultra Taq polimerasi (2.5U/μl) | 1 | 1 | 1 |
| DNA plasmidico (20ng/μl) | 1 | 1 | 1 |
Tabella 1. Combinazione di reagenti utilizzati con successo per mutare e amplificare il GC-ricchi KISS1R
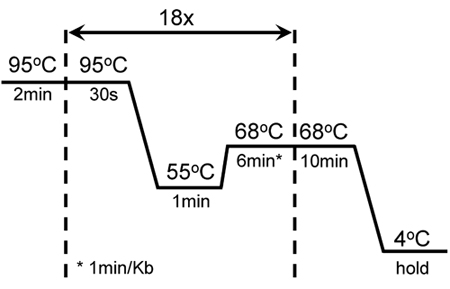
Figura 1. Condizioni di ciclismo di mutagenesi successo e amplificazione di GC ricchi KISS1R: Un inizio 2min a caldo è stata seguita da 18 cicli di 30 sec di fusione a 95 ° C, 1 min annealing a 55 ° C min estensione e 6 a 68 ° C. Un ulteriore 10 min estensione a 68 ° C è stata aggiunta alla fine del ciclo. Queste impostazioni sono stati adeguati dal protocollo di mutagenesi originale del II QuikChange XL-mutagenesi sito-diretta kit (Stratagene).
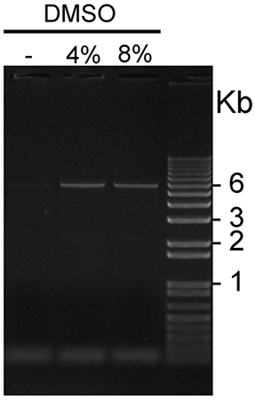
Figura 2. Visualizzazione di GC ricchi pCS2 + Myc KISS1R amplificato in presenza o in assenza di DMSO: Cinque aliquote l di pCS2 + KISS1R Myc amplificato in presenza di 0, 4 o 8% DMSO sono stati caricati in questa rappresentativa 1% gel colorati con etidio bromuro e visualizzati utilizzando raggi UV. Le bande di plasmide 6Kb sono visibili su entrambe le corsie di carico di prodotti della PCR amplificati in presenza del 4% e l'8% DMSO, ma non sulla corsia di prima, che è stato caricato con una PCRprodotto amplificato in assenza di DMSO.
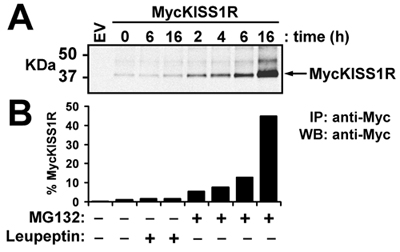
Figura 3. Effetto della leupeptina o MG132 sui livelli di proteina Myc KISS1R in cellule HEK-293: HEK-293 cellule che esprimono Myc KISS1R sono stati trattati con 100μg/ml leupeptina o 10μM MG132 a 37 ° C per i tempi designato. Myc KISS1R su 400μg di lisato cellulare è stata immunoprecipitati con 2.5μg di agarosio coniugata con anticorpi anti-myc e analizzati mediante western blot. (A) LI-COR rilevazione Odissea di KISS1R Myc dopo incubazione di immunoblot con coniglio anti-Myc-tag anticorpi seguito da incubazione con IRDye 800CW marcato anti-coniglio; (B) Quantificazione delle bande KISS1R Myc mostrato in (A) utilizzando il LI-COR Odissea software di quantificazione. I risultati sono rappresentati come fold-aumento rispetto cellule non trattate (tempo 0).
Discussione
Mutagenesi sito-diretta è stata utilizzata per studiare la funzione delle proteine con l'introduzione di cambiamenti nucleotide nella sequenza codificante del gene targeting per oltre tre decenni. La tecnica originale è stata descritta nel 1978 dal chimico britannico-canadese e Premio Nobel Michael Smith 10. Michael Smith condiviso il Premio Nobel 1993 per la chimica Kary Mullis, con il biochimico americano che ha inventato la reazione a catena della polimerasi (PCR) 11. Il metodo origi...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato parzialmente finanziato dalla Filiale di riproduzione del National Institute of Child Health e lo Sviluppo Umano (NICHD - R21 HD059015) e dalla Charles H. Hood Fondazione Premio Giovani Bambino Investigator Research Salute (Boston, MA).
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | |
| 10x PCRx Enhancer Solution | Invitrogen | 52391 | |
| PfuUltra ad alta fedeltà DNA polimerasi detergente alternativo | Stratagene | 600385 | |
| DPN-I | New England Biolabs | R0176 | |
| XL10-Gold E. Ultracompetent coli cellule | Stratagene | 200314 | |
| DMEM | Cellgro | 10-013-CV | |
| Siero fetale bovino | Atlanta Biologicals | S11550 | |
| Geneporter Transfection Reagent | Genlantis | T201007 | |
| Leupeptina | Calbiochem | 108975 | |
| MG132 | Calbiochem | 47491 | |
| 10xPBS | Ambion | AM9625 | |
| Inibitori della proteasi cocktail e PMSF | Santa Cruz Biotechnology | Sc-24948 | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| anti-Myc tag (clone 4A6) agarosio coniugato | Millipore | 16-219 | |
| 2x tampone di caricamento | BioRad | 161-0737 | |
| Criterio di Tris-HCl prefabbricati gel, 4-15% di pendenza | BioRad | 345-0028 | |
| Immobilon-FL membrana PVDF | Millipore | IPFL00010 | |
| Odissea tampone di bloccaggio | LI-COR Biosciences | 927-40000 | |
| 10xTBS | BioRad | 170-6435 | |
| Coniglio anti-myc anticorpi | Segnalazione cellulare | 2272 | |
| Capra anti-coniglio IRDye 800CW | LI-COR Biosciences | 926-32211 | |
| Odissea Imaging System infrarossi | LI-COR Biosciences | ||
| Halt inibitore della proteasi Cocktail (100x) | Thermo Scientific | 78430 |
Riferimenti
- Seminara, S. B., Messager, S., Chatzidaki, E. E. The GPR54 Gene as a Regulator of Puberty. N Engl J Med. 349, 1614-1627 (2003).
- de Roux, N., Genin, E., Carel, J. C. Hypogonadotropic Hypogonadism Due to Loss of Function of the KiSS1-Derived Peptide Receptor GPR54. Proc Natl Acad Sci U S A. 100, 10972-10976 (2003).
- Messager, S., Chatzidaki, E. E., Ma, D. Kisspeptin Directly Stimulates Gonadotropin-Releasing Hormone Release via G Protein-Coupled Receptor 54. Proc Natl Acad Sci U S A. 102, 1761-1766 (2005).
- Teles, M. G., Bianco, S. D., Brito, V. N. A GPR54-Activating Mutation in a Patient with Central Precocious Puberty. N Engl J Med. 358, 709-715 (2008).
- Tenenbaum-Rakover, Y., Commenges-Ducos, M., Iovane, A. Neuroendocrine Phenotype Analysis in Five Patients with Isolated Hypogonadotropic Hypogonadism due to a L102P Inactivating Mutation of GPR54. J Clin Endocrinol Metab. 92, 1137-1144 (2007).
- Semple, R. K., Achermann, J. C., Ellery, J. Two Novel Missense Mutations in G Protein-Coupled Receptor 54 in a Patient with Hypogonadotropic Hypogonadism. J Clin Endocrinol Metab. 90, 1849-1855 (2005).
- Wacker, J. L., Feller, D. B., Tang, X. B. Disease-Causing Mutation in GPR54 Reveals the Importance of the Second Intracellular Loop for Class A G-Protein-Coupled Receptor Function. J Biol Chem. 283, 31068-31078 (2008).
- Szereszewski, J. M., Pampillo, M., Ahow, M. R. GPR54 regulates ERK1/2 activity and hypothalamic gene expression in a Galpha(q/11) and beta-arrestin-dependent manner. PLoS One. 5, e12964-e12964 (2010).
- Bianco, S. D. C., Vandepas, L., Correa-Medina, M., Gereben, B., Mukherjee, A., Kuohung, W., Carroll, R., Teles, M. G., Latronico, A. C., Kaiser, U. B. KISS1R Intracellular Trafficking and Degradation: Effect of the Arg386Pro Disease-Associated Mutation. Endocrinology. , (2011).
- Hutchison, C. A., Phillips, S., Edgell, M. H. Mutagenesis at a Specific Position in a DNA Sequence. J Biol Chem. 253, 6551-6560 (1978).
- Mullis, K. B. The Unusual Origin of the Polymerase Chain Reaction. Sci Am. 262, 56-61 (1990).
- Henke, W., Herdel, K., Jung, K., Schnorr, D., Loening, S. A. Betaine improves the PCR amplification of GC-rich DNA sequences. Nucleic Acids Res. 25, 3957-3958 (1997).
- Sahdev, S., Saini, S., Tiwari, P., Saxena, S., Singh Saini, K. Amplification of GC-rich genes by following a combination strategy of primer design, enhancers and modified PCR cycle conditions. Mol Cell Probes. 21, 303-307 (2007).
- Kong, K. C., Poyner, D. R., Wheatly, M. Chapter 10. MSTA. in G Protein-Coupled Receptors: Essential Methods. , 197-204 (2010).
- Hall, R. A., George, S. R., O'Dowd, B. F. Chapter 9. G protein-Coupled Receptor-Protein Interactions. , 170-171 (2005).
- Chaturvedi, K., Bandari, P., Chinen, N., Howells, R. D. Proteasome Involvement in Agonist-Induced Down-Regulation of Mu and Delta Opioid Receptors. J Biol Chem. 276, 12345-12355 (2001).
- Shenoy, S. K., McDonald, P. H., Kohout, T. A., Lefkowitz, R. J. Regulation of Receptor Fate by Ubiquitination of Activated Beta 2-Adrenergic Receptor and Beta-Arrestin. Science. 294, 1307-1313 (2001).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon