Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mutagenèse et d'analyse des mutations génétiques dans la séquence riches en GC récepteur KISS1 identifiés chez l'homme souffrant de troubles de la reproduction
Dans cet article
Résumé
Des mutations dans le récepteur kisspeptine (KISS1R) sont associés à des troubles de la reproduction chez les patients. Nous décrivons ici comment introduire des mutations d'intérêt dans la séquence riches en GC d'KISS1R ainsi que l'utilisation de constructions KISS1R de caractériser la voie de dégradation du récepteur par immunoprécipitation et Western blot
Résumé
Le récepteur kisspeptine (KISS1R) est un G récepteurs couplés aux protéines reconnu comme l'élément déclencheur de la puberté et un régulateur de la compétence de reproduction à l'âge adulte 1,2,3. Mutations d'inactivation dans KISS1R identifiées chez des patients ont été associés à un hypogonadisme hypogonadotrophique iodiopathic (IHH) 1,2 et une puberté précoce 4. Les études fonctionnelles de ces mutants sont cruciaux pour notre compréhension des mécanismes impliqués dans la régulation de la reproduction par ce récepteur ainsi que ceux qui façonnent les résultats des maladies qui résultent de la signalisation KISS1R anormale et la fonction. Cependant, la séquence très riches en GC du gène KISS1R rend plutôt difficile d'introduire ou d'amplifier les mutations du gène codant pour ce récepteur par PCR.
Nous décrivons ici une méthode pour introduire des mutations de l'intérêt dans cette séquence très riches en GC qui a été utilisée avec succès pour générer plus d'un douzaine de mutants KISS1R dans notre laboratoire. Nous avons optimisé les conditions de PCR afin de faciliter l'amplification d'une gamme de mutants KISS1R qui incluent substitutions, délétions ou des insertions dans la séquence KISS1R. L'ajout d'une solution de PCR Enhancer, ainsi que d'un faible pourcentage de DMSO ont été particulièrement utiles pour améliorer l'amplification. Cette procédure optimisée peut être utile pour d'autres riches en GC modèles ainsi.
Le vecteur d'expression codant pour la KISS1R est été utilisée pour caractériser la signalisation et la fonction de ce récepteur afin de comprendre comment les mutations peuvent modifier KISS1R fonction et conduire à la reproduction phénotypes associés. En conséquence, les applications potentielles de mutants générés par KISS1R mutagenèse dirigée peut être illustrée par de nombreuses études 1,4,5,6,7,8. À titre d'exemple, la mutation gain-de-fonction dans le KISS1R (Arg386Pro), qui est associée à la puberté précoce, a montré une prolongation de la réactivité du récepteur à 4 stimulation de ligand ainsi que de modifier le taux de dégradation des KISS1R 9 . Fait intéressant, nos études indiquent que KISS1R est dégradée par le protéasome, par opposition à la dégradation lysosomale classiques décrites pour la plupart G récepteurs couplés aux protéines 9. Dans l'exemple présenté ici, la dégradation de l'KISS1R est étudiée dans cellules rénales embryonnaires humaines (HEK-293) exprimant de manière transitoire Myc-taggés KISS1R (MycKISS1R) et traités avec des inhibiteurs du protéasome ou lysosome. Les lysats cellulaires sont immunoprécipitées en utilisant une agarose conjugué d'anticorps anti-myc suivie par une analyse Western blot. Détection et quantification des MycKISS1R sur des transferts est effectuée en utilisant le système Odyssey infrarouge LI-COR. Cette approche peut être utile dans l'étude de la dégradation d'autres protéines d'intérêt aussi bien.
Protocole
1. La mutagenèse dirigée de la séquence de gène très riches en GC KISS1R
- Modèle: pleine séquence d'ADNc de l'humain avec une KISS1R Myc-étiquette fusionnée à son extrémité N-terminale. Cette séquence est clonée dans le vecteur d'expression pCS2 +, qui est compatible avec les lignées cellulaires de mammifères utilisée ensuite pour transfections. Ce vecteur d'expression est appelé aux présentes pCS2 KISS1R + Myc.
- Conception d'amorce: amorces sont conçus pour transporter des mutations souhaitées, conformément aux instructions de l'QuikChange kit de mutagenèse dirigée (Stratagene). En résumé:
- Les deux amorces (marche avant et arrière) doit contenir la mutation désirée et recuit à la même séquence sur les brins opposés du plasmide (par exemple avant et arrière amorces sont complémentaires les unes aux autres)
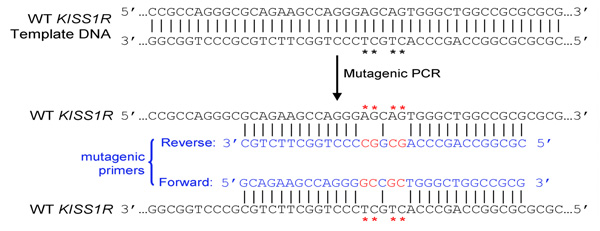
- Primaires devrait être de 25 à 45 bases de long et fin en une ou plusieurs bases de C ou G
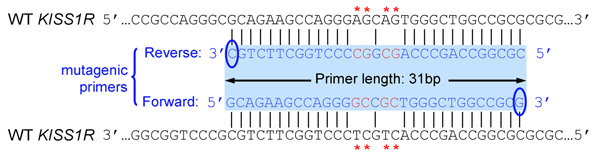

- Mutation introduite (s) doit être au milieu d'apprêt et flanqué de ~ 10-15 bases de la séquence correcte des deux côtés
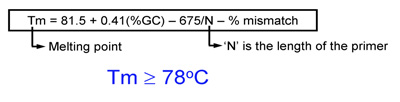
- La température de fusion (Tm) des amorces doit être égale ou supérieure à 78 ° C. Utilisez les formules suivantes pour estimer Tm:
- Lors de l'introduction de mutations: Tm = 81,5 + 0,41 (% GC) - 675 / N - discordance% (N est la longueur d'amorce dans des bases)
- Lors de l'introduction insertions ou délétions: Tm = 81,5 + 0,41 (% GC) - 675 / N (N n'inclut pas les bases qui sont insérés ou supprimés)
- Utilisez amorces dessalé (pas de purifications supplémentaires nécessaires).
- Les deux amorces (marche avant et arrière) doit contenir la mutation désirée et recuit à la même séquence sur les brins opposés du plasmide (par exemple avant et arrière amorces sont complémentaires les unes aux autres)
- Les meilleures conditions d'amplification comprennent l'ajout d'PCRx Enhancer solution (Invitrogen) et le DMSO. Au moins deux concentrations de DMSO, tel que 4% et 8%, devrait être testé. Les conditions de PCR sont indiquées dans le tableau I et dans la figure 1, et les résultats d'une amplification par PCR de représentant pCS2 + MycKISS1R est montré dans la figure 2.
- Éliminer l'ADN des parents sur les produits d'amplification par la digestion de l'ADN méthylé avec Dpnl pour 1h 30min à 37 ° C: mélanger dans un tube à centrifuger 17.5μl du produit PCR avec des 2μl NEBuffer 10x 4 et 0.5μl des Dpnl (10U; New England Biolabs).
- Transformez Dpnl-traitée produit PCR: 2μl mélange de Dpnl traités ADN avec 45μl d'XL10-Gold Ultracompetent E. coli (Stratagene) en pré-refroidi 15 ml tube de culture cellulaire. Ajouter 2μl du β-mercaptoéthanol et suivez les instructions Stratagene. Plate 100-400μl de bactéries transformées en LB-agar contenant 100μg/ml ampicilline et incuber à 37 ° C.
- Miniprep 2-4 colonies individuelles pour isoler l'ADN plasmidique. Confirmer l'introduction réussie des mutations souhaitées par le séquençage et l'analyse de l'ADN isolé.
2. Transfection transitoire de MycKISS1R en cellules HEK-293
- Les expériences suivantes sont effectuées dans des cellules humaines embryonnaires de rein (HEK-293) en culture dans un incubateur à CO 2 (5% de CO 2) à 37 ° C dans du DMEM supplémenté avec 10% sérum de veau fœtal (FBS) et 1% de pénicilline / streptomycine.
- Graine cellules HEK-293 au 2.5x10 5 cellules / ml dans des plaques 6 puits et de les laisser pousser une nuit à 37 ° C avant la transfection. NOTE: (i) l'utilisation des puits en triple pour chaque condition expérimentale, (ii) la confluence des cellules idéale au moment de la transfection est 30% -50%.
- Transfecter cellules HEK-293 utilisant le réactif de transfection GenePorter (Genlantis), selon les instructions du fabricant: Mélanger la moitié de l'DMEM sans sérum avec 0.5μg pCS2 + MycKISS1R ainsi que le contrôle 0.5μg (vide) vecteur pour totaliser 1 pg d'ADN / puits. Mélanger l'autre moitié avec 10 ul réactif de transfection par pg d'ADN transfecté. NOTE: Idéal de concentration d'ADN plasmidique peut varier.
3. Traitement des cellules et la lyse
- 24h après transfection, remplacez milieu cellulaire avec 1ml DMEM contenant 2,5% de FBS (pour diminuer le métabolisme cellulaire). NOTE: Cette diminution dans le sérum peut faciliter et / ou d'amplifier la détection de résultats.
- Ajouter inhibiteur de lysosome (100 ug / puits de leupeptine) directement dans chaque puits d'un ensemble de 6 puits assiette. Incuber à 37 ° C pendant 6 ou 16 h (ou un autre moment souhaité)
- Ajouter fraîchement préparés inhibiteur de protéasome (10 uM / puits de MG132) directement dans tous les puits de deux ensemble des plaques 6 puits. Incuber à 37 ° C pendant 2, 4, 6 ou 16h (ou parfois souhaité). Ajouter des véhicules à tous les puits de la quatrième à 6 puits plaque (0 point dans le temps) et incuber à 37 ° C pendant 16h
- Quand l'incubation est terminée, déplacer les plaques de glace et d'effectuer cette procédure de lyse ensemble sur la glace pour empêcher la dégradation des protéines:
- Pour augmenter le rendement en protéines, de combiner les trois exemplaires sur les plaques de 6 puits dans un centr seuleTube ifuge
- Aspirer et laver les cellules moyenne une fois avec 1 ml d'glacée tampon phosphate salin (PBS)
- Ajouter 100 pi de tampon glacé de lyse (20 mM HEPES, pH 7,4, 1% NP-40, 150 mM NaCl, 1 mM EDTA, le désoxycholate de sodium à 0,25%) contenant des inhibiteurs de protéase (1x cocktail contenant 100 mM AEBSF-HCl, 80μM aprotinine, bestatine 5mM, E-64 1,5 mM, EDTA 0,5 M, 2 mM de leupeptine et 1 mM de pepstatine A, plus 2 mM PMSF) dans chaque puits
- Retirer les cellules avec un grattoir cellulaire et le transfert de lysats de centrifuger les tubes
- Passez les cellules ~ 10 fois par une aiguille de calibre 20. REMARQUE: Ne pas soniquer échantillons destinés à la détection par Western blot des protéines membranaires. Sonication conduit à l'agrégation des protéines membranaires, qui ne migrent pas correctement pendant l'électrophorèse
- Incuber lysats cellulaires pour 1h à 4 ° C sur une plate-forme à bascule
- Lysats cellulaires Centrifuger à 4 ° C pendant 10 min à 10000 rpm et les surnageants x transfert à de nouveaux tubes. REMARQUE: Ne pas déranger granulés lors de cette étape
- Déterminer la concentration de protéines dans 10 ul de surnageant en utilisant la méthode BCA (Pierce)
- Diluer lysats à 1mg/ml avec un tampon de lyse contenant des inhibiteurs de protéase.
4. Immunoprécipitation et Western blot de détection d'MycKISS1R
- Effectuez les étapes suivantes immunoprécipitation sur la glace (ou à 4 ° C):
- Laver la quantité appropriée d'agarose conjugué d'anticorps anti-myc (2.5μg / échantillon) à deux reprises avec PBS glacé et ajoutez ceci à 400μg de protéines lysat.
- Immunoprécipiter l'MycKISS1R sur des lysats nuit à 4 ° C dans une plate-forme à bascule avec une agitation douce.
- Isoler des billes d'agarose par centrifugation d'impulsion à 4 ° C (jusqu'à 10 000 x rpm)
- Aspirer et jeter surnageants (sans déranger les boulettes)
- Laver une fois avec des perles glacée tampon de lyse et deux fois avec PBS glacé. Inverser les tubes doucement avant de filer
- Perles Ressuspend contenant de l'anticorps lié au MycKISS1R tampon d'échantillon de chargement 2x contenant 10% de β-mercaptoéthanol.
- Western blot d'immunocomplexes MycKISS1R:
- Échantillons de chaleur pendant 30 min à 37 ° C. NOTE: Ne pas faire bouillir les échantillons destinés à la détection par Western blot des protéines membranaires. Comme sonication, ébullition conduit également à l'agrégation de ces protéines
- Déplacer les tubes immédiatement dans la glace pendant 5 min
- Séparer les protéines par SDS-PAGE sur un gel gradient 4-15%.
- Transfert à Immobilon-FL membrane de PVDF (pour la détection infra-rouge) à 25V pendant 30 min dans un tampon de transfert (Tris base 48mm, 39mm de glycine, 1,2 SDS, 20% de méthanol, pH 9,2) en utilisant le Bio-Rad semi-sec Transfert un appareil
- Laver les membranes pendant 5 min à température ambiante avec une solution saline tamponnée Tris (TBS) et le bloc pour 1h à température ambiante avec Odyssey Licor de blocage sur une plate-forme à bascule (alternativement, 5% de lait dans du TBS peut être utilisé pour bloquer la liaison non spécifique).
- Membranes Incuber une nuit à 4 ° C avec le lapin d'anticorps anti-myc (1:500) dans la solution de blocage contenant 0,1% de Tween-20
- Retirer anticorps primaire et lavez les membranes 3 fois de 5 min chacune avec du TBS contenant 0,1% de Tween-20 (TBST)
- Incuber les membranes pour 1h à température ambiante avec de chèvre Infra-RedDye ® 800CW-anticorps anti-IgG de lapin (1:10.000) en bloquant tampon contenant 0,1% de Tween-20 et 0,01% de SDS
- Retirer anticorps secondaire, se laver les membranes 3 fois de 5 min chacune avec TBST et une dernière fois avec le SCT seulement (pour enlever les Tween-20)
- Imagerie et quantification des MycKISS1R utilisant le LI-COR Odyssey imageur infra-rouge:
- Le MycKISS1R sur les membranes seront imagées par le LI-COR Odyssey imageur infra-rouge. Pour commencer, placez la membrane sur le coin inférieur gauche de scanner Odyssey, en l'alignant sur la grille. Couvrir avec le tapis de caoutchouc, lisser les bulles avec un rouleau et fermez le couvercle
- Créer un nouveau fichier de projet sur l'ordinateur. Nommez le fichier, cliquez sur «Terminé», puis entrez le login scanner sur "scan". Taille du boîtier de console scanner pour s'adapter à votre membrane, puis choisir la résolution de 169μm et une qualité d'image moyenne
- Choisissez les paramètres d'intensité pour 700 (rouge) et 800 (vert) des canaux selon la force attendue de chaque signal. C'est à des fins de visualisation du signal et ne seront pas d'influence sur la quantification. Cliquez sur "Start Scan"
- Nom et enregistrer la numérisation, puis cliquez sur "OK" pour l'ouvrir dans une nouvelle fenêtre pour la quantification. Monomères MycKISS1R devrait être visible à environ 43kD
- Utilisation de la «boîte» de l'outil (sur la barre latérale de gauche), tracez une boîte autour de la première bande. Faites glisser la boîte autour pour s'assurer que toutes les bandes s'intégrer, puis "copier" et "coller" la boîte sur toutes les bandes
- Sélectionnez toutes les cases en utilisant "Ctrl + A", puis sélectionnez l'option de soustraire de fond médiane. Cliquez sur "rapport" sur le menu en haut et d'un tableur avec les valeurs de quantification apparaît. Les résultats représentatifs sont présentés ici represented que rabattable augmenter au fil des cellules non traitées (temps zéro)
5. Les résultats représentatifs:
- La mutagenèse dirigée de la séquence du gène KISS1R très riches en GC:
Tableau 1. Montre la combinaison de réactifs pour améliorer l'efficacité de l'amplification KISS1R. Cette combinaison est utilisée avec succès pour introduire plusieurs mutations dirigées contre des régions distinctes de l'KISS1R séquence d'ADNc, ainsi que pour l'amplification de ce gène très riches en GC. La figure 1 montre les conditions de cyclisme et l'amplification de la mutagenèse de KISS1R. Ces conditions ont été modifiées par l'QuikChange kit de mutagenèse dirigée (Stratagene).
Figure 2. Montre un résultat représentatif en utilisant ce protocole optimisé. L'ajout de 4% ou 8% de DMSO combiné avec l'Enhancer améliore considérablement le rendement PCRx d'amplification de la GC riche KISS1R ADNc contenant le plasmide. Dans cette expérience représentative, le DMSO 4% prévu conditions d'amplification légèrement meilleur par rapport à 8% de DMSO. Transformation des produits d'amplification Dpnl-traitées avec ultracompetent E. coli donne généralement de 100 à plus de 1000 colonies, et le taux d'introduction réussie des mutations souhaitées est de 80-90% déterminée par séquençage de l'ADN. - Utiliser des constructions MycKISS1R pour étudier la physiologie des récepteurs:
Dans cette expérience représentative, le type sauvage pCS2 + MycKISS1R amplifié selon le protocole optimisé décrite ici est utilisée pour étudier la dégradation in vivo des récepteurs KISS1 dans une lignée cellulaire pertinents (HEK-293) exprimant de manière transitoire MycKISS1R. Après le traitement des transfectées cellules HEK-293 avec les lysosomes (leupeptine) ou du protéasome (MG132) inhibiteurs selon le protocole décrit dans les méthodes, les cellules sont lysées et traitées pour western blot. Le panneau supérieur de la figure 3 montre le scan de monomères MycKISS1R tandis que le panneau du bas montre la quantification des bandes indiquées sur le panneau supérieur. La quantification des bandes indique que ni ni 6h 16h de traitement leupeptine affecté les niveaux de protéines MycKISS1R. Inversement, le traitement avec MG132 a entraîné une augmentation dépendant du temps en protéines MycKISS1R dans ces cellules, qui culmine avec une accumulation de 45 fois du récepteur après 16h d'incubation avec MG132. Ces observations indiquent que, contrairement à la plupart G récepteurs couplés aux protéines, KISS1R est dégradée par le protéasome (plutôt que le lysosome).
| DMSO | |||
| Réactifs | 0 | 4% | 8% |
| Eau | 35 | 33 | 31 |
| 10x Pfu Ultra tampon Taq | 5 | 5 | 5 |
| mélange de dNTP (10 mM) | 1 | 1 | 1 |
| Sens Primer (25pmol/μl) | 1 | 1 | 1 |
| Antisens Primer (25pmol/μl) | 1 | 1 | 1 |
| 10x PCRx Enhancer Solution | 5 | 5 | 5 |
| DMSO | 0 | 2 | 4 |
| Pfu Ultra Taq polymérase (2.5U/μl) | 1 | 1 | 1 |
| ADN plasmidique (20ng/μl) | 1 | 1 | 1 |
Tableau 1. Combinaison de réactifs utilisés avec succès à muter et à amplifier les KISS1R riches en GC
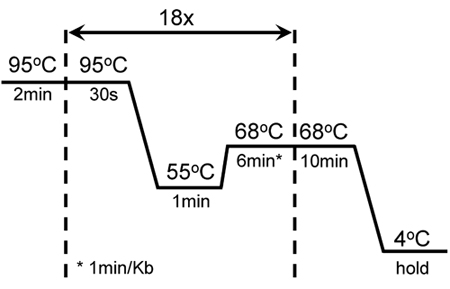
Figure 1. Cyclisme conditions de la mutagenèse et l'amplification du succès riches en GC KISS1R: Un début 2min chaude a été suivie par 18 cycles de 30 sec fondre à 95 ° C; 1 min de recuit à 55 ° d'extension C et 6 min à 68 ° C. Un supplément de 10 min d'extension à 68 ° C a été ajouté à la fin du dernier cycle. Ces paramètres ont été ajustés par rapport au protocole original de la mutagenèse QuikChange II kit XL-mutagénèse dirigée (Stratagene).
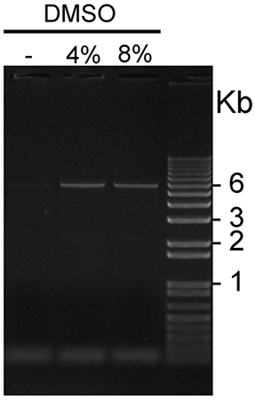
Figure 2. Visualisation des riches en GC pCS2 + Myc KISS1R amplifié en présence ou en absence de DMSO: Cinq aliquotes des pCS2 + KISS1R Myc amplifié en présence de 0, 4 ou 8% de DMSO ont été chargés dans ce représentant de gel d'agarose 1% coloré avec éthidium bromure et visualisés en utilisant la lumière UV. Les bandes de plasmide de 6Kb sont visibles sur les deux voies chargées avec des produits de PCR amplifié en présence de 4% et 8% de DMSO, mais pas sur la première voie, qui a été chargé avec une PCRproduit amplifié en l'absence de DMSO.
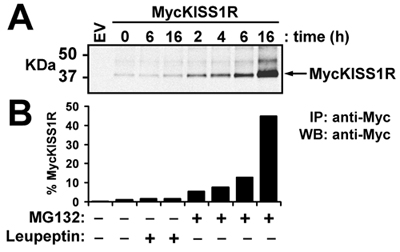
Figure 3. Effet de la leupeptine ou MG132 sur les niveaux de Myc KISS1R protéines dans les cellules HEK-293: cellules HEK-293 exprimant Myc KISS1R ont été traités avec 100μg/ml leupeptine ou 10 uM MG132 à 37 ° C pendant le temps désigné. Myc KISS1R sur 400μg de lysat cellulaire a été immunoprécipité avec 2.5μg d'agarose-anticorps anti-myc anticorps et analysés par western blot. (A) de détection de l'Odyssée LI-COR du KISS1R Myc après une incubation d'immunoblots de lapin anti-Myc-tag anticorps suivie d'une incubation avec IRDye 800CW-anticorps anti-lapin (B) Quantification des bandes KISS1R Myc montré en (A) en utilisant le LI-COR logiciel de quantification Odyssée. Les résultats sont représentés en tant rabattable augmenter au fil des cellules non traitées (temps 0).
Discussion
La mutagenèse dirigée a été utilisée pour étudier la fonction des protéines par l'introduction de changements de nucléotides dans la séquence codante des gènes cibles de plus de trois décennies. La technique originale a été décrite en 1978 par le chimiste britannique-canadien et lauréat du Prix Nobel Michael Smith 10. Michael Smith a partagé le Prix Nobel 1993 de chimie avec Kary Mullis, le biochimiste américain qui a inventé la Polymerase Chain Reaction (PCR) 11. La méthode ...
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Ce travail a été partiellement financé par la Direction de la reproduction de l'Institut national de la santé infantile et le développement humain (NICHD - R21 HD059015) et par la Charles H. Bois Fondation Enfant Young Investigator Award de recherche en santé (Boston, MA).
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Société | Numéro de catalogue | |
| 10x PCRx Enhancer Solution | Invitrogen | 52391 | |
| PfuUltra haute fidélité Détergent alternative ADN polymérase | Stratagene | 600385 | |
| DPN-I | New England Biolabs | R0176 | |
| XL10-Gold Ultracompetent E. cellules coli | Stratagene | 200314 | |
| DMEM | Cellgro | 10 à 013-CV | |
| Sérum fœtal bovin | Atlanta Biologicals | S11550 | |
| Réactif de transfection Geneporter | Genlantis | T201007 | |
| Leupeptine | Calbiochem | 108975 | |
| MG132 | Calbiochem | 47491 | |
| 10xPBS | Ambion | AM9625 | |
| Inhibiteur de la protéase cocktail et PMSF | Santa Cruz Biotechnology | Sc-24948 | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| anti-Myc tag (clone 4A6) agarose conjugué | Millipore | 16-219 | |
| Tampon de charge 2x | BioRad | 161-0737 | |
| Critère de Tris-HCl préfabriqué gel gradient 4-15% | BioRad | 345-0028 | |
| Immobilon-FL membrane PVDF | Millipore | IPFL00010 | |
| Tampon de blocage Odyssey | LI-COR Biosciences | 927-40000 | |
| 10xTBS | BioRad | 170-6435 | |
| Lapin d'anticorps anti-myc | Signalisation cellulaire | 2272 | |
| Chèvre anti-lapin IRDye 800CW | LI-COR Biosciences | 926-32211 | |
| Imagerie infrarouge Odyssey système | LI-COR Biosciences | ||
| Cocktail inhibiteur de protéase Halt (100x) | Thermo Scientific | 78430 |
Références
- Seminara, S. B., Messager, S., Chatzidaki, E. E. The GPR54 Gene as a Regulator of Puberty. N Engl J Med. 349, 1614-1627 (2003).
- de Roux, N., Genin, E., Carel, J. C. Hypogonadotropic Hypogonadism Due to Loss of Function of the KiSS1-Derived Peptide Receptor GPR54. Proc Natl Acad Sci U S A. 100, 10972-10976 (2003).
- Messager, S., Chatzidaki, E. E., Ma, D. Kisspeptin Directly Stimulates Gonadotropin-Releasing Hormone Release via G Protein-Coupled Receptor 54. Proc Natl Acad Sci U S A. 102, 1761-1766 (2005).
- Teles, M. G., Bianco, S. D., Brito, V. N. A GPR54-Activating Mutation in a Patient with Central Precocious Puberty. N Engl J Med. 358, 709-715 (2008).
- Tenenbaum-Rakover, Y., Commenges-Ducos, M., Iovane, A. Neuroendocrine Phenotype Analysis in Five Patients with Isolated Hypogonadotropic Hypogonadism due to a L102P Inactivating Mutation of GPR54. J Clin Endocrinol Metab. 92, 1137-1144 (2007).
- Semple, R. K., Achermann, J. C., Ellery, J. Two Novel Missense Mutations in G Protein-Coupled Receptor 54 in a Patient with Hypogonadotropic Hypogonadism. J Clin Endocrinol Metab. 90, 1849-1855 (2005).
- Wacker, J. L., Feller, D. B., Tang, X. B. Disease-Causing Mutation in GPR54 Reveals the Importance of the Second Intracellular Loop for Class A G-Protein-Coupled Receptor Function. J Biol Chem. 283, 31068-31078 (2008).
- Szereszewski, J. M., Pampillo, M., Ahow, M. R. GPR54 regulates ERK1/2 activity and hypothalamic gene expression in a Galpha(q/11) and beta-arrestin-dependent manner. PLoS One. 5, e12964-e12964 (2010).
- Bianco, S. D. C., Vandepas, L., Correa-Medina, M., Gereben, B., Mukherjee, A., Kuohung, W., Carroll, R., Teles, M. G., Latronico, A. C., Kaiser, U. B. KISS1R Intracellular Trafficking and Degradation: Effect of the Arg386Pro Disease-Associated Mutation. Endocrinology. , (2011).
- Hutchison, C. A., Phillips, S., Edgell, M. H. Mutagenesis at a Specific Position in a DNA Sequence. J Biol Chem. 253, 6551-6560 (1978).
- Mullis, K. B. The Unusual Origin of the Polymerase Chain Reaction. Sci Am. 262, 56-61 (1990).
- Henke, W., Herdel, K., Jung, K., Schnorr, D., Loening, S. A. Betaine improves the PCR amplification of GC-rich DNA sequences. Nucleic Acids Res. 25, 3957-3958 (1997).
- Sahdev, S., Saini, S., Tiwari, P., Saxena, S., Singh Saini, K. Amplification of GC-rich genes by following a combination strategy of primer design, enhancers and modified PCR cycle conditions. Mol Cell Probes. 21, 303-307 (2007).
- Kong, K. C., Poyner, D. R., Wheatly, M. Chapter 10. MSTA. in G Protein-Coupled Receptors: Essential Methods. , 197-204 (2010).
- Hall, R. A., George, S. R., O'Dowd, B. F. Chapter 9. G protein-Coupled Receptor-Protein Interactions. , 170-171 (2005).
- Chaturvedi, K., Bandari, P., Chinen, N., Howells, R. D. Proteasome Involvement in Agonist-Induced Down-Regulation of Mu and Delta Opioid Receptors. J Biol Chem. 276, 12345-12355 (2001).
- Shenoy, S. K., McDonald, P. H., Kohout, T. A., Lefkowitz, R. J. Regulation of Receptor Fate by Ubiquitination of Activated Beta 2-Adrenergic Receptor and Beta-Arrestin. Science. 294, 1307-1313 (2001).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon