Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
El estudio de ADN Looping por FRET sola molécula
En este artículo
Resumen
Este estudio presenta un procedimiento experimental detallado para medir la dinámica de bucle de ADN de doble cadena utilizando una sola molécula de transferencia de energía de resonancia fluorescente (FRET). El protocolo también se describe cómo extraer la densidad de probabilidad de bucle llamado factor J.
Resumen
De flexión de ADN de doble cadena (dsDNA) se asocia con muchos procesos biológicos importantes, tales como el reconocimiento de DNA-proteína y el empaquetamiento del ADN en nucleosomas. Termodinámica de ADN de doble cadena de flexión se ha estudiado mediante un método denominado ciclación que se basa en la ADN ligasa para unir covalentemente los extremos pegajosos cortos de un ADN de doble cadena. Sin embargo, la eficiencia de la ligadura puede verse afectada por muchos factores que no están relacionados con dsDNA bucle tales como la estructura del ADN que rodea los extremos pegajosos unidas, y ligasa también puede afectar a la velocidad de bucle evidente a través de mecanismos tales como la unión no específica. Aquí, se muestra la forma de medir la cinética de bucle de ADN bicatenario sin ligasa mediante la detección de la formación de bucle de ADN transitoria por FRET (transferencia de energía de resonancia fluorescente). moléculas de ADN de doble cadena se construyen usando un simple protocolo basado en PCR con un par de FRET y un enlazador biotina. La densidad de probabilidad de bucle conocido como el factor de J se extrae de la tasa de bucle y la tasa de recocido entre dos desconexiónted extremos pegajosos. Al poner a prueba dos dsDNAs con diferentes curvaturas intrínsecas, se muestra que el factor J es sensible a la forma intrínseca de la ADN de doble cadena.
Introducción
La comprensión de las propiedades mecánicas de ADN de doble cadena es de importancia fundamental en las ciencias básicas y aplicaciones de ingeniería. La estructura de ADN de doble cadena es más complicado que una escalera helicoidal recto debido ángulos de balanceo, inclinación y giro entre pares de bases sucesivas pueden variar con la secuencia. Fluctuaciones térmicas pueden provocar dsDNA a someterse a diversos modos de fluctuaciones conformacionales como la flexión, torsión y estiramiento. Transiciones de fusión tales como el retorcimiento y también pueden ocurrir en condiciones extremas.
Entre estos movimientos, dsDNA flexión tiene el impacto biológico más sensible 1. ADN de doble cadena de flexión está asociada con la represión de genes o la activación por la unión de dos sitios distantes cerca uno del otro. También juega un papel importante en el empaquetamiento del ADN dentro del núcleo de la célula o de una cápside viral. Deformación por flexión de ADN de doble cadena se puede visualizar experimentalmente por microscopía de alta resolución (AFM 2 y TEM 3), y la Thermodynamics y cinéticas pueden ser estudiadas mediante ensayos de bucle, que enlazan químicamente sitios yuxtapuestos del ADN de doble cadena.
Uno de tales ensayos es ligasa dependiente de ciclación 4. En este ensayo, las moléculas de ADN de doble cadena con extremos 'pegajosos' (cohesivos) se circularized o dimerizados por ADN ligasa. Mediante la comparación de las tasas de círculo y la formación de dímero, se puede obtener una concentración molar eficaz de uno de los extremos del ADN en la proximidad del otro extremo, que se conoce como el factor de J. Este factor J es dimensionalmente equivalente a la densidad de probabilidad de encontrar un extremo del ADN a una distancia corta desde el otro extremo, y por lo tanto refleja la flexibilidad del ADN. Medir el factor de J como una función de la longitud del ADN revela muchas características acerca de la mecánica de ADN incluyendo el 4,5 longitud de persistencia.
La cadena de gusano-como modelo (WLC) ha sido ampliamente considerado como el modelo canónico de polímero para la mecánica dsDNA en función de su éxito en la explicaciónnando las curvas fuerza-extensión obtenidos en el ADN tirando experimentos 6 y predecir correctamente los factores de J dsDNAs más de 200 pb 7. Sin embargo, usando el ensayo de ciclación en las moléculas de ADN de doble cadena tan cortos como 100 pb, Cloutier y Widom midieron los factores J ser varios órdenes de magnitud mayor que la predicción del modelo WLC 8. Un año más tarde, Du et al. producido J factores de acuerdo con el modelo WLC usando el ensayo de ciclación con concentraciones más bajas de ligasa y atribuido el resultado anómalo del grupo Widom a concentraciones altas ligasa utilizada 9. Esta controversia es un ejemplo de la influencia inevitable de la ADN ligasa en la cinética de ciclación cuando se utiliza el ensayo convencional 9. Por otra parte, la ligasa de ADN también puede afectar a la estructura del ADN y la rigidez a través inespecífica 10,11 vinculante.
Para eliminar los problemas técnicos de los ensayos de bucles de proteínas dependientes, hemos demostrado una protensayo de bucle ein-gratuito basado en energía de resonancia fluorescente Transfer (FRET) 12. En este método, las conformaciones en bucle se detectan por FRET entre el donante y aceptor unido cerca de los extremos pegajosos de una molécula de ADN. Un microscopio de fluorescencia de reflexión total interna de tipo objetivo (TIRFM) se utiliza para registrar las trayectorias de bucle reversible y eventos unlooping de moléculas individuales de ADN inmovilizado en la superficie por un período de tiempo prolongado. Este método cuenta con el montaje basado en la PCR de moléculas de ADN para generar moléculas de ADN-desajuste libre, que es una mejora crucial en un método similar por Vafabakhsh y Ha 13. El aspecto de una sola molécula de este protocolo permite la medición de distribuciones en adición a Ensemble promedios mientras que el aspecto de FRET permite medir la dinámica de bucle de ADN repetidamente de la misma molécula, incluso en condiciones que pueden afectar la actividad ligasa.
La configuración TIRFM se muestra en la Figura 1. Una costumbrePlatina de diseño se coloca sobre un cuerpo del microscopio Olympus IX61. 532 nm y 640 nm láser se introducen desde el lado y son reflejados por espejos diminutos elípticas 14 en el alta NA objetivo de lograr ángulo crítico de incidencia en la interfaz agua-cubreobjetos. Observamos que más generalizada-a través de objetivo TIR utilizando espejos dicroicos o configuraciones de TIR a base de prisma también se puede utilizar para esta aplicación de FRET. La imagen de fluorescencia formado por el microscopio se divide en donante y aceptor de imágenes por un espejo dicroico. Ellos se vuelven a reflejados en dos mitades de una EMCCD. Filtros de emisión de paso de largo adicionales se utilizan para reducir la señal de fondo.
El control de temperatura es esencial para la adquisición de los datos cinéticos reproducibles. Para el control de la temperatura, el objetivo se separa de la pieza de nariz del cuerpo del microscopio para minimizar la transferencia de calor, y el agua de un enfriador controlada / calentador de la temperatura se hace circular a través de un collar de latón que encaja herméticamentealrededor del metal interior debajo de la chaqueta objetivo. Esta configuración es capaz de alcanzar control de la temperatura robusta en la superficie cubreobjetos entre 15 y 50 ° C (Figura 2). En este trabajo, la temperatura de la muestra se mantuvo a 24 ° C.
El protocolo siguiente se presenta el procedimiento paso a paso para la construcción de ADN, la estimación de la forma de ADN, el experimento de una sola molécula, y la determinación del factor J.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparación de la muestra de ADN bicatenario
- Diseñar ADNs curvas a nivel mundial mediante la repetición de una secuencia de 10-mer. Por ejemplo, 5'-GTGCCAGCAACAGATAGC - (TTTATCATCCTTTATCATCC X) 7 - TTTCATTCGAGCTCGTTGTTG-3 'es un ADN curvada 186-pb donde X es una base adicional al azar y la secuencia que flanquea la secuencia de 10-mer repitiendo son secuencias adaptadoras.
NOTA: En este ejemplo se seleccionaron dos 10-meros con preferencias opuestas a la formación de nucleosomas en base a un estudio a gran escala de ocupación nucleosoma por Kaplan et al 15.. Desde la repetición helicoidal de ADN de doble cadena es cerca de 10 pb, cualquier desviación neta del eje helicoidal de la 10-mer se acumulan para producir una forma como un arco circular (Figura 3A). Desde el período helicoidal está más cerca de 10,5 pb, una base extra se inserta después de cada dos repeticiones para mantener la estructura curvada como plana como sea posible. Estas secuencias más cortas de 200 pb se pueden pedir a companies que el servicio de síntesis de genes oferta. Es conveniente para flanquear estas secuencias con secuencias adaptadoras comunes para las etapas subsiguientes. El procedimiento se esquematiza en la Figura 3B. - Lleve a cabo PCR con cebador 1 (GTGCCAGCAACAGATAGC) y cebador 2 (/ 5Cy3/TAAATTCCTACAACAACGAGCTCGAATG). NOTA: Cebador 2 se etiqueta con el Cy3 donante de FRET en el extremo 5 '. Una receta típica PCR y protocolo de ciclos se presentan en las tablas 1 y 2.
- Lleve a cabo PCR con Primer 3 (/ 5BioTEG/GAAACATAG/iCy5/GAATTTACCGTGCCAGCAACAGATAGC) y Primer 4 (CAACAACGAGCTCGAATG). NOTA: Cebador 3 está etiquetado con el aceptor Cy5 FRET a través de la columna vertebral y la biotina-enlazador en el extremo 5 '. Receta PCR y protocolo de ciclos son como anteriormente.
- Purificar los productos de PCR utilizando un kit de limpieza de PCR.
- Mezclar el producto marcado con Cy3 y el producto marcado con Cy5 en un tampón para el intercambio de cadenas (100 mM NaCl, 10 mM de Tris-HCl, pH 7,0, EDTA 1 mM) a concentraciones finales de 0,4 y# 181; M y 0,1 M, respectivamente. NOTA: El exceso de Cy3-ADN aumenta la concentración del dúplex que lleva tanto Cy3 y Cy5 como resultado de la línea de cambio.
- Tipo de hebras por incubación a 98,5 ° C durante 2 min, enfriar gradualmente a 5 ° C con velocidad de rampa de 0,1 º C / seg, e incubando a 5 ° C durante 2 horas.
2. Electroforesis en gel para detectar ADN de doble cadena Curvatura
- Verter el gel de poliacrilamida 16,17 mezclando soluciones de acrilamida y bis-acrilamida en relación 29.2:0.8, 5% (w / v) en 1X Tris / borato / EDTA (TBE) tampón a pH 8,0. Nota: 10 ml de solución de gel contiene: 1,217 ml de 40% de acrilamida, 0,667 ml de 2% de bis-acrilamida, 1 ml de TBE 10X, 100 persulfato L de amonio (APS) 10%, 10 L de TEMED, y el resto es dH 2 O. Solidificación completa del gel tarda unos 30 min.
- Cargar el gel de poliacrilamida con muestras de ADN y la escalera de ADN en tampón de carga 1X (5% de glicerol, 0,03% (w / v) de bromofenol BLue) y ejecutar el gel a 5-8 V / cm a 4 ° C durante 45 min o hasta que el frente del colorante se acerca el final del gel.
- Tinción del gel usando tampón TBE 1X que contiene 0,5 g / ml de bromuro de etidio durante 30 min. Identificar las bandas de ADN bajo iluminación UV. Comparar posiciones de las bandas con el marcador de tamaño (100 pb escalera de ADN) para calcular los tamaños aparentes de las moléculas de ADN. NOTA: Curved ADN generalmente se mueven más lento que los ADN rectas.
3. De flujo Preparación de células
- Perforar 6-7 pares de agujeros a lo largo de dos bordes opuestos de una lámina de vidrio (3'' x 1'') usando un taladro y brocas de diamante. Después de la perforación, frote la diapositiva de agua que fluye para quitar polvo de vidrio visible. NOTA: Los agujeros sirven como entradas de perfusión y puntos de venta. Durante la perforación, el enfriamiento de la diapositiva con agua es importante para prevenir el agrietamiento.
- Colocar los portaobjetos en posición vertical en un frasco de vidrio y llenarlo con agua. Déjelos en remojo durante 15 min y transferirlos a otro frasco de vidrio dedicated para la limpieza acetona. Rellenar con acetona y déjelos en remojo durante 15 min. Enjuagar los portaobjetos con etanol utilizando una botella de spray y luego con agua. Colóquelos en un recipiente de polipropileno, llenarlo de 5 M de hidróxido de potasio y déjelos en remojo durante 15 min. Por último, sonicar las diapositivas de agua durante 15 min. Limpiar los cubreobjetos (n º 1, 24 x 40 mm) utilizando el mismo protocolo. NOTA: diapositivas y cubreobjetos limpios pueden almacenarse en dH 2 O para el uso a largo plazo.
- Mezclar 1 mg de biotina-PEG-silano (MW 3400) con 80 mg de mPEG-silano (MW 2000) en 340 L de solución de bicarbonato de sodio 0,1 M. Mezclar bien y centrifugar la mezcla brevemente para eliminar las burbujas. NOTA: La funcionalización de la superficie con polietilenglicol (PEG) ayuda a reducir la unión no específica de ADN a la superficie.
- Ponga 80 L de la solución de PEG en cada diapositiva y bajar suavemente un cubreobjetos sobre ella. Espere 45 min. Separe el cubreobjetos de la diapositiva con pinzas, enjuáguelos con abundante cantidad de dH 2 O y dejar que tseco dobladillo al aire libre.
- Coloque tiras delgadas de cinta adhesiva de doble cara en toda la diapositiva para formar canales. Alinear un cubreobjetos sobre ella y presione firmemente el cubreobjetos contra la corredera para formar canales estancos a los líquidos. Utilice 5 minutos epoxi para sellar los bordes de los canales.
4. Preparación de la solución de Trolox
- Ponga ~ 30 mg Trolox y 10 ml de dH2O en un matraz y usar una barra de agitación imán para agitar la solución al aire libre durante 18 horas.
- Filtrar la solución usando un filtro de 0,2 micras y ajustar el pH a 7 mediante la adición de ~ 6 l de 1 M Tris base (pH 11). Nota: Trolox es un reactivo anti-parpadeo que se utiliza comúnmente en una sola molécula estudia 18. La acción antifading de Trolox proviene de su derivado oxidado que está presente en la solución de Trolox parcialmente degradada 19. Disolviendo rápidamente Trolox en metanol o una solución de Tris pH alto se debe evitar debido a la oxidación ineficiente.
5. Individual-molImaging ecule
- Inyectar 15 l de solución de NeutrAvidin (0,5 mg / ml) en el canal y esperar durante 2 minutos antes de enjuagar con 100 l de tampón de T50 (10 mM de Tris-HCl, NaCl 50 mM, pH 7,0).
- Inyectar 50 l de muestra de ADN (50-100 pM) en el canal. Esperar 5 minutos y enjuagar el ADN no unido de distancia con 100 L de tampón de T50. NOTA: moléculas de ADN se unirán específicamente a la superficie a través de la interacción biotina-NeutrAvidin.
- Llenar el canal con el buffer de imágenes que contiene un sistema de eliminación de oxígeno 20 (100 mM de PCD, 5 mM de PCA, 1 mM de Trolox, y NaCl 500 mM).
- Ajuste el EMCCD en modo de transferencia de fotogramas para transmitir 2 x 2 imágenes agrupadas (256 x 256) a la computadora a 25 fotogramas por segundo.
- Poner el aceite de inmersión sobre el objetivo del microscopio, y fijar la celda de flujo en la platina del microscopio utilizando clips de muestra. De grano grueso, ajuste el enfoque mirando el patrón de reflexión láser en la pared. Ajuste con precisión el enfoque utilizando la visualización en vivo de fluorescencia imlas edades en el monitor.
- Comience la adquisición de datos con el láser de 532 nm en. Compruebe la vista en vivo y ajuste el enfoque si es necesario. Deje de adquisición de datos cuando la mayoría de las moléculas han photobleached.
NOTA: En esta instalación, se utiliza un programa de laboratorio-escrito C para controlar el microscopio y visualizar imágenes en directo en el monitor, ya que se guardan en el disco duro.
6. Procesamiento de imágenes y análisis de datos
NOTA: Una serie de tiempo de 256 x 256 imágenes son procesadas por un código de MATLAB para generar una sola molécula de rastros de tiempo de Cy3 y Cy5 intensidades. Para emparejar píxeles entre el canal de donante y el canal aceptor de la imagen de vista dividida, 6-7 pares de Cy3 y Cy5 manchas, cada par de la misma molécula uniformemente dispersado a través del campo de vista, son recogidos manualmente, y una transformación afín se calcula utilizando las coordenadas de estos lugares como puntos de anclaje.
- El uso de un script de MATLAB, mirar a través de todos los tiempos de una sola molécula traza tsombrero muestran múltiples transiciones entre las señales de baja y alta traste. Identificar los estados en bucle y unlooped.
NOTA: La señal de FRET se define como la intensidad de Cy5 (I a) dividido por la suma de Cy3 y Cy5 intensidades (I A + Id). Estado en bucle tiene un alto valor FRET mientras que el estado unlooped tiene valor bajo FRET. El histograma de las señales de FRET de una sola molécula se distribuye bimodal porque de bucle reversible y unlooping. - Encontrar el umbral que separa las dos distribuciones mediante la determinación de la intersección entre las dos curvas gaussianas armarios.
- Calcular la eficiencia de FRET como
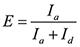 y asignar los estados en bucle con altos valores de FRET y los estados unlooped con valores bajos de FRET.
y asignar los estados en bucle con altos valores de FRET y los estados unlooped con valores bajos de FRET. - El uso de un script de MATLAB, analizar el número acumulado de moléculas (N (t)) que en bucle (oalcanzado el estado alto FRET) en diferentes lapsos de tiempo desde el inicio de la adquisición de datos. NOTA: Dado que una molécula de ADN de doble cadena puede iniciar en cualquier conformación del estado unlooped en el inicio de adquisición de datos, la tasa de aumento en la población en bucle refleja la tasa de bucle medios promediados sobre conformaciones iniciales. Extraer el bucle tasa de bucle K mediante el ajuste de N (t) con una función exponencial:
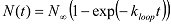 Si aumenta bifásica, que puede ser equipado con una función doble exponencial:
Si aumenta bifásica, que puede ser equipado con una función doble exponencial: 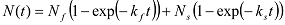 En este caso, el bucle k se obtiene a partir de:
En este caso, el bucle k se obtiene a partir de: 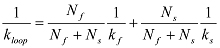 NOTA: En teoría, la probabilidad de supervivencia que un polímero no ha enrollado en el tiempo t no es un simple o doble función exponencial 12. Fitt Exponencialción se utiliza como un medio práctico para extraer el promedio de tiempo de bucle.
NOTA: En teoría, la probabilidad de supervivencia que un polímero no ha enrollado en el tiempo t no es un simple o doble función exponencial 12. Fitt Exponencialción se utiliza como un medio práctico para extraer el promedio de tiempo de bucle.
7. Determinación del factor J
NOTA: El factor J representa cómo concentrada uno de los extremos de un ADN de doble cadena es alrededor del otro extremo. Se puede determinar por interpolación la concentración de un segmento extremo del ADN que produciría la misma velocidad de reacción con el otro segmento final como la tasa de bucle medido. Experimentalmente, un segmento extremo está inmovilizado sobre la superficie, y el otro segmento extremo se introduce a una cierta concentración c. Si la tasa de recocido medida entre los dos extremos es k recocido, entonces el factor de J 21 está dada por 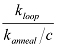 . La constante de velocidad de recocido (k = k recocido recocido / C) es independiente de la concentración de la sonda.
. La constante de velocidad de recocido (k = k recocido recocido / C) es independiente de la concentración de la sonda.
- Caudal 20 l de 30-50 pM biotina-Cy5 oligo (Primer 3) iONT un canal recubierto de NeutrAvidin. Enjuague el canal con 100 l T50 para lavar oligos no consolidados.
- Preparar el tampón de formación de imágenes tal como se describe en la parte 5 con la adición de Cy3 oligo (cebador 2) a una concentración final de 50 nM. Este flujo de buffer de imágenes en el canal.
- Mientras se mantiene el láser 532 nm, gire brevemente el láser de 640 nm a identificar la ubicación de la superficie determinada Cy5 oligos. Apague el láser de 640 nm y empezar a monitorizar la señal de FRET.
NOTA: Tras la hibridación de un oligo Cy3 a un Cy5 oligonucleótido unido a la superficie, Cy5 fluorescencia surgirá de FRET. - El uso de un script de MATLAB, analizar el número de moléculas que comienzan en el estado no ligado (baja intensidad de Cy5), pero más tarde se convierten en el estado (alta intensidad de Cy5) recocido como una función del tiempo a partir de los restos de intensidad Cy5.
- Trace ese número de moléculas en función del tiempo de recocido. Ajuste esta curva con una sola función exponencial (
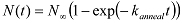 ) Para obtener la tasa de recocido (k recocido).
) Para obtener la tasa de recocido (k recocido). - Repita este experimento a diferentes concentraciones Cy3 oligo (60, 100, y 180 nm) para confirmar la linealidad entre la tasa de recocido y la concentración de reactivo. Extracto de la constante de velocidad de recocido de segundo orden (k recocido ) A partir de la pendiente.
- Calcular el factor J de
 , Donde bucle k es la tasa de bucle medido en las mismas condiciones tampón.
, Donde bucle k es la tasa de bucle medido en las mismas condiciones tampón.
Access restricted. Please log in or start a trial to view this content.
Resultados
Moléculas de ADN utilizados para el estudio de bucle consisten en una región dúplex de la secuencia y longitud variables y salientes monocatenarios que son complementarios entre sí. Los salientes, que son 7 bases de longitud, pueden hibridar entre sí para capturar el estado de bucle. Cada saliente contiene ya sea Cy3 o Cy5 que está vinculado en la columna vertebral del ADN a través de la química amidita. El Cy5-alero también está vinculado con biotina-TEG (15-átomo de Tetra-etilenglicol espaciador) para la in...
Access restricted. Please log in or start a trial to view this content.
Discusión
Se utilizó un ensayo simple de una sola molécula basado en FRET para estudiar la cinética de bucle de ADN de diferentes formas intrínsecas. ADNs curvados se pueden preparar mediante la repetición de una secuencia de 10-mer, en fase con el período helicoidal de 10,5 pb, y sus curvaturas pueden ser estimados utilizando la página. Estos dsDNAs están diseñados con extremos pegajosos para permitir la estabilización transitoria de bucle. Se extrajo la tasa de bucle desde el aumento exponencial en el número de molé...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores declaran no tener ningún conflicto de intereses.
Agradecimientos
Damos las gracias a James Waters, Gable Wadsworth y Bo Broadwater para la lectura crítica del manuscrito. También agradecemos a cuatro revisores anónimos por proporcionar comentarios útiles. Reconocemos el apoyo financiero del Instituto de Tecnología de Georgia, el premio a la Trayectoria Fondo Burroughs Wellcome en la interfaz de la Ciencia y la concesión de la red de investigación de estudiantes de la NSF Física de Sistemas Vivos.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Small DNA FRAG Extract Kit-100PR | VWR | 97060-558 | |
| Acrylamide 40% solution 500 ml | VWR | 97064-522 | |
| Bis-acrylamide 2% (w/v) solution 500 ml | VWR | 97063-948 | |
| GeneRuler 100 bp DNA Ladder, 100-1,000 bp | Fermentas | SM0241 | |
| Mini Vertical PAGE System | VWR | 89032-300 | |
| Syringe filter 0.2 μm CS50 | VWR | A2666 | |
| Trolox | Sigma-Aldrich | 238813-1G | triplet state quencher |
| Protocatechuic acid (PCA) | Sigma-Aldrich | 08992-50MG | oxygen scavenging system |
| Protocatechuate 3,4-Dioxygenase (PCD) | Sigma-Aldrich | P8279-25UN | oxygen scavenging system |
| mPEG-silane, MW 2,000 1 g | Laysan Bio | MPEG-SIL-2000-1g | |
| Biotin-PEG-Silane, MW 3,400 | Laysan Bio | Biotin-PEG-SIL-3400-1g | |
| Avidin, NeutrAvidin Biotin-binding Protein | Invitrogen | A2666 | |
| Phusion Hot Start High-Fidelity DNA Polymerase | New England Biolabs | F-540L | |
| Gel/PCR DNA Fragments Extraction Kit | IBI Scientific | IB47020 | |
| Premium plain glass microscope slides | Fisher Scientific | 12-544-1 | |
| VWR micro cover glass, rectangular, no. 1 | VWR | 48404-456 | |
| Fisher Scientific Isotemp 1006S Recirculating Chiller/Heater | Fisher Scientific | temperature control | |
| Objective Cooling Collar | Bioptechs | 150303 | temperature control |
| KMI53 Biological Micrometer Measuring Stage | Semprex | KMI53 | |
| High Performance DPSS Laser 532 nm 50 mW | Edmund optics | NT66-968 | Cy3 excitation |
| CUBE Fiber Pigtailed 640 nm, 30 mW, Fiber, FC/APC Connector | Coherent | 1139604 | Cy5 excitation |
| 650 nm BrightLine Dichroic Beamsplitter | Semrock | FF650-Di01-25x36 | splitting dichroic |
| LaserMUX Beam Combiner, reflects 514.5, 532, & 543.5 nm lasers, 25 mm | Semrock | LM01-552-25 | combining dichroic |
| Brightline Fluorescence Filter 593/40 | Semrock | FF01-593/40-25 | Cy3 emission filter |
| 635 nm EdgeBasic LWP longpass Filter, 25 mm | Semrock | BLP01-635R-25 | Cy5 emission filter |
| EMCCD iXon+ | Andor Technology | DU-897E-CS0-#BV | |
| IX51 inverted microscope frame | Olympus | ||
| Objective UApo N 100X/1.49 Oil TIRF | Olympus | ||
| Immersion oil type-F for fluorescence microscopy | Olympus | IMMOIL-F30CC | |
| 2 mm Diameter 45° Rod Lens Aluminum Coated | Edmund optics | 54-092 | miniature mirror |
| 1/4" Travel Single-Axis Translation Stage | Thorlabs | MS-1 | translation of miniature mirror |
| Ø1" Achromatic Doublet, ARC: 400-700 nm, f=200 mm | Thorlabs | AC254-200-A | focusing lens |
| Adjustable Mechanical Slit | Thorlabs | VA100 | |
| Dielectric Mirror | Thorlabs | BB1-E02 | |
| Ø1" Achromatic Doublet, f = 100 mm | Thorlabs | AC254-100-A | relay lens |
| Lens Mount for Ø1" Optics | Thorlabs | LMR1 | |
| Dichroic Filter Mount | Thorlabs | FFM1 | |
| Fixed Cage Cube Platform | Thorlabs | B3C | |
| Kinematic Mount for Ø1" Optics | Thorlabs | KM100 | |
| N-BK7 Plano-Convex Lens, Ø1", f = 40 mm | Thorlabs | LA1422-A | collimating lens |
| N-BK7 Plano-Convex Lense, Ø6.0 mm, f = 15 mm | Thorlabs | LA1222-A | telescope lens |
| N-BK7 Plano-Convex Lense, Ø6.0 mm, f = 150 mm | Thorlabs | LA1433-A | telescope lens |
Referencias
- Garcia, H. G., et al. Biological consequences of tightly bent DNA: The other life of a macromolecular celebrity. Biopolymers. 85, 115-130 (2007).
- Wiggins, P. A., et al. High flexibility of DNA on short length scales probed by atomic force microscopy. Nature Nanotechnology. 1, (2006).
- Lionberger, T. A., et al. Cooperative kinking at distant sites in mechanically stressed DNA. Nucleic Acids Research. 41, 6785-6792 (2011).
- Shore, D., et al. DNA flexibility studied by covalent closure of short fragments into circles. Proc Natl Acad Sci U S A. 78, 4833-4837 (1981).
- Geggier, S., Vologodskii, A. Sequence dependence of DNA bending rigidity. Proc Nat Acad Sci U S A. 107, 15421-15426 (1992).
- Smith, S. B., et al. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258, 1122-1126 (1992).
- Peters, J. P., Maher, L. J. DNA curvature and flexibility in vitro and in vivo. Quarterly Reviews of Biophysics. 43, 23-63 (2010).
- Cloutier, T. E., Widom, J. Spontaneous sharp bending of double-stranded DNA. Molecular Cell. 14, 355-362 (2004).
- Du, Q., et al. Cyclization of short DNA fragments and bending fluctuations of the double helix. Proc Natl Acad Sci U S A. 102, 5397-5402 (2005).
- Yuan, C., et al. T4 DNA ligase is more than an effective trap of cyclized dsDNA. Nucl. Acids Res. 35, 5294-5302 (2007).
- Manzo, C., et al. The effect of nonspecific binding of lambda repressor on DNA looping dynamics. Biophysical Journal. 103, 1753-1761 (2012).
- Le, T. T., Kim, H. D. Measuring shape-dependent looping probability of DNA. Biophys. J. 104, 2068-2076 (2013).
- Vafabakhsh, R., Ha, T. Extreme bendability of DNA less than 100 base pairs long revealed by single-molecule cyclization. Science. 337, 1097-1101 (2012).
- Friedman, L., et al. Viewing dynamic assembly of molecular complexes by multi-wavelength single-molecule fluorescence. Biophysical Journal. 91, 1023-1031 (2006).
- Kaplan, N., et al. The DNA-encoded nucleosome organization of a eukaryotic genome. Nature. 458, 362-366 (2009).
- Koo, H. S., Crothers, D. M. Calibration of DNA curvature and a unified description of sequence-directed bending. Proc Nat Acad Sci U S A. 85, 1763-1767 (1988).
- Prosseda, G., et al. A temperature-induced narrow DNA curvature range sustains the maximum activity of a bacterial promoter in vitro. Biochemistry. 49, 2778-2785 (2010).
- Rasnik, I., et al. Nonblinking and long-lasting single-molecule fluorescence imaging. Nature Methods. 3, 891-893 (2006).
- Cordes, T., et al. On the mechanism of Trolox as antiblinking and antibleaching reagent. J. Am. Chem. Soc. 131, 5018-5019 (2009).
- Aitken, C. E., et al. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophys J. 94, 1826-1835 (2008).
- Taylor, W. H., Hagerman, P. J. Application of the method of phage T4 DNA ligase-catalyzed ring-closure to the study of DNA structure: II. NaCl-dependence of DNA flexibility and helical repeat. Journal of Molecular Biology. 212, 363-376 (1990).
- Bolshoy, A., et al. Curved DNA without A-A: experimental estimation of all 16 DNA wedge angles. Proc Natl Acad Sci U S A. 88, 2312-2316 (1991).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucl. Acids Res. 37, 6984-6990 (2009).
- Vologodskii, A., et al. Bending of short DNA helices. Artif DNA PNA XNA. 4, (2013).
- Hoover, D. M., Lubkowski, J. DNAWorks: an automated method for designing oligonucleotides for PCR-based gene synthesis. Nucl. Acids Res. 30, (2002).
- Waters, J. T., Kim, H. D. Equilibrium Statistics of a Surface-Pinned Semiflexible Polymer. Macromolecules. 46, 6659-6666 (2013).
- Mills, J. B., et al. Electrophoretic evidence that single-stranded regions of 1 or more nucleotides dramatically increase the flexibility of DNA. Biochemistry. 33, 1797-1803 (1994).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados