È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Studiare Looping DNA a singola molecola FRET
In questo articolo
Riepilogo
Questo studio presenta una procedura sperimentale dettagliato per misurare le dinamiche di loop di DNA a doppio filamento con singola molecola Fluorescence Resonance Energy Transfer (FRET). Il protocollo descrive inoltre come estrarre il ciclo di densità di probabilità chiamato il fattore J.
Abstract
Curvatura del DNA a doppia elica (dsDNA) è associato con molti importanti processi biologici quali il riconoscimento DNA-proteine e confezionamento DNA in nucleosomi. Termodinamica di dsDNA curvatura è stata studiata da un metodo chiamato ciclizzazione che si basa sul DNA ligasi di unirsi covalentemente brevi estremità coesive di un dsDNA. Tuttavia, l'efficienza legatura può essere influenzata da molti fattori che non sono legati a dsDNA loop come la struttura del DNA che circonda le estremità appiccicose uniti, e ligasi può anche influenzare il tasso looping apparente attraverso meccanismi quali il legame non specifico. Qui mostriamo come misurare dsDNA cinetica looping senza ligasi rilevando formazione transitoria ciclo DNA mediante FRET (Fluorescence Resonance Energy Transfer). molecole di dsDNA sono costruiti utilizzando un protocollo basato su PCR semplice con un paio FRET e un linker biotina. La densità di probabilità loop noto come fattore J viene estratto dal prezzo loop e il tasso di ricottura tra due sezionatoreTed estremità appiccicose. Testando due dsDNAs con curvature diverse intrinseche, mostriamo che il fattore J è sensibile alla forma intrinseca del dsDNA.
Introduzione
Comprendere le proprietà meccaniche di dsDNA è di fondamentale importanza nelle scienze di base e applicazioni di ingegneria. La struttura di dsDNA è più complicato di una scala elicoidale dritto, perché angoli di rollio, inclinazione e torsione tra coppie di basi successive possono variare con la sequenza. Fluttuazioni termiche possono causare dsDNA di sottoporsi a diversi modi di fluttuazioni conformazionali come la flessione, torsione e stretching. Transizioni come fusione e attorcigliamenti possono verificarsi anche in condizioni estreme.
Tra questi movimenti, dsDNA flessione ha l'impatto biologico più evidente 1. dsDNA piegatura è associato repressione genica o attivazione portando due siti distanti vicini tra loro. Esso gioca un ruolo importante nella confezione DNA all'interno del nucleo cellulare o un capside virale. Deformazione piegatura di dsDNA possono essere visualizzati sperimentalmente microscopia ad alta risoluzione (AFM 2 e 3 TEM), e il Thermodynamics e cinetiche possono essere studiati mediante saggi di looping, che legano chimicamente siti giustapposte del dsDNA.
Un tale saggio è ligasi-dipendente ciclizzazione 4. In questo saggio, le molecole di dsDNA con (coesivi) estremita 'adesive' sono circolarizzato o dimerizzate da DNA ligasi. Confrontando i tassi di cerchio e la formazione di dimeri, si può ottenere una concentrazione molare efficace di una delle estremità del DNA in prossimità dell'altra estremità, che è conosciuto come il fattore J. Questo fattore J è dimensionalmente equivalente alla densità di probabilità di trovare una estremità del DNA a breve distanza dall'altra estremità, e riflette quindi la flessibilità del DNA. Misurare il fattore J in funzione della lunghezza del DNA rivela molte caratteristiche di meccanica DNA compresa la lunghezza di persistenza 4,5.
La catena vermiforme (WLC) modello è stato ampiamente considerato come il modello di polimero canonico per la meccanica dsDNA in base al suo successo nel spiegazioneining le curve forza-estensione ottenuti in DNA tirare esperimenti 6, e correttamente predire i fattori di J dsDNAs più di 200 bp 7. Tuttavia, utilizzando il test ciclizzazione su molecole dsDNA più brevi 100 bp, Cloutier e Widom misurato i fattori J di essere diversi ordini di grandezza superiori rispetto al modello WLC previsione 8. Un anno dopo, Du et al. prodotta fattori J in accordo con il modello WLC utilizzando il test ciclizzazione con concentrazioni più basse di ligasi e attribuito il risultato anomalo dal gruppo Widom ad alte concentrazioni ligasi utilizzati 9. Questa controversia esemplifica l'influenza inevitabile di DNA ligasi sulla cinetica di ciclizzazione quando si utilizza il test convenzionali 9. Inoltre, DNA ligasi può anche influenzare la struttura del DNA e la rigidità attraverso legame non specifico 10,11.
Per eliminare le preoccupazioni tecniche di analisi looping proteina-dipendente, abbiamo recentemente dimostrato una prottest looping ein-libero basato sul Fluorescence Resonance Energy Transfer (FRET) 12. In questo metodo, conformazioni loop vengono rilevati dal FRET tra donatore e accettore divisoria in prossimità delle estremità appiccicose di una molecola di DNA. Un microscopio a fluorescenza a riflessione interna totale obiettivo di tipo (TIRFM) viene utilizzato per registrare traiettorie di ciclaggio reversibile e eventi unlooping di molecole di DNA a singola superficie immobilizzato per un periodo prolungato di tempo. Questo metodo offre assemblaggio basato su PCR di molecole di DNA per generare molecole di DNA senza disadattamento, che è un miglioramento fondamentale per un metodo simile per Vafabakhsh e Ha 13. L'aspetto unico molecola di questo protocollo permette misura delle distribuzioni oltre Ensemble medie mentre l'aspetto FRET permette di misurare la dinamica di ciclica DNA ripetutamente dalla stessa molecola, anche in condizioni che possono alterare ligasi attività.
La configurazione TIRFM è mostrato in Figura 1. A personalizzatoPortaoggetti progettato è posto sopra un corpo del microscopio Olympus IX61. 532 nm e 640 nm laser vengono introdotti dal lato e sono riflessi da specchi ellittici minuscoli 14 nella alta obiettivo NA per ottenere l'angolo di incidenza critico all'interfaccia coprioggetto-acqua. Notiamo che più diffusa passante obiettivo TIR utilizzando specchi dicroici o configurazioni TIR basato prisma può essere utilizzato anche per questa applicazione FRET. L'immagine di fluorescenza formata dal microscopio è suddiviso in immagini donatore e accettore da uno specchio dicroico. Vengono poi ri-masterizzata su due metà di un EMCCD. Ulteriori filtri di emissione passa-lungo sono utilizzati per ridurre segnale di fondo.
Il controllo della temperatura è essenziale per acquisire dati cinetici riproducibili. Per il controllo della temperatura, l'obiettivo è separato dal boccaglio del corpo del microscopio per ridurre al minimo il trasferimento di calore, e l'acqua da un raffreddatore / riscaldatore di temperatura controllata è distribuito attraverso un collare di ottone che si adatta strettamenteattorno al metallo interno sotto la giacca obiettivo. Questa configurazione è in grado di ottenere il controllo della temperatura solido a superficie coprioggetto tra 15 e 50 ° C (Figura 2). In questo lavoro, la temperatura del campione è stata mantenuta a 24 ° C.
Il protocollo seguente presenta la procedura step-by-step per la costruzione del DNA, la stima di forma del DNA, esperimento di singola molecola, e J determinazione fattore.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Preparazione dsDNA campione
- Progettare DNA curve globalmente ripetendo una sequenza di 10-mer. Ad esempio, 5'-GTGCCAGCAACAGATAGC - (TTTATCATCCTTTATCATCC X) 7 - TTTCATTCGAGCTCGTTGTTG-3 'è un DNA curvo 186-bp, dove X è una base supplementare casuale e la sequenza fiancheggiante la sequenza ripetuta 10-mer sono sequenze adattatore.
NOTA:. In questo esempio 15 sono stati scelti due 10-mers con preferenze opposte alla formazione dei nucleosomi sulla base di una larga scala studio occupazione nucleosomi da Kaplan et al. Poiché la ripetizione dell'elica di dsDNA è vicino a 10 bp, qualsiasi deviazione netto dell'asse elicoidale del 10-mer si accumulerà per produrre una forma ad arco di cerchio (Figura 3A). Poiché il periodo elicoidale è più vicino a 10,5 bp, una base supplementare è inserito dopo ogni due ripetizioni di mantenere la struttura curva come planare possibile. Queste sequenze più brevi di 200 bp possono essere ordinati da companies che offrono gene servizio di sintesi. E 'conveniente per affiancare queste sequenze con sequenze adattatori comuni per i passi successivi. La procedura è schematizzata in Figura 3B. - Eseguire la PCR con Primer 1 (GTGCCAGCAACAGATAGC) e Primer 2 (/ 5Cy3/TAAATTCCTACAACAACGAGCTCGAATG). NOTA: Primer 2 è etichettato con il Cy3 donatore FRET all'estremità 5 '. Una tipica ricetta PCR e protocollo di ciclismo sono presentati nelle tabelle 1 e 2.
- Eseguire la PCR con Primer 3 (/ 5BioTEG/GAAACATAG/iCy5/GAATTTACCGTGCCAGCAACAGATAGC) e Primer 4 (CAACAACGAGCTCGAATG). NOTA: Primer 3 è etichettato con l'accettante Cy5 FRET attraverso la spina dorsale e la biotina-linker all'estremità 5 '. PCR ricetta e il protocollo ciclismo sono come sopra.
- Purificare i prodotti di PCR utilizzando un kit di pulizia PCR.
- Mescolare il prodotto Cy3 marcato e il prodotto Cy5 marcato in un buffer di scambio strand (NaCl 100 mM, 10 mM Tris-HCl pH 7,0, 1 mM EDTA) a concentrazioni finali di 0,4 &# 181, M e 0,1 micron, rispettivamente. NOTA: L'eccesso Cy3-DNA aumenta la concentrazione del duplex trasportare sia Cy3 e Cy5 per effetto dello scambio filamento.
- Scambio trefoli incubando a 98,5 ° C per 2 min, gradualmente raffreddamento a 5 ° C con velocità di rampa di 0,1 ° C / sec, e incubando a 5 ° C per 2 ore.
2. Elettroforesi su gel per rilevare dsDNA curvatura
- Versare il gel poliacrilammide 16,17 mescolando acrilammide e soluzioni bis-acrilammide a 29.2:0.8 rapporto, 5% (w / v) in 1X Tris / Borato / EDTA (TBE) tampone a pH 8,0. Nota: 10 ml di soluzione di gel contiene: 1.217 ml 40% di acrilammide, 0.667 ml 2% bis-acrilammide, 1 ml di TBE 10X, 100 L persolfato di ammonio (APS) 10%, 10 L TEMED, e il resto è dH 2 O. Solidificazione completa del gel dura circa 30 min.
- Caricare il gel di poliacrilammide con campioni di DNA e il DNA ladder in 1X tampone di caricamento (5% glicerolo, 0,03% (w / v) di bromofenolo blue) ed eseguire il gel a 5-8 V / cm a 4 ° C per 45 minuti o fino a quando il fronte del colorante si avvicina alla fine del gel.
- Macchiare il gel utilizzando tampone 1X TBE contenente 0,5 g / ml di etidio bromuro per 30 min. Identificare le bande di DNA sotto illuminazione UV. Confronta le posizioni di banda con il marcatore di formato (100 bp DNA ladder) per calcolare le dimensioni apparenti delle molecole di DNA. NOTA: curvo DNA generalmente si muovono più lentamente rispetto DNA dritto.
3. Flusso Preparazione cellulare
- Praticare 6-7 coppie di fori lungo due lati opposti di un vetrino (3'' x 1'') utilizzando un trapano a colonna e punte di diamante. Dopo la foratura, strofinare lo scivolo in acqua corrente per eliminare polvere di vetro visibile. NOTA: I fori servono come ingressi di perfusione e punti vendita. Durante la perforazione, raffreddando il vetrino con acqua è importante per evitare fessurazioni.
- Porre i vetrini in posizione verticale in un barattolo di vetro e riempirlo con acqua. Ultrasuoni per 15 min e trasferirli in un altro vaso di vetro dedicated per pulizia acetone. Riempire con acetone e ultrasuoni per 15 min. Sciacquare i vetrini con etanolo utilizzando un flacone spray e poi con acqua. Metterli in un barattolo di polipropilene, riempirlo con idrossido di potassio 5 M, e ultrasuoni per 15 min. Infine, sonicare i vetrini in acqua per 15 minuti. Pulire i coprioggetti (n. 1, 24 x 40 mm) utilizzando lo stesso protocollo. NOTA: diapositive e vetrini puliti possono essere memorizzati in dH 2 O per l'uso a lungo termine.
- Mescolare 1 mg di biotina-PEG-silano (MW 3400) con 80 mg di MPEG-silano (MW 2000) in 340 L di soluzione di bicarbonato 0,1 M di sodio. Mescolare bene e centrifugare la miscela brevemente per sbarazzarsi di bolle. NOTA: La funzionalizzazione della superficie con polietilene glicole (PEG) aiuta a ridurre il legame non specifico del DNA alla superficie.
- Mettere 80 L della soluzione di PEG in ogni diapositiva e abbassare delicatamente un coprioggetto su di esso. Attendere 45 min. Separare il vetrino dalla diapositiva con le pinzette, sciacquare con abbondante dH 2 O e lasciare torlo asciugare all'aria aperta.
- Mettere sottili strisce di nastro biadesivo in tutta la diapositiva di formare canali. Allineare un coprioggetto su di esso e premere con decisione il vetrino contro la diapositiva di formare canali a tenuta stagna. Utilizzare 5 minuti epossidica per sigillare i bordi dei canali.
4. Preparazione della Soluzione Trolox
- Mettere ~ 30 mg Trolox e 10 ml di dH 2 O in un pallone e utilizzare una barra di magnete scalpore per mescolare la soluzione all'aria aperta per 18 ore.
- Filtrare la soluzione con un filtro da 0,2 micron e regolare il pH a 7 con l'aggiunta di ~ 6 microlitri di 1 base M Tris (pH 11). Nota: Trolox è un reagente anti-lampeggiante che viene comunemente usato in singola molecola studi 18. L'azione antiscolorimento di Trolox deriva dal suo derivato ossidato che è presente in parzialmente degradata soluzione Trolox 19. Sciogliendo rapidamente Trolox in metanolo o ad alto pH della soluzione Tris deve essere evitato a causa dell'ossidazione inefficiente.
5. Single-molImaging ecule
- Iniettare 15 ml di soluzione NeutrAvidin (0,5 mg / ml) nel canale e attendere 2 minuti prima di risciacquare con 100 microlitri di tampone T50 (10 mM Tris-HCl, 50 mM NaCl, pH 7,0).
- Iniettare 50 ml di campione di DNA (50-100 pM) nel canale. Attendere 5 minuti e risciacquare il DNA non legato via con 100 L di tampone T50. NOTA: molecole di DNA saranno specificatamente legarsi alla superficie attraverso l'interazione NeutrAvidin-biotina.
- Riempire il canale con il buffer di imaging che contiene un sistema che assorbe ossigeno 20 (PCD 100 mM, 5 mM PCA, 1 Trolox mM e NaCl 500 mM).
- Impostare la EMCCD in modalità a trasferimento di quadro per lo streaming di 2 x 2 immagini categorizzate (256 x 256) al computer a 25 fotogrammi al secondo.
- Mettete l'olio per immersione sull'obiettivo microscopio, e fissare la cella di flusso sul palco microscopio utilizzando clip campioni. Coarse-regolare il fuoco osservando il modello riflessione laser sulla parete. Fine-regolare la messa a fuoco utilizzando la visualizzazione in tempo reale della fluorescenza imetà sul monitor.
- Iniziare acquisizione dati con il laser 532 nm su. Controllare il live view e regolare la messa a fuoco, se necessario. Smettere di acquisizione dati quando la maggior parte delle molecole sono fotodecolorate.
NOTA: In questa struttura, viene utilizzato un programma-lab scritta C per controllare il microscopio e visualizzare le immagini in diretta sul monitor in quanto vengono salvati sul disco rigido.
6. Elaborazione delle immagini e analisi dei dati
NOTA: Una serie temporale di 256 x 256 le immagini vengono elaborate da un codice MATLAB per generare singola molecola tempo tracce di Cy3 e Cy5 intensità. Per accoppiare pixel tra il canale donatore e accettore canale dell'immagine split-view, 6-7 coppie di Cy3 e Cy5 macchie, ciascuna coppia dalla stessa molecola uniformemente dispersa attraverso il campo di vista, vengono raccolte manualmente, e una trasformazione affine viene calcolato utilizzando le coordinate di questi punti come punti di ancoraggio.
- Utilizzo di uno script MATLAB, guardare attraverso tutti i tempi singola molecola tracce tcappello mostrano più transizioni tra i segnali alte e basse tasto. Identificare gli stati in loop e in loop.
NOTA: Il segnale FRET è definita come l'intensità Cy5 (I a) diviso per la somma di Cy3 e Cy5 intensità (I a + I d). Stato Looped ha un alto valore FRET mentre lo stato in loop ha un valore basso FRET. L'istogramma dei segnali FRET da una singola molecola è bimodale distribuito a causa di looping reversibile e unlooping. - Trova la soglia che separa le due distribuzioni determinando l'intersezione tra le due curve gaussiane muro.
- Calcolare l'efficienza FRET come
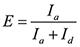 e assegnare gli stati ansa con alti valori di FRET e gli stati in loop con valori bassi FRET.
e assegnare gli stati ansa con alti valori di FRET e gli stati in loop con valori bassi FRET. - Utilizzo di uno script MATLAB, analizzare il numero cumulativo di molecole (N (t)), che in loop (oraggiunto lo stato alto FRET) a diversi time-lapse da inizio acquisizione dati. NOTA: Dal momento che una molecola di dsDNA può iniziare in qualsiasi conformazione dello stato in loop all'inizio di acquisizione dati, il tasso di aumento della popolazione loop riflette il tasso medio di ciclo media di oltre conformazioni iniziali. Estrarre il looping ciclo tasso k montando N (t) con una funzione esponenziale:
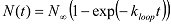 Se aumenta bifasico, può essere dotato di una doppia funzione esponenziale:
Se aumenta bifasico, può essere dotato di una doppia funzione esponenziale: 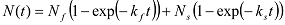 In questo caso, k ciclo è ottenuta da:
In questo caso, k ciclo è ottenuta da: 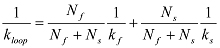 NOTA: Teoricamente, la probabilità di sopravvivenza che un polimero non ha loop al tempo t non è una singola o doppia funzione esponenziale 12. FITT esponenzialeING è usato come un mezzo pratico per estrarre il tempo medio looping.
NOTA: Teoricamente, la probabilità di sopravvivenza che un polimero non ha loop al tempo t non è una singola o doppia funzione esponenziale 12. FITT esponenzialeING è usato come un mezzo pratico per estrarre il tempo medio looping.
7. Determinazione del fattore J
NOTA: Il fattore J rappresenta come concentrato un'estremità di un dsDNA è intorno all'altra estremità. Può essere determinato interpolando la concentrazione di una estremità del segmento di DNA che produrrebbe la stessa velocità di reazione con l'altra estremità segmento come tasso loop misurata. Sperimentalmente, un segmento finale viene immobilizzato sulla superficie, e l'altra estremità segmento viene introdotto ad una certa concentrazione c. Se il tasso di ricottura misurata tra le due estremità è k ricottura, allora il fattore J 21 è dato da 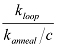 . La costante di velocità di ricottura (k = k ricottura ricottura / C) è indipendente dalla concentrazione sonda.
. La costante di velocità di ricottura (k = k ricottura ricottura / C) è indipendente dalla concentrazione sonda.
- Flusso 20 l di 30-50 pM biotina-Cy5 oligo (Primer 3) into un canale NeutrAvidin rivestita. Sciacquare il canale con 100 microlitri T50 per lavare via oligo non legato.
- Preparare il tampone di imaging come descritto nella parte 5 con l'aggiunta di Cy3 oligo (Primer 2) ad una concentrazione finale di 50 nM. Flusso questo buffer di imaging nel canale.
- Pur mantenendo il laser 532 nm su, girare brevemente il laser 640 nm a identificare le posizioni di associazione superficie Cy5 oligo. Spegnere il laser 640 nm e iniziare a monitorare il segnale FRET.
NOTA: Su ibridazione di un oligo Cy3 ad un Cy5 oligo-bound superficie, Cy5 fluorescenza derivare da FRET. - Utilizzo di uno script MATLAB, analizzare il numero di molecole che si aprono nello stato non legato (bassa intensità Cy5) ma poi si trasformano in stato (intensità alta Cy5) ricotto in funzione del tempo per l'intensità tracce Cy5.
- Tracciare questo numero di molecole vs tempo ricottura. Montare questa curva con una singola funzione esponenziale (
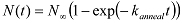 ) Per ottenere il tasso di ricottura (k tempra).
) Per ottenere il tasso di ricottura (k tempra). - Ripetere questo esperimento a diverse concentrazioni Cy3 oligo (60, 100 e 180 nm) per confermare la linearità tra il tasso di ricottura e la concentrazione reagente. Estrarre la costante di velocità di ricottura secondo ordine (k tempra ) Dal pendio.
- Calcolare il fattore J da
 , Dove k ciclo è il tasso loop misurata nelle stesse condizioni buffer.
, Dove k ciclo è il tasso loop misurata nelle stesse condizioni buffer.
Access restricted. Please log in or start a trial to view this content.
Risultati
Molecole di DNA utilizzati per lo studio loop consistono in una regione duplex di sequenza e lunghezza variabile e sporgenze a singolo filamento che sono complementari tra loro. Le sporgenze, che sono lunghe 7-base, possono ricombinarsi tra loro per acquisire lo stato in loop. Ogni sbalzo contiene sia Cy3 o Cy5 che è collegato nella spina dorsale del DNA attraverso la chimica amidite. Il Cy5-sporgenza è anche collegata con biotina-TEG (15-atomo Tetra-glicole etilenico distanziale) per l'immobilizzazione di superfi...
Access restricted. Please log in or start a trial to view this content.
Discussione
Un semplice test single-molecule basato su FRET è stato usato per studiare la cinetica di loop di DNA di diverse forme intrinseche. DNA curve possono essere preparati ripetendo una sequenza di 10-mer in fase con il periodo elicoidale di 10,5 bp, e le curvature possono essere stimati usando PAGE. Questi dsDNAs sono progettati con le estremità adesive per permettere la stabilizzazione ciclo transitorio. Abbiamo estratto il tasso loop dalla crescita esponenziale del numero di molecole in loop nel tempo. La costante di ve...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano assenza di conflitti di interesse.
Riconoscimenti
Ringraziamo James Waters, Gable Wadsworth e Bo Broadwater per criticamente la lettura del manoscritto. Ringraziamo anche quattro revisori anonimi per fornire commenti utili. Noi riconosciamo il sostegno finanziario Georgia Institute of Technology, il Burroughs Wellcome Fund Award Career l'interfaccia scientifico, e la borsa di rete di ricerca studente dalla NSF Fisica dei sistemi viventi.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Small DNA FRAG Extract Kit-100PR | VWR | 97060-558 | |
| Acrylamide 40% solution 500 ml | VWR | 97064-522 | |
| Bis-acrylamide 2% (w/v) solution 500 ml | VWR | 97063-948 | |
| GeneRuler 100 bp DNA Ladder, 100-1,000 bp | Fermentas | SM0241 | |
| Mini Vertical PAGE System | VWR | 89032-300 | |
| Syringe filter 0.2 μm CS50 | VWR | A2666 | |
| Trolox | Sigma-Aldrich | 238813-1G | triplet state quencher |
| Protocatechuic acid (PCA) | Sigma-Aldrich | 08992-50MG | oxygen scavenging system |
| Protocatechuate 3,4-Dioxygenase (PCD) | Sigma-Aldrich | P8279-25UN | oxygen scavenging system |
| mPEG-silane, MW 2,000 1 g | Laysan Bio | MPEG-SIL-2000-1g | |
| Biotin-PEG-Silane, MW 3,400 | Laysan Bio | Biotin-PEG-SIL-3400-1g | |
| Avidin, NeutrAvidin Biotin-binding Protein | Invitrogen | A2666 | |
| Phusion Hot Start High-Fidelity DNA Polymerase | New England Biolabs | F-540L | |
| Gel/PCR DNA Fragments Extraction Kit | IBI Scientific | IB47020 | |
| Premium plain glass microscope slides | Fisher Scientific | 12-544-1 | |
| VWR micro cover glass, rectangular, no. 1 | VWR | 48404-456 | |
| Fisher Scientific Isotemp 1006S Recirculating Chiller/Heater | Fisher Scientific | temperature control | |
| Objective Cooling Collar | Bioptechs | 150303 | temperature control |
| KMI53 Biological Micrometer Measuring Stage | Semprex | KMI53 | |
| High Performance DPSS Laser 532 nm 50 mW | Edmund optics | NT66-968 | Cy3 excitation |
| CUBE Fiber Pigtailed 640 nm, 30 mW, Fiber, FC/APC Connector | Coherent | 1139604 | Cy5 excitation |
| 650 nm BrightLine Dichroic Beamsplitter | Semrock | FF650-Di01-25x36 | splitting dichroic |
| LaserMUX Beam Combiner, reflects 514.5, 532, & 543.5 nm lasers, 25 mm | Semrock | LM01-552-25 | combining dichroic |
| Brightline Fluorescence Filter 593/40 | Semrock | FF01-593/40-25 | Cy3 emission filter |
| 635 nm EdgeBasic LWP longpass Filter, 25 mm | Semrock | BLP01-635R-25 | Cy5 emission filter |
| EMCCD iXon+ | Andor Technology | DU-897E-CS0-#BV | |
| IX51 inverted microscope frame | Olympus | ||
| Objective UApo N 100X/1.49 Oil TIRF | Olympus | ||
| Immersion oil type-F for fluorescence microscopy | Olympus | IMMOIL-F30CC | |
| 2 mm Diameter 45° Rod Lens Aluminum Coated | Edmund optics | 54-092 | miniature mirror |
| 1/4" Travel Single-Axis Translation Stage | Thorlabs | MS-1 | translation of miniature mirror |
| Ø1" Achromatic Doublet, ARC: 400-700 nm, f=200 mm | Thorlabs | AC254-200-A | focusing lens |
| Adjustable Mechanical Slit | Thorlabs | VA100 | |
| Dielectric Mirror | Thorlabs | BB1-E02 | |
| Ø1" Achromatic Doublet, f = 100 mm | Thorlabs | AC254-100-A | relay lens |
| Lens Mount for Ø1" Optics | Thorlabs | LMR1 | |
| Dichroic Filter Mount | Thorlabs | FFM1 | |
| Fixed Cage Cube Platform | Thorlabs | B3C | |
| Kinematic Mount for Ø1" Optics | Thorlabs | KM100 | |
| N-BK7 Plano-Convex Lens, Ø1", f = 40 mm | Thorlabs | LA1422-A | collimating lens |
| N-BK7 Plano-Convex Lense, Ø6.0 mm, f = 15 mm | Thorlabs | LA1222-A | telescope lens |
| N-BK7 Plano-Convex Lense, Ø6.0 mm, f = 150 mm | Thorlabs | LA1433-A | telescope lens |
Riferimenti
- Garcia, H. G., et al. Biological consequences of tightly bent DNA: The other life of a macromolecular celebrity. Biopolymers. 85, 115-130 (2007).
- Wiggins, P. A., et al. High flexibility of DNA on short length scales probed by atomic force microscopy. Nature Nanotechnology. 1, (2006).
- Lionberger, T. A., et al. Cooperative kinking at distant sites in mechanically stressed DNA. Nucleic Acids Research. 41, 6785-6792 (2011).
- Shore, D., et al. DNA flexibility studied by covalent closure of short fragments into circles. Proc Natl Acad Sci U S A. 78, 4833-4837 (1981).
- Geggier, S., Vologodskii, A. Sequence dependence of DNA bending rigidity. Proc Nat Acad Sci U S A. 107, 15421-15426 (1992).
- Smith, S. B., et al. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258, 1122-1126 (1992).
- Peters, J. P., Maher, L. J. DNA curvature and flexibility in vitro and in vivo. Quarterly Reviews of Biophysics. 43, 23-63 (2010).
- Cloutier, T. E., Widom, J. Spontaneous sharp bending of double-stranded DNA. Molecular Cell. 14, 355-362 (2004).
- Du, Q., et al. Cyclization of short DNA fragments and bending fluctuations of the double helix. Proc Natl Acad Sci U S A. 102, 5397-5402 (2005).
- Yuan, C., et al. T4 DNA ligase is more than an effective trap of cyclized dsDNA. Nucl. Acids Res. 35, 5294-5302 (2007).
- Manzo, C., et al. The effect of nonspecific binding of lambda repressor on DNA looping dynamics. Biophysical Journal. 103, 1753-1761 (2012).
- Le, T. T., Kim, H. D. Measuring shape-dependent looping probability of DNA. Biophys. J. 104, 2068-2076 (2013).
- Vafabakhsh, R., Ha, T. Extreme bendability of DNA less than 100 base pairs long revealed by single-molecule cyclization. Science. 337, 1097-1101 (2012).
- Friedman, L., et al. Viewing dynamic assembly of molecular complexes by multi-wavelength single-molecule fluorescence. Biophysical Journal. 91, 1023-1031 (2006).
- Kaplan, N., et al. The DNA-encoded nucleosome organization of a eukaryotic genome. Nature. 458, 362-366 (2009).
- Koo, H. S., Crothers, D. M. Calibration of DNA curvature and a unified description of sequence-directed bending. Proc Nat Acad Sci U S A. 85, 1763-1767 (1988).
- Prosseda, G., et al. A temperature-induced narrow DNA curvature range sustains the maximum activity of a bacterial promoter in vitro. Biochemistry. 49, 2778-2785 (2010).
- Rasnik, I., et al. Nonblinking and long-lasting single-molecule fluorescence imaging. Nature Methods. 3, 891-893 (2006).
- Cordes, T., et al. On the mechanism of Trolox as antiblinking and antibleaching reagent. J. Am. Chem. Soc. 131, 5018-5019 (2009).
- Aitken, C. E., et al. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophys J. 94, 1826-1835 (2008).
- Taylor, W. H., Hagerman, P. J. Application of the method of phage T4 DNA ligase-catalyzed ring-closure to the study of DNA structure: II. NaCl-dependence of DNA flexibility and helical repeat. Journal of Molecular Biology. 212, 363-376 (1990).
- Bolshoy, A., et al. Curved DNA without A-A: experimental estimation of all 16 DNA wedge angles. Proc Natl Acad Sci U S A. 88, 2312-2316 (1991).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucl. Acids Res. 37, 6984-6990 (2009).
- Vologodskii, A., et al. Bending of short DNA helices. Artif DNA PNA XNA. 4, (2013).
- Hoover, D. M., Lubkowski, J. DNAWorks: an automated method for designing oligonucleotides for PCR-based gene synthesis. Nucl. Acids Res. 30, (2002).
- Waters, J. T., Kim, H. D. Equilibrium Statistics of a Surface-Pinned Semiflexible Polymer. Macromolecules. 46, 6659-6666 (2013).
- Mills, J. B., et al. Electrophoretic evidence that single-stranded regions of 1 or more nucleotides dramatically increase the flexibility of DNA. Biochemistry. 33, 1797-1803 (1994).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon