É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Estudar DNA Looping por Single-Molecule FRET
Neste Artigo
Resumo
Este estudo apresenta um procedimento experimental detalhado para medir a dinâmica de loop de DNA de cadeia dupla usando uma única molécula de fluorescência Resonance Energy Transfer (FRET). O protocolo também descreve como extrair a densidade de probabilidade looping chamado o factor de J.
Resumo
Dobrando de ADN de cadeia dupla (dsDNA) está associado com muitos processos biológicos importantes, tais como o reconhecimento de DNA-proteína e embalagem de ADN em nucleossomas. A termodinâmica da dobragem ADNcd foi estudado por um método chamado de ciclização que se baseia na ADN-ligase para se juntar covalentemente extremidades pegajosas curtas de um ADNcd. No entanto, a eficiência ligadura pode ser afetada por vários fatores que não estão relacionados com dsDNA looping, como a estrutura do DNA em torno das extremidades pegajosas unidas e ligase também pode afetar a taxa de looping aparente através de mecanismos como a ligação não específica. Aqui, vamos mostrar como medir dsDNA cinética looping sem ligase por detectar a formação transitória laço DNA por FRET (Fluorescence Resonance Energy Transferência). moléculas de dsDNA são construídos utilizando um protocolo baseado em PCR simples, com um par de FRET e um ligante de biotina. A densidade de probabilidade looping conhecido como o factor de J é extraído a partir da taxa de looping e a taxa de hibridação entre duas desconexãoted extremidades pegajosas. Ao testar dois dsDNAs com diferentes curvaturas intrínsecas, mostra-se que o factor de J é sensível à forma intrínseca do dsDNA.
Introdução
Compreender as propriedades mecânicas do dsDNA é de fundamental importância em ciências básicas e aplicações de engenharia. A estrutura do dsDNA é mais complicado do que uma escada helicoidal reto porque os ângulos de rolo, de inclinação e torção entre pares de bases sucessivas pode variar de acordo com seqüência. Flutuações térmicas podem causar dsDNA se submeter a diversos modos de flutuações conformacionais tais como flexão, torção e alongamento. Transições, como fusão e dobras também pode ocorrer em condições extremas.
Entre esses movimentos, dsDNA flexão tem o impacto biológico mais perceptível 1. dsDNA flexão está associada com a repressão ou a activação de genes, trazendo dois locais distantes próximos uns dos outros. Também desempenha um papel importante no empacotamento do ADN no interior do núcleo da célula ou uma cápside viral. Dobrando deformação de dsDNA pode ser visualizado experimentalmente por microscopia de alta resolução (AFM 2 e TEM 3), e o thermodynamics cinética e podem ser estudados por meio de ensaios de loop, que ligam quimicamente sítios justapostas do dsDNA.
Um tal ensaio é dependente da ligase de ciclização 4. Neste ensaio, as moléculas de dsDNA com (coesas) extremidades 'sticky' são circularizado ou dimerizada por DNA ligase. Ao comparar as taxas de círculo e a formação de dímero, pode-se obter uma concentração molar eficaz de uma das extremidades do DNA na vizinhança da outra extremidade, que é conhecido como o factor de J. Este factor de J é dimensionalmente equivalente à densidade de probabilidade de encontrar uma extremidade do ADN a uma curta distância a partir da outra extremidade, e, portanto, reflecte a flexibilidade do ADN. Medindo o factor J como uma função do comprimento do DNA revela várias características sobre a mecânica de ADN, incluindo o comprimento de 4,5 persistência.
A cadeia sem-fim-como modelo (WLC) tem sido amplamente considerado como o modelo de polímero canônico para a mecânica dsDNA com base em seu sucesso na explicaçãoIning as curvas de força-de extensão obtidos no ADN puxando as experiências 6 e predizer correctamente os factores J dsDNAs de mais do que 200 pb 7. No entanto, utilizando o ensaio de ciclização em moléculas de dsDNA tão curtos como 100 pb, Cloutier e Widom medidos os factores J ser várias ordens de magnitude maiores do que a previsão do modelo WLC 8. Um ano depois, Du et al. produzido factores J de acordo com o modelo WLC utilizando o ensaio de ciclização com concentrações mais baixas de ligase e atribuído o resultado anómalo a partir do grupo Widom a altas concentrações de ligase utilizada 9. Esta controvérsia exemplifica a influência inevitável de DNA-ligase em cinética de ciclização quando se utiliza o ensaio convencional 9. Além disso, a DNA ligase também pode afetar a estrutura do DNA e rigidez através inespecífica 10,11 vinculativo.
Para eliminar as preocupações técnicas de ensaios looping dependentes de proteína, que recentemente demonstrou um protensaio looping ein-livre baseado em fluorescência Resonance Energy Transfer (FRET) 12. Neste método, as conformações laçados são detectados por FRET entre o doador e receptor ligado perto das extremidades coesivas de uma molécula de DNA. Um total de microscópio de fluorescência de reflexão interna de tipo de objectivo (TIRFM) é utilizado para registar trajectórias de looping reversível e eventos unlooping de moléculas de DNA únicas imobilizado à superfície, por um período de tempo prolongado. Este método apresenta a montagem baseado em PCR de moléculas de ADN para gerar moléculas de ADN livre de desadaptação, o que é uma melhoria essencial ao longo de um método semelhante por Vafabakhsh e Ha 13. O aspecto de molécula única deste protocolo permite a medição da distribuição, para além Ensemble médias enquanto que o aspecto de FRET permite medir a dinâmica de looping de ADN repetidamente a partir da mesma molécula, mesmo em condições que possam prejudicar a actividade de ligase.
A configuração TIRFM é mostrado na Figura 1. Um costumeConcebido fase amostra é colocada sobre um corpo do microscópio Olympus IX61. 532 nm e 640 nm lasers são introduzidos a partir do lado e são reflectidos por pequenos espelhos elípticas 14 para o objectivo de NA elevado para atingir o ângulo crítico de incidência na interface lamela de água. Notamos que mais difundido através TIR-objectivo usando espelhos dicroicos ou configurações TIR-base de prisma também pode ser utilizado para esta aplicação de FRET. A imagem de fluorescência formada pelo microscópio é dividido em dadores e aceitadores de imagens por um espelho dicróico. Eles são, então, re-trabalhada em duas metades de um EMCCD. Filtros de emissão adicionais passa-longa são usados para reduzir o sinal de fundo.
O controle de temperatura é essencial para a aquisição de dados cinéticos reprodutíveis. Por controlo da temperatura, o objectivo é separada da peça de nariz do corpo do microscópio para minimizar a transferência de calor e de água a partir de uma temperatura controlada resfriador / aquecedor é feito circular através de uma gola de bronze que se encaixa firmementeem todo o interior metal embaixo do casaco objetivo. Esta configuração é capaz de alcançar o controlo robusto temperatura na superfície lamela entre 15 e 50 ° C (Figura 2). Neste trabalho, a temperatura da amostra foi mantida a 24 ° C.
O protocolo a seguir apresenta o procedimento passo-a-passo para a construção de DNA, a estimativa de forma DNA, molécula única experiência, e J determinação do fator.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparação dsDNA Amostra
- Projete DNAs globalmente curvas repetindo uma seqüência de 10-mer. Por exemplo, 5'-GTGCCAGCAACAGATAGC - (TTTATCATCCTTTATCATCC X) 7 - TTTCATTCGAGCTCGTTGTTG-3 'é um ADN curvo 186 pb, em que X é uma base aleatória adicional e a sequência que flanqueia a sequência de 10-mer de repetição são sequências de adaptador.
NOTA:. Neste exemplo, dois 10-mers com preferências opostas à formação nucleosome com base em um estudo de larga escala ocupação nucleosome por Kaplan et al 15 foram escolhidos. Uma vez que a repetição helicoidal de ADNcd é perto de 10 pb, qualquer desvio líquido do eixo helicoidal do 10-mer se acumulam para produzir uma forma como um arco circular (Figura 3A). Uma vez que o período helicoidal está mais perto de 10,5 pb, uma base adicional é inserido depois de cada duas repetições para manter a estrutura curva planar como possível. Essas seqüências mais curtas do que 200 pb pode ser encomendado a partir companies que o serviço de síntese de genes oferta. É conveniente para flanquear essas seqüências com seqüências de adaptadores comuns para as etapas subseqüentes. O procedimento é esquematizado na Figura 3B. - Realizar PCR com iniciador de síntese 1 (GTGCCAGCAACAGATAGC) e iniciador 2 (/ 5Cy3/TAAATTCCTACAACAACGAGCTCGAATG). NOTA: Iniciador 2 é marcado com o Cy3 dador FRET na extremidade 5 '. Uma receita típica de PCR e ciclismo protocolo, são apresentados nas Tabelas 1 e 2.
- Realizar PCR com Primer 3 (/ 5BioTEG/GAAACATAG/iCy5/GAATTTACCGTGCCAGCAACAGATAGC) e Primer 4 (CAACAACGAGCTCGAATG). NOTA: Iniciador 3 é marcado com o aceitador FRET Cy5 através da espinha dorsal e a biotina-ligante na extremidade 5 '. Receita PCR e ciclismo protocolo são como acima.
- Purifica-se os produtos de PCR utilizando um kit de limpeza de PCR.
- Misture o produto Cy3 marcado e o produto marcado com Cy5 em um tampão para a troca de fio (100 mM de NaCl, 10 mM Tris-HCl pH 7,0, EDTA 1 mM) em concentrações finais de 0,4 e# 181; M e 0,1 M, respectivamente. NOTA: O excesso Cy3 ADN aumenta a concentração do duplex que transporta tanto Cy3 e Cy5 como resultado da troca de fio.
- Taxas de fios por incubação a 98,5 ° C por 2 min, arrefecimento gradual para 5 ° C com uma taxa de rampa de 0,1 ° C / seg, e incubar em 5 ° C durante 2 horas.
2. Eletroforese em Gel para detectar dsDNA Curvatura
- Despeje a gel de poliacrilamida a 16,17 por mistura de acrilamida e soluções de bis-acrilamida numa proporção de 29.2:0.8, 5% (w / v) em 1X Tris / borato / EDTA (TBE), tampão a pH 8,0. Nota: 10 ml de solução de gel contém: 1,217 ml de 40% de acrilamida, 0,667 ml de 2% de bis-acrilamida, 1 ml de TBE 10X, 100 L de persulfato de amónio (APS) 10%, 10 l de TEMED, e o resto é dH2O Solidificação completa do gel leva cerca de 30 min.
- Carregar o gel de poliacrilamida com amostras de DNA e a escada de DNA em 1X tampão de carga (5% de glicerol, 0,03% (w / v) bl bromofenolue) e correr o gel a 5-8 V / cm a 4 ° C durante 45 minutos ou até a frente do corante aproxima-se da extremidade do gel.
- Mancha do gel utilizando tampão 1X TBE contendo brometo de 0,5 g / ml de etídio durante 30 min. Identificar as bandas de DNA sob iluminação UV. Comparar as posições da banda com o marcador de tamanho (escada de 100 pb de ADN) para calcular os tamanhos aparentes das moléculas de DNA. NOTA: Curved DNAs geralmente se movem mais lentamente do que DNAs retas.
3. Caudal Preparação das células
- Fure 6-7 pares de furos ao longo de dois lados opostos de uma lâmina de vidro (3'' x 1'') usando uma furadeira e brocas de diamante. Após a perfuração, esfregue o slide em água corrente para remover o pó de vidro visível. NOTA: Os furos servem como entradas e saídas de perfusão. Durante a perfuração, o arrefecimento da lâmina com água é importante para evitar a fissuração.
- Colocar as lâminas na posição vertical em uma jarra de vidro e encha-o com água. Sonicar por 15 min e transferi-los para outra jarra de vidro dedicated para limpeza acetona. Encha-o com acetona e sonicado por 15 min. Lavar as lâminas com etanol usando um borrifador e depois com água. Coloque-os em um frasco de polipropileno, preenchê-lo com 5 M de hidróxido de potássio e sonicado por 15 min. Finalmente, sonicate as lâminas em água por 15 min. Limpar as lamelas (# 1, 24 x 40 mm), utilizando o mesmo protocolo. NOTA: lâminas e lamínulas limpos podem ser armazenados em dH 2 O para uso a longo prazo.
- Mistura-se 1 mg de biotina-PEG-silano (MW 3400), com 80 mg de mPEG-silano (MW 2000) em 340 L de solução de bicarbonato de sódio 0,1 M. Misture bem e centrifugar a mistura rapidamente para se livrar das bolhas. NOTA: A funcionalização da superfície de polietileno glicol (PEG) ajuda a reduzir a ligação não específica de ADN na superfície.
- Coloque 80 L da solução de PEG em cada slide e delicadamente abaixar uma lamela sobre ele. Aguarde 45 min. Separe a lamela do slide com uma pinça, lave-os com uma quantidade abundante de dH 2 O e deixe them secar ao ar livre.
- Coloque tiras finas de fita dupla-pau em todo o slide para formar canais. Alinhe uma lamela sobre ele e pressione firmemente o lamela contra o slide para formar canais estanques. O uso de 5 minutos de epóxi para vedar as extremidades dos canais.
4. Preparação de Solução de Trolox
- Coloque ~ 30 mg de Trolox e 10 ml de dH2O num balão e usar uma barra magnética de agitação para agitar a solução ao ar livre durante 18 horas.
- Filtrar a solução através de um filtro de 0,2 um e ajustar o pH a 7 por adição de ~ 6 ul de base Tris 1 M. (pH 11). Nota: Trolox é um reagente anti-piscar, que é comumente usado em uma única molécula estuda 18. A acção antifading de Trolox vem de um seu derivado oxidado, que está presente na solução de Trolox parcialmente degradado 19. Dissolver rapidamente Trolox em metanol ou uma solução de pH elevado Tris devem ser evitadas por causa da oxidação ineficaz.
5. Single-molImagem ecule
- Injectar 15 mL de solução de NeutrAvidin (0,5 mg / ml) para dentro do canal e esperar durante 2 min antes da lavagem com 100 mL de tampão de T50 (10 mM Tris-HCl, 50 mM de NaCl, pH 7,0).
- Injectar 50 mL de amostra de DNA (50-100 pM) para dentro do canal. Aguarde 5 minutos e enxaguar o DNA não ligado afastado com 100 L de tampão T50. NOTA: as moléculas de ADN irá ligar-se especificamente à superfície através da interacção biotina-NeutrAvidin.
- Encher o canal com o tampão de imagem que contém um sistema de eliminação de oxigénio 20 (PCD 100 mM, APC 5, Trolox 1 mM, e NaCl 500 mM).
- Defina o EMCCD no modo de transferência de quadros para transmitir 2 x 2 imagens Guardado (256 x 256) para o computador a 25 quadros por segundo.
- Coloque o óleo de imersão sobre a objetiva do microscópio, e fixar a célula de fluxo no palco microscópio usando clipes de amostras. Grosseiro-ajustar o foco, olhando para o padrão de reflexão do laser na parede. Fine-ajustar o foco usando a exibição ao vivo de fluorescência imidades no monitor.
- Comece a aquisição de dados com o laser de 532 nm, em. Verifique a visualização ao vivo e ajustar o foco, se necessário. Pare de aquisição de dados quando a maioria das moléculas têm Foto-descoloração.
NOTA: Nesta instalação, um programa escrito em laboratório C para controlar o microscópio e exibir imagens ao vivo no monitor como eles são salvos no disco rígido é usado.
6. Processamento de Imagem e Análise de Dados
NOTA: A série temporal de 256 x 256 imagens são processadas por um código MATLAB para gerar traços de Cy3 e Cy5 intensidades tempo de moléculas individuais. Para emparelhar pixels entre o canal doador eo canal receptor da imagem split-view, 6-7 pares de Cy3 e Cy5 pontos, cada par de uma mesma molécula uniformemente dispersas em todo o campo de visão, são colhidos manualmente, e uma transformação afim é calculada usando as coordenadas desses pontos como pontos de ancoragem.
- Usando um script MATLAB, olhar através de todos os tempos uma única molécula traça tchapéu mostra várias transições entre os sinais de baixa e alta traste. Identificar os estados em loop e unlooped.
NOTA: O sinal de FRET é definida como a intensidade de Cy5 (Ia) dividido pela soma de Cy3 e Cy5 intensidades (I-a + I d). Estado loop tem alto valor FRET enquanto estado unlooped tem baixo valor FRET. O histograma dos sinais de FRET uma única molécula é bimodalmente distribuídas por causa de looping reversível e unlooping. - Encontre o limiar que separa as duas distribuições, determinando a intersecção entre as duas curvas de Gauss embutidos.
- Calcule a eficiência FRET como
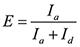 e atribuir os estados em loop com altos valores de FRET e os estados unlooped com baixos valores de traste.
e atribuir os estados em loop com altos valores de FRET e os estados unlooped com baixos valores de traste. - Usando um script MATLAB, analisar o número acumulado de moléculas (N (t)) que dava (ouatingiu o elevado estado FRET) em diferentes lapsos de tempo desde o início da aquisição de dados. Observação: Uma vez que uma molécula de dsDNA pode começar em qualquer conformação do estado unlooped no início da aquisição de dados, a taxa de aumento na população anelada reflete a taxa de looping média média mais conformações iniciais. Extraia o looping ciclo taxa k ajustando N (t) com uma função exponencial:
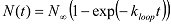 Se aumenta bifasicamente, ele pode ser equipado com uma função exponencial dupla:
Se aumenta bifasicamente, ele pode ser equipado com uma função exponencial dupla: 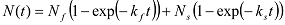 Neste caso, k é obtido a partir de ciclo:
Neste caso, k é obtido a partir de ciclo: 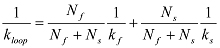 NOTA: Teoricamente, a probabilidade de sobrevivência que um polímero não tem loop no tempo t não é um simples ou dupla função exponencial 12. Fitt Exponencialing é usado como um meio prático para extrair o tempo médio de looping.
NOTA: Teoricamente, a probabilidade de sobrevivência que um polímero não tem loop no tempo t não é um simples ou dupla função exponencial 12. Fitt Exponencialing é usado como um meio prático para extrair o tempo médio de looping.
7. Determinação do Fator J
NOTA: O factor J representa como concentrado numa extremidade de um ADNcd é em torno da outra extremidade. Ele pode ser determinado por interpolação a concentração de um segmento de extremidade do DNA que produzir a mesma taxa de reacção com o outro segmento de extremidade como a taxa de looping medido. Experimentalmente, um segmento de extremidade é imobilizado sobre a superfície, e o outro segmento de extremidade é introduzido numa determinada concentração c. Se a taxa de recozimento medido entre as duas extremidades é k emparelhar, então o factor de J 21 é dada pela 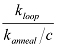 . A constante de velocidade de recozimento (k = k recozimento de emparelhamento / C) é independente da concentração da sonda.
. A constante de velocidade de recozimento (k = k recozimento de emparelhamento / C) é independente da concentração da sonda.
- Fluxo 20 l de 30-50 pM biotina-Cy5 oligo (Primer 3) into um canal NeutrAvidin-revestido. Lavar o canal com 100 T50 mL para lavar oligos não ligados.
- Preparar o tampão de formação de imagens conforme descrito na parte 5 com a adição de Cy3 oligo (iniciador 2) a uma concentração final de 50 nM. Fluxo este buffer de imagem para o canal.
- Mantendo o laser de 532 nm, gire rapidamente o laser de 640 nm para identificar os locais de oligos Cy5 ligado à superfície. Desligue o laser de 640 nm e começar a monitorar o sinal de FRET.
NOTA: Após a hibridização de um oligo Cy3 a um Cy5 oligo-bound superfície, Cy5 fluorescência surgirão FRET. - Usando um script MATLAB, analisar o número de moléculas que se iniciam no estado livre (baixa intensidade Cy5), mas mais tarde se transformar em Estado (alta intensidade de Cy5) recozido em função do tempo a partir de vestígios de intensidade Cy5.
- Traçar este número de moléculas em função do tempo recozidos. Coloque esta curva com uma única função exponencial (
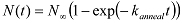 ) Para obter a taxa de recozimento (k recozimento).
) Para obter a taxa de recozimento (k recozimento). - Repetir esta experiência a diferentes concentrações de Cy3 oligo (60, 100, e 180 nm) para confirmar a linearidade entre a velocidade de recozimento e a concentração de reagente. Extrai-se a constante de velocidade de segunda ordem de recozimento (k recozimento ) A partir da inclinação.
- Calcular o factor de J
 , Onde k é a taxa de ciclo de looping medido nas mesmas condições de tampão.
, Onde k é a taxa de ciclo de looping medido nas mesmas condições de tampão.
Access restricted. Please log in or start a trial to view this content.
Resultados
As moléculas de ADN utilizadas para o estudo de looping consistem de uma região dúplex de sequência variável, e o comprimento e saliências em cadeia simples que sejam complementares uma da outra. As saliências, que são 7 bases de comprimento, pode emparelhar com o outro para capturar o estado anelada. Cada saliência contém ou Cy3 ou Cy5 que está ligado na espinha dorsal do DNA através da química amidite. O Cy5 saliência está também relacionada com biotina-TEG (15 átomos-Tetra-Etileno Glicol espaçador) ...
Access restricted. Please log in or start a trial to view this content.
Discussão
Um ensaio de uma única molécula simples baseado em FRET foi utilizado para estudar a cinética de looping de ADN de diferentes formas intrínsecas. DNAs curvos podem ser preparados por uma sequência de repetição de 10-mer em fase com o período helicoidal de 10,5 pb, e suas curvaturas pode ser estimada utilizando PAGE. Estes dsDNAs são projetados com extremidades pegajosas para permitir a estabilização laço transitória. Foram extraídos a taxa de looping do aumento exponencial do número de moléculas em loop ...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Agradecemos James Waters, Gable Wadsworth e Bo Broadwater pela leitura crítica do manuscrito. Agradecemos também a quatro revisores anônimos por fornecer observações úteis. Reconhecemos o apoio financeiro do Georgia Institute of Technology, o Prémio Carreira Burroughs Wellcome Fund na Interface Científica, ea pesquisa estudante concessão de rede de NSF Physics dos sistemas vivos.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Small DNA FRAG Extract Kit-100PR | VWR | 97060-558 | |
| Acrylamide 40% solution 500 ml | VWR | 97064-522 | |
| Bis-acrylamide 2% (w/v) solution 500 ml | VWR | 97063-948 | |
| GeneRuler 100 bp DNA Ladder, 100-1,000 bp | Fermentas | SM0241 | |
| Mini Vertical PAGE System | VWR | 89032-300 | |
| Syringe filter 0.2 μm CS50 | VWR | A2666 | |
| Trolox | Sigma-Aldrich | 238813-1G | triplet state quencher |
| Protocatechuic acid (PCA) | Sigma-Aldrich | 08992-50MG | oxygen scavenging system |
| Protocatechuate 3,4-Dioxygenase (PCD) | Sigma-Aldrich | P8279-25UN | oxygen scavenging system |
| mPEG-silane, MW 2,000 1 g | Laysan Bio | MPEG-SIL-2000-1g | |
| Biotin-PEG-Silane, MW 3,400 | Laysan Bio | Biotin-PEG-SIL-3400-1g | |
| Avidin, NeutrAvidin Biotin-binding Protein | Invitrogen | A2666 | |
| Phusion Hot Start High-Fidelity DNA Polymerase | New England Biolabs | F-540L | |
| Gel/PCR DNA Fragments Extraction Kit | IBI Scientific | IB47020 | |
| Premium plain glass microscope slides | Fisher Scientific | 12-544-1 | |
| VWR micro cover glass, rectangular, no. 1 | VWR | 48404-456 | |
| Fisher Scientific Isotemp 1006S Recirculating Chiller/Heater | Fisher Scientific | temperature control | |
| Objective Cooling Collar | Bioptechs | 150303 | temperature control |
| KMI53 Biological Micrometer Measuring Stage | Semprex | KMI53 | |
| High Performance DPSS Laser 532 nm 50 mW | Edmund optics | NT66-968 | Cy3 excitation |
| CUBE Fiber Pigtailed 640 nm, 30 mW, Fiber, FC/APC Connector | Coherent | 1139604 | Cy5 excitation |
| 650 nm BrightLine Dichroic Beamsplitter | Semrock | FF650-Di01-25x36 | splitting dichroic |
| LaserMUX Beam Combiner, reflects 514.5, 532, & 543.5 nm lasers, 25 mm | Semrock | LM01-552-25 | combining dichroic |
| Brightline Fluorescence Filter 593/40 | Semrock | FF01-593/40-25 | Cy3 emission filter |
| 635 nm EdgeBasic LWP longpass Filter, 25 mm | Semrock | BLP01-635R-25 | Cy5 emission filter |
| EMCCD iXon+ | Andor Technology | DU-897E-CS0-#BV | |
| IX51 inverted microscope frame | Olympus | ||
| Objective UApo N 100X/1.49 Oil TIRF | Olympus | ||
| Immersion oil type-F for fluorescence microscopy | Olympus | IMMOIL-F30CC | |
| 2 mm Diameter 45° Rod Lens Aluminum Coated | Edmund optics | 54-092 | miniature mirror |
| 1/4" Travel Single-Axis Translation Stage | Thorlabs | MS-1 | translation of miniature mirror |
| Ø1" Achromatic Doublet, ARC: 400-700 nm, f=200 mm | Thorlabs | AC254-200-A | focusing lens |
| Adjustable Mechanical Slit | Thorlabs | VA100 | |
| Dielectric Mirror | Thorlabs | BB1-E02 | |
| Ø1" Achromatic Doublet, f = 100 mm | Thorlabs | AC254-100-A | relay lens |
| Lens Mount for Ø1" Optics | Thorlabs | LMR1 | |
| Dichroic Filter Mount | Thorlabs | FFM1 | |
| Fixed Cage Cube Platform | Thorlabs | B3C | |
| Kinematic Mount for Ø1" Optics | Thorlabs | KM100 | |
| N-BK7 Plano-Convex Lens, Ø1", f = 40 mm | Thorlabs | LA1422-A | collimating lens |
| N-BK7 Plano-Convex Lense, Ø6.0 mm, f = 15 mm | Thorlabs | LA1222-A | telescope lens |
| N-BK7 Plano-Convex Lense, Ø6.0 mm, f = 150 mm | Thorlabs | LA1433-A | telescope lens |
Referências
- Garcia, H. G., et al. Biological consequences of tightly bent DNA: The other life of a macromolecular celebrity. Biopolymers. 85, 115-130 (2007).
- Wiggins, P. A., et al. High flexibility of DNA on short length scales probed by atomic force microscopy. Nature Nanotechnology. 1, (2006).
- Lionberger, T. A., et al. Cooperative kinking at distant sites in mechanically stressed DNA. Nucleic Acids Research. 41, 6785-6792 (2011).
- Shore, D., et al. DNA flexibility studied by covalent closure of short fragments into circles. Proc Natl Acad Sci U S A. 78, 4833-4837 (1981).
- Geggier, S., Vologodskii, A. Sequence dependence of DNA bending rigidity. Proc Nat Acad Sci U S A. 107, 15421-15426 (1992).
- Smith, S. B., et al. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258, 1122-1126 (1992).
- Peters, J. P., Maher, L. J. DNA curvature and flexibility in vitro and in vivo. Quarterly Reviews of Biophysics. 43, 23-63 (2010).
- Cloutier, T. E., Widom, J. Spontaneous sharp bending of double-stranded DNA. Molecular Cell. 14, 355-362 (2004).
- Du, Q., et al. Cyclization of short DNA fragments and bending fluctuations of the double helix. Proc Natl Acad Sci U S A. 102, 5397-5402 (2005).
- Yuan, C., et al. T4 DNA ligase is more than an effective trap of cyclized dsDNA. Nucl. Acids Res. 35, 5294-5302 (2007).
- Manzo, C., et al. The effect of nonspecific binding of lambda repressor on DNA looping dynamics. Biophysical Journal. 103, 1753-1761 (2012).
- Le, T. T., Kim, H. D. Measuring shape-dependent looping probability of DNA. Biophys. J. 104, 2068-2076 (2013).
- Vafabakhsh, R., Ha, T. Extreme bendability of DNA less than 100 base pairs long revealed by single-molecule cyclization. Science. 337, 1097-1101 (2012).
- Friedman, L., et al. Viewing dynamic assembly of molecular complexes by multi-wavelength single-molecule fluorescence. Biophysical Journal. 91, 1023-1031 (2006).
- Kaplan, N., et al. The DNA-encoded nucleosome organization of a eukaryotic genome. Nature. 458, 362-366 (2009).
- Koo, H. S., Crothers, D. M. Calibration of DNA curvature and a unified description of sequence-directed bending. Proc Nat Acad Sci U S A. 85, 1763-1767 (1988).
- Prosseda, G., et al. A temperature-induced narrow DNA curvature range sustains the maximum activity of a bacterial promoter in vitro. Biochemistry. 49, 2778-2785 (2010).
- Rasnik, I., et al. Nonblinking and long-lasting single-molecule fluorescence imaging. Nature Methods. 3, 891-893 (2006).
- Cordes, T., et al. On the mechanism of Trolox as antiblinking and antibleaching reagent. J. Am. Chem. Soc. 131, 5018-5019 (2009).
- Aitken, C. E., et al. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophys J. 94, 1826-1835 (2008).
- Taylor, W. H., Hagerman, P. J. Application of the method of phage T4 DNA ligase-catalyzed ring-closure to the study of DNA structure: II. NaCl-dependence of DNA flexibility and helical repeat. Journal of Molecular Biology. 212, 363-376 (1990).
- Bolshoy, A., et al. Curved DNA without A-A: experimental estimation of all 16 DNA wedge angles. Proc Natl Acad Sci U S A. 88, 2312-2316 (1991).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucl. Acids Res. 37, 6984-6990 (2009).
- Vologodskii, A., et al. Bending of short DNA helices. Artif DNA PNA XNA. 4, (2013).
- Hoover, D. M., Lubkowski, J. DNAWorks: an automated method for designing oligonucleotides for PCR-based gene synthesis. Nucl. Acids Res. 30, (2002).
- Waters, J. T., Kim, H. D. Equilibrium Statistics of a Surface-Pinned Semiflexible Polymer. Macromolecules. 46, 6659-6666 (2013).
- Mills, J. B., et al. Electrophoretic evidence that single-stranded regions of 1 or more nucleotides dramatically increase the flexibility of DNA. Biochemistry. 33, 1797-1803 (1994).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados