A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
לימוד ה-DNA Looping ידי סריג Single-מולקולה
In This Article
Summary
מחקר זה מציג הליך ניסיון מפורט כדי למדוד דינמיקת לולאות של גדילי הדנ"א כפול באמצעות פלואורסצנטי התהודה העברת אנרגיה מולקולה בודדת (סריג). הפרוטוקול מתאר גם כיצד לחלץ את צפיפות הסתברות הלולאה נקראת גורם J.
Abstract
כיפוף של גדילי הדנ"א כפול (dsDNA) קשור עם הרבה תהליכים ביולוגיים חשובים כגון זיהוי חלבון דנ"א ואריזת ה-DNA לתוך nucleosomes. תרמודינמיקה של כיפוף dsDNA נחקרה על ידי cyclization שיטה הנקראת אשר מסתמך על האנזים DNA להצטרף קוולנטית קצוות דביקים קצרים של dsDNA. עם זאת, יעילות קשירה יכולה להיות מושפעת מגורמים רבים שאינם קשורים לdsDNA לולאה כגון מבנה ה-DNA המקיף את הקצוות הדביקים הצטרפו, ואנזים יכול להשפיע גם על שיעור לולאות לכאורה באמצעות מנגנונים כגון ספציפי מחייב. כאן, אנו מראים כיצד למדוד קינטיקה לולאות dsDNA ללא האנזים על ידי איתור היווצרות ה-DNA לולאה חולפת ידי סריג (פלואורסצנטי התהודה העברת אנרגיה). מולקולות dsDNA נבנות באמצעות פרוטוקול המבוסס על ה-PCR פשוט עם זוג סריג ומקשר ביוטין. צפיפות הסתברות הלולאה הידועה כגורם J מופק שיעור לולאות ושיעור חישול בין שתי disconnecקצוות דביקים טד. על ידי בדיקת שתי dsDNAs עם עקמומיות פנימית שונה, אנו מראים כי גורם J הוא רגיש לצורה הפנימית של dsDNA.
Introduction
הבנת התכונות מכאניות של dsDNA היא בעל חשיבות עליונה במדעים בסיסיים ויישומים הנדסיים. המבנה של dsDNA הוא מסובך יותר מאשר סולם סליל ישר בגלל זוויות גלגול, הטיה ופיתול בין זוגות בסיסים רצופים יכולות להשתנות עם רצף. תנודות תרמיות יכולות לגרום dsDNA לעבור מצבים שונים של תנודות קונפורמציה כגון כיפוף, פיתול ומתיחות. מעברים כגון היתוך ומסתלסל יכולים להתרחש גם בתנאים קיצוניים.
בין תנועות אלה, יש כיפוף dsDNA ההשפעה הביולוגית הבולטת ביותר 1. כיפוף dsDNA קשור לדיכוי גן או הפעלה על ידי הבאת שני אתרים מרוחקים קרובים אחד לשני. זה גם משחק תפקיד חשוב באריזת ה-DNA בתוך גרעין התא או הקופסית נגיפית. עיוות כיפוף של dsDNA ניתן דמיינו באופן ניסיוני על ידי מיקרוסקופ ברזולוציה גבוהה (AFM 2 וTEM 3), וthermodynamics וקינטיקה ניתן ללמוד על ידי מבחני לולאות, אשר כימי קישור אתרים זה לצד זה של dsDNA.
assay אחד כזה הוא cyclization האנזים תלוי 4. ב assay זה, מולקולות dsDNA עם קצוות "דביקים" (מגובש) הן circularized או dimerized ידי האנזים ה-DNA. על ידי השוואת המחירים של המעגל והיווצרות דימר, ניתן לקבל ריכוז טוחנת יעיל של קצה אחד של ה-DNA בקרבת הקצה השני, אשר ידוע כגורם J. גורם J זה ממדי שווה לצפיפות הסתברות למציאת קצה אחד של ה-DNA במרחק קצר מהקצה השני, ובכך משקף את הגמישות של ה-DNA. מדידת גורם J כפונקציה של אורך ה-DNA מגלה מאפיינים רבים על מכניקת DNA כולל 4,5 אורך ההתמדה.
השרשרת דמוית תולעת המודל (WLC) כבר נחשב למודל הפולימר הקנונית למכניקת dsDNA מבוססת על הצלחתה בexplaining עקומות כוח ההארכה שהושגו ב-DNA מושך ניסויי 6, ובצורה נכונה לניבוי גורמי J של dsDNAs זמן רב יותר מאשר 200 נ"ב 7. עם זאת, באמצעות assay cyclization על מולקולות dsDNA קצרות ככל 100 נ"ב, קלוטייה וWidom מדדו את גורמי J להיות בכמה סדרי גודל גבוהים יותר מהתחזית של מודל WLC 8. שנה לאחר מכן, דו et al. מיוצר גורמי J בהסכם עם מודל WLC באמצעות assay cyclization עם ריכוזים נמוכים יותר של האנזים וייחס את התוצאה החריגה מקבוצת Widom לריכוזי האנזים גבוהים בשימוש 9. מחלוקת זו מדגימה את ההשפעה הבלתי נמנעת של האנזים ה-DNA על קינטיקה cyclization בעת שימוש assay הקונבנציונלי 9. יתר על כן, האנזים DNA יכול להשפיע גם על מבנה ה-DNA ונוקשות דרך 10,11 מחייב ספציפי.
כדי למנוע חששות הטכניים של מבחני לולאות חלבון תלוי, לאחרונה הפגינו protassay לולאות עין בחינם מבוסס על פלואורסצנטי התהודה העברת אנרגיה (סריג) 12. בשיטה זו, תצורות כרך מזוהות על ידי סריג בין תורם acceptor המצורף ליד הקצוות הדביקים של מולקולת ה-DNA. מיקרוסקופ אובייקטיבי מסוג הקרינה השתקפות הכל הפנימית (TIRFM) משמש להקלטת מסלולים של לולאות הפיכים ואירועי unlooping ממולקולות דנ"א חד משותק פני השטח לתקופה ממושכת של זמן. שיטה זו כוללת הרכבה של מולקולות ה-DNA המבוססת על ה-PCR לייצור מולקולות ה-DNA ללא חוסר התאמה, שהוא שיפור מכריע על שיטה דומה על ידי Vafabakhsh ו13 הא. היבט המולקולה בודדת של פרוטוקול זה מאפשר מדידה של הפצות בנוסף לאנסמבל ממוצע ואילו היבט סריג מאפשר למדוד דינמיקת לולאות DNA שוב ושוב מאותה המולקולה, גם במצבים שעלולים לפגוע בפעילות האנזים.
התקנת TIRFM מוצגת באיור 1. מותאמת אישיתשלב דגימה מעוצבת ממוקם מעל גוף מיקרוסקופ אולימפוס IX61. 532 ננומטר ו640 לייזרי ננומטר הם הציגו מהצד ובאים לידי הביטוי במראות סגלגלות זעירות 14 למטרת NA הגבוהה כדי להשיג זווית קריטית של שכיחות בממשק coverslip המים. נציין כי נפוץ יותר TIR דרך אובייקטיבי באמצעות מראות dichroic או setups TIR מבוסס פריזמה יכול לשמש גם עבור יישום סריג הזה. תמונת הקרינה שהוקמה על ידי מיקרוסקופ מחולקת לתמונות תורם acceptor ידי מראה dichroic. הם לאחר מכן מחדש הדמיה על שני חצאים של EMCCD. מסנני פליטה ארוך לעבור נוספים נמצאים בשימוש כדי להפחית את אות רקע.
בקרת טמפרטורה היא חיונית לרכישת נתונים הקינטית לשחזור. לבקרת טמפרטורה, המטרה היא להפריד את מזנקים של גוף מיקרוסקופ כדי למזער העברת חום, ומים מנשלט ילר / תנור בטמפרטורה מופצת דרך צווארון פליז שחוזקה התקפיםסביב מתכת הפנים מתחת למעייל האובייקטיבי. התקנה זו היא מסוגלת להשיג שליטה בטמפרטורה יציבה על פני השטח coverslip בין 15 50 ° C (איור 2). בעבודה זו, מדגם הטמפרטורה נשמרה ב24 ° C.
הפרוטוקול שלהלן מציג את הליך צעד אחר צעד לבניית ה-DNA, הערכה של צורת ה-DNA, ניסוי מולקולה בודדת, ונחישות גורם J.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. לדוגמא הכנה dsDNA
- עיצוב DNAs מעוקל ברחבי העולם על ידי חזרה על רצף 10-Mer. לדוגמא, 5'-GTGCCAGCAACAGATAGC - (TTTATCATCCTTTATCATCC X) 7 - TTTCATTCGAGCTCGTTGTTG-3 "הוא ה-DNA 186-bp מעוקלת כאשר X הוא בסיס נוסף אקראי ורצף איגוף רצף 10-mer החוזר הם רצפי מתאם.
הערה:. בדוגמא זו שני 10 מרס עם העדפות הפוכות להיווצרות הנוקלאוזום מבוססות על מחקר תפוסת הנוקלאוזום בקנה מידה גדולה על ידי קפלן ואח' 15 נבחרו. מאז חוזר הסליל של dsDNA הוא קרוב ל10 נ"ב, כל סטיה נקי של ציר הסליל של 10-mer תצטבר לייצר צורה כמו קשת עגולה (איור 3 א). מאחר שתקופת הסליל היא קרובה יותר ל10.5 נ"ב, בסיס נוסף מוכנס אחרי כל שתי חזרות כדי לשמור על המבנה המעוגל כמישוריים ככל האפשר. ניתן להזמין ברצפים אלה קצרים יותר מאשר 200 נ"ב מcompanieים ששירות סינתזת גן ההצעה. זה נוח לאגף רצפים אלה עם רצפי מתאם משותפים לשלבים הבאים. הליך schematized באיור 3. - לבצע PCR עם 1 פריימר (GTGCCAGCAACAGATAGC) ופריימר 2 (/ 5Cy3/TAAATTCCTACAACAACGAGCTCGAATG). הערה: פריימר 2 מתויג עם Cy3 תורם סריג בסוף "5. מתכון PCR טיפוסי ופרוטוקול רכיבה על אופניים מוצגים בלוחות 1 ו -2.
- לבצע PCR עם פריימר 3 (/ 5BioTEG/GAAACATAG/iCy5/GAATTTACCGTGCCAGCAACAGATAGC) ו -4 פריימר (CAACAACGAGCTCGAATG). הערה: פריימר 3 מתויג עם Cy5 acceptor סריג דרך עמוד השדרה וביוטין-מקשר בסוף "5. מתכון PCR ופרוטוקול רכיבה על אופניים הם כאמור לעיל.
- לטהר את מוצרי ה-PCR באמצעות ערכת ניקוי ה-PCR.
- מערבבים את המוצר שכותרתו Cy3 ומוצר שכותרתו Cy5 בחיץ להחלפת גדיל (100 mM NaCl, 10 מ"מ טריס-HCl pH 7.0, 1 mM EDTA) בריכוזים סופיים של 0.4 &181 #; M ו0.1 מיקרומטר, בהתאמה. הערה: Cy3-DNA העודף מגביר את הריכוז של דופלקס ביצוע שני Cy3 וCy5 כתוצאה מחילופי גדיל.
- קווצות שערי על ידי דוגרים על 98.5 מעלות צלזיוס למשך 2 דקות, קירור בהדרגה עד 5 מעלות צלזיוס עם שיעור רמפה של 0.1 ° C / sec, ודוגר על 5 ° C למשך 2 שעות.
2. ג'ל אלקטרופורזה לזיהוי dsDNA עקמומיות
- יוצקים את הג'ל polyacrylamide 16,17 על ידי ערבוב acrylamide ופתרונות bis-acrylamide ב29.2:0.8 יחס, 5% (w / v) במאגר 1X טריס / Borate / EDTA (TBE) ב-pH 8.0. הערה: 10 מיליליטר של תמיסת ג'ל מכיל: 1.217 acrylamide מיליליטר 40%, 0.667 מיליליטר 2% bis-acrylamide, 1 מיליליטר TBE 10X, 100 persulfate אמוניום L (APS) 10%, 10 L TEMED, וכל השאר הוא DH 2 O. התמצקות מלאה של הג'ל לוקחת בערך 30 דקות.
- טען את ג'ל polyacrylamide עם דגימות DNA וסולם ה-DNA בחיץ טעינת 1X (5% גליצרול, 0.03% (w / v bl bromophenol)UE) ולהפעיל את הג'ל על 5-8 V / סנטימטר ב-C ° 4 דקות 45 או עד שצבען הקדמי מתקרב לסוף של הג'ל.
- כתם ג'ל באמצעות חיץ 1X TBE המכיל 0.5 גר '/ מיליליטר ethidium ברומיד ל30 דקות. זהה את להקות ה-DNA תחת תאורת UV. שקלו את עמדות להקה עם סמן הגודל (100 סולם DNA נ"ב) כדי לחשב את הגדלים, לכאורה, של מולקולות ה-DNA. הערה: DNAs המעוקל בדרך כלל איטי יותר מאשר להעביר DNAs הישר.
3. זרימת הכנת תא
- מקדחה 6-7 זוגות של חורים בשני קצוות מנוגדים של שקופיות זכוכית (3'' x 1'') באמצעות מקדחה ומקדחי יהלום. לאחר קידוח, לשפשף את השקופית במים זורמים כדי להסיר אבקת זכוכית גלויה. הערה: החורים משמשים כפתחי כניסת זלוף ושקעים. במהלך קידוח, קירור השקופית עם מים חשוב כדי למנוע פיצוח.
- מניחים את השקופיות זקופות בצנצנת זכוכית ולמלא אותו במים. Sonicate במשך 15 דקות ולהעביר אותם לעוד דה צנצנת זכוכיתdicated לניקוי אצטון. מלא אותו עם אצטון sonicate במשך 15 דקות. יש לשטוף את השקופיות עם אתנול באמצעות בקבוק תרסיס ולאחר מכן עם מים. למקם אותם בצנצנת פוליפרופילן, למלא אותו עם 5 הידרוקסיד M אשלגן, sonicate במשך 15 דקות. לבסוף, sonicate השקופיות במים במשך 15 דקות. נקה את coverslips (מס '1, 24 x 40 מ"מ) משתמש באותו פרוטוקול. הערה: שקופיות וcoverslips ניקה יכולים להיות מאוחסנים ב DH 2 O לשימוש לטווח ארוך.
- מערבבים 1 מ"ג של ביוטין-PEG-silane (MW 3400) עם 80 מ"ג של MPEG-silane (MW 2000) ב340 L של 0.1 פתרון ביקרבונט M נתרן. מערבבים היטב וצנטריפוגות התערובת בקצרה להיפטר מבועות. הערה: functionalization של פני השטח עם פוליאתילן גליקול (PEG) מסייע להפחית ספציפי מחייב של הדנ"א אל פני השטח.
- שים 80 L של פתרון PEG בכל שקופית ועדינות להוריד את coverslip על זה. חכה 45 דקות. הפרד את coverslip מהשקופית עם פינצטה, לשטוף אותם עם כמות העצומה של DH 2 O ולתת tיבש מכפלת באוויר פתוח.
- מניחים רצועות דקות של קלטת דו מקל על פני השקופית כדי ליצור ערוצים. יישר coverslip על זה ולחץ בחוזקה נגד coverslip לשקופית כדי ליצור ערוצים נוזל הדוקה. השתמש אפוקסי 5 דקות כדי לאטום את הקצוות של הערוצים.
4. הכנת Trolox הפתרון
- שים ~ 30 מ"ג Trolox ו10 מיליליטר DH 2 O בבקבוק ולהשתמש בבר ומערבב מגנט לעורר את הפתרון באוויר פתוח במשך 18 שעה.
- סנן את הפתרון באמצעות מסנן 0.2 מיקרומטר ולהתאים את ה-pH ל 7 על ידי הוספה ~ 6 μl של 1 בסיס M טריס (pH 11). הערה: Trolox הוא מגיב אנטי מהבהב המשמש בדרך כלל במולקולה בודדת לומד 18. פעולת antifading של Trolox מגיעה מנגזרת חמצון שלה, אשר נמצא בפתרון Trolox המושפל באופן חלקי 19. מהירות המסת Trolox במתנול או פתרון ה-pH טריס גבוה יש להימנע בגלל חמצון יעיל.
5. Single-molהדמיה ecule
- הזרק 15 μl של פתרון NeutrAvidin (0.5 מ"ג / מיליליטר) לתוך הערוץ ולחכות 2 דקות לפני השטיפה עם של חיץ T50 100 μl (10 מ"מ טריס-HCl, 50 mM NaCl, pH 7.0).
- הזרק 50 μl של דגימת DNA (50-100 PM) לתוך התעלה. מתן 5 דקות ולשטוף את ה-DNA מאוגד משם עם של חיץ T50 100 L. הערה: מולקולות דנ"א ספציפיות תהיה לאגד את פני השטח באמצעות האינטראקציה NeutrAvidin-ביוטין.
- מלא ערוץ עם חיץ ההדמיה המכיל מערכת הדחה חמצן 20 (100 מ"מ PCD, 5 מ"מ PCA, 1 מ"מ Trolox, ו500 מ"מ NaCl).
- הגדר את EMCCD במצב מסגרת העברה להזרים 2 x 2 תמונות זרקת לפח (256 x 256) למחשב ב25 מסגרות לשנייה.
- שים שמן טבילה במטרת מיקרוסקופ, ולתקן את תא הזרימה על הבמה מיקרוסקופ באמצעות קליפים דגימה. גס להתאים את הפוקוס ע"י הסתכלות בדפוס השתקפות לייזר על הקיר. עדין להתאים את הפוקוס באמצעות התצוגה החיה של im הקרינהגילים על המסך.
- בגין רכישת נתונים עם לייזר 532 ננומטר ב. בדוק את התצוגה החיה ולהתאים את הפוקוס במידת צורך. תפסיק רכישת נתונים כאשר רוב המולקולות photobleached.
הערה: במתקן זה, משמשת תכנית C-נכתב מעבדה כדי לשלוט במיקרוסקופ ולהציג תמונות חיות על המסך כפי שהם נשמרים לדיסק הקשיח.
6. עיבוד תמונה וניתוח נתונים
הערה: סדרת זמן של 256 x 256 תמונות מעובדות על ידי קוד MATLAB ליצור עקבות זמן מולקולה בודדת של עוצמות Cy3 וCy5. כדי לשייך פיקסלים בין ערוץ התורם וערוץ acceptor של התמונה מפוצלת נוף, 6-7 זוגות של כתמי Cy3 וCy5, כל זוג מאותו המולקולה המפוזרת באופן שווה על פני שדה הראייה, הם הרימו ידני, ושינוי affine מחושב באמצעות הקואורדינטות של נקודות אלה כנקודות עיגון.
- באמצעות תסריט MATLAB, להסתכל דרך כל זמן המולקולה בודדת עקבות tמעברים מרובים מופע כובע בין אותות סריג נמוכים וגבוהים. זהה את מדינות כרך וunlooped.
הערה: אות סריג מוגדרת כעוצמת Cy5 (אני), מחולק בסכום של עוצמות Cy3 וCy5 (אני + אני D). יש מדינה בלולאה ערך סריג גבוה תוך מדינת unlooped יש ערך סריג נמוך. היסטוגרמה של אותות סריג ממולקולה בודדת מופצת bimodally בגלל לולאות וunlooping הפיכים. - מצא את הסף המפריד בין שתי הפצות על ידי קביעת הצומת שבין שתי עקומות גאוס מצוידים.
- לחשב את יעילות סריג כמו
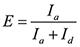 ולהקצות את המדינות בלולאה עם ערכים גבוה סריג ומדינות unlooped עם ערכי סריג נמוכים.
ולהקצות את המדינות בלולאה עם ערכים גבוה סריג ומדינות unlooped עם ערכי סריג נמוכים. - באמצעות תסריט MATLAB, לנתח את המספר המצטבר של מולקולות (N (t)) שכרך (אוהגיע למצב סריג הגבוה) בפערי זמן שונים מאז תחילת רכישת נתונים. הערה: מאחר שמולקולת dsDNA יכולה להתחיל בכל קונפורמציה של מדינת unlooped בתחילת רכישת נתונים, שיעור הגידול באוכלוסיית הכרך משקף את שיעור הלולאה הממוצע בממוצע לכל תצורות ראשוניות. לחלץ את k שיעור לולאת לולאה על ידי התאמת N (t) עם פונקציה מעריכית:
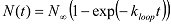 אם עליות biphasically, זה יכול להיות מצויד עם פונקציה כפולה מעריכי:
אם עליות biphasically, זה יכול להיות מצויד עם פונקציה כפולה מעריכי: 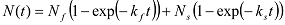 במקרה זה, k לולאה מתקבלת:
במקרה זה, k לולאה מתקבלת: 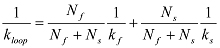 הערה: באופן תיאורטי, את הסתברות ההישרדות שפולימר לא בלולאה בזמן t היא לא פונקציה מעריכית יחידה או כפולה 12. Fitt מעריכיing משמש כאמצעים מעשיים כדי לחלץ את זמן לולאות הממוצע.
הערה: באופן תיאורטי, את הסתברות ההישרדות שפולימר לא בלולאה בזמן t היא לא פונקציה מעריכית יחידה או כפולה 12. Fitt מעריכיing משמש כאמצעים מעשיים כדי לחלץ את זמן לולאות הממוצע.
7. קביעת פקטור J
הערה: גורם J מייצג איך מרוכז קצה אחד של dsDNA הוא מעבר לקצה השני. זה יכול להיקבע על ידי ביון הריכוז של קטע קצה אחד של ה-DNA שמייצר את אותו קצב התגובה עם מגזר הקצה השני כשיעור לולאות שנמדד. בניסוי, קטע קצה אחד הוא משותק על פני השטח, ומגזר הקצה השני הוא הציג בריכוז ג מסוים. אם שיעור חישול נמדד בין שני הקצוות הוא לחשל k, אז גורם J 21 ניתן על ידי 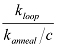 . השיעור הקבוע חישול (לחשל k = לחשל k / C) אינו תלוי בריכוז הבדיקה.
. השיעור הקבוע חישול (לחשל k = לחשל k / C) אינו תלוי בריכוז הבדיקה.
- לזרום 20 μl של אוליגו ביוטין-Cy5 30-50 PM (פריימר 3) in כדי ערוץ מצופה NeutrAvidin. יש לשטוף את הערוץ עם 100 T50 μl לשטוף oligos מאוגד.
- הכן את חיץ ההדמיה כפי שמתואר בחלק 5 עם התוספת של אוליגו Cy3 (פריימר 2) בריכוז סופי של 50 ננומטר. לזרום חיץ הדמיה זו לתוך התעלה.
- תוך שמירה על לייזר 532 ננומטר ב, פונה בקצרה לייזר 640 ננומטר למקומות של oligos Cy5 מאוגד משטח לזהות. כבה את לייזר 640 ננומטר ולהתחיל ניטור אות סריג.
הערה: עם הכלאה של אוליגו Cy3 לאוליגו Cy5 מאוגד פני השטח, הקרינה Cy5 תנבע מסריג. - באמצעות תסריט MATLAB, לנתח את מספר המולקולות שתתחלנה במדינה מאוגדת (עוצמת Cy5 נמוכה), אך מאוחר יותר יהפכו למדינה (בעצימות גבוהה Cy5) annealed כפונקציה של זמן מעקבות עוצמת Cy5.
- מגרש מספר זה של מולקולות לעומת זמן annealed. להתאים עקומה זו עם פונקציה מעריכית יחידה (
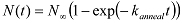 ) כדי להשיג את שיעור חישול (לחשל k).
) כדי להשיג את שיעור חישול (לחשל k). - חזור על הניסוי הזה בריכוזים שונים Cy3 אוליגו (60, 100, ו180 ננומטר) כדי לאשר את הליניאריות בין שיעור חישול והריכוז מגיב. חלץ את שיעור קבוע חישול מסדר השנייה (לחשל k ) מהמדרון.
- חשב את גורם J מ
 , כאשר k לולאה היא שיעור הלולאות שנמדד באותו מצב החיץ.
, כאשר k לולאה היא שיעור הלולאות שנמדד באותו מצב החיץ.
Access restricted. Please log in or start a trial to view this content.
תוצאות
מולקולות ה-DNA משמשות למחקר הלולאה מורכבת של אזור דופלקס של רצף משתנה ובאורך וסככות חד גדילים שהם משלימים אחד את השני. הסככות, שהן ארוכות 7 בסיס, יכולות לחשל זה לזה כדי ללכוד את מצב הכרך. כל הסככה מכילה גם Cy3 או Cy5 כי הוא מקושר בעמוד שדרת ה-DNA באמצעות הכימיה amidite. Cy5-הסככה קשו...
Access restricted. Please log in or start a trial to view this content.
Discussion
Assay מולקולה בודדת פשוט המבוסס על סריג שימש ללמוד קינטיקה לולאות של DNAs של צורות פנימיות שונות. ניתן להכין DNAs מעוקל על ידי חזרה על רצף 10-mer בשלב עם תקופת הסליל של 10.5 נ"ב, וניתן להעריך עקמומיות שלהם באמצעות עמוד. dsDNAs אלה נועדו עם קצוות דביקים על מנת לאפשר ייצוב לולאה חולף...
Access restricted. Please log in or start a trial to view this content.
Disclosures
המחברים מצהירים שום ניגוד עניינים.
Acknowledgements
אנו מודים לג'יימס ווטרס, גייבל Wadsworth ובי Broadwater לקריאה ביקורתי של כתב היד. אנו מודים גם לארבעה מבקרים האנונימיים על מתן הערות מועילות. אנו מכירים תמיכה כספית מהמכון הטכנולוגי של ג'ורג'יה, פרס הקריירה בורוז Wellcome הקרן בממשק המדעי, ומענק רשת מחקר הסטודנט מNSF הפיזיקה של מערכות חיים.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Small DNA FRAG Extract Kit-100PR | VWR | 97060-558 | |
| Acrylamide 40% solution 500 ml | VWR | 97064-522 | |
| Bis-acrylamide 2% (w/v) solution 500 ml | VWR | 97063-948 | |
| GeneRuler 100 bp DNA Ladder, 100-1,000 bp | Fermentas | SM0241 | |
| Mini Vertical PAGE System | VWR | 89032-300 | |
| Syringe filter 0.2 μm CS50 | VWR | A2666 | |
| Trolox | Sigma-Aldrich | 238813-1G | triplet state quencher |
| Protocatechuic acid (PCA) | Sigma-Aldrich | 08992-50MG | oxygen scavenging system |
| Protocatechuate 3,4-Dioxygenase (PCD) | Sigma-Aldrich | P8279-25UN | oxygen scavenging system |
| mPEG-silane, MW 2,000 1 g | Laysan Bio | MPEG-SIL-2000-1g | |
| Biotin-PEG-Silane, MW 3,400 | Laysan Bio | Biotin-PEG-SIL-3400-1g | |
| Avidin, NeutrAvidin Biotin-binding Protein | Invitrogen | A2666 | |
| Phusion Hot Start High-Fidelity DNA Polymerase | New England Biolabs | F-540L | |
| Gel/PCR DNA Fragments Extraction Kit | IBI Scientific | IB47020 | |
| Premium plain glass microscope slides | Fisher Scientific | 12-544-1 | |
| VWR micro cover glass, rectangular, no. 1 | VWR | 48404-456 | |
| Fisher Scientific Isotemp 1006S Recirculating Chiller/Heater | Fisher Scientific | temperature control | |
| Objective Cooling Collar | Bioptechs | 150303 | temperature control |
| KMI53 Biological Micrometer Measuring Stage | Semprex | KMI53 | |
| High Performance DPSS Laser 532 nm 50 mW | Edmund optics | NT66-968 | Cy3 excitation |
| CUBE Fiber Pigtailed 640 nm, 30 mW, Fiber, FC/APC Connector | Coherent | 1139604 | Cy5 excitation |
| 650 nm BrightLine Dichroic Beamsplitter | Semrock | FF650-Di01-25x36 | splitting dichroic |
| LaserMUX Beam Combiner, reflects 514.5, 532, & 543.5 nm lasers, 25 mm | Semrock | LM01-552-25 | combining dichroic |
| Brightline Fluorescence Filter 593/40 | Semrock | FF01-593/40-25 | Cy3 emission filter |
| 635 nm EdgeBasic LWP longpass Filter, 25 mm | Semrock | BLP01-635R-25 | Cy5 emission filter |
| EMCCD iXon+ | Andor Technology | DU-897E-CS0-#BV | |
| IX51 inverted microscope frame | Olympus | ||
| Objective UApo N 100X/1.49 Oil TIRF | Olympus | ||
| Immersion oil type-F for fluorescence microscopy | Olympus | IMMOIL-F30CC | |
| 2 mm Diameter 45° Rod Lens Aluminum Coated | Edmund optics | 54-092 | miniature mirror |
| 1/4" Travel Single-Axis Translation Stage | Thorlabs | MS-1 | translation of miniature mirror |
| Ø1" Achromatic Doublet, ARC: 400-700 nm, f=200 mm | Thorlabs | AC254-200-A | focusing lens |
| Adjustable Mechanical Slit | Thorlabs | VA100 | |
| Dielectric Mirror | Thorlabs | BB1-E02 | |
| Ø1" Achromatic Doublet, f = 100 mm | Thorlabs | AC254-100-A | relay lens |
| Lens Mount for Ø1" Optics | Thorlabs | LMR1 | |
| Dichroic Filter Mount | Thorlabs | FFM1 | |
| Fixed Cage Cube Platform | Thorlabs | B3C | |
| Kinematic Mount for Ø1" Optics | Thorlabs | KM100 | |
| N-BK7 Plano-Convex Lens, Ø1", f = 40 mm | Thorlabs | LA1422-A | collimating lens |
| N-BK7 Plano-Convex Lense, Ø6.0 mm, f = 15 mm | Thorlabs | LA1222-A | telescope lens |
| N-BK7 Plano-Convex Lense, Ø6.0 mm, f = 150 mm | Thorlabs | LA1433-A | telescope lens |
References
- Garcia, H. G., et al. Biological consequences of tightly bent DNA: The other life of a macromolecular celebrity. Biopolymers. 85, 115-130 (2007).
- Wiggins, P. A., et al. High flexibility of DNA on short length scales probed by atomic force microscopy. Nature Nanotechnology. 1, (2006).
- Lionberger, T. A., et al. Cooperative kinking at distant sites in mechanically stressed DNA. Nucleic Acids Research. 41, 6785-6792 (2011).
- Shore, D., et al. DNA flexibility studied by covalent closure of short fragments into circles. Proc Natl Acad Sci U S A. 78, 4833-4837 (1981).
- Geggier, S., Vologodskii, A. Sequence dependence of DNA bending rigidity. Proc Nat Acad Sci U S A. 107, 15421-15426 (1992).
- Smith, S. B., et al. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258, 1122-1126 (1992).
- Peters, J. P., Maher, L. J. DNA curvature and flexibility in vitro and in vivo. Quarterly Reviews of Biophysics. 43, 23-63 (2010).
- Cloutier, T. E., Widom, J. Spontaneous sharp bending of double-stranded DNA. Molecular Cell. 14, 355-362 (2004).
- Du, Q., et al. Cyclization of short DNA fragments and bending fluctuations of the double helix. Proc Natl Acad Sci U S A. 102, 5397-5402 (2005).
- Yuan, C., et al. T4 DNA ligase is more than an effective trap of cyclized dsDNA. Nucl. Acids Res. 35, 5294-5302 (2007).
- Manzo, C., et al. The effect of nonspecific binding of lambda repressor on DNA looping dynamics. Biophysical Journal. 103, 1753-1761 (2012).
- Le, T. T., Kim, H. D. Measuring shape-dependent looping probability of DNA. Biophys. J. 104, 2068-2076 (2013).
- Vafabakhsh, R., Ha, T. Extreme bendability of DNA less than 100 base pairs long revealed by single-molecule cyclization. Science. 337, 1097-1101 (2012).
- Friedman, L., et al. Viewing dynamic assembly of molecular complexes by multi-wavelength single-molecule fluorescence. Biophysical Journal. 91, 1023-1031 (2006).
- Kaplan, N., et al. The DNA-encoded nucleosome organization of a eukaryotic genome. Nature. 458, 362-366 (2009).
- Koo, H. S., Crothers, D. M. Calibration of DNA curvature and a unified description of sequence-directed bending. Proc Nat Acad Sci U S A. 85, 1763-1767 (1988).
- Prosseda, G., et al. A temperature-induced narrow DNA curvature range sustains the maximum activity of a bacterial promoter in vitro. Biochemistry. 49, 2778-2785 (2010).
- Rasnik, I., et al. Nonblinking and long-lasting single-molecule fluorescence imaging. Nature Methods. 3, 891-893 (2006).
- Cordes, T., et al. On the mechanism of Trolox as antiblinking and antibleaching reagent. J. Am. Chem. Soc. 131, 5018-5019 (2009).
- Aitken, C. E., et al. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophys J. 94, 1826-1835 (2008).
- Taylor, W. H., Hagerman, P. J. Application of the method of phage T4 DNA ligase-catalyzed ring-closure to the study of DNA structure: II. NaCl-dependence of DNA flexibility and helical repeat. Journal of Molecular Biology. 212, 363-376 (1990).
- Bolshoy, A., et al. Curved DNA without A-A: experimental estimation of all 16 DNA wedge angles. Proc Natl Acad Sci U S A. 88, 2312-2316 (1991).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucl. Acids Res. 37, 6984-6990 (2009).
- Vologodskii, A., et al. Bending of short DNA helices. Artif DNA PNA XNA. 4, (2013).
- Hoover, D. M., Lubkowski, J. DNAWorks: an automated method for designing oligonucleotides for PCR-based gene synthesis. Nucl. Acids Res. 30, (2002).
- Waters, J. T., Kim, H. D. Equilibrium Statistics of a Surface-Pinned Semiflexible Polymer. Macromolecules. 46, 6659-6666 (2013).
- Mills, J. B., et al. Electrophoretic evidence that single-stranded regions of 1 or more nucleotides dramatically increase the flexibility of DNA. Biochemistry. 33, 1797-1803 (1994).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved