このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
1分子FRETによるDNAループを研究
要約
この研究は、単一分子蛍光共鳴エネルギー移動(FRET)を用いて二本鎖DNAのループダイナミクスを測定するための詳細な実験手順を提示する。プロトコルは、Jファクターと呼ばれるループする確率密度を抽出する方法を説明します。
要約
二本鎖DNA(dsDNA)の屈曲は、ヌクレオソーム内に、例えばDNA-タンパク質認識およびDNAのパッケージングのような多くの重要な生物学的プロセスに関連している。二本鎖DNAの曲げの熱力学は、共有結合dsDNAの短い付着末端を結合するDNAリガーゼに依存している環化と呼ばれる方法で研究されている。しかし、ライゲーション効率は、そのような接合粘着末端を取り囲むDNA構造としてループdsDNAに関係のない多くの要因によって影響され得る、およびリガーゼはまた、非特異的結合の機構を通じて明らかなループ速度に影響を与えることができる。ここでは、FRET(蛍光共鳴エネルギー移動)による一過性DNAループの形成を検出することによってリガーゼなしでのdsDNAのループ速度を測定する方法を示している。二本鎖DNA分子は、FRET対とビオチンリンカーを有する単純なPCRベースのプロトコルを使用して構築される。 J因子として知られているループする確率密度は2 disconnec間をループレートとアニール速度から抽出されるテッド粘着末端。異なる固有の曲率を有する2つのdsDNAsを試験することによって、我々は、J因子はdsDNAの固有の形状に敏感であることを示している。
概要
dsDNAの機械的性質を理解することは、基礎科学や工学アプリケーションで基本的に重要である。連続した塩基対間のロール、チルト、およびねじれ角がシーケンスで変化することができるので、二本鎖DNAの構造はストレートヘリカル梯子よりも複雑です。熱揺らぎは、二本鎖DNAは、曲げねじれやストレッチングなどの立体配座の変動の多様なモードを受ける可能性があります。このような溶融やねじれなどの遷移はまた、極端な条件で発生する可能性があります。
これらの動きの中では、二本鎖DNA曲がりが最も顕著な生物学的影響は1を持っています。二本鎖DNAの曲がりは互いに近接2遠隔部位にすることによって遺伝子抑制または活性化に関連している。それはまた、細胞核又はウイルスキャプシドの内側DNAパッケージングにおいて重要な役割を果たしている。 dsDNAの曲げ変形は、高解像度顕微鏡(AFM及びTEM 2 3)、およびthermodynにより実験的に可視化することができるamics及び動力学は、化学的にdsDNAの並置部位をリンクループアッセイによって研究することができる。
1つのそのようなアッセイはリガーゼ依存環化4である。このアッセイでは、「スティッキー」(凝集)末端を有する二本鎖DNA分子は、DNAリガーゼにより環状又は二量体化される。サークル及び二量体形成の速度を比較することによって、J因子として知られている他端の近傍のDNAの一端の有効モル濃度を得ることができる。このJ係数は、他端から短い距離でDNAの一端を見出す確率密度と寸法的に等価であり、したがって、DNAの柔軟性を反映する。 DNAの長さの関数としてのJ係数を測定することは持続長4,5含むDNA力学に関する多くの特性を明らかにする。
ワームのようなチェーン(WLC)のモデルが広くexplaでの成功に基づいて二本鎖DNA力学の正規ポリマーモデルと見なされてきた力伸長曲線ining実験6を引いたDNAで得られ、かつ正確に、より長い200塩基対7よりdsDNAsのJ要因を予測する。しかし、100bpのできるだけ短い二本鎖DNA分子上の環化アッセイを用いて、クルーティエとWidomは、WLCのモデル予測8よりも桁違いに高くなるようにJ因子を測定した。一年後、ドゥら 。リガーゼの低い濃度で環化アッセイを使用してWLCモデルと一致J因子を生産し、Widomグループから9リガーゼを使用する高濃度に異常な結果に起因。従来のアッセイ9を使用する場合、この論争が環化速度に及ぼすDNAリガーゼの不可避の影響を例示する。また、DNAリガーゼはまた、非特異的な結合10,11を介してDNA構造および剛性に影響を与えることができる。
タンパク質依存ループアッセイの技術的な問題を解消するために、我々は最近、PROTが実証蛍光共鳴エネルギー移動(FRET)12に基づいてEINのないループアッセイ。この方法では、ループのコンフォメーションは、DNA分子の付着末端付近に取り付けられたドナーとアクセプターの間のFRETにより検出される。対物レンズ型全反射蛍光顕微鏡(TIRFM)は、可逆ループと、長期間にわたって表面固定単一DNA分子からunloopingイベントの軌跡を記録するために使用される。この方法は、Vafabakhshとハ13による同様の方法上の重要な改良で、ミスマッチのないDNA分子を生成するために、DNA分子のPCRベースのアセンブリを提供しています。このプロトコルの単一分子の態様は、FRETの態様であってもリガーゼ活性を損なう可能な条件では、1つは、同じ分子から繰り返しDNAのループダイナミクスを測定することを可能にしながら平均をアンサンブルに加えて分布の測定を可能にする。
TIRFMのセットアップを図1に示す。カスタム設計された試料ステージは、オリンパスIX61顕微鏡本体上に配置される。 532 nmおよび640 nmのレーザーは、側面から導入され、カバーガラス-水界面における臨界入射角を達成するために、高NA対物レンズ内に微小な楕円鏡14で反射される。私たちは、より広範なダイクロイックミラーやプリズムベースのTIR設定を行うスルー客観TIRもこのFRET用途に使用できることに注意してください。顕微鏡によって形成された蛍光像は、ダイクロイックミラーによりドナーとアクセプターのイメージに分割される。そして、彼らはEMCCDの両半分に再度結像される。追加のロングパス放出フィルタは、バックグラウンドシグナルを減少させるために使用される。
温度制御は、再現性の速度論的データを取得するために不可欠である。温度制御のために、目的は、熱伝達を最小にするために、顕微鏡本体のノーズピースから分離され、温度制御された冷却器/加熱器からの水が密に嵌真鍮カラーを通って循環される客観的なジャケットの下にあるインテリア金属の周り。このセットアップでは、15〜50°C( 図2)との間のカバーガラス表面に強固な温度制御を達成することができる。本研究では、試料温度を24℃に維持した。
以下のプロトコルは、DNAの構築、DNA形状の推定、単一分子実験、およびJ要因判定するためのステップバイステップ手順を提示する。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1。二本鎖DNAサンプル調製
- デザインは、全体的に10マー配列を繰り返すことによりDNAを湾曲。例えば、5'-GTGCCAGCAACAGATAGC - (TTTATCATCCTTTATCATCC X)7 - TTTCATTCGAGCTCGTTGTTG-3 'は、Xがランダム余分な塩基であり、繰り返し10量体配列に隣接する配列は、アダプター配列で186 bpの湾曲したDNAである。
注:カプランらによる大規模なヌクレオソーム占有研究に基づいてヌクレオソームの形成とは逆の好みにこの例では、2つの10量体15が選ばれた。二本鎖DNAの螺旋の繰り返しが10塩基対に近いので、10 -マーの螺旋軸のいずれかの正味たわみは円弧( 図3A)のような形状を生成するために蓄積されます。ヘリカル期間は10.5 BPに近いので、余分な塩基が可能な平面として湾曲した構造を維持するために、すべての2の繰り返しの後に挿入されます。 200 BPより短いこれらの配列は、companieのから注文することができます遺伝子合成サービスを提供していますS。これは、後続のステップのための共通のアダプター配列をこれらの配列に隣接するのが便利である。手順を図3Bに図式化されている。 - プライマー1(GTGCCAGCAACAGATAGC)及びプライマー2(/ 5Cy3/TAAATTCCTACAACAACGAGCTCGAATG)を用いてPCRを行う。 NOTE:プライマー2は、その5 '末端にFRETドナーCy3で標識される。典型的なPCRレシピやサイクリングプロトコルは、 表1および表2に示す。
- プライマー3(/ 5BioTEG/GAAACATAG/iCy5/GAATTTACCGTGCCAGCAACAGATAGC)及びプライマー4(CAACAACGAGCTCGAATG)を用いてPCRを行う。 NOTE:プライマー3を5 '末端に骨格とビオチン - リンカーを介してFRETアクセプターCy5で標識される。 PCRのレシピ、サイクリングプロトコルは、上記の通りである。
- PCRクリーンアップキットを用いてPCR産物を精製する。
- 0.4&最終濃度鎖交換用緩衝液(100mMのNaCl、10mMのトリス-HCl、pH7.0、1mMのEDTA)中のCy3標識産物およびCy5標識生成物を混合しそれぞれMと0.1μM;#181。注:過剰のCy3-DNAは、鎖交換の結果として、Cy3およびCy5の両方を運ぶ二重鎖の濃度が増加します。
- 徐々に5まで冷却し、2分間98.5℃でインキュベートすることにより為替ストランド ランプ0.1°C /秒の速度、および5でのインキュベーションと°C 2時間Cと°。
2。ゲル電気泳動は、二本鎖DNAの曲率を検出するための
- 29.2:0.8の割合でアクリルアミドとビスアクリルアミドの溶液を混合することによりポリアクリルアミドゲル16,17を注ぎ、5%(w / v)のpH8.0の1Xトリス/ボレート/ EDTA(TBE)緩衝液中。注:ゲル溶液10mlを含有する:1.217ミリリットルの40%アクリルアミド、0.667ミリリットルの2%ビス-アクリルアミドを1ml TBE 10Xを、100 Lの過硫酸アンモニウム(APS)10%、10 L TEMED、残りはをdH 2 Oである。ゲルの全固体化には約30分かかります。
- DNA試料および1Xローディング緩衝液中のDNAラダー(5%グリセロール、0.03%(w / v)のブロモフェノールblを用いてポリアクリルアミドゲルをロードするUE)と45分間、又は染料前線がゲルの端に近づくまで4℃で5-8 V / cmとゲルを実行する。
- 30分間、0.5μg/ mlの臭化エチジウムを含有する1X TBE緩衝液を用いてゲルを染色する。 UV照射下でDNAバンドを識別します。 DNA分子の見かけの大きさを計算するためにサイズマーカー(100 bpのDNAラダー)を用いてバンドの位置を比較する。注:湾曲したDNAは、ほぼ直線状のDNAよりも遅く移動する。
3。細胞調製フロー
- ドリルドリルプレス、ダイヤモンドドリルビットを用いてガラススライドの二つの対向するエッジ(3×1'''')に沿った正孔の対6-7。掘削した後、目に見えるガラス粉末を除去するために水を流すことでスライドをこする。 NOTE:穴は、灌流入口及び出口として機能する。掘削時には、水でスライドを冷却して割れを防ぐことが重要です。
- ガラスジャーに直立スライドを置き、水でそれを埋める。 15分間超音波処理し、別のガラスジャードにそれらを転送アセトン洗浄にdicated。 15分間のアセトンと超音波処理でそれを埋める。スプレーボトルを使用してエタノールでスライドを洗い流してから水と。 、ポリプロピレンジャーに配置し、5Mの水酸化カリウムとそれを記入し、15分間超音波処理。最後に、15分間水中でスライドを超音波処理する。同じプロトコルを使用してカバーグラス(1号、24×40 mm)を清掃してください。 NOTE:クリーンスライドとカバースリップは、長期間の使用のためのdH 2 Oに保存することができる。
- 0.1 M重炭酸ナトリウム溶液の340 LでのmPEG-シラン(MW 2000)の80 mgのビオチン-PEG-シラン(MW 3400)1mgを混合する。よく混ぜ、気泡を取り除くために簡単に混合し遠心する。注:ポリエチレングリコール(PEG)で表面の機能化は、表面へのDNAの非特異的結合を減らすことができます。
- 各スライド上のPEG溶液80 Lを入れて、ゆっくりとその上にカバースリップを下げる。 45分間待ちます。 、ピンセットでスライドからカバーガラスを分離するのdH 2 Oを多量のでそれらを洗い流しとletトンオープンエアでの裾ドライ。
- チャネルを形成するスライド全体に両面テープの薄いストリップを配置します。その上にカバーガラスを合わせ、しっかりと液密チャネルを形成するスライドに対してカバースリップを押してください。チャンネルのエッジをシールするために5分エポキシを使用してください。
4。トロロックス溶液の調製を
- フラスコの中で〜30mgのトロロックスと10ミリリットルのdH 2 Oを入れて、18時間オープンエアでソリューションを攪拌し、マグネット攪拌棒を使用しています。
- 0.2μmのフィルターを用いて溶液を濾過し、1Mトリス塩基(pHが11)の〜6μLを加えることによりpHを7に調整。注意:トロロックスは、単一分子は18の研究で一般的に使用されている抗点滅試薬である。トロロックスの褪色防止作用は、部分的に分解ロックス液19中に存在し、その酸化誘導体から来ている。すぐにメタノールや高pH Tris溶液中トロロックスを溶解しているため、非効率的酸化のは避けるべきである。
5。シングルモルeculeイメージング
- チャンネルにニュートラ溶液(0.5 mg / ml)を15μlのを注入し、T50緩衝液100μl(10mMトリス-HCl、50mMのNaCl、pH7.0)でリンスする前に、2分間待ちます。
- チャネルにDNAサンプル(50〜PM)の50μLを注入。 5分間待ってから、T50バッファの100リットルで、未結合するDNAを洗い流す。 NOTE:DNA分子が特異的にニュートラアビジン - ビオチン相互作用を介して表面に結合する。
- 酸素除去システム20(100 mMのPCD、5 mMのPCA、1 mMのトロロックス、および500のNaCl)が含まれているイメージングバッファーでチャンネルを入力します。
- 毎秒25フレームの速度でパソコンに2×2ビニングされた画像(256×256)をストリーミングするために、フレーム転送モードでEMCCDを設定します。
- 顕微鏡対物レンズにイマージョンオイルを入れて、試験片のクリップを使用して、顕微鏡ステージ上でフローセルを修正。壁にレーザー反射パターンを見ることで、フォーカスを粗調整します。蛍光IMのライブビューを使ってピントを微調整モニター上の年齢層。
- 上の532 nmのレーザーを使用してデータ収集を開始します。ライブビューを確認し、必要に応じてフォーカスを調整します。ほとんどの分子が退色している時にデータ収集を停止します。
注:この機能では、顕微鏡を制御し、これらはハードディスクに保存されるようにモニターにライブ映像を表示するためのラボが作成したCプログラムが使用されます。
6。画像処理とデータ解析
NOTE:256×256画像の時系列を、Cy3およびCy5強度の単一分子タイムトレースを生成するために、MATLABコードによって処理される。ドナーチャネルとスプリットビュー画像の受容チャネルとの間のピクセルを対にする、Cy3およびCy5スポットの6-7組、均等に視野に分散し、同じ分子からの各ペアは、アフィン変換を手動で拾い、アールアンカーポイントとしてこれらのスポットの座標を用いて計算される。
- MATLABスクリプトを使用して、すべての単一分子の時間に目を通すトンをトレース帽子は高低のFRETシグナルの間に複数の遷移を示しています。ループ状にループしない状態を識別します。
NOTE:FRET信号(I a)は、Cy3およびCy5強度(I dはI +)との和で割ったCy5の強度として定義される。ループしない状態が低いFRET値を有しながらループ状態が高いFRET値を有する。単一分子からのFRETシグナルのヒストグラムが二峰性のため、可逆ループしunlooping分散されている。 - 2フィットガウス曲線との交点を決定することにより、二つの分布を分離しきい値を検索します。
- FRET効率などを計算する
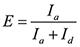 と高いFRET値と低いのFRET値がループしない状態でループ状態を割り当てます。
と高いFRET値と低いのFRET値がループしない状態でループ状態を割り当てます。 - MATLABスクリプトを使用して、分子(N(t))との累積数を分析することは、ループ(またはデータ収集の開始から異なる時間が経過においてFRETハイ状態)に達した。注:二本鎖DNA分子は、データ収集の開始時にループしない状態のいずれかのコンフォメーションで起動することができますので、ループの人口の増加率は平均ルーピング率は初期のコンフォメーションで平均化が反映されます。指数関数で、N(t)を当てはめることによって、ループ速度k ループを抽出します。
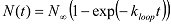 二相性が増加すると、二重指数関数を取り付けることができる。
二相性が増加すると、二重指数関数を取り付けることができる。 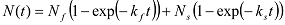 この場合、k個のループはから得られる。
この場合、k個のループはから得られる。 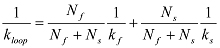 注:理論的には、ポリマーは時刻tにループしていないことを生存確率は、単一または二重の指数関数12ではありません。指数関数的なFITTING平均ループ時間を抽出するための実用的な手段として使用される。
注:理論的には、ポリマーは時刻tにループしていないことを生存確率は、単一または二重の指数関数12ではありません。指数関数的なFITTING平均ループ時間を抽出するための実用的な手段として使用される。
7。、J係数を決定
NOTE:J係数はdsDNAの一端が他端の周囲にどのように濃縮表す。これは、測定された速度ループなどの他の端部セグメントと同じ反応速度を産生するDNAの一方の端部セグメントの濃度を補間することによって決定することができる。実験的には、一方の端部セグメントが表面上に固定され、他方の端部セグメントは、特定の濃度cで導入される。両端の測定されたアニール速度がk アニールである場合、J因子21は次式で与えられる。 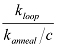 。アニール速度定数(k = kのアニールアニール / C)は、プローブ濃度とは無関係である。
。アニール速度定数(k = kのアニールアニール / C)は、プローブ濃度とは無関係である。
- 30〜50 pMのビオチン-Cy5のオリゴの20μL(プライマー3)を流れ、IニュートラコートされたチャネルNTO。未結合のオリゴを洗い流すために、100μlのT50とのチャネルを洗浄します。
- 50nMの最終濃度のCy3オリゴ(プライマー2)を添加した部分5に記載のように撮像緩衝液を調製する。チャンネルにこの画像バッファフロー。
- 上の532 nmのレーザーを維持しながら、簡単に表面に結合したCy5のオリゴの位置を識別するために640 nmのレーザーをオンにします。 640 nmのレーザーをオフにして、FRETシグナルの監視を開始。
注:表面に結合したCy5のオリゴへのCy3オリゴのハイブリダイゼーションの際、Cy5の蛍光は、FRETから発生します。 - MATLABスクリプトを使用して、結合していない状態(低Cy5の強度)で起動が、後にCy5の強度トレースから時間の関数としてアニールした状態(高Cy5の強度)に変わる分子の数を分析する。
- アニールされた分子対時間のこの数をプロットします。 (単一指数関数でこの曲線にフィット
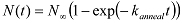 )アニーリング速度(k アニール ) を得た。
)アニーリング速度(k アニール ) を得た。 - アニール速度と反応物濃度との間の直線性を確認するために、さまざまなCy3のオリゴ濃度(60、100、および180 nm)のこの実験を繰り返します。二次アニール速度定数(k アニールを抽出する )の傾きから。
- Jからの係数を計算する
 k個のループは同じ緩衝液条件で測定ループレートである。
k個のループは同じ緩衝液条件で測定ループレートである。
Access restricted. Please log in or start a trial to view this content.
結果
ループ試験のために使用されるDNA分子は、可変配列および長さと互いに相補的な一本鎖オーバーハングの二本鎖領域からなる。 7塩基の長さである突出部は、ループ状態をキャプチャするために互いにアニールすることができる。各オーバーハングはダイト化学によりDNA骨格にリンクされているのCy3またはCy5のいずれかが含まれます。 Cy5標識オーバーハングもまた、ビオチン-TEG 表面固?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
FRETに基づく単純な単一分子アッセイは、異なる固有形状のDNAのループ動態を研究するために使用した。湾曲DNAは10.5塩基の螺旋周期と同位相で10マー配列を繰り返すことにより調製することができ、それらの曲率は、PAGEを用いて推定することができる。これらdsDNAs過渡ループの安定化を可能にするように粘着末端を有するように設計されている。私たちは、時間をかけてループされる分子の?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者らは、利害の衝突を宣言していません。
謝辞
私たちは、批判的に原稿を読み取るためのジェームズ·ウォーターズ、ゲーブルワズワースとボーブロードウォーターに感謝します。我々はまた、有益なコメントを提供するための4の匿名査読に感謝します。私たちは、ジョージア工科大学、科学的なインタフェースでバローズWellcomeの基金のキャリア賞、生物系のNSFの物理学の学生の研究ネットワークの助成金からの財政支援を認める。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Small DNA FRAG Extract Kit-100PR | VWR | 97060-558 | |
| Acrylamide 40% solution 500 ml | VWR | 97064-522 | |
| Bis-acrylamide 2% (w/v) solution 500 ml | VWR | 97063-948 | |
| GeneRuler 100 bp DNA Ladder, 100-1,000 bp | Fermentas | SM0241 | |
| Mini Vertical PAGE System | VWR | 89032-300 | |
| Syringe filter 0.2 μm CS50 | VWR | A2666 | |
| Trolox | Sigma-Aldrich | 238813-1G | triplet state quencher |
| Protocatechuic acid (PCA) | Sigma-Aldrich | 08992-50MG | oxygen scavenging system |
| Protocatechuate 3,4-Dioxygenase (PCD) | Sigma-Aldrich | P8279-25UN | oxygen scavenging system |
| mPEG-silane, MW 2,000 1 g | Laysan Bio | MPEG-SIL-2000-1g | |
| Biotin-PEG-Silane, MW 3,400 | Laysan Bio | Biotin-PEG-SIL-3400-1g | |
| Avidin, NeutrAvidin Biotin-binding Protein | Invitrogen | A2666 | |
| Phusion Hot Start High-Fidelity DNA Polymerase | New England Biolabs | F-540L | |
| Gel/PCR DNA Fragments Extraction Kit | IBI Scientific | IB47020 | |
| Premium plain glass microscope slides | Fisher Scientific | 12-544-1 | |
| VWR micro cover glass, rectangular, no. 1 | VWR | 48404-456 | |
| Fisher Scientific Isotemp 1006S Recirculating Chiller/Heater | Fisher Scientific | temperature control | |
| Objective Cooling Collar | Bioptechs | 150303 | temperature control |
| KMI53 Biological Micrometer Measuring Stage | Semprex | KMI53 | |
| High Performance DPSS Laser 532 nm 50 mW | Edmund optics | NT66-968 | Cy3 excitation |
| CUBE Fiber Pigtailed 640 nm, 30 mW, Fiber, FC/APC Connector | Coherent | 1139604 | Cy5 excitation |
| 650 nm BrightLine Dichroic Beamsplitter | Semrock | FF650-Di01-25x36 | splitting dichroic |
| LaserMUX Beam Combiner, reflects 514.5, 532, & 543.5 nm lasers, 25 mm | Semrock | LM01-552-25 | combining dichroic |
| Brightline Fluorescence Filter 593/40 | Semrock | FF01-593/40-25 | Cy3 emission filter |
| 635 nm EdgeBasic LWP longpass Filter, 25 mm | Semrock | BLP01-635R-25 | Cy5 emission filter |
| EMCCD iXon+ | Andor Technology | DU-897E-CS0-#BV | |
| IX51 inverted microscope frame | Olympus | ||
| Objective UApo N 100X/1.49 Oil TIRF | Olympus | ||
| Immersion oil type-F for fluorescence microscopy | Olympus | IMMOIL-F30CC | |
| 2 mm Diameter 45° Rod Lens Aluminum Coated | Edmund optics | 54-092 | miniature mirror |
| 1/4" Travel Single-Axis Translation Stage | Thorlabs | MS-1 | translation of miniature mirror |
| Ø1" Achromatic Doublet, ARC: 400-700 nm, f=200 mm | Thorlabs | AC254-200-A | focusing lens |
| Adjustable Mechanical Slit | Thorlabs | VA100 | |
| Dielectric Mirror | Thorlabs | BB1-E02 | |
| Ø1" Achromatic Doublet, f = 100 mm | Thorlabs | AC254-100-A | relay lens |
| Lens Mount for Ø1" Optics | Thorlabs | LMR1 | |
| Dichroic Filter Mount | Thorlabs | FFM1 | |
| Fixed Cage Cube Platform | Thorlabs | B3C | |
| Kinematic Mount for Ø1" Optics | Thorlabs | KM100 | |
| N-BK7 Plano-Convex Lens, Ø1", f = 40 mm | Thorlabs | LA1422-A | collimating lens |
| N-BK7 Plano-Convex Lense, Ø6.0 mm, f = 15 mm | Thorlabs | LA1222-A | telescope lens |
| N-BK7 Plano-Convex Lense, Ø6.0 mm, f = 150 mm | Thorlabs | LA1433-A | telescope lens |
参考文献
- Garcia, H. G., et al. Biological consequences of tightly bent DNA: The other life of a macromolecular celebrity. Biopolymers. 85, 115-130 (2007).
- Wiggins, P. A., et al. High flexibility of DNA on short length scales probed by atomic force microscopy. Nature Nanotechnology. 1, (2006).
- Lionberger, T. A., et al. Cooperative kinking at distant sites in mechanically stressed DNA. Nucleic Acids Research. 41, 6785-6792 (2011).
- Shore, D., et al. DNA flexibility studied by covalent closure of short fragments into circles. Proc Natl Acad Sci U S A. 78, 4833-4837 (1981).
- Geggier, S., Vologodskii, A. Sequence dependence of DNA bending rigidity. Proc Nat Acad Sci U S A. 107, 15421-15426 (1992).
- Smith, S. B., et al. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258, 1122-1126 (1992).
- Peters, J. P., Maher, L. J. DNA curvature and flexibility in vitro and in vivo. Quarterly Reviews of Biophysics. 43, 23-63 (2010).
- Cloutier, T. E., Widom, J. Spontaneous sharp bending of double-stranded DNA. Molecular Cell. 14, 355-362 (2004).
- Du, Q., et al. Cyclization of short DNA fragments and bending fluctuations of the double helix. Proc Natl Acad Sci U S A. 102, 5397-5402 (2005).
- Yuan, C., et al. T4 DNA ligase is more than an effective trap of cyclized dsDNA. Nucl. Acids Res. 35, 5294-5302 (2007).
- Manzo, C., et al. The effect of nonspecific binding of lambda repressor on DNA looping dynamics. Biophysical Journal. 103, 1753-1761 (2012).
- Le, T. T., Kim, H. D. Measuring shape-dependent looping probability of DNA. Biophys. J. 104, 2068-2076 (2013).
- Vafabakhsh, R., Ha, T. Extreme bendability of DNA less than 100 base pairs long revealed by single-molecule cyclization. Science. 337, 1097-1101 (2012).
- Friedman, L., et al. Viewing dynamic assembly of molecular complexes by multi-wavelength single-molecule fluorescence. Biophysical Journal. 91, 1023-1031 (2006).
- Kaplan, N., et al. The DNA-encoded nucleosome organization of a eukaryotic genome. Nature. 458, 362-366 (2009).
- Koo, H. S., Crothers, D. M. Calibration of DNA curvature and a unified description of sequence-directed bending. Proc Nat Acad Sci U S A. 85, 1763-1767 (1988).
- Prosseda, G., et al. A temperature-induced narrow DNA curvature range sustains the maximum activity of a bacterial promoter in vitro. Biochemistry. 49, 2778-2785 (2010).
- Rasnik, I., et al. Nonblinking and long-lasting single-molecule fluorescence imaging. Nature Methods. 3, 891-893 (2006).
- Cordes, T., et al. On the mechanism of Trolox as antiblinking and antibleaching reagent. J. Am. Chem. Soc. 131, 5018-5019 (2009).
- Aitken, C. E., et al. An oxygen scavenging system for improvement of dye stability in single-molecule fluorescence experiments. Biophys J. 94, 1826-1835 (2008).
- Taylor, W. H., Hagerman, P. J. Application of the method of phage T4 DNA ligase-catalyzed ring-closure to the study of DNA structure: II. NaCl-dependence of DNA flexibility and helical repeat. Journal of Molecular Biology. 212, 363-376 (1990).
- Bolshoy, A., et al. Curved DNA without A-A: experimental estimation of all 16 DNA wedge angles. Proc Natl Acad Sci U S A. 88, 2312-2316 (1991).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucl. Acids Res. 37, 6984-6990 (2009).
- Vologodskii, A., et al. Bending of short DNA helices. Artif DNA PNA XNA. 4, (2013).
- Hoover, D. M., Lubkowski, J. DNAWorks: an automated method for designing oligonucleotides for PCR-based gene synthesis. Nucl. Acids Res. 30, (2002).
- Waters, J. T., Kim, H. D. Equilibrium Statistics of a Surface-Pinned Semiflexible Polymer. Macromolecules. 46, 6659-6666 (2013).
- Mills, J. B., et al. Electrophoretic evidence that single-stranded regions of 1 or more nucleotides dramatically increase the flexibility of DNA. Biochemistry. 33, 1797-1803 (1994).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved