Method Article
Adherencia de bacterias a las superficies de midieron en el laboratorio de la planta
En este artículo
Resumen
Un método sencillo para medir y caracterizar la adherencia bacteriana a las plantas, especialmente las raíces y brotes, se describe en este artículo.
Resumen
Este manuscrito describe un método para medir la Unión bacteriana a las superficies de la planta axénicos en el microscopio óptico y mediante el uso de recuento de células viables. Materiales de planta usados incluyen raíces, brotes, hojas y cortan de frutas. Los métodos descritos son baratos, fácil y conveniente para pequeños tamaños de muestra. Enlace se mide en el laboratorio y puede utilizarse una variedad de medios de incubación y las condiciones. Puede determinarse el efecto de inhibidores. Situaciones que promueven e inhiben la Unión también pueden ser evaluadas. En algunos casos es posible distinguir si varias condiciones modificar enlace debido principalmente a sus efectos sobre la planta o en las bacterias.
Introducción
La medición de Unión bacteriana a las superficies de la planta se ha convertido en importante en tres situaciones diferentes. La primera situación es el examen de la transmisión de los patógenos humanos en planta superficies1,2,3. El objetivo aquí es para prevenir el atascamiento bacteriano o eliminar o matar bacterias encuadernadas y así reducir la transmisión de la enfermedad en material vegetal. La segunda situación es el examen de la Unión de patógenos de plantas a sembrar superficies4. Una vez más el objetivo aquí es para evitar vinculante o eliminar o matar bacterias encuadernadas y así reducir la enfermedad. La tercera situación es el examen de la fijación de las bacterias simbióticas o promoción de crecimiento vegetal5,6. El objetivo aquí es promover bacterias vinculante y así al aumento de la salud de las plantas y cultivos los rendimientos.
Las técnicas para medir la Unión bacteriana a superficies descritas en este artículo la planta son baratos y relativamente fáciles de realizar. Los únicos requisitos son un microscopio y materiales encontrados generalmente en un laboratorio de bacteriología. Para algunas técnicas un sonicador de baño es útil. Las técnicas descritas están diseñadas para atar los experimentos realizados con muestras relativamente pequeñas. De Unión se realizan en el laboratorio, aunque es posible modificar algunas de estas técnicas para el uso en el invernadero o en el campo.
Estas técnicas se han utilizado para medir la Unión bacteriana a las raíces, brotes, hojas de corte, corte frutas y tomates intactos en el laboratorio7,8,9,10,11, 12,13,14,15. Se han utilizado también para medir la colonización de raíces de plantas que crecen en el suelo o arena en el laboratorio16. Las técnicas se han utilizado muchas especies bacterianas incluyendo tumefaciens de la agrobacteria, Sinorhizobium meliloti, Escherichia coli, Salmonella enterica y Pseudomonas fluorescens. Una útil descripción de métodos para evaluar la interacción de a. tumefaciens con superficies puede encontrarse en Morton y Fuqua (2012)17. En todos los casos los tamaños de la muestra participantes eran pequeños, generalmente menos que las plantas de 25-50. Las técnicas descritas son adecuadas para uso con patógenos humanos que deben mantenerse independientes durante los experimentos.
Protocolo
1. preparación del Material vegetal axénicos
- Preparar las plántulas cultivadas en agua.
- Coloque una pequeña cantidad de semillas (menos de 30) en un 30, 50, 100 o vaso de vidrio de 150 mL. Hemos utilizado tomate, alfalfa, Arabidopsis thaliana, guisante, haba, tabaco, lechuga y semillas de zanahoria con este protocolo.
Nota: Para determinar cuantas semillas se pueden esterilizar juntos sin separarse de una semilla a otro tipo de contaminación, ver paso 1.1.2. - Cubra las semillas con etanol al 80% y agitar brevemente. Remojar las semillas durante 1 minuto.
- Quite el etanol y cubrir las semillas con una solución de 50% de lejía comercial volumen (ClONa) y 0,1% Tritón X-100 en agua del grifo. Remojar las semillas por 20 min.
Nota: Si las semillas son grandes, como las semillas de frijol, puede ser necesario alargar el tiempo de remojo para eliminar totalmente los hongos en la superficie de la semilla. - Retirar la mezcla de lejía y lavar las semillas 3 veces con agua estéril. Remojar las semillas en agua durante 1 minuto para cada lavado.
- Para obtener plántulas axénicos añadir una pequeña cantidad de agua estéril entre 5 y 25 mL dependiendo del tamaño de las semillas. Vierta el agua y las semillas en una placa Petri estéril para la germinación de la semilla.
Nota: El agua debe cubrir el fondo del plato pero no cubre las semillas para fomentar la formación de pelos radicales. Axénicos describen una cultura en la que sólo hay una sola especie de organismo. - Incubar en la oscuridad hasta que las plántulas alcancen el tamaño deseado entre 1 cm y 10 cm (aproximadamente 5 días para tomate y a. thaliana y 1 a 3 días para alfalfa). Para las semillas más grandes utilice un recipiente estéril cubierto con una capacidad de más de 100 mL como un plato de cristal cubierto con papel de aluminio.
- Coloque una pequeña cantidad de semillas (menos de 30) en un 30, 50, 100 o vaso de vidrio de 150 mL. Hemos utilizado tomate, alfalfa, Arabidopsis thaliana, guisante, haba, tabaco, lechuga y semillas de zanahoria con este protocolo.
- Determinar la frecuencia de contaminación de un lote de semillas especialmente con microorganismos que permanecen viables en o dentro de las semillas después de la esterilización de superficie.
Nota: Esto es necesario porque ocasionalmente semillas llevan microorganismos debajo de la capa de semilla que no puede morir por la esterilización de superficie. Utilizar la frecuencia de semillas contaminadas en un lote de semillas para determinar cuantas semillas para esterilizar en un momento sin un alto riesgo del grupo de semillas se contamine debido a una semilla con bacterias u hongos que sobreviven al tratamiento.- Llevar a cabo medidas 1.1.1 a la 1.1.3.
- Coloque las semillas en una placa Petri de agar nutritivo. Poner 10-30 semillas en cada plato los espaciado hacia fuera por lo que puede ser marcados individualmente. Sello de los platos con cinta o sellado de la película.
- Incubar durante 3-5 días a 25 ° C, anotando cada día por consecuencia visible de microorganismos a partir de las semillas.
- Preparar las plántulas cultivadas en arena. Las plantas de semillero se pueden cultivar en arena para el uso en experimentos de adherencia bacteriana.
- Llevar a cabo medidas 1.1.1 a la 1.1.3.
- Esterilizar la arena de cuarzo o sea en el autoclave. Ambos contienen algún material orgánico. Si esto influirá en el experimento, lave la arena cubriendo con dos veces su volumen de 0.1 M HCl. mezclar durante 10 minutos deje que la arena para colocar y retirar el líquido. Enjuague la arena 3 veces con agua, y una vez con 80% etanol seguido de dos enjuagues de agua adicional usando el mismo protocolo en cuanto a lo 0.1 M HCl. esterilizar la arena lavada en la autoclave.
- Esterilizar envases por sumergirse en lejía de 50% durante 5 minutos y enjuagar 5 veces con agua estéril por sumersión. Permita que seque y selle la parte inferior con la película. La película obtenida de los fabricantes es generalmente libre de microorganismos en el interior lado que debe ser colocado hacia el interior del contenedor.
- Mezclar la arena estéril con suficiente agua para mojar que los palillos de arena. La cantidad dependerá de qué tan seca la arena es. 10-35% del volumen de la arena suele ser suficiente. Coloque la arena húmeda en el recipiente permitiendo suficiente profundidad para la longitud de raíces necesaria y suficiente espacio por encima de la arena para el crecimiento de los brotes. Las distancias exactas dependen de la especie y la variedad de planta que se utiliza.
- Semillas de la planta.
- Hacer un agujero poco profundo en la arena con una varilla de vidrio estéril sólo lo suficientemente profunda para colocar la semilla debajo de la superficie de la arena (aproximadamente 1-5 mm). Colocar la semilla en el agujero. Cubrir con una fina capa de arena. Selle la parte superior del recipiente con película para evitar pérdida de agua y entrada de microbios adicionales.
- Crecer las plantas en el laboratorio bajo una luz o en el invernadero a una temperatura adecuada y duración del día para las especies y la variedad de planta. Para el tomate, alfalfa, o Arabidopsis thaliana uso temperatura y ciclos de luz/oscuridad de 12 h.
- Planta de semillero en un agujero en la arena (1-2 mm de diámetro y de profundidad suficiente que la raíz se cubrirá con arena). Hacer el agujero con una varilla de vidrio estéril. Guía la raíz cuidadosamente en el orificio usando un gancho de crochet de acero inoxidable estéril si es necesario y los lados del agujero se llenan de arena.
- Por otra parte, crecen plantas axenically o en agar que contienen una mezcla de sales tales como sales de MS. Uso de brotes de plantas axénicos en agar directamente. Evite las raíces crecidas con agar agar se adhiere a la superficie de la planta y las bacterias. Esto puede causar una impresión falsa de la adherencia bacteriana a la superficie de la planta.
2. preparación de otros materiales vegetales
- Lavar el material vegetal cultivado en invernadero con agua antes de usarla para reducir el número de microbios indígenas. Habrá bacterias y hongos en las plantas. Protozoos estará presentes en el suelo, en las raíces y, posiblemente, en las hojas. Lavar con lejía o etanol alterará la superficie de la planta y no se recomienda.
- Si después de lavado con agua un número considerable de microbios siguen siendo, tratar las plantas con el jabón de mano líquido diluido o diluir el peróxido de hidrógeno (0.01%) para eliminar o matar los microbios. Esto es generalmente menos perjudicial para la planta de blanqueo o etanol.
- Como material vegetal comprado en el mercado local es difícil de descontaminar, elegir el material que parece no han sido sujetos a almacenamiento prolongado. Evitar partes del material que aparecen dañados o marrón.
- Lavar con agua y cortes frescos en los extremos del material que previamente se han cortado antes de usarlo, a menos que las interacciones de las bacterias de prueba con las bacterias (y eucariotas) que haya acumulado en los sitios de corte son de interés. No lavar con lejía o etanol como alterará la superficie de la planta.
- Tratamiento de sitios de la herida. Sitios de la herida a menudo acceso las bacterias al interior del material de planta18.
- Bloquear o reducir la adhesión bacteriana a y movimiento a través, de herida o corte sitios creados en la preparación del material por sumergir el borde de corte en parafina derretida o por el sitio de la pintura con parafina derretida usando un cepillo pequeño. No utilice una espátula o con cualquier instrumento agudo ya que puede dañar el tejido.
- Sello vástago cicatrices en frutas como tomates con parafina. Algunas bacterias nadarán en el área debajo de la parafina, pero sus números son generalmente pequeños.
- Si la entrada bacteriana en sitios de la herida es de interés, estimación de la distancia que las bacterias pueden moverse llevada por corrientes de agua o difusión observando el movimiento de un colorante añadido a la solución. Marque las bacterias móviles mediante la introducción de un gen que codifica una proteína fluorescente como la proteína verde fluorescente (GFP) en ellos y trazarlos usando microscopía de fluorescencia se describe en paso 6.1.219.
3. preparación de las bacterias
- Crecen las bacterias. Uso un medio que más se aproxime a las condiciones de las bacterias es probable que hayan estado expuestas a inmediatamente antes de encontrar la planta en el mundo real fuera del laboratorio. Carbono y nitrógeno las fuentes así como la presencia de iones, cationes particularmente divalentes (Ca, Mg, Fe, Mn y Zn) y fosfato y el pH del medio son importantes.
- Use caldo de Luria o medio AB mínimo para a. tumefaciens y otras bacterias de suelo20. Para e. coli, que pueden originarse en el intestino, utilizar caldo de Luria.
- Añadir inductores tales como exudados de la raíz o extractos de plantas comerciales como soytone o azúcares como la sacarosa o xilosa con la media21. Si estas sustancias se utilizan como inductores, añadir una concentración baja, por ejemplo 0,01%. Si son fuentes de carbono usado, añadir una mayor concentración, por ejemplo, 0.1%.
- Preparar el inóculo bacteriano. Diluir las bacterias en agua estéril o en el medio en el que la incubación se realizará y añadir al material de planta. La dilución se discute en los pasos 4.2 y 4.3.
- Para eliminar el medio de crecimiento antes de utilizar la bacteria, centrifugar la suspensión bacteriana a 10.000 x g durante 2 minutos, retirar el sobrenadante y resuspender las bacterias con un vórtex en el mismo medio que se utilizará para la incubación de material vegetal. Este método puede quitar o disminuir el número o la cantidad de material extracelular y apéndices como exopolisacáridos, cápsulas, flagellae y pili. Si es importante que estas estructuras superficiales permanecen intactas, simplemente diluir las bacterias antes de inocularlos o utilizar el método alternativo descrito en el paso 3.2.2.
- Como un método alternativo para eliminar el medio de cultivo, recoger los gérmenes en un filtro de nitrocelulosa o policarbonato con un tamaño de poro de 0,2 μm o menos. Lave las bacterias con el medio de incubación y resuspender por agitación suave o vórtex del filtro en un recipiente estéril del medio.
4. inoculación de las bacterias
- Determinar el número de bacterias para inocular con referencia a la medición que se utilizará para determinar la adherencia bacteriana y la duración de la incubación.
- Para estudios microscópicos con incubación veces menos de 1 día inocular cantidades relativamente grandes de bacterias. Añadir una cantidad de la cultura para alcanzar una concentración bacteriana final de más de 106 bacterias / mL. Para tiempos más largos de incubación disminuyen el tamaño del inóculo bacteriano.
- Para estudios en el que se medirá la adherencia bacteriana por recuentos de células viables, agregue una cantidad de la cultura para alcanzar una concentración bacteriana final de 103 106 bacterias / mL.
- Evitar la adición de tantas bacterias que su metabolismo cambia la concentración de pH u oxígeno en el medio de incubación. Medir el pH con papel pH o un electrodo. Medir la concentración de oxígeno usando un electrodo de oxígeno.
- Para plantas cultivadas en arena, inocular en tres maneras posibles.
- Inocular la semilla con las bacterias antes de la siembra remojando la semilla durante 1 min en una suspensión en agua de alrededor de 106 bacterias / mL.
- Inocular la raíz por germinar plántulas axénicos como se describe en la sección 1 y la raíz de la plántula es cerca de 1 cm de largo que lo sumerge o colocar la plántula entera en una suspensión de 106 bacterias / mL en agua durante 1 minuto.
- Inocular la arena por las bacterias de la mezcla con la arena antes de plantar para obtener una concentración final de aproximadamente 103 bacterias por mL o por regar las plántulas con una suspensión de 106 bacterias / mL después de la siembra.
5. incubación de las bacterias con el Material vegetal
- Para la incubación en medio líquido, incubar las bacterias con el material vegetal en agua estéril o mineral y la sacarosa sales o medio de cultivo de tejido de la planta (como un 1:10 dilución de sales MS)22,23.
- Utilice un recipiente para que las bacterias no se adhieren. Trate de mantener la superficie de la planta continuamente cubierto por inmersión o por agitación suave. Agitación vigorosa puede prevenir la adherencia o incluso eliminar las bacterias de la superficie de la planta.
- Observar el material vegetal en el microscopio óptico después de diferentes intervalos de tiempo para determinar cuándo detener la incubación y realizar mediciones. Un curso de tiempo de adherencia suele ser valioso con muestras tomadas cada 1 a 4 h o cada día dependiendo de la velocidad de la interacción.
- Para incubar en la arena, se aplican las mismas consideraciones descritas para la incubación en medio líquido en el paso 5.1.
6. medición de la adherencia mediante microscopía
- Hacer mediciones microscópicas con óptica Nomarski o contraste de fase para una fácil visualización de las bacterias en las superficies. Sin embargo, puede utilizarse cualquier microscopio de campo brillante con la ampliación de 20 X o superior.
- Utilizar la observación en el microscopio para determinar si las bacterias están aleatoriamente distribuidas o ubicadas en sitios específicos. También Compruebe si están obligados por sí solos o en racimos. Buscar la presencia de microcolonias sugiriendo crecimiento bacteriano o captura después de la adhesión. Comprobar si las bacterias parecen formar un biofilm en la superficie.
Nota: El biofilm es una gran cantidad de bacterias atados a la superficie y rodeados por una matriz extracelular23,24,25. La estructura puede ser liso y uniforme o tienen una arquitectura más compleja. Métodos para el estudio de biofilms asociados con superficies de planta han sido descritas17. - Uso de bacterias con la etiqueta con un marcador fluorescente. Si están presentes otras bacterias y las bacterias de interés son identificadas por una etiqueta fluorescente como la proteína verde fluorescente, uso de microscopía de fluorescencia para determinar la presencia de la bacteria etiquetada en clusters de otras bacterias26. Para GFP, use un filtro con 490 excitación nm y 520 nm emisión.
- Compruebe que la etiqueta fluorescente es no perjudicar las bacterias mediante la observación de la adherencia al material axénico de una mezcla de bacterias de tipo silvestre y etiquetado de la misma cepa con óptica Nomarski y fluorescencia. Si las bacterias fluorescentes y oscuras son aleatoriamente mezclados y presente en números iguales entonces la etiqueta no interfirió con el ensayo.
- Utilizar la observación en el microscopio para determinar si las bacterias están aleatoriamente distribuidas o ubicadas en sitios específicos. También Compruebe si están obligados por sí solos o en racimos. Buscar la presencia de microcolonias sugiriendo crecimiento bacteriano o captura después de la adhesión. Comprobar si las bacterias parecen formar un biofilm en la superficie.
- Determinar el número de bacterias adjuntos.
Nota: Es muy difícil determinar el número de bacterias adjuntos en el microscopio. Cuando la Unión está a superficies de planta irregular, generalmente no es posible obtener una medida cuantitativa. Microscopía electrónica de barrido (no discutido en este artículo) se puede utilizar para hacer estas mediciones.- Cuando las bacterias están limitadas a una superficie lisa como un pelo de raíz, contar el número de bacterias a la orilla del pelo de raíz por mm de longitud de pelo de la raíz. Tenga cuidado de utilizar pelos de la raíz de aproximadamente el mismo tamaño y la forma en la comparación de las mediciones.
- Para determinar el tamaño de los objetos en el microscopio, utilice una diapositiva comercial con marcas de medida en él. Observar y fotografiar esta diapositiva en la misma configuración que utiliza para el material experimental y uso de las imágenes resultantes para determinar el tamaño de los objetos en las fotomicrografías.
- Preparar la muestra para microscopía.
- Lavar la muestra. Hacia la muestra una gota de agua o incubación medio en un portaobjetos de microscopio y observar directamente.
Nota: Esto tiene la ventaja de que si hubo ningún crecimiento bacteriano o muerte bacteriana real allí es improbable que muchas bacterias libres. Tomar la ausencia de bacterias libres como una señal de advertencia que puede haber habido muerte bacteriana o atascamiento bacteriano al contenedor en el que la incubación se llevó a cabo. El efecto de lavado de la muestra se muestra en la figura 1. - Lave suavemente la muestra en medio de agua o de incubación colocando en un frasco de líquido y invertir el frasco suavemente. Luego coloque la muestra en el portaobjetos del microscopio en fresco líquido para la observación.
- Montaje de la muestra en líquido usando un ordinario cubreobjetos y portaobjetos.
- Si la muestra es gruesa y así haría un bulto bajo el cubreobjetos, utilizar un cubreobjetos de press-apply. Estos vidrios de cobertura tienen un anillo de goma o de plástico alrededor del borde de la hoja de cubierta. Colocar el líquido y la muestra en el pozo en el cubreobjetos y colocar el portaobjetos en la parte superior y presione suavemente para sellar el cubreobjetos a la diapositiva. Invertir y examinar.
- Como alternativa utilizar un alga con portaobjetos y cubreobjetos de manera similar. Nota que se mueve con esta profundidad generalmente no se puede examinar con un objetivo de más de 20 aumentos.
- Lavar la muestra. Hacia la muestra una gota de agua o incubación medio en un portaobjetos de microscopio y observar directamente.

Figura 1 : Pasos en la preparación de una muestra para determinar el número de bacterias encuadernados. Unión de a. tumefaciens a pelos de la raíz de tomate (A, B y C) y a hilos de nylon (D, E y F). En las muestras montadas en agua sin lavar ambos obligados bacterias (flechas negras) y las bacterias libres (flechas blancas) se pueden ver (A y D). Después de lavar que las bacterias límite siendo pero las bacterias libres ya no son actualmente (B y E). Después de la sonicación se han eliminado las bacterias dependientes de la superficie de la muestra (C y F). Haga clic aquí para ver una versión más grande de esta figura.
- Si se utilizaron bacterias fluorescentes, examinar la muestra en el microscopio después de retirar la arena. Las raíces de las plantas cultivadas en arena generalmente no son adecuadas para la microscopia como las partículas de arena interfieren con la observación de las bacterias.
- Retire la planta de un recipiente de crecimiento según el paso 7. Coloque las raíces en un recipiente de agua y mezclar suavemente para quitar la arena que se asentarán en el fondo del recipiente.
- Retire el agua de lavado de la planta y montaje de la muestra como en el paso 6.3.3.
7. medición de la adherencia utilizando células viables cuenta
- Determinar el número de bacterias viables adjuntos utilizando un sonicador.
- Eliminar las bacterias. Colocar la muestra en un frasco con suficiente agua, tampón o incubación medio para cubrir el material de planta e invertir el frasco varias veces.
- Si hay más de 10 bacterias libres3 por mL presente en la incubación inicial, realizar lavados secuenciales para eliminar a todos ellos. Ver en un microscopio de luz (ver paso 6.3) para determinar la presencia de bacterias libres.
- Lavar hasta que hay una reducción sustancial en el número de bacterias libres pero recuerda que existe un equilibrio entre bacterias consolidadas y no consolidadas, por lo que el número de bacterias libres nunca disminuya a cero.
- Retire la muestra de la cubeta de lavado líquido con una espátula o pinzas.
- Determinar el número de bacterias encuadernados en incubaciones de plantas en líquido.
- Suspender la muestra lavada en un frasco y cubrirla con un volumen medido de agua, el medio de incubación o tampón de lavado. Use suficiente líquido para cubrir la muestra. Colocar el frasco en un sonicador de baño y someter a ultrasonidos para un minuto.
- Retire la muestra y examinar bajo el microscopio óptico para determinar si la bacteria dependiente ha sido removida de la planta. La eliminación de bacterias de una muestra por sonicación se muestra en la figura 1. Si hay bacterias aún consolidadas presentes continúan sonicando la muestra hasta que tal uno que no limite las bacterias quedan en la superficie de la muestra. Si la bacteria dependiente no puede extraerse por sonicación, añadir arena de cuarzo estéril de 1-10 mg/mL y repetir el baño de ultrasonidos y examen microscópico.
- Determinar que el procedimiento de sonicación que parece más eficaz en el paso 7.1.3.2 no reduce la viabilidad de las bacterias mediante la suspensión de bacterias de un cultivo líquido de la solución que se utilizará para la sonicación (Añadir arena de cuarzo, si fuera necesario). Determinar el número de células viables. Someter a ultrasonidos las bacterias y determinar nuevamente el conteo de células viables.
Nota: Si hay una reducción en el recuento de células viables, modificar el procedimiento cambiando la composición del líquido o sonicación tiempo hasta que el tratamiento no tiene ningún efecto en el recuento de células viables. - Determinar que el tampón de dilución utilizado para el recuento de células viables no afecta la viabilidad de las bacterias que han sido incubados bajo condiciones utilizadas comparando un recuento viable utilizando búferes de dilución diferentes o de agua.
- Eliminar las bacterias. Colocar la muestra en un frasco con suficiente agua, tampón o incubación medio para cubrir el material de planta e invertir el frasco varias veces.
- Determinar el número de bacterias viables adjuntos mediante homogeneización.
- Colocar la muestra en un mortero estéril o licuadora u otro dispositivo de homogeneización. Cubrirlo con un volumen medido de agua estéril, medio de incubación o lavado Tampón27. Utilizar un volumen suficiente para cubrir la muestra. Utilice una licuadora 100 mL.
- Moler la muestra hasta que está bien homogeneizada. Comprobar el pH después de homogeneización para asegurarse que ácido liberado de los tejidos de la planta no ha causado una fuerte caída en el pH debajo de 7. Si el pH ha bajado utiliza un búfer como tampón fosfato en el líquido de homogeneización para mantener el pH.
- Determinar el número de células viables.
- Uso de bacterias con resistencia a los antibióticos.
- En aquellas situaciones en las que más de un tipo de bacteria presente, marca las cepas bacterianas usando espontánea resistencia a los antibióticos (generalmente rifampicina y ácido nalidíxico).
- Determinar el nivel de antibiótico a utilizar. Si se dispone de las culturas de los otros organismos debe estar presente en la incubación, la placa de estas culturas cultivadas bajo las condiciones de la incubación prevista con plantas en placas, con un rango de concentraciones de los seleccionados antibióticas. Determinar la concentración mínima que no permite crecimiento de cualquiera de estos organismos. Esta es la menor concentración de antibiótico a la cual las bacterias de prueba tendrá que ser resistente.
- Obtener a espontáneas mutantes resistentes a los antibióticos. Cultivar una cultura de las bacterias en medio rico para registro tardío o fase estacionaria. 0,1 mL sin diluir sobre una placa que contiene el antibiótico deseado en una concentración adecuada de la placa. Determinar las concentraciones a utilizar como se describe en el paso 7.3.1.1. Mantener y purificar las bacterias que crecen y por lo tanto son resistentes a los antibióticos.
- Determinar que la resistencia a los antibióticos no reduce el crecimiento de las bacterias haciendo una curva de crecimiento en medio líquido de la matriz y las cepas resistentes a los antibióticos. Determinar que las bacterias resistentes a los antibióticos muestran el mismo nivel de colonización de material vegetal axénicos como la cepa parental, si es posible.
- En aquellas situaciones en las que más de un tipo de bacteria presente, marca las cepas bacterianas usando espontánea resistencia a los antibióticos (generalmente rifampicina y ácido nalidíxico).
- Determinar el número de bacterias viables a las plantas cultivadas en arena.
- Retirar la planta del contenedor y la arena. Para retirar las plantas de contenedores, primero retire el material de sellado en la tapa y el fondo del recipiente. Coloque el recipiente sobre un trozo de papel estéril y suavemente Retire la arena entera o suelo y planta como un gran cilindro cónico golpeando suavemente el recipiente contra una superficie para aflojar el material.
- Si es necesario, utilice una espátula o varilla para aflojar el material alrededor de los bordes va la parte inferior del envase.
- Cuando el cilindro de arena o tierra que contiene la planta es libre en el papel, partirla por la mitad para revelar la raíz de la planta. Si lo desea, tomar muestras de la arena cerca del borde del contenedor, así como cerca de la raíz. Esto puede ser útil para determinar la extensión y acumulación y crecimiento de las bacterias.
- Mida la longitud de la raíz. Coger la raíz y eliminar la arena y bacterias adherirse libremente a la raíz (el material de la rizosfera) por inmersión de la raíz en un volumen medido de agua o buffer y agitarlo suavemente. Determinar el recuento de células viables de la bacteria en la suspensión resultante por la galjanoplastia en un medio adecuado tal como agar Luria. Esto representa el número de bacterias libremente asociada con la raíz.
- Retirar la planta del contenedor y la arena. Para retirar las plantas de contenedores, primero retire el material de sellado en la tapa y el fondo del recipiente. Coloque el recipiente sobre un trozo de papel estéril y suavemente Retire la arena entera o suelo y planta como un gran cilindro cónico golpeando suavemente el recipiente contra una superficie para aflojar el material.
- Quite las bacterias firmemente consolidadas por sonicación y determinar sus números tal como se describe en el paso 7.1.
- Por otra parte, para determinar la ubicación de la raíz de las bacterias firmemente consolidadas Coloque la raíz lavada en la superficie de una placa de Petri que contienen agar nutritivo u otro medio adecuado. Observe la ubicación de colonias de bacterias en la raíz durante los próximos 3 días usando un microscopio de disección o lupa.
- Expresar los resultados como número de bacterias por planta, por cm2 de superficie, por cm longitud de la raíz o por gramo de peso fresco de tejido. Hacer varias repeticiones el mismo día y también hacer repeticiones en diferentes días utilizando diferentes culturas bacterianas y diferentes lotes de material vegetal.
8. determinar si un efecto de las condiciones de incubación en adherencia debido a una respuesta de las bacterias o la planta
- Utilizar material vegetal vivo o muerto.
- Someter la planta a una gran variedad de productos químicos, fijadores u otros tratamientos como el calor con el fin de acabar con él. Lave bien el material vegetal en el medio de incubación y agua después del uso de cualquiera de estos tratamientos. Luego inocular la bacteria. Esto no va a destruir la superficie de la planta, pero lo hará metabólicamente inactivos para que no se puede responder a las bacterias.
- Medir la adherencia bacteriana como se describe en los pasos 6 y 7. También determinar el número de células viables a la incubación con el material de planta al principio de la incubación y el número de células viables presentes en el final de la incubación para asegurar que no hay sustancias químicas tóxicas estaban presentes durante la incubación.
- Uso de material inanimado.
- Adherencia bacteriana uso a material inanimado para determinar si un efecto sobre la adherencia bacteriana al material vegetal es debido a un efecto sobre la planta o las bacterias. Elegir un material inanimado para que las bacterias se unen y que es similar en forma y tamaño para el material vegetal estudiado. Las posibilidades incluyen papeles de filtro de todos los tipos (celulosa, nitrocelulosa, fibra de vidrio, policarbonato, etcetera), hilos (nylon, algodón, poliéster, lana de vidrio, etcetera), vidrio o plástico cubreobjetos, cupones de acero inoxidable y diálisis membranas.
- Lavar el material inanimado completamente en el agua y el medio en el que la incubación se realizará y esterilizar antes de uso. La mayoría de los materiales listados en el paso 8.2.1 es estable a la esterilización en autoclave.
- Incubar el material inanimado bajo las condiciones deseadas y cuenta como se describe en los pasos 6 y 7. Un ejemplo del uso de hilos de nylon para determinar a que obedece el atar reducido de a. tumefaciens a pelos de la raíz en las concentraciones de calcio alto (al menos en parte) a un efecto del calcio sobre la bacteria se muestra en la figura 428.
Resultados
A. tumefaciens coloniza las superficies radiculares. Con el fin de determinar si la producción bacteriana de la celulosa desempeña un papel en la colonización de la raíz, los efectos de las mutaciones bacterianas que impiden la síntesis de celulosa fueron examinados16. Se utilizaron las técnicas descritas en los pasos 1,3 y 7.1. Semillas de tomate eran superficie esterilizada y germinadas en agua estéril. Cuando las raíces fueron aproximadamente 2 cm de largo fueron sumergidas en una suspensión de 105 bacterias / mL y plantados en el suelo pasteurizado en contenedores. Las plantas fueron cultivadas durante 14 días a 25 ° C en un ciclo oscuro h 12 h luz/12. Después de la indicada veces las plantas fueron removidos de los contenedores. Las raíces fueron lavadas y sonicadas en un sonicador de baño para eliminar las bacterias encuadernadas. Números de bacterianos se determinaron utilizando el recuento de células viables. La figura 2 muestra el efecto de dos diversas mutaciones de celulosa-menos en la capacidad de las bacterias para colonizar las raíces de tomate. Aunque las desviaciones estándar de las mediciones eran tan altas como 0.9 log10 (un problema común con este tipo de medición) la reducción en el atascamiento de los mutantes menos celulosa es claramente evidente y podemos concluir que la producción bacteriana de celulosa ayuda a la bacteria en la colonización de raíces de tomate.
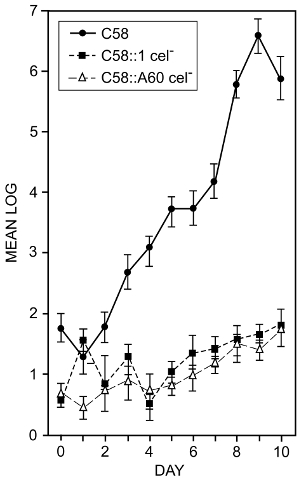
Figura 2 : Raíz de la colonización de tipo salvaje y menos celulosa mutantes de a. tumefaciens. Log10 número total de bacterias por cm longitud de la raíz de raíces de tomate inoculadas con cepa de tipo salvaje a. tumefaciens C58 y mutantes menos celulosa C58:1 y C58:A60. Los números que aparecen son el medio de un mínimo de cuatro experimentos separados. Las barras indican las desviaciones estándar de los medios. Las raíces se inocularon por inmersión en una suspensión de 105 bacterias / mL para un minuto. Las plantas fueron cultivadas en contenedores y las bacterias libremente adherentes fueron quitadas por el lavado de las raíces en lavado tampón en un frasco de vidrio. Bacterias firmemente adheridas fueron quitadas usando un sonicador de baño y la suspensión resultante plateado para determinar recuento viable16. Esta figura ha sido modificada de Matthysse y McMahan. Haga clic aquí para ver una versión más grande de esta figura.
Se examinó el papel de exopolisacáridos en la Unión de e. coli y otras bacterias a brotes de alfalfa. Algunos brotes de enfermedad diarreica por e. coli O157: H7 se trazaron a brotes de alfalfa contaminada. Unión de las bacterias de tipo silvestre y mutantes incapaces de hacer distintos exopolisacáridos se midió usando los métodos descritos en los pasos 1.1, 5.1 y 7.2. Brotes de alfalfa fueron superficie esterilizada y germinó un día en agua estéril a 25 ° C en la oscuridad. Cuatro brotes con las capas de semilla adjuntadas fueron colocados en platos de plástico estériles que contiene 5 mL de agua. Las bacterias cultivadas en caldo Luria fueron agregadas a una concentración final de aproximadamente 5 x 103 / mL. Los brotes inoculados se incubaron a 25 ° C en la oscuridad durante 3 días. Los brotes fueron lavados dos veces en 5 mL de agua estéril en un vial por inversión vigorosa y homogeneizó en lavado Tampón utilizando un homogeneizador de vidrio teflón motorizado. Experimentos anteriores utilizando bacterias marcan con un plásmido portador del gen GFP demostrado ningunas bacterias internalizadas aunque fácilmente se observaron bacterias superficiales. Los resultados se muestran en la tabla 1. Examinaron a dos cepas de e. coli O157: H7. En ambas cepas la producción de poli-β-1, 6-glucurónico acid(PGA) parece hacer la contribución más grande a la Unión del patógeno e. coli a las superficies de la planta. Ácido colónico también desempeñó un papel significativo en la Unión. Mientras que la reducción en el atascamiento en celulosa menos mutantes fue significativa no era tan grande como para los otros dos polisacáridos.
| Efectos de las mutaciones en los Genes de producción de exopolisacárido en encuadernación de E. coli O157: H7 brotes y capas de semilla abiertas | |||
| Cepa bacteriana | Mutación o genotipo (fenotipo relevante) | Sesión número10 de bacterias límite por brote o Testa | |
| Alfalfa brotesb | Capas de semilla abiertas | ||
| 86-24 | Ninguno (tipo salvaje) | 4.7 ± 0.6 | 5.6 ± 0,2 |
| 8624N | yhjN (celulosa-minus) | 2,9 ± 0,7c | 3.5 ± 0.6c |
| C 8624 | wcaD (colon menos ácido) | de 1,8 ± 0,7c | 2.4 ± 0,5c |
| 8624P | Camp (PGA-minus) | < 1.0c | ± 1.0 1.0c |
| DEC4A | Ninguno (tipo salvaje) | 5.6 ± 0,2 | 6.1 ± 0.3 |
| DEC4AN | yhjN (celulosa-minus) | 4.8 ± 0.8d | 4.1 ± 0,8d |
| DEC4AC | wcaD (colon menos ácido) | 3,9 ± 0,5c | 4.8 ± 0.8d |
| DEC4AP | Camp (PGA-minus) | < 1.0c | 1,2 ± 0,7c |
| una media ± desviación estándar de un mínimo de tres mediciones de la cantidad (log10) de bacterias límite después de 3 días. | |||
| b brotes fueron lavados antes de la medición. | |||
| c significativamente diferente del tipo silvestre: P < 0.01. | |||
| d significativamente diferente del tipo silvestre: P < 0.05. | |||
| Esta tabla ha sido modificada de Matthysse, Deora, Mishra y Torres10. | |||
Tabla 1: efectos de mutaciones en genes de producción de exopolisacárido en Unión de E. coli O157: H7 a brotes. Para determinar el papel de exopolisacáridos y lipopolisacáridos en la Unión del patógeno e. coli O157: H7 cepas de brotes de alfalfa, la Unión de un conjunto de mutantes para los brotes y abiertas capas de semilla fue examinada usando los métodos se describe en el paso 6. Los resultados mostraron que poli-ß-1, 6 -N-acetil-D-glucosamina (PGA) parece ser esencial para la Unión a brotes y eso ácido de la celulosa y colanic son necesarios para la máxima unión de e. coli O157. Esta tabla ha sido modificada de Matthysse, Deora, Mishra y Torres10.
Con el fin de determinar si la producción de PGA es suficiente causar el atascamiento bacteriana a las superficies, un plásmido (pMM11) llevando el operón clonado los genes necesarios para la PGA de codificación producción fue introducida en dos diferentes bacterias que no Normalmente podrá enlazar a las raíces de tomate10. A. tumefaciens A1045 es un mutante de la cepa de tipo salvaje C58 que no puede hacer el glucano cíclico-β-1, 2 y tampoco se unen para plantar superficies29. Sinorhizobium meliloti 1021 que forma nódulos radiculares en alfalfa es incapaz de enlazar no leguminosas incluyendo tomate12. Las técnicas descritas en los pasos 1.1, 5.1, 6.3 y 7.1 se utilizan para determinar si la capacidad de hacer PGA generalmente aumentar Unión bacteriana a las superficies radiculares. Semillas de tomate eran superficie esterilizada y germinadas en agua estéril. Raíces se corta en segmentos de 1 cm de longitud y se colocaron en agua estéril y se inocularon las bacterias. Como estas dos especies de bacterias crecen a tasas diferentes, Unión se midió en diferentes momentos para permitir que cantidades aproximadamente iguales de crecimiento bacteriano. La presencia de la pMM11 del plásmido causó un aumento significativo similar en el número de bacterias dependientes de ambas especies (tabla 2)10. También se observó un aumento significativo en la Unión en el microscopio óptico, pero el enlace fue muy diferente para las dos especies (figura 3). A. tumefaciens A1045 obligado como bacterias individuales a la superficie de la raíz. S. meliloti atado en grandes grupos en los que sólo algunas de las bacterias fueron Unidos directamente a la raíz y la mayoría de las bacterias fueron unida a otras bacterias. Este ejemplo muestra que el simplemente analizando los números de destino sin incluir observaciones microscópicas de bacterias puede dar una impresión errónea de los resultados de un experimento. Aunque ambos métodos (recuento de células viables y observaciones microscópicas) muestran que pMM11 mayor atascamiento bacteriano a las raíces de tomate, el tipo de enlace por la producción de PGA fue diferente para las dos especies bacterianas10.
| El efecto de la pMM11 del plásmido en la Unión de bacterias a las raíces de tomate | ||
| Cepa bacteriana | Plásmido | Limitado número de bacterias por mm de longitud de raíz |
| A. tumefaciens A1045un | ninguno | 0.25 x 103 ± 0.25 x 103 |
| pBBR1mcs (vector) | 0.25 x 103 ± 0.25 x 103 | |
| pMM11 (síntesis de PGA) | 10 x 103 ± 0.25 x 103 | |
| S. meliloti 1021b | ninguno | ninguno detecta |
| pBBR1mcs (vector) | ninguno detecta | |
| pMM11 (síntesis de PGA) | 50 x 103 ± 5 x 103 | |
| un atascamiento bacteriano se midió después de 2 horas | ||
| b Unión bacteriana fue medida después de 18 horas | ||
| Esta tabla ha sido modificada de Matthysse, Deora, Mishra y Torres10. | ||
Tabla 2: el efecto de un plásmidos portadores de genes para la síntesis de PGA en Unión de A. tumefaciens A1045 y S. meliloti 1021 a segmentos de raíz de tomate. Para examinar la capacidad de poli-ß-1, 6 -N-acetil-D-glucosamina (PGA) para promover la Unión de las bacterias a las raíces, el efecto de un plásmido que confiere la habilidad de hacer PGA (pMM11) en la Unión de dos cepas de bacterias a la planta las raíces del tomate fue examinado. Ninguna cepa de bacterias demostró Unión significativa a las raíces de tomate en la ausencia del plásmido o ante la presencia del plásmido sin los genes que codifican la síntesis de la PGA (pBBR1mcs). La adición de los plásmidos portadores de genes de síntesis de PGA aumentó vinculante por ambos tipos de bacterias. Porque a. tumefaciens crece más rápido que el enlace de S. meliloti se midió después de 2 h de incubación para a. tumefaciens y 18 h de S. meliloti. Las técnicas utilizadas son las que se describen en el paso 7. Esta tabla ha sido modificada de Matthysse, Deora, Mishra y Torres10.
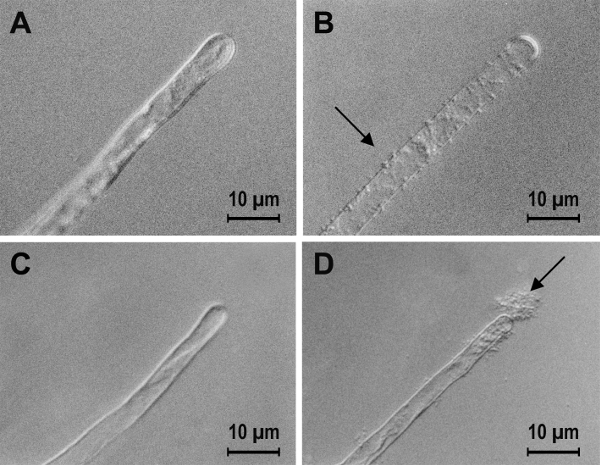
Figura 3 : El efecto de la pMM11 del plásmido lleva los genes de biosíntesis de la PGA en la Unión de a. tumefaciens A1045 S. meliloti 1021 a pelos de la raíz de tomate. Enlace a pelos de la raíz del tomate de la A) a. tumefaciens A1045, B) pMM11 de a. tumefaciens A1045, C) S. meliloti 1021 y D) pMM11 de S. meliloti 1021. Aunque el aumento en la Unión de las dos especies bacterianas es más o menos similar, los detalles del enlace como se ve en el microscopio óptico son muy diferentes. Las técnicas utilizadas son las que se describen en el paso 6. Esta figura ha sido modificada de Matthysse, Deora, Mishra y Torres10. Haga clic aquí para ver una versión más grande de esta figura.
A veces es posible utilizar Unión a superficies no-biológicos para ayudar a distinguir la contribución de las bacterias y de la planta en una interacción específica. La polysaccharide(UPP) unipolar de a. tumefaciens ha demostrado ser capaz de mediar la Unión bacteriana a una variedad de ambas superficies biológicas y no biológicas30. Calcio se observó que inhiben el atascamiento de a. tumefaciens a sembrar superficies mediadas por la UPP28. Con el fin de determinar si la inhibición por los iones del calcio de Unión bacteriana a las superficies de la planta es debido a un efecto sobre las bacterias o sobre la superficie de la planta, se examinó la Unión de las bacterias a hilos de nylon. Las técnicas se describe en el paso 8.2 fueron utilizados. Semillas de tomate eran superficie esterilizada y germinó en el agua como se describe en el paso 1. Las bacterias fueron cultivadas en medio mínimo con sacarosa y añadidas a las raíces de tomate o hilos de nylon a una concentración final de aproximadamente 105/ml como se describe en el paso 5.1. El efecto del agregado de CaCl2 en Unión bacteriana en raíces de tomate e hilos de nylon fue examinado en el microscopio. La figura 4 muestra una inhibición similar de unión de calcio utilizando cualquier superficie lo que sugiere que el efecto del calcio es principalmente en las bacterias10.

Figura 4 : El efecto del calcio sobre el atascamiento de a. tumefaciens a pelos de la raíz de tomate e hilos de nylon. A. tumefaciens se incubó con raíces de tomate (A y B) o hilos de nylon (C y D) durante 24 h en un 1:10 dilución de sales MS y una 1:20 dilución de medio mínimo AB, 0.4% de sacarosa (A y C) o en un 1:10 dilución de sales MS y 1:20 dilución de medio mínimo AB , 0.4% de sacarosa que contiene 60 mM CaCl2 (B y D)31. El agregado de CaCl2 inhibe Unión bacteriana a raíces e hilos de nylon, lo que sugiere que la inhibición era principalmente debido a un efecto en las bacterias y no en la superficie de la planta. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Es importante ser consciente de todas las superficies que las bacterias pueden adherirse durante el experimento. Así se pueden subestimar las bacterias que son capaces de unirse a cristal si se realiza un recuento viable usando tubos de vidrio y pipetas. Si las plantas se cultivan en agar o suelo y algunos del agar o suelo permanece en las plantas las bacterias pueden enlazar el material adherido en lugar de a las plantas. Por otro lado, lavar la superficie de la planta, particularmente en el caso de las raíces, puede quitar capas superficiales naturales tales como mucosa y alterar así los resultados de pruebas de adherencia.
Es importante estar seguro de que las bacterias se añade a la mezcla de incubación siguen vivas durante el experimento. Así rutinariamente se hagan recuentos de células viables de bacterias libres como adjuntadas. Algunas mutaciones bacterianas o tratamientos pueden reducir la tasa de crecimiento bacteriano o causan la muerte de una fracción de la población bacteriana. Bacterias vivas y muertas no sean distinguibles en el microscopio a menos que se utilizan tinciones especiales. Existe un kit de tinción útil para bacterias muertas o vivir que depende de la exclusión de los tintes de las bacterias vivas. Sin embargo, si una población mixta de especies bacterianas entonces recuento viable de la especie de interés es probablemente el método más fácil para determinar si la incubación ha resultado en la muerte bacteriana.
Composición medio influirá en crecimiento y supervivencia bacteriana. Exudado de raíz y materiales liberados de la herida y cortar los sitios proporcionará sustrato para crecimiento bacteriano modesto. Fosfato, nitrógeno y hierro tienden a ser limitante en estas condiciones. Cationes divalentes como calcio y magnesio pueden influir en la adherencia. En algunos casos fuente de carbono puede influir en la adherencia. pH es también importante. En general el pH de la rizosfera es entre 5.5 y 6.5.
Es necesario tener cuidado en el uso de bacterias con resistencia a los antibióticos. Los antibióticos más frecuentemente utilizados son la rifampicina y ácido nalidíxico. La resistencia a estos antibióticos es generalmente debido a mutaciones en genes cromosómicos (ARN polimerasa y la girasa, respectivamente) y por lo tanto no puede ser fácilmente transferido a otra cepa durante la incubación. Este tipo de resistencia no resulta en la degradación o modificación del antibiótico. Las bacterias de la marca con un marcador de genes portados por el plásmido no se recomienda a menos que el plásmido no puede transferirse a otras bacterias. La resistencia a los antibióticos no debe ser debido a la degradación o química modificación del antibiótico como antibióticos bacterias sensibles entonces serán capaces de crecer en placas de antibióticos si se ubican cerca de las bacterias resistentes.
Los métodos descritos en este documento son útiles para pequeños tamaños de muestra o experimentos donde las muestras deban ser contenida (por ejemplo, experimentos con patógenos humanos). Para tamaños de muestra grande (sobre 100 g de material o de más de 50 plantas) otros métodos o modificación drástica de estos métodos serían necesarios10,19,32,19,33, 34 , 35 , 36. la presencia de grandes cantidades de microorganismos distintos de las especies estudiadas también puede plantear problemas importantes. Posibles soluciones incluyen el uso de bacterias marcadas con una proteína fluorescente o resistencia a los antibióticos como se describe en los pasos 6.1.2 y 7.3. Sin embargo, cuando las bacterias de interés son raros individuos en una población grande de otros microorganismos estos marcadores no pueden ser suficientes para permitir una evaluación clara de los números de las bacterias estudiadas.
Todos los métodos descritos aquí son métodos de laboratorio en. Modificaciones menores se necesitarían estudios de invernadero. Más modificaciones mayores están probable que sean necesarios para estudios de campo donde protozoos, insectos y otros animal variación de depredación y el clima complican la provisión de condiciones definidas para los experimentos. En el futuro estos métodos pueden ampliarse para incluir las interacciones de dos o más microorganismos en la superficie de la planta.
Divulgaciones
El autor declara que ella no tiene competencia financieras intereses.
Agradecimientos
El autor agradece a Susan Whitfield para la preparación de las figuras y Camille Martin y Hillary Samagaio para asistencia con algunos de los experimentos.
Materiales
| Name | Company | Catalog Number | Comments |
| light microscope | any | N/A | phase contrast or Nomarski optics are helpful, for studies using fluorescent markers a fluorescence microscope is required. |
| seeds | any | N/A | make a note of the seed lot number and the cultivar |
| bleach | any | N/A | |
| bath sonicator | any scientific supply company | N/A | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| nutrient agar | Difco | 001-01-8 | |
| soytone | Difco | 24360 | |
| Sand | sea sand Fisher Scientific | S25 | |
| sand | Sigma-Aldrich | S9887 | |
| conetainers | Stuewe & Sons, Inc. | Ray-Leach cone-tainers | many different sizes are available to suit the type of plant you wish to grow |
| parafilm | any scientific supply company | N/A | |
| MS salts | Sigma-Aldrich | M5524 | |
| parrafin | any | N/A | |
| centrigfuge | any scientific company | N/A | to pellet most bacteri only a small centrifuge with a max force of 10,000 XG is needed |
| vortex mixer | any scientific company | ||
| micrometer | ACCU-SCOPE Accessories | A3145 | |
| Sedgwick-rafter counting cell | Hauser Scientific | HS3800 | |
| probe-clip press-seal incubatin chamber | Sigma-Aldrich | Z359483 | |
| rifampicin | Sigma-Aldrich | R3501 | |
| naladixic acid | Sigma-Aldrich | n8878 | |
| Live/dead stain | In Vitrogen Molecular Bioprobes | L7007 |
Referencias
- Heaton, J. C., Jones, K. Microbial contamination of fruit and vegetables and the behaviour of enteropathogens in the phyllosphere: a review. J. Appl. Microbiol. 104, 613-626 (2008).
- Beuchat, L. R. Ecological factors influencing survival and growth of human pathogens on raw fruit and vegetables. Microbes Infect. 4, 413-423 (2002).
- Berger, C. N., et al. Fresh fruit and vegetables as vehicles for the transmission of human pathogens. Environ. Microbiol. 12, 2385-2397 (2010).
- Lugtenberg, B. J., Chin, A. W. T., Bloemberg, G. V. Microbe-plant interactions: principles and mechanisms. Antonie Van Leeuwenhoek. 81, 373-383 (2002).
- Preston, G. M., Haubold, B., Rainey, P. B. Bacterial genomics and adaptation to life on plants: implications for the evolution of pathogenicity and symbiosis. Curr. Opin. Microbiol. 1, 589-597 (1998).
- Compant, S., Duffy, B., Nowak, J., Clement, C., Barka, E. A. Use of Plant Growth-Promoting Bacteria for Biocontrol of Plant Diseases: Principles, Mechanisms of Action, and Future Prospects. Appl. Environ. Microbiol. 71, 4951-4959 (2005).
- Mathews, S. L., Smith, R. B., Matthysse, A. G. A comparison of the retention of pathogenic Escherichia coli. O157 by sprouts, leaves and fruits. Microb. Biotechnol. 7, 570-579 (2014).
- Fuqua, C., Matthysse, A. G. Methods for studying bacterial biofilms associated with plants. Methods Enzymol. 337, 3-18 (2001).
- Torres, A. G., Jeter, C., Langley, W., Matthysse, A. G. Differential binding of Escherchia coli.O157:H7 to alfalfa, human epithelial cells, and plastic is mediated by a variety of surface structures. Appl. Environ. Microbiol. 71, 8008-8015 (2005).
- Matthysse, A. G., Deora, R., Mishra, M., Torres, A. G. Polysaccharides cellulose, poly-beta-1,6-n-acetyl-D-glucosamine, and colanic acid are required for optimal binding of Escherichia coli O157:H7 strains to alfalfa sprouts and K-12 strains to plastic but not for binding to epithelial cells. Appl. Environ. Microbiol. 74, 2384-2390 (2008).
- Matthysse, A. G., et al. The effect of cellulose overproduction on binding and biofilm formation on roots by Agrobacterium tumefaciens. Mol. Plant Microbe Interact. 18, 1002-1010 (2005).
- Matthysse, A. G., Kijne, J. W., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. Attachment of Rhizobiaceae to plant cells. The Rhizobiaceae. , 235-249 (1998).
- Matthysse, A. G. Conditioned medium promotes the attachment of Agrobacterium tumefaciens.strain NT1 to carrot cells. Protoplasma. 183, 131-136 (1994).
- Matthysse, A. G., McMahan, S. The effect of the Agrobacterium tumefaciens. attR mutation on attachment and root colonization differs between legumes and other dicots. Appl. Environ. Microbiol. 67, 1070-1075 (2001).
- Jeter, C., Matthysse, A. G. Characterization of the binding of diarrheagenic strains of E. coli.to plant surfaces and the role of curli in the interaction of the bacteria with alfalfa sprouts. Mol. Plant Microbe Interact. 18, 1235-1242 (2005).
- Matthysse, A. G., McMahan, S. Root colonization by Agrobacterium tumefaciens is reduced in cel, attB, attD, and attR mutants. Appl. Environ. Microbiol. 64, 2341-2345 (1998).
- Morton, E. R., Fuqua, C. Phenotypic analyses of Agrobacterium. Curr. Protoc. Microbiol. , (2012).
- Brandl, M. T. Plant lesions promote the rapid multiplication of Escherichia coli O157:H7 on postharvest lettuce. Appl. Environ. Microbiol. 74, 5285-5289 (2008).
- Franz, E., et al. Quantification of contamination of lettuce by GFP-expressing Escherichia coli O157:H7 and Salmonella enterica serovar Typhimurium. Food Microbiol. 24, 106-112 (2007).
- Morton, E. R., Fuqua, C. Laboratory maintenance of Agrobacterium. Curr. Protoc. Microbiol. , (2012).
- Lugtenberg, B. J., Kravchenko, L. V., Simons, M. Tomato seed and root exudate sugars: composition, utilization by Pseudomonas biocontrol strains and role in rhizosphere colonization. Environ. Microbiol. 1, 439-446 (1999).
- Murashige, T., Skoog, F. A Revised Medium for Rapid Growth and Bio Assays with Tobacco Tissue Cultures. Physiologia Plantarum. 15, 473-497 (1962).
- Danhorn, T., Fuqua, C. Biofilm formation by plant-associated bacteria. Annu. Rev. Microbiol. 61, 401-422 (2007).
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- O'Toole, G., Kaplan, H. B., Kolter, R. Biofilm formation as microbial development. Annu. Rev. Microbiol. 54, 49-79 (2000).
- Charkowski, A. O., Barak, J. D., Sarreal, C. Z., Mandrell, R. E. Differences in growth of Salmonella enterica and Escherichia coli O157:H7 on alfalfa sprouts. Appl. Environ. Microbiol. 68, 3114-3120 (2002).
- Loper, J., Suslow, T., Schroth, M. Lognormal distribution of bacterial populations in the rhizosphere. Phytopathology. 74, 1454-1460 (1984).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front Plant Sci. 5, 252 (2014).
- Douglas, C. J., Halperin, W., Nester, E. W. Agrobacterium tumefaciens mutants affected in attachment to plant cells. J. Bacteriol. 152, 1265-1275 (1982).
- Tomlinson, A. D., Fuqua, C. Mechanisms and regulation of polar surface attachment in Agrobacterium tumefaciens. Curr. Opin. Microbiol. 12, 708-714 (2009).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front Plant Sci. 5, (2014).
- Wright, K. M., et al. The endophytic lifestyle of Escherichia coli O157:H7: quantification and internal localization in roots. Phytopathology. 103, 333-340 (2013).
- Berger, C. N., et al. Fresh fruit and vegetables as vehicles for the transmission of human pathogens. Environ. Microbiol. 12, 2385-2397 (2010).
- Erickson, M. C., et al. Internalization and Fate of Escherichia coli O157:H7 in Leafy Green Phyllosphere Tissue Using Various Spray Conditions. J. Food Prot. 77, 713-721 (2014).
- McKellar, R. C., et al. Evaluation of different approaches for modeling Escherichia coli O157:H7 survival on field lettuce. Int. J. Food Microbiol. 184, 75-85 (2014).
- Wu, F. M., et al. Factors influencing the detection and enumeration of Escherichia coli O157:H7 on alfalfa seeds. Int. J. Food Microbiol. 71, 93-99 (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados