Method Article
Einhaltung von Bakterien auf Flächen, die im Labor ermittelten Pflanzen
In diesem Artikel
Zusammenfassung
Eine einfache Methode zur Messung und Charakterisierung von bakteriellen Adhäsion zu Pflanzen, besonders Wurzeln und Sprossen, wird in diesem Artikel beschrieben.
Zusammenfassung
Dieses Manuskript beschreibt eine Methode zur Messung der bakterielle Bindung an axenic pflanzlicher Oberflächen im Lichtmikroskop und durch den Einsatz von lebensfähigen Zellzahlen. Pflanzlichen Stoffen verwendet gehören Wurzeln, Sprossen, Blätter und Früchte schneiden. Die beschriebenen Methoden sind kostengünstig, leicht und eignet sich für kleine Probenmengen. Bindung wird im Labor gemessen und eine Vielzahl von Medien Inkubation und Bedingungen verwendet werden. Der Einfluss von Inhibitoren kann ermittelt werden. Situationen, die zu fördern und hemmen die Bindung können auch beurteilt werden. In einigen Fällen ist es möglich zu unterscheiden, ob verschiedene Bedingungen verbindlich in erster Linie aufgrund ihrer Wirkung auf die Pflanze oder die Bakterien verändern.
Einleitung
Die Messung der bakterielle Bindung an Oberflächen zu Pflanzen ist in drei unterschiedlichen Situationen sehr wichtig geworden. Die erste Situation ist die Prüfung der Übertragung von menschlichen Krankheitserreger am Werk Oberflächen1,2,3. Das Ziel hier ist bakterielle Bindung zu verhindern oder zu entfernen oder gebundene Bakterien abtöten und damit die Übertragung von Krankheiten durch Pflanzenmaterial zu reduzieren. Im zweiten Fall ist die Untersuchung der Bindung der Pflanzenpathogene Oberflächen4Pflanzen. Wieder einmal das Ziel hier ist verbindlich zu verhindern oder zu entfernen oder gebundene Bakterien abtöten und damit zur Krankheit zu reduzieren. Die dritte Lage ist die Untersuchung der Bindung von symbiotischen oder Pflanze-wachstumsfördernde Bakterien5,6. Das Ziel dabei ist die Förderung bakterielle verbindlich und somit zur Erhöhung der Pflanzengesundheit und Ernte ergibt.
Die Techniken zur Messung der bakterielle Bindung an Oberflächen, die in diesem Artikel beschriebenen Pflanzen sind preiswert und relativ einfach durchzuführen. Die einzigen Voraussetzungen sind ein Mikroskop und Materialien, die in der Regel in einem Bakteriologie Labor gefunden. Für einige Techniken eignet sich ein Bad sonikator. Die beschriebenen Techniken eignen sich für verbindliche Experimente mit relativ kleinen Stichprobengrößen durchgeführt. Messungen von Bindung sind im Labor durchgeführt, obwohl es vielleicht möglich, einige dieser Techniken für den Einsatz im Gewächshaus oder im Feld zu ändern.
Diese Techniken wurden verwendet, um bakterielle Bindung an Wurzeln, Sprossen, geschnittene Blätter, geschnittenen Früchten und intakte Cherry-Tomaten im Labor7,8,9,10,11Messen, 12,13,14,15. Sie wurden auch zur Wurzel Besiedlung der Pflanzen wachsen im Boden zu messen oder sand in den Labor-16. Die Techniken wurden mit vielen Bakterienarten Agrobacterium Tumefaciens, Sinorhizobium Meliloti, Escherichia coli, Salmonella Enterica und Pseudomonas Fluorescenseinschließlich verwendet. Eine nützliche Beschreibung der Methoden für die Beurteilung der Wechselwirkung von A. Tumefaciens mit Oberflächen finden Sie in Morton und Fuqua (2012)17. In allen Fällen wurden die beteiligten Stichprobengrößen klein, in der Regel weniger als 25-50 Pflanzen. Die beschriebenen Techniken sind geeignet für den Einsatz mit menschlichen Krankheitserreger, die während der Experimente enthaltenen gehalten werden müssen.
Protokoll
1. Vorbereitung des Axenic Pflanzenmaterial
- Bereiten Sie Setzlinge in Wasser gewachsen.
- Legen Sie eine kleine Anzahl von Samen (weniger als 30) in einem 30, 50, 100 oder 150 mL Becherglas. Wir haben Tomaten, Luzerne, Arabidopsis Thaliana, Erbsen, Bohnen, Tabak, Salat und karottensamen mit diesem Protokoll verwendet.
Hinweis: Um festzustellen, wie viele Samen ohne Verunreinigung aus einem Samenkorn zum anderen verbreiten zusammen sterilisiert werden können, siehe Schritt 1.1.2. - Bedecken Sie die Samen mit 80 % igem Ethanol und kurz schwenken. Lassen Sie die Samen für 1 min einweichen.
- Gießen Sie das Ethanol und bedecken Sie die Samen mit einer Lösung aus 50 % durch Volumen kommerzielle Bleichmittel (NaClO) und 0,1 % Triton x-100 im Leitungswasser. Lassen Sie die Samen für 20 Minuten einweichen.
Hinweis: Wenn die Samen groß, wie z. B. Bohnensamen sind, möglicherweise es verlängert die Einwirkzeit um die Pilze auf der Oberfläche der Samen vollständig zu töten. - Die Bleichmittel Mischung abgießen und die Samen 3 Mal mit sterilem Wasser zu waschen. Lassen Sie die Samen in Wasser für 1 min für jede Wäsche einweichen.
- Um zu erhalten fügen axenic Sämlinge eine kleine Menge steriles Wasser zwischen 5 und 25 mL je nach Größe der Samen. Gießen Sie die Samen und Wasser in eine sterile Petrischale für die Keimung der Samen.
Hinweis: Sollte das Wasser bedecken Sie den Boden der Schale aber nicht decken die Samen um die Bildung der Wurzelhaare zu fördern. Axenic beschreibt eine Kultur, in der nur eine einzige Art des Organismus vorhanden ist. - Inkubieren Sie im Dunkeln, bis die Sämlinge die gewünschte Größe zwischen 1 cm und 10 cm (ca. 5 Tage für Tomaten und A. Thaliana und 1 bis 3 Tage für Luzerne) erreichen. Verwenden Sie für größere Samen einen überdachten sterilen Behälter mit einem Fassungsvermögen von mehr als 100 mL wie eine Glasschale mit Folie abgedeckt.
- Legen Sie eine kleine Anzahl von Samen (weniger als 30) in einem 30, 50, 100 oder 150 mL Becherglas. Wir haben Tomaten, Luzerne, Arabidopsis Thaliana, Erbsen, Bohnen, Tabak, Salat und karottensamen mit diesem Protokoll verwendet.
- Bestimmen Sie die Frequenz der Kontamination von einem bestimmten Saatgutpartie mit Mikroorganismen, die auf oder innerhalb der Samen nach der Oberfläche-Sterilisation Lebensfähigkeit.
Hinweis: Dies ist notwendig, da gelegentliche Samen Mikroorganismen unter der Samenschale tragen, die durch Oberfläche-Sterilisation nicht getötet werden kann. Verwenden Sie die Häufigkeit der kontaminierten Samen in einer Saatgutpartie um zu bestimmen, wie viele Samen um zu sterilisieren auf einmal ohne ein hohes Risiko für die Gruppe der Samen immer kontaminiert durch einen Samen mit Bakterien oder Pilzen, die die Behandlung überleben.- Führen Sie Schritte 1.1.1 bis 1.1.3.
- Legen Sie die Samen auf ein Nähragar Petrischale. Habe ca. 10-30 Samen in jedem Gericht Abstand so dass sie einzeln bewertet werden können. Versiegeln Sie die Gerichte mit Klebeband oder Abdichtung Film.
- Ca. 3-5 Tage bei 25 ° C erzielte täglich sichtbaren Auswuchs von Mikroorganismen aus den Samen inkubieren.
- Bereiten Sie Setzlinge in Sand angebaut. Setzlinge können im Sand für den Einsatz in bakterielle Adhäsion Experimente.
- Führen Sie Schritte 1.1.1 bis 1.1.3.
- Sterilisieren Sie Quarz oder Meer Sand in den Autoklaven. Beide enthalten einige organisches Material. Wenn dies das Experiment auswirkt, waschen Sie den Sand durch abdecken mit zweimal sein Volumen von 0,1 M HCl. Mix für 10 min. erlauben den Sand zum absetzen und die Flüssigkeit abgießen. Spülen Sie den Sand 3 Mal mit Wasser und einmal mit 80 % Ethanol gefolgt von zwei zusätzlichen Wasser spült mit dem gleichen Protokoll wie bei 0,1 M HCl. entkeimen die gewaschenen Sand in den Autoklaven.
- Sterilisieren von Containern durch Untertauchen sie in 50 % Bleichmittel für 5 min und spülen Sie sie 5-Mal mit sterilem Wasser durch Untertauchen. Damit sie trocknen und Dichtung unten mit dichten Film. Der Film, wie vom Hersteller zu beziehen ist in der Regel frei von Mikroorganismen auf der Innenseite Seite, die mit Blick auf das Innere des Containers platziert werden soll.
- Mischen Sie die sterilen Sand mit genug sterilem Wasser bis es nass genug, dass die Sand-Sticks. Die Höhe hängt davon ab, wie trocken der Sand ist. 10-35 % des Volumens des Sandes ist in der Regel ausreichend. Legen Sie den nassen Sand in den Behälter so dass ausreichend Tiefe für die Länge der Wurzeln notwendig und ausreichend Raum über dem Sand für das Wachstum des Sprosses. Die genauen Abstände richten sich nach Art und Sorte der Pflanze verwendet wird.
- Pflanzensamen.
- Stellen Sie ein flaches Loch in den Sand mit einem sterilen Glasstab nur tief genug, um das Saatgut unter der Oberfläche des Sandes (ca. 1-5 mm) platzieren. Legen Sie die Samen in das Loch. Bedecken Sie es mit einer dünnen Schicht von Sand. Verschließen Sie den oberen Rand des Containers mit Abdichtung Film um Wasserverlust und Eingang der zusätzlichen Mikroben zu verhindern.
- Wachsen Sie die Pflanzen im Labor unter einem Licht oder im Gewächshaus bei geeigneter Temperatur und Tageslänge für Art und Sorte der Pflanze. Verwenden Sie für Tomaten, Luzerne oder Arabidopsis Thaliana Raumtemperatur und 12 h hell/dunkel-Zyklen.
- Pflanzen Sie die Setzlinge in ein Loch in den Sand (ca. 1-2 mm im Durchmesser und tief genug, dass die Wurzel mit Sand abgedeckt werden). Machen Sie das Loch mit einem sterilen Glasstab. Führen Sie die Wurzel vorsichtig in das Loch mit einer sterilen Edelstahl Häkelnadel wenn benötigt wird und die Seiten des Loches mit Sand zu füllen.
- Alternativ, wachsen Sie Pflanzen axenically auf oder im Agar enthält eine Mischung von Salzen wie z. B. MS Salze. Verwenden Sie Triebe der axenic Pflanzen in Agar direkt. Vermeiden Sie Wurzeln gewachsen mit Agar Agar klebt an der Pflanzenoberfläche und die Bakterien. Dadurch kann ein falscher Eindruck von bakterielle Adhäsion auf der Pflanzenoberfläche.
2. Vorbereitung des anderen pflanzlichen Material
- Waschen Sie Pflanzenmaterial im Gewächshaus mit Wasser vor dem Gebrauch die Reduzierung der indigenen Mikroben angebaut. Es werden Bakterien und Pilze auf die Pflanzen. Protozoen werden im Boden, an den Wurzeln, und möglicherweise auf den Blättern vorhanden sein. Waschen mit Bleichmittel oder Ethanol ändert der Pflanzenoberfläche und wird nicht empfohlen.
- Sollte nach Wasserreinigung bedeutende Zahlen von Mikroben bleiben, behandeln die Pflanzen mit verdünnten flüssigen Handseife oder Wasserstoffperoxid (0,01 %) zu entfernen oder zu töten Mikroben zu verdünnen. Dies ist in der Regel weniger schädlich für die Pflanze als Bleichmittel oder Ethanol.
- Als Pflanze ist auf einem lokalen Markt gekauften Materials schwierig zu dekontaminieren, wählen Material, das nicht angezeigt wird, zu lange Lagerung unterzogen wurden. Vermeiden Sie Teile des Materials, die braune oder beschädigt angezeigt.
- Waschen Sie ihn mit Wasser und frische Schnitte an den Enden des Materials die zuvor geschnitten worden sind, bevor Sie es verwenden, es sei denn, schneiden Sie die Wechselwirkungen der testbakterien mit Bakterien (und Eukaryoten) die auf angesammelt haben, werden Websites von Interesse sind. Waschen Sie nicht mit Bleichmittel oder Ethanol, wie sie der Pflanzenoberfläche verändern werden.
- Websites der Wunde zu behandeln. Wunde Seiten bieten oft Bakterien Zugang in das Innere der Pflanze Material18.
- Blockieren Sie oder verringern Sie bakterielle Bindung an und Bewegung durch Wunde oder geschnittenen Websites erstellt bei der Vorbereitung des Materials durch die Schnittkante in geschmolzenem Paraffin eintauchen oder durch die Website mit geschmolzenem Paraffin mit einem kleinen Pinsel malen. Verwenden Sie keinem Spatel oder irgendwelche scharfen umzusetzen, da dies das Gewebe beschädigen kann.
- Siegel stammen Narben auf Früchte wie Tomaten mit Paraffin. Einige Bakterien werden in den Bereich unter dem Paraffin Schwimmen, aber ihre Zahlen sind in der Regel klein.
- Wenn bakterielle Inkrafttreten Wunde Websites von Interesse, Schätzung, die Distanz, die Bakterien gelangen durch Wasserläufe oder Diffusion ausgeführt, wird durch die Beobachtung der Bewegung eines Farbstoffes der Projektmappe hinzugefügt. Markieren Sie bewegliche Bakterien durch die Einführung eines Gens Codierung ein fluoreszierendes Protein wie grün fluoreszierendes Protein (GFP) hinein und sie verfolgen mit Fluoreszenz-Mikroskopie 6.1.2 Schritt19beschrieben.
3. Vorbereitung der Bakterien
- Wachsen Sie die Bakterien. Verwenden Sie ein Medium, das am ehesten die Bedingungen der Bakterien approximiert werden voraussichtlich die ausgesetzt worden sind, unmittelbar vor der Begegnung mit dem Werk in der realen Welt außerhalb des Labors. Kohlenstoff und Stickstoff Quellen sowie die Anwesenheit von Ionen, insbesondere zweiwertigen Kationen (Ca, Mg, Fe, Mn und Zn) und Phosphat und der pH-Wert des Mediums sind wichtig.
- Verwenden Sie minimal AB Mittel "oder" Luria Brühe für A. Tumefaciens und andere Boden-Bakterien-20. Verwenden Sie für E. Coli, die im Darm entstehen können, Luria Brühe.
- Die mittlere21Induktoren z. B. Wurzelausscheidungen oder kommerzielle Pflanzenextrakte wie Soytone oder Zucker wie Saccharose oder Xylose hinzufügen. Wenn diese Stoffe als Induktoren verwendet werden, fügen Sie eine niedrige Konzentration, z. B. 0,01 %. Wenn sie gebrauchte Kohlenstoffquellen sind, fügen Sie eine höhere Konzentration, z. B. 0,1 %.
- Bereiten Sie das bakterielle Inokulum. Verdünnen Sie die Bakterien in sterilem Wasser oder das Medium, in dem die Inkubation wird nachgeholt, und fügen sie Sie das Pflanzenmaterial. Die entsprechende Verdünnung wird in Schritten 4.2 und 4.3 diskutiert.
- Um die Wachstumsmedien entfernen, bevor die Bakterien, die Bakteriensuspension bei 10.000 x g für 2 min zentrifugieren, überstand abgießen und Aufschwemmen der Bakterien durch Vortexen Sie in das gleiche Medium, welches für die Inkubation mit dem Pflanzenmaterial verwendet wird. Diese Methode kann zu entfernen oder verringern Sie die Anzahl oder Menge der extrazellulären Material und Anhängsel wie exopolysaccharide, Kapseln, Geißeln und/oder Pili. Wenn es wichtig ist, dass diese Oberflächenstrukturen intakt bleiben, dann einfach verdünnen Sie die Bakterien zu, bevor sie impfen oder verwenden Sie die alternative Methode, die im Schritt 3.2.2 beschrieben.
- Als eine alternative Methode zum Entfernen Wachstumsmedium, sammeln Sie die Bakterien auf einem Nitrozellulose oder Polycarbonat Filter mit einer Porengröße von 0,2 µm oder weniger. Waschen Sie die Bakterien mit dem Medium Inkubation und Aufschwemmen sie durch sanftes schütteln oder aufschütteln des Filters in einen sterilen Behälter des Mediums.
4. Impfung der Bakterien
- Bestimmen Sie die Anzahl der Bakterien unter Bezugnahme auf die Messung verwendet werden, um festzustellen, bakterielle Adhäsion geimpft werden und die Dauer der Inkubation.
- Mikroskopische Studien mit Inkubation mal weniger als 1 Tag sind relativ große Anzahl von Bakterien zu impfen. Fügen Sie eine Menge an Kultur, eine bakterielle Endkonzentration von mehr als 106 Bakterien pro mL zu erreichen. Für längere Inkubationszeiten verkleinern Sie bakterielle Inokulum.
- Für Studien, in denen bakterielle Adhäsion wird daran gemessen werden tragfähige Zellzahlen, fügen Sie eine Menge an Kultur, eine bakterielle Endkonzentration von 10 zu erreichen3 bis 106 Bakterien pro mL.
- Vermeiden Sie so viele Bakterien, dass ihr Stoffwechsel ändert sich die pH-Wert oder Sauerstoff-Konzentration im Inkubation Medium hinzufügen. PH-Wert mit pH-Papier oder einer Elektrode zu messen. Sauerstoff-Konzentration mit einer Sauerstoffelektrode zu messen.
- Für Pflanzen in Sand in drei mögliche Wege zu impfen.
- Impfen Sie die Samen mit den Bakterien vor der Pflanzung durch Einweichen der Samen für 1 min in einer Suspension in Wasser etwa 106 Bakterien pro mL.
- Die Wurzel von axenic Sämlinge Keimen, wie in Abschnitt 1 beschrieben, als Sämling Root ist etwa 1 cm lang eintauchen oder die ganze Sämling in eine Suspension von 106 Bakterien pro mL Wasser für 1 min zu impfen.
- Impfen Sie Sand, durch Mischen der Bakterien mit dem Sand vor dem Einpflanzen um eine Endkonzentration von etwa 103 Bakterien pro mL zu geben oder durch Bewässerung des Keimlings mit einer Suspension von 106 Bakterien pro mL nach der Pflanzung.
(5) Inkubation der Bakterien mit dem Pflanzenmaterial
- Für die Inkubation in flüssigen Medien, brüten die Bakterien mit dem Pflanzenmaterial in sterilem Wasser oder Saccharose und mineralische Salze oder pflanzlichen Gewebe Kulturmedium (z. B. eine 01:10 Verdünnung MS Salze)22,23.
- Verwenden Sie einen Behälter, auf dem sich die Bakterien nicht halten. Versuchen zu der Pflanzenoberfläche bedeckt kontinuierlich durch Eintauchen oder durch sanfte Bewegung. Kräftig schütteln kann verhindern Haftung oder sogar Bakterien aus der Pflanzenoberfläche entfernen.
- Beobachten Sie das Pflanzenmaterial in dem Lichtmikroskop nach unterschiedlichen Zeitintervallen zu bestimmen, wann man aufhören die Inkubation und Messungen machen. Ein zeitlichen Verlauf der Adhäsion ist oft wertvolle mit Proben, die jeweils 1 bis 4 h oder jeden Tag abhängig von der Geschwindigkeit der Interaktion.
- Um im Sand zu inkubieren, gelten Sie die gleichen Überlegungen zur Inkubation im flüssigen Medium im Schritt 5.1 beschrieben.
6. Messung der Adhäsion mit Mikroskopie
- Machen Sie mikroskopische Messungen mit Nomarski oder Phasenkontrast-Optiken für die einfache Anzeige der Bakterien auf Oberflächen. Jedoch kann jedes Hellfeld Mikroskop mit Vergrößerung von 20 X oder höher verwendet werden.
- Verwenden Sie mikroskopische Beobachtung, um festzustellen, ob die Bakterien sind nach dem Zufallsprinzip verteilt oder in bestimmte Websites. Auch überprüfen Sie, ob sie einzeln oder in Büscheln gebunden sind. Achten Sie auf das Vorhandensein von mikrokolonien was bakterielles Wachstum oder Einklemmung nach Haftung. Überprüfen Sie, ob die Bakterien scheinen einen Biofilm auf der Oberfläche bilden.
Hinweis: Ein Biofilm ist eine große Anzahl von Bakterien an der Oberfläche gebunden und umgeben von einer extrazellulären Matrix23,24,25. Die Struktur kann glatt und gleichmäßig oder haben eine kompliziertere Architektur. Methoden zur Untersuchung von Biofilms pflanzlicher Oberflächen zugeordnet wurden beschrieben17. - Verwenden Sie Bakterien, die mit einem fluoreszierenden Marker markiert. Wenn andere Bakterien vorhanden sind, und die Bakterien von Interesse werden durch einen fluoreszierenden Tag wie z. B. grün fluoreszierendes Protein identifiziert, verwenden Sie Fluoreszenz-Mikroskopie um zu bestimmen, das Vorhandensein von tagged Bakterien in Clustern mit andere Bakterien-26. Verwenden Sie für GLP einen Filter mit 490 nm Anregung und 520 nm Emission.
- Überprüfen Sie, dass das fluoreszierende Tag nicht die Bakterien beeinträchtigt ist durch die Beobachtung der Einhaltung von axenic Material eine gleiche Mischung von Wildtyp-Bakterien und tagged Bakterien von dem gleichen Stamm mit Nomarski und Fluoreszenz-Optik. Wenn die fluoreszierenden und dunklen Bakterien nach dem Zufallsprinzip gemischt und in gleicher Anzahl vorhanden sind der Tag mit dem Assay nicht stören.
- Verwenden Sie mikroskopische Beobachtung, um festzustellen, ob die Bakterien sind nach dem Zufallsprinzip verteilt oder in bestimmte Websites. Auch überprüfen Sie, ob sie einzeln oder in Büscheln gebunden sind. Achten Sie auf das Vorhandensein von mikrokolonien was bakterielles Wachstum oder Einklemmung nach Haftung. Überprüfen Sie, ob die Bakterien scheinen einen Biofilm auf der Oberfläche bilden.
- Bestimmen Sie die Anzahl der angeschlossenen Bakterien.
Hinweis: Es ist sehr schwierig, die Anzahl der angeschlossenen Bakterien im Mikroskop bestimmen. Wenn die Bindung zu unregelmäßigen pflanzlicher Oberflächen ist, ist es in der Regel nicht möglich, eine quantitative Messung zu erhalten. Rasterelektronenmikroskopie (nicht in diesem Artikel beschriebenen) kann verwendet werden, um solche Messungen zu machen.- Wenn die Bakterien zu einer glatten Oberfläche wie ein Wurzelhaare gebunden sind, die Anzahl der Bakterien an den Rand der Wurzelhaare pro mm Wurzelhaare Länge gebunden. Achten Sie darauf, um Wurzelhaare etwa die gleiche Größe und Form im Vergleich zu verwenden.
- Um die Größe von Objekten im Mikroskop zu bestimmen, verwenden Sie eine kommerzielle Folie mit gemessenen Markierungen drauf. Beobachten Sie und fotografieren Sie diese Folie auf die gleichen Einstellungen wie für das experimentelle Material verwendet und verwenden Sie die dabei entstandenen Bilder, um festzustellen, die Größe der Objekte in die Vergößerung.
- Bereiten Sie die Probe für die Mikroskopie.
- Waschen Sie die Probe. Bewegen Sie die Probe in einem Tropfen Wasser oder Inkubation Medium auf einen Objektträger zu und beobachten Sie es direkt.
Hinweis: Dies hat den Vorteil, dass gäbe es kein Bakterienwachstum oder bakterielle Tod es unwahrscheinlich viele freie Bakterien sein. Nehmen Sie das Fehlen von freien Bakterien als ein Warnzeichen, dass möglicherweise wurden bakterielle Tod oder bakterielle Bindung an den Container, in dem die Inkubation durchgeführt wurde. Die Wirkung des Waschens der Probenmaterials ist in Abbildung 1dargestellt. - Waschen Sie die Probe vorsichtig in Wasser oder Inkubation Medium indem man es in ein Fläschchen mit Flüssigkeit und invertieren das Fläschchen vorsichtig. Dann legen Sie die Probe auf die Objektträger in frische Flüssigkeit zur Beobachtung.
- Montieren Sie die Probe in Flüssigkeit mit einem gewöhnlichen Deckglas und Mikroskop-Objektträger.
- Verwenden Sie wenn die Probe dick ist und so eine Beule unter dem Deckglas machen würde, ein Press-apply Deckglas. Diese Abdeckung Slips haben einen Ring aus Kautschuk oder Kunststoff am Rand des Deckglases. Legen Sie die Flüssigkeit und die Probe in den Brunnen in das Deckglas und dann legen Sie die Folie an der Spitze und drücken Sie an, um das Deckglas auf die Folie zu versiegeln. Umkehren und zu untersuchen.
- Alternativ verwenden Sie eine Alge, die Rutsche und Deckglas in ähnlicher Weise zu zählen. Beachten Sie, dass Folien mit dieser Tiefe können nicht generell mit einem Objektiv von mehr als 20 X Vergrößerung untersucht werden.
- Waschen Sie die Probe. Bewegen Sie die Probe in einem Tropfen Wasser oder Inkubation Medium auf einen Objektträger zu und beobachten Sie es direkt.

Abbildung 1 : Schritte bei der Vorbereitung einer Probe zur Bestimmung der Anzahl der gebundenen Bakterien. A. Tumefaciens Bindung Tomate Wurzelhaare (A, B und C) und Nylonfäden (D, E und F). In Proben im Wasser montiert, ohne Waschen beides Bakterien (Schwarze Pfeile) gebunden und freie Bakterien (weisse Pfeile) zu sehen (A und D). Nach dem Waschen die gebundenen Bakterien bleiben aber die freie Bakterien sind nicht mehr vorhanden (B und E). Nach Anwendung von Ultraschall wurden die gebundenen Bakterien von der Probenoberfläche (C und F) entfernt. Bitte klicken Sie hier für eine größere Version dieser Figur.
- Wenn fluoreszierende Bakterien verwendet wurden, untersuchen Sie die Probe unter dem Mikroskop nach der Sand entfernt worden ist. Wurzeln der Pflanzen in Sand sind in der Regel nicht geeignet für Mikroskopie, als die Beobachtung der Bakterien die Sandpartikel stören.
- Entfernen Sie die Pflanze von einem Wachstum Container nach Schritt 7. Legen Sie die Wurzeln in ein Gefäß mit Wasser und mischen Sie vorsichtig, um den Sand zu entfernen, die auf den Boden des Behälters absetzen wird.
- Entfernen Sie die Pflanze aus dem Waschwasser und montieren Sie die Probe wie in Schritt 6.3.3.
7. Messung der Adhäsion mit lebensfähigen Zelle zählt
- Bestimmen Sie die Anzahl der lebensfähigen beigefügten Bakterien mit einem sonikator.
- Die ungebundenen Bakterien zu entfernen. Legen Sie die Probe in ein Fläschchen mit ausreichend Wasser, Puffer oder Inkubation waschmedium bedecken das Pflanzenmaterial und invertieren das Fläschchen einige Male.
- Gäbe es mehr als 103 freie Bakterien pro mL in der ersten Inkubation vorhanden, führen Sie sequentielle Waschungen um alle zu entfernen. Check-in einem Lichtmikroskop (siehe Punkt 6.3) auf das Vorhandensein von Bakterien frei bestimmen.
- Waschen, bis es eine erhebliche Verringerung der Zahl der freien Bakterien gibt, aber denken Sie daran, dass es ein Gleichgewicht zwischen gebundenen und ungebundenen Bakterien so die Anzahl der freien Bakterien nie auf Null sinken kann.
- Entfernen Sie die Probe aus dem Fläschchen der Waschflüssigkeit mit einer Zange oder einem Spachtel.
- Bestimmen Sie die Anzahl der gebundenen Bakterien in Inkubationen von Pflanzen in Flüssigkeit.
- Auszusetzen Sie die gewaschene Probe in einer Durchstechflasche und bedecken Sie es mit einem gemessenen Volumen von Wasser, Inkubation Medium oder waschen Puffer. Verwenden Sie genügend Flüssigkeit, um die Probe abzudecken. Legen Sie das Fläschchen in einem Bad sonikator und für eine min beschallen.
- Entfernen Sie die Probe und prüfen sie unter dem Lichtmikroskop zu bestimmen, ob die gebundenen Bakterien aus der Anlage entfernt wurden. Die Entfernung von Bakterien aus einer Stichprobe durch Ultraschallbehandlung ist in Abbildung 1dargestellt. Gibt es noch vorhandenen Bakterien gebunden weiter beschallen die Probe bis so eine Zeit, die keine gebundenen Bakterien auf der Oberfläche der Probe bleiben. Wenn die gebundenen Bakterien durch Ultraschallbehandlung entfernt werden können, fügen Sie 1 bis 10 mg/mL sterile Quarzsand und wiederholen Sie die Anwendung von Ultraschall und mikroskopische Untersuchung.
- Bestimmen, dass die Beschallung-Verfahren, das am effektivsten im Schritt 7.1.3.2 erscheint die Aussetzung von Bakterien aus einer Flüssigkultur in der Lösung für Beschallung verwendet werden nicht die Lebensfähigkeit der Bakterien verringert (Quarzsand, fügen, wenn es nötig war). Bestimmen Sie die Zellenanzahl der lebensfähigen. Beschallen Sie die Bakterien und bestimmen Sie die Zellenanzahl der lebensfähigen wieder.
Hinweis: Ist ein Rückgang der Anzahl der lebensfähigen Zellen, ändern Sie das Verfahren durch eine Änderung der Zusammensetzung der Flüssigkeit und/oder Beschallung Zeit, bis die Behandlung keinen Einfluss auf die Anzahl der lebensfähigen Zellen hat. - Bestimmen Sie, dass die Verdünnungspuffer für tragfähige zellenzahlen verwendet die Lebensfähigkeit der Bakterien nicht beeinflusst, die unter den Bedingungen verwendet, durch den Vergleich von lebensfähigen Zellzahlen unter Verwendung unterschiedlicher Verdünnung Puffer und/oder Wasser inkubiert worden.
- Die ungebundenen Bakterien zu entfernen. Legen Sie die Probe in ein Fläschchen mit ausreichend Wasser, Puffer oder Inkubation waschmedium bedecken das Pflanzenmaterial und invertieren das Fläschchen einige Male.
- Bestimmen Sie die Anzahl der lebensfähigen beigefügten Bakterien mit Homogenisierung.
- Legen Sie die Probe in einem sterilen Mörser oder Mixer oder ein anderes Gerät Homogenisierung. Bedecken Sie es mit einem gemessenen Volumen von sterilem Wasser, Inkubation Mittel oder waschen Puffer27. Verwenden Sie ein Volumen die Probe zu decken. Verwenden Sie für einen Mixer 100 mL.
- Schleifen Sie die Probe, bis es gut homogenisiert wird. Überprüfen Sie den pH-Wert nach Homogenisierung sicher sein, dass Säure befreit das Pflanzengewebe einen drastischen Einbruch pH-Wert unter 7 nicht verursacht hat. Wenn der pH-Wert gesunken verwenden Sie einen Puffer wie Phosphatpuffer in der Homogenisierung Flüssigkeit um den pH-Wert beizubehalten.
- Bestimmen Sie die Zellenanzahl der lebensfähigen.
- Verwenden Sie Bakterien mit Antibiotika-Resistenz gekennzeichnet.
- Markieren Sie in Situationen, in denen mehr als eine Art von Bakterium vorhanden ist Bakterienstämme mit spontanen Widerstand gegen Antibiotika (in der Regel Rifampicin und Nalidixic Säure).
- Bestimmen Sie das Antibiotikum zu verwenden. Wenn Kulturen anderer Organismen, die voraussichtlich in die Inkubation vorkommen verfügbar sind, Platte dieser Kulturen angebaut unter den Bedingungen der beabsichtigten Inkubation mit Pflanzen auf Platten, die eine Reihe von Konzentrationen der gewählten Antibiotika. Bestimmen Sie die niedrigste Konzentration, die Wachstum aller dieser Organismen nicht zulässt. Dies ist die niedrigste Konzentration des Antibiotikums, die testbakterien resistent sein müssen.
- Erhalten Sie spontane Antibiotika-resistenten Mutanten. Wachsen Sie eine Kultur des Bakteriums in reiche Mittel zu spät Log oder stationären Phase. Platte 0,1 mL unverdünnt auf einen Teller, die das gewünschte Antibiotikum bei einer entsprechenden Konzentration enthalten. Bestimmen Sie die Konzentrationen zu verwenden, wie in Schritt 7.3.1.1 beschrieben. Zu halten und die Bakterien, die wachsen und somit resistent gegen das Antibiotikum zu reinigen.
- Bestimmen Sie, dass Antibiotika-Resistenz nicht das Wachstum der Bakterien reduziert auf diese Weise eine Wachstumskurve im flüssigen Medium der übergeordneten und Antibiotika-resistente Stämme. Bestimmen Sie, dass die Antibiotika-resistente Bakterien das gleiche Maß an Besiedlung des axenic Pflanzenmaterial als Elternstamm, zeigen wenn dies möglich ist.
- Markieren Sie in Situationen, in denen mehr als eine Art von Bakterium vorhanden ist Bakterienstämme mit spontanen Widerstand gegen Antibiotika (in der Regel Rifampicin und Nalidixic Säure).
- Bestimmen Sie die Anzahl der lebensfähigen Bakterien an Pflanzen in Sand gebunden.
- Entfernen Sie die Pflanze aus dem Container und Sand. Um Pflanzen aus Behältern zu entfernen, entfernen Sie zunächst Dichtungsmaterial auf der Ober- und Unterseite des Behälters. Stellen Sie den Behälter über einen Zettel steril und entfernen Sie vorsichtig den gesamten Sand oder Boden und Pflanze als eine große konische Zylinder durch sanft klopfen die Container gegen eine Fläche um das Material zu lösen.
- Verwenden Sie ggf. einen Spatel oder Stab, um das Material an den Rändern durchlaufen den Boden des Behälters zu lösen.
- Wenn der Zylinder von Sand oder Erde, enthält das Werk frei auf dem Papier ist, teilen Sie es in der Mitte, die Pflanzenwurzel zu offenbaren. Falls gewünscht, nehmen Sie Proben des Sandes von nahe dem Rand des Containers sowie in der Nähe der Wurzel. Dies kann nützlich zur Bestimmung der Ausbreitung (und Ansammlung und Wachstum) der Bakterien sein.
- Messen Sie die Länge der Wurzel. Abholen der Wurzelverzeichnis und entfernen Sie den Sand und Bakterien locker an die Wurzel (die Rhizosphäre Material) durch Eintauchen der Wurzel in einem gemessenen Volumen von Wasser oder Puffer und vorsichtig schütteln. Bestimmen Sie die tragfähige Zellzahl der Bakterien in die entstandene Suspension durch Ausplattieren auf einem geeigneten Datenträger wie Luria Agar. Dies entspricht der Anzahl der Bakterien, die locker mit der Wurzel verbunden.
- Entfernen Sie die Pflanze aus dem Container und Sand. Um Pflanzen aus Behältern zu entfernen, entfernen Sie zunächst Dichtungsmaterial auf der Ober- und Unterseite des Behälters. Stellen Sie den Behälter über einen Zettel steril und entfernen Sie vorsichtig den gesamten Sand oder Boden und Pflanze als eine große konische Zylinder durch sanft klopfen die Container gegen eine Fläche um das Material zu lösen.
- Entfernen Sie die eng gebundenen Bakterien durch Ultraschallbehandlung und bestimmen Sie ihre Zahl zu, wie unter Punkt 7.1 beschrieben.
- Alternativ, um zu bestimmen die Lage an der Wurzel des eng gebundenen Bakterien platzieren die gewaschene Wurzel auf der Oberfläche einer Petrischale mit Nähragar oder einem anderen geeigneten Medium. Beobachten Sie den Standort von Bakterienkolonien auf die Wurzel in den nächsten 3 Tagen mit einem sezierenden Mikroskop oder Lupe.
- Express-Ergebnisse als Anzahl der Bakterien pro Pflanze, pro cm2 Fläche, pro cm Wurzellänge oder pro Gramm Frischgewicht des Gewebes. Mehrere Wiederholungen am selben Tag zu tun und auch Wiederholungen an verschiedenen Tagen mit verschiedenen Bakterienkulturen und verschiedenen viel Pflanzenmaterial.
(8) festzustellen, ob ein Effekt der Inkubationsbedingungen auf Haftung wegen einer Antwort die Bakterien oder die Anlage
- Verwenden Sie tot oder Toten Pflanzenmaterial.
- Unterziehen Sie die Pflanze Material für eine Vielzahl von Chemikalien, Fixiermittel oder andere Behandlungen wie Hitze um es zu töten. Waschen Sie das Pflanzenmaterial in Wasser und Inkubation Medium nach der Verwendung dieser Behandlungen. Dann die Bakterien zu impfen. Dies wird nicht der Pflanzenoberfläche zerstören, aber das würde es metabolisch inaktiv machen, so dass es nicht auf die Bakterien reagieren kann.
- Bakterielle Adhäsion zu messen, wie in den Schritten 6 und 7 beschrieben. Außerdem bestimmen Sie die Anzahl der lebensfähigen Zellen hinzugefügt, um die Inkubation mit Pflanzenmaterial zu Beginn der Inkubation und die Anzahl der lebensfähigen Zellen am Ende der Inkubation um sicherzustellen, dass keine giftigen Chemikalien anwesend während der Inkubation waren vorhanden.
- Verwenden Sie lebloses Material.
- Verwendung bakteriellen Einhaltung lebloses Material zu ermitteln, ob ein Effekt gesehen auf bakterielle festhalten, Material zu Pflanzen ist aufgrund der Auswirkungen auf die Anlage oder die Bakterien. Wählen Sie ein leblose Material an die Bakterien binden und ähnelt in Form und Größe auf das Pflanzenmaterial studierte. Die Möglichkeiten umfassen Filterpapiere aller Arten (Zellulose, Nitrozellulose, Glasfaser, Polycarbonat, etc.), Threads (Nylon, Baumwolle, Polyester, Glaswolle, etc.), Glas oder Kunststoff Deckgläsern, Edelstahl-Gutscheine und Dialyse Membranen.
- Waschen Sie die leblose Material gründlich mit Wasser und das Medium, in dem die Inkubation durchgeführt werden und vor Gebrauch sterilisieren. Die meisten in Schritt 8.2.1 aufgeführten Materialien sind stabil, Sterilisation durch Autoklavieren.
- Inkubieren Sie die leblose Material unter den gewünschten Bedingungen und Gäste wie in den Schritten 6 und 7 beschrieben. Ein Beispiel für die Verwendung von Nylonfäden zu bestimmen, dass die reduzierte Bindung von A. Tumefaciens an Wurzelhaare bei hohen Kalzium-Konzentrationen zurückzuführen ist (zumindest teilweise) zu einer Wirkung von Kalzium auf die Bakterien ist in Abbildung 4-28dargestellt.
Ergebnisse
A. Tumefaciens kolonisiert Wurzeloberflächen. Um festzustellen, ob bakterielle Produktion von Zellulose eine Rolle bei der Kolonisierung der Wurzel spielt, die Auswirkungen der bakteriellen Mutationen, die Zellulose-Synthese zu verhindern wurden geprüft,16. In den Schritten 1.3 und 7.1 beschriebenen Techniken wurden verwendet. Tomaten-Samen wurden Oberfläche sterilisiert und in sterilem Wasser keimten. Wenn die Wurzeln ca. 2 cm lang waren waren sie eingetaucht in eine Suspension von 105 Bakterien pro mL und in pasteurisierter Boden in Container gepflanzt. Die Pflanzen wurden gezüchtet für 14 Tage bei 25 ° C auf einem 12 h Licht/12 h-Dunkel-Zyklus. Nach der angegebenen Zeit die Pflanzen wurden aus den Behältern entfernt. Die Wurzeln wurden gewaschen und beschallt in einem Bad sonikator gebundene Bakterien zu entfernen. Bakterielle Zahlen wurden mit lebensfähigen Zellen bestimmt. Abbildung 2 zeigt die Wirkung von zwei verschiedenen Zellulose-Minus-Mutationen auf die Fähigkeit der Bakterien, Tomate Wurzeln zu kolonisieren. Obwohl die Standardabweichungen der einige Messungen waren so hoch wie 0,910 (ein häufiges Problem bei dieser Art der Messung) melden Sie sich der Rückgang der Bindung der Zellulose-Minus Mutanten ist klar ersichtlich und wir können feststellen, dass die bakterielle Produktion von Zellulose hilft die Bakterien bei der Besiedlung der Tomate Wurzeln.
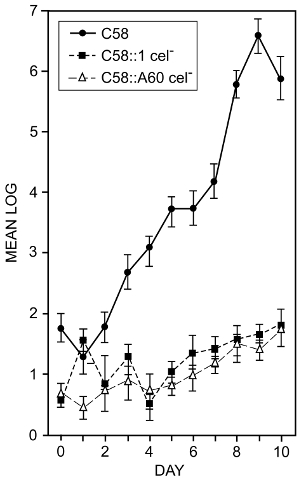
Abbildung 2 : Root Besiedlung durch Wildtyp und Mutanten Zellulose-Minus von A. Tumefaciens. Log10 Gesamtzahl der Bakterien pro cm Wurzellänge erholte sich von Tomaten Wurzeln mit Wildtyp-Stamm A. Tumefaciens C58 und Zellulose-Minus Mutanten, C58:1 und C58:A60 geimpft. Die Nummern sind die Mittel von einem Minimum von vier separate Experimente. Balken zeigen Standardabweichungen vom Mittel. Wurzeln wurden durch Eintauchen in eine Suspension von 105 Bakterien pro mL für 1 min geimpft. Die Pflanzen waren in Containern angebaut und die lose anhaftenden Bakterien wurden durch die Wurzeln in Waschpuffer in eine Glasflasche Waschen entfernt. Fest anhaftende Bakterien wurden entfernt, mit einem Bad sonikator und die entstandene Suspension vergoldet um festzustellen, dass lebensfähige Zelle16zählt. Diese Zahl wurde von Matthysse und McMahan geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
Die Rolle der exopolysaccharide in der Bindung von E. Coli und andere Bakterien, Alfalfasprossen wurde untersucht. Einige Ausbrüche von Durchfallerkrankungen durch E. Coli O157: H7 haben auf kontaminierte Alfalfasprossen zurückzuführen. Bindung von Wildtyp Bakterien und Mutanten, die nicht in der Lage zu verschiedenen exopolysaccharide wurde mittels in Schritte 1.1, 5.1 und 7.2 beschriebenen Methoden gemessen. Alfalfasprossen waren Oberfläche sterilisiert und für einen Tag in sterilem Wasser bei 25 ° C im Dunkeln gekeimt. Vier Sprossen mit angehängten samenhüllen wurden in sterilen Plastikschalen mit 5 mL Wasser platziert. Bakterien in Luria Brühe angebaut wurden, eine Endkonzentration von etwa 5 x 103 pro mL hinzugefügt. Die beimpften Sprossen waren für 3 Tage bei 25 ° C im Dunkeln inkubiert. Die Sprossen wurden zweimal in 5 mL sterilem Wasser in einer Phiole durch kräftige Umkehrung gewaschen und in Waschpuffer mit einer motorbetriebenen Teflon Glas Homogenisator homogenisiert. Bisherigen Experimente mit Hilfe von Bakterien markiert mit einem Plasmid trägt das GFP-gen zeigten keine internalisierten Bakterien zwar Oberfläche Bakterien leicht beobachtet wurden. Die Ergebnisse sind in Tabelle 1dargestellt. Zwei Stämme von E. Coli O157: H7 wurden untersucht. Beide Stämme erschien die Produktion von Poly-β-1,6-glucuron-acid(PGA) zu den größten Beitrag zur Bindung von pathogenen E. Coli , Oberflächen zu Pflanzen. Kolon Säure spielte auch eine bedeutende Rolle in Bindung. Während der Rückgang der Bindung in Zellulose-Minus Mutanten erhebliche war es nicht so groß wie für die anderen zwei Polysaccharide.
| Auswirkungen von Mutationen in den Genen Exopolysaccharide Produktion auf die Bindung von E. coli O157: H7, Sprossen und offene samenhüllen | |||
| Bakterienstamm | Mutation oder Genotyp (entsprechenden Phänotyp) | Log10 Anzahl der Bakterien pro Spross oder Samenschale gebunden | |
| Alfalfa Sprossenb | Offenen samenhüllen | ||
| 86-24 | Keiner (Wildtyp) | 4,7 ± 0,6 | 5,6 ± 0,2 |
| 8624N | yhjN (Zellulose-Minus) | 2.9 ± 0,7c | 3,5 ± 0,6c |
| 8624C | wcaD (Kolon Säure-Minus) | 1,8 ± 0,7c | 2.4 ± 0,5c |
| 8624P | pgaC (PGA-Minus) | < 1.0c | 1,0 ± 1.0c |
| DEC4A | Keiner (Wildtyp) | 5,6 ± 0,2 | 6.1 ± 0,3 |
| DEC4AN | yhjN (Zellulose-Minus) | 4,8 ± 0,8d | 4.1 ± 0,8d |
| DEC4AC | wcaD (Kolon Säure-Minus) | 3.9 ± 0,5c | 4,8 ± 0,8d |
| DEC4AP | pgaC (PGA-Minus) | < 1.0c | 1.2 ± 0,7c |
| ein Mittelwert ± Standardabweichung von mindestens drei Messungen der Anzahl (Log10) der Bakterien nach 3 Tagen gebunden. | |||
| b-Sprossen wurden vor der Messung gewaschen. | |||
| Deutlich anders als der Wildtyp c: P < 0,01. | |||
| d signifikant verschieden von dem Wildtyp: P < 0,05. | |||
| Diese Tabelle wurde von Matthysse, Deora, Mishra und Torres10geändert. | |||
Tabelle 1: Auswirkungen von Mutationen in den Genen Exopolysaccharide Produktion auf die Bindung von E. Coli O157: H7, Sprossen. Um festzustellen, die Rolle der verschiedenen exopolysaccharide und Lipopolysaccharid in der Bindung von pathogenen E. Coli O157: H7-Stämme, Alfalfasprossen, die Bindung einer Gruppe von Mutanten, die Sprossen und offene samenhüllen wurde untersucht, mit den Methoden in Schritt 6 beschrieben. Die Ergebnisse zeigten, dass Poly-ß-1, 6 -N-Acetyl-D-Glucosamin (PGA) scheint wesentlich für die Bindung an Sprossen und Zellulose und Colanic Säure sind für maximale Bindung von E. Coli O157 erforderlich. Diese Tabelle wurde von Matthysse, Deora, Mishra und Torres10geändert.
Um festzustellen, ob die Produktion von PGA ausreichend, um bakterielle Bindung an Oberflächen, ein Plasmid (pMM11) tragen die geklonten Operon Codierung die Gene für PGA erforderlichen Pflanzen verursachen wurde Produktion in zwei verschiedenen Bakterien eingeführt, die nicht werden Sie normalerweise Tomaten Wurzeln10binden. A. Tumefaciens A1045 ist eine Mutante der Wildtyp-Stamm C58 nicht zyklische-β-1,2-Glucan machen und auch nicht an Oberflächen29Pflanzen binden. Sinorhizobium Meliloti 1021 bildet WURZELKNÖLLCHEN auf Luzerne scheitert an nicht-Leguminosen binden darunter Tomaten12. In Schritte 1.1, 6.3, 5.1 und 7.1 beschriebenen Techniken wurden verwendet, um festzustellen, ob die Fähigkeit zur PGA in der Regel bakterielle Bindung an Wurzeloberflächen erhöht. Tomaten-Samen wurden Oberfläche sterilisiert und in sterilem Wasser keimten. Wurzeln in Segmente 1 cm Länge geschnitten und in sterilem Wasser gelegt wurden und die Bakterien geimpft wurden. Da diese beiden Arten von Bakterien unterschiedlich schnell wachsen, wurde Bindung zu verschiedenen Zeiten, um etwa gleiche Mengen von Bakterienwachstum ermöglichen gemessen. Das Vorhandensein von Plasmid pMM11 verursacht eine ähnliche deutliche Erhöhung der Anzahl der gebundenen Bakterien von beiden Arten (Tabelle 2)10. Eine deutliche Zunahme der Bindung galt auch in dem Lichtmikroskop die Bindung war jedoch ganz anders für die beiden Arten (Abbildung 3). A. Tumefaciens A1045 als einzelne Bakterien an der wurzeloberflächegebunden. S. Meliloti gebunden in großen Clustern, in denen nur ein paar der Bakterien wurden direkt an der Wurzel und die Mehrheit der Bakterien auf andere Bakterien befestigt waren. Dieses Beispiel zeigt, dass einfach die Zahl der Bakterien gebunden ohne einschließlich mikroskopische Beobachtungen analysiert einen irreführenden Eindruck der Ergebnisse eines Experiments geben kann. Obwohl beide Methoden (tragfähige Zellzahlen und mikroskopische Beobachtungen) zeigen, dass pMM11 bakterielle Bindung an Tomaten Wurzeln erhöht, war die Art der Bindung verursacht bei der Herstellung von PGA für zwei Bakterienarten10unterschiedlich.
| Die Wirkung von Plasmid pMM11 auf die Bindung von Bakterien zu Tomaten Wurzeln | ||
| Bakterienstamm | Plasmid | Anzahl der Bakterien pro mm Wurzellänge gebunden |
| A. Tumefaciens A1045ein | keine | 0,25 x 103 ± 0,25 x 103 |
| pBBR1mcs (Vektor) | 0,25 x 103 ± 0,25 x 103 | |
| pMM11 (PGA-Synthese) | 10 x 103 ± 0,25 x 103 | |
| S. Meliloti 1021b | keine | keine erkannt |
| pBBR1mcs (Vektor) | keine erkannt | |
| pMM11 (PGA-Synthese) | 50 x 103 ± 5 x 103 | |
| eine bakterielle Bindung wurde nach 2 Stunden gemessen. | ||
| b bakterielle Bindung war Maßnahme nach 18 Stunden | ||
| Diese Tabelle wurde von Matthysse, Deora, Mishra und Torres10geändert. | ||
Tabelle 2: die Wirkung von ein Plasmid mit Genen für die Synthese von PGA auf Bindung der A. Tumefaciens A1045 und S. Meliloti 1021 Tomate Wurzel Segmenten. Um zu prüfen, die Fähigkeit der Poly-ß-1, 6 -N-Acetyl-D-Glucosamin (PGA) zur Förderung der Bindung von Bakterien, Pflanzen Wurzeln, die Wirkung von ein Plasmid verleiht die Fähigkeit, PGA (pMM11) auf die Bindung von zwei Bakterienstämme Anlage verbunden sind, Tomaten-Wurzeln wurde untersucht. Weder Bakterienstamm zeigten signifikante Bindung an Tomaten Wurzeln in Abwesenheit des Plasmids oder im Beisein der Plasmid ohne den Genen Codierung PGA-Synthese (pBBR1mcs). Die Zugabe des Plasmids mit PGA Synthese Genen erhöhte Bindung von beiden Arten von Bakterien. Da A. Tumefaciens schneller wächst als S. Meliloti Bindung nach 2 h Inkubation für A. Tumefaciens und nach 18 h für S. Melilotigemessen wurde. Die verwendeten Techniken sind in Schritt 7 beschrieben. Diese Tabelle wurde von Matthysse, Deora, Mishra und Torres10geändert.
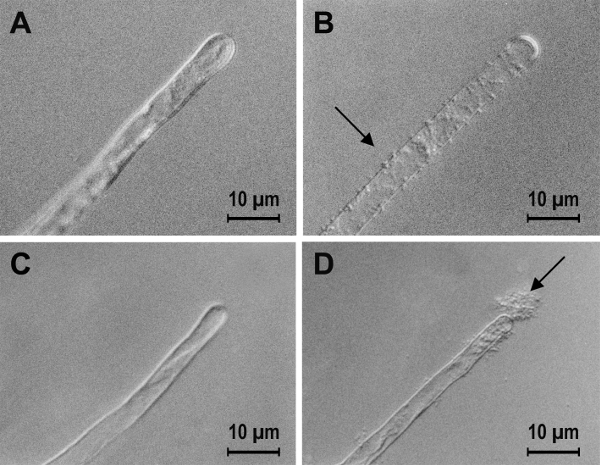
Abbildung 3 : Die Wirkung von Plasmid pMM11 tragen die PGA-Biosynthese-Gene auf die Bindung von A. Tumefaciens A1045 und S. Meliloti 1021, Tomate Wurzelhaare. A. Tumefaciens A1045 Bindung an Tomaten Wurzelhaare von A), B) A. Tumefaciens A1045 pMM11, C) S. Meliloti 1021 und D) S. Meliloti 1021 pMM11. Obwohl die Zunahme der Bindung von zwei Bakterienarten ungefähr ähnlich ist sind Details der Bindung wie in dem Lichtmikroskop gesehen ganz anders. Die verwendeten Techniken sind in Schritt 6 beschrieben. Diese Zahl wurde von Matthysse, Deora, Mishra und Torres10geändert. Bitte klicken Sie hier für eine größere Version dieser Figur.
Es ist manchmal möglich, Bindung an nicht-biologischen Oberflächen verwenden, um Hilfe bei der Unterscheidung des Beitrags der Bakterien und des Werkes in eine spezifische Interaktion. Die unipolaren polysaccharide(UPP) von A. Tumefaciens hat sich gezeigt, um bakterielle Bindung an eine Vielzahl von beiden biologischen und nicht-biologischen Oberflächen30vermitteln zu können. Calcium wurde beobachtet, um die Bindung von A. Tumefaciens , Oberflächen, vermittelt durch die UPP28Pflanzen hemmen. Die Bindung der Bakterien an Nylonfäden wurde untersucht, um festzustellen, ob die Hemmung von Calcium-Ionen der bakterielle Bindung zu Flächen Pflanzen durch Wirkung auf Bakterien oder auf der Pflanzenoberfläche ist. Beschreibt die Techniken im Schritt 8.2 dienten. Tomaten-Samen wurden Oberfläche sterilisiert und gekeimt im Wasser wie in Schritt 1 beschrieben. Die Bakterien wurden in minimaler Medium mit Saccharose angebaut und zur Tomate Wurzeln oder Nylonfäden an eine Endkonzentration von etwa 105/mL hinzugefügt, wie im Schritt 5.1 beschrieben. Die Wirkung der zusätzlichen CaCl2 auf bakterielle Bindung an Tomaten-Wurzeln und Nylonfäden war im Mikroskop untersucht. Abbildung 4 zeigt eine ähnliche Hemmung der Bindung von Kalzium mit entweder Oberfläche vorschlagen, die die Wirkung des Kalziums in erster Linie auf die Bakterien10ist.

Abbildung 4 : Die Wirkung von Kalzium auf die Bindung von A. Tumefaciens an Tomaten Wurzelhaare und Nylonfäden. A. Tumefaciens wurde mit Tomate Wurzeln (A und B) oder Nylonfäden (C und D) für 24 h inkubiert, in einem 01:10 Verdünnung von MS Salzen und ein 01:20 Verdünnung AB minimaler Medium, 0,4 % Saccharose (A und C) oder in einem 01:10 Verdünnung von MS Salzen und 01:20 Verdünnung von minimaler Medium AB , 0,4 % Saccharose mit 60 mM CaCl2 (B und D)31. Die zusätzlichen CaCl2 gehemmt bakterielle Bindung zu den Wurzeln und Nylonfäden, die darauf hindeutet, dass die Hemmung in erster Linie durch einen Effekt auf die Bakterien nicht auf der Pflanzenoberfläche war. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Es ist wichtig zu wissen für alle Oberflächen, die Bakterien während des Experiments eingehalten werden können. So können Bakterien, die in der Lage, Bindung an Glas unterschätzt werden, wenn tragfähige zellenzahlen erfolgen mittels Glasröhren und Pipetten. Wenn Pflanzen im Agar oder Boden und einige der Agar wachsen oder Boden auf den Anlagen bleibt können die Bakterien auf das anhaftende Material nicht auf die Pflanzen binden. Auf der anderen Seite Waschen der Pflanzenoberfläche, insbesondere im Falle von Wurzeln, kann entfernen Sie natürliche Oberflächenbeschichtungen wie Schleim und verändern damit die Ergebnisse der Prüfungen der Einhaltung.
Es ist wichtig, sicher sein, dass die Bakterien, die Inkubation Mischung hinzugefügt während des Experiments am Leben zu bleiben. Somit sollten praktikable zellenzahlen von freien sowie beigefügte Bakterien routinemäßig erfolgen. Einige Behandlungen oder bakterielle Mutationen können bakterielles Wachstumsrate reduzieren oder tatsächlich dazu führen, den Tod eines Bruchteils der Bakterienpopulation. Lebende und tote Bakterien möglicherweise nicht im Mikroskop zu unterscheiden, es sei denn, Spezialfärbungen eingesetzt werden. Gibt es ein nützlich Fleck-Kit für die lebenden/Toten Bakterien die den Ausschluss von Farbstoffen aus der lebenden Bakterien abhängig. Allerdings, wenn eine gemischte Bevölkerung von Bakterienarten vorhanden ist dann lebensfähig Zellzahlen der Arten von Interesse ist wahrscheinlich die einfachste Methode um festzustellen, ob die Inkubation bakterielle Tod geführt hat.
Mittlere Zusammensetzung beeinflussen Bakterien überleben und Wachstum. Wurzel Exsudat und Materialien aus der Wunde entlassen und schneiden Websites bieten Substrat um bescheidene bakterielles Wachstum zu unterstützen. Stickstoff, Phosphat und Eisen neigen dazu, unter diesen Bedingungen zu begrenzen. Zweiwertige kationen wie Calcium und Magnesium können die Haftung beeinflussen. In manchen Fällen kann Kohlenstoffquelle Adhäsion wirkt. pH ist ebenfalls von Bedeutung. Im Allgemeinen ist der pH-Wert der Rhizosphäre zwischen 5,5 und 6,5.
Es ist notwendig, vorsichtig im Umgang mit Bakterien mit Antibiotika-Resistenz gekennzeichnet werden. Die am häufigsten verwendeten Antibiotika sind Rifampicin und Nalidixic Säure. Resistenz gegen diese Antibiotika ist in der Regel aufgrund von Mutationen in den chromosomalen Genen (RNA-Polymerase und Gyrase, beziehungsweise) und nicht so leicht auf eine andere Belastung während der Inkubation übertragen werden. Diese Art von Widerstand führt auch nicht in die Zerstörung oder Veränderung des Antibiotikums. Kennzeichnung von Bakterien mit einem Plasmid übertragene gen Marker wird nicht empfohlen, es sei denn, das Plasmid auf andere Bakterien übertragen werden kann. Die Antibiotikaresistenz darf nicht durch Abbau oder chemische Modifikation des Antibiotikums wie Antibiotika empfindlichere Bakterien dann in der Lage werden, auf Antibiotika Platten zu wachsen, wenn sie sich in der Nähe die resistenten Bakterien sind.
In diesem Artikel beschriebenen Methoden eignen sich für kleine Stichprobengrößen bzw. Experimente sollten die Proben (z. B. Experimente mit menschlichen Krankheitserreger) enthalten sein. Für große Stichproben (über 100 g Material oder mehr als 50 Pflanzen) wäre andere Methoden oder drastische Änderung dieser Methoden benötigt10,19,32,19,33, 34 , 35 , 36. die Anwesenheit einer großen Anzahl von Mikroorganismen als die untersuchten Arten kann auch erhebliche Probleme aufwerfen. Mögliche Lösungen umfassen die Verwendung von Bakterien mit einem fluoreszierenden Proteins oder Antibiotika-Resistenz gekennzeichnet, wie in den Schritten 6.1.2 und 7.3 beschrieben. Wenn die Bakterien von Interesse selten Individuen in einer großen Population von anderen Mikroorganismen sind können diese Markierungen nicht ausreichend, um die Zahl der untersuchten Bakterien eindeutig bewerten jedoch.
Alle die hier beschriebenen Methoden sind Labormethoden basiert. Geringfügige Änderungen wären für gewächshausstudien erforderlich. Weitere wesentliche Änderungen werden voraussichtlich für Feldstudien verlangt werden, wo Protozoen, Insekten und andere Tiere Prädation und Klima-Variante erschweren die Bereitstellung von definierten Bedingungen für die Experimente. In Zukunft können diese Methoden erweitert werden die Wechselwirkungen von zwei oder mehr Mikroorganismen auf der Pflanzenoberfläche.
Offenlegungen
Der Autor erklärt, dass sie keine finanziellen widerstreitenden Interessen hat.
Danksagungen
Der Autor dankt Susan Whitfield für Vorbereitung der Figuren und Camille Martin und Hillary Samagaio für Hilfe mit einigen der Experimente.
Materialien
| Name | Company | Catalog Number | Comments |
| light microscope | any | N/A | phase contrast or Nomarski optics are helpful, for studies using fluorescent markers a fluorescence microscope is required. |
| seeds | any | N/A | make a note of the seed lot number and the cultivar |
| bleach | any | N/A | |
| bath sonicator | any scientific supply company | N/A | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| nutrient agar | Difco | 001-01-8 | |
| soytone | Difco | 24360 | |
| Sand | sea sand Fisher Scientific | S25 | |
| sand | Sigma-Aldrich | S9887 | |
| conetainers | Stuewe & Sons, Inc. | Ray-Leach cone-tainers | many different sizes are available to suit the type of plant you wish to grow |
| parafilm | any scientific supply company | N/A | |
| MS salts | Sigma-Aldrich | M5524 | |
| parrafin | any | N/A | |
| centrigfuge | any scientific company | N/A | to pellet most bacteri only a small centrifuge with a max force of 10,000 XG is needed |
| vortex mixer | any scientific company | ||
| micrometer | ACCU-SCOPE Accessories | A3145 | |
| Sedgwick-rafter counting cell | Hauser Scientific | HS3800 | |
| probe-clip press-seal incubatin chamber | Sigma-Aldrich | Z359483 | |
| rifampicin | Sigma-Aldrich | R3501 | |
| naladixic acid | Sigma-Aldrich | n8878 | |
| Live/dead stain | In Vitrogen Molecular Bioprobes | L7007 |
Referenzen
- Heaton, J. C., Jones, K. Microbial contamination of fruit and vegetables and the behaviour of enteropathogens in the phyllosphere: a review. J. Appl. Microbiol. 104, 613-626 (2008).
- Beuchat, L. R. Ecological factors influencing survival and growth of human pathogens on raw fruit and vegetables. Microbes Infect. 4, 413-423 (2002).
- Berger, C. N., et al. Fresh fruit and vegetables as vehicles for the transmission of human pathogens. Environ. Microbiol. 12, 2385-2397 (2010).
- Lugtenberg, B. J., Chin, A. W. T., Bloemberg, G. V. Microbe-plant interactions: principles and mechanisms. Antonie Van Leeuwenhoek. 81, 373-383 (2002).
- Preston, G. M., Haubold, B., Rainey, P. B. Bacterial genomics and adaptation to life on plants: implications for the evolution of pathogenicity and symbiosis. Curr. Opin. Microbiol. 1, 589-597 (1998).
- Compant, S., Duffy, B., Nowak, J., Clement, C., Barka, E. A. Use of Plant Growth-Promoting Bacteria for Biocontrol of Plant Diseases: Principles, Mechanisms of Action, and Future Prospects. Appl. Environ. Microbiol. 71, 4951-4959 (2005).
- Mathews, S. L., Smith, R. B., Matthysse, A. G. A comparison of the retention of pathogenic Escherichia coli. O157 by sprouts, leaves and fruits. Microb. Biotechnol. 7, 570-579 (2014).
- Fuqua, C., Matthysse, A. G. Methods for studying bacterial biofilms associated with plants. Methods Enzymol. 337, 3-18 (2001).
- Torres, A. G., Jeter, C., Langley, W., Matthysse, A. G. Differential binding of Escherchia coli.O157:H7 to alfalfa, human epithelial cells, and plastic is mediated by a variety of surface structures. Appl. Environ. Microbiol. 71, 8008-8015 (2005).
- Matthysse, A. G., Deora, R., Mishra, M., Torres, A. G. Polysaccharides cellulose, poly-beta-1,6-n-acetyl-D-glucosamine, and colanic acid are required for optimal binding of Escherichia coli O157:H7 strains to alfalfa sprouts and K-12 strains to plastic but not for binding to epithelial cells. Appl. Environ. Microbiol. 74, 2384-2390 (2008).
- Matthysse, A. G., et al. The effect of cellulose overproduction on binding and biofilm formation on roots by Agrobacterium tumefaciens. Mol. Plant Microbe Interact. 18, 1002-1010 (2005).
- Matthysse, A. G., Kijne, J. W., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. Attachment of Rhizobiaceae to plant cells. The Rhizobiaceae. , 235-249 (1998).
- Matthysse, A. G. Conditioned medium promotes the attachment of Agrobacterium tumefaciens.strain NT1 to carrot cells. Protoplasma. 183, 131-136 (1994).
- Matthysse, A. G., McMahan, S. The effect of the Agrobacterium tumefaciens. attR mutation on attachment and root colonization differs between legumes and other dicots. Appl. Environ. Microbiol. 67, 1070-1075 (2001).
- Jeter, C., Matthysse, A. G. Characterization of the binding of diarrheagenic strains of E. coli.to plant surfaces and the role of curli in the interaction of the bacteria with alfalfa sprouts. Mol. Plant Microbe Interact. 18, 1235-1242 (2005).
- Matthysse, A. G., McMahan, S. Root colonization by Agrobacterium tumefaciens is reduced in cel, attB, attD, and attR mutants. Appl. Environ. Microbiol. 64, 2341-2345 (1998).
- Morton, E. R., Fuqua, C. Phenotypic analyses of Agrobacterium. Curr. Protoc. Microbiol. , (2012).
- Brandl, M. T. Plant lesions promote the rapid multiplication of Escherichia coli O157:H7 on postharvest lettuce. Appl. Environ. Microbiol. 74, 5285-5289 (2008).
- Franz, E., et al. Quantification of contamination of lettuce by GFP-expressing Escherichia coli O157:H7 and Salmonella enterica serovar Typhimurium. Food Microbiol. 24, 106-112 (2007).
- Morton, E. R., Fuqua, C. Laboratory maintenance of Agrobacterium. Curr. Protoc. Microbiol. , (2012).
- Lugtenberg, B. J., Kravchenko, L. V., Simons, M. Tomato seed and root exudate sugars: composition, utilization by Pseudomonas biocontrol strains and role in rhizosphere colonization. Environ. Microbiol. 1, 439-446 (1999).
- Murashige, T., Skoog, F. A Revised Medium for Rapid Growth and Bio Assays with Tobacco Tissue Cultures. Physiologia Plantarum. 15, 473-497 (1962).
- Danhorn, T., Fuqua, C. Biofilm formation by plant-associated bacteria. Annu. Rev. Microbiol. 61, 401-422 (2007).
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- O'Toole, G., Kaplan, H. B., Kolter, R. Biofilm formation as microbial development. Annu. Rev. Microbiol. 54, 49-79 (2000).
- Charkowski, A. O., Barak, J. D., Sarreal, C. Z., Mandrell, R. E. Differences in growth of Salmonella enterica and Escherichia coli O157:H7 on alfalfa sprouts. Appl. Environ. Microbiol. 68, 3114-3120 (2002).
- Loper, J., Suslow, T., Schroth, M. Lognormal distribution of bacterial populations in the rhizosphere. Phytopathology. 74, 1454-1460 (1984).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front Plant Sci. 5, 252 (2014).
- Douglas, C. J., Halperin, W., Nester, E. W. Agrobacterium tumefaciens mutants affected in attachment to plant cells. J. Bacteriol. 152, 1265-1275 (1982).
- Tomlinson, A. D., Fuqua, C. Mechanisms and regulation of polar surface attachment in Agrobacterium tumefaciens. Curr. Opin. Microbiol. 12, 708-714 (2009).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front Plant Sci. 5, (2014).
- Wright, K. M., et al. The endophytic lifestyle of Escherichia coli O157:H7: quantification and internal localization in roots. Phytopathology. 103, 333-340 (2013).
- Berger, C. N., et al. Fresh fruit and vegetables as vehicles for the transmission of human pathogens. Environ. Microbiol. 12, 2385-2397 (2010).
- Erickson, M. C., et al. Internalization and Fate of Escherichia coli O157:H7 in Leafy Green Phyllosphere Tissue Using Various Spray Conditions. J. Food Prot. 77, 713-721 (2014).
- McKellar, R. C., et al. Evaluation of different approaches for modeling Escherichia coli O157:H7 survival on field lettuce. Int. J. Food Microbiol. 184, 75-85 (2014).
- Wu, F. M., et al. Factors influencing the detection and enumeration of Escherichia coli O157:H7 on alfalfa seeds. Int. J. Food Microbiol. 71, 93-99 (2001).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten