Method Article
Adhérence des bactéries à planter des Surfaces mesurées en laboratoire
Dans cet article
Résumé
Une méthode simple permettant de mesurer et de caractériser l’adhérence bactérienne aux plantes, en particulier les racines et les pousses, est décrit dans cet article.
Résumé
Cet article décrit une méthode pour mesurer la liaison bactérienne aux surfaces de plantes axéniques au microscope et grâce à l’utilisation du nombre de cellules viables. Matières végétales utilisées comprennent des racines, des pousses, des feuilles et coupez les fruits. Les méthodes décrites sont peu coûteux, facile et adapté pour la petite taille des échantillons. Liaison est mesuré en laboratoire et une variété de conditions et du milieu d’incubation peut être utilisée. Les effets des inhibiteurs peuvent être déterminées. Des situations qui favorisent et inhibent la liaison peuvent également être évaluées. Dans certains cas, il est possible de distinguer si diverses conditions modifient liaison principalement en raison de leurs effets sur la plante ou sur les bactéries.
Introduction
La mesure de fixation bactérienne de planter des surfaces est devenu importante dans trois situations disparates. La première situation est l’examen de la transmission d’agents pathogènes humains sur plante surfaces1,2,3. Le but ici est d’empêcher les bactéries ou d’enlever ou tuer les bactéries liées et donc de réduire la transmission de la maladie de matériel végétal. La deuxième situation est l’examen de la fixation des agents phytopathogènes pour planter des surfaces4. Une fois de plus, le but ici est d’empêcher la liaison ou pour enlever ou tuer les bactéries liées et donc de réduire la maladie. La troisième situation est l’examen de la fixation des bactéries symbiotiques ou favorisant la croissance des plantes5,6. Le but ici est de promouvoir les bactéries contraignant et donc à l’augmentation de la santé des plantes et des cultures donne.
Les techniques pour mesurer la liaison bactérienne de planter des surfaces décrites dans cet article sont relativement faciles à réaliser et peu coûteux. Les seules exigences sont un microscope et des matériaux trouvés généralement dans un laboratoire de bactériologie. Pour certaines techniques un sonicateur bain est utile. Les techniques décrites sont conçus pour la liaison d’expériences réalisées à l’aide de relativement petite taille des échantillons. Mesures de liaison sont effectuées en laboratoire, bien qu’il peut être possible de modifier certaines de ces techniques pour utilisation en serre ou dans le domaine.
Ces techniques ont été utilisées pour mesurer la fixation bactérienne aux racines, pousses, feuilles coupées, fruits coupés et des tomates cerises intacts dans le laboratoire7,8,9,10,11, 12,13,14,15. Ils ont également été utilisés pour mesurer la colonisation des racines des plantes qui poussent dans le sol ou le sable dans le laboratoire16. Les techniques ont été utilisées avec de nombreuses espèces bactériennes, y compris les Agrobacterium tumefaciens, Sinorhizobium meliloti, Escherichia coli, Salmonella enterica et Pseudomonas fluorescens. Une description utile des méthodes d’évaluation de l’interaction d’a. tumefaciens avec surfaces se trouvent à Morton et Antoine Fuqua (2012)17. Dans tous les cas la taille de l’échantillon impliquée était petite, généralement moins que 25 à 50 plantes. Les techniques décrites sont adaptés pour une utilisation avec des agents pathogènes humains qui doivent rester confinée au cours des expériences.
Protocole
1. préparation du matériel végétal axénique
- Préparer les plants cultivés dans l’eau.
- Placer un petit nombre de graines (moins de 30) dans un 30, 50, 100 ou carafe en verre 150 mL. Nous avons utilisé des tomates, luzerne, Arabidopsis thaliana, pois, haricots, tabac, laitue et graines de carotte avec ce protocole.
Remarque : Pour déterminer combien de graines peut être stérilisés ensemble sans contamination s’étalant de semence d’une à l’autre, reportez-vous à l’étape 1.1.2. - Couvrir les graines avec l’éthanol à 80 % et agiter brièvement. Laissez les graines tremper pendant 1 min.
- Décanter l’éthanol et de couvrir les graines avec une solution de 50 % de l’eau de Javel commerciale volume (NaClO) et 0,1 % Triton X-100 dans l’eau du robinet. Laissez les graines tremper pendant 20 min.
Remarque : Si les graines sont volumineux, tels que les graines de haricot, il peut être nécessaire d’allonger la durée de trempage complètement tuer les champignons sur la surface de la graine. - Décanter le mélange d’eau de Javel et lavez les graines 3 fois avec de l’eau stérile. Laissez les graines tremper dans l’eau pendant 1 minute pour chaque lavage.
- Pour obtenir des semis axéniques ajoutent une petite quantité d’eau stérile entre 5 et 25 mL selon la taille des graines. Versez les graines et l’eau dans une boîte de Petri stérile pour la germination des graines.
NOTE : L’eau doit recouvrir le fond du plat mais pas couvrir les graines afin d’encourager la formation des poils absorbants. Axéniques décrit une culture dans laquelle seulement une seule espèce d’organisme est présente. - Incuber dans l’obscurité jusqu'à ce que les plants atteignent la taille souhaitée entre 1 et 10 cm (environ 5 jours pour la tomate et a. thaliana et 1 à 3 jours pour la luzerne). Pour les plus grosses graines utiliser un récipient stérile couvert d’une capacité de plus de 100 mL, comme un plat en verre recouvert de papier.
- Placer un petit nombre de graines (moins de 30) dans un 30, 50, 100 ou carafe en verre 150 mL. Nous avons utilisé des tomates, luzerne, Arabidopsis thaliana, pois, haricots, tabac, laitue et graines de carotte avec ce protocole.
- Déterminer la fréquence de contamination d’un lot de semences particulière avec les micro-organismes qui demeurent viables sur ou dans les graines après la stérilisation de la surface.
NOTE : Ceci est nécessaire parce que les graines occasionnels transportent micro-organismes sous les téguments de la graine qui ne peut être tué par la surface-stérilisation. Utiliser la fréquence de graines contaminées dans un lot de semences pour déterminer combien de graines pour stériliser simultanément sans un risque élevé du groupe des graines contamination due à une seule graine par des bactéries ou des champignons qui survivent au traitement.- Réalisez les étapes 1.1.1 à 1.1.3.
- Placer les graines sur une gélose nutritive Pétri. Mettre environ 10-30 graines dans chaque plat leur espacement afin qu’ils peuvent être marqués individuellement. Sceller les plats avec du ruban ou étanchéité film.
- Incuber pendant 3-5 jours à 25 ° C, marquant chaque jour pour l’excroissance visible des micro-organismes des graines.
- Préparer les plants cultivés dans du sable. Semis peuvent être cultivés dans le sable pour une utilisation dans des expériences de l’adhérence bactérienne.
- Réalisez les étapes 1.1.1 à 1.1.3.
- Stériliser le sable de quartz ou de la mer dans l’autoclave. Ces deux contiennent des matières organiques. Si cela va affecter l’expérience, laver le sable en le couvrant avec deux fois son volume de 0,1 M HCl. mélanger pendant 10 min. autoriser le sable pour s’installer et décanter le liquide. Rincer le sable 3 fois avec de l’eau, et une fois avec 80 % éthanol suivie deux rinçages ajoutant de l’eau en utilisant le même protocole que pour le 0,1 M HCl. stériliser le sable lavé dans l’autoclave.
- Stériliser les récipients en les immergeant dans bleach 50 % pendant 5 min et rincez-les 5 fois avec de l’eau stérile par submersion. Laissez-les sécher et sceller le fond avec film d’étanchéité. Le film obtenu à partir du fabricant est généralement exempt de micro-organismes à l’intérieur côté qui doit être placée face à l’intérieur du conteneur.
- Mélanger le sable stérile avec de l’eau assez stérile à humide, il suffit que les bâtons de sable. Le montant dépendra comment sécher le sable est. Généralement 10 à 35 % du volume de sable est suffisante. Placer le sable humide dans le bac permettant une profondeur suffisante pour la longueur des racines, espace nécessaire et suffisant au-dessus du sable pour la croissance de la pousse. Les distances exactes dépendent de l’espèce et la variété de plante utilisée.
- Graines de plantes.
- Faire un trou peu profond dans le sable avec une baguette de verre stérile juste assez profonde pour placer les semences sous la surface du sable (environ 1-5 mm). Placez les graines dans le trou. Recouvrir d’une fine couche de sable. Sceller le haut du conteneur avec étanchéité film afin d’éviter la perte d’eau et entrée de microbes supplémentaires.
- Cultiver les plantes en laboratoire sous une lumière ou dans la serre à une température appropriée et la durée du jour pour l’espèce et la variété de plante. Pour la tomate, luzerne ou Arabidopsis thaliana utilisent la température ambiante et cycles de lumière/obscurité de 12 h.
- Planter les semis dans un trou dans le sable (1-2 mm de diamètre et assez profondément que la racine est recouverts de sable). Faire le trou avec une baguette de verre stérile. Introduire la racine dans le trou à l’aide d’un stérile en acier inoxydable crochet si nécessaire et remplir les côtés du trou avec du sable.
- Vous pouvez également faire pousser des plantes axéniques sur ou dans la gélose contenant un mélange de sels tels que les sels de MS. Utiliser des pousses de plantes axéniques cultivées sur gélose directement. Éviter les racines cultivées avec agar comme agar adhère à la surface de la plante et les bactéries. Cela peut entraîner une fausse impression de l’adhérence bactérienne à la surface de la plante.
2. Elaboration d’autres matières végétales
- Laver les végétaux cultivé en serre avec de l’eau avant utilisation pour réduire le nombre de microbes indigènes. Il y aura des bactéries et des champignons sur les plantes. Protozoaires sera présent dans le sol, sur les racines et éventuellement sur les feuilles. Lavage avec eau de Javel ou de l’éthanol va modifier la surface de la plante et n’est pas recommandé.
- Si après lavage l’eau un nombre important de microbes reste, traiter les plantes avec un savon liquide dilué ou diluer l’eau oxygénée (0,01 %) d’enlever ou de tuer les microbes. C’est généralement moins dommageable pour la plante que l’eau de Javel ou de l’éthanol.
- Comme plante, matériel acheté sur un marché local est difficile à décontaminer, choisir le matériel qui ne semble pas avoir fait l’objet d’entreposage à long. Éviter les parties du matériau qui apparaissent marron ou endommagés.
- Laver à l’eau et faire des coupes fraîches sur les extrémités du matériau qui ont précédemment été coupées avant de l’utiliser, à moins que les interactions entre les bactéries de test avec les bactéries (et eucaryotes) qui aura accumulé à couper les sites présentent un intérêt. Ne pas laver avec eau de Javel ou de l’éthanol comme ils modifieront la surface de la plante.
- Traiter les sites de plaie. Sites de plaie offrent souvent des bactéries l’accès à l’intérieur de la matière végétale18.
- Bloquer ou réduire l’attachement des bactéries aux et le mouvement par le biais, plaie ou coupes sites créés lors de la préparation du matériel en trempant le bord coupé à la paraffine fondue ou en peignant le site avec de la paraffine fondue à l’aide d’une petite brosse. Ne pas utiliser une spatule ou tout mettre en œuvre forte car cela pourrait endommager le tissu.
- Joint tige cicatrices sur les fruits comme les tomates avec de la paraffine. Certaines bactéries seront nagées dans la zone située sous la paraffine, mais leur nombre est généralement de petite taille.
- Si une entrée bactérienne en sites de plaie présente un intérêt, estimation de la distance qui peuvent déplacer des bactéries transportée par les cours d’eau ou diffusion en observant le mouvement d’un colorant ajouté à la solution. Marquer des bactéries motiles en introduisant un gène codant pour une protéine fluorescente comme la protéine fluorescente verte (GFP) en eux et tracer à l’aide de la microscopie en fluorescence, décrit à l’étape 6.1.219.
3. préparation des bactéries
- Se développer les bactéries. Utiliser un support qui se rapproche de plus près les conditions, les bactéries sont susceptibles d’avoir été exposés au juste avant de rencontrer la plante dans le monde réel en dehors du laboratoire. Carbone et azote des sources ainsi que la présence d’ions, cations divalents particulièrement (Ca, Mg, Fe, Mn et Zn) et phosphate et le pH du milieu est importante.
- Utiliser un milieu minimal AB ou bouillon Luria pour a. tumefaciens et autres bactéries de sol20. Pour e. coli, qui peuvent être fournis dans le tube digestif, utiliser du bouillon de Luria.
- Ajouter inducteurs tels que les exsudats des racines ou des extraits de plantes commerciales telles que soytone ou sucres comme le saccharose ou le xylose à la moyenne de21. Si ces substances sont utilisées comme inducteurs, ajouter une concentration faible, par exemple de 0,01 %. Si elles sont sources de carbone utilisé, ajouter une concentration plus élevée, par exemple de 0,1 %.
- Préparation de l’inoculum bactérien. Diluer les bactéries dans l’eau stérile ou dans le milieu dans lequel l’incubation s’effectuera et ajoutez-les à la matière végétale. La dilution appropriée est examinée à l’étape 4.2 et 4.3.
- Pour retirer le support de croissance avant d’utiliser les bactéries, centrifuger la suspension bactérienne à 10 000 x g pendant 2 min, décanter le liquide surnageant et remettre en suspension les bactéries au vortex dans le même milieu qui sera utilisé pour l’incubation avec le matériel végétal. Cette méthode peut supprimer ou diminuer le nombre ou la quantité de matériel extracellulaire et appendices comme les exopolysaccharides, capsules, flagelles ou pili. S’il est important que ces structures de surface restent intacts, puis simplement diluer les bactéries avant d’inoculer les ou utiliser la méthode alternative indiquée au point 3.2.2.
- Comme méthode alternative pour enlever le milieu de culture, récolter les germes sur un filtre de nitrocellulose ou en polycarbonate avec une taille de pores de 0,2 µm ou moins. Laver les bactéries avec le milieu d’incubation et les remettre en suspension en agitant doucement ou vortex du filtre dans un contenant stérile du milieu.
4. l’inoculation de la bactérie
- Déterminer le nombre de bactéries à ensemencer en ce qui concerne la mesure de référence pour déterminer l’adhésion bactérienne et la durée de l’incubation.
- Pour les études microscopiques concernant l’incubation fois inférieure à 1 jour inoculer un nombre relativement élevé de bactéries. Ajouter un montant de culture pour atteindre une concentration bactérienne finale de plus de 106 bactéries / mL. Pour plus longues périodes d’incubation diminue la taille de l’inoculum bactérien.
- Pour les études dans lequel adhésion bactérienne sera mesurée par le nombre de cellules viables, ajouter un montant de culture pour atteindre une concentration bactérienne finale de 103 106 bactéries / mL.
- Évitez d’ajouter des bactéries tant que leur métabolisme modifie la concentration de pH ou de l’oxygène dans le milieu d’incubation. Mesurer le pH à l’aide de papier pH ou une électrode. Mesurer la concentration d’oxygène à l’aide d’une électrode à oxygène.
- Pour les plantes cultivées dans le sable, inoculer de trois manières possibles.
- Inoculer les semences avec les bactéries avant la plantation en faisant tremper les graines pendant 1 min en suspension dans l’eau environ 106 bactéries / mL.
- Ensemencer la racine par la germination des semis axéniques tel que décrit à l’article 1 et lorsque les racines des semis est environ 1 cm de long plongeant ou placer le Plant entier dans une suspension de 106 bactéries / mL dans l’eau pendant 1 min.
- Inoculer le sable par les bactéries de mélange avec le sable avant la plantation pour donner une concentration finale de 10 environ3 bactéries / mL ou en arrosant les plantules avec une suspension de 106 bactéries / mL après la plantation.
5. l’incubation des bactéries avec le matériel végétal
- Pour l’incubation dans un milieu liquide, incuber les bactéries avec le matériel végétal dans l’eau stérile ou de sels de sucrose et minérale ou végétale de milieu de culture tissulaire (par exemple un 01:10 dilution des sels MS)22,23.
- Utiliser un récipient à laquelle les bactéries n’adhèrent pas. Essayez de garder la surface de la plante en permanence couvert par immersion ou par agitation douce. Une agitation vigoureuse peut empêcher l’adhérence ou même éliminer les bactéries de la surface de la plante.
- Observer le matériel végétal au microscope après différents intervalles de temps pour déterminer le moment d’arrêter l’incubation et de faire des mesures. Une évolution temporelle de l’adhérence est souvent précieuse avec des échantillons prélevés toutes les 1 à 4 h ou tous les jours selon la vitesse de l’interaction.
- Pour incuber dans le sable, appliquer les mêmes considérations décrites à l’incubation dans un milieu liquide à l’étape 5.1.
6. mesure de l’adhérence à l’aide de la microscopie
- Effectuer des mesures microscopiques avec optique Nomarski ou contraste de phase pour le visionnement facile des bactéries sur des surfaces. Cependant, n’importe quel microscope en champ lumineux avec un grossissement de 20 X ou version ultérieure peut être utilisé.
- L’observation microscopique permet de déterminer si les bactéries sont aléatoirement distribués ou situés dans des sites spécifiques. Vérifiez également si elles sont tenues individuellement ou par groupes. Recherchez la présence de microcolonies suggérant la croissance bactérienne ou happé après adhésion. Vérifier si les bactéries semblent former un biofilm sur la surface.
Remarque : Un biofilm est un grand nombre de bactéries à la surface et entouré d’une matrice extracellulaire23,24,25. La structure peut être lisse et uniforme ou avoir une architecture plus complexe. Méthodes pour l’étude des biofilms associés aux surfaces de la plante ont été décrits17. - Utiliser des bactéries contenant le tag un marqueur fluorescent. Si les autres bactéries sont présentes et les bactéries d’intérêt sont identifiés par un marqueur fluorescent comme protéine fluorescente verte, utiliser la microscopie de fluorescence pour déterminer la présence de la bactérie étiquetée en grappes d’autres bactéries26. Pour GFP, utiliser un filtre avec une excitation 490 nm et d’émission à 520 nm.
- Vérifiez que la balise fluorescente n’affecte pas négativement les bactéries en observant l’adhérence au matériau axénique d’un mélange égal de bactéries de type sauvage et marqués de la même souche utilisant optique Nomarski et de fluorescence. Si les bactéries fluorescentes et sombres sont aléatoirement mixtes et présents en nombre égal, puis la balise n’interférence pas avec le test.
- L’observation microscopique permet de déterminer si les bactéries sont aléatoirement distribués ou situés dans des sites spécifiques. Vérifiez également si elles sont tenues individuellement ou par groupes. Recherchez la présence de microcolonies suggérant la croissance bactérienne ou happé après adhésion. Vérifier si les bactéries semblent former un biofilm sur la surface.
- Déterminer le nombre de bactéries fixées.
Remarque : Il est très difficile de déterminer le nombre de bactéries fixées au microscope. Lorsque la liaison est à surfaces irrégulières végétales, il n’est généralement pas possible d’obtenir une mesure quantitative. Microscopie électronique (ne pas abordées dans cet article) peut être utilisé pour effectuer ces mesures.- Lorsque les bactéries sont liés à une surface lisse comme un cheveu de la racine, compter le nombre de bactéries lié au bord de la chevelure de la racine par la longueur des poils absorbants mm. Prendre soin d’utiliser des poils absorbants d’à peu près la même taille et la forme en comparant les mesures.
- Pour déterminer la taille des objets dans le microscope, utilisez une diapositive commerciale avec marquages mesurées à ce sujet. Observer et photographier cette diapositive avec les mêmes paramètres que celui utilisé pour le matériel expérimental et utiliser les images obtenues pour déterminer la taille des objets dans les photomicrographies.
- Préparation des échantillons pour la microscopie.
- Laver l’échantillon. Passer l’échantillon à une goutte d’eau ou incubation moyen sur une lame de microscope et observer directement.
NOTE : Ceci a l’avantage que si il n’y avait aucune croissance bactérienne ou la mort bactérienne réelle il n’est peu probable que beaucoup de bactéries libres. Prendre l’absence de bactéries libres comme un signe d’avertissement que peut il y avoir été mort bactérienne ou bactérienne liaison vers le conteneur dans lequel l’incubation a été réalisée. L’effet de l’échantillon de lavage est illustré à la Figure 1. - Laver l’échantillon doucement dans l’eau ou incubation moyenne en le plaçant dans un flacon de liquide et en retournant le flacon doucement. Puis placez l’échantillon sur la lame de microscope en fraîche liquide pour l’observation.
- Monter l’échantillon dans un liquide à l’aide d’une lamelle couvre-objet ordinaire et une lame de microscope.
- Si l’échantillon est épais et donc ferait un renflement sous la lamelle couvre-objet, utilisez une lamelle de press-apply. Ces lamelles ont un anneau de caoutchouc ou de plastique autour du bord de la lamelle couvre-objet. Placer les échantillons et les liquides dans le puits de la lamelle couvre-objet et ensuite placer la lame sur le dessus et appuyez doucement pour sceller la lamelle couvre-objet de la diapositive. Inverser et examiner.
- Sinon utiliser une algue comptage de lame et lamelle couvre-objet de la même manière. Remarque qui se glisse avec cette profondeur ne peut généralement être examinée avec un objectif de plus de 20 X grossissement.
- Laver l’échantillon. Passer l’échantillon à une goutte d’eau ou incubation moyen sur une lame de microscope et observer directement.

Figure 1 : Les étapes de la préparation d’un échantillon pour déterminer le nombre de bactéries liés. A. tumefaciens liaison aux poils absorbants de tomate (A, B et C) et de fils de nylon (D, E et F). Dans les échantillons montés dans l’eau sans lavage tous les deux liés (flèches noires) les bactéries et les bactéries libres (flèches blanches) peuvent être vu (A et D). Après que lavage, que la bactérie liée demeure mais les bactéries libres ne sont plus présents (B et E). Après sonication les bactéries liées ont été retirés de la surface de l’échantillon (C et F). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
- Si les bactéries fluorescentes ont été utilisés, examiner l’échantillon au microscope après avoir retiré le sable. Racines de plantes cultivées dans le sable ne sont généralement pas appropriés pour la microscopie comme les particules de sable gênent l’observation des bactéries.
- Retirez la plante d’un conteneur de croissance selon les directives de l’étape 7. Placer les racines dans un récipient d’eau et mélanger doucement pour enlever le sable qui se déposent au fond du récipient.
- Retirer la plante de l’eau de lavage et monter l’échantillon comme au point 6.3.3.
7. mesure de l’adhérence à l’aide de cellules viables compte
- Déterminer le nombre de bactéries viables de joints en utilisant un sonicateur.
- Enlever les bactéries non liés. Placer l’échantillon dans un flacon avec suffisamment d’eau, lavage tampon ou incubation moyenne pour couvrir le matériel végétal et inverser la fiole plusieurs fois.
- S’il n’y a plus de 10 bactéries libres3 / mL présent lors de l’incubation initiale, effectuer des lavages séquentielles pour supprimer tous les. Vérifier au microscope optique (Voir l’étape 6.3) pour déterminer la présence de bactéries libres.
- Rincer jusqu'à ce qu’il y a une réduction substantielle du nombre de bactéries libres mais n’oubliez pas qu’il y a un équilibre entre les bactéries liées et non liées donc le nombre de bactéries libres ne peut jamais réduire à zéro.
- Retirer l’échantillon dans le flacon de lavage liquide avec une pince ou une spatule.
- Déterminer le nombre de bactéries liées dans des incubations de plantes dans un liquide.
- Suspendre l’échantillon lavé dans un flacon et couvrez-la avec un volume mesuré de l’eau, milieu d’incubation ou tampon de lavage. Utiliser suffisamment de liquide pour couvrir l’échantillon. Placer la fiole dans un sonicateur bain et laisser agir il pendant un min.
- Supprimer l’exemple et l’examiner sous microscope photonique pour déterminer si la bactérie liée ont été retirée de l’usine. L’élimination des bactéries d’un échantillon par sonication est illustrée à la Figure 1. S’il y a toujours lié bactéries présentes continuent sonification l’échantillon jusqu'à tel un temps pendant lequel aucune bactérie liée ne reste sur la surface de l’échantillon. Si la bactérie liée ne peut pas être supprimée par sonication, ajouter du sable de quartz stérile de 1 à 10 mg/mL et répétez la sonication et l’examen microscopique.
- Déterminer que la procédure de sonication qui semble plus efficace à l’étape 7.1.3.2 ne réduit pas la viabilité de la bactérie en suspendant des bactéries d’une culture liquide dans la solution à utiliser pour la sonication (ajouter du sable de quartz, si c’était nécessaire). Déterminer le nombre de cellules viables. Laisser agir la bactérie et déterminer le nombre de cellules viables à nouveau.
Remarque : S’il y a une diminution du nombre de cellules viables, modifiez la procédure en modifiant la composition de l’époque sonication et/ou liquide jusqu'à ce que le traitement n’a aucun effet sur le nombre de cellules viables. - Déterminer que le tampon de dilution utilisé pour le nombre de cellules viables n’altère pas la validité des bactéries qui ont été incubées dans les conditions utilisées en comparant le nombre de cellules viables réalisé à l’aide de tampons de différentes dilution et/ou d’eau.
- Enlever les bactéries non liés. Placer l’échantillon dans un flacon avec suffisamment d’eau, lavage tampon ou incubation moyenne pour couvrir le matériel végétal et inverser la fiole plusieurs fois.
- Déterminer le nombre de bactéries viables de joints à l’aide d’homogénéisation.
- Placer l’échantillon dans un mortier stérile ou mélangeur ou tout autre dispositif d’homogénéisation. Couvrez-la avec un volume mesuré de l’eau stérile, milieu d’incubation ou27de la mémoire tampon de lavage. Utiliser un volume suffisant pour couvrir l’échantillon. Pour un blender utiliser 100 mL.
- Broyer l’échantillon jusqu'à ce qu’elle soit bien homogène. Vérifier le pH après que homogénéisation pour être sûr qu’acide libérée des tissus végétaux n’a pas causé une chute de pH inférieur à 7. Si le pH a baissé utiliser un tampon comme un tampon de phosphate dans le liquide d’homogénéisation pour maintenir le pH.
- Déterminer le nombre de cellules viables.
- Utiliser des bactéries marquées avec la résistance aux antibiotique.
- Dans les cas où plus d’un type de bactérie est présent, marquer les souches bactériennes à l’aide de résistance spontanée aux antibiotiques (en général la rifampicine et à l’acide nalidixique).
- Déterminer le niveau de l’antibiotique à utiliser. S’il existent des cultures des autres organismes devrait être présent lors de l’incubation, plaque de ces cultures cultivées dans les conditions de l’incubation prévue avec des plantes sur des plaques contenant une gamme de concentrations de l’élu aux antibiotiques. Déterminer la concentration la plus faible qui ne permet pas la croissance de le quelconque de ces organismes. Il s’agit de la plus faible concentration d’antibiotique auquel la bactérie test devra être résistant.
- Obtenir des mutants résistants aux antibiotiques spontanés. Atteindre une culture de la bactérie en milieu riche fin journal ou de la phase stationnaire. Plaque 0,1 mL non dilué sur une plaque contenant l’antibiotique désiré à une concentration appropriée. Déterminer les concentrations d’utiliser tel que décrit à l’étape 7.3.1.1. Conservez et purifier les bactéries qui se développent et sont donc résistants à l’antibiotique.
- Déterminer que la résistance aux antibiotique ne réduit pas la croissance des bactéries en faisant une courbe de croissance dans un milieu liquide de la société mère et les souches résistantes aux antibiotiques. Déterminer que les bactéries résistantes aux antibiotiques montrent le même niveau de colonisation du matériel végétal axéniques comme la souche parentale, si cela est possible.
- Dans les cas où plus d’un type de bactérie est présent, marquer les souches bactériennes à l’aide de résistance spontanée aux antibiotiques (en général la rifampicine et à l’acide nalidixique).
- Déterminer le nombre de bactéries viables lié aux plantes cultivées dans le sable.
- Retirer la plante du conteneur et sable. Pour supprimer les plantes de conteneurs, retirez d’abord matériau d’étanchéité sur le dessus et le fond du récipient. Placer le récipient sur un morceau de papier stérile et retirer doucement le sable ensemble ou le sol et la plante comme un grand cylindre conique en frappant doucement le récipient contre une surface pour desserrer le matériel.
- Si nécessaire, utilisez une spatule ou une tige pour desserrer le matériau autour des bords traverse le fond du récipient.
- Quand la bouteille de sable ou de terre contenant la plante est libre sur le papier, il fendu par le milieu pour révéler la racine de la plante. Si vous le souhaitez, prélever des échantillons de sable de près du bord du conteneur ainsi que près de la racine. Cela peut être utile pour déterminer la propagation (et accumulation et la croissance) de la bactérie.
- Mesurez la longueur de la racine. Prenez la racine et enlever le sable et les bactéries adhérant plus ou moins à la racine (le matériel de la rhizosphère) par trempage de la racine dans un volume mesuré de l’eau ou de la mémoire tampon et secouant doucement. Déterminer le nombre de cellules viables de bactéries dans la suspension obtenue par culture sur un milieu approprié comme la gélose Luria. Cela représente le nombre de bactéries faiblement associés à la racine.
- Retirer la plante du conteneur et sable. Pour supprimer les plantes de conteneurs, retirez d’abord matériau d’étanchéité sur le dessus et le fond du récipient. Placer le récipient sur un morceau de papier stérile et retirer doucement le sable ensemble ou le sol et la plante comme un grand cylindre conique en frappant doucement le récipient contre une surface pour desserrer le matériel.
- Enlever les bactéries étroitement liés par sonication et déterminer leur nombre comme indiqué au point 7.1.
- Sinon, pour déterminer l’emplacement sur la racine des bactéries étroitement liés placer la racine lavée sur la surface d’une boîte de Pétri contenant la gélose nutritive ou autre moyen approprié. Observez l’emplacement des colonies bactériennes sur la racine sur les 3 prochains jours à l’aide d’un microscope à dissection ou une loupe.
- Exprimer les résultats sous forme de nombre de bactéries par plante, par cm2 de surface, par la longueur des racines cm ou par gramme de poids frais de tissu. Faire des répétitions multiples sur le même jour et aussi faire des répétitions à des jours différents à l’aide de différentes cultures bactériennes et différents lots de matières végétales.
8. déterminer si l’effet des conditions d’incubation sur l’adhérence est due à une réponse de la bactérie ou la plante
- Utiliser des végétaux morts ou tués.
- Sous réserve de l’usine de matériel d’une variété de produits chimiques, fixatifs ou d’autres traitements comme la chaleur afin de le tuer. Laver soigneusement le matériel végétal dans le milieu de l’eau et d’incubation après l’utilisation de le quelconque de ces traitements. Alors inoculer la bactérie. Cela ne détruira pas la surface de la plante, mais il fera métaboliquement inactives afin qu’il ne peut pas répondre à la bactérie.
- Mesurer l’adhérence bactérienne comme décrit aux étapes 6 et 7. Également déterminer le nombre de cellules viables, ajouté à l’incubation avec le matériel végétal au début de l’incubation et le nombre de cellules viables présents à la fin de l’incubation pour s’assurer qu’aucune substance chimique toxique n’était présents lors de l’incubation.
- Utiliser des matières inanimées.
- Adhérence des bactéries utilisation de matériaux inanimé pour déterminer si un effet observé sur l’adhérence des bactéries à la matière végétale est due à un effet sur les plantes ou les bactéries. Choisissez une matière inanimée à laquelle lient les bactéries et qui est semblable dans la forme et la taille pour le matériel végétal étudié. Les possibilités incluent papier-filtre de tous types (cellulose, nitrocellulose, fibre de verre, polycarbonate, etc..), fils (nylon, coton, polyester, laine de verre, etc..), verre ou lamelles couvre-objet en plastique, acier inoxydable coupons et dialyse membranes.
- Laver la matière inanimée soigneusement à l’eau et le milieu dans lequel l’incubation s’effectuera et stériliser avant usage. La plupart des matières énumérées à l’étape 8.2.1 est stable à la stérilisation à l’autoclave.
- Incuber la matière inanimée sous les conditions souhaitées et le score comme décrit aux étapes 6 et 7. Un exemple de l’utilisation de filets en nylon pour déterminer que la liaison réduite d’a. tumefaciens aux poils absorbants à des concentrations de calcium élevé est due (au moins en partie) à un effet du calcium sur les bactéries est indiqué à la Figure 428.
Résultats
A. tumefaciens colonise la surface des racines. Afin de déterminer si la production bactérienne de la cellulose joue un rôle dans la colonisation des racines, les effets de mutations bactériennes qui empêchent la synthèse de la cellulose ont été examiné16. Les techniques décrites aux étapes 1.3 et 7.1 ont été utilisés. Graines de tomate étaient en surface stérilisé et germent dans l’eau stérile. Lorsque les racines étaient environ 2 cm de long, ils étaient trempés dans une suspension de 105 bactéries / mL et plantés dans le sol pasteurisé dans des conteneurs. Les plantes ont été cultivées pendant 14 jours à 25 ° C sur un cycle foncé h de lumière/12 12 h. Après indiqué temps les plantes ont été récoltées dans les conteneurs. Les racines ont été lavés et sonication dans un sonicateur bain pour éliminer les bactéries liés. Nombre de bactéries ont été déterminées à l’aide du nombre de cellules viables. La figure 2 montre l’effet de deux mutations différentes de cellulose-moins sur la capacité des bactéries à coloniser les racines de la tomate. Bien que les écarts de quelques mesures ont été aussi hauts que 0,9 log10 (un problème courant avec ce type de mesure) la réduction obligatoire des mutants cellulose-minus est évidente et nous pouvons conclure que la production bactérienne de cellulose favorise les bactéries la colonisation des racines de la tomate.
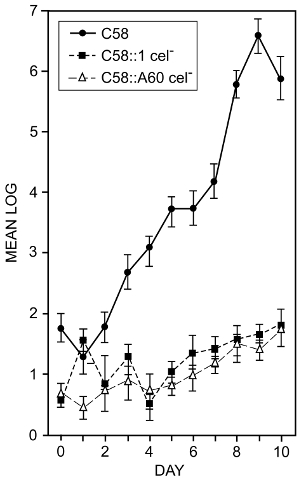
Figure 2 : Racine de colonisation de type sauvage et des mutants de cellulose-moins d’a. tumefaciens. Log10 nombre total de bactéries par cm de longueur racine extraite des racines de tomates inoculés avec la souche de type sauvage a. tumefaciens C58 et mutants de cellulose-moins C58:1 et C58:A60. Les chiffres indiqués sont les moyens d’un minimum de quatre expériences distinctes. Les barres indiquent des écarts-types des moyens. Racines ont été inoculés par leur immersion dans une suspension de 105 bactéries / mL pour un min. Les plantes ont été cultivées dans des récipients et la bactérie faiblement adhérente est enlevée par lavage des racines dans le tampon non contrôlé dans un flacon en verre de lavage. Bactéries étroitement adhérentes ont été supprimés en utilisant un sonicateur de bain et de la suspension qui en résulte plaqué pour déterminer la numération de cellules viables16. De Matthysse et McMahan, ce chiffre a été modifié. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
A examiné le rôle des exopolysaccharides dans la liaison d’e. coli et autres bactéries aux germes de luzerne. Certains foyers de maladie diarrhéique due à e. coli O157 : H7 ont été attribuées aux germes de luzerne contaminés. Liaison des bactéries de type sauvage et des mutants incapables de faire diverses exopolysaccharides a été mesurée en utilisant les méthodes décrites dans les étapes 1.1, 5.1 et 7.2. Germes de luzerne ont été surface stérilisé et mises à germer pendant une journée dans l’eau stérile à 25 ° C dans l’obscurité. Quatre choux avec des téguments ci-joint ont été placés dans des plats en plastique stériles contenant 5 mL d’eau. Bactéries cultivées dans un bouillon Luria ont été ajoutés à une concentration finale d’environ 5 x 103 / mL. Les germes inoculés ont été incubées à 25 ° C dans l’obscurité pendant 3 jours. Les pousses ont été lavés deux fois dans 5 mL d’eau dans un flacon stérile par inversion vigoureuse et homogénéisés dans le tampon à l’aide d’un homogénéisateur de verre téflon moteur de lavage. Des expériences antérieures à l’aide de bactéries marquées avec un plasmide porteur du gène de la GFP ont montré aucune bactérie intériorisée, même si les bactéries de surface ont été observées facilement. Les résultats figurent dans le tableau 1. Deux souches d’e. coli O157 : H7 ont été examinés. Dans les deux souches de la production de poly-β-1, 6-glucuronique acid(PGA) semblait contribuent le plus à la liaison du pathogène e. coli de planter des surfaces. L’acide colique a également joué un rôle significatif dans la liaison. Alors que la réduction obligatoire chez les mutants de cellulose-minus était significative, il n’était pas aussi importante que celle pour les deux autres polysaccharides.
| Effets des Mutations dans les gènes de la Production d’exopolysaccharides sur reliure d’e. coli O157 : H7 aux choux et ouvert des téguments | |||
| Souche bactérienne | Mutation ou génotype (phénotype pertinente) | Numéro10 journal de bactéries lié par sprout ou tégument | |
| Luzerneb | Téguments ouverts | ||
| 86-24 | Aucun (type sauvage) | 4,7 ± 0,6 | 5,6 ± 0,2 |
| 8624N | yhjN (cellulose-minus) | de 2,9 ± 0,7c | 3,5 ± 0,6c |
| C 8624 | wcaD (colique acide-minus) | 1.8 ± 0,7c | 2,4 ± 0,5c |
| 8624P | ACGP (PGA-minus) | < 1,0c | 1,0 ± 1,0c |
| DEC4A | Aucun (type sauvage) | 5,6 ± 0,2 | 6.1 ± 0,3 |
| DEC4AN | yhjN (cellulose-minus) | 4,8 ± 0,8d | 4,1 ± 0,8d |
| DEC4AC | wcaD (colique acide-minus) | 3,9 ± 0,5c | 4,8 ± 0,8d |
| DEC4AP | ACGP (PGA-minus) | < 1,0c | 1,2 ± 0,7c |
| un moyenne ± écart-type d’un minimum de trois mesures du nombre de bactéries (log10) lié après 3 jours. | |||
| Choux de b ont été lavés avant la mesure. | |||
| c significativement différente de la souche sauvage : P < 0,01. | |||
| d significativement différente de la souche sauvage : P < 0,05. | |||
| Cette table a été modifiée de Matthysse, Desclaux, Mishra et Torres10. | |||
Tableau 1 : effets des mutations dans les gènes de la production d’exopolysaccharides sur fixation de E. coli O157 : H7 pour les choux. Afin de déterminer le rôle des divers exopolysaccharides et lipopolysaccharide dans la liaison du pathogène souches d’e. coli O157 : H7 aux germes de luzerne, la liaison d’une série de mutants pour les choux et les téguments ouverts a été étudiée en utilisant les méthodes décrit à l’étape 6. Les résultats ont montré que poly-ß-1, 6 -N-acétyl-D-glucosamine (PGA) semble être essentiel pour la liaison aux germes et que la cellulose et colanique l’acide sont nécessaires pour la liaison maximale d’e. coli O157. Cette table a été modifiée de Matthysse, Desclaux, Mishra et Torres10.
Afin de déterminer si la production de PGA est suffisante pour provoquer la liaison bactérienne de planter des surfaces, un plasmide (pMM11) portant l’opéron cloné codant les gènes nécessaires à la PGA, la production a été introduite dans deux bactéries différentes qui ne seraient pas normalement être capable de se lier à la tomate racines10. A. tumefaciens A1045 est un mutant de la souche de type sauvage C58 qui omet de faire 1,2 cyclique-β glucanes et aussi ne peut pas lier de planter des surfaces29. Sinorhizobium meliloti 1021 formant des nodules racinaires sur luzerne ne parvient pas à lier les non-légumineuses dont12de la tomate. Les techniques décrites dans les étapes 1.1, 5.1, 6,3 et 7,1 servaient à déterminer si la capacité de faire PGA généralement augmentée fixation bactérienne à la surface des racines. Graines de tomate étaient en surface stérilisé et germent dans l’eau stérile. Racines ont été coupés en segments de 1 cm de longueur et placés dans l’eau stérile et les bactéries ont été inoculés. Comme ces deux espèces de bactéries croissent à des taux différents, contraignant a été mesurée à des moments différents pour permettre à peu près égales de la croissance bactérienne. La présence de le pMM11 de plasmide entraîne une augmentation significative similaire dans le nombre de bactéries dépendant de ces deux espèces (tableau 2)10. Une augmentation significative de la liaison a été également vu au microscope, mais la liaison était très différente chez les deux espèces (Figure 3). A. tumefaciens A1045 lié comme des bactéries à la surface de la racine. S. meliloti lié en larges bouquets où seulement quelques uns des bactéries ont été liés directement à la racine et la majorité des bactéries était attachée à d’autres bactéries. Cet exemple montre que l’analyse simplement le nombre de bactéries liés sans inclure l’observation au microscope peut donner une impression trompeuse des résultats d’une expérience. Bien que les deux méthodes (nombre de cellules viables et observation au microscope) montrent que pMM11 a augmenté fixation bactérienne aux racines de la tomate, le type de liaison causée par la production de PGA était différent pour les deux espèces bactériennes10.
| L’effet de la pMM11 de plasmide sur la liaison des bactéries aux racines de la tomate | ||
| Souche bactérienne | Plasmide | Nombre de bactéries lié par la longueur des racines mm |
| A. tumefaciens A1045un | aucun | 0,25 x 103 ± 0,25 x 103 |
| pBBR1mcs (vector) | 0,25 x 103 ± 0,25 x 103 | |
| pMM11 (synthèse de la PGA) | 10 x 103 ± 0,25 x 103 | |
| S. meliloti 1021b | aucun | aucun détecté |
| pBBR1mcs (vector) | aucun détecté | |
| pMM11 (synthèse de la PGA) | 50 x 103 ± 5 x 103 | |
| une liaison bactérienne a été mesurée après 2 heures | ||
| b fixation bactérienne était mesure après 18 heures | ||
| Cette table a été modifiée de Matthysse, Desclaux, Mishra et Torres10. | ||
Tableau 2 : l’effet d’un plasmide porteur de gènes pour la synthèse de PGA sur la liaison de A. tumefaciens A1045 et S. meliloti 1021 à sections de racines de tomates. Afin d’examiner la possibilité de poly-ß-1, 6 -N-acétyl-D-glucosamine (PGA) pour favoriser la fixation de bactéries pour planter des racines, l’effet d’un plasmide qui confère la capacité de faire PGA (pMM11) sur la liaison de deux souches de bactéries associées aux plantes à racines de tomates a été examinée. Aucune souche de la bactérie ont montré une liaison significative aux racines de la tomate en l’absence du plasmide ou en présence de plasmide sans les gènes codant la synthèse PGA (pBBR1mcs). L’ajout du plasmide porteur de gènes de synthèse de PGA a augmenté la fixation par les deux types de bactéries. Parce qu’a. tumefaciens se développe plus vite que la liaison de S. meliloti a été mesurée après 2 h d’incubation pour a. tumefaciens et après 18 h pour S. meliloti. Les techniques utilisées sont celles qui sont indiquées à l’étape 7. Cette table a été modifiée de Matthysse, Desclaux, Mishra et Torres10.
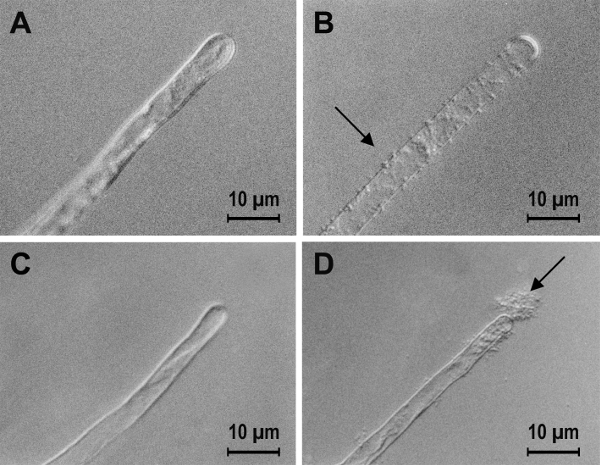
Figure 3 : L’effet de la pMM11 du plasmide portant les gènes de biosynthèse de PGA sur la liaison d’a. tumefaciens A1045 et S. meliloti 1021 aux poils absorbants tomate. Liaison aux poils absorbants tomate de A) a. tumefaciens A1045, B) a. tumefaciens A1045 pMM11, C) S. meliloti 1021 et D) S. meliloti 1021 pMM11. Bien que l’augmentation dans la liaison des deux espèces bactériennes est à peu près similaire, les détails de la liaison comme vu au microscope sont très différentes. Les techniques utilisées sont celles qui sont indiquées à l’étape 6. Ce chiffre a été modifié par Matthysse, Desclaux, Mishra et Torres10. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Il est parfois possible d’utiliser la liaison à surfaces non biologiques pour aider à distinguer l’apport de bactéries et de la plante dans une interaction spécifique. Le polysaccharide(UPP) unipolaire d’a. tumefaciens a démontré pouvoir de médiation bactérienne contraignant à une variété de ces deux surfaces biologiques et non biologiques30. Calcium a été observée à inhiber la liaison de a. tumefaciens de planter des surfaces médiées par l' UPP28. Afin de déterminer si l’inhibition par les ions calcium de fixation bactérienne de planter des surfaces est due à un effet sur les bactéries ou sur la surface de la plante, la fixation des bactéries sur les filets en nylon a été examinée. Les techniques décrit à l’étape 8.2 ont été utilisés. Graines de tomate étaient en surface stérilisé et germent dans l’eau tel que décrit à l’étape 1. Les bactéries ont été cultivés sur un milieu minimal avec saccharose et ajoutés aux racines de tomates ou de fils de nylon à une concentration finale d’environ 105/ml comme indiqué au point 5.1. A examiné l’effet de l’ACIC ajouté2 sur fixation bactérienne aux racines de tomates et de fils de nylon au microscope. La figure 4 montre une inhibition similaire de liaison par le calcium à l’aide soit surface ce qui suggère que l’effet du calcium est principalement sur les bactéries10.

Figure 4 : L’effet du calcium sur la liaison d’a. tumefaciens à poils absorbants tomate et fils nylon. A. tumefaciens est incubé avec les racines de la tomate (A et B) ou de fils de nylon (C et D) pendant 24 h dans une 01:10 dilution des sels de MS et une 01:20 dilution du milieu minimal AB, 0,4 % de sucrose (A et C) ou dans un 01:10 dilution des sels MS et 01:20 dilution du milieu minimal AB , 0,4 % de sucrose contenant 60 mM CaCl2 (B et D)31. L’ajout CaCl2 inhibent la liaison bactérienne à la fois des racines et des filets en nylon, ce qui suggère que l’inhibition est principalement due à un effet sur les bactéries plutôt que sur la surface de la plante. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Il est important d’être conscient de toutes les surfaces à laquelle les bactéries peuvent adhérer pendant l’expérience. Ainsi les bactéries qui sont capables de se lier au verre peuvent être sous-estimée si le nombre de cellules viables est effectué à l’aide de pipettes et tubes de verre. Si les plantes sont cultivées dans la gélose ou sol et certains de la gélose ou de saleté sur les plantes, puis les bactéries peuvent lier à la matière adhérente, plutôt qu’aux plantes. D’autre part, laver la surface de la plante, en particulier dans le cas des racines, peut enlever les revêtements de surface naturelles tels que muqueux et altèrent ainsi les résultats des tests d’adhérence.
Il est important de s’assurer que les bactéries ajoutées au mélange d’incubation restent en vie pendant l’expérience. Ainsi du nombre de cellules viables de bactéries libres ainsi que joints devrait systématiquement effectué. Certains traitements ou mutations bactériennes peuvent réduire le taux de croissance bactérienne ou effectivement causer la mort d’une fraction de la population bactérienne. Bactéries vivantes et mortes ne soient pas reconnaissables au microscope, à moins que les colorations spéciales sont utilisées. Il y a un kit de tache utile pour vivre/morts bactéries qui dépend de l’exclusion des colorants des bactéries vivantes. Toutefois, si une population mixte d’espèces bactériennes est présente du nombre de cellules viables des espèces d’intérêt est susceptible d’être la méthode la plus simple pour déterminer si l’incubation a entraîné la mort bactérienne.
Composition du milieu influeront sur la croissance et la survie bactérienne. Exsudat racinaire et matériaux libérés de la plaie et couper les sites fourniront substrat pour soutenir la croissance bactérienne modeste. Phosphates, azote et le fer ont tendance à se limiter dans ces conditions. Des cations divalents comme le calcium et le magnésium peuvent influencer l’adhérence. Dans certains cas, source de carbone peut influencer adhérence. pH est également important. En général, le pH de la rhizosphère est entre 5,5 et 6,5.
Il est nécessaire d’être prudent en utilisant marquées avec la résistance aux antibiotique des bactéries. Antibiotiques les plus souvent utilisées sont la rifampicine et à l’acide nalidixique. La résistance à ces antibiotiques est généralement due à des mutations dans des gènes chromosomiques (ARN polymérase et gyrase, respectivement) et donc ne peut facilement être transférée à une autre souche durant l’incubation. Ce type de résistance aussi n’entraîne pas la dégradation ou la modification de l’antibiotique. Marquage des bactéries avec un plasmide gène marqueur n’est pas recommandée à moins que le plasmide peut s’appliquer à toutes les autres bactéries. La résistance aux antibiotique ne doit pas être dû à une modification chimique ou de dégradation de l’antibiotique, comme des bactéries sensibles aux antibiotiques sera alors capables de croître sur plaques d’antibiotiques si elles sont situées à proximité de la bactérie résistante.
Les méthodes décrites dans cet article sont utiles pour la petite taille des échantillons et/ou des expériences où les échantillons doivent être contenus (par exemple, dans les expériences impliquant des agents pathogènes humains). Pour les grands échantillons (au-dessus de 100 g de matériau ou de plus de 50 plants) autres méthodes ou modification drastique de ces méthodes seraient nécessaires10,19,32,19,33, 34 , 35 , 36. la présence d’un grand nombre de micro-organismes autres que les espèces étudiées peut également poser des problèmes importants. Les solutions possibles comprennent l’utilisation de bactéries marquée d’une protéine fluorescente ou la résistance aux antibiotique comme décrit aux étapes 6.1.2 et 7.3. Toutefois, lorsque les bactéries d’intérêt sont rares individus dans une population importante d’autres micro-organismes ces marqueurs ne peut-être pas suffisantes pour permettre une évaluation claire du nombre de bactéries étudiées.
Toutes les méthodes décrites ici sont des méthodes de laboratoire. Des modifications seraient nécessaires pour les études à effet de serre. Des modifications plus importantes sont susceptibles d’être requis pour les études sur le terrain où les protozoaires, insectes et autre animale variation de la prédation et le climat compliquent la fourniture de conditions définies pour les expériences. À l’avenir ces méthodes peuvent être prolongés pour inclure les interactions entre deux ou plusieurs microorganismes sur la surface de la plante.
Déclarations de divulgation
L’auteur déclare qu’elle n’a aucun intérêts financiers concurrents.
Remerciements
L’auteur remercie Susan Whitfield pour la préparation des figures et Camille Martin et Hillary Samagaio pour assistance avec quelques-unes des expériences.
matériels
| Name | Company | Catalog Number | Comments |
| light microscope | any | N/A | phase contrast or Nomarski optics are helpful, for studies using fluorescent markers a fluorescence microscope is required. |
| seeds | any | N/A | make a note of the seed lot number and the cultivar |
| bleach | any | N/A | |
| bath sonicator | any scientific supply company | N/A | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| nutrient agar | Difco | 001-01-8 | |
| soytone | Difco | 24360 | |
| Sand | sea sand Fisher Scientific | S25 | |
| sand | Sigma-Aldrich | S9887 | |
| conetainers | Stuewe & Sons, Inc. | Ray-Leach cone-tainers | many different sizes are available to suit the type of plant you wish to grow |
| parafilm | any scientific supply company | N/A | |
| MS salts | Sigma-Aldrich | M5524 | |
| parrafin | any | N/A | |
| centrigfuge | any scientific company | N/A | to pellet most bacteri only a small centrifuge with a max force of 10,000 XG is needed |
| vortex mixer | any scientific company | ||
| micrometer | ACCU-SCOPE Accessories | A3145 | |
| Sedgwick-rafter counting cell | Hauser Scientific | HS3800 | |
| probe-clip press-seal incubatin chamber | Sigma-Aldrich | Z359483 | |
| rifampicin | Sigma-Aldrich | R3501 | |
| naladixic acid | Sigma-Aldrich | n8878 | |
| Live/dead stain | In Vitrogen Molecular Bioprobes | L7007 |
Références
- Heaton, J. C., Jones, K. Microbial contamination of fruit and vegetables and the behaviour of enteropathogens in the phyllosphere: a review. J. Appl. Microbiol. 104, 613-626 (2008).
- Beuchat, L. R. Ecological factors influencing survival and growth of human pathogens on raw fruit and vegetables. Microbes Infect. 4, 413-423 (2002).
- Berger, C. N., et al. Fresh fruit and vegetables as vehicles for the transmission of human pathogens. Environ. Microbiol. 12, 2385-2397 (2010).
- Lugtenberg, B. J., Chin, A. W. T., Bloemberg, G. V. Microbe-plant interactions: principles and mechanisms. Antonie Van Leeuwenhoek. 81, 373-383 (2002).
- Preston, G. M., Haubold, B., Rainey, P. B. Bacterial genomics and adaptation to life on plants: implications for the evolution of pathogenicity and symbiosis. Curr. Opin. Microbiol. 1, 589-597 (1998).
- Compant, S., Duffy, B., Nowak, J., Clement, C., Barka, E. A. Use of Plant Growth-Promoting Bacteria for Biocontrol of Plant Diseases: Principles, Mechanisms of Action, and Future Prospects. Appl. Environ. Microbiol. 71, 4951-4959 (2005).
- Mathews, S. L., Smith, R. B., Matthysse, A. G. A comparison of the retention of pathogenic Escherichia coli. O157 by sprouts, leaves and fruits. Microb. Biotechnol. 7, 570-579 (2014).
- Fuqua, C., Matthysse, A. G. Methods for studying bacterial biofilms associated with plants. Methods Enzymol. 337, 3-18 (2001).
- Torres, A. G., Jeter, C., Langley, W., Matthysse, A. G. Differential binding of Escherchia coli.O157:H7 to alfalfa, human epithelial cells, and plastic is mediated by a variety of surface structures. Appl. Environ. Microbiol. 71, 8008-8015 (2005).
- Matthysse, A. G., Deora, R., Mishra, M., Torres, A. G. Polysaccharides cellulose, poly-beta-1,6-n-acetyl-D-glucosamine, and colanic acid are required for optimal binding of Escherichia coli O157:H7 strains to alfalfa sprouts and K-12 strains to plastic but not for binding to epithelial cells. Appl. Environ. Microbiol. 74, 2384-2390 (2008).
- Matthysse, A. G., et al. The effect of cellulose overproduction on binding and biofilm formation on roots by Agrobacterium tumefaciens. Mol. Plant Microbe Interact. 18, 1002-1010 (2005).
- Matthysse, A. G., Kijne, J. W., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. Attachment of Rhizobiaceae to plant cells. The Rhizobiaceae. , 235-249 (1998).
- Matthysse, A. G. Conditioned medium promotes the attachment of Agrobacterium tumefaciens.strain NT1 to carrot cells. Protoplasma. 183, 131-136 (1994).
- Matthysse, A. G., McMahan, S. The effect of the Agrobacterium tumefaciens. attR mutation on attachment and root colonization differs between legumes and other dicots. Appl. Environ. Microbiol. 67, 1070-1075 (2001).
- Jeter, C., Matthysse, A. G. Characterization of the binding of diarrheagenic strains of E. coli.to plant surfaces and the role of curli in the interaction of the bacteria with alfalfa sprouts. Mol. Plant Microbe Interact. 18, 1235-1242 (2005).
- Matthysse, A. G., McMahan, S. Root colonization by Agrobacterium tumefaciens is reduced in cel, attB, attD, and attR mutants. Appl. Environ. Microbiol. 64, 2341-2345 (1998).
- Morton, E. R., Fuqua, C. Phenotypic analyses of Agrobacterium. Curr. Protoc. Microbiol. , (2012).
- Brandl, M. T. Plant lesions promote the rapid multiplication of Escherichia coli O157:H7 on postharvest lettuce. Appl. Environ. Microbiol. 74, 5285-5289 (2008).
- Franz, E., et al. Quantification of contamination of lettuce by GFP-expressing Escherichia coli O157:H7 and Salmonella enterica serovar Typhimurium. Food Microbiol. 24, 106-112 (2007).
- Morton, E. R., Fuqua, C. Laboratory maintenance of Agrobacterium. Curr. Protoc. Microbiol. , (2012).
- Lugtenberg, B. J., Kravchenko, L. V., Simons, M. Tomato seed and root exudate sugars: composition, utilization by Pseudomonas biocontrol strains and role in rhizosphere colonization. Environ. Microbiol. 1, 439-446 (1999).
- Murashige, T., Skoog, F. A Revised Medium for Rapid Growth and Bio Assays with Tobacco Tissue Cultures. Physiologia Plantarum. 15, 473-497 (1962).
- Danhorn, T., Fuqua, C. Biofilm formation by plant-associated bacteria. Annu. Rev. Microbiol. 61, 401-422 (2007).
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- O'Toole, G., Kaplan, H. B., Kolter, R. Biofilm formation as microbial development. Annu. Rev. Microbiol. 54, 49-79 (2000).
- Charkowski, A. O., Barak, J. D., Sarreal, C. Z., Mandrell, R. E. Differences in growth of Salmonella enterica and Escherichia coli O157:H7 on alfalfa sprouts. Appl. Environ. Microbiol. 68, 3114-3120 (2002).
- Loper, J., Suslow, T., Schroth, M. Lognormal distribution of bacterial populations in the rhizosphere. Phytopathology. 74, 1454-1460 (1984).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front Plant Sci. 5, 252 (2014).
- Douglas, C. J., Halperin, W., Nester, E. W. Agrobacterium tumefaciens mutants affected in attachment to plant cells. J. Bacteriol. 152, 1265-1275 (1982).
- Tomlinson, A. D., Fuqua, C. Mechanisms and regulation of polar surface attachment in Agrobacterium tumefaciens. Curr. Opin. Microbiol. 12, 708-714 (2009).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front Plant Sci. 5, (2014).
- Wright, K. M., et al. The endophytic lifestyle of Escherichia coli O157:H7: quantification and internal localization in roots. Phytopathology. 103, 333-340 (2013).
- Berger, C. N., et al. Fresh fruit and vegetables as vehicles for the transmission of human pathogens. Environ. Microbiol. 12, 2385-2397 (2010).
- Erickson, M. C., et al. Internalization and Fate of Escherichia coli O157:H7 in Leafy Green Phyllosphere Tissue Using Various Spray Conditions. J. Food Prot. 77, 713-721 (2014).
- McKellar, R. C., et al. Evaluation of different approaches for modeling Escherichia coli O157:H7 survival on field lettuce. Int. J. Food Microbiol. 184, 75-85 (2014).
- Wu, F. M., et al. Factors influencing the detection and enumeration of Escherichia coli O157:H7 on alfalfa seeds. Int. J. Food Microbiol. 71, 93-99 (2001).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon