Method Article
Aderência de bactérias para plantar superfícies medidas em laboratório
Neste Artigo
Resumo
Um método fácil para medir e caracterizar adesão bacteriana em plantas, principalmente raízes e brotos, é descrito neste artigo.
Resumo
Este manuscrito descreve um método para medir a ligação bacteriana às superfícies da planta axénica no microscópio de luz e através do uso de contagem de células viáveis. Materiais vegetais usados incluem raízes, brotos, folhas e cortar frutas. Os métodos descritos são de baixo custo, fácil e adequado para amostras pequenas. Vinculação é medida em laboratório e uma variedade de condições e meios de incubação pode ser usada. O efeito de inibidores pode ser determinado. Situações que promovem e inibem a ligação também podem ser avaliadas. Em alguns casos é possível distinguir-se várias condições alteram ligação principalmente devido aos seus efeitos na planta ou sobre as bactérias.
Introdução
A medição de vinculação bacteriana para plantar superfícies tornou-se importante em três situações diferentes. A primeira situação é o exame da transmissão de agentes patogénicos humanos na planta superfícies1,2,3. O objetivo aqui é para impedir a ligação de bactérias ou para remover ou matar as bactérias vinculadas e, portanto, para reduzir a transmissão da doença pelo material de planta. A segunda situação é o exame da ligação de patógenos vegetais para plantar superfícies4. Mais uma vez, o objetivo aqui é para impedir a ligação ou para remover ou matar as bactérias vinculadas e, portanto, para reduzir a doença. A terceira situação é o exame da ligação de bactérias simbióticas ou planta-growth-promoting5,6. O objetivo aqui é promover bacteriano vinculativo e, assim, para aumento de fitossanidade e cultura produz.
As técnicas para medir a ligação bacteriana para plantar superfícies descritas neste artigo são baratos e relativamente fáceis de realizar. Os únicos requisitos são um microscópio e materiais encontrados geralmente em um laboratório de bacteriologia. Para algumas técnicas um sonicador de banho é útil. As técnicas descritas são projetadas para experiências realizadas usando tamanhos de amostra relativamente pequeno de vinculação. Medições de vinculação são feitas em laboratório, embora seja possível modificar algumas dessas técnicas para o uso na estufa ou no campo.
Estas técnicas têm sido utilizadas para medir a ligação bacteriana de raízes, brotos, folhas cortadas, frutas cortadas e tomatinhos intactos na laboratório7,8,9,10,11, 12,13,14,15. Tem também sido costumavam medir a colonização de raiz de plantas que crescem no solo ou areia no laboratório16. As técnicas têm sido utilizadas com muitas espécies bacterianas, incluindo Agrobacterium tumefaciens, tem Sinorhizobium, Escherichia coli, Salmonella typhi e Pseudomonas fluorescens. Uma descrição útil de métodos para avaliar a interação da . tumefaciens com superfícies pode ser encontrada em Morton e Fuqua (2012)17. Em todos os casos as dimensões das amostras envolvidas eram pequenas, geralmente menos do que 25-50 plantas. As técnicas descritas são adequadas para uso com agentes patogénicos humanos que precisam ser mantidos contido durante os experimentos.
Protocolo
1. preparação de Material vegetal axénica
- Prepare mudas cultivadas em água.
- Coloque um pequeno número de sementes (menos de 30) em um 30, 50, 100 ou copo de vidro de 150 mL. Usamos tomate, alfafa, Arabidopsis thaliana, ervilha, feijão, tabaco, alface e sementes de cenoura com este protocolo.
Nota: Para determinar quantas sementes podem ser esterilizadas juntos sem contaminação propagação da semente de um para outro, consulte a etapa 1.1.2. - Cubra as sementes com 80% de etanol e agite brevemente. -As sementes de molho durante 1 min.
- Despeje o etanol e cobrir as sementes com uma solução de 50% por alvejante comercial de volume (NaClO) e 0,1% Triton X-100 na água da torneira. -As sementes de molho por 20 min.
Nota: Se as sementes são grandes, tais como sementes de feijão, pode ser necessário aumentar o tempo de imersão completamente matar os fungos na superfície da semente. - Despeje a mistura de água sanitária e lavar as sementes 3 vezes com água estéril. -As sementes de molho na água por 1 min para cada lavagem.
- Para obter mudas axénica adicionar uma pequena quantidade de água estéril entre 5 e 25 mL dependendo do tamanho das sementes. Despeje a água e as sementes em uma placa de Petri estéril para germinação de sementes.
Nota: A água deve cobrir o fundo do prato, mas não cubra as sementes para incentivar a formação de pelos radiculares. Axénica descreve uma cultura em que somente uma única espécie de organismo está presente. - Incube no escuro até as mudas atinjam o tamanho desejado, entre 1 cm e 10 cm (cerca de 5 dias para o tomate e a. thaliana e 1 a 3 dias para alfafa). Para sementes maiores use um recipiente estéril e coberto com uma capacidade de mais de 100 mL, tais como um prato de vidro coberto com papel alumínio.
- Coloque um pequeno número de sementes (menos de 30) em um 30, 50, 100 ou copo de vidro de 150 mL. Usamos tomate, alfafa, Arabidopsis thaliana, ervilha, feijão, tabaco, alface e sementes de cenoura com este protocolo.
- Determine a frequência de contaminação de um lote de sementes particular com microorganismos que permanecem viáveis ou dentro as sementes após a esterilização de superfície.
Nota: Isto é necessário porque sementes ocasionais carregam microorganismos sob o revestimento de semente que não pode ser morto por superfície-esterilização. Use a frequência de sementes contaminadas em um lote de sementes para determinar quantas sementes para esterilizar ao mesmo tempo sem um risco elevado do grupo de sementes, tornando-se contaminada devido a uma semente com bactérias ou fungos que sobrevivem ao tratamento.- Realize etapas 1.1.1 através de 1.1.3.
- Coloque as sementes sobre um prato de Petri ágar. Colocar sementes de 10-30 em cada prato, espaçando-as para fora, para que eles podem ser marcados individualmente. Sele os pratos com fita ou selagem da película.
- Incube durante 3-5 dias a 25 ° C, marcando cada dia por consequência visível de microorganismos das sementes.
- Prepare mudas cultivadas em areia. Mudas podem ser cultivadas em areia para uso em experimentos de adesão bacteriana.
- Realize etapas 1.1.1 através de 1.1.3.
- Esterilize a areia de quartzo ou mar na autoclave. Ambos contêm algum material orgânico. Se isso afetará o experimento, lave a areia cobrindo-o com duas vezes o seu volume de 0.1 M HCl. Mix de 10 min. permitir a areia para resolver e decantar o líquido. Lave a areia 3 vezes com água, e uma vez com 80% etanol seguido de duas lavagens de água adicional usando o mesmo protocolo quanto a 0.1 M HCl. esterilize em autoclave, a areia lavada.
- Esterilizar os recipientes submergindo-los em água sanitária 50% por 5 min e enxague 5 vezes com água esterilizada por submersão. Permita-lhes secar e selar o fundo com película de vedação. O filme como obtidas do fabricante é geralmente livre de microorganismos no interior lado que deve ser colocado virado para o interior do recipiente.
- Misture a areia estéril com água estéril suficiente para molhar o suficiente para que as varas de areia. A quantidade dependerá como seco é de areia. Geralmente, 10-35% do volume de areia é suficiente. Coloque a areia molhada no recipiente permitindo profundidade suficiente para o comprimento das raízes necessário e suficiente espaço acima da areia para o crescimento das filmagens. As distâncias exactas dependem da espécie e variedade da planta a ser usada.
- Plantar sementes.
- Faça um buraco raso na areia com uma vareta de vidro estéril profunda o suficiente para colocar a semente na superfície da areia (aproximadamente 1-5 mm). Coloque a semente no buraco. Cubra com uma camada fina de areia. Sele o topo do recipiente com filme para evitar a perda de água e a entrada de micróbios adicionais de vedação.
- Crescem as plantas no laboratório sob uma luz ou na estufa a uma temperatura adequada e a duração do dia para a espécie e variedade da planta. Para o tomate, alfafa ou Arabidopsis thaliana use temperatura e ciclos de luz/escuro de 12 h.
- Plantio de mudas em um buraco na areia (1-2mm de diâmetro e profundidade suficiente que a raiz será coberta com areia). Faça o furo com uma vareta de vidro estéril. Guia a raiz cuidadosamente no orifício usando um estéril em aço inoxidável de crochê se necessário e enchem os lados do buraco de areia.
- Alternativamente, cultivar plantas axenically ou em ágar contendo uma mistura de sais, tais como sais de MS. Use tiros de axénica plantas cultivadas em ágar diretamente. Evite as raízes crescidas com ágar como ágar varas para tanto a superfície da planta e as bactérias. Isso pode resultar em uma falsa impressão de adesão bacteriana à superfície da planta.
2. preparação de outro Material de planta
- Lave o material de planta cultivada em estufa com água antes do uso para reduzir o número de micróbios indígenas. Vai haver bactérias e fungos nas plantas. Protozoários, estará presentes no solo, as raízes e, possivelmente nas folhas. Lavar com água sanitária ou etanol irá alterar a superfície da planta e não é recomendado.
- Se após a lavagem da água, um número significativo de micróbios permanece, trata as plantas com sabonete líquido diluído ou diluir o peróxido de hidrogênio (0,01%) para remover ou matar os micróbios. Isto é geralmente menos prejudicial para a planta do que água sanitária ou álcool etílico.
- Como planta material comprado em um mercado local é difícil descontaminar, escolha o material que parece não ter sido objecto de armazenamento longo. Evite partes do material que aparecem marrom ou danificados.
- Lave com água e fazer cortes frescos nas extremidades do material que anteriormente foram cortadas antes de usá-lo, a menos que as interações das bactérias teste com as bactérias (e eucariontes) que irão ter acumulado em locais do corte são de interesse. Não lave com água sanitária ou álcool etílico como eles irão alterar a superfície da planta.
- Trate a ferida de sites. Sites de ferida frequentemente fornecem bactérias acesso ao interior da planta material18.
- Bloquear ou reduzir bacteriano anexo a e movimento através de, ferida ou cortadas sites criados na preparação do material, mergulhando a borda cortada em parafina derretida ou pintando o site com parafina derretida, usando uma escova pequena. Não use uma espátula ou qualquer instrumento afiado que isso poderá danificar o tecido.
- Vedação da haste cicatrizes em frutos tais como tomates com parafina. Algumas bactérias vão nadar para a área sob a parafina, mas seus números são geralmente pequenos.
- Se a entrada bacteriana em sites de ferida é de interesse, estimativa da distância que as bactérias podem mover transportada por fluxos de água ou difusão, observando o movimento de um corante adicionado à solução. Marcar motile bactérias através da introdução de um gene que codifica uma proteína fluorescente como proteína verde fluorescente (GFP)-los e localizá-los usando microscopia de fluorescência, descrita no passo 6.1.219.
3. preparação das bactérias
- Crescem as bactérias. Utilização de um meio que mais se aproxima das condições, as bactérias são susceptíveis de ter sido exposto a imediatamente antes de encontrar a planta no mundo real, fora do laboratório. Carbono e nitrogênio fontes bem como a presença de íons, cátions divalentes particularmente (Ca, Mg, Fe, Mn e Zn) e fosfato e o pH do meio são importantes.
- Use o meio mínimo AB ou caldo Luria para a. tumefaciens e outras bactérias de solo20. Para Escherichia coli, que pode ter origem no intestino, use caldo de Luria.
- Adicione indutores como exsudados radiculares ou extratos de plantas comerciais tais como soytone ou açúcares como a sacarose ou xilose o médio21. Se essas substâncias são usadas como indutores, adicione uma baixa concentração, por exemplo, 0,01%. Se eles são fontes de carbono usado, adicione uma maior concentração, por exemplo, 0,1%.
- Prepare o inóculo bacteriano. Dilua as bactérias em água estéril ou no meio em que a incubação será realizado e adicioná-los para o material de planta. A diluição adequada é discutida em passos 4.2 e 4.3.
- Para remover a mídia de crescimento antes de utilizar as bactérias, centrifugar a suspensão bacteriana 10.000 x g por 2 min, decantar o sobrenadante e ressuspender as bactérias vortexing-los na mesma mídia que será utilizada para a incubação com o material de planta. Esse método pode remover ou diminuir o número ou a quantidade de material extracelular e apêndices como exopolissacarídeo, cápsulas, flagelos, e/ou pili. Se é importante que essas estruturas de superfície permanecem intactas, então simplesmente diluir as bactérias antes de inoculá-los ou usar o método alternativo descrito no passo 3.2.2.
- Como um método alternativo para remover o meio de crescimento, recolha as bactérias em um filtro de nitrocelulose ou policarbonato com um tamanho de poro de 0,2 µm ou menos. Lavar as bactérias com o meio de incubação e ressuspendê-los por agitação suave ou num Vortex do filtro em um recipiente estéril de médio porte.
4. a inoculação das bactérias
- Determine o número de bactérias para ser inoculadas com referência a medição deve ser usado para determinar a adesão bacteriana e a duração da incubação.
- Para estudos microscópicos envolvendo incubação vezes menos de 1 dia inocular relativamente grande número de bactérias. Adicione uma quantidade de cultura para atingir uma concentração bacteriana final de mais de 106 bactérias / mL. Para os tempos de incubação mais diminuir o tamanho do inóculo bacteriano.
- Para estudos em que adesão bacteriana será medido pela contagem de células viáveis, adicione uma quantidade de cultura para atingir uma concentração bacteriana final de 103 106 bactérias / mL.
- Evite adicionar tantas bactérias que seu metabolismo muda a concentração de pH ou oxigênio no meio de incubação. Medir o pH com papel de pH ou um eletrodo. Medir a concentração de oxigênio, utilizando um eletrodo de oxigênio.
- Para plantas cultivadas em areia, inocule em três maneiras possíveis.
- Inocule as sementes com a bactéria antes do plantio por semente por 1 min de imersão em suspensão em água de cerca de 106 bactérias / mL.
- Inocule a raiz germinando axénica mudas conforme descrito na seção 1 e, quando a raiz de plântulas é mergulhando-o cerca de 1 cm de comprimento ou colocando o seedling todo em uma suspensão de 106 bactérias / mL na água por 1 min.
- Inocule a areia misturando as bactérias com a areia antes de plantar para dar uma concentração final de aproximadamente 103 bactérias / mL ou regando as mudas com uma suspensão de 106 bactérias / mL após o plantio.
5. incubação das bactérias com o Material de planta
- Para a incubação em meio líquido, incubar as bactérias com o material vegetal em água estéril ou sais minerais e de sacarose ou meio de cultura de tecidos de plantas (tais como um 01:10 diluição dos sais de MS)22,23.
- Use um recipiente para o qual as bactérias não aderir. Tente manter a superfície da planta continuamente coberto por submersão ou por agitação suave. Agitação vigorosa pode impedir a adesão ou até mesmo remover as bactérias da superfície da planta.
- Observe o material vegetal no microscópio de luz após diferentes intervalos de tempo para determinar quando parar a incubação e fazer medições. Um curso de tempo de adesão muitas vezes é valioso com amostras colhidas a cada 1 a 4 h ou diariamente dependendo da velocidade da interação.
- Para incubar em areia, aplicam-se as mesmas considerações descritas para incubação em meio líquido na etapa 5.1.
6. medição de aderência usando microscopia
- Fazer medições microscópicas com fibra óptica Nomarski ou contraste de fase para facilitar a visualização das bactérias em superfícies. No entanto, pode ser usado qualquer microscópio de campo claro com ampliação de 20 X ou superior.
- Use a observação microscópica para determinar se as bactérias são aleatoriamente distribuídas ou localizadas em locais específicos. Verifique também se eles estão ligados individualmente ou em grupos. Olha para a presença de microcolonies, sugerindo que o crescimento bacteriano ou uma armadilha após a adesão. Verifique se as bactérias parecem formar um biofilme na superfície.
Nota: Um biofilme é um grande número de bactérias vinculado à superfície e rodeado por uma matriz extracelular23,24,25. A estrutura pode ser lisa e uniforme ou ter uma arquitetura mais complicada. Métodos para o estudo de biofilmes associados com as superfícies da planta têm sido descritas17. - Use bactérias marcadas com um marcador fluorescente. Se outras bactérias estão presentes e as bactérias de interesse são identificadas por uma etiqueta fluorescente como proteína verde fluorescente, use microscopia de fluorescência para determinar a presença da bactéria marcadas em clusters de outras bactérias26. Para as boas práticas agrícolas, use um filtro com 490 nm de excitação e emissão de 520 nm.
- Verifica que a tag fluorescente é não prejudicar as bactérias observando a aderência ao material axénica de uma mistura igual de bactérias do tipo selvagem e etiquetadas bactérias da mesma estirpe usando óptica tanto Nomarski e fluorescência. Se as bactérias fluorescentes e escuras são aleatoriamente misturadas e presente em números iguais então a tag não interferiu com o ensaio.
- Use a observação microscópica para determinar se as bactérias são aleatoriamente distribuídas ou localizadas em locais específicos. Verifique também se eles estão ligados individualmente ou em grupos. Olha para a presença de microcolonies, sugerindo que o crescimento bacteriano ou uma armadilha após a adesão. Verifique se as bactérias parecem formar um biofilme na superfície.
- Determine o número de bactérias anexados.
Nota: É muito difícil determinar o número de bactérias anexados no microscópio. Quando a ligação é para superfícies irregulares da planta, isso geralmente não é possível obter uma medida quantitativa. Microscopia eletrônica (não discutidos neste artigo) pode ser usado para fazer tais medições.- Quando as bactérias são vinculadas a uma superfície lisa como uma raiz do cabelo, conte o número de bactérias vinculado à borda do cabelo raiz por raiz cabelo mm de comprimento. Tome cuidado para usar os cabelos de raiz de aproximadamente o mesmo tamanho e forma em comparando as medições.
- Para determinar o tamanho de objetos no microscópio, use um slide comercial com marcações medidas. Observar e fotografar este slide com as mesmas configurações usadas para o material experimental e usar as imagens resultantes para determinar o tamanho dos objetos nas fotomicrografias.
- Prepare a amostra para microscopia.
- Lave a amostra. Mover a amostra para uma gota de água ou incubação médio em uma corrediça do microscópio e observá-lo diretamente.
Nota: Isto tem a vantagem que se não houvesse nenhum crescimento bacteriano ou morte bacteriana real lá são susceptíveis de ser muitas bactérias livre. Leve a ausência das bactérias livre como um sinal de aviso que pode ter havido morte bacteriana ou bacteriana vinculação para o recipiente em que foi realizada a incubação. O efeito de lavagem da amostra é mostrado na Figura 1. - Lave a amostra suavemente na água ou incubação médio colocando-o em um frasco de líquido e invertendo o frasco suavemente. Em seguida, coloque a amostra do slide de microscópio em líquido fresco para observação.
- Monte a amostra em líquido usando uma lamela ordinária e microscópio.
- Se a amostra for espessa e assim faria uma protuberância sob a lamínula, use um deslizamento da tampa de press-apply. Estes lamínulas têm um anel de borracha ou de plástico ao redor da borda da lamínula. Coloque o líquido e a amostra no poço em lamínula e então coloque o slide em cima e pressione suavemente para selar o deslizamento da tampa para o slide. Inverter e examinar.
- Como alternativa, use um algas contando slide e deslizamento da tampa de forma semelhante. Nota que desliza com esta profundidade geralmente não pode ser examinada com uma objectiva de maior ampliação de 20 X.
- Lave a amostra. Mover a amostra para uma gota de água ou incubação médio em uma corrediça do microscópio e observá-lo diretamente.

Figura 1 : Passos na preparação de uma amostra para determinar o número de bactérias acoplados. A. tumefaciens vinculação para cabelos de raiz de tomate (A, B e C) e fios de nylon (D, E e F). Em amostras montadas na água sem lavar ambos ligados a bactérias (setas pretas) e bactérias gratuito (setas brancas) podem ser vistas (A e D). Depois de lavar que as limite bactérias permanecem, mas as bactérias gratuito não estão mais presentes (B e E). Depois sonication as bactérias limite tem sido removidas da superfície da amostra (C e F). Clique aqui para ver uma versão maior desta figura.
- Se foram usadas bactérias fluorescentes, examine a amostra no microscópio, depois que a areia foi removida. As raízes das plantas cultivadas na areia geralmente não são adequadas para microscopia como as partículas de areia interferem com a observação das bactérias.
- Remova a planta de um recipiente de crescimento conforme o passo 7. Coloque as raízes em um recipiente com água e misture suavemente para remover a areia que vai resolver para o fundo do recipiente.
- Retire a planta da água de lavagem e montar a amostra como na etapa 6.3.3.
7. medição de aderência usando célula viável conta
- Determine o número de bactérias anexados viáveis, utilizando um sonicador.
- Remova as bactérias não acopladas. Coloca a amostra em um frasco com água suficiente, lavagem de buffer ou incubação médio para cobrir o material vegetal e inverta o frasco várias vezes.
- Se houvesse mais de 103 livre bactérias / mL presente na incubação inicial, realize lavagens sequenciais para remover todos eles. Verificar em um microscópio de luz (ver passo 6.3) para determinar a presença de bactérias gratuito.
- Lave até que haja uma redução substancial do número de bactérias livre, mas lembre-se que há um equilíbrio entre as bactérias acopladas e desacopladas, então o número de bactérias gratuito nunca pode diminuir para zero.
- Retire a amostra do frasco de líquido com uma pinça ou espátula de lavagem.
- Determine o número de bactérias acoplados na incubação do botânico em líquido.
- Suspender a amostra lavada em um frasco e cubra-o com um volume de água, meio de incubação ou tampão de lavagem. Use o líquido suficiente para cobrir a amostra. Coloque o frasco em um sonicador de banho e proceda à sonicação-por um minuto.
- Retirar a amostra e examiná-la sob o microscópio para determinar se as bactérias limite tem sido removidas da planta. A remoção de bactérias a partir de uma amostra por sonication é mostrada na Figura 1. Se houver ainda sujeito a bactérias presentes continue sonicating a amostra até tal, uma vez que nenhum limite bactérias permanecem na superfície da amostra. Se as bactérias limite não podem ser removidas por sonication, adicionar areia de quartzo estéril de 1-10 mg/mL e repetir o sonication e exame microscópico.
- Determinar que o procedimento de sonication que aparece mais eficaz na etapa 7.1.3.2 não reduz a viabilidade das bactérias, suspendendo as bactérias de uma cultura líquida na solução a ser usado para sonication (adicionar areia de quartzo, se fosse necessário). Determine a contagem de células viáveis. Proceda à sonicação as bactérias e determinar a contagem de células viáveis novamente.
Nota: Se houver uma redução na contagem de células viáveis, modifique o procedimento, alterando a composição do líquido e/ou sonication tempo até que o tratamento não tem efeito sobre contagem de células viáveis. - Determine que o tampão de diluição usado para contagem de células viáveis não afeta a viabilidade das bactérias que têm sido incubado sob as condições usadas comparando a contagem de células viáveis feita usando buffers de diluição diferente e/ou água.
- Remova as bactérias não acopladas. Coloca a amostra em um frasco com água suficiente, lavagem de buffer ou incubação médio para cobrir o material vegetal e inverta o frasco várias vezes.
- Determine o número de bactérias de anexado viáveis usando homogeneização.
- Coloca a amostra num almofariz estéril ou liquidificador ou outro dispositivo de homogeneização. Cubra-o com um volume de água estéril, meio de incubação ou tampão27de lavagem. Use um volume suficiente para cobrir a amostra. Para um liquidificador use 100 mL.
- Triture até que é bem homogeneizado. Verificar o pH após homogeneização para ter certeza que o ácido liberada a partir do tecido de planta não tenha causado uma queda acentuada em pH abaixo de 7. Se o pH caiu use um buffer como tampão de fosfato no líquido homogeneização para manter o pH.
- Determine a contagem de células viáveis.
- Utilização de bactérias marcadas com resistência aos antibióticos.
- Em situações em que mais de um tipo de bactéria está presente, marca estirpes bacterianas usando espontânea resistência a antibióticos (geralmente rifampicina e ácido nalidíxico).
- Determine o nível de antibiótico para usar. Se as culturas dos outros organismos deverá estar presente em incubação são disponíveis, placa essas culturas cultivadas sob as condições de incubação pretendida com plantas em placas contendo uma gama de concentrações dos escolhidos antibióticos. Determine a menor concentração que não permite o crescimento de qualquer um desses organismos. Esta é a menor concentração de antibiótico para o qual as bactérias de teste precisa ser resistente.
- Obter espontâneos mutantes resistentes aos antibióticos. Cresce uma cultura de bactérias em meio rico log final ou fase estacionária. Placa de 0,1 mL, sem diluição em uma placa contendo o antibiótico desejado em uma concentração adequada. Determine as concentrações usar conforme descrito na etapa 7.3.1.1. Manter e purificar as bactérias que crescem e, portanto, são resistentes ao antibiótico.
- Determine que resistência aos antibióticos não reduz o crescimento das bactérias, fazendo uma curva de crescimento em meio líquido do pai e estirpes resistentes aos antibióticos. Determine que as bactérias resistentes aos antibióticos mostram o mesmo nível de colonização de material vegetal axénica como a estirpe, se isso for possível.
- Em situações em que mais de um tipo de bactéria está presente, marca estirpes bacterianas usando espontânea resistência a antibióticos (geralmente rifampicina e ácido nalidíxico).
- Determine o número de bactérias viáveis vinculado a plantas cultivadas na areia.
- Retire a planta do recipiente e areia. Para remover as plantas de recipientes, primeiro remova o material de selagem na parte superior e inferior do recipiente. Coloque o recipiente sobre um pedaço de papel estéril e remova cuidadosamente a areia toda ou o solo e a planta como um grande cilindro cônico batendo suavemente o recipiente contra uma superfície, para soltar o material.
- Se necessário, utilize uma espátula ou haste para soltar o material em torno das bordas passando fundo do recipiente.
- Quando o cilindro de areia ou solo contendo a planta está livre no papel, dividi ao meio para revelar a raiz da planta. Se desejado, colher amostras de areia de perto da borda do recipiente, assim como perto da raiz. Isso pode ser útil para determinar a propagação (e acumulação e crescimento) das bactérias.
- Meça o comprimento da raiz. Pegue a raiz e remover a areia e as bactérias aderem frouxamente para a raiz (o material da rizosfera) mergulhando a raiz de um volume medido de água ou buffer e sacudi-la suavemente. Determine a contagem de células viáveis da bactéria na suspensão resultante por chapeamento em meio adequado como ágar Luria. Isso representa o número de bactérias associadas frouxamente com a raiz.
- Retire a planta do recipiente e areia. Para remover as plantas de recipientes, primeiro remova o material de selagem na parte superior e inferior do recipiente. Coloque o recipiente sobre um pedaço de papel estéril e remova cuidadosamente a areia toda ou o solo e a planta como um grande cilindro cônico batendo suavemente o recipiente contra uma superfície, para soltar o material.
- Remover as bactérias limita firmemente pelo sonication e determinar seus números, conforme descrito no passo 7.1.
- Como alternativa, para determinar o local na raiz das bactérias limita firmemente Coloque a raiz lavada na superfície de uma placa de Petri contendo ágar nutriente ou outro meio adequado. Observe a localização de colônias bacterianas na raiz durante os próximos 3 dias usando uma dissecação de microscópio ou lupa.
- Resultados expressos como o número de bactérias por planta, por cm2 de área de superfície, por cm de comprimento de raiz ou por grama de peso fresco do tecido. Fazer várias repetições no mesmo dia e também fazer repetições em dias diferentes, usando diferentes culturas bacterianas e diferentes lotes de material vegetal.
8. determinando se um efeito de condições de incubação na adesão é devido a uma resposta da bactéria ou a planta
- Use o material vegetal vivo ou morto.
- Sujeito a planta material para uma variedade de produtos químicos, fixadores ou outros tratamentos, como o calor a fim de matá-lo. Lave o material vegetal em água e incubação médio após o uso de qualquer um destes tratamentos. Em seguida, inocule as bactérias. Isto não destruirá a superfície da planta, mas será mais metabolicamente inativos para que ele não pode responder às bactérias.
- Medir a adesão bacteriana como descrito nos passos 6 e/ou 7. Também determine o número de células viáveis adicionado para a incubação com o material de planta no início da incubação e o número de células viáveis presentes no final de incubação para garantir que nenhum produtos químicos tóxicos estavam presentes durante a incubação.
- Use o material inanimado.
- Aderência bacteriana de uso de material inanimado, para determinar se um efeito visto na aderência bacteriana para plantar material é devido a um efeito sobre a planta ou as bactérias. Escolher um material inanimado a que ligam as bactérias e que é semelhante em forma e tamanho para o material de planta estudado. Possibilidades incluem papéis de filtro de todos os tipos (celulose, nitrocelulose, fibra de vidro, policarbonato, etc.), tópicos (nylon, algodão, poliéster, lã de vidro, etc.), vidro ou plásticas lamelas, cupons de aço inoxidável e diálise membranas.
- Lave o material inanimado completamente em água e o meio no qual a incubação será realizado e esterilizar antes do uso. A maioria dos materiais listados na etapa 8.2.1 é estável à esterilização em autoclave.
- Incube o material inanimado sob as condições desejadas e pontuação, conforme descrito nas etapas 6 e 7. Um exemplo do uso de fios de nylon para determinar que a ligação reduzida da . tumefaciens para cabelos de raiz em concentrações elevadas de cálcio é devida (pelo menos em parte) a um efeito de cálcio sobre as bactérias é mostrado na Figura 428.
Resultados
A. tumefaciens coloniza as superfícies da raiz. A fim de determinar se a produção bacteriana da celulose desempenha um papel na colonização de raiz, os efeitos de mutações de bactérias que impedem a síntese de celulose foram examinadas16. Foram utilizadas as técnicas descritas em etapas 1.3 e 7.1. Sementes de tomate foram superfície esterilizada e germinadas em água estéril. Quando as raízes estavam cerca de 2 cm de comprimento foram mergulhadas em uma suspensão de 105 bactérias / mL e plantadas em solo pasteurizado em recipientes. As plantas foram cultivadas por 14 dias a 25 ° C num ciclo escuro h luz/12 de 12 h. Depois que as vezes as plantas indicadas foram removidas dos recipientes. As raízes foram lavadas e lisadas em um sonicador de banho para remover as bactérias acopladas. Bacterianos números foram determinados usando a contagem de células viáveis. A Figura 2 mostra o efeito das mutações de celulose-menos dois diferentes sobre a capacidade das bactérias para colonizar raízes de tomate. Embora os desvios-padrão de algumas medidas foram tão alto quanto 0.9 log10 (um problema comum com este tipo de medida) a redução na ligação da celulose-menos mutantes é claramente evidente e podemos concluir que a produção bacteriana de celulose auxilia as bactérias na colonização de raízes de tomate.
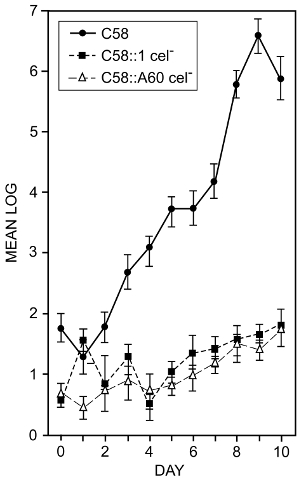
Figura 2 : Raiz de colonização por tipo selvagem e mutantes de celulose-menos da . tumefaciens. Log10 número total de bactérias por cm de comprimento de raiz recuperado de raízes de tomate inoculadas com estirpe de tipo selvagem a. tumefaciens C58 e celulose-menos mutantes, C58:1 e C58:A60. Os números mostrados são os meios de um mínimo de quatro experimentos separados. As barras indicam desvios-padrão dos meios. As raízes foram inoculadas mergulhando-os em uma suspensão de 105 bactérias / mL para um minuto. As plantas foram cultivadas em recipientes e as bactérias frouxamente aderentes foram removidas lavando as raízes no buffer em um frasco de vidro de lavagem. Firmemente aderentes bactérias foram removidas usando um sonicador de banho e a suspensão resultante chapeado para determinar a célula viável conta16. Esta figura foi modificada de Matthysse e McMahan. Clique aqui para ver uma versão maior desta figura.
Analisou-se o papel de exopolissacarídeo na ligação de e. coli e outras bactérias de brotos de alfafa. Alguns surtos de doenças diarreicas devido a e. coli O157: H7 têm sido rastreados para brotos de alfafa contaminada. Ligação das bactérias do tipo selvagem e mutantes incapazes de fazer vários exopolissacarídeo foi medida usando os métodos descritos nas etapas 1.1, 5.1 e 7.2. Rebentos de alfafa foram superfície esterilizada e germinadas por um dia em água estéril a 25 ° C, no escuro. Quatro brotos com os revestimentos de semente anexados foram colocados em pratos de plástico estéril contendo 5 mL de água. Bactérias cultivadas em caldo Luria foram adicionadas a uma concentração final de aproximadamente 5 x 103 / mL. Os brotos inoculados foram incubados a 25 ° C, no escuro por 3 dias. Os brotos foram lavados duas vezes em 5 mL de água estéril em um frasco por inversão vigorosa e homogeneizados no buffer usando um homogeneizador de vidro do Teflon motorizado de lavagem. Experiências anteriores usando bactérias marcado com um plasmídeo que carreg o gene GFP não mostrou nenhuma bactéria interiorizada embora superfície bactérias observaram-se facilmente. Os resultados são mostrados na tabela 1. Foram examinadas duas estirpes de Escherichia coli O157: H7. Em ambas as cepas a produção de acid(PGA) poli-β-1,6-glucurônico apareceu para fazer a maior contribuição para a ligação de patogênicas Escherichia coli para plantar superfícies. Ácido do cólon também desempenhou um papel significativo na ligação. Enquanto a redução da ligação em celulose-menos mutantes foi significativa não era tão grande como para os outros dois polissacarídeos.
| Efeitos das mutações nos Genes de produção de exopolissacarídeo na vinculação de E. coli O157: H7 de brotos e revestimentos de semente abertos | |||
| Estirpe bacteriana | Mutação ou genótipo (fenótipo relevante) | 10 número de bactérias vinculado por broto ou semente casaco de log | |
| B de brotos de alfafa | Revestimentos de semente abertos | ||
| 86-24 | Nenhum (tipo selvagem) | 4,7 ± 0,6 | 5,6 ± 0.2 |
| 8624N | yhjN (celulose-subtração) | 2,9 ± 0,7c | 3,5 ± 0,6c |
| 8624C | wcaD (menos ácido do cólon) | 1,8 ± 0,7c | 2.4 ± 0.5c |
| 8624P | pgaC (PGA-subtração) | < 1,0c | 1,0 ± 1.0c |
| DEC4A | Nenhum (tipo selvagem) | 5,6 ± 0.2 | 6.1 ± 0,3 |
| DEC4AN | yhjN (celulose-subtração) | de 4,8 ± 0,8d | 4.1 ± 0,8d |
| DEC4AC | wcaD (menos ácido do cólon) | 3,9 ± 0.5c | de 4,8 ± 0,8d |
| DEC4AP | pgaC (PGA-subtração) | < 1,0c | 1.2 ± 0,7c |
| média ± desvio padrão de um mínimo de três medições do número de bactérias (log10) vinculado depois de 3 dias. | |||
| b brotos foram lavados antes da medição. | |||
| c significativamente diferente do tipo selvagem: P < 0,01. | |||
| d significativamente diferente do tipo selvagem: P < 0,05. | |||
| Esta tabela foi modificada de Matthysse, Deora, Mishra e Torres10. | |||
Tabela 1: efeitos das mutações nos genes de produção de exopolissacarídeo na ligação de Escherichia coli O157: H7 de brotos. A fim de determinar o papel de vários exopolissacarídeo e lipopolissacarídeo na ligação de patogenicidade estirpes de Escherichia coli O157: H7 de brotos de alfafa, a vinculação de um conjunto de mutantes para os brotos e revestimentos de semente abertos foi examinado usando os métodos descrito na etapa 6. Os resultados mostraram que poli-ß-1, 6 -N-acetil-D-glucosamina (PGA) parece ser essencial para a ligação com as couves e esse ácido tanto celulose e colanic são necessários para ligação máxima de Escherichia coli O157. Esta tabela foi modificada de Matthysse, Deora, Mishra e Torres10.
A fim de determinar se a produção de PGA é suficiente para causar ligação bacteriana plantar superfícies, um plasmídeo (pMM11) carregando o operon clonado codificação os genes necessários para PGA produção foi introduzida em duas bactérias diferentes que não Normalmente, seria capaz de ligar a tomate raízes10. A. tumefaciens A1045 é um mutante da estirpe selvagem tipo C58 que não efectuar cíclico-1,2-β-glucan e também falha ao ligar para plantar superfícies29. Sinorhizobium tem 1021 que forma nódulos na alfafa falha ao ligar para não-leguminosas incluindo tomate12. As técnicas descritas em etapas 1.1, 5.1, 7.1 e 6.3 foram usadas para determinar se a capacidade de fazer PGA geralmente maior vinculação bacteriana às superfícies de raiz. Sementes de tomate foram superfície esterilizada e germinadas em água estéril. As raízes foram cortadas em segmentos de 1 cm de comprimento e colocadas em água estéril e as bactérias foram vacinadas. Como estas duas espécies de bactérias crescem em ritmos diferentes, vinculação foi medida em momentos diferentes para permitir quantidades aproximadamente iguais de crescimento bacteriano. A presença de pMM11 o plasmídeo causada semelhante aumento significativo no número de bactérias limite de ambas as espécies (tabela 2)10. Um aumento significativo na ligação também foi visto no microscópio de luz, mas a ligação era muito diferente para as duas espécies (Figura 3). A. tumefaciens A1045 associadas como bactérias individuais para a superfície da raiz. S. tem ligado em grandes aglomerados em que somente algumas das bactérias eram conectadas diretamente à raiz, e a maioria das bactérias foram anexada a outras bactérias. Este exemplo mostra que simplesmente analisar os números de bactérias vinculados sem incluir observações microscópicas pode dar uma ideia errada dos resultados de um experimento. Embora ambos os métodos (contagem de células viáveis e observações microscópicas) mostram que pMM11 aumentou bacteriana ligação às raízes de tomate, o tipo de ligação causada pela produção de PGA foi diferente para as duas espécies bacterianas10.
| O efeito da pMM11 plasmídeo sobre a ligação de bactérias às raízes de tomate | ||
| Estirpe bacteriana | Plasmídeo | Limite de número de bactérias por mm de comprimento de raiz |
| A. tumefaciens A1045um | nenhum | 0,25 x 103 ± 0,25 x 103 |
| pBBR1mcs (vector) | 0,25 x 103 ± 0,25 x 103 | |
| pMM11 (síntese de PGA) | 10 x 103 ± 0,25 x 103 | |
| S. tem 1021b | nenhum | Não detectado |
| pBBR1mcs (vector) | Não detectado | |
| pMM11 (síntese de PGA) | 50 x 103 ± 5 x 103 | |
| uma associação bacteriana foi medida após 2 horas | ||
| b ligação bacteriana foi medida após 18 horas | ||
| Esta tabela foi modificada de Matthysse, Deora, Mishra e Torres10. | ||
Tabela 2: O efeito de um plasmídeo carregando os genes para a síntese de PGA na ligação de A. tumefaciens A1045 e S. tem 1021 para segmentos de raiz de tomate. A fim de examinar a capacidade de poli-ß-1, 6 -N-acetil-D-glucosamina (PGA) para promover a ligação de bactérias para plantar raízes, o efeito de um plasmídeo confere a capacidade de fazer PGA (pMM11) na ligação de duas cepas de bactérias associadas a planta para raízes de tomate foi examinado. Nem cepa de bactérias mostrou significativa ligação às raízes de tomate na ausência do plasmídeo ou na presença do plasmídeo sem os genes que codificam a síntese de PGA (pBBR1mcs). A adição do plasmídeo com genes de síntese PGA aumentou a vinculação por ambos os tipos de bactérias. Porque a. tumefaciens cresce mais rápido do que o S. tem ligação foi medida após 2 h de incubação para a. tumefaciens e após 18 h para S. tem. As técnicas utilizadas são as descritas na etapa 7. Esta tabela foi modificada de Matthysse, Deora, Mishra e Torres10.
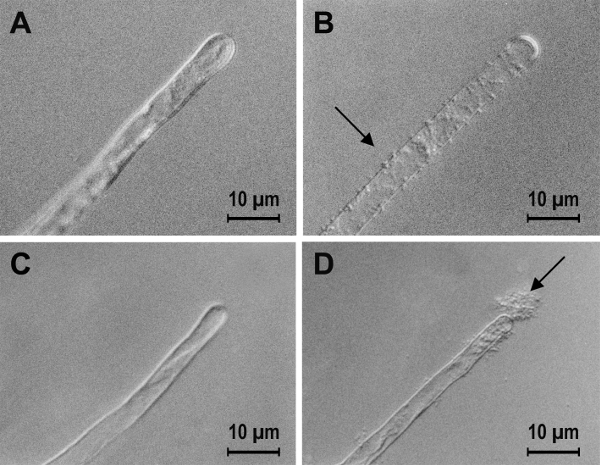
Figura 3 : O efeito da pMM11 plasmídeo carregando os genes de biossíntese de PGA na ligação da . tumefaciens A1045 e S. tem 1021 para cabelos de raiz tomate. Ligação para cabelos de raiz de tomate da) a. tumefaciens A1045, B) pMM11 a. tumefaciens A1045, C) tem S. 1021 e D) tem S. 1021 pMM11. Embora o aumento da vinculação das duas espécies bacterianas é mais ou menos semelhante os detalhes da ligação como visto no microscópio luz são completamente diferentes. As técnicas utilizadas são as descritas na etapa 6. Esta figura foi modificada de Matthysse, Deora, Mishra e Torres10. Clique aqui para ver uma versão maior desta figura.
Às vezes é possível usar a ligação de superfícies não-biológicos para auxiliar na distinção entre a contribuição das bactérias e da planta em uma interação específica. O polysaccharide(UPP) unipolar da . tumefaciens tem demonstrado ser capaz de mediar bacteriana vinculação a uma variedade de ambas as superfícies biológicos e não biológicos30. Cálcio, observou-se inibir a ligação da a. tumefaciens plantar superfícies mediadas por UPP28. A fim de determinar se a inibição por íons de cálcio da vinculação bacteriana para plantar superfícies é devido a um efeito sobre as bactérias ou sobre a superfície da planta, analisou-se a vinculação das bactérias de fios de nylon. As técnicas se descreve no passo 8.2 foram usados. Sementes de tomate foram superfície esterilizada e germinadas em água conforme descrito na etapa 1. As bactérias foram cultivadas em meio mínimo com sacarose e adicionadas a raízes de tomate ou fios de nylon em uma concentração final de aproximadamente 105/mL, conforme descrito na etapa 5.1. O efeito da adição de CaCl2 sobre vinculação bacteriana a raízes de tomate e fios de nylon foi examinado no microscópio. A Figura 4 mostra uma semelhante inibição da ligação de cálcio usando qualquer superfície sugerindo que o efeito do cálcio é principalmente sobre as bactérias10.

Figura 4 : O efeito do cálcio na ligação da . tumefaciens para cabelos de raiz de tomate e fios de nylon. A. tumefaciens foi incubada com raízes de tomate (A e B) ou fios de nylon (C e D) por 24 h em um 01:10 diluição dos sais de MS e um 01:20 diluição do meio mínimo AB, 0,4% de sacarose (A e C) ou em um 01:10 diluição dos sais de MS e 01:20 diluição do meio mínimo AB , 0,4% de sacarose que contém 60 mM CaCl2 (B e D)31. O adicionado CaCl2 inibiu bacteriana vinculação a raízes e fios de nylon, sugerindo que a inibição foi principalmente devido a um efeito sobre as bactérias, e não sobre a superfície da planta. Clique aqui para ver uma versão maior desta figura.
Discussão
É importante estar ciente de todas as superfícies, para o qual as bactérias podem aderir durante o experimento. Assim, as bactérias que são capazes de se ligar ao vidro podem ser subestimadas se a contagem de células viáveis é feitas usando pipetas e tubos de vidro. Se as plantas são cultivadas em ágar ou solo e alguns do ágar ou solo permanece nas plantas, em seguida, as bactérias podem se ligar ao material aderente ao invés de plantas. Por outro lado, lavar a superfície da planta, particularmente no caso de raízes, pode remover revestimentos superficiais naturais tais como mucosa e, assim, alterar os resultados dos testes de aderência.
É importante ter certeza de que a bactéria adicionada à mistura de incubação permanecem viva durante o experimento. Assim, contagem de células viáveis de bactérias gratuito, bem como anexadas deve ser feitas rotineiramente. Alguns tratamentos ou mutações bacterianas podem reduzir a taxa de crescimento bacteriano ou mesmo causar a morte de uma fração da população bacteriana. Bactérias vivas e mortas não podem ser distinguíveis no microscópio, a menos que as manchas especiais são usadas. Há um kit de mancha útil para viver/mortos bactérias que depende a exclusão dos corantes de bactérias vivas. No entanto, se uma população mista de espécies bacterianas está presente então, contagem de células viáveis da espécie de interesse é provável que seja o método mais fácil para determinar se a incubação resultou em morte bacteriana.
Composição média irá influenciar o crescimento e sobrevivência bacteriana. Exsudado de raiz e materiais libertado da ferida e cortar sites irão fornecer substrato para apoiar o modesto crescimento bacteriano. Fosfato, nitrogênio e ferro tendem a ser um fator limitante nessas condições. Cátions divalentes, tais como cálcio e magnésio podem influenciar a adesão. Em alguns casos a fonte de carbono pode influenciar a adesão. pH também é importante. Em geral, o pH da rizosfera é entre 5,5 e 6,5.
É preciso ter cuidado na utilização de bactérias marcadas com resistência aos antibióticos. Os antibióticos mais frequentemente utilizados são a rifampicina e ácido nalidíxico. Resistência a estes antibióticos geralmente é devido a mutações nos genes cromossômicos (RNA polimerase e girase, respectivamente) e, portanto, não podem facilmente ser transferidas para outra estirpe durante a incubação. Este tipo de resistência também não resulta na degradação ou modificação do antibiótico. Marcação de bactérias com um plasmídeo-carregadas gene marcador não é recomendado a menos que o plasmídeo não pode ser transferido para qualquer outras bactérias. A resistência aos antibióticos não deve ser devido à degradação ou químicos, modificação do antibiótico como bactérias sensíveis aos antibióticos, então será capazes de crescer em placas de antibióticas, se eles estão localizados perto de bactérias resistentes.
Os métodos descritos neste artigo são úteis para amostras pequenas e/ou experimentos onde as amostras precisam ser contidos (por exemplo, experimentos envolvendo agentes patogénicos humanos). Para tamanhos de amostra grande (acima de 100 g de material ou de mais de 50 plantas) outros métodos ou drástica modificação desses métodos seria necessário10,19,32,19,33, 34 , 35 , 36. a presença de um grande número de microorganismos que não a espécie a ser estudada também pode representar problemas significativos. Possíveis soluções incluem o uso de bactérias marcadas com uma proteína fluorescente ou resistência aos antibióticos, como descrito nos passos 6.1.2 e 7.3. No entanto, quando as bactérias de interesse são raros indivíduos em uma população grande de outros microorganismos estes marcadores podem não ser adequados para permitir uma avaliação objetiva dos números da bactéria em estudo.
Todos os métodos descritos aqui são métodos laboratoriais com base. Pequenas modificações seriam necessários estudos com efeito de estufa. Mais grandes modificações são susceptíveis de ser necessária para estudos de campo onde, protozoários, insetos e outra animal variação de predação e clima complicam a provisão de condições definidas para os experimentos. No futuro esses métodos podem ser ampliados para incluir as interações de dois ou mais microorganismos na superfície da planta.
Divulgações
O autor declara que ela tem sem interesses financeiros concorrentes.
Agradecimentos
O autor agradece a Susan Whitfield para preparação da Hillary Samagaio e Camille Martin e figuras para assistência com alguns dos experimentos.
Materiais
| Name | Company | Catalog Number | Comments |
| light microscope | any | N/A | phase contrast or Nomarski optics are helpful, for studies using fluorescent markers a fluorescence microscope is required. |
| seeds | any | N/A | make a note of the seed lot number and the cultivar |
| bleach | any | N/A | |
| bath sonicator | any scientific supply company | N/A | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| nutrient agar | Difco | 001-01-8 | |
| soytone | Difco | 24360 | |
| Sand | sea sand Fisher Scientific | S25 | |
| sand | Sigma-Aldrich | S9887 | |
| conetainers | Stuewe & Sons, Inc. | Ray-Leach cone-tainers | many different sizes are available to suit the type of plant you wish to grow |
| parafilm | any scientific supply company | N/A | |
| MS salts | Sigma-Aldrich | M5524 | |
| parrafin | any | N/A | |
| centrigfuge | any scientific company | N/A | to pellet most bacteri only a small centrifuge with a max force of 10,000 XG is needed |
| vortex mixer | any scientific company | ||
| micrometer | ACCU-SCOPE Accessories | A3145 | |
| Sedgwick-rafter counting cell | Hauser Scientific | HS3800 | |
| probe-clip press-seal incubatin chamber | Sigma-Aldrich | Z359483 | |
| rifampicin | Sigma-Aldrich | R3501 | |
| naladixic acid | Sigma-Aldrich | n8878 | |
| Live/dead stain | In Vitrogen Molecular Bioprobes | L7007 |
Referências
- Heaton, J. C., Jones, K. Microbial contamination of fruit and vegetables and the behaviour of enteropathogens in the phyllosphere: a review. J. Appl. Microbiol. 104, 613-626 (2008).
- Beuchat, L. R. Ecological factors influencing survival and growth of human pathogens on raw fruit and vegetables. Microbes Infect. 4, 413-423 (2002).
- Berger, C. N., et al. Fresh fruit and vegetables as vehicles for the transmission of human pathogens. Environ. Microbiol. 12, 2385-2397 (2010).
- Lugtenberg, B. J., Chin, A. W. T., Bloemberg, G. V. Microbe-plant interactions: principles and mechanisms. Antonie Van Leeuwenhoek. 81, 373-383 (2002).
- Preston, G. M., Haubold, B., Rainey, P. B. Bacterial genomics and adaptation to life on plants: implications for the evolution of pathogenicity and symbiosis. Curr. Opin. Microbiol. 1, 589-597 (1998).
- Compant, S., Duffy, B., Nowak, J., Clement, C., Barka, E. A. Use of Plant Growth-Promoting Bacteria for Biocontrol of Plant Diseases: Principles, Mechanisms of Action, and Future Prospects. Appl. Environ. Microbiol. 71, 4951-4959 (2005).
- Mathews, S. L., Smith, R. B., Matthysse, A. G. A comparison of the retention of pathogenic Escherichia coli. O157 by sprouts, leaves and fruits. Microb. Biotechnol. 7, 570-579 (2014).
- Fuqua, C., Matthysse, A. G. Methods for studying bacterial biofilms associated with plants. Methods Enzymol. 337, 3-18 (2001).
- Torres, A. G., Jeter, C., Langley, W., Matthysse, A. G. Differential binding of Escherchia coli.O157:H7 to alfalfa, human epithelial cells, and plastic is mediated by a variety of surface structures. Appl. Environ. Microbiol. 71, 8008-8015 (2005).
- Matthysse, A. G., Deora, R., Mishra, M., Torres, A. G. Polysaccharides cellulose, poly-beta-1,6-n-acetyl-D-glucosamine, and colanic acid are required for optimal binding of Escherichia coli O157:H7 strains to alfalfa sprouts and K-12 strains to plastic but not for binding to epithelial cells. Appl. Environ. Microbiol. 74, 2384-2390 (2008).
- Matthysse, A. G., et al. The effect of cellulose overproduction on binding and biofilm formation on roots by Agrobacterium tumefaciens. Mol. Plant Microbe Interact. 18, 1002-1010 (2005).
- Matthysse, A. G., Kijne, J. W., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. Attachment of Rhizobiaceae to plant cells. The Rhizobiaceae. , 235-249 (1998).
- Matthysse, A. G. Conditioned medium promotes the attachment of Agrobacterium tumefaciens.strain NT1 to carrot cells. Protoplasma. 183, 131-136 (1994).
- Matthysse, A. G., McMahan, S. The effect of the Agrobacterium tumefaciens. attR mutation on attachment and root colonization differs between legumes and other dicots. Appl. Environ. Microbiol. 67, 1070-1075 (2001).
- Jeter, C., Matthysse, A. G. Characterization of the binding of diarrheagenic strains of E. coli.to plant surfaces and the role of curli in the interaction of the bacteria with alfalfa sprouts. Mol. Plant Microbe Interact. 18, 1235-1242 (2005).
- Matthysse, A. G., McMahan, S. Root colonization by Agrobacterium tumefaciens is reduced in cel, attB, attD, and attR mutants. Appl. Environ. Microbiol. 64, 2341-2345 (1998).
- Morton, E. R., Fuqua, C. Phenotypic analyses of Agrobacterium. Curr. Protoc. Microbiol. , (2012).
- Brandl, M. T. Plant lesions promote the rapid multiplication of Escherichia coli O157:H7 on postharvest lettuce. Appl. Environ. Microbiol. 74, 5285-5289 (2008).
- Franz, E., et al. Quantification of contamination of lettuce by GFP-expressing Escherichia coli O157:H7 and Salmonella enterica serovar Typhimurium. Food Microbiol. 24, 106-112 (2007).
- Morton, E. R., Fuqua, C. Laboratory maintenance of Agrobacterium. Curr. Protoc. Microbiol. , (2012).
- Lugtenberg, B. J., Kravchenko, L. V., Simons, M. Tomato seed and root exudate sugars: composition, utilization by Pseudomonas biocontrol strains and role in rhizosphere colonization. Environ. Microbiol. 1, 439-446 (1999).
- Murashige, T., Skoog, F. A Revised Medium for Rapid Growth and Bio Assays with Tobacco Tissue Cultures. Physiologia Plantarum. 15, 473-497 (1962).
- Danhorn, T., Fuqua, C. Biofilm formation by plant-associated bacteria. Annu. Rev. Microbiol. 61, 401-422 (2007).
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- O'Toole, G., Kaplan, H. B., Kolter, R. Biofilm formation as microbial development. Annu. Rev. Microbiol. 54, 49-79 (2000).
- Charkowski, A. O., Barak, J. D., Sarreal, C. Z., Mandrell, R. E. Differences in growth of Salmonella enterica and Escherichia coli O157:H7 on alfalfa sprouts. Appl. Environ. Microbiol. 68, 3114-3120 (2002).
- Loper, J., Suslow, T., Schroth, M. Lognormal distribution of bacterial populations in the rhizosphere. Phytopathology. 74, 1454-1460 (1984).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front Plant Sci. 5, 252 (2014).
- Douglas, C. J., Halperin, W., Nester, E. W. Agrobacterium tumefaciens mutants affected in attachment to plant cells. J. Bacteriol. 152, 1265-1275 (1982).
- Tomlinson, A. D., Fuqua, C. Mechanisms and regulation of polar surface attachment in Agrobacterium tumefaciens. Curr. Opin. Microbiol. 12, 708-714 (2009).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front Plant Sci. 5, (2014).
- Wright, K. M., et al. The endophytic lifestyle of Escherichia coli O157:H7: quantification and internal localization in roots. Phytopathology. 103, 333-340 (2013).
- Berger, C. N., et al. Fresh fruit and vegetables as vehicles for the transmission of human pathogens. Environ. Microbiol. 12, 2385-2397 (2010).
- Erickson, M. C., et al. Internalization and Fate of Escherichia coli O157:H7 in Leafy Green Phyllosphere Tissue Using Various Spray Conditions. J. Food Prot. 77, 713-721 (2014).
- McKellar, R. C., et al. Evaluation of different approaches for modeling Escherichia coli O157:H7 survival on field lettuce. Int. J. Food Microbiol. 184, 75-85 (2014).
- Wu, F. M., et al. Factors influencing the detection and enumeration of Escherichia coli O157:H7 on alfalfa seeds. Int. J. Food Microbiol. 71, 93-99 (2001).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados