Method Article
Medición de las interacciones de las proteínas globulares y filamentosas por espectroscopia de resonancia magnética Nuclear (RMN) y microescala Thermophoresis (MST)
En este artículo
Resumen
Aquí, presentamos un protocolo para la producción y purificación de proteínas que están marcados con isótopos estables y posterior caracterización de las interacciones de la proteína-proteína usando la espectroscopia de resonancia magnética Nuclear (RMN) y microescala Los experimentos de Thermophoresis (MST).
Resumen
Proteínas filamentosas como vimentina proporcionan organización dentro de las células proporcionando un andamio estructural con los sitios que se unen a las proteínas que contienen plakin repite. Aquí, un protocolo para la detección y medición de tales interacciones se describe usando el dominio repetición del plakin globulares de envoplakin y la bobina helicoidal de vimentina. Esto proporciona una base para determinar si una proteína une vimentin (o proteínas filamentosas similares) y para la medida de la afinidad de la interacción. La proteína globular del interés marcada con 15N y tituló con vimentina proteína en solución. Un espectro de RMN bidimensional es adquirido para detectar las interacciones mediante la observación de cambios en forma de pico o cambios químicos y para dilucidar los efectos de las condiciones de la solución incluyendo los niveles de sal, que influyen en la estructura cuaternaria de la vimentina. Si la proteína de interés une el ligando filamentoso, se cuantifica la interacción vinculante por MST con las proteínas purificadas. El enfoque es una manera sencilla para determinar si una proteína de interés une a un filamento y para evaluar cómo alteraciones, tales como condiciones de la solución, o las mutaciones afectan la interacción.
Introducción
Las interacciones entre las proteínas permiten la formación de máquinas moleculares que crear orden dentro de las células. Las interacciones individuales son a menudo débiles pero generalmente contribuyen a complejos polivalentes que pueden ser cooperativos y dinámicamente reglamentados. Ensayos sensibles que ofrecen resolución atómica e información cuantitativa acerca de tales interacciones complejas se necesitan deducir mecanismos y diseñar intervenciones como droga-como de moléculas. Espectroscopia de RMN es un método eficiente para la obtención de información sobre las interacciones entre proteínas y también se utiliza para la detección rápida de ligandos, incluyendo aquellos que se unen débilmente1. Los métodos de NMR se pueden categorizar en los que se observan proteínas o ligando observar. Este manuscrito utiliza el enfoque anterior en el que se adquiere un espectro de un isótopo estable etiquetado como proteína que es comparativamente pequeña (generalmente debajo de 20 kDa) y se titula el ligando sin etiquetar. Esto permitirá el etiquetado residuos implicados en la interacción que se asignará en casos favorables. Una vez que las formas complejas, hay cambios en los ambientes químicos de residuos de interacción que se manifiestan como cambios en la cambio química y forma de sus señales NMR. La magnitud de tales cambios se correlaciona con el grado de participación de estos grupos en la interacción. Perturbaciones de la cambio química (CSP) se pueden medir comparando una serie de espectros de RMN de la proteína en la ausencia y la presencia de cantidades variables del ligand. Para ligandos más grandes o complejas interacciones, el cambio en forma de pico o intensidad puede medirse para deducir las interacciones.
El experimento 2D más comun utilizado para la detección de las interacciones ligando es 15N heteronuclear quantum sola correlación (HSQC) experimento2. Esto requiere que una proteína uniformemente etiquetarse con 15N, que normalmente se consigue expresando como versiones etiquetadas con afinidad en cultivos bacterianos e. coli cultivadas en 15medios enriquecidos en N. Vinculante es evidente cuando se superponen los espectros HSQC reunidos durante la valoración, revelando cambios de pico para un subconjunto de los residuos implicados en la formación de complejos. La interacción puede ocurrir en el régimen de intercambio rápido donde las señales de estado libre y saturado de ligando colapsan en una población promediada pico. Por otra parte, en el caso de intercambio lento entre los Estados, ambas señales se observan con integrales que representan sus cantidades relativas. Mientras que el análisis de NMR lineshape pueden utilizarse para estimar las afinidades de enlace en algunos casos, métodos como el MST también han demostrado ser convenientes y proporcionan validación cruzada de las interacciones genuinas.
El ejemplo es de dos proteínas que se encuentran dentro de desmosomas. Median las ensambladuras entre las superficies de la célula y citoesqueleto y median interacciones multivalentes entre máquinas de adherencia de célula y los filamentos intermedios para mantener la integridad de la piel y los tejidos del corazón y soportar de las fuerzas de cizalla. Enfermedades pueden causar cuando desmosomal proteínas vimentina como desmoplaquina son comprometidas por mutaciones o autoanticuerpos, llevando a la desestabilización de las ensambladuras de la célula, y por lo tanto, sus interacciones son de importancia crítica3. La base estructural de ligando por desmosomal proteínas se caracteriza por espectroscopia de RMN, mientras que la interacción puede ser cuantificada por el MST. En este documento los métodos fueron utilizados para caracterizar las interacciones entre los dominios repetición plakin (PRDs) que a menudo están presentes como sistemas tándem que ofrecen canales básicos y el vimentin, un filamento intermedio que interactúa a través de una superficie ácida ofrece su helicoidal paquete4. Estos complejos se forman en la membrana celular donde ancla para filamentos intermedios del citoesqueleto de la célula a los desmosomas que unen a las células adyacentes, formando así una red de lazos adhesivos que irradia a lo largo de un tejido.
Protocolo
1. expresión de la proteína recombinante
-
Expresión del PRD Envoplakin (E-PRD) y el Vimentin 99-249 (VimRod)
- Transformar e. coli BL21(DE3) las células con el plásmido que contiene el gen deseado. Propagación de las células en placas de agar que contiene 100 ampicilina μg/mL. Incubar las placas a 37 ° C durante la noche.
- Escoger una sola Colonia y sembrar 20 mL de caldo buenísimo (TB) que contiene 100 ampicilina μg/mL para seleccionar para el plásmido. Crece el cultivo a 37 ° C con agitación (180 rpm) durante la noche.
- Transferencia de la cultura entera 20 mL a 1 L de TB que contiene 50 ampicilina μg/mL. Incubar el cultivo a 37 ° C con agitación a 180 rpm hasta el OD600 = 0.6-0.8.
- Reducir la temperatura a 18 º C e inducen la expresión de proteínas mediante la adición de isopropilo β-D-1-tiogalactopiranósido (IPTG) a una concentración final de 1 mM. Continuar la incubación a 18 ° C con agitación a 160 rpm durante la noche para permitir la expresión de la proteína.
- La cosecha de las células por centrifugación la cultura a 8.000 x g por 15 min, decantar y desechar el sobrenadante.
- Lavar las células cosechadas por Resuspender el precipitado de células en aproximadamente 40 mL de fosfato solución salina amortiguadora (PBS: 20 mM de tampón fosfato, pH 7,4, 120 mM de NaCl). Transfiera la resuspensión a un tubo de 50 mL. Centrifugar nuevamente a 8.000 x g durante 15 minutos.
- Decante y descarte el sobrenadante. Inmediatamente comienza el protocolo de purificación o congelar los pellets de células a-20 ° C para su uso futuro.
-
Expresión de proteína isotópicamente etiquetada
- Transformar células de e. coli BL21(DE3) y preparar una cultura de arrancador 20 mL como en 1.1.1-1.1.2.
- Transferencia de la cultura de todo 20 mL a 1 L de TB enriquecido que contiene un adicional 4,0 g triptona, 5.0 g de NaCl y 100 de μg/mL ampicilina. Incubar el cultivo a 37 ° C con agitación a 160 rpm hasta el OD600 = 1.6-1.9.
- Cosechar el cultivo de 1 L por centrifugación a 8000 x g por 15 min, decantar y desechar el sobrenadante.
- Lavar el precipitado de células por resuspender suavemente en aproximadamente 40 mL de PBS y la resuspensión de transferencia a un tubo de 50 mL.
- Centrifugar nuevamente a 8.000 x g por 15 min, decantar y desechar el sobrenadante.
- Resuspender el precipitado de células en 20 mL de media mínimo M9 (tabla 1) y transferencia al resto de los 950 mL de media mínimo M9 conteniendo ampicilina μg/mL 100.
- Añadir 50 mL de la mezcla nutriente filtro esterilizado (tablas 2 y 3).
- Aclimatarse a la cultura a 18 ° C por 30 min antes de agregar IPTG a una concentración final de 1 mM.
- Incubar durante una noche a 18 ° C con agitación a 160 rpm.
- Las células como en la sección 1.1.4-1.1.6 de la cosecha.
2. inmovilizado Metal afinidad cromatografía (IMAC) purificación de VimRod y E-PRD
-
Purificación de His6 etiquetados VimRod
- Resuspender el precipitado de células en 5 mL/g de PBS con una proteasa inhibidor cóctel EDTA. Homogeneizar con 12 golpes en un homogeneizador de tejidos Dounce mejorar la lisis celular en el siguiente paso.
- En el hielo, someter a ultrasonidos la suspensión celular en un pulso de 1 s 1/s off, amplitud de 80% para un total de 1,5 min repetir sonicación adicional dos veces, girando suavemente en el hielo entre tramos para evitar el sobrecalentamiento.
- Centrifugar la muestra a 75.000 x g durante 45 minutos decantar y filtrar el sobrenadante utilizando un filtro de jeringa (0.45 μm).
- Equilibrar una columna IMAC de 5 mL con 5 volúmenes de columna (CV) de tampón de unión (20 mM HEPES, pH 7,5, 500 mM NaCl, imidazol de 10 mM) con un caudal de 1 mL/min utilizando un sistema de cromatografía líquida (FPLC) proteína rápida.
- Cargar el sobrenadante filtrado en la columna a un flujo de 0,5 mL/min.
- Lavar la columna con tampón 5 CV de lavar (20 mM HEPES, pH 7,5, 500 mM NaCl, imidazol de 50 mM) con un caudal de 1 mL/min.
- Eluir la proteína con 3 CV de tampón de elución (20 mM HEPES, pH 7,5, 500 mM NaCl, imidazol de 350 mM) a un flujo de 0,5 mL/min 1.5 recoger fracciones de mL. Si está disponible, seleccione el modo de caudal ascendente elución para aumentar la concentración de proteínas eluídas.
- Identificar las fracciones del cromatograma FPLC que contienen la proteína del interés por SDS-PAGE y utilizan métodos estándar para medir la concentración de proteínas5.
- Piscina y concentrar las fracciones elución que contienen las cantidades más altas de la proteína usando un dispositivo de ultrafiltración centrífugas (MWCO 3 kDa, 5 mL) a 2 mL. Centrifugue a 21.000 x g para eliminar cualquier precipitado y pasar a través de un filtro de 0,22 μm.
- Equilibrar una columna de cromatografía (S) exclusión de 120 mL tamaño con tampón de CV de S 2 (20 mM HEPES, 150 mM NaCl, pH 7.5, 0,5 mM TCEP) con un caudal de 1 mL/min utilizando un FPLC.
- Inyectar la proteína concentrada de 2.1.9 sobre la columna y eluir con tampón de CV de S 1 a una velocidad de flujo de 0,5 mL/min, recogiendo fracciones de 1 mL.
- Identificar las fracciones que contiene la proteína de interés como antes.
- Las fracciones que contiene la proteína de cantidades más alta de la piscina.
- Almacenar a 4 ° C para uso a corto plazo o añadir glicerol al 20% y conservar a-80 ° C en pequeñas alícuotas.
-
Purificación de la proteína E PRD con la etiqueta de His6 eliminada
- Siga los pasos en 2.1.1-2.1.8 para purificar la proteína His6 tagged E-PRD. Piscina las fracciones pico y determinar la concentración de proteína.
- Añadir tabaco etch proteasa del virus (TEV) (1 mg/mL) en 2 μl/mg de proteína combinada. Transferencia para diálisis (6 kDa) de la tubería y dializo en buffer de S durante la noche a 4 ° C. Este paso permite la ruptura de la etiqueta de His6 y eliminación de lo imidazol que interfiere con el atascamiento a la resina de Ni-NTA en el paso siguiente.
- Equilibre los 5 mL de resina Ni-NTA en una columna de gravedad con tampón de CV de S 3. Drenar el exceso de tampón de la resina.
- Pour la proteína E-PRD hendida en la resina e incubar durante 1 hora en una plataforma oscilante para permitir uncleaved His6 tagged E-PRD y la etiqueta de His6 exfoliada enlazar. La proteasa TEV es también etiquetado His6 y será enlazar la resina. Recoge el flujo a través, que contiene la etiqueta-libre E-PRD. Lavar la resina con 2 buffer de CV de S para todos lo E-PRD se recupera.
- E-PRD se concentran en el flujo a través de 2 ml con un dispositivo de ultrafiltración centrífugas (MWCO 3 kDa, 5 mL). Centrifugar a 21 000 x g para eliminar cualquier precipitado y pasar a través de un filtro de 0,22 μm.
- Equilibrar una columna S de 120 mL con tampón de CV de S 2 (20 mM HEPES, 150 mM NaCl, pH 7.5, TCEP MST o 20 mM Tris-HCl, 1 mM TDT, pH 7 para NMR de 0,5 mM) con un caudal de 1 mL/min utilizando un FPLC.
- Inyectar la proteína concentrada de E-PRD de 2.2.5 a la columna y eluir con tampón de CV de S 1 a una velocidad de flujo de 0,5 mL/min, recogiendo fracciones de 1 mL.
- Identificar las fracciones que contiene la proteína de interés como antes.
- Las fracciones que contiene la proteína de cantidades más alta de la piscina.
- Almacenar a 4 ° C para uso a corto plazo o añadir glicerol al 20% y conservar a-80 ° C en pequeñas alícuotas.
3. RMN los métodos
- Preparación de la muestra de RMN
- Purificar 15tipo marcado con N o R1914E E-PRD proteína como previamente se describió utilizando 20 mM Tris-HCl, 1 mM TDT, pH 7 como el S Paso 2.2.6. Soluciones stock de proteína generalmente van de 0.3 a 1 mM con un volumen de aproximadamente 1 mL.
Nota: La proteína puede ser concentrada a > 100 μm utilizando un dispositivo de ultrafiltración centrífuga 5 mL, MWCO 3 kDa para la concentración en un rango adecuado para la preparación de la muestra. - Purificar una muestra de proteína VimRod de 20 mM Tris-HCl, 1 mM TDT, pH 7 como el S Paso 2.1.10.
- En un volumen final de 500 μl, añadir tipo salvaje o mutante de la proteína E PRD a una concentración final de 100μm, óxido de deuterio (D2O) a una concentración final del 10% (v/v) y DSS (ácido 4,4-dimethyl-4-silapentane-1-sulfonic) a una concentración final de 20 μm. traer el volumen hasta 500 μl de muestra con 20 mM Tris-HCl, 1 mM TDT, pH 7. Una preparación de la muestra representativa se describe en la tabla 4.
Nota: El 0 ppm resonancia de DSS se utiliza para calibrar los desplazamientos químicos de 1H como para referencia indirecta de la sustancia 15N cambios de la proteína6. D2O se utiliza para la señal de bloqueo de deuterio para mantener el espectrómetro en un campo magnético neto constante. - Hacer una segunda muestra del E-PRD, D2O y DSS como en el paso anterior y añadir VimRod a una concentración final de 50 μm antes de llevar el volumen hasta 500 μl.
- Transferir las 500 μl muestras a un 5 mm ancho NMR los tubos para el experimento.
- Purificar 15tipo marcado con N o R1914E E-PRD proteína como previamente se describió utilizando 20 mM Tris-HCl, 1 mM TDT, pH 7 como el S Paso 2.2.6. Soluciones stock de proteína generalmente van de 0.3 a 1 mM con un volumen de aproximadamente 1 mL.
- Disposición Experimental de RMN
- Encender el flujo de aire con el comando eject "ej"; Esto abrirá la muestra del imán. Ahora, coloque la muestra dentro de una ruleta en la parte superior el imán por la abertura y coloque con el comando "ij". Espere hasta que la muestra se coloca dentro el imán antes de proceder.
- Crear un nuevo conjunto de datos utilizando el comando "edc" y carga estándar 1H NMR parámetros seleccionando experimentan "ZGPR" (figura 1). Rellene los campos de nombre, EXPNO (número de experimento) y PROCNO (número de la carpeta de datos). Seleccionar el solvente en el campo "Set solvente" y haga clic en "Ejecutar 'getprosol'" para leer probehead estándar y parámetros dependientes solvente (prosol).
- La muestra a la deuterados solvente, es decir, D2O la cerradura, el comando "lock" y esperar a que termine arrasando y logra bloqueo.
- Corregir la frecuencia de la resonancia del imán girando la muestra utilizando el comando sintonización automático "atma". Seguimiento de la curva de oscilación hasta que finalice la sintonización automática.
- Acuñe el campo magnético usando TOPSHIM (comando "topshim"). Equilibrado es el proceso de ajustes en campo magnético para lograr uniformidad en la muestra. Es una buena práctica para almacenar los valores de la cuña con el comando "Ass" y leerlos usando "rsh" antes de topshim, si usando las muestras de igual o similares.
- Ajustar la ganancia del receptor con el comando "rga" para lograr la máxima relación señal a ruido.
- Coloque el centro del espectro sobre el desplazamiento de resonancia del agua (o1) y el pulso de protón de 90 grados (p1) en alta potencia con "calibo1p1".
- Recoge el espectro de protón usando el cero ir comando "zg" y con "efp" que incluye multiplicación exponencial ("em"), la decadencia de inducción libre (FID) incorporando la ampliación de la línea, "ft" transformación de fourier de FID y "pk" aplicar fase corrección.
- Aplicar el corrección de fase automática "apk" y la corrección de línea base automática "absn" usando el polinomio sin opción de integración.
- Crear un nuevo conjunto de datos (como en 3.2.2) para el experimento HMBC SOFAST seleccionando "SFHMQC3GPPH" en el experimento.
- Copia optimizado P1 y O1 del espectro de protón y rellenar impulsos dependientes P1 mediante comando "getprosol 1H p1 plw1", donde p1 es el valor optimizado de P1 y plw1 es el nivel de potencia P1.
- Optimizar la constante CNST54 para establecer el desplazamiento para cambio química amida y CNST55 para definir el ancho de banda con el fin de abarcar las regiones espectrales de interés que permite el aumento del receptor ser optimizado (figura 2). Para seleccionar estos parámetros, extraer el primer FID (decadencia de inducción libre) el espectro de dos dimensiones y buscar la señal observada para definirlos. Además, variar el retardo de la relajación (D1), número de exploraciones (NS) y exploraciones simuladas (DS) para obtener la sensibilidad de la señal aceptable con el comando "gs", que permite ir y análisis para monitorear la calidad de los datos en tiempo real.
- Registrar los espectros que utilizando cero ir "zg".
- Procesamiento de datos de RMN
- Configurar los parámetros de procesamiento para el tamaño de la F2 directa (1H) y F1 indirecta (15N) dimensiones de la utilización del espectro "SI F2 = 2048, F1 = 512" con opcional predicción lineal en dimensión indirecta (figura 3).
- Seleccione "QSINE" como la función de ventana e introduzca un cambio de campana del seno (SSB) de 2 para procesar el espectro de dos dimensiones.
- Introduzca el comando "xfb" para procesar los datos en ambas direcciones con la transformación de la función y transformada de fourier de ventana.
- Utilice el comando "apk2d" para llevar a cabo la corrección de fase automática en ambas direcciones. Si el proceso automático no alcanza un nivel satisfactorio de corrección de fase, extraer FIDs con el comando "rser", calcular los valores de la fase de procesamiento de 1D y aplicarlos a los datos 2D.
- Corregir la línea de base con la función de corrección automática de línea de base "abs2" para datos 2D. Esto aplica a una función polinomial entre los valores de ppm definidos en los parámetros de proceso y producirá un espectro 2D para su posterior análisis.
- Si planea realizar el procesamiento serial para la comparación de datos de interacción con otra molécula, almacenar los parámetros de proceso con el comando "wpar" y recordar con "rpar". De esta manera que todos los conjuntos de datos se procesarán con los mismos parámetros y variaciones no se presentará debido a las diferencias de procesamiento.
- Análisis de los datos de RMN
- Introduzca el comando "pp" para iniciar el proceso de recolección de pico.
- Definir el ppm rango y mínima intensidad/número máximo de picos basados en picos esperados (figura 4). Haga clic en aceptar y verificar los resultados mediante inspección visual. Si es necesario, vuelva a ejecutar el proceso hasta que los resultados son satisfactorios basado en espectros de calidad.
- Generar una peaklist con el comando "pp".
Nota: Este peaklist contiene información de sobre intensidad altura/pico de datos por defecto y puede ser exportado a espectros posterior y puede ser leído por otros programas. - Observar cambios en las intensidades de pico o movimiento en los cambios químicos en los espectros HSQC proteínas que indican la interacción con otra molécula. Si la interacción de la molécula es grande, esperar reducciones en intensidades pico junto con la desaparición de algunos picos.
- Importar la lista de pico para el siguiente conjunto de datos haciendo clic en la pestaña de "picos" y seleccionando "importar" con un clic derecho en la ventana de picos.
- Visualizar los picos en el espectro y si es necesario cambiar a nuevas posiciones. Haga clic en "reset intensidades" para el "cuadro completo" para generar un peaklist para el espectro con intensidades (figura 5). Esta lista de pico se trasladará la información de posición de lista de pico almacenados.
- Exportar las listas de pico de diferentes conjuntos de datos a una hoja de cálculo u otro programa de matemática para el análisis seleccionando la función "Exportar".
- Calcular el cambio en la intensidad de pico con la función "intensidad de pico en la intensidad del complejo espectro/pico en el espectro de proteínas" para cada pico. Valores se pueden convertir en porcentaje de cambio por la multiplicación de 100. Tenga en cuenta que los volúmenes pico también son útiles aunque intensidades de pico son más fáciles de medir para los picos que están situados cerca uno del otro, como suele ser el caso de proteínas de alta densidad de picos relativamente amplios.
4. microescala Thermophoresis (MST)
-
Preparación de la proteína ligando E-PRD
- Intercambio de ligando en un buffer compatible del MST por diálisis hasta 800 μL de proteína en una unidad de diálisis mini 3.5 kDa suspendida en 1 L de 20 mM HEPES, pH 7,5, 10 mM NaCl, revolviendo lentamente a 4 ° C durante la noche.
- El ligando utilizando una unidad de ultrafiltración centrífugas (MWCO de 3 kDa) se concentran por centrifugación a 14 000 x g durante 10 min. traslado de la proteína concentrada a un tubo limpio.
- Centrifugue el ligando a 21.000 x g por 10 min y cuidadosamente transfiera el sobrenadante a un tubo nuevo para quitar cualquier proteína precipitada. Determinar la concentración del ligando utilizando la absorbancia a 280 nm y el coeficiente de extinción de ligando. Agregar 10% Tween-20 para dar una concentración final de 0.015%. Tween-20 se añade al tampón de ensayo para evitar la adsorción a los tubos capilares. El buffer de ensayo final que se utiliza para los experimentos de MST es 20 mM HEPES, pH 7,5, 10 mM NaCl, 0.015% Tween-20.
-
Preparación del objetivo marcado con tinte de proteína VimRod
- Reconstituir el tinte rojo-tris-NTA añadiendo 50 μL de 1 x PBS-T se suministra con el rojo-tris-NTA para obtener una concentración de 5 μM. Dispensar alícuotas μL 2 en los tubos de 200 μL y almacenar a-20 ° C.
- Diluir la proteína diana a 0.34 μM con tampón de ensayo. Añadir 58 μL de blanco a una alícuota de 2 μL de colorante de rojo-tris-NTA e incubar durante 30 min a temperatura ambiente. La concentración final de marcado tinte objetivo es 0.33 μM. Centrifugue el objetivo marcado a 21.000 x g por 10 min y cuidadosamente transfiera el sobrenadante a un tubo nuevo para quitar cualquier precipitado. El tinte rojo-tris-NTA se une a las proteínas a través de la etiqueta de His6 y tiene una constante de disociación de enlace (KD) en el rango nanomolar sub. Efectivamente es 100% ligada a la proteína diana por lo que no se requiere ninguna otra purificación.
- Encienda el instrumento MST y abra el Software de Control. Seleccione el ajuste rojo para el tinte rojo-tris-NTA. Gire el control de temperatura a 25 ° C.
- Seleccione la prueba preliminar para validar el etiquetado de la blanco y marque para la agregación o adsorción en las cubetas. Una evaluación de estos parámetros es proporcionar de forma automática.
- Mix 17 μL de tampón de ensayo y 3 μL de proteína diana y mezclar mediante pipeteo. Llene dos tubos capilares estándar por inmersión en el objetivo de diluido y elaboración de líquido en el centro del tubo capilar. Coloque los tubos capilares en la bandeja y en los instrumentos. Iniciar la medición.
- Revisar los resultados buscando un nivel suficiente de fluorescencia y sin signos de adsorción (distorsión en la forma del tubo capilar) o la agregación (distorsiones en el rastro de MST) que se marcan en el análisis de software. Si los resultados son positivo hacia el ligando debe probarse pasos, si no un tampón diferente o puede aumentar la cantidad de Tween 20 al 0.05% o superior.
-
Preparación de la serie de E-PRD ligando doble dilución
- Preparar una serie de diluciones por etiquetado 16, tubos para 200 μL de 1-16.
- Añadir 17 μL del ligand en 1.17 x mayor concentración que la concentración máxima ligando deseada Tube1. Este volumen es dos veces la cantidad necesaria (8,5 μL) como una parte alícuota se transfieren al siguiente tubo de serie. El volumen final de la prueba es 10 μL 8,5 μL de concentración de x 1.17 de ligando se diluirá a 1 x por la adición de 1,5 μL de la proteína diana, VimRod. Esta concentración máxima elegida debe ser al menos 20 veces el valor estimado de KD .
- Añadir el 8,5 μL de tampón de ensayo a Tubes2-16, usando una punta de pipeta nueva para cada alícuota. Reutilización de puntas de pipeta puede afectar la precisión (para asesoría precisa pipetear vea las instrucciones). Transferencia de 8,5 μL de ligando de Tube1 a Tube2 liberan lentamente la solución en el buffer de ensayo sin generar burbujas. Mezclar el ligando y el buffer mediante pipeteo arriba y abajo por lo menos 6 veces, otra vez sin generar burbujas. El PRD E en el tubo más concentrado fue 1,5 mM y la etiqueta VimRod estuvo presente en una concentración final de 50 nM.
- Transferir 8,5 μL de ligando de Tube2 a Tube3 y mezclar con el tampón de ensayo. Repetir la dilución seriada hasta que todos los tubos han tenido ligando añadido. Deseche 8,5 μL de Tube16 para que todos los tubos contengan 8,5 μL de ligando en una serie de diluciones dobles.
-
Preparación del experimento reacción vinculante y MST
- Añadir 1,5 μL de la proteína diana etiquetado a cada uno de los tubos y mezclar suavemente mediante pipeteo arriba y abajo, teniendo cuidado de evitar las burbujas. Incubar durante 15 minutos.
- Seleccionar el modo experto para la prueba enlace y entrar en los parámetros de una serie de dilución seriada. Asegúrese de ajustar el control de la temperatura a 25 ° C, la energía de excitación se establece en 40% y el poder del MST al medio. Estos parámetros deben optimizarse para otros socios de enlace están estudiadas.
- Los capilares se llenan de las reacciones de la Unión y coloque en la bandeja del capilar. Cargar la cubeta en el instrumento, espere que la temperatura para recuperar 25 ° C para iniciar la medición.
-
Análisis de datos
- Abra el archivo de ensayo de enlace en el software de análisis de afinidad. Revisar el análisis capilares; Los niveles de fluorescencia no deben variar más del 10% de la media, no hay agregación o adsorción debe detectar como se describe en la sección 3.6.
- Seleccione el análisis del MST en el panel derecho y arrastre los datos del ensayo en el sistema de análisis. Si hay varios funcionamientos del ensayo de Unión ligando-objetivo mismo en condiciones idénticas puede combinar dejando caer en el mismo conjunto. Alternativamente, cada ejecución puede ser caído análisis independientemente.
- Desplazamos hasta la pestaña ajuste de dosis respuesta a graficar los datos. Elija el modelo de KD si se prevé un sitio único obligatorio. El modelo de Hill es también una opción para múltiples sitios de unión con el comportamiento cooperativo. Puntos aislados que no pasaron control de calidad pueden extraerse el ajuste en esta etapa. Combinar conjuntos con múltiples ensayos serán promediados y errores se calcularon como la desviación estándar.
- Abra la ficha final y comparar resultados entre todas las parcelas en una sola gráfica. Recuperar resultados de montaje para cada curva de la tabla que se genera. Exportar los datos o curvas equipadas a otro software de presentación o análisis si así lo desea.
Resultados
El dominio E-PRD (residuos 1822-2014 clonados en HTC pProEX) del gene humano envoplakin y el dominio de VimRod (residuos 99-249 clonados en pET21a) de vimentin humano4 fueron expresadas con las etiquetas de His6 y purificada. Figura 6 y figura 7 muestran los niveles de pureza de VimRod (18,8 kDa) y E-PRD (21,8 kDa) obtenidos de este método de purificación de proteínas. La eliminación de His6 tag desde la construcción del PRD E es necesario para los experimentos de MST como la proteína VimRod se etiqueta con un tinte de His6 etiqueta obligatoria y cualquier E-PRD conserva su etiqueta de His6 puede competir para el atascamiento del tinte. La segunda columna IMAC después de la ruptura de la etiqueta con proteasa TEV elimina la proteasa TEV, la etiqueta exfoliada y cualquier uncleaved His6-E-PRD que seguía siendo. El paso de pulido final de la purificación es cromatografía por exclusión de tamaño. A pesar de ambas proteínas de tamaño similar, el VimRod elutes de la columna en un mL 51 mientras que el pico de elución E-PRD se centra en 72 mL donde se esperaría un monómero de la proteína de este tamaño. El aparente aumento de tamaño de VimRod es probable que debido a la sus características como una varilla larga filamentosa en forma de proteína analíticamente ultracentrífuga experimentos demostraron que la VimRod era monomérica4. Menores rendimientos de proteína se obtienen de las culturas cultivadas en M9 que de caldo rico debido a una menor cantidad de células que se producen en los medios de comunicación mínima. El crecimiento inicial de los cultivos más grandes para las preparaciones de M9 en TB permite la mejora de los rendimientos de la célula manteniendo la medida de 15N etiquetado es necesario para los experimentos de NMR.
El 15N -1HSQCs H fueron adquiridas para el tipo salvaje y mutante R1914E de E-PRD en presencia o ausencia de VimRod (figura 8A-8 D). El espectro del E-PRD en la figura 8A muestra el número esperado de picos bien resueltos, indicativos de una proteína bien plegada. En presencia de VimRod (figura 8B) el espectro muestra amplia línea ensanchamiento y desaparición de pico, correspondiente al enlace entre el E-PRD y VimRod. Este enlace se pierde por la mutación de R1914E según lo evidenciado por la comparación de la figura 8 y 8 D. Se observa poco cambio en el espectro con adición de VimRod a la mutante R1914E que indica una falta de unión entre este mutante E PRD y VimRod. Las intensidades de pico E-PRD en la presencia/ausencia de VimRod se compararon y trazadas como las intensidades pico relativo en figura 8E, que indica el rango de ampliación de pico en el complejo E-PRD. El mutante de R1914E E-PRD (no mostrado) retuvo cerca del 97% de los picos en 20% o más altas intensidades de pico en presencia de VimRod en comparación con alrededor del 20% para el tipo salvaje (figura 8E). Esto representa una pérdida del mutante de punto de función, con mutantes adicionales con efectos intermedios también después de haber sido estudiados4.
Para validar y cuantificar el atascamiento de análisis VimRod y PRD E MST utilizando His6-VimRod marcada con colorante fluorescente rojo-tris-NTA como destino mezclado con la disminución de las concentraciones de ligando E PRD de 1,28 mM a 39.1 nM fueron realizados. Tres titulaciones de Unión se llevaron a cabo y los resultados son un promedio y se muestra en la figura 9. Los datos se ajustaron con un modelo estándar del sitio de un ligando y dieron un KD de 25,7 μM ± 2.1. Evaluación de las uniones entre VimRod y E-PRD por la resonancia de plasmón superficial dio un valor de KD similar de 19,1 ± 1.3 μM4.

Figura 1: pantalla de captura de la configuración del experimento NMR. La ventana mostrada se usa para configurar un experimento estándar para recoger un conjunto de datos HSQC. Parámetros del experimento se leen en junto al experimento. El experimento ZGPR que se muestra es escogido como un experimento inicial para cargar los parámetros estándar y solvente de protones dependiente. La ventana de título se usa para detalles experimentales para fines de mantenimiento de registros de entrada. Para recoger el espectro HSQC el experimento ZGPR se sustituye por SFHMQC3GPPH. Haga clic aquí para ver una versión más grande de esta figura.
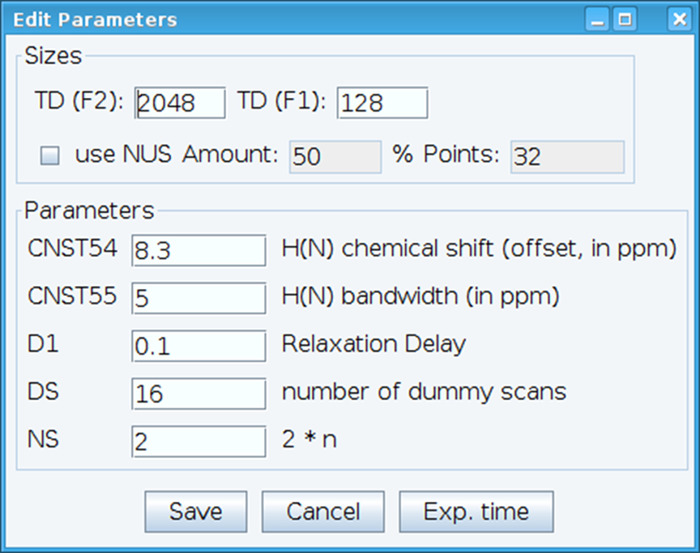
Figura 2: ajuste de parámetros experimentales RMN. La ventana mostrada se usa para introducir los parámetros básicos para la secuencia de pulso NMR para optimizar la señal. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: RMN de procesamiento de datos. Parámetros utilizados para el procesamiento de cada una de las dos dimensiones del espectro NMR se muestran con flechas indicando que por lo general se ajustan. Haga clic aquí para ver una versión más grande de esta figura.
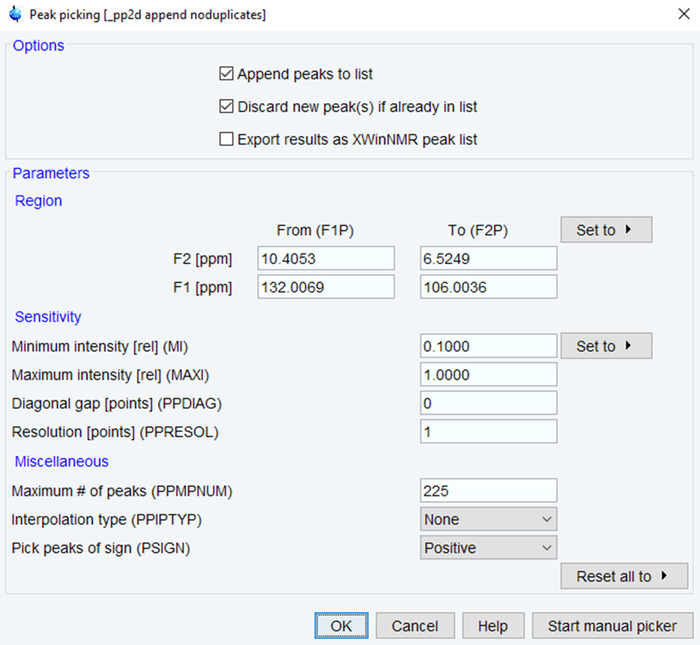
Figura 4: parámetros de cosecha pico RMN. Se muestran los parámetros utilizados para escoger NMR picos en el espectro NMR procesado con valores típicos. Ajustar la gama del ppm, la intensidad y el número de picos para optimizar los espectros. Haga clic aquí para ver una versión más grande de esta figura.
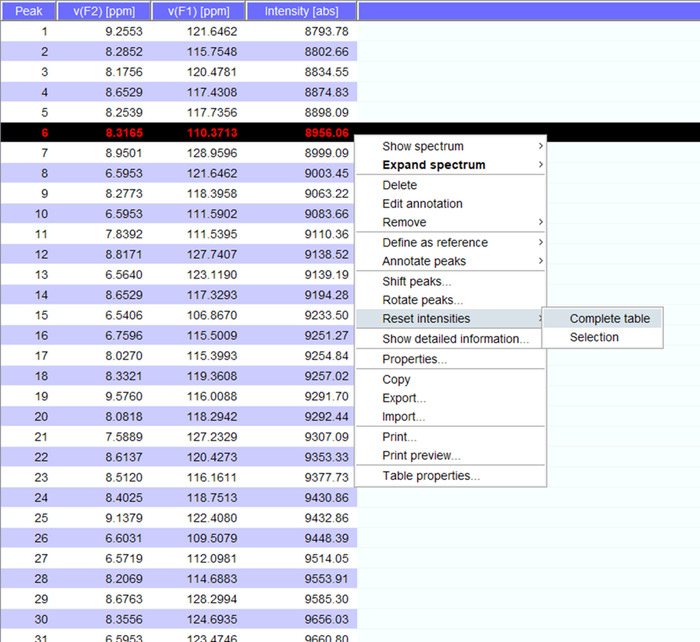
Figura 5: Representante Peaklist con intensidades de. Cada pico que se levanta en el espectro de RMN es dado un número y su 1H y 15N química cambia y se muestran la intensidad de la señal. Este peaklist entonces puede utilizarse para comparar los espectros obtenidos en ausencia y presencia de un socio de interacción. Haga clic aquí para ver una versión más grande de esta figura.
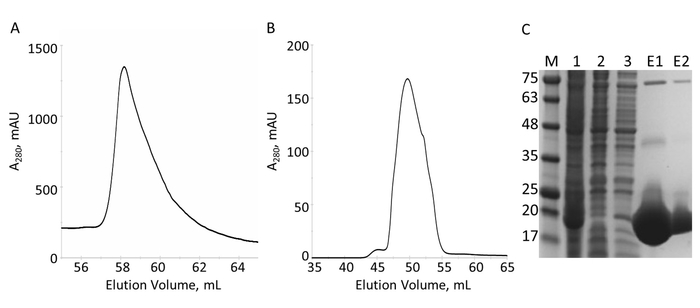
Figura 6: purificación del VimRod His6 tagged IMAC y S. A. El cromatograma para la elución de la columna IMAC muestra un pico importante de VimRod. B. el cromatograma para la elución de la columna S muestra un pico importante. C. SDS-PAGE de las fracciones recogidas a lo largo de purificación: normas MW con el MW indicaron en kDa a la izquierda del gel (M), lisado de células (1), fluir por IMAC (2), lavado (3), elución combinado (E1), agrupada S elución (E2). Bandas visibles en mayores pesos moleculares en líneas E1 y E2 son oligómeros de VimRod puro según lo confirmado por western blot (datos no mostrados). Haga clic aquí para ver una versión más grande de esta figura.
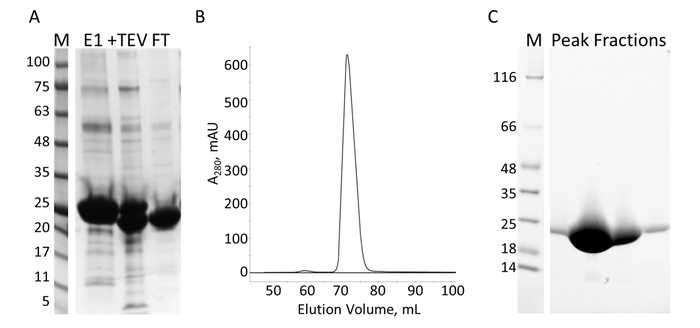
Figura 7: purificación del E-PRD por IMAC y S. A. SDS-PAGE de la purificación de IMAC con los estándares de peso molecular con el MW indicados en kDa a la izquierda del gel (M) y el efluente de la primera columna IMAC (E1), la TEV escote productos (+ TEV) y el flujo a través de la segunda columna IMAC (FT). B. la cromatografía de la columna S muestra un pico importante. C. SDS-PAGE de los estándares de peso molecular (M) y las fracciones de pico S. Haga clic aquí para ver una versión más grande de esta figura.
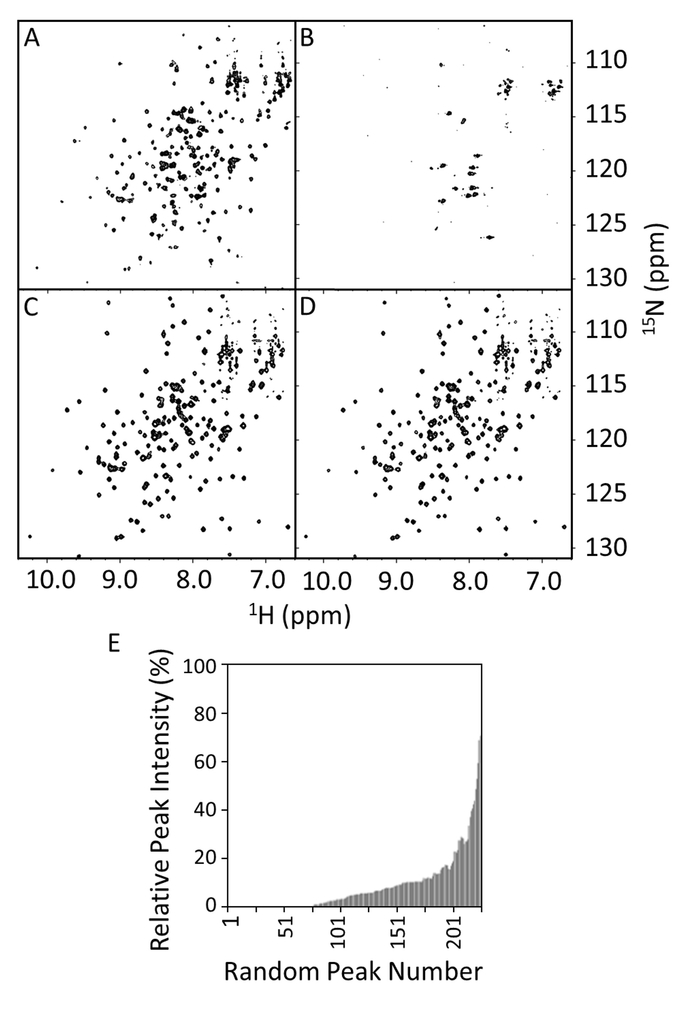
Figura 8: HSQC espectros de tipo salvaje y mutante R1914E de E-PRD en presencia y ausencia de VimRod. Los espectros HSQC Mostrar tipo salvaje E-PRD (100 μm) en 20 mM Tris-HCl, 150 mM NaCl, 1 mM TDT, pH 7 en la ausencia (A) o presencia de 50 μm VimRod (B). Los paneles C y D son los espectros HSQC del mutante R1914E (100μm) en la ausencia o presencia de 50 μm VimRod, respectivamente. En grupo E el pico de relativo 1H -15N intensidades del PRD E con o sin el atascamiento de VimRod se muestran en función del número máximo, que es asignado arbitrariamente y no se basa en la posición de la secuencia. Estos valores pueden utilizarse para definir un atajo de importancia para la reducción de la intensidad de pico sobre la adición de un ligando. Si las tareas están disponibles, a menudo se observan valores significativos para asignar a una zona de Unión. Haga clic aquí para ver una versión más grande de esta figura.
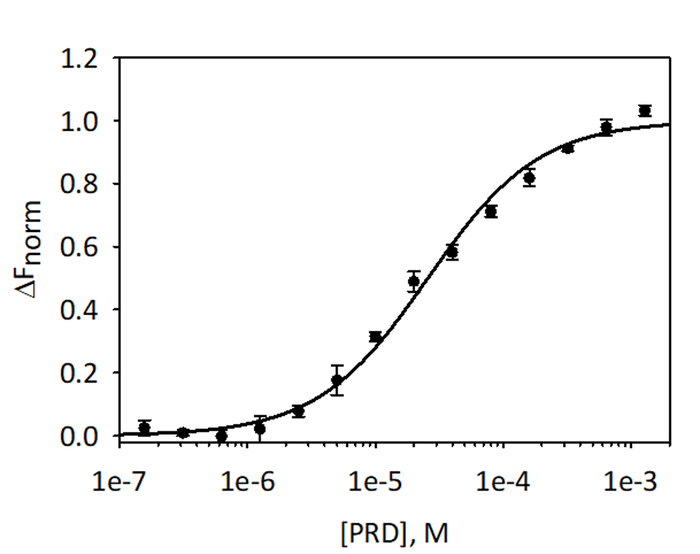
Figura 9: enlace de E-PRD a VimRod. E-PRD se diluyó en una serie de diluciones dobles de 1,28 mM a 39.1 nM y se incubaron con VimRod etiquetado antes de realizar el análisis de MST. Se combinaron los datos de tres ensayos independientes. Los datos se ajustaron a un modelo de KD dar un KD de 25,7 μM con una confianza de KD del μM ± 2.1. Haga clic aquí para ver una versión más grande de esta figura.
| Reactivo de | Cantidad |
| Fosfato de sodio, dibásico (anhidro) | 6,0 g |
| Fosfato de potasio, fosfato monobásico (anhidro) | 3.0 g |
| Cloruro de sodio | 0.5 g |
| H2O | Hasta 950 mL |
Tabla 1. Media M9 para el etiquetado isotópico.
| Reactivo de | Cantidad |
| 15 NH4Cl | 1.0 g |
| Glucosa (o 13C-glucosa) | 2.0 g |
| 1 M MgSO4 | 2 mL |
| 50 mM CaCl2 | 4 mL |
| 20 mg/mL tiamina | 1,0 mL |
| FECLAS de 3 mM3 | 400 ΜL |
| Metal Mix (cuadro 3) | 500 ΜL |
| H2O | Hasta 50 mL |
Tabla 2. Mezcla de nutrientes para medios de suplementación de M9.
| Reactivo de | Cantidad |
| 4 mM ZnSO4 | 323 mg |
| 1 mM MnSO4 | 75,5 mg |
| 4,7 mM H3BO3 | 145 mg |
| 0,7 mM CuSO4 | 55,9 mg |
| H2O | Hasta 500 mL |
Tabla 3. Metal Mix suplemento para enriquecer la mezcla de nutrientes de MT.
| Muestra | 1 mM E-PRD en tampón A1 (μL) | VimRod de 1mM en tampón A (μL) | Buffer (μL) | 200 μm DSS en D2O (μL) | Tampón B2 (μL) | Total volumen (μL) |
| E-PRD solo | 50 | 0 | 50 | 50 | 350 | 500 |
| E-PRD + VimRod | 50 | 50 | 0 | 50 | 350 | 500 |
| 1 Tampón A: 20 mM Tris-HCl, 1 mM TDT, pH 7 | ||||||
| 2 Tampón B: 23 mM Tris-HCl, 1,14 mM DTT, pH 7 | ||||||
Tabla 4. Preparación de la muestra de RMN.
Discusión
2D 15experimento NMR resuelto N es uno de los más utilizados métodos para mostrar cómo dos moléculas interactúan. Es el método más rica en información que permite que las señales de ambos socios continuamente monitorizada durante un experimento de titulación en estado de solución. Aunque típicamente cualitativo en el caso de grandes complejos, el método puede también utilizarse en casos favorables para medir las afinidades de enlace donde se pueden rastrear señales NMR en espectros de alta resolución. Donde hacen las asignaciones pueden ser convenientemente, como en el caso de muchas proteínas bajo 20 KD de tamaño, los sitios de Unión pueden también asignarse. Ensayos complementarios como MST proporcionan información cuantitativa sobre interacciones en solución y requieren menos proteína en Estados sin etiqueta. Comparación de datos de enlace mutante es útil para proporcionar controles para asegurar que las interacciones que se evidencia por la ampliación de la línea de NMR son genuinas y no artefactos de, por ejemplo, cambios de agregación o la viscosidad.
Expresión de la proteína
Agilizar el proceso de expresión reduce la cantidad de producción de proteína intensiva de mano de obra. Parte de este proceso de optimización consiste en la identificación de una cepa apropiada de e. coli para la expresión recombinante de la proteína. Preferencia de cepa depende de elementos como la naturaleza del vector en uso y, más específicamente, la máxima estabilidad de la proteína recombinante expresada7. El riesgo de degradación de la proteína heteróloga por endógeno e. coli pueden reducirse por el uso de proteasas proteasas deficiente e. coli como la cepa BL21. Para los genes que contienen codones raros, se puede preferir una cepa como RIPL BL21-CodonPlus (DE3). Esta variedad combina el carácter deficiente de la proteasa de la cepa BL21 con copias adicionales de endógenas de codón raro tRNAs para arginina, isoleucina, prolina y leucina. Alternativamente, codones raros que pueden comprometer la sobreexpresión pueden evitarse ordenando una construcción optimizada de codón de una fuente comercial. Muchas cepas de e. coli están disponibles para la expresión de genes recombinantes, cada uno optimizado para la elusión de un problema particular en la expresión7. En el caso de este estudio, la cepa deficiente BL21(DE3) de proteasa estándar produce cantidades suficientes de proteína soluble para la posterior purificación y análisis.
Purificación de proteínas
El protocolo de purificación de una proteína dada es a menudo único en el sentido que cada proteína sigue siendo estable y soluble en diferentes condiciones tales como temperatura, concentración de sales o pH. La eficacia global de purificación mediante cromatografía de afinidad también es sensible a la concentración de especies liberador como imidazol en varios pasos durante el proceso de purificación. En este trabajo, las condiciones de amortiguamiento crítico para IMAC fueron pH para el E-PRD, imidazol concentración y para la VimRod. Un pH de 7.5 fue necesario para evitar la precipitación del PRD E después de elución inicial de la columna IMAC. Para la purificación de la IMAC de VimRod, aumentando la concentración de imidazol de 30 a 50 mM durante la etapa de lavado de columna fue encontrado para tener una mejora sustancial en la pureza de las fracciones elución final. La etapa de elución, aumentando la concentración de imidazol desde 250 hasta 350 m m también se han encontrado para mejorar el rendimiento de la elución final. Intentos iniciales a fin de eluir proteínas usando imidazol 250 mM llevaron a elución incompleta de VimRod según lo revelado por una tira de imidazol de 1 M final de la columna (datos no mostrados). Aumento de la concentración de imidazol hasta 350 m m para la elución fue suficiente para recuperar todo de la proteína enlazada a la columna. S puede servir a un propósito dual porque actúa como un paso de pulido para la purificación de la proteína mientras que simultáneamente realiza cambio de buffer. Cambio de buffer es un paso crítico para el análisis subsecuente de la Unión ya que elimina el imidazólico utilizado a fin de eluir proteínas etiquetadas His6. También sirve como una oportunidad para cambiar las condiciones tales como concentración de sal o pH, que puede afectar la eficacia de ciertas técnicas de aguas abajo o ensayos. Proteína de cambio térmico (PTS) puede utilizarse para identificar reservas óptimo para ensayos de aguas abajo, especialmente para aquellos que requieren proteínas estables para períodos prolongados de tiempo a temperatura ambiente8,9.
Análisis de enlace
Proteína que recién preparada es fundamental para los ensayos de Unión exacta, aunque congelado proteínas también pueden usarse siempre y cuando los resultados son comparados. Proteínas filamentosas como vimentina multimerize de una sal y pH dependiente, y por lo tanto la solución condicionan la necesidad de ser optimizado y el estado oligomérico estimada por un método como SEC10,11, dispersión de luz dinámica 12 o Ultracentrifugación analítica13,14,15. Espectroscopia de RMN es ideal para la medición de las interacciones ligando de pequeñas proteínas a resolución atómica. Sin embargo, cuando una proteína interactúa con la molécula más grande, sobreviene de secado más lento, y esto resulta en la pérdida de las señales, que pueden confirmar vinculante aunque no necesariamente permiten la asignación de sitios, que también requiere asignación de por lo menos de Unión columna vertebral resonancias. En este escenario, los experimentos NMR no permiten identificación del sitio de interacción. Por lo tanto sitio dirigido mutagénesis se aplica para identificar los residuos críticos necesarios para el enlace. Por lo tanto, estos mutantes no exhiben pérdida de señal. En el presente Protocolo, una forma mutante con una substitución en la posición 1914 conserva la intensidad de pico en la presencia de VimRod y confirma, por tanto, la interrupción de la interacción de E-PRD y VimRod. Asignación de las resonancias de columna vertebral y sidechain añadiría valor a este enfoque, particularmente como se ha resuelto la estructura de la E gratis-PRD por cristalografía de rayos x4. Futuras aplicaciones de la RMN incluyen la caracterización de las interacciones complejas entre las moléculas más grandes y se beneficiará de ultra alta imanes de campo y el uso de otros grupos observables como 13C-etiquetados y trifluoro metil grupos como reporteros.
MST tiene un número de ventajas para el estudio de las interacciones de enlace16. Los socios de la Unión son libres en solución y no inmovilizados. Análisis de la calidad de las muestras está incorporado en el software con control de calidad informes de agregación, adsorción en los capilares o etiquetado fluorescente insuficiente de la molécula objetivo. Se emplean pequeñas cantidades de la blanco, la concentración de la etiqueta meta es generalmente entre 20-50 nM en un 10-20 μL volumen de reacción. Este protocolo utiliza volúmenes de reacción muy pequeños (10 μL) para maximizar la concentración de ligando que se logra en las valoraciones que permite interacciones de enlace débil ser caracterizado. Esto requiere un pipeteo preciso y cuidado para evitar la introducción de burbujas durante la mezcla aún. Una mezcla adecuada es fundamental para la medición precisa y consistente de la fluorescencia a lo largo de la serie de diluciones seriadas. La cantidad de Tween-20 en los experimentos de MST se redujo de un estándar de 0.05% al 0.015% para disminuir la tendencia a crear burbujas y mejorar la mezcla.
El tinte rojo-Tris-NTA proporciona una manera rápida, fácil y conveniente de fluorescencia etiqueta cualquier proteína que tiene una etiqueta. El etiquetado es con eficacia completo en sólo 30 minutos y está muy apretado para que ningún procedimiento de eliminación de tinte es necesario. No hay modificaciones a residuos del aminoácido en la proteína que puede alterar las propiedades de enlace de ligando. Una advertencia es que solamente la proteína a ser etiquetados como debe tener una etiqueta de His6. Esto requiere la ruptura de la etiqueta de la proteína ligando, E-PRD y la eliminación de la etiqueta y uncleaved E-PRD con un segundo paso de columna IMAC. Si es posible, se preparara la proteína ligando sin el uso de una etiqueta. Por otra parte, las proteínas pueden ser covalente etiquetados como con un fluoróforo a amina a residuos de lisina o tiol a residuos de cisteína. Sin embargo, se debe tener cuidado al uso de estos sistemas desde el accesorio covalente de un fluoróforo puede afectar las interacciones de enlace electrostático o polar basándose en residuos de lisina o cisteína. La cuantificación de atar afinidad entre el VimRod y el E-PRD por MST era inusualmente sensible a la concentración de la sal. Este problema fue mitigado por diálisis inicialmente el objetivo y el ligando en el mismo lote de tampón de ensayo. Sin embargo, saturación de la curva de enlace de MST no podría ser alcanzado cuando se realiza el ensayo de MST en presencia de 150 mM de NaCl debido al comportamiento complejo de VimRod. Se obtuvieron datos confiables, completados una vez que la concentración de NaCl se redujo a 10 mM que permite el cálculo exacto de KD. Por lo tanto, se recomienda cuidadosa optimización de las condiciones de la solución y comparación con ensayos complementarios para lograr resultados robustos. Además, el MST puede utilizarse para cuantificar la dependencia sal de una determinada interacción, cuantificar las propiedades estequiométricas de las interacciones entre proteínas, plegamiento de la proteína del monitor y sonda en enzima cinética17.
Divulgaciones
Los autores no divulgar conflictos de interés.
Agradecimientos
Este proyecto ha sido apoyado por NSERC RGPIN-2018-04994, programa de innovación Campus Alberta (RCP-12-002 C) e Instituto de investigación de Alberta del prión / Alberta Innova Bio Solutions (201600018), otorgado a M.O y genoma Canadá y Fundación de Canadá para Becas de innovación otorgados al centro de Metabolómica de innovación (TMIC) y NANUC.
Materiales
| Name | Company | Catalog Number | Comments |
| Monolith NT.115, includes control and analysis software | NanoTemper Technologies | MO-G008 | Instrument for microscal thermophoresis |
| His-Tag Labeling Kit RED-Tris-NTA | NanoTemper Technologies | MO-L008 | RED-tris-NTA dye for MST |
| Standard capillaries | NanoTemper Technologies | MO-K022 | capillaries for MST |
| HisTrap HP, 5 mL | GE Healthcare | 17524801 | IMAC column |
| HisPur Ni-NTA resin, 100 mL | Thermo Fisher Scientific | 88222 | IMAC resin |
| HiLoad 16/600 Superdex 75 pg | GE Healthcare | 28989333 | SEC column |
| TEV protease | Sigma Aldrich | T4455 | cleavage of his tag from E-PRD |
| BL21(DE3) Competent E. coli | New England Biolabs | C2527H | cells for protein expression |
| cOmplete Protease Inhibitor Cocktail Tablets EDTA-Free | Sigma Aldrich | 11873580001 | protease inhibitors for protein purification by IMAC |
| TCEP, Tris(2-carboxyethyl)phosphine hydrochloride | Sigma Aldrich | C4706 | reducing agent for protein purification |
| Reagents (HEPES, NaCl, etc) | Sigma Aldrich | various | Preparation of media and buffers |
| Ammonium chloride (15N, 99%) | Cambridge Isotope Laboratories | NLM-467 | isotope labelling for NMR |
| D2O, Deuterium Oxide (D, 99.8%) | Cambridge Isotope Laboratories | DLM-2259 | NMR sample preparation |
| DSS, Sodium 2,2-dimethyl-2-silapentane-5-sulfonate-D6 (D,98%) | Cambridge Isotope Laboratories | DLM-8206 | reference for NMR |
| Precision 5 mm NMR Tubes, 7” long | SJM/Deuterotubes | BOROECO-5-7 | NMR tubes |
| NMR spectrometer (14.1 Tesla) | Bruker | acquisition of NMR data | |
| TCI 5mm z-PFG cryogenic probe | Bruker | acquisition of NMR data | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Bruker TopSpin 4.0.1 | Bruker | processing of NMR data | |
| MO.Control | NanoTemper Technologies | included with Monlith NT.115 | |
| MO.Affinity Analysis | NanoTemper Technologies | included with Monlith NT.115 |
Referencias
- Vinogradova, O., Qin, J. NMR as a unique tool in assessment and complex determination of weak protein-protein interactions. Topics in Current Chemistry. 326, 35-45 (2012).
- McKercher, M. A., Wuttke, D. S. NMR Chemical Shift Mapping of SH2 Peptide Interactions. Methods in Molecular Biology. 1555, 269-290 (2017).
- Al-Jassar, C., Bikker, H., Overduin, M., Chidgey, M. Mechanistic Basis of Desmosome-Targeted Diseases. Journal of Molecular Biology. 425 (21), 4006-4022 (2013).
- Fogl, C., et al. Mechanism of intermediate filament recognition by plakin repeat domains revealed by envoplakin targeting of vimentin. Nature Communications. 7, (2016).
- Walker, J. M. . The Protein Protocols Handbook. , (2002).
- Wishart, D. S., et al. 1H, 13C and 15N chemical shift referencing in biomolecular NMR. J. Biomol. NMR. 6, 135-140 (1995).
- Casali, N. Escherichia coli Host Strains. E. coli Plasmid Vectors. , 27-48 (2003).
- Ericsson, U. B., Hallberg, B. M., DeTitta, G. T., Dekker, N., Nordlund, P. Thermofluor-based high-throughput stability optimization of proteins for structural studies. Analytical Biochemistry. 357 (2), 289-298 (2006).
- Kozak, S., et al. Optimization of protein samples for NMR using thermal shift assays. Journal of Biomolecular Nmr. 64, 281-289 (2016).
- Burgess, R. R. A brief practical review of size exclusion chromatography: Rules of thumb, limitations, and troubleshooting. Protein Expression and Purification. , (2018).
- Kunji, E. R. S., Harding, M., Butler, P. J. G., Akamine, P. Determination of the molecular mass and dimensions of membrane proteins by size exclusion chromatography. Methods. 46 (2), 62-72 (2008).
- Stetefeld, J., McKenna, S. A., Patel, T. R. Dynamic light scattering: a practical guide and applications in biomedical sciences. Biophysical Reviews. 8 (4), 409-427 (2016).
- Patel, T. R., Winzor, D. J., Scott, D. J. Analytical ultracentrifugation: A versatile tool for the characterisation of macromolecular complexes in solution. Methods. 95, 55-61 (2016).
- Pearson, J. Z., Cole, J. L., et al. Chapter One - Next-Generation AUC Adds a Spectral Dimension: Development of Multiwavelength Detectors for the Analytical Ultracentrifuge. Methods in Enzymology. 562, 1-26 (2015).
- Gorbet, G. E., Pearson, J. Z., Demeler, A. K., Cölfen, H., Demeler, B., Cole, J. L. Chapter Two - Next-Generation AUC: Analysis of Multiwavelength Analytical Ultracentrifugation Data. Methods in Enzymology. 562, 27-47 (2015).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nature Communications. 1, 100 (2010).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados