Method Article
核磁気共鳴分光法 (NMR) とマイクロ働く (MST) による球状と糸状のタンパク質の相互作用を測定
要約
ここで、安定同位体と核磁気共鳴 (NMR) 分光法とマイクロ スケールを用いたタンパク質間相互作用の後の評価と生産のラベルが付いている蛋白質の浄化のためのプロトコルを紹介します。働く (MST) 実験。
要約
ビメンチンなどの糸状のタンパク質は、ヘミデスモソーム斑を含んでいる蛋白質の結合サイトと構造の足場を提供することで細胞内組織の繰り返しを提供します。ここでは、検出し、このような相互作用を測定するためのプロトコルは、envoplakin の球状ヘミデスモソーム斑リピート ドメインおよびビメンチンのヘリカル コイルを使用して説明します。これは相互作用の親和性の測定、タンパク質結合ビメンチン (または同じような糸状の蛋白質) かどうかを決定するための基礎を提供します。興味の球状蛋白質は15N 標識し、ビメンチン タンパク質溶液で滴定します。二次元 NMR スペクトルは、化学シフトやピーク形状の変化を観察することによって相互作用を検出してビメンチン第四紀構造に影響を及ぼす塩のレベルを含むソリューションの条件の影響を明らかにする取得されます。興味の蛋白質のリガンドを糸状に、結合の相互作用は浄化された蛋白質を使用して MST によって定量化されます。アプローチは興味の蛋白質が、フィラメントをバインドするかどうかを決定しての相互作用に影響を与える突然変異やソリューションの条件などの変化を評価するための簡単な方法です。
概要
タンパク質間相互作用は、細胞内で順序を作成する分子機械の形成を許可します。個々 の相互作用は弱いことが多いが、通常協調と動的に調整することができます多価錯体に貢献。敏感な試金原子分解能およびそのような複雑な相互作用についての定量的な情報を提供するメカニズムを推測して薬のような分子などの介入を設計が必要です。NMR 分光学は蛋白質の相互作用の完全な情報を取得するための効率的な方法ですおよびまた含め弱1を結合する配位子の高速スクリーニングのために使用されます。使用される NMR 法は、タンパク質の観察や配位子を観察するに分類できます。本稿では、前者のアプローチは比較的小さい (通常 20 kDa) の下でタンパク質をラベルの安定同位元素のスペクトルを取得し、ラベルのない配位子は滴定を使用します。これにより、ラベル付きの残基は有利な例で、マップする相互作用に関与します。一度複雑なフォーム、ケミカル シフトの変化として表れる相互作用する残基の化学的環境とその NMR 信号の形状に変更があります。このような変化の範囲の相互作用でこれらのグループの関与の度合いと相関します。化学シフト摂動 (Csp) は、一連の不在とリガンドのさまざまな量の存在で収集された蛋白質の NMR スペクトルを比較することによって測定できます。大きな配位子や複雑な相互作用は、相互作用を推測するピーク形状や強度の変化を測定できます。
リガンド相互作用を検出するために使用される最も一般的な 2 D の実験は、 15N 25pxn-1 異核単一量子相関 (HSQC) 実験2です。1 つのタンパク質が15N、エシェリヒア属大腸菌細菌培養15N 濃縮メディアにし、バージョンのタグ付きの親和性として表現することによって達成される通常均一にラベルを付けることが必要です。バインディングは、明白な滴定の中に収集された HSQC スペクトルを重ね合せを明らかに複雑な形成に関わる残基のサブセットのピークの変化です。無料とリガンドで飽和状態の信号が 1 つの人口の平均ピークに崩壊高速為替制度の相互作用が発生します。また、状態の間遅い交換した場合では、両方の信号は彼らの相対的な量を表す積分で観察されます。NMR ラインシェイプ解析は、いくつかのケースで結合親和性を推定する使用できますが、MST などの方法も便利な証明しているし、本物の相互作用のクロス検証を提供します。
例は、デスモゾーム内で見つかった 2 つの蛋白質です。彼らは接合細胞表面と細胞骨格との間を仲介し、細胞接着装置と皮膚の整合性を維持するために中間径フィラメントの多価の相互作用を仲介して心臓組織との耐えるせん断力。Desmoplakin やビメンチンなど特異的蛋白質が変異や細胞接合の不安定化につながる自己抗体によって侵害され、そのための相互作用は非常に重要の3疾患が起因できます。デスモソーム タンパク質によるリガンド結合の構造基盤は、MST での相互作用を定量化することができますしながら NMR 分光法による特徴付けられます。ヘミデスモソーム斑リピート ドメイン (PRDs) 多くの場合提供する基本的な溝、タンデム セットとして存在しているとビメンチンのヘリカルによって提供される酸性表面を介して相互作用する中間径フィラメント間の相互作用を特徴付けるためのメソッド本4をバンドルします。これらの複合体は、デスモゾーム ティッシュを通して放射する接着のネットワークを形成、隣接する細胞に接続する細胞の細胞骨格の中間径フィラメントの停泊細胞膜で形成されています。
プロトコル
1. 組換え蛋白質の表現
-
Envoplakin PRD (E-PRD) およびビメンチン 99年-249 (VimRod) の発現
- 目的遺伝子を含むプラスミドを持つ大腸菌BL21(DE3) セルを変換します。100 μ g/mL アンピシリンを含む寒天培地プレート上のセルを広めます。37 ° C でプレートを一晩インキュベートします。
- 単一コロニーをピックアップし、プラスミッドの選択 100 μ g/mL アンピシリンを含む素晴らしいスープ (TB) の 20 mL を接種します。(180 rpm) を一晩中揺れを 37 ° C で文化を育てます。
- 全体 20 mL 文化を 50 μ G/ml のアンピシリンを含む TB の 1 L に転送します。外径 φ600まで 180 rpm で振とうしながら 37 ° C で文化をインキュベート = 0.6 0.8。
- 18 ° C に温度を減らし、最終濃度 1 mM にイソプロピル β-D-1-thiogalactopyranoside (IPTG) を追加することでタンパク質の発現を誘導します。蛋白質の表現を許可する一晩 160 rpm で振とうしながら 18 ° C で潜伏を続けてください。
- 8,000 の x g で 15 分間デカントで文化を遠心分離によって細胞を収穫し、上澄みを廃棄します。
- リン酸塩の約 40 mL の細胞ペレットを再で収穫された細胞緩衝食塩洗浄 (PBS: 20 mM リン酸緩衝、pH 7.4 では、120 mM の NaCl)。再懸濁を 50 mL チューブに転送します。8,000 の x g で再度遠心して、15 分間。
- デカントし、上澄みを廃棄します。すぐに浄化のプロトコルを開始または将来使用するための-20 ° C で細胞ペレットを凍結します。
-
同位体分類された蛋白質の発現
- 大腸菌由来BL21(DE3) 細胞を変形し、1.1.1-1.1.2 のように 20 mL スターター文化を準備します。
- 全体 20 mL 文化を追加 4.0 g トリプトン 5.0 g、塩化ナトリウム、100 μ g/mL アンピシリンを含む豊かな TB の 1 L に転送します。外径 φ600まで 160 rpm で振とうしながら 37 ° C で文化をインキュベート 1.6 1.9 を =。
- 15 分デカント 8000 × g で遠心分離によって 1 L 文化を収穫し、上澄みを廃棄します。
- 約 40 mL の PBS で優しく再細胞ペレットを洗浄し、再懸濁を 50 mL チューブに転送します。
- 再び 15 分デカントの 8,000 の x g で遠心し、上澄みを廃棄します。
- M9 最小媒体を (表 1) と 100 μ g/mL アンピシリンを含む M9 最小媒体の 950 mL の残りの部分への転送の 20 mL の細胞ペレットを再懸濁します。
- フィルター滅菌栄養組合せ (表 2 および 3) の 50 mL を追加します。
- 最終濃度 1 mM の IPTG を追加する前に 18 ° C、30 分に文化を慣らします。
- 160 rpm で振とうしながら 18 ° C で一晩インキュベートします。
- セクション 1.1.4-1.1.6 のように細胞を収穫します。
2. VimRod と E PRD の金属アフィニ ティー ・ クロマトグラフィー (IMAC) 精製を固定化
-
タグ付きの His6 VimRod の精製
- プロテアーゼ阻害剤カクテル欠けている EDTA を含む PBS の 5 mL/ト細胞ペレットを再懸濁します。次の手順でセル換散を改善するために Dounce 組織ホモジナイザの 12 ストロークで均質します。
- 氷の上を 1/1 の s のパルスで細胞懸濁液を超音波、1.5 分の合計の 80% 振幅超音波処理追加 2 回、過熱を防ぐために実行の間氷の上ゆっくり旋回を繰り返します。
- 75,000 x g で 45 分間デカントでサンプルを遠心し、シリンジ フィルター (0.45 μ m) を使用して上清をフィルターします。
- タンパク質の高速液体クロマトグラフィー (FPLC) システムを使用して 1 mL/分の流量で結合バッファー (20 mM HEPES、pH 7.5、500 mM の NaCl、10 mM のイミダゾール) の 5 列ボリューム (CV) と 5 mL の IMAC のコラムを平衡させ。
- 0.5 mL/min の流速で列にフィルター処理された上澄みをロードします。
- 5 CV の洗浄バッファー (20 mM HEPES、pH 7.5、500 mM の NaCl、50 mM のイミダゾール) 1 mL/min の流速で洗い流します。
- 3 タンパク質を溶出溶出バッファー (20 mM HEPES、pH 7.5、500 mM の NaCl、350 mM のイミダゾール) 0.5 mL/分収集 1.5 mL の一部分の流量での CV。可能な場合、溶出蛋白質の濃度を増加する最大フロー溶出モードを選択します。
- SDS-PAGE によって興味の蛋白質を含み、タンパク質濃度5を測定する標準的な方法を使用して FPLC クロマト グラムから分数を識別します。
- プール、2 mL を遠心限外濾過装置 (MWCO 3 kDa、5 mL) を使用して蛋白質の最高額を含む溶出画分を集中します。任意の沈殿物を削除し、0.22 μ m フィルターを通過する 21,000 × g で遠心分離機します。
- 2 S CV バッファー (20 mM HEPES、150 mM の NaCl、pH 7.5、0.5 mM TCEP)、FPLC を使用して 1 mL/分の流量で 120 mL サイズ排除クロマトグラフィー (S) コラムを平衡させ。
- 列に 2.1.9 から高濃度のタンパク質を注入し、流量を 0.5 mL/min、1 mL の一部分を収集で 1 S CV バッファーで溶出します。
- 前に、として興味の蛋白質を含有する画分を識別します。
- 最高の量のタンパク質を含有する画分をプールします。
- 短期的使用のための 4 ° C で保存または 20% と小さい因数で-80 ° C で保存場所にグリセリンを追加します。
-
削除 His6 タグで E PRD タンパク質の精製
- 2.1.1-2.1.8 His6 タグ E PRD 蛋白質を浄化するための手順に従います。ピーク画分をプール、タンパク質濃度を決定します。
- タバコを追加でプールされた蛋白質の 2 μ L/mg のウイルス (TEV) プロテアーゼ (1 mg/mL) をエッチングします。透析の管 (6 kDa) に移し、4 ° C で一晩 S バッファー内 dialyzeこの手順では、His6 タグの胸の谷間と、次のステップで Ni NTA 樹脂へのバインドに干渉イミダゾールの除去をことができます。
- 3 S CV バッファーを持つ重力列で Ni NTA 樹脂 5 mL を平衡します。樹脂から余分なバッファーをドレインします。
- 樹脂に劈開 E PRD タンパク質を注ぎ、分解されていないタグ付きの His6 の E-PRD と劈開の His6 タグをバインドするを許可するロッキング プラットフォームで 1 時間インキュベートします。TEV のプロテアーゼは His6 タグはまた、樹脂をバインドします。タグ無料 E-PRD を含む、流れを収集します。E PRD のすべてを確保するため洗浄 2 S CV バッファーで樹脂を回収します。
- 遠心限外濾過装置 (MWCO 3 kDa、5 mL) を使用して 2 mL に流れの E-PRD を集中します。任意の沈殿物を削除し、0.22 μ m フィルターを通過する 21000 × g で遠心分離機します。
- 2 S CV バッファー (20 mM HEPES、150 mM の NaCl、pH 7.5、0.5 mM TCEP MST または 20 mM トリス-HCl、1 mM DTT、NMR 用 pH 7)、FPLC を使用して 1 mL/分の流量での 120 mL S 列を平衡します。
- 列に 2.2.5 から集中 E PRD タンパク質を注入し、0.5 mL/min、1 mL の一部分を収集の流量で 1 S CV バッファーで溶出します。
- 前に、として興味の蛋白質を含有する画分を識別します。
- 最高の量のタンパク質を含有する画分をプールします。
- 短期的使用のための 4 ° C で保存または 20% と小さい因数で-80 ° C で保存場所にグリセリンを追加します。
3. NMR 法
- NMR サンプル準備
- 15N 標識野生型または 20 ミリメートル トリス-HCl、1 mM DTT、pH 7 ステップ 2.2.6 S バッファーとしてを使用して前述のよう R1914E E-PRD 蛋白質を浄化します。蛋白質の貯蔵液は通常約 1 mL のボリュームで 1 mm 0.3 から範囲です。
注: 蛋白質に集中することができます > 100 μ M 試料の適切な範囲への集中をもたらすため MWCO 3 kDa、5 mL 遠心限外濾過装置を使用しています。 - 20 mM トリス-HCl、1 mM DTT、pH 7 ステップ 2.1.10 S バッファーとしてを使用してラベルの VimRod 蛋白質のサンプルを浄化します。
- 500 μ L の最終巻、最終濃度 100 μ m、20 μ M の最終濃度 10% (v/v)、および DSS (4,4-dimethyl-4-silapentane-1-sulfonic 酸) の最終的な集中に重水素酸化物 (D2O) に野生型と変異体の E-PRD タンパク質を追加持参500 μ L までサンプル ボリューム 20 ミリメートル pH 7 1 mM DTT、トリス-HCl を使用しています。代表的なサンプル準備は表 4で説明します。
注: 0 ppm DSS の共鳴1H 化学シフトを調整するために使用し同様、タンパク質6シフト15N 化学の間接参照するため。D2O は、一定の純磁気フィールドで動作分析計を維持する重水素ロック信号に使用されます。 - 前の手順のように O と DSS を E-PRD、D2の 2 番目のサンプルを作り、500 μ L にボリュームを持って来る前に 50 μ M の最終的な集中に VimRod を追加します。
- 500 μ L のサンプルを実験用 5 mm 広い NMR チューブに転送します。
- 15N 標識野生型または 20 ミリメートル トリス-HCl、1 mM DTT、pH 7 ステップ 2.2.6 S バッファーとしてを使用して前述のよう R1914E E-PRD 蛋白質を浄化します。蛋白質の貯蔵液は通常約 1 mL のボリュームで 1 mm 0.3 から範囲です。
- NMR 実験のセットアップ
- イジェクト コマンド"ej"; と空気の流れをオンにこれは磁石からサンプルを持ち出すでしょう。今、開始によって磁石の上にスピナーでサンプルを配置し、コマンド"ij"と挿入します。サンプルが進む前に磁石の内部安定するまでを待ちます。
- 「Edc」コマンドを使用して新しいデータセットを作成し、"ZGPR"(図 1) 実験をクリックして標準の1H-NMR パラメーターを読み込みます。名前、EXPNO (実験数) および PROCNO (処理されたデータ フォルダー番号) フィールドに入力します。「溶媒を設定」フィールドに溶媒を選択し、標準的な probehead と溶媒依存性 (prosol) パラメーターを読み取れません「'getprosol' を実行する」をクリックします。
- サンプルで、重水素化溶媒、すなわちD2O をロック、「ロック」コマンドを使用しそれが完了するまで待つにスイープしロックを実現。
- 「アートマ」コマンドの自動チューニングを使用してサンプルをチューニングすることによって磁石の共振周波数を修正します。自動調整が完了するまでは、ウォブル曲線を監視します。
- TOPSHIM (コマンド"topshim") を使用して磁場をシムします。Shim は、サンプルのまわりの均一性を達成するために磁場を調整のプロセスです。コマンド「wsh」と shim 値を格納およびそれらを読むことをお勧め同じまたは同じようなサンプルを使用して場合 topshim 前に"rsh の"を使用しています。
- 最大信号対雑音比を達成するために「rga」コマンドでレシーバーの感度を調整します。
- 水共振オフセット (o1) のスペクトルの中心を置き、高出力"calibo1p1"を使用して 90 度プロトン パルス (p1) を設定します。
- 陽子のエネルギー スペクトルのゼロを使用して行く「zg」コマンドと"efp"指数乗算 ("em")、線幅を組み込む自由誘導減衰 (FID)、"フィート"プロセスで収集フェーズを適用する FID と"pk"のフーリエ変換補正。
- 自動フェーズ補正"apk"と"absn"統合オプションを使用せず多項式を用いた自動ベースライン補正を適用します。
- SOFAST 非実験 (3.2.2) のように新しいデータセットを作成するには、実験で"SFHMQC3GPPH"を選択します。
- コピー陽子のエネルギー スペクトルから P1 と O1 を最適化し、コマンド"getprosol 1 H p1 plw1"、p1 は最適化された P1 の値、plw1、P1 のパワー ・ レベルを使用して、P1 依存パルス。
- アミド化学シフトのオフセットを設定する CNST54 定数を最適化し、するレシーバーの感度を可能にする興味のスペクトル領域を包含するために帯域幅を定義する CNST55 最適化 (図 2)。これらのパラメーターを選択するには、二次元スペクトルから最初の FID (自由誘導減衰) を抽出し、それらを定義する, 観測信号を探します。さらに、緩和遅延 (D1)、スキャン (NS) とダミー スキャン コマンド"gs"と許容可能な信号感度を得るため (DS) を有効に行くとリアルタイムでデータの品質を監視するスキャンの数をによって異なります。
- ゼロ「zg」Go を使用してスペクトルを記録します。
- NMR データ処理
- 直接 F2 のサイズに処理パラメーターを設定 (1H) と間接 F1 (15N) スペクトルによる寸法"SI F2 = 2048、F1 = 512"間接ディメンション (図 3) でオプションの線形予測を用いた。
- 窓関数として"QSINE"を選択し、二次元スペクトルを処理する 2 の正弦ベル シフト (SSB) を入力します。
- ウィンドウ関数とフーリエ変換と双方向でデータを処理するコマンド"xfb"を入力します。
- コマンド"apk2d"を使用して、両方向で自動フェーズ補正を実施します。自動プロセスは、位相補正の満足のいくレベルを達成しない場合、「rser」コマンドで Fid を抽出、1 次元処理から相値を計算し、2 D データに適用。
- 2 D データの自動ベースライン補正機能「abs2」とベースラインを修正します。これは処理パラメーターで定義されている ppm 値間多項式関数を適用し、さらなる分析のための 2次元スペクトルになります。
- 別の分子との相互作用データの比較のためシリアル処理を実行する場合は、コマンド"wpar"と処理パラメーターを保存し、リコール「rpar」。すべてのデータセットが同じパラメーターとバリエーションで処理されますこの方法では導入されません処理方法の違いのため。
- NMR データ分析
- ピーク ピッキングのプロセスを開始するコマンド"pp"を入力します。
- 予想されるピーク (図 4) に基づくピークの ppm の範囲と最小強度/最大数を定義します。[Ok] をクリックし、目視検査による結果を確認します。必要な場合は、スペクトル品質を基に処理結果が満足されるまで再実行します。
- "Pp の"コマンドで実測を生成します。
メモ: この実測既定データ高さ/ピーク強度情報を含まれていますと後続のスペクトルにエクスポートすることができます、他のプログラムで読み取ることができます。 - 別の分子との相互作用を示すタンパク質 HSQC スペクトルにおける化学シフトの動きやピーク強度の変化を観察します。相互作用する分子のサイズが大きい場合は、いくつかのピークの消失と共にピーク強度で削減を期待します。
- ピーク ウィンドウで右クリックで「ピーク」タブをクリックして、「インポート」を選択することによって次のデータ セットにピーク リストをインポートします。
- スペクトルのピークを可視化する新しい位置にそれらを移すために必要な場合。一覧」の「強度をリセットする」をクリックして (図 5) の強度とスペクトル実測を生成します。このピーク ・ リストは、ストアドのピーク ・ リストから位置情報に持ち越されます。
- 「エクスポート」機能を選択して別のデータセットからスプレッドシートまたは他の数学的な解析プログラムのピークのリストをエクスポートします。
- 関数「各ピークのタンパク質スペクトルの複雑なスペクトル/ピーク強度のピーク強度」とピーク強度の変化を計算します。値は、掛け算の 100 の変化率に変換できます。ピーク強度が比較的広いピークの高密度タンパク質の場合は、通常、お互いの近くに配置されているピークの測定は容易ですが、ピーク ボリュームは便利なもに注意してください。
4. マイクロ働く (MST)
-
Ligand 蛋白質の E PRD の準備
- 1 l 20 mM HEPES、pH 7.5、10 mM の NaCl、一晩ゆっくりと 4 ° C で攪拌の中断 3.5 kDa ミニ透析室におけるタンパク質の 800 μ L までおこし、MST 互換性のあるバッファーにリガンドを交換します。
- 14 000 x g で 10 分間クリーン チューブへの転送高濃度タンパク質で遠心分離遠心限外濾過ユニット (MWCO 3 kDa) を用いたリガンドを集中します。
- 21,000 × g 10 分間に存在するリガンドを遠心し、慎重に任意の沈殿したタンパク質を削除する新しいチューブに上清を転送します。280 吸光度を用いたリガンドの濃度を決定する nm と配位子吸光係数。10% 追加トゥイーン-20 0.015% の最終的な集中を与えるため。トゥイーン 20 は、毛細血管への吸着を防ぐためにアッセイバッファーに追加されます。MST の実験に使用される最終的なアッセイ バッファーが 20 mM HEPES、pH 7.5、10 mM の NaCl、0.015% Tween 20。
-
ターゲットの色素標識タンパク質 VimRod の作製
- 1x PBS T が 5 μ M の濃度を与える赤トリス NTA に付属の 50 μ L を追加することで赤トリス NTA 染料を再構成します。200 μ L のチューブに 2 μ 因数を省略し、-20 ° C で保存
- アッセイバッファーで 0.34 μ m までターゲット蛋白質を希釈します。赤トリス NTA 色素の 2 μ 因数にターゲットの 58 μ L を追加し、室温で 30 分間インキュベートします。染め分類されたターゲットの最終濃度は 0.33 μ M です。10 分 21,000 x g でラベル付けされたターゲットを遠心し、慎重に任意の沈殿物を削除する新しいチューブに上清を転送します。赤トリス NTA 色素は His6 タグによるタンパク質に結合し、結合解離定数 (KD) は、サブ ナノモル範囲。それは効果的に 100% ターゲット蛋白質に連結されているので、さらに浄化する必要はありません。
- MST の楽器、コントロール ソフトウェアを開きます。赤トリス NTA 色素の赤の設定を選択します。25 ° c. に温度制御をオンに
- ターゲットのラベルを検証する予備を選択し、集約やキュヴェットへの吸着を確認します。これらのパラメーターの評価が自動的に提供されます。
- ミックス 17 μ L アッセイバッファーのターゲット蛋白質のピペッティングによるミックス 3 μ L。希薄化後のターゲットのそれらを浸すことと毛細血管の中心に液体を描画によって 2 つの標準的な毛細血管を記入してください。トレイと楽器には、毛細血管を配置します。測定を開始します。
- 蛍光の十分なレベルと吸着 (キャピラリー ライン形状の歪み) の兆候を探して結果を確認または集計 (MST トレースの歪み) ソフトウェアの分析でフラグが設定されます。結果が肯定的な移動場合手順、そうでない場合は別のバッファーにリガンドを試して必要があります。 または 0.05% 以上、トゥイーン 20 の量を増やすことができます。
-
E PRD 配位子の 2 倍希釈系列の作製
- 16 の分類によって希釈系列を調製する 1 月 16 日から 200 μ L チューブ。
- Tube1 に必要な最大リガンド濃度よりも高い濃度 × 1.17 でリガンドの 17 μ L を追加します。このボリュームは、2 倍量必要 (8.5 μ L)、因数は次の管を直列に転送されます。アッセイの最終巻は、ターゲット蛋白質の VimRod の 1.5 μ L 添加による 1 倍に希釈され 1.17 x リガンド濃度 8.5 μ 10 μ L です。選択この最大濃度は、20 回以上推定 KDの値をする必要があります。
- Tubes2-16 新しいピペット チップを用いた各因数にアッセイバッファーの 8.5 μ L を追加します。ピペット チップを再利用 (正確な分注を参照してください製造元の指示に関するアドバイス) の精度に影響することができます。ゆっくりと泡を生成することがなく、アッセイバッファーにソリューションをリリース Tube2 に Tube1 からリガンドの 8.5 μ L を転送します。泡を発生させることがなく、上下に少なくとも 6 回を再びにピペッティングによるリガンドとバッファーをミックスします。最も集中管 E PRD 1.5 mM, ラベル付きの VimRod 50 の最終濃度に存在していた nM。
- Tube2 からリガンドの 8.5 μ L を Tube3 に転送し、アッセイバッファー ミックスします。シリアル希釈を繰り返してすべてのチューブを持っている配位子を追加します。すべてのチューブの 2 倍希釈系列のリガンドの 8.5 μ L が含まれるように Tube16 から 8.5 μ L を破棄します。
-
MST と結合反応実験の準備
- チューブのそれぞれにラベル付きターゲット蛋白質の 1.5 μ L を追加、軽く泡を避けるように注意しながら上下ピペッティングで混ぜます。15 分間インキュベートします。
- 結合の試金のためのエキスパート モードを選択し、シリアル希釈系列のパラメーターに入力します。温度制御の設定を確認してください 25 ° C、励振電力 40% と MST 電源を媒体に設定されています。これらのパラメーターは、研究されている他の結合パートナーの最適化する必要があります。
- 結合反応で毛細血管を埋めるし、キャピラリーのトレイに配置。楽器にトレイをロードは、温度 25 ° C を取り戻すし、測定を開始するを待ちます。
-
データ分析
- アフィニ ティー解析ソフトウェアでバインディング アッセイ ファイルを開きます。キャピラリーのスキャンを確認します。蛍光レベル平均値からの 10% 以上に変わらないように、セクション 3.6 で説明されているようにする必要がありますが検出されない集計または吸着。
- 右側のパネルに MST 分析を選択して、分析データを分析セットにドラッグします。同じ条件下で同じターゲット-リガンド結合アッセイ法の複数の実行がある場合は、同じセットにドロップすることによって結合できます。また、実行のたびにドロップできるない解析独立。
- データのグラフを用量応答フィット] タブに移動します。単一のバインディング サイトが予想される場合は、KDモデルを選択します。丘モデルはまた協調行動を持つ複数の結合サイトのためのオプションです。品質管理を通過しなかった外れ値のポイントは、この段階でフィットから削除できます。マージ複数のアッセイとセットが平均してエラーは標準偏差として計算されます。
- 最後のタブを開き、1 つのグラフのすべてのプロット間で結果を比較します。生成されるテーブルから各曲線の近似の結果を取得します。必要な場合に、データまたは近似曲線を他のプレゼンテーションや分析ソフトウェアにエクスポートします。
結果
人間 envoplakin 遺伝子と VimRod ドメイン (99-249 pET21a にクローン残基) の E-PRD ドメイン (pProEX HTC に複製残留 1822-2014 年) 人間の vimentin の4 His6 タグで表現され、精製します。図 6と図 7は、VimRod の純度のレベルを示す (18.8 kDa) と E PRD (21.8 kDa) は蛋白質の浄化のこの方法から得られます。E PRD コンストラクトのタグは VimRod 蛋白質は His6 タグ結合色素を使用してラベル付けし、染料の結合、His6 タグを保持すべての E-PRD を奪い合います MST 実験に不可欠な His6 の除去。TEV プロテアーゼ、劈開のタグと、分解されていない His6-E-PRD 残った TEV プロテアーゼとタグの胸の谷間の後 2 番目の IMAC のコラムを削除します。浄化の最終的な研磨段階は、サイズ排除クロマトグラフィーです。にもかかわらず、両方蛋白質同じようなサイズの E PRD 溶出ピークはこのサイズのタンパク質モノマーが期待される 72 mL を中心としながら、VimRod を 51 mL の列で示してください。VimRod のサイズの明らかな増加は可能性が高いために、糸状の長い棒とその特性形蛋白質の解析的遠心実験実証単量体4を VimRod にいた。タンパク質の低い利回りは、M9 最小媒体で生産されて細胞の低い量のため豊富な流体培養基からのそれらより栽培文化から取得されます。TB で M9 の準備のための大きいスターター文化の初期生育では、 15N NMR 実験に必要なラベルの範囲を維持しながら細胞収率の改善をことができます。
15n-1H HSQCs に E PRD R1914E 変異体と野生型の VimRod (図 8 a-8 D) の有無で買収されました。図 8 aに E PRD のスペクトルは、うまく解決ピーク、適切に折り畳まれたタンパク質を示す数の期待値を示しています。VimRod (図 8 b) 存在下では、スペクトルは、広範なラインを広げると E-PRD と VimRod 間のバインディングに対応するピーク消失を示しています。このバインディングは、図 8 と 8 Dの比較では明らかに R1914E の突然変異によって失われます。小さな変更は、この突然変異体 E PRD と VimRod 間のバインドの欠如を示す R1914E 変異体 VimRod の添加時にスペクトルで観察されます。VimRod の有無で E PRD のピーク強度を比較し、図 8 e、ピーク拡がり E PRD 複合体の範囲を示す相対ピーク強度としてプロットされます。(図示せず) E PRD R1914E 変異株は野生型 (図 8 e) の約 20% に比べると VimRod の存在下で 20% またはより高いピーク強度のピークの約 97% を保持しました。研究4であって機能点変異体、また中間の効果を持っている追加の突然変異体の損失を表します。
39.1 まで 1.28 mM から E PRD リガンドの濃度を減少させると混合を検証し、ターゲットとして赤トリス NTA 色素の付いた His6 VimRod を使用して VimRod と E-PRD MST 分析のバインディングを量的に nM を行った。3 つのバインディングの滴定を行い、結果は平均、図 9に示します。データは 1 サイト リガンド結合の標準モデルとピッタリでした、25.7 ± 2.1 μ M の KDを与えた。表面プラズモン共鳴による VimRod と E-PRD の間結合の評価は、19.1 ± 1.3 μ m 帯4のような KD値を与えた。

図 1: NMR 実験のセットアップのスクリーン キャプチャします。HSQC のデータセットを収集するために標準的な実験を設定するウィンドウが表示されますが使用されます。実験パラメーターが読み込まれる実験に隣接します。示されている ZGPR の実験は、標準および溶媒依存陽子パラメーターを読み込む初期実験として選択されます。タイトル ウィンドウを使用して、記録保持の目的のための実験の詳細を入力します。HSQC スペクトルを収集するには、ZGPR の実験は SFHMQC3GPPH に置き換えられます。この図の拡大版を表示するのにはここをクリックしてください。
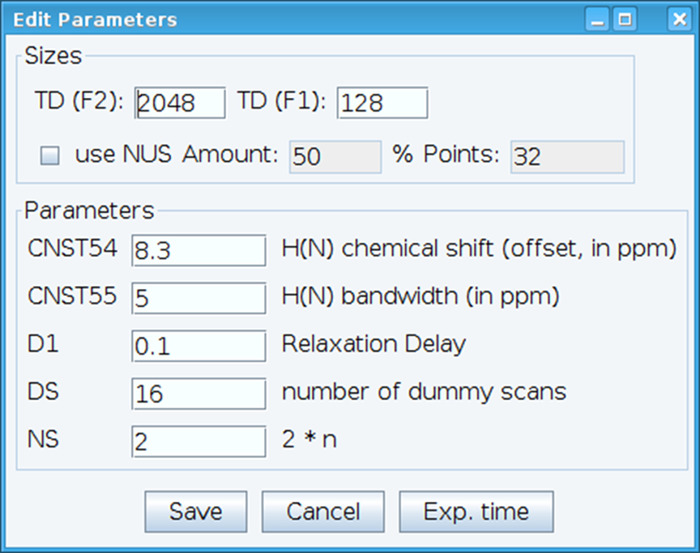
図 2: NMR 実験パラメーターを調整します。ウィンドウが表示されますは、NMR のパルス シーケンスの基本パラメーターを入力するため信号を最適化するために使用されます。この図の拡大版を表示するのにはここをクリックしてください。

図 3: NMR データ処理します。通常調整されるそれらを表す矢印を持つ各 NMR スペクトルの 2 つのディメンションを処理するために使用されるパラメーターが表示されます。この図の拡大版を表示するのにはここをクリックしてください。
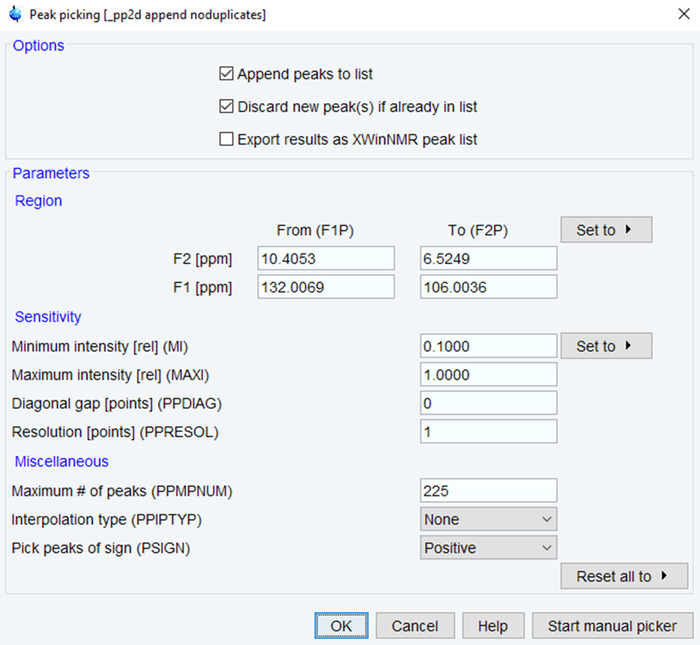
図 4: NMR ピーク選択するためのパラメーターです。加工の NMR スペクトルで NMR ピークを拾うために使用パラメーターは、典型的な値で表示されます。Ppm の範囲、強度およびスペクトルを最適化するためにピークの数を調整します。この図の拡大版を表示するのにはここをクリックしてください。
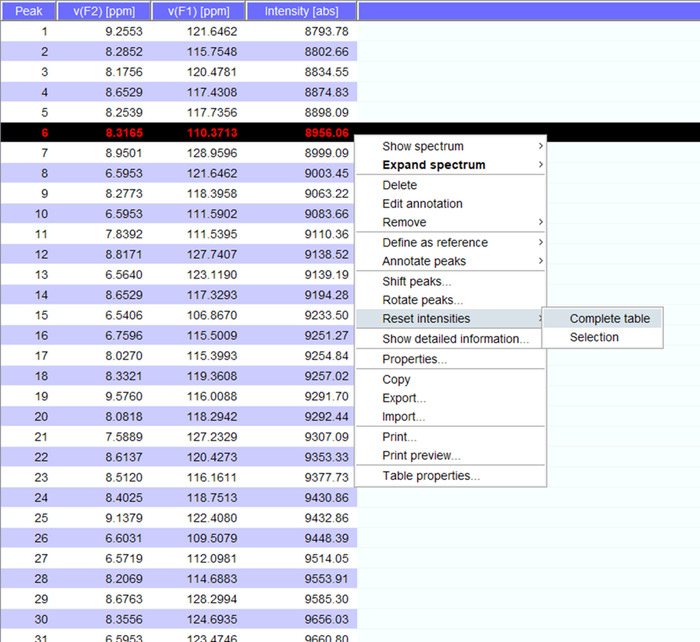
図 5: 代表実測強度と。数値、およびその1H NMR スペクトルで選んだそれぞれのピークがあると15N 化学シフト、信号強度が表示されます。この実測は、相互作用のパートナーの有無で得られたスペクトルを比較する使用できます。この図の拡大版を表示するのにはここをクリックしてください。
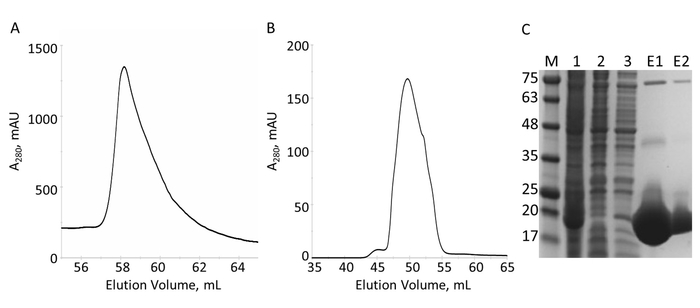
図 6: IMAC と s. A. His6 タグ VimRod の精製IMAC のコラムからの溶出のクロマト グラムは、VimRod の 1 つの主要なピークを示しています。B. S カラムからの溶出のクロマト グラムは、1 つの主要なピークを示しています。浄化の過程で収集した分数の C. SDS-PAGE: プールのゲル (M) 細胞ライセート (1)、IMAC の流れを通して (2)、洗浄 (3)、プールされた溶出 (E1)、左に kDa S 溶出 (E2) で MW と MW 基準が示されました。E1 と E2 のレーンで高い分子量で目に見えるバンドは、西部のしみ (データは示されていない) によって確定した純粋な VimRod のオリゴマーです。この図の拡大版を表示するのにはここをクリックしてください。
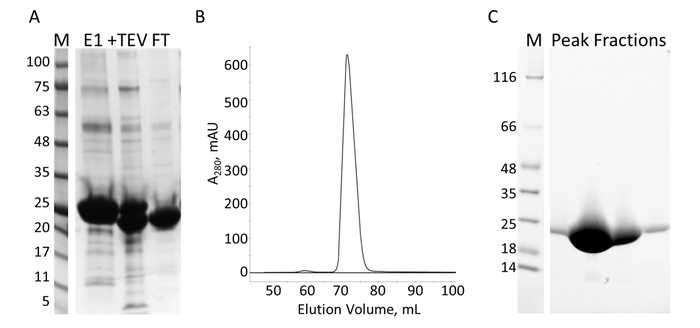
図 7: IMAC と s. A. E PRD の精製MW に分子量の基準を示す IMAC 浄化の SDS ページは、最初 IMAC 列 (E1)、TEV 胸の谷間製品 (+ TEV) から kDa のゲル (M) と溶出液の左側に示されている、2 番目の IMAC のコラム (フィート) から流れる。B. S 列からクロマト グラフでは、1 つの主要なピークを示しています。C. SDS-PAGE 分子量規格 (M) の S のピークからの分数。この図の拡大版を表示するのにはここをクリックしてください。
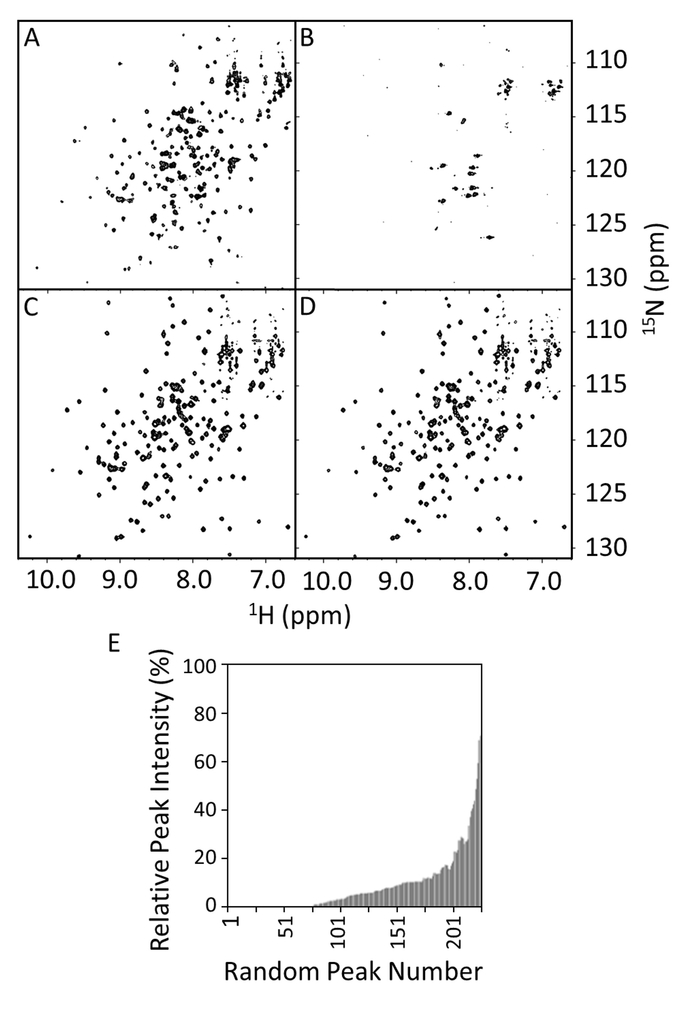
図 8: 野生型および E-PRD 存在下での R1914E 変異体と VimRod の欠如の HSQC スペクトル。HSQC スペクトル表示野生型 E PRD (100 μ M) で 20 mM のトリス-HCl、150 mM の NaCl、1 mM DTT、pH 7 (A) 有無 50 μ M の VimRod (B)。C と D のパネル 50 μ M の有無が HSQC スペクトル R1914E 変異体 (100 μ m) の VimRod、それぞれ。パネル E 相対1H-15N ピークの強度 E PRD VimRod バインディングの有無は任意に割り当てられた、シーケンスの位置に基づいていないピーク数の関数として表示されます。これらの値は、配位子の添加によってピーク強度低減の意義カットオフを定義する使用できます。割り当てが利用可能な場合バインド領域にマップする重要な値をよく見かけることができます。この図の拡大版を表示するのにはここをクリックしてください。
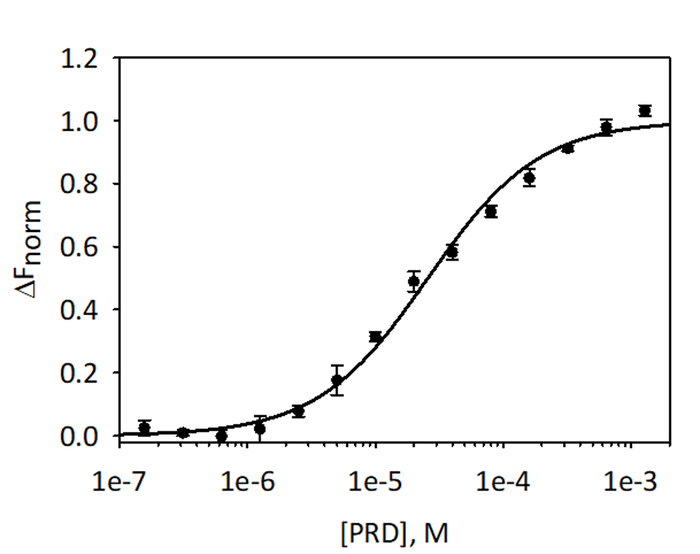
図 9: VimRod E-珠江デルタのバインドします。E PRD は 39.1 まで 1.28 mM から 2 倍希釈のシリーズで希釈した nM とラベル付き VimRod と MST 分析を実行する前に。3 つの独立した測定からのデータを結合しました。データは、μ m ± 2.1 KD自信を持って 25.7 μ M の KDを与える KDモデルにピッタリだった。この図の拡大版を表示するのにはここをクリックしてください。
| 試薬 | 数量 |
| 二塩基性リン酸ナトリウム (無水) | 6.0 g |
| リン酸-カリウム (無水) | 3.0 g |
| 塩化ナトリウム | 0.5 g |
| H2O | 950 mL |
表 1。M9 媒体を同位体の分類のため。
| 試薬 | 数量 |
| 15NH4Cl | 1.0 g |
| グルコース (または13C グルコース) | 2.0 g |
| 1 M MgSO4 | 2 mL |
| 50 mM CaCl2 | 4 mL |
| 20 mg/mL チアミン | 1.0 mL |
| 3 mM した FeCl3 | 400 Μ L |
| 金属のミックス (表 3) | 500 Μ L |
| H2O | 50 mL |
表 2。栄養補充の M9 媒体ミックス。
| 試薬 | 数量 |
| 4 mM ZnSO4 | 323 mg |
| 1 mM MnSO4 | 75.5 mg |
| 4.7 mM H3ボー3 | 145 mg |
| 0.7 mM CuSO4 | 55.9 mg |
| H2O | 500 mL |
表 3。メタル ミックス MT 栄養組合せを豊かにするサプリメント。
| サンプル | 1 mM E PRD バッファー A1 (μ L) | 1 mM VimRod バッファー A (μ L) | バッファー (μ L) | 200 μ M DSS は D2O (μ L) | バッファー B2 (μ L) | 合計量 (μ L) |
| E-PRD だけで | 50 | 0 | 50 | 50 | 350 | 500 |
| E PRD + VimRod | 50 | 50 | 0 | 50 | 350 | 500 |
| 1バッファー a: 20 mM トリス-HCl、1 mM DTT、pH 7 | ||||||
| 2バッファー b: 23 mM トリス-HCl、1.14 mM DTT、pH 7 | ||||||
表 4。NMR サンプル準備。
ディスカッション
2 D の15N 分解 NMR 実験は最も広く使用されているの一つか 2 つの分子を表示するメソッドのやり取り。両方のパートナーの信号ソリューション状態で滴定実験を通して継続的に監視することができる最も情報豊富な方法です。通常質的に大きな複合体の場合、メソッドも使える有利な例の結合親和性を測定する NMR 信号を高分解能スペクトルで追跡できます。割り当ては、便利に行うことができます、場所など 20 kDa のサイズの下で多くの蛋白質の場合結合部位もマップできます。MST など補完的な試金ソリューションでは、相互作用に関する定量的な情報を提供、ラベルのない状態のより少ない蛋白質を必要とします。変異バインド データの比較は NMR 線によって証明される相互作用が本物であることを確認するためのコントロール、たとえば、集計や粘度の変化のない成果物を提供するために役立ちます。
蛋白質の表現
労働集約的なタンパク質を合成する量が減ります式プロセスの合理化します。この最適化プロセスの一部には、組換えタンパク質発現のためのエシェリヒア属大腸菌の適切な株の識別が含まれます。使用するベクターの性質などの要素に依存するひずみの好みより具体的には、組換え蛋白質の究極の安定性を表明7。危険性内因性大腸菌による異種タンパク質の分解のプロテアーゼをプロテアーゼの使用によって減らすことが欠損大腸菌BL21 系統など。珍しいコドンを含む遺伝子、BL21 CodonPlus (DE3) RIPL のひずみは好まれるかもしれない。この株は、アルギニン、イソロイシン、プロリン、ロイシンのまれなコドン Trna の内因性コピーと BL21 系統のプロテアーゼ欠乏性質を兼ね備えています。また、商業ソースからコドン最適化構築の発注によって過剰発現を危険にさらすことができるまれのコドンを避けることができます。組換え遺伝子の発現はエシェリヒア属大腸菌の多くの菌株、式7の中に特定の問題の回避に最適各。この研究の場合は、標準プロテアーゼ欠損株 BL21(DE3) はそれに続く浄化および分析のため可溶性タンパク質の十分な量を生産しました。
蛋白質の浄化
特定の蛋白質の浄化のプロトコルは各蛋白質のまま安定しており、温度、塩分濃度、pH などさまざまな条件の下で溶けるという意味でユニークな多くの場合。アフィニ ティー ・ クロマトグラフィーによって精製の全体的な効果はまた精製過程など様々 なステップでイミダゾール溶出種の濃度に敏感。今回、IMAC の重要なバッファー条件は E-PRD の pH と、VimRod のイミダゾールの集中だった。7.5 の pH は、IMAC のコラムから次の初期溶出 E PRD の沈殿物を避けるために必要でした。VimRod の IMAC 浄化、最終的な溶出画分の純度で大幅な改善をして発見されたカラム洗浄ステップの間に 30 から 50 mm のイミダゾールの濃度を増加させます。溶出のステップのまた最終の溶出の収量を改善するために発見された 250 から 350 mm のイミダゾールの濃度を増加させます。250 mM のイミダゾールを用いたタンパク質を溶出する初期の試みは、列 (データは示されていない) の最終的な 1 M イミダゾール ストリップによって明らかにされた VimRod の不完全な溶出をもたらした。溶出用 350 mm のイミダゾールの集中の増加は、列にバインドされている蛋白質のすべてを回復するのに十分だった。同時にバッファー交換を実行中蛋白質の浄化のための研磨段階として機能するため、S は二重目的に役立つことができます。His6 タグ付きタンパク質を溶出に使用されるイミダゾールを削除しますので、バッファー交換はその後結合解析のための重要なステップです。また、塩分濃度や特定の下流のテクニックやアッセイの有効性に影響を与える可能性があります pH などの条件を変更する機会として機能します。タンパク質の安定なタンパク質を必要とするそれらのために特に下流の試金のための最適なバッファーを識別するために温度変化 (PTS) を使用できますに長時間室温8,9時。
結合解析
結果比較されます限り、固定された蛋白質を使用もできますが、正確なバインディング アッセイには作りたてのタンパク質が欠かせません。塩、pH 依存の方法でビメンチン multimerize など糸状のタンパク質、したがって解決条件を最適化する必要性とオリゴマーの状態推定秒10、など11、動的光散乱法による12または分析超遠心法13,14,15。NMR 分光法による原子分解能で小さな蛋白質のリガンド相互作用を測定に適しています。ただし、大きい分子と相互作用タンパク質と遅いタンブリングのすさまじい、この結果バインディングを確認できますが、それは必ずしも、信号の損失、許可の結合部位も少なくともの割り当てに必要となるマッピング バックボーン共鳴。このシナリオでは、NMR 実験に相互作用部位の同定はできません。それ故にサイトは、突然変異誘発は、連結に必要な重要な残基を識別するために適用を指示しました。したがって、このような突然変異体には信号の損失は起きません。このプロトコルでは 1914 位置置換変異フォームは VimRod の存在下でピーク強度を保持し、したがって E PRD と VimRod の相互作用の中断を確認します。バックボーンと側鎖の共鳴の割り当ては、無料 E-PRD の構造は x 線結晶構造解析4で解決されている、特にとしてこのアプローチに値を追加します。NMR の将来のアプリケーションより大きな分子間の複雑な相互作用の特性が含まれます、超高い分野磁石や13など他の観察可能なグループの使用の恩恵 C 標識と記者としてのみメチル グループ。
MST には、結合相互作用16の勉強のための利点の数があります。結合パートナーがソリューションで自由でない固定化です。サンプルの質の分析は品質管理報告の集計、毛細血管や標的分子の不十分な蛍光標識への吸着とソフトウェアに組み込まれます。ターゲットの少量が用いられます、ラベル付けされたターゲットの濃度は通常 10-20 μ L のボリューム/反応で 20 ~ 50 nM の間。このプロトコルは、特徴付けられる弱い結合相互作用を許可する滴定で実現できるリガンドの濃度を最大化する、非常に小さな反応ボリューム (10 μ L) を使用します。これは、正確なピペッティングおよびまだ十分に混合しながら気泡を導入することを避けるために注意が必要となります。適切な混合シリアル希薄のセットに沿って、一貫性のある正確な蛍光測定が重要です。MST の実験でトゥイーン 20 の量が標準的な泡を作成、混合を強化する傾向を下げるために 0.015% 0.05% から減少しました。
赤トリス NTA 染料に彼を持つタンパク質を蛍光ラベルを簡単迅速かつ便利な方法が用意されていますタグ。ラベルがわずか 30 分で効果的に完了し、色素除去する必要はありません、非常にタイトです。リガンド結合特性を変えるかもしれない蛋白質のアミノ酸残基には、変更は行われません。注意点は、His6 タグをラベルを付けるだけのタンパク質が必要です。これは配位子蛋白質や E-PRD、タグと 2 番目の IMAC 列ステップで分解されていない E PRD の除去からタグの胸の谷間が必要です。可能であれば、ligand 蛋白質は彼の使用せず準備されるべきタグです。また、リジン残基またはチオールのシステイン残基に結合する結合アミンでの蛍光と共有蛋白質を分類する可能性があります。ただし、リジンまたはシステイン残基に依存する静電または極性結合の相互作用に影響を与える可能性があります、fluorophore の共有結合以来このようなシステムを使用して注意が必要があります。結合 VimRod と E-PRD MST での親和性の定量化は、塩濃度に異常に敏感だった。この問題は、当初アッセイバッファーの同じバッチにターゲットとリガンドをおこしによって軽減されました。それにもかかわらず、MST 結合曲線の彩度は、VimRod の複雑な挙動に起因する 150 ミリメートル塩化ナトリウムの存在下で MST 分析を実行するときにない達成できます。KDの正確な計算を許可する 10 mm NaCl の濃度を下げた後、信頼性の高い、完全なデータが得られました。したがって、慎重に最適化ソリューションの条件と相補的な試金との比較は堅牢な結果を達成するためにお勧めします。また、特定の相互作用のための塩濃度依存性を定量化、酵素反応速度論17に蛋白質の相互作用、モニター タンパク質の折り畳み、およびプローブの化学量論的特性を定量化する MST を使用可能性があります。
開示事項
著者は利益相反を開示ないです。
謝辞
このプロジェクトは、アルバータ州プリオン研究所、キャンパス アルバータ州イノベーション プログラム (RCP-12-002 C) レベル RGPIN-2018-04994 に支えられてきた M.O とゲノムのカナダのためのカナダの財団にアルバータ州革新バイオ ソリューション (201600018) を受賞/革新のメタボロミクス技術革新センター (TMIC) と NANUC に授与された補助金。
資料
| Name | Company | Catalog Number | Comments |
| Monolith NT.115, includes control and analysis software | NanoTemper Technologies | MO-G008 | Instrument for microscal thermophoresis |
| His-Tag Labeling Kit RED-Tris-NTA | NanoTemper Technologies | MO-L008 | RED-tris-NTA dye for MST |
| Standard capillaries | NanoTemper Technologies | MO-K022 | capillaries for MST |
| HisTrap HP, 5 mL | GE Healthcare | 17524801 | IMAC column |
| HisPur Ni-NTA resin, 100 mL | Thermo Fisher Scientific | 88222 | IMAC resin |
| HiLoad 16/600 Superdex 75 pg | GE Healthcare | 28989333 | SEC column |
| TEV protease | Sigma Aldrich | T4455 | cleavage of his tag from E-PRD |
| BL21(DE3) Competent E. coli | New England Biolabs | C2527H | cells for protein expression |
| cOmplete Protease Inhibitor Cocktail Tablets EDTA-Free | Sigma Aldrich | 11873580001 | protease inhibitors for protein purification by IMAC |
| TCEP, Tris(2-carboxyethyl)phosphine hydrochloride | Sigma Aldrich | C4706 | reducing agent for protein purification |
| Reagents (HEPES, NaCl, etc) | Sigma Aldrich | various | Preparation of media and buffers |
| Ammonium chloride (15N, 99%) | Cambridge Isotope Laboratories | NLM-467 | isotope labelling for NMR |
| D2O, Deuterium Oxide (D, 99.8%) | Cambridge Isotope Laboratories | DLM-2259 | NMR sample preparation |
| DSS, Sodium 2,2-dimethyl-2-silapentane-5-sulfonate-D6 (D,98%) | Cambridge Isotope Laboratories | DLM-8206 | reference for NMR |
| Precision 5 mm NMR Tubes, 7” long | SJM/Deuterotubes | BOROECO-5-7 | NMR tubes |
| NMR spectrometer (14.1 Tesla) | Bruker | acquisition of NMR data | |
| TCI 5mm z-PFG cryogenic probe | Bruker | acquisition of NMR data | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Bruker TopSpin 4.0.1 | Bruker | processing of NMR data | |
| MO.Control | NanoTemper Technologies | included with Monlith NT.115 | |
| MO.Affinity Analysis | NanoTemper Technologies | included with Monlith NT.115 |
参考文献
- Vinogradova, O., Qin, J. NMR as a unique tool in assessment and complex determination of weak protein-protein interactions. Topics in Current Chemistry. 326, 35-45 (2012).
- McKercher, M. A., Wuttke, D. S. NMR Chemical Shift Mapping of SH2 Peptide Interactions. Methods in Molecular Biology. 1555, 269-290 (2017).
- Al-Jassar, C., Bikker, H., Overduin, M., Chidgey, M. Mechanistic Basis of Desmosome-Targeted Diseases. Journal of Molecular Biology. 425 (21), 4006-4022 (2013).
- Fogl, C., et al. Mechanism of intermediate filament recognition by plakin repeat domains revealed by envoplakin targeting of vimentin. Nature Communications. 7, (2016).
- Walker, J. M. . The Protein Protocols Handbook. , (2002).
- Wishart, D. S., et al. 1H, 13C and 15N chemical shift referencing in biomolecular NMR. J. Biomol. NMR. 6, 135-140 (1995).
- Casali, N. Escherichia coli Host Strains. E. coli Plasmid Vectors. , 27-48 (2003).
- Ericsson, U. B., Hallberg, B. M., DeTitta, G. T., Dekker, N., Nordlund, P. Thermofluor-based high-throughput stability optimization of proteins for structural studies. Analytical Biochemistry. 357 (2), 289-298 (2006).
- Kozak, S., et al. Optimization of protein samples for NMR using thermal shift assays. Journal of Biomolecular Nmr. 64, 281-289 (2016).
- Burgess, R. R. A brief practical review of size exclusion chromatography: Rules of thumb, limitations, and troubleshooting. Protein Expression and Purification. , (2018).
- Kunji, E. R. S., Harding, M., Butler, P. J. G., Akamine, P. Determination of the molecular mass and dimensions of membrane proteins by size exclusion chromatography. Methods. 46 (2), 62-72 (2008).
- Stetefeld, J., McKenna, S. A., Patel, T. R. Dynamic light scattering: a practical guide and applications in biomedical sciences. Biophysical Reviews. 8 (4), 409-427 (2016).
- Patel, T. R., Winzor, D. J., Scott, D. J. Analytical ultracentrifugation: A versatile tool for the characterisation of macromolecular complexes in solution. Methods. 95, 55-61 (2016).
- Pearson, J. Z., Cole, J. L., et al. Chapter One - Next-Generation AUC Adds a Spectral Dimension: Development of Multiwavelength Detectors for the Analytical Ultracentrifuge. Methods in Enzymology. 562, 1-26 (2015).
- Gorbet, G. E., Pearson, J. Z., Demeler, A. K., Cölfen, H., Demeler, B., Cole, J. L. Chapter Two - Next-Generation AUC: Analysis of Multiwavelength Analytical Ultracentrifugation Data. Methods in Enzymology. 562, 27-47 (2015).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nature Communications. 1, 100 (2010).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved