Method Article
Mesurer les Interactions des protéines globulaires et filamenteuses par spectroscopie de résonance magnétique nucléaire (RMN) et thermophorèse a petite Echelle (MST)
Dans cet article
Résumé
Nous présentons ici un protocole pour la production et la purification des protéines qui sont marqués avec des isotopes stables et ultérieure caractérisation des interactions protéine-protéine à l’aide de la spectroscopie de résonance magnétique nucléaire (RMN) et micro-échelle Expériences de thermophorèse (MST).
Résumé
Protéines filamenteuses Comme vimentine fournissent répète d’organisation au sein de cellules en fournissant un échafaudage structurels avec les sites permettant de lier des protéines contenant des plakin. Ici, un protocole pour la détection et la mesure de ces interactions est décrite en utilisant le domaine globulaire plakin d’envoplakin et la bobine hélicoïdale de vimentine. Cela fournit une base pour déterminer si une protéine lie vimentine (ou protéines filamenteuses similaires) et mesure de l’affinité de l’interaction. La protéine globulaire d’intérêt est étiquetée avec 15N et titrée avec la vimentine protéine en solution. Un spectre de RMN bidimensionnel est acquis pour détecter des interactions en observant les changements dans la forme de pic ou déplacements chimiques et élucider les effets des conditions de solution, y compris les niveaux de sel, qui influencent la structure quaternaire de vimentine. Si la protéine d’intérêt lie le ligand filamenteux, la réaction de liaison est quantifiée par MST en utilisant les protéines purifiées. L’approche est un moyen simple pour déterminer si une protéine d’intérêt lie à un filament et d’évaluer l’incidence des altérations, comme les mutations ou les conditions de la solution, sur l’interaction.
Introduction
Interactions entre les protéines permettent la formation des machines moléculaires qui créent l’ordre à l’intérieur des cellules. Les interactions individuelles sont souvent faibles mais contribuent généralement à multivalents complexes qui peuvent être coopératif et dynamiquement réglementé. Les épreuves sensibles qui fournissent la résolution atomique et des informations quantitatives sur ces interactions complexes sont nécessaires pour déduire les mécanismes et concevoir des interventions comme la drogue-comme des molécules. Spectroscopie RMN est une méthode efficace pour obtenir les informations sur les interactions entre protéines et est aussi utilisée pour le dépistage rapide des ligands y compris ceux qui se lient faiblement1. Les méthodes de RMN utilisés peuvent être classés en ceux qui sont les protéines observer ou observer de ligand. Ce manuscrit utilise l’ancienne approche dans laquelle un spectre d’une protéine qui est relativement faible (généralement sous 20 kDa) marquée des isotopes stables est acquise et le ligand non étiqueté est titré. Ceci permet les résidus étiquetées impliqués dans l’interaction à mapper dans les cas favorables. Une fois les formes complexes, il y a des changements dans les environnements chimiques des résidus interdépendants qui se manifestent par des changements dans le déplacement chimique et la forme de leurs signaux de RMN. L’ampleur de ces changements est corrélée avec le degré d’implication de ces groupes dans l’interaction. Perturbations de déplacement chimique (EFPC) peuvent être mesurées en comparant une série de spectres RMN de la protéine recueillies en absence et en présence de quantités variables du ligand. Pour les plus gros ligands ou interactions complexes, le changement de forme de la pointe ou l’intensité peut être mesuré pour déduire des interactions.
L’expérience 2D couramment utilisé pour la détection des interactions ligand est le 15N-hétéronucléaire corrélation (HSQC) quantique unique expérience2. Cela exige qu’une protéine être uniformément étiquetés avec 15N, qui est généralement réalisé en les exprimant en tant que versions affinité-tag dans les cultures bactériennes Escherichia coli en 15médias enrichis en N. La liaison est apparente lorsque les spectres HSQC recueillis durant le titrage sont superposent, révélant les changements de pointe pour un sous-ensemble de résidus impliqués dans la formation du complexe. L’interaction peut se produire dans le régime d’échange rapide où les signaux de l’État libre et saturée en ligand sombrer dans une population moyenne de crête. Par ailleurs, dans le cas d’échanges lents entre les États, les deux signaux est observés avec intégrales qui représentent leurs quantités relatives. Analyse lineshape RMN peut être utilisés pour estimer les affinités de liaison dans certains cas, des méthodes telles que MST se sont également révélés commodes et fournissent une validation croisée de véritables interactions.
L’exemple fourni est de deux protéines retrouvées dans les desmosomes. Ils véhiculent des jonctions entre les surfaces de la cellule et le cytosquelette et médient MULTIVALENTES interactions entre les machines d’adhérence cellulaire et des filaments intermédiaires pour maintenir l’intégrité de la peau et les tissus cardiaques et résister à des forces de cisaillement. Maladies peuvent se produire lorsque desmosomes protéines comme desmoplakine ou vimentine sont compromises par les mutations ou les auto-anticorps, conduisant à la déstabilisation des jonctions cellule-cellule, et leurs interactions sont donc d’une importance cruciale,3. La base structurelle de liaison du ligand par des desmosomes protéines peut être caractérisée par la spectroscopie RMN, tandis que les interactions peuvent être quantifiées en MST. Méthodes ci-après ont été utilisées pour caractériser les interactions entre domaines de répéter plakin (PRDs) qui sont souvent présents sous forme d’ensembles de tandem qui offrent des rainures base et vimentine, un filament intermédiaire qui interagit à travers une surface acide offerte par son hélice forfait4. Ces complexes sont formées à la membrane cellulaire où ils l’ancre pour les filaments intermédiaires du cytosquelette cellulaire aux desmosomes qui se connectent à des cellules adjacentes, formant ainsi un réseau de liaisons adhésifs qui rayonne dans un tissu.
Protocole
1. recombinant Protein Expression
-
Expression de Envoplakin PRD (E-PRD) et la vimentine 99-249 (VimRod)
- Transformer les cellules d’e. coli BL21 (DE3) avec le plasmide contenant le gène désiré. Étaler les cellules sur milieu gélosé contenant 100 ampicilline µg/mL. Incuber les plaques à 37 ° C pendant la nuit.
- Prélever une colonie unique et inoculer 20 mL de bouillon formidable (TB) contenant le 100 µg/mL ampicilline à sélectionner pour le plasmide. Cultiver la culture à 37 ° C sous agitation (180 tr/min) pendant la nuit.
- Transférer la culture entière 20 mL à 1 L de la tuberculose contenant 50 ampicilline µg/mL. Incuber la culture à 37 ° C sous agitation à 180 tr/min jusqu'à ce que l' OD600 = 0,6 à 0,8.
- Réduire la température à 18 ° C et induisent l’expression de la protéine en ajoutant l’isopropyl β-D-1-thiogalactopyranoside (IPTG) à une concentration finale de 1 mM. Continuer d’incubation à 18 ° C sous agitation à 160 tours/minute pendant la nuit afin de permettre l’expression de la protéine.
- Récolter les cellules par centrifugation de la culture à 8 000 x g pendant 15 min. décanter et éliminer le surnageant.
- Laver les cellules récoltées par resuspendant le culot cellulaire dans environ 40 mL de phosphate solution saline tamponnée (PBS : un tampon phosphate 20 mM, pH 7,4, 120 mM NaCl). Transférer la remise en suspension dans un tube de 50 mL. Centrifuger à nouveau à 8 000 x g pendant 15 min.
- Décanter et éliminer le surnageant. Immédiatement commencer le protocole de purification ou geler les granules cellulaires à-20 ° C pour une utilisation future.
-
Expression de la protéine isotopiquement étiqueté
- Transformer des cellules e. coli BL21 (DE3) et préparer une culture de départ de 20 mL comme dans 1.1.1-1.1.2.
- Transférer la culture entière 20 mL à 1 L de la tuberculose enrichi contenant un supplémentaires 4,0 g tryptone 5,0 g de NaCl et 100 ampicilline µg/mL. Incuber la culture à 37 ° C sous agitation à 160 tr/min jusqu'à ce que l' OD600 = 1,6 à 1,9.
- La récolte de la culture de 1 L par centrifugation à 8000 x g pendant 15 min. décanter et éliminer le surnageant.
- Laver le culot cellulaire par doucement resuspendant dans environ 40 mL de PBS et transférer la remise en suspension dans un tube de 50 mL.
- Centrifuger à nouveau à 8 000 x g pendant 15 min. décanter et éliminer le surnageant.
- Resuspendre le culot dans 20 mL de milieu minimal M9 (tableau 1) et transfert vers le reste de la 950 mL de milieu minimal M9 contenant 100 ampicilline µg/mL.
- Ajouter 50 mL du mélange nutritif filtre stérilisé (tableaux 2 et 3).
- S’acclimater à la culture à 18 ° C pendant 30 min avant d’ajouter l’IPTG à une concentration finale de 1 mM.
- Incuber pendant la nuit à 18 ° C sous agitation à 160 tours/minute.
- Récolter les cellules comme dans l’article 1.1.4-1.1.6.
2. immobilisé Metal Affinity Chromatography (IMAC) Purification de VimRod et E-PRD
-
Purification de His6-le tag VimRod
- Resuspendre le culot dans 5 mL/g de PBS contenant un EDTA manque cocktail inhibiteur de protéase. Homogénéiser avec 12 coups dans un homogénéisateur de tissu Dounce afin d’améliorer la lyse de la cellule à l’étape suivante.
- Sur la glace, laisser agir la suspension cellulaire à une impulsion de 1 s à 1/s éteint, amplitude de 80 % pour un total de 1,5 min. répéter la sonication supplémentaires deux fois, en agitant doucement sur la glace entre les pistes pour éviter la surchauffe.
- Centrifuger l’échantillon à 75 000 x g pendant 45 min. décanter et filtrer le surnageant à l’aide d’un filtre de seringue (0,45 µm).
- Equilibrer une colonne d’IMAC de 5 mL avec 5 volumes de colonne (CV) de tampon de liaison (20 mM HEPES, pH 7.5, 500 mM NaCl, imidazole de 10 mM) à un débit de 1 mL/min à l’aide d’un système de chromatographie en phase liquide (FPLC) protéine rapide.
- Charger le surnageant filtré dans la colonne à un débit de 0,5 mL/min.
- Laver la colonne avec le tampon de 5 CV de lavage (20 mM HEPES, pH 7.5, 500 mM NaCl, imidazole de 50 mM) à un débit de 1 mL/min.
- Éluer la protéine avec 3 CV du tampon d’élution (20 mM HEPES, pH 7.5, 500 mM NaCl, imidazole 350 mM) à un débit de fractions de 0,5 mL/min. recueillir 1,5 mL. Le cas échéant, sélectionnez le mode de débit ascendant d’élution d’augmenter la concentration de protéine éluée.
- Identifier les fractions du chromatogramme FPLC qui contiennent la protéine d’intérêt par SDS-PAGE et utilisent des méthodes normalisées pour mesurer la concentration de protéine5.
- Piscine et concentrer les fractions d’élution contenant les plus fortes teneurs en protéines en utilisant un dispositif centrifuge ultrafiltration (MWCO 3 kDa, 5 mL) à 2 mL. Centrifuger à 21 000 g pour enlever n’importe quel précipité et passer à travers un filtre de 0,22 µm.
- Equilibrer une colonne de chromatographie (S) exclusion de taille de 120 mL avec le tampon de CV de S 2 (20 mM HEPES, 150 mM NaCl, pH 7.5, 0,5 mM TCEP) à un débit de 1 mL/min à l’aide d’un FPLC.
- Injecter le concentré de protéine de 2.1.9 sur la colonne et éluer avec 1 tampon CV de S à un débit de 0,5 mL/min, rassemblant des fractions de 1 mL.
- Identifier les fractions contenant la protéine d’intérêt, comme avant.
- Mettre en commun les fractions contenant la protéine de montants plus élevée.
- Conserver à 4 ° C pour usage à court terme ou ajouter glycérol à 20 % et à conserver à-80 ° C dans de petites parties aliquotes.
-
Purification de la protéine E-PRD avec la balise His6 supprimée
- Suivez les étapes de 2.1.1-2.1.8 pour purifier la protéine E-PRD-le tag His6. Mettre en commun les fractions de pic et déterminer la concentration de protéine.
- Ajouter tabac etch protéase de virus (TEV) (1 mg/mL) à 2 µL/mg de protéine mis en commun. Transfert à la dialyse tube (6 kDa) et de dialyser dans un tampon S toute la nuit à 4 ° C. Cette étape permet le clivage de la balise His6 et l’enlèvement de l’imidazole qui interfère avec la liaison à la résine de Ni-NTA à l’étape suivante.
- Equilibrer les 5 mL de résine Ni-NTA dans une colonne de gravité avec le tampon de CV de S 3. Égoutter l’excès de tampon de la résine.
- Versez la protéine E-PRD clivée sur la résine et incuber pendant 1 heure sur une plate-forme bascule pour permettre le PRD-His6-tag E clivé et la balise His6 clivée lier. La protéase TEV est également His6-tag et va se pour lier à la résine. Recueillir le débit à travers, qui contient la balise-free E-PRD. Laver la résine avec un tampon 2 CV de S pour s’assurer que tous le E-PRD est récupéré.
- Concentré de E-PRD dans le flux par le biais de 2 mL à l’aide d’un dispositif centrifuge ultrafiltration (MWCO 3 kDa, 5 mL). Centrifuger à g 21 000 pour enlever n’importe quel précipité et passer à travers un filtre de 0,22 µm.
- Equilibrer une colonne de 120 mL S avec un tampon 2 CV de S (20 mM HEPES, 150 mM NaCl, pH 7.5, 0,5 mM PTCE pour MST ou 20 mM Tris-HCl, 1 millimètre DTT, pH 7 pour NMR) à un débit de 1 mL/min à l’aide d’un FPLC.
- Injecter la protéine E-PRD concentrée de 2.2.5 sur la colonne et éluer avec 1 tampon CV de S à un débit de 0,5 mL/min, rassemblant des fractions de 1 mL.
- Identifier les fractions contenant la protéine d’intérêt, comme avant.
- Mettre en commun les fractions contenant la protéine de montants plus élevée.
- Conserver à 4 ° C pour usage à court terme ou ajouter glycérol à 20 % et à conserver à-80 ° C dans de petites parties aliquotes.
3. méthodes de RMN
- Préparation des échantillons de la RMN
- Purifier 15N-étiqueté sauvage ou une protéine R1914E E-PRD comme décrit précédemment, à l’aide de 20 mM Tris-HCl, 1 millimètre DTT, pH 7 comme le tampon de S pour étape 2.2.6. Solutions courantes de protéines se situent généralement entre 0,3 à 1 mM avec des volumes d’environ 1 mL.
Remarque : La protéine peut être concentré à > 100 µM en utilisant un MWCO 3 kDa, dispositif centrifuge ultrafiltration 5 mL pour abaisser la concentration en une gamme appropriée pour la préparation de l’échantillon. - Purifier un échantillon de protéine de VimRod sans étiquette à l’aide de 20 mM Tris-HCl, 1 millimètre DTT, pH 7 comme le tampon de S pour étape 2.1.10.
- Dans un volume final de 500 µL, ajoutez la protéine E-PRD sauvage ou mutante à une concentration finale de 100µM, oxyde de deutérium (D2O) à une concentration finale de 10 % (v/v) et DSS (acide 4,4-dimethyl-4-silapentane-1-sulfonic) à une concentration finale de 20 µM. Bring le volume de l’échantillon jusqu'à 500 µL avec 20 mM Tris-HCl, 1 millimètre DTT, pH 7. Une préparation de l’échantillon représentatif est décrit dans le tableau 4.
Remarque : Le 0 ppm résonance du Mas est utilisée pour calibrer les déplacements chimiques de 1H ainsi que pour le référencement indirect de la substance chimique 15N décale de la protéine6. D2O est utilisé pour le signal de verrouillage du deutérium pour garder le spectromètre opérant à un champ magnétique net constant. - Forment un second échantillon de E-PRD, D2O et DSS comme dans l’étape précédente et ajoutez VimRod à une concentration finale de 50 µM, avant de porter le volume jusqu'à 500 µL.
- Transférer les échantillons de 500 µL à un tubes de NMR large de 5 mm pour l’expérience.
- Purifier 15N-étiqueté sauvage ou une protéine R1914E E-PRD comme décrit précédemment, à l’aide de 20 mM Tris-HCl, 1 millimètre DTT, pH 7 comme le tampon de S pour étape 2.2.6. Solutions courantes de protéines se situent généralement entre 0,3 à 1 mM avec des volumes d’environ 1 mL.
- RMN du montage expérimental
- Allumez le débit d’air avec la commande eject « ej » ; Cela fera apparaître l’échantillon de l’aimant. Maintenant, placer l’échantillon dans un cône d’hélice sur le dessus de l’aimant par l’ouverture et l’insérer avec la commande « ij ». Attendez que l’échantillon s’installe à l’intérieur de l’aimant avant de continuer.
- Créez un nouveau dataset à l’aide de la commande « edc » et charge standard 1H RMN paramètres en sélectionnant experiment « ZGPR » (Figure 1). Renseignez les champs nom de, EXPNO (numéro de l’expérience) et PROCNO (numéro de dossier de données traitées). Sélectionnez le solvant dans le champ « Set solvant » et cliquez sur « Exécuter « getprosol » » pour lire la sonde standard et des paramètres dépendant du solvant (prosol).
- Verrouiller l’échantillon à la deutéré solvables, c'est-à-dire, D2O, à l’aide de la commande « verrouiller » et attendez qu’il est complètement balayant et réalise la serrure.
- Corriger la fréquence de résonance de l’aimant par le réglage de l’échantillon à l’aide de la commande de syntonisation automatique « atma ». Surveiller la courbe de wobble jusqu'à ce que le réglage automatique est terminé.
- Caler le champ magnétique à l’aide de TOPSHIM (commande « topshim »). Calage est le processus d’ajustements au champ magnétique à l’obtention d’une uniformité autour de l’échantillon. Il est conseillé de stocker les valeurs de cale avec la commande « wsh » et les lire à l’aide de « rsh » avant topshim, si vous utilisez les échantillons identiques ou similaires.
- Ajuster le gain du récepteur avec la commande « rga » pour atteindre maximale du signal / bruit.
- Placer le centre du spectre sur le décalage de résonance de l’eau (o1) et la valeur de l’impulsion de proton de 90 degrés (p1) à forte puissance à l’aide de « calibo1p1 ».
- Recueillir le spectre du proton en utilisant le zéro aller « zg » commande et processus avec « efp », qui comprend la multiplication exponentielle (« em »), la décomposition de l’induction libre (FID) intégrant l’élargissement de la ligne, « ft » transformée de Fourier de la FID et « pk » d’appliquer la phase correction.
- Appliquer la correction automatique de phase « apk » et la correction automatique de la base « RSEA » en utilisant le polynôme sans l’option d’intégration.
- Créer un nouveau dataset (comme en 3.2.2) pour l’expérience SOFAST HMBC en sélectionnant « SFHMQC3GPPH » dans l’expérience.
- Copie optimisée P1 et O1 de spectre protonique et remplir P1 impulsions dépendantes à l’aide de la commande « getprosol 1H p1 plw1 », où p1 est la valeur de P1 optimisée et plw1 est le niveau de puissance pour P1.
- Optimiser la constante CNST54 pour définir le décalage pour les déplacements chimiques amide et CNST55 pour définir la bande passante afin d’englober les régions d’intérêt qui permet le gain du récepteur à être optimisé (Figure 2). Pour sélectionner ces paramètres, extrait la première FID (free induction decay) le spectre bidimensionnel et chercher le signal observé pour les définir. De plus, varier le délai de relaxation (D1), nombre d’analyses (NS) et les analyses factices (DS) pour obtenir une sensibilité acceptable de signal avec la commande « gs », qui permet de passer et scan pour surveiller la qualité des données en temps réel.
- Enregistrer les spectres à l’aide de Zero aller « zg ».
- Traitement des données RMN
- Réglez les paramètres de traitement à la taille de la F2 direct (1H) et indirecte F1 (15N) dimensions du spectre en utilisant « SI F2 = 2048, F1 = 512 » avec option prédiction linéaire en dimension indirecte (Figure 3).
- Sélectionnez « QSINE » que la fonction de fenêtre et entrez une Maj de bell Sine (SSB) de 2 à traiter le spectre bidimensionnel.
- Entrez la commande « xfb » pour traiter les données dans les deux sens avec la transformation de fourier et de fonction fenêtre.
- Utilisez la commande « apk2d » pour effectuer correction automatique de phase dans les deux sens. Si le processus automatique n’atteint pas un niveau satisfaisant de correction de phase, extraire FIDs avec la commande « SSIE », calculer les valeurs de la phase de transformation 1D et appliquez-les aux données 2D.
- Corriger la base avec la fonction de correction automatique de la base « abs2 » de données 2D. Cela s’applique à une fonction polynomiale entre valeurs ppm définies dans les paramètres de traitement et produira un spectre 2D pour une analyse ultérieure.
- Si l’intention d’exécuter le traitement de la série pour la comparaison des données sur l’interaction avec une autre molécule, stocker les paramètres de traitement avec la commande « wpar » et les rappeler avec « rpar ». De cette façon que tous les ensembles de données seront traitées avec les mêmes paramètres et variations ne sera pas introduite en raison de différences de traitement.
- Analyse des données RMN
- Entrez la commande « pp » pour lancer le processus de cueillette de pointe.
- Définissez le nombre d’intensité maximale ppm plage et minimum de pics issu des pics attendus (Figure 4). Cliquez sur OK et vérifier les résultats de l’inspection visuelle. Le cas échéant, relancez le processus jusqu'à ce que les résultats sont satisfaisants basé sur la qualité des spectres.
- Générer un peaklist avec la commande « pp ».
Remarque : Ce peaklist contient des informations de hauteur/pic intensité par défaut et peut être exportée vers les spectres et peut être lu par d’autres programmes. - Observer les changements dans les intensités maximales ou mouvement des déplacements chimiques dans les spectres HSQC protéine qui indiquent une interaction avec une autre molécule. Si la molécule interagissant est grande, s’attendre à des réductions dans les intensités maximales ainsi que de la disparition de certains sommets.
- Importer la liste de pointe sur le jeu de données suivant en cliquant sur l’onglet « pics » et sélectionner « importer » avec un clic droit dans la fenêtre de pics.
- Visualiser les pics sur le spectre et si nécessaire de passer à un nouveau poste. Cliquez sur « Réinitialiser les intensités » pour « tableau complet » pour générer un peaklist pour le spectre avec des intensités (Figure 5). Cette liste pointe porteront sur les informations de position de la liste de crête mémorisée.
- Exporter les listes de la crête de différents ensembles de données vers une feuille de calcul ou un autre programme de mathématique pour l’analyse en sélectionnant la fonction « Exporter ».
- Calculer la variation des intensités maximales avec la fonction « pic d’intensité dans le spectre complexe/pic d’intensité dans le spectre de la protéine » pour chaque pic. Les valeurs peuvent être convertis au taux de variation par multiplication de 100. Notez que les volumes de pointe sont utiles aussi bien que les intensités maximales sont plus faciles à mesurer pour les pics qui sont positionnés à proximité de l’autre, comme c’est généralement le cas pour les protéines avec une haute densité de sommets relativement larges.
4. MicroScale thermophorèse (MST)
-
Préparation du Ligand protéine E-PRD
- Échanger le ligand dans un tampon compatible MST par dialyse jusqu'à 800 μL de protéine dans une unité de dialyse Mini 3,5 kDa en suspension dans 1 L de 20 mM HEPES, pH 7.5, 10 mM NaCl, en remuant lentement à 4 ° C durant la nuit.
- Concentrer le ligand à l’aide d’une unité d’ultrafiltration centrifuge (3 kDa MWCO) par centrifugation à de 14 000 x g pendant 10 min. Transférez le concentré de protéine dans un tube propre.
- Centrifuger le ligand à 21 000 x g pendant 10 min et transvaser avec soin le surnageant dans un nouveau tube pour enlever toute protéine précipité. Déterminer la concentration de ligand à l’aide de l’absorbance à 280 nm et le coefficient d’extinction de ligand. Ajouter 10 % Tween-20 pour donner une concentration finale de 0,015 %. Tween-20 est ajoutée au tampon de dosage pour éviter d’adsorption des capillaires. Le tampon final qui est utilisé pour les expériences de MST est 20 mM HEPES, pH 7.5, 10 mM NaCl, 0,015 % Tween-20.
-
Préparation du colorant-étiquetés de cible protéique VimRod
- Reconstituer le colorant rouge-tris-NTA en ajoutant 50 μL de 1 x PBS-T fourni avec le rouge-tris-NTA pour donner une concentration de 5 μM. Diluer 2 aliquotes μL dans des 200 μL tubes et conserver à-20 ° C.
- Diluer la protéine cible à 0,34 μM avec du tampon d’essai. Ajouter 58 μL de cible à une portion de 2 μl de colorant rouge-tris-NTA et incuber 30 min à température ambiante. La concentration finale de colorant-étiquetés de cible est 0,33 μM. Centrifuger la cible marquée à 21 000 x g pendant 10 min et transvaser avec soin le surnageant dans un nouveau tube pour enlever n’importe quel précipité. Le colorant rouge-tris-NTA se lie aux protéines par le biais de la balise His6 et a une constante de dissociation de liaison (KD) dans la gamme de sup-nanomolaires. C’est effectivement 100 % lié à la protéine cible donc aucune purification plus poussée n’est nécessaire.
- Allumez l’instrument de MST et ouvrez le logiciel de contrôle. Sélectionnez le paramètre rouge pour la teinture rouge-tris-NTA. Activer le contrôle de la température à 25 ° C.
- Choisissez le prétest pour valider le marquage de la cible et laissez d’agrégation ou adsorption sur les cuvettes. Une évaluation de ces paramètres est fournie automatiquement.
- Mix 17 μL de tampon et 3 μl du mélange de pipetage et de protéine cible. Remplir deux capillaires standards en les trempant dans la cible diluée et en dessinant liquide au centre du capillaire. Placez les capillaires dans la barre d’État et aux instruments. Démarrer la mesure.
- Dresser un bilan à la recherche d’un niveau suffisant de fluorescence et aucun signe d’adsorption (distorsion dans la forme de la ligne capillaire) ou l’agrégation (distorsions dans la trace de MST), qui sera signalée dans les logiciels d’analyse. Si les résultats sont positif sur le ligand des étapes, si ce n’est pas un tampon différent doit être jugé ou le montant de Tween-20 peut être majoré à 0,05 % ou plus.
-
Préparation de la série E-PRD de double Dilution Ligand
- Préparer une série de dilutions par marquage 16, 200 tubes μL de 1-16.
- Ajouter 17 μL du ligand à 1,17 x concentration supérieure à la concentration de ligand maximal désirée Tube1. Ce volume est deux fois le montant nécessaire (8,5 μL) comme une partie aliquote sera ensuite transférée au prochain tube de série. Le volume final de l’essai est 10 μL 8,5 μL de 1,17 concentration x du ligand sera diluée à 1 x par addition de 1,5 μL de la protéine cible, VimRod. Cette concentration maximale choisie doit être au moins 20 fois la valeur estimée de la KD .
- Ajouter les 8,5 μL de tampon de Tubes2-16 en utilisant un nouvel embout de la pipette pour chaque aliquote. Réutilisation des pointes de pipette peut affecter la précision (pour obtenir des conseils sur précise pipetage voir indications). Transférer 8,5 μL de ligand du Tube1 Tube2 relâchant lentement la solution dans le tampon sans générer des bulles. Mélanger le ligand et le tampon de pipetage, haut en bas, au moins 6 fois, encore une fois sans générer des bulles. E-PRD dans le tube plus concentré était de 1,5 mM et le VimRod marqué était présent à une concentration finale de 50 nM.
- Transférer 8,5 μL de ligand de Tube2 à Tube3 et mélanger avec le tampon. Répétez la dilution en série jusqu'à ce que tous les tubes ont eu ligand ajouté. Jeter les 8,5 μl de Tube16 afin que tous les tubes contiennent 8,5 μL de ligand dans une série de dilutions de double.
-
Préparation de l’expérience de la réaction de liaison et de MST
- Ajouter 1,5 μL de la protéine cible marquée dans chacun des tubes et mélanger doucement en pipettant également, haut et bas en prenant soin d’éviter les bulles. Incuber pendant 15 min.
- Sélectionnez le Mode Expert pour l’essai de liaison et entrez dans les paramètres pour une série de dilutions. Assurez-vous que le contrôle de température est réglé à 25 ° C, la puissance d’excitation est définie à 40 % et la puissance de MST au milieu. Ces paramètres doivent être optimisées pour autres partenaires de liaison à l’étude.
- Remplissez les capillaires avec les réactions de liaison puis dans la barre d’état capillaire. Charger le bac dans l’instrument, attendez que la température se retrouver 25 ° C et démarrer la mesure.
-
Analyse des données
- Ouvrez le fichier de test de liaison dans le logiciel d’analyse de l’affinité. Examiner les analyses capillaires ; Niveaux de fluorescence ne devraient pas varier de plus de 10 % par rapport à la moyenne, aucune agrégation ou adsorption ne devrait être détectée comme décrit à la section 3.6.
- Sélectionnez l’analyse MST sur le panneau de droite et faites glisser les données de test dans l’ensemble de l’analyse. S’il y a plusieurs parcours de l’essai de liaison ligand-cible même dans des conditions identiques, elles peuvent être fusionnées en laissant tomber dans le même ensemble. Par ailleurs, chaque série peut être encastré dans analyse indépendamment.
- Accédez à l’onglet Dose réponse correspondent à des données de graphique. Choisissez le modèle KD si un site de liaison unique est prévu. Le modèle Hill est également une option pour plusieurs sites de fixation avec un comportement coopératif. Points de valeurs aberrantes qui ne passent pas le contrôle de la qualité peuvent être supprimés de l’ajustement à ce stade. Ensembles de fusion avec plusieurs tests seront moyennés et erreurs calculé comme l’écart-type.
- Ouvrez l’onglet finale et comparer les résultats entre tous les tracés sur un graphique unique. Récupérer les résultats de raccord pour chaque courbe de la table qui est générée. Exporter les données ou les courbes ajustées à d’autres logiciels de présentation ou d’analyse si vous le souhaitez.
Résultats
Le domaine de la E-PRD (résidus 1822-2014 clonés dans pProEX-HTC) du gène humain envoplakin et le domaine VimRod (résidus 99-249 clonés dans pET21a) de vimentine humaine4 ont été exprimées avec les balises His6 et purifiées. Figure 6 et Figure 7 montrent que les niveaux de pureté de VimRod (18,8 kDa) et E-PRD (21,8 kDa) obtenues par cette méthode de purification des protéines. La suppression de la His6 tag de la construction de l’E-PRD est indispensable pour les expériences de MST comme la protéine VimRod est étiquetée à l’aide d’un colorant de liaison His6 tag et tout E-PRD conservant son étiquette His6 peut-être concourir pour la fixation du colorant. La deuxième colonne IMAC après clivage de la balise avec la protéase TEV supprime la protéase TEV, la balise clivée et toute clivé His6-E-PRD qui restaient. La dernière étape de polissage de la purification est la chromatographie d’exclusion. Malgré les deux protéines étant d’une taille similaire, le VimRod élue de la colonne à un 51 mL alors que le pic d’élution E-PRD est centré à 72 mL où s’attendrait un monomère de protéine de cette taille. L’augmentation apparente de la taille de VimRod est probablement due à la ses caractéristiques comme une longue tige filamenteuse en forme de protéine comme analytiquement ultracentrifugeuse expériences ont démontré que le VimRod était monomère4. Baisse des rendements de protéine sont obtenus à partir des cultures faites en M9 que ceux d’un bouillon riche en raison d’un montant inférieur de cellules étant produites dans les médias un minimum. La croissance initiale de la plus grande et ferments lactiques pour les préparations de M9 TB permet l’amélioration des rendements cellulaires tout en conservant l’étendue de 15N étiquetage nécessaires pour les expériences de RMN.
Le 15N -1H HSQCs ont été acquis pour le type sauvage et le mutant R1914E E-PRD en présence ou en absence de VimRod (Figure 8 a-8D). Le spectre de E-PRD dans la Figure 8 montre le nombre attendu de pics bien résolues, indicatives d’une protéine correctement pliée. En présence de VimRod (Figure 8 b), le spectre montre élargissement de la gamme étendue et de la disparition de pointe, correspondant à la liaison entre le E-PRD et VimRod. Cette liaison est perdue par mutation de R1914E comme en témoigne en comparaison de la Figure 8 et 8d. Peu de changement est observée dans le spectre à l’addition de VimRod pour le mutant R1914E indiquant un manque de liaison entre ce mutant E-PRD et VimRod. Les intensités maximales de E-PRD dans la présence ou l’absence de VimRod ont été comparées et tracées comme les intensités relatives des pics en 8E de la Figure, qui indique la portée de l’élargissement de pic dans le complexe de E-PRD. Le mutant R1914E de E-PRD (non illustré) conservé environ 97 % des pics à 20 % ou des intensités maximales plus élevées en présence de VimRod, comparée à environ 20 % pour le type sauvage (Figure 8F). Cela représente une perte de mutant de point de fonction, avec les autres mutants ayant des effets intermédiaires aussi avoir été étudié4.
À valider et à quantifier la liaison de ces facteurs VimRod et E-PRD MST His6-VimRod marqué par fluorescence rouge-tris-NTA agent comme cible mélangé avec une diminution des concentrations du ligand E-PRD de 1,28 mM à 39,1 nM ont été effectuées. Trois titrages de liaison ont été réalisées et les résultats sont en moyenne et illustrés à la Figure 9. Les données ont été ajustées avec un modèle standard de liaison du ligand du site unique et a donné un KD de 25,7 ± 2.1 μM. Évaluation de la liaison entre VimRod et E-PRD par résonance plasmonique de surface a donné une valeur de KD similaire de 19,1 ± 1,3 μM4.

Figure 1 : Capture d’écran de la configuration de l’expérience de RMN. La fenêtre illustrée est utilisée pour mettre en place une expérience standard pour recueillir un dataset HSQC. Paramètres de l’expérience sont lus dans adjacente à l’expérience. L’expérience ZGPR montré est choisie comme une première expérience pour charger les paramètres standard et solvant protonique dépendants. La fenêtre titre est utilisée pour entrer les détails expérimentaux aux fins de conservation des données. Pour percevoir le spectre HSQC l’expérience ZGPR est remplacé par SFHMQC3GPPH. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
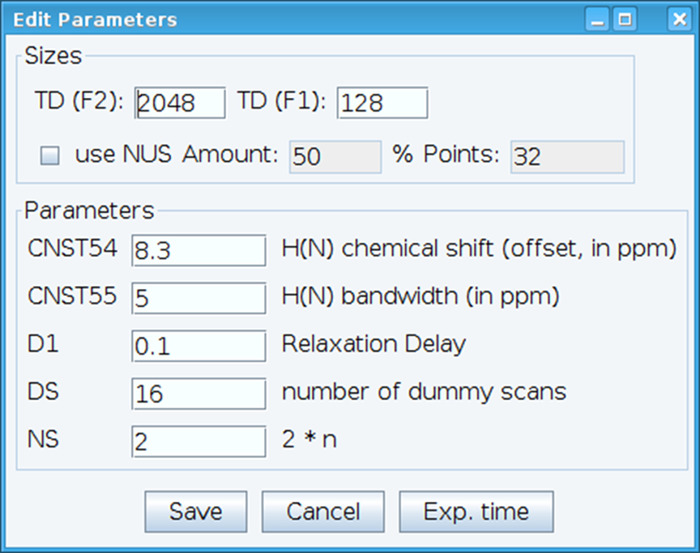
Figure 2 : réglage des paramètres expérimentaux RMN. La fenêtre illustrée sert à entrer les paramètres fondamentaux pour la séquence d’impulsions NMR afin d’optimiser le signal. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.

Figure 3 : traitement des données RMN. Paramètres utilisés pour le traitement de chacune des deux dimensions du spectre RMN du apparaissent, avec des flèches indiquant ceux qui sont en général réglés. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
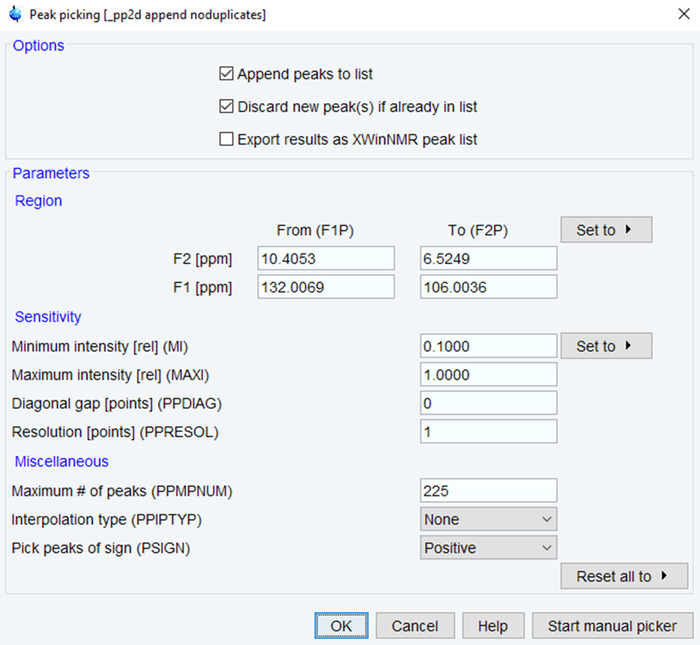
Figure 4 : paramètres pour le prélèvement de RMN pic. Les paramètres utilisés pour la cueillette des NMR pics dans le spectre RMN transformé sont affichés avec des valeurs typiques. Régler la gamme de ppm, l’intensité et la nombre de pics pour optimiser les spectres. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
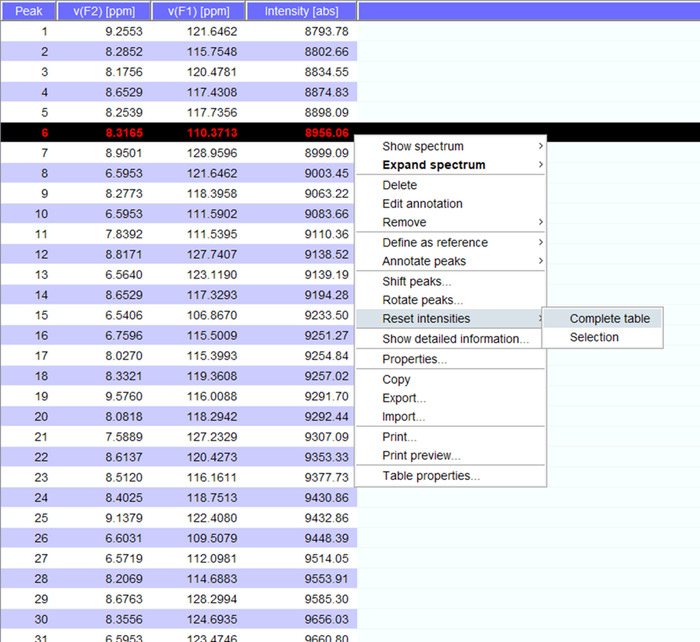
Figure 5 : représentant des Peaklist avec des intensités. Chaque pic qui est prélevée dans le spectre RMN reçoit un nombre et ses 1H et déplacements chimiques 15N et intensité du signal sont affichés. Cette peaklist permet ensuite de comparer les spectres obtenus dans la présence/absence d’un partenaire d’interaction. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
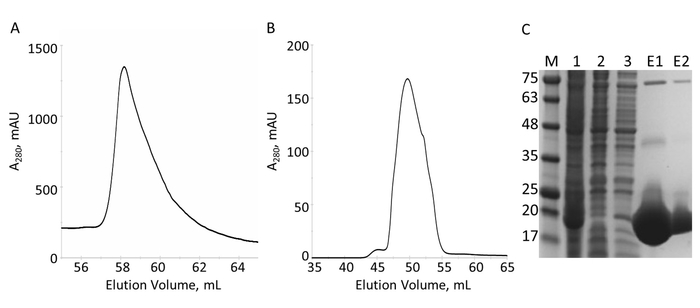
Figure 6 : Purification de VimRod His6-tag IMAC et S. A. Le chromatogramme pour l’élution de la colonne IMAC montre un pic majeur de VimRod. B. le chromatogramme pour l’élution de la colonne S montre un pic majeur. C. SDS-PAGE des fractions collectées au cours de la purification : normes MW avec la MW figurant aux points kDa à gauche du gel (M), lysat cellulaire (1), IMAC intermédiaire (2), lavage (3), mise en commun d’élution (E1), mis en commun S d’élution (E2). Bandes visibles à un poids moléculaire plus élevé dans les couloirs E1 et E2 sont des oligomères de VimRod pur tel que confirmé par western blot (données non présentées). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
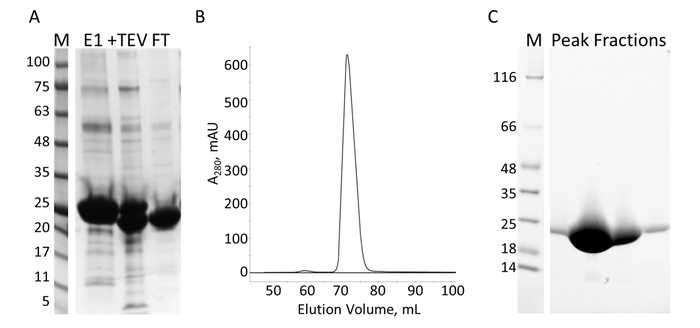
Figure 7 : Purification du E-PRD par IMAC et S. A. SDS-PAGE, de la purification de l’IMAC montrant des étalons de masse moléculaire avec la MW indiqué dans kDa à gauche du gel (M) et l’éluat de la première colonne IMAC (E1), le TEV clivage produits (+ TEV) et le flux par le biais de la deuxième colonne de l’IMAC (FT). B. le chromatographe de la colonne S montre un pic majeur. SDS-PAGE C. les normes de poids moléculaire (M) et les fractions de sommet S. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
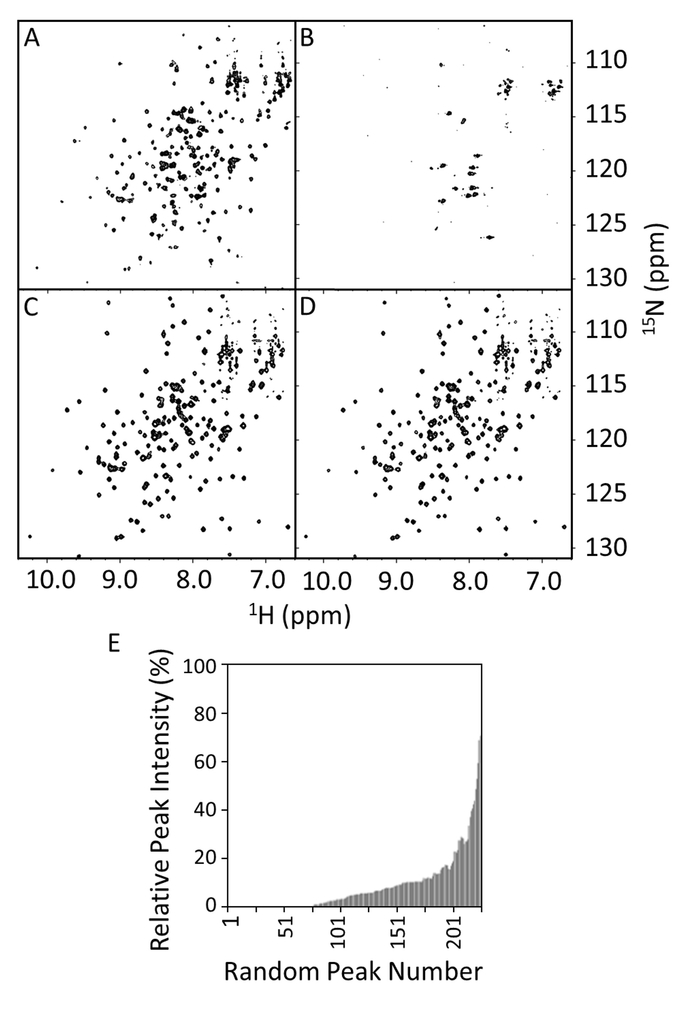
Figure 8 : HSQC spectres de type sauvage et R1914E Mutant de E-PRD en présence et en Absence de VimRod. Les spectres HSQC montrent sauvage E-PRD (100 µM) en 20 mM Tris-HCl, pH 7, en l’absence (A) ou en présence de 50 µM de 150 mM NaCl, 1 millimètre DTT, VimRod (B). Séries C et D sont les spectres HSQC du mutant R1914E (100µM) en l’absence ou la présence de 50 µM VimRod, respectivement. Dans le groupe E le pic de15N relative 1H - intensités du PRD-E avec ou sans liaison de VimRod sont indiqués en fonction du nombre maximum, qui est arbitrairement assigné et non basée sur la position de la séquence. Ces valeurs peuvent être utilisées pour définir un seuil de signification pour réduction d’intensité maximale lors de l’addition d’un ligand. Si les affectations sont disponibles, les valeurs importantes peuvent souvent être vus pour mapper vers une zone de liaison. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
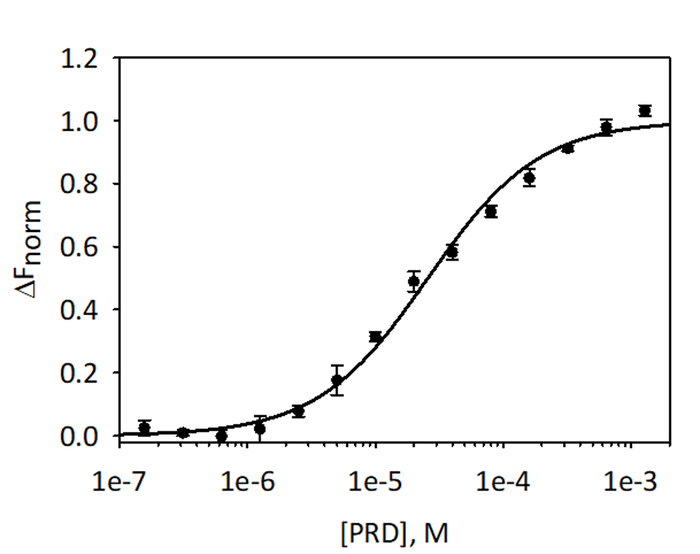
Figure 9 : liaison de E-PRD à VimRod. E-PRD a été dilué dans une série de dilutions de double de 1,28 mM pour 39,1 nM et incubées avec VimRod marqué avant d’effectuer l’analyse de MST. Les données de trois essais indépendants ont été combinées. Les données ont été ajustées à un modèle KD donnant un KD de 25,7 μM avec une confiance KD de ± 2.1 μM. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
| Réactif | Quantité |
| Phosphate de sodium dibasique (anhydre) | 6,0 g |
| Phosphate de potassium monobasique (anhydre) | 3.0 g |
| Chlorure de sodium | 0,5 g |
| H2O | Jusqu'à 950 mL |
Le tableau 1. M9 médias pour marquage isotopique.
| Réactif | Quantité |
| 15 NH4Cl | 1,0 g |
| Glucose (ou 13C-glucose) | 2,0 g |
| 1 M MgSO4 | 2 mL |
| 50 mM CaCl2 | 4 mL |
| 20 mg/mL Thiamine | 1,0 mL |
| 3 mM FeCl3 | 400 ΜL |
| Metal Mix (tableau 3) | 500 ΜL |
| H2O | Jusqu'à 50 mL |
Le tableau 2. Mélange nutritif pour les médias de la supplémentation de M9.
| Réactif | Quantité |
| 4 mM ZnSO4 | 323 mg |
| 1 mM MnSO4 | 75,5 mg |
| 4. 7 mM H3BO3 | 145 mg |
| 0,7 mM CuSO4 | 55,9 mg |
| H2O | Jusqu'à 500 mL |
Tableau 3. Supplément de metal Mix pour enrichir le mélange nutritif de MT.
| Échantillon | 1 mM E-PRD de tampons A1 (µL) | 1mM VimRod de tampon A (µL) | Tampon une (µL) | 200 µM DSS en D2O (µL) | Tampon B2 (µL) | Total Volume (µL) |
| E-PRD seul | 50 | 0 | 50 | 50 | 350 | 500 |
| E-PRD + VimRod | 50 | 50 | 0 | 50 | 350 | 500 |
| 1 Mémoire tampon A: 20 mM Tris-HCl, 1 millimètre DTT, pH 7 | ||||||
| 2 Mémoire tampon B: 23 mM Tris-HCl, 1,14 mM DTT, pH 7 | ||||||
Tableau 4. Préparation de l’échantillon de la RMN.
Discussion
Le 2D 15expérience N-résolu NMR est un du plus largement utilisé des méthodes pour montrer comment deux molécules interagissent. C’est la méthode de la plus riche en informations qui permet aux signaux des deux partenaires à être surveillée de façon continue tout au long d’une expérience de titrage en état de solution. Bien que généralement qualitatives dans le cas de grands complexes, la méthode peut aussi servir dans les cas favorables pour mesurer les affinités de liaison où les signaux RMN peuvent être suivis dans des spectres de haute résolution. Lorsque les attributions peuvent être effectuées facilement, comme dans le cas de beaucoup de protéines sous 20 kDa dans la taille, les sites de fixation peuvent aussi être mappés. Des tests complémentaires comme MST fournissent des informations quantitatives sur les interactions en solution et exigent moins de protéines dans les États sans étiquette. Comparaison de mutant de liaison de données est utile pour fournir des contrôles pour s’assurer que les interactions mis en évidence par l’élargissement des raies de NMR sont authentiques et pas d’artefacts de, par exemple, des changements de viscosité ou de l’agrégation.
Expression de la protéine
Rationalisation du processus d’expression réduit la quantité de production de protéines intensive de travail. Cadre de ce processus d’optimisation implique l’identification d’une souche appropriée d’Escherichia coli d’expression de la protéine recombinante. Préférence de souche dépend des éléments, y compris la nature du vecteur en cours d’utilisation et, plus précisément, la stabilité ultime de la protéine recombinante est exprimé7. Le risque de dégradation de la protéine hétérologue par endogène d’e. coli protéases peuvent être réduits par l’utilisation de protéase déficient e. coli comme la souche BL21. Gènes contenant des codons rares, une souche comme RIPL BL21-CodonPlus (DE3) peut être privilégiée. Cette souche allie la nature déficiente de protéase de la souche BL21 des exemplaires supplémentaires d’endogènes des ARNt codons rares pour l’arginine, isoleucine, proline et la leucine. Alternativement, les codons rares qui peuvent compromettre la surexpression peuvent être évités en ordonnant une construction optimisée codon provenant d’une source commerciale. De nombreuses souches d’Escherichia coli sont disponibles pour l’expression du gène recombinant, chacun optimisé pour le contournement d’un problème au cours de l’expression7. Dans le cas de cette étude, la souche déficiente en protéase standard BL21 (DE3) produit en quantité suffisante de protéine soluble pour purification ultérieure et l’analyse.
Purification de protéine
Le protocole de purification d’une protéine donnée est souvent unique en ce sens que chaque protéine reste stable et soluble dans des conditions différentes telles que température, concentration en sel ou pH. L’efficacité globale de purification par chromatographie d’affinité est également sensible à la concentration des espèces élution comme imidazole à différentes étapes au cours du processus de purification. Dans ce travail, les conditions de tampon critique pour IMAC étaient pH pour le E-PRD et concentration imidazole pour le VimRod. Un pH de 7.5 devait éviter la précipitation du PRD-E après élution initiale de la colonne de l’IMAC. Pour la purification de l’IMAC de VimRod, augmentation de la concentration de l’imidazole de 30 à 50 mM au cours de l’étape de lavage de colonne s’est avéré pour avoir une amélioration substantielle dans la pureté des fractions d’élution finale. Pour l’étape d’élution, augmentation de la concentration de l’imidazole de 250 à 350 mM s’est avéré pour améliorer le rendement de l’élution finale. Les premières tentatives pour éluer les protéines à l’aide d’imidazole de 250 mM a conduit à l’élution incomplète du VimRod telle que révélée par une bande d’imidazole 1 M final de la colonne (données non présentées). Augmentation de la concentration de l’imidazole à 350 mM pour l’élution suffisait récupérer l’ensemble de la protéine liée à la colonne. S pouvant servir un double objectif, car il agit comme une étape de polissage pour la purification de protéines tout en effectuant simultanément change de tampon. Change de tampon est une étape essentielle pour l’analyse ultérieure de liaison car il supprime l’imidazole utilisé pour Éluer la protéine His6-étiquetée. Il sert également une occasion de changer les conditions telles que la concentration en sel ou pH, ce qui peut affecter l’efficacité de certaines techniques en aval ou essais. Protéine décalage thermique (PTS) peut être utilisé pour identifier les tampons optimales pour les tests en aval, en particulier pour ceux qui ont besoin de protéines stable pour de longues périodes de temps à la température de la pièce8,9.
Analyse de liaison
Protéine qui est fraîchement préparé est critique pour essais de liaison exacte, bien que la protéine congelé peut également être utilisé tant que les résultats sont comparés. Les protéines filamenteuses Comme vimentine multimerize d’une manière dépendante de sel et pH, et par conséquent la solution il fallait être optimisé et l’État oligomère estimée par une méthode telle que SEC10,11, dynamique de diffusion de lumière 12 ou ultracentrifugation analytique13,14,15. Spectroscopie RMN est bien adaptée pour la mesure des interactions ligand de petites protéines à résolution atomique. Cependant, quand une protéine interagit avec une molécule plus grande, il s’ensuit un culbutage plus lent et cela se traduit par la perte des signaux qui peuvent confirmer liaison, bien qu’il ne le fait pas nécessairement permettre cartographie des sites, ce qui nécessitent également affectation d’au moins de fixation dorsale résonances. Dans ce scénario, des expériences RMN ne permettent pas d’identifier le site de l’interaction. Donc site réalisé mutagénèse est appliqué pour identifier les résidus importants nécessaires pour la liaison. C’est pourquoi ces mutants ne présentent pas de perte de signal. Dans ce protocole, une forme mutante avec une substitution en position 1914 conserve les intensités maximales en présence de VimRod et confirme donc la perturbation de l’interaction des E-PRD et VimRod. Affectation des résonances épine dorsale et sidechain ajouterais valeur à cette approche, en particulier comme la structure pour le PRD-E libre a été résolue par cristallographie aux rayons x4. Futures applications de la RMN comprennent la caractérisation des interactions complexes entre les molécules plus grosses et bénéficieront d’ultra hautes aimants de champ et l’utilisation des autres groupes observables telles que 13C-étiquetés et groupes méthyles trifluoro comme journalistes.
MST a un certain nombre d’avantages pour l’étude des interactions de liaison16. Les partenaires de liaison sont libres en solution et pas immobilisées. Analyse de la qualité des échantillons est intégré dans le logiciel avec contrôle de la qualité reporting d’agrégation, l’adsorption sur les capillaires ou marquage fluorescent insuffisante de la molécule cible. De petites quantités de la cible sont généralement utilisés, la concentration de la cible marquée est généralement entre 20 et 50 nM en 10-20 μL volume/réaction. Ce protocole utilise des volumes de réaction très faible (10 μL) afin d’optimiser la concentration de ligand qui peut être réalisé dans les titrages permettant des interactions de liaison faible à caractériser. Cela exige que l'on précise pipetage et prenant soin d’éviter d’introduire des bulles tout en mélangeant toujours soigneusement. Un mélange adéquat est essentiel pour les mesures de fluorescence précise et uniforme le long de la série de dilutions en série. Le montant de Tween-20 dans les expériences de MST a été réduit à une norme de 0,05 % à 0,015 % pour réduire la tendance à créer des bulles et d’améliorer le mélange.
Le colorant rouge-Tris-NTA offre un moyen rapide, facile et pratique d’étiqueter fluorescent toute protéine qui a un son tag. L’étiquetage est effectivement terminé en 30 minutes seulement et est très serré de sorte qu’aucune procédure de retrait de colorant n’est nécessaire. Aucune modifications ne sont apportées aux résidus d’acides aminés dans la protéine qui seraient susceptibles de modifier les propriétés de liaison du ligand. Une mise en garde est que seulement les protéines doivent être étiquetées doivent avoir une balise His6. Cela nécessitait le clivage de la balise de la protéine ligand, E-PRD et la suppression de la balise et clivé E-PRD avec une deuxième étape de colonne IMAC. Si possible, la protéine ligand doit être préparée sans l’utilisation d’une balise. Alternativement, protéines peuvent être étiquetés de façon covalente avec un fluorophore par amine coupler à des résidus de lysine ou thiol de couplage pour les résidus de cystéine. Cependant, il faut quand à l’aide de ces systèmes depuis la fixation covalente d’un fluorophore peut-être affecter les interactions de liaison électrostatique ou polaire en s’appuyant sur les résidus de lysine ou la cystéine. La quantification de l’affinité entre les VimRod et les E-PRD de MST a été particulièrement sensible à la concentration de sel. Ce problème a été atténué par dialyse initialement la cible et le ligand dans le même lot de tampon. Néanmoins, saturation de la courbe de liaison MST ne pourrait être atteint que lorsque vous effectuez les tests de MST en présence de NaCl 150 mM en raison du comportement complex de VimRod. Données fiables et complètes a été obtenues une fois que la concentration de NaCl a été abaissée à 10 mM, ce qui permet un calcul précis de la KD. Donc, optimisation minutieuse des conditions de la solution et comparaison avec des analyses complémentaires sont recommandés afin d’atteindre des résultats robustes. En outre, les MST peut-être servir à quantifier la dépendance de sel pour une interaction donnée, de quantifier les propriétés stoechiométriques des interactions protéine, moniteur le repliement des protéines et de la sonde en enzyme cinétique17.
Déclarations de divulgation
Les auteurs ne divulguer aucun conflit d’intérêt.
Remerciements
Ce projet a été soutenu par le CRSNG RGPIN-2018-04994, Campus Alberta Innovation Program (RCP-002-12 C) et l’Alberta Prion Research Institute / Alberta Innovates Bio Solutions (201600018), attribué à tibus et Génome Canada, Fondation canadienne pour Subventions de l’innovation décernées à la métabolomique Innovation Centre (ICTM) et NANUC.
matériels
| Name | Company | Catalog Number | Comments |
| Monolith NT.115, includes control and analysis software | NanoTemper Technologies | MO-G008 | Instrument for microscal thermophoresis |
| His-Tag Labeling Kit RED-Tris-NTA | NanoTemper Technologies | MO-L008 | RED-tris-NTA dye for MST |
| Standard capillaries | NanoTemper Technologies | MO-K022 | capillaries for MST |
| HisTrap HP, 5 mL | GE Healthcare | 17524801 | IMAC column |
| HisPur Ni-NTA resin, 100 mL | Thermo Fisher Scientific | 88222 | IMAC resin |
| HiLoad 16/600 Superdex 75 pg | GE Healthcare | 28989333 | SEC column |
| TEV protease | Sigma Aldrich | T4455 | cleavage of his tag from E-PRD |
| BL21(DE3) Competent E. coli | New England Biolabs | C2527H | cells for protein expression |
| cOmplete Protease Inhibitor Cocktail Tablets EDTA-Free | Sigma Aldrich | 11873580001 | protease inhibitors for protein purification by IMAC |
| TCEP, Tris(2-carboxyethyl)phosphine hydrochloride | Sigma Aldrich | C4706 | reducing agent for protein purification |
| Reagents (HEPES, NaCl, etc) | Sigma Aldrich | various | Preparation of media and buffers |
| Ammonium chloride (15N, 99%) | Cambridge Isotope Laboratories | NLM-467 | isotope labelling for NMR |
| D2O, Deuterium Oxide (D, 99.8%) | Cambridge Isotope Laboratories | DLM-2259 | NMR sample preparation |
| DSS, Sodium 2,2-dimethyl-2-silapentane-5-sulfonate-D6 (D,98%) | Cambridge Isotope Laboratories | DLM-8206 | reference for NMR |
| Precision 5 mm NMR Tubes, 7” long | SJM/Deuterotubes | BOROECO-5-7 | NMR tubes |
| NMR spectrometer (14.1 Tesla) | Bruker | acquisition of NMR data | |
| TCI 5mm z-PFG cryogenic probe | Bruker | acquisition of NMR data | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Bruker TopSpin 4.0.1 | Bruker | processing of NMR data | |
| MO.Control | NanoTemper Technologies | included with Monlith NT.115 | |
| MO.Affinity Analysis | NanoTemper Technologies | included with Monlith NT.115 |
Références
- Vinogradova, O., Qin, J. NMR as a unique tool in assessment and complex determination of weak protein-protein interactions. Topics in Current Chemistry. 326, 35-45 (2012).
- McKercher, M. A., Wuttke, D. S. NMR Chemical Shift Mapping of SH2 Peptide Interactions. Methods in Molecular Biology. 1555, 269-290 (2017).
- Al-Jassar, C., Bikker, H., Overduin, M., Chidgey, M. Mechanistic Basis of Desmosome-Targeted Diseases. Journal of Molecular Biology. 425 (21), 4006-4022 (2013).
- Fogl, C., et al. Mechanism of intermediate filament recognition by plakin repeat domains revealed by envoplakin targeting of vimentin. Nature Communications. 7, (2016).
- Walker, J. M. . The Protein Protocols Handbook. , (2002).
- Wishart, D. S., et al. 1H, 13C and 15N chemical shift referencing in biomolecular NMR. J. Biomol. NMR. 6, 135-140 (1995).
- Casali, N. Escherichia coli Host Strains. E. coli Plasmid Vectors. , 27-48 (2003).
- Ericsson, U. B., Hallberg, B. M., DeTitta, G. T., Dekker, N., Nordlund, P. Thermofluor-based high-throughput stability optimization of proteins for structural studies. Analytical Biochemistry. 357 (2), 289-298 (2006).
- Kozak, S., et al. Optimization of protein samples for NMR using thermal shift assays. Journal of Biomolecular Nmr. 64, 281-289 (2016).
- Burgess, R. R. A brief practical review of size exclusion chromatography: Rules of thumb, limitations, and troubleshooting. Protein Expression and Purification. , (2018).
- Kunji, E. R. S., Harding, M., Butler, P. J. G., Akamine, P. Determination of the molecular mass and dimensions of membrane proteins by size exclusion chromatography. Methods. 46 (2), 62-72 (2008).
- Stetefeld, J., McKenna, S. A., Patel, T. R. Dynamic light scattering: a practical guide and applications in biomedical sciences. Biophysical Reviews. 8 (4), 409-427 (2016).
- Patel, T. R., Winzor, D. J., Scott, D. J. Analytical ultracentrifugation: A versatile tool for the characterisation of macromolecular complexes in solution. Methods. 95, 55-61 (2016).
- Pearson, J. Z., Cole, J. L., et al. Chapter One - Next-Generation AUC Adds a Spectral Dimension: Development of Multiwavelength Detectors for the Analytical Ultracentrifuge. Methods in Enzymology. 562, 1-26 (2015).
- Gorbet, G. E., Pearson, J. Z., Demeler, A. K., Cölfen, H., Demeler, B., Cole, J. L. Chapter Two - Next-Generation AUC: Analysis of Multiwavelength Analytical Ultracentrifugation Data. Methods in Enzymology. 562, 27-47 (2015).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nature Communications. 1, 100 (2010).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon