Method Article
Interazioni di proteine globulari e filamentose di spettroscopia di risonanza magnetica nucleare (NMR) e Microscala submicronica (MST) di misura
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per la produzione e purificazione di proteine che sono etichettati con isotopi stabili e successiva caratterizzazione delle interazioni proteina-proteina mediante spettroscopia di risonanza magnetica nucleare (NMR) e Microscala Esperimenti di Termoforesi (MST).
Abstract
Proteine filamentose come vimentin forniscono organizzazione all'interno delle cellule fornendo un'impalcatura strutturale con siti che legano proteine contenenti plakin si ripete. Qui, un protocollo per la rilevazione e la misurazione di tali interazioni è descritto utilizzando il dominio di ripetere plakin globulare di envoplakin e la bobina elicoidale di vimentina. Questo fornisce una base per determinare se una proteina si lega il vimentin (o simili proteine filamentose) e per la misura dell'affinità dell'interazione. La proteina globulare di interesse è etichettata con 15N e titolata con il vimentin proteina in soluzione. Uno spettro NMR bidimensionale viene acquisito per rilevare interazioni osservando cambiamenti in forma di picco o spostamenti chimici e per chiarire gli effetti delle condizioni di soluzione tra cui livelli di sale, che influenzano la struttura quaternaria vimentin. Se la proteina di interesse si lega il ligando filamentoso, l'interazione di associazione è quantificato dal MST utilizzando le proteine purificate. L'approccio è un modo semplice per determinare se una proteina di interesse viene associata un filamento e valutare come alterazioni, quali mutazioni o condizioni di soluzione, influenzano l'interazione.
Introduzione
Interazioni tra proteine permettono la formazione di macchine molecolari che creano ordine all'interno delle cellule. Le interazioni individuali sono spesso deboli ma solitamente contribuiscono a complessi polivalenti che possono essere cooperativo e dinamicamente regolato. Saggi sensibili che forniscono risoluzione atomica e informazioni quantitative su tali interazioni complesse sono necessari meccanismi di dedurre e progettare interventi quali molecole di droga-come. La spettroscopia NMR è un metodo efficiente per ottenere tali informazioni sulle interazioni della proteina e inoltre è usata per lo screening rapido per ligandi compresi quelli che legano debolmente1. I metodi di NMR utilizzati possono essere classificati in quelli che sono proteine osservare o ligando osservare. Questo manoscritto utilizza il primo approccio in cui viene acquisito un spettro di un isotopo stabile con l'etichetta della proteina che è relativamente piccola (solitamente sotto 20 kDa) e il ligando adenoida è titolato. In questo modo i residui con etichettati coinvolti nell'interazione deve essere mappato in casi favorevoli. Una volta le forme complesse, ci sono cambiamenti in ambienti chimici dei residui d'interazione che si manifestano come variazioni lo spostamento chimico e la forma dei loro segnali NMR. La portata di tali modifiche si correla con il grado di coinvolgimento di questi gruppi nell'interazione. Perturbazioni di spostamento chimico (CSP) possono essere misurati confrontando una serie di spettri NMR della proteina raccolti in assenza e in presenza di quantità variabili di ligando. Per ligandi più grandi o complesse interazioni, il cambiamento nella forma di picco o l'intensità può essere misurato per dedurre le interazioni.
L'esperimento 2D più comune utilizzato per la rilevazione di interazioni ligando è l'esperimento di 15N-eteronucleari singolo quantum correlazione (HSQC)2. Questo richiede che una proteina uniformemente essere etichettati con 15N, che è tipicamente realizzato di esprimerle come affinità-tagged versioni in colture batteriche di Escherichia coli coltivate in 15media N-arricchita. Associazione è apparente quando gli spettri HSQC raccolti durante la titolazione si sovrappongono, rivelando le modifiche di picco per un sottoinsieme dei residui coinvolti nella formazione complessa. L'interazione può verificarsi in regime di scambio veloce dove i segnali di stato libero e ligando-saturi collassano in una popolazione media di picco. In alternativa, nel caso di lenti scambi tra gli Stati, entrambi i segnali sono osservati con integrali che rappresentano i loro importi relativi. Mentre analisi lineshape NMR possono essere utilizzato per stimare le affinità di legame in alcuni casi, metodi quali MST inoltre hanno provato comodi e forniscono la convalida incrociata delle interazioni genuini.
L'esempio fornito è di due proteine che si trovano all'interno di desmosomi. Essi mediare giunzioni tra superfici delle cellule e del citoscheletro e mediare multivalenti interazioni tra macchine di adesione delle cellule e filamenti intermedi per mantenere l'integrità della pelle e tessuti del cuore e sopportare di elevate forze di taglio. Quando le proteine desmosomiali come desmoplakina o il vimentin sono compromessi da mutazioni o autoanticorpi, che conduce alla destabilizzazione delle giunzioni cellula-cellula, e quindi le loro interazioni sono di importanza critica3, possono provocare malattie. La base strutturale del ligando di proteine desmosomiali può essere caratterizzata dalla spettroscopia NMR, mentre le interazioni possono essere quantificate da MST. Metodi nel presente documento sono stati usati per caratterizzare le interazioni tra domini ripetere plakin (MDP) che spesso sono presenti come insiemi di tandem che offrono scanalature di base e il vimentin, un filamento intermedio che interagisce attraverso una superficie acida offerta dalla sua elicoidale Bundle4. Questi complessi sono formati alla membrana cellulare dove di ancoraggio per i filamenti intermedi del citoscheletro delle cellule a desmosomi che collegano alle celle adiacenti, formando così una rete di legami adesivi che si irradia durante un tessuto.
Protocollo
1. espressione recombinant della proteina
-
Espressione di Envoplakin PRD (E-PRD) e il Vimentin 99-249 (VimRod)
- Trasformare le cellule di e. coli BL21 (DE3) con il plasmide contenente il gene desiderato. Diffondere le cellule su piastre di agar contenente ampicillina di 100 µ g/mL. Incubare le piastre a 37 ° C durante la notte.
- Scegli una singola Colonia e inoculare 20 mL di brodo formidabile (TB) contenente 100 ampicillina µ g/mL per selezionare per il plasmide. Far crescere la cultura a 37 ° C con agitazione (180 giri/min) durante la notte.
- Trasferire la cultura intera da 20 mL a 1 L di TB contenente ampicillina di 50 µ g/mL. Incubare la coltura a 37 ° C con agitazione a 180 giri/min fino a quando il OD600 = 0.6-0.8.
- Ridurre la temperatura a 18 ° C e indurre l'espressione della proteina aggiungendo isopropilico β-D-1-thiogalactopyranoside (IPTG) ad una concentrazione finale di 1 mM. Continuare incubazione a 18 ° C con agitazione a 160 giri/min durante la notte per consentire l'espressione della proteina.
- Raccogliere le cellule mediante centrifugazione la cultura a 8.000 x g per 15 min. decantare e scartare il surnatante.
- Lavare le cellule prelevate da risospendere il pellet cellulare in circa 40 mL di fosfato soluzione salina tamponata (PBS: tampone fosfato 20 mM, pH 7.4, 120 mM NaCl). Trasferire la risospensione in una provetta 50 mL. Centrifugare nuovamente a 8.000 x g per 15 min.
- Decantare e scartare il surnatante. Immediatamente iniziare il protocollo di purificazione o congelare il pellet di cellule a-20 ° C per un uso futuro.
-
Espressione della proteina isotopicamente etichettata
- Trasformare le cellule di e. coli BL21 (DE3) e preparare una coltura starter 20 mL come in 1.1.1-1.1.2.
- Trasferire la cultura intera da 20 mL a 1 L di TB arricchito contenente un tryptone ulteriore g 4,0, 5,0 g NaCl e 100 µ g/mL ampicillina. Incubare la coltura a 37 ° C con agitazione a 160 giri/min fino a quando il OD600 = 1.6-1.9.
- La cultura di 1 L di raccolta mediante centrifugazione a 8000 x g per 15 min. decantare e scartare il surnatante.
- Lavare il pellet cellulare da Risospendere delicatamente in circa 40 mL di PBS e trasferire la risospensione in un tubo da 50 mL.
- Centrifugare nuovamente a 8.000 x g per 15 min. decantare e scartare il surnatante.
- Risospendere il pellet cellulare in 20 mL di media minimi M9 (tabella 1) e trasferimento al resto del 950 mL di media minimi M9 contenente ampicillina di 100 µ g/mL.
- Aggiungere 50 mL di miscela nutriente filtro sterilizzato (tabelle 2 e 3).
- Acclimatare la cultura a 18 ° C per 30 min prima dell'aggiunta di IPTG ad una concentrazione finale di 1 mM.
- Incubare per una notte a 18 ° C con agitazione a 160 giri/min.
- Raccogliere le cellule come nella sezione 1.1.4-1.1.6.
2. purificazione di cromatografia (IMAC) di affinità del metallo di VimRod ed E-PRD immobilizzato
-
Purificazione di His6-Tag VimRod
- Risospendere il pellet cellulare in 5 mL/g di PBS contenente un inibitore di proteasi cocktail EDTA carente. Omogeneizzare con 12 colpi in un omogeneizzatore del tessuto lisata per migliorare la lisi delle cellule nel passaggio seguente.
- Su ghiaccio, Sonicare sospensione delle cellule ad un impulso di 1 s su/1 s off, ampiezza di 80% per un totale di 1,5 min. Ripetere la sonicazione altre due volte, agitando delicatamente il ghiaccio tra le esecuzioni per evitare il surriscaldamento.
- Centrifugare il campione a 75.000 x g per 45 min. decantare e filtrare il surnatante utilizzando un filtro per siringa (0,45 µm).
- Equilibrare una colonna IMAC 5ml con 5 volumi di colonna (CV) del buffer obbligatorio (20 mM HEPES, pH 7.5, 500 mM NaCl, imidazolo 10 mM) con una portata di 1 mL/min utilizzando un sistema di cromatografia in fase liquida (FPLC) proteine veloci.
- Caricare il surnatante filtrato sulla colonna ad una portata di 0,5 mL/min.
- Lavare la colonna con 5 CV di lavare buffer (20 mM HEPES, pH 7.5, 500 mM NaCl, imidazolo di 50 mM) con una portata di 1 mL/min.
- Eluire la proteina con 3 CV del tampone di eluizione (20 mM HEPES, pH 7.5, 500 mM NaCl, imidazolo 350 mM) ad un flusso di frazioni di 0,5 mL/min raccogliere 1,5 mL. Se disponibile, selezionare la modalità getto eluizione per aumentare la concentrazione delle proteine eluite.
- Identificare le frazioni dal cromatogramma FPLC che contengono la proteina di interesse da SDS-PAGE e utilizzare metodi standard per misurare la concentrazione di proteina5.
- Piscina e le frazioni di eluizione che contengono le quantità elevate di proteina usando un dispositivo centrifugo ultrafiltrazione (kDa MWCO 3, 5ml) a 2 mL di concentrato. Centrifugare a 21.000 x g per rimuovere qualsiasi precipitato e passare attraverso un filtro di 0,22 μm.
- Equilibrare una colonna di cromatografia (S) esclusione dimensioni 120 mL con 2 CV di S buffer (20 mM HEPES, 150 mM NaCl, pH 7.5, 0,5 mM TCEP) ad un flusso di 1 mL/min utilizzando un FPLC.
- Iniettare il concentrato della proteina da 2.1.9 sulla colonna ed eluire con 1 tampone di CV di S ad una portata di 0,5 mL/min, raccogliendo frazioni di 1 mL.
- Identificare le frazioni contenenti la proteina di interesse come prima.
- Piscina le frazioni contenenti la proteina di importi più elevata.
- Conservare a 4 ° C per uso a breve termine o aggiungere glicerolo al 20% e conservare a-80 ° C in piccole aliquote.
-
Purificazione della proteina E-PRD con l'etichetta His6 rimosso
- Seguire i passaggi in 2.1.1-2.1.8 per purificare la proteina His6-etichetta E-PRD. Piscina le frazioni di picco e determinare la concentrazione di proteina.
- Aggiungere il tabacco etch proteasi virus (TEV) (1 mg/mL) a 2 µ l/mg di proteina in pool. Trasferimento a dialisi tubi (6 kDa) e Dializzare nel buffer di S durante la notte a 4 ° C. Questo passaggio consente di clivaggio del tag His6 e rimozione dell'imidazolo che interferisca con l'associazione alla resina Ni-NTA nel passaggio successivo.
- Portare i 5 mL di resina Ni-NTA in una colonna di gravità con 3 CV di S buffer. Tampone in eccesso di scarico dalla resina.
- Versare la proteina E-PRD spaccata sulla resina e incubare per 1 ora su una piattaforma a dondolo per consentire l'ApoAlert His6-etichetta E-PRD e il tag di His6 spaccato da associare. La proteasi TEV è anche His6-etichetta e volontà di associare la resina. Raccogliere il flusso attraverso, che contiene il tag-gratuita E-PRD. Lavare la resina con 2 CV di S buffer per garantire tutti i E-PRD è recuperato.
- Concentrarsi E-PRD nel flusso attraverso a 2 mL utilizzando un dispositivo centrifugo ultrafiltrazione (kDa MWCO 3, 5ml). Centrifugare a 21 000 x g per rimuovere qualsiasi precipitato e passare attraverso un filtro di 0,22 μm.
- Equilibrare una colonna 120 mL S con 2 CV di S buffer (20 mM HEPES, 150 mM NaCl, pH 7.5, 0,5 mM TCEP per MST o 20 mM Tris-HCl, 1 millimetro DTT, pH 7 per NMR) ad un flusso di 1 mL/min utilizzando un FPLC.
- Iniettare il concentrato della proteina E-PRD da 2.2.5 sulla colonna ed eluire con 1 tampone di CV di S ad una portata di 0,5 mL/min, raccogliendo frazioni di 1 mL.
- Identificare le frazioni contenenti la proteina di interesse come prima.
- Piscina le frazioni contenenti la proteina di importi più elevata.
- Conservare a 4 ° C per uso a breve termine o aggiungere glicerolo al 20% e conservare a-80 ° C in piccole aliquote.
3. NMR metodi
- Preparazione del campione NMR
- Purifica i 15N-labeled selvaggio-tipo o proteina di R1914E E-PRD come descritto in precedenza utilizzando 20 mM Tris-HCl, 1 millimetro DTT, pH 7 come il buffer di S per passaggio 2.2.6. Soluzioni di riserva di proteine, solitamente vanno da 0,3 a 1 mM con volumi di circa 1 mL.
Nota: Proteina possa essere concentrata a > 100 µM utilizzando un kDa MWCO 3, 5ml centrifugo ultrafiltrazione dispositivo per portare la concentrazione in una gamma adatta per la preparazione del campione. - Purificare un campione della proteina di VimRod senza etichetta utilizzando 20 mM Tris-HCl, 1 millimetro DTT, pH 7 come il buffer di S per passaggio 2.1.10.
- In un volume finale di 500 µ l, aggiungere wild-type o mutante della proteina E-PRD a una concentrazione finale di 100 µm, ossido di deuterio (D2O) a una concentrazione finale di 10% (v/v) e di DSS (acido 4,4-dimethyl-4-silapentane-1-sulfonic) a una concentrazione finale di 20 µM. Bring il volume del campione fino a 500 µ l utilizzando 20 mM Tris-HCl, 1 millimetro DTT, pH 7. Una preparazione del campione rappresentativo è descritta nella tabella 4.
Nota: 0 ppm risonanza del DSS è usato per calibrare gli spostamenti chimici 1H così come per il riferimento indiretto del prodotto chimico 15N sposta delle proteine6. D2O è utilizzato per il segnale di blocco di deuterio per mantenere lo spettrometro operante a un campo magnetico netto costante. - Portare un secondo campione di E-PRD, D2O e DSS come nel passaggio precedente e aggiungere VimRod a una concentrazione finale di 50 µM prima di portare il volume fino a 500 µ l.
- Trasferire i campioni µ l 500 a un tubi NMR ampia di 5 mm per l'esperimento.
- Purifica i 15N-labeled selvaggio-tipo o proteina di R1914E E-PRD come descritto in precedenza utilizzando 20 mM Tris-HCl, 1 millimetro DTT, pH 7 come il buffer di S per passaggio 2.2.6. Soluzioni di riserva di proteine, solitamente vanno da 0,3 a 1 mM con volumi di circa 1 mL.
- Setup sperimentale NMR
- Attivare il flusso d'aria con il comando di espulsione "ej"; verrà visualizzata l'esempio dal magnete. Ora, posto il campione all'interno di una casella di selezione sopra il magnete dall'apertura e inserire con il comando "ij". Attendere che il campione si deposita all'interno del magnete prima di procedere.
- Creare un nuovo dataset utilizzando il comando "edc" e caricare i parametri standard 1H NMR selezionando esperimento "ZGPR" (Figura 1). Compilare i campi nome, EXPNO (esperimento numero) e PROCNO (numero di cartella di dati trattati). Selezionare il solvente nel campo "Imposta solvente" e clicca su "Execute 'getprosol'" per leggere probehead standard e parametri dipendenti dal solvente (prosol).
- Bloccare il campione al deuterati solvente, vale a dire, D2O, utilizzando il comando "blocca" e attendere fino a quando è finito spazzare e raggiunge blocco.
- Correggere la frequenza di risonanza del magnete di tuning l'esempio utilizzando il comando Sintonizzazione automatico "atma". Monitorare la curva di oscillazione fino al termine della sintonizzazione automatica.
- Il campo magnetico usando TOPSHIM (comando "topshim") di spessore. Spessoramento è processo di aggiustamenti al campo magnetico per ottenere uniformità intorno al campione. È buona norma per memorizzare i valori di spessore con il comando "wsh" e leggerli con "rsh" prima di topshim, se usando i campioni uguali o simili.
- Regolare il guadagno del ricevitore con comando "rga" per raggiungere il massimo rapporto segnale-rumore.
- Posizionare il centro dello spettro sull'offset della risonanza di acqua (o1) e impostare l'impulso di protoni (p1) di 90 gradi ad alta potenza utilizzando "calibo1p1".
- Raccogliere lo spettro del protone usando lo zero andare processo con "efp" che comprende la moltiplicazione esponenziale ("em"), free induction decay (FID) che incorporano allargamento di riga, "ft" e "zg" comando trasformazione del fourier di FID e "pk" per applicare la fase correzione.
- Applicare la correzione automatica di fase "apk" e la correzione automatica linea di base "absn" utilizzando il polinomio senza opzione di integrazione.
- Creare un nuovo dataset (come 3.2.2) per l'esperimento SOFAST HMBC selezionando "SFHMQC3GPPH" nell'esperimento.
- Copia ottimizzata P1 e O1 dallo spettro del protone e popolare P1 dipendenti impulsi utilizzando il comando "getprosol 1h p1 plw1", dove p1 è il valore di P1 ottimizzato e plw1 è il livello di potenza per P1.
- Ottimizzare la costante CNST54 per impostare l'offset per spostamento chimico ammide e CNST55 per definire la larghezza di banda al fine di comprendere le regioni spettrali di interesse che permette il guadagno del ricevitore essere ottimizzato (Figura 2). Per selezionare questi parametri, estrarre il primo FID (decadimento libero induzione) dallo spettro bidimensionale e cercare il segnale osservato per definirli. Inoltre, variare il ritardo di rilassamento (D1), numero di scansioni (NS) e fittizi scansioni (DS) per ottenere la sensibilità del segnale accettabile con il comando "gs", che consente di andare e scansione per monitorare la qualità dei dati in tempo reale.
- Registrare gli spettri con Zero Go "zg".
- Elaborazione dati NMR
- Impostare i parametri di elaborazione per la dimensione della F2 diretto (1H) e indiretta F1 (15N) dimensioni del spettro utilizzando "SI F2 = 2048, F1 = 512" con opzionale predizione lineare nella dimensione indiretta (Figura 3).
- Selezionare "QSINE" come la funzione di finestra e immettere uno spostamento di bell Sine (SSB) di 2 per elaborare lo spettro bidimensionale.
- Immettere il comando "xfb" per elaborare i dati in entrambe le direzioni con trasformazione di funzione e trasformata di fourier di finestra.
- Utilizzare il comando "apk2d" per eseguire la correzione automatica di fase in entrambe le direzioni. Se il processo automatico non raggiunge un livello soddisfacente di correzione di fase, estrarre FIDs con il comando "rser", calcolare i valori di fase dall'elaborazione 1D e applicarli ai dati 2D.
- Correggere la linea di base con la funzione di correzione automatica linea di base "abs2" per dati 2D. Questo si applica una funzione polinomiale tra ppm valori definiti nei parametri di elaborazione e produrrà uno spettro 2D per ulteriori analisi.
- Se si prevede di eseguire l'elaborazione seriale per il confronto di dati di interazione con un'altra molecola, memorizza i parametri di elaborazione con il comando "wpar" e richiamarli con "rpar". In questo modo che tutti i set di dati saranno trattati con stessi parametri e le variazioni non verrà introdotto a causa di differenze di elaborazione.
- Analisi dei dati NMR
- Immettere il comando "pp" per iniziare il processo di picking di picco.
- Definire il numero di intensità/massima gamma e minimo del ppm di picchi in base ai previsti picchi (Figura 4). Fare clic su OK e verificare i risultati mediante ispezione visiva. Se necessario, eseguire nuovamente il processo fino a quando i risultati sono soddisfacenti basato su qualità di spettri.
- Generare un peaklist con il comando "pp".
Nota: Questo peaklist contiene dati altezza/picco intensità informazioni per impostazione predefinita e possono essere esportati in spettri successivi e possono essere letti da altri programmi. - Osservare le modifiche nelle intensità di picco o movimento in spostamenti chimici negli spettri HSQC proteine che indicano l'interazione con un'altra molecola. Se la molecola interagente è grande, è possibile aspettarsi riduzioni delle intensità di picco insieme a scomparsa di alcune vette.
- Importare l'elenco di picco per il prossimo set di dati facendo clic sulla scheda "picchi" e selezionando "importazione" con un clic destro nella finestra picchi.
- Visualizzare le cime sopra lo spettro e se necessario spostare loro in nuove posizioni. Clicca su "reset intensità" per "tabella completa" per generare un peaklist per lo spettro con intensità (Figura 5). Questo elenco di picco riporto le informazioni di posizione dall'elenco stored picco.
- Esportare gli elenchi di picco da diversi set di dati in un foglio di calcolo o altro programma di matematico per l'analisi selezionando la funzione "Esporta".
- Calcolare la variazione delle intensità di picco con la funzione "intensità di picco in intensità di complesso spettro/picco nello spettro di proteina" per ogni picco. I valori possono essere convertiti alla variazione percentuale da moltiplicazione di 100. Si noti che picchi di lavoro sono anche utili, anche se l'intensità di picco sono più facili da misurare per cime che sono posizionati vicino a vicenda, come sono solitamente il caso per le proteine con un'alta densità di picchi relativamente ampie.
4. su microscala submicronica (MST)
-
Preparazione del ligando E proteina-PRD
- Scambiare il ligand in un buffer compatibile MST di dialisi fino a 800 μL di proteina in un'unità di mini-dialisi 3,5 kDa sospesa in 1 L di 20 mM HEPES, pH 7.5, 10 millimetri di NaCl, mescolando lentamente a 4 ° C durante la notte.
- Concentrare il ligando utilizzando un'unità di ultrafiltrazione centrifugo (3 kDa MWCO) mediante centrifugazione a 14 000 x g per 10 min trasferimento proteine concentrate in una provetta pulita.
- Centrifugare il ligando a 21.000 x g per 10 min e attentamente trasferire il surnatante in una provetta nuova per rimuovere qualsiasi proteina precipitata. Determinare la concentrazione del ligando utilizzando l'assorbanza a 280 nm e il coefficiente di estinzione ligando. Aggiungere 10% Tween-20 per ottenere una concentrazione finale di 0,015%. Tween-20 è aggiunto il buffer di analisi per impedire l'adsorbimento non specifico ai capillari. Il tampone del saggio finale che viene utilizzato per gli esperimenti di MST è 20 mM HEPES, pH 7.5, 10 millimetri di NaCl, 0.015% Tween-20.
-
Preparazione di tingere-contrassegnati Target della proteina VimRod
- Ricostituire il colorante rosso-tris-NTA aggiungendo 50 μL di 1X PBS-T in dotazione con il rosso-tris-NTA dare una concentrazione di 5 μM. Pipettare 2 aliquote del μL nei tubi di 200 μL e conservare a-20 ° C.
- Diluire la proteina dell'obiettivo a 0,34 μM con tampone del saggio. Aggiungere 58 μL di destinazione ad un'aliquota di 2 μL di colorante rosso-tris-NTA e incubare per 30 min a temperatura ambiente. La concentrazione finale di destinazione tingere-contrassegnati è 0,33 μM. Centrifugare il contrassegnati dell'obiettivo a 21.000 x g per 10 min e attentamente trasferire il surnatante in una provetta nuova per rimuovere qualsiasi precipitato. Il colorante rosso-tris-NTA si lega alle proteine tramite il tag His6 e ha una costante di dissociazione di legame (KD) nella gamma sub-nanomolari. È efficace al 100% associato alla proteina bersaglio quindi nessun ulteriore purificazione è necessaria.
- Accendere lo strumento MST e aprire il Software di controllo. Selezionare l'impostazione di rosso per il colorante rosso-tris-NTA. Attivare il controllo della temperatura a 25 ° C.
- Il pre-test per convalidare l'etichettatura del bersaglio e visualizzate per aggregazione o adsorbimento per la cuvette. Una valutazione di questi parametri viene fornita automaticamente.
- 17 mix μL di tampone del saggio e 3 µ l di proteina bersaglio e mescolare pipettando. Riempire due capillari standard immergendoli nella destinazione diluita e aspirare il liquido al centro del capillare. Posizionare i capillari nel vassoio e negli strumenti. Avviare la misurazione.
- Esaminare i risultati alla ricerca di un livello sufficiente di fluorescenza e nessun segno di adsorbimento (distorsione nella forma di capillare) o l'aggregazione (distorsioni nella traccia MST) che verrà contrassegnata in software di analisi. Se i risultati sono positivo mossa verso il ligando passaggi, se non un buffer diversi deve essere provato o la quantità di Tween-20 può essere aumentata a 0.05% o superiore.
-
Preparazione della serie di diluizioni di ligando E-PRD
- Preparare una serie di diluizioni di etichettatura 16, 200 tubi μL da 1-16.
- Aggiungere 17 μL del ligando a 1.17 x una maggiore concentrazione rispetto alla concentrazione di ligando massimo desiderata di 1. Questo volume è due volte la quantità necessaria (8,5 μL) come un'aliquota sarà quindi trasferita al tubo successivo nella serie. Il volume finale del dosaggio è 10 μL quindi 8,5 μL di 1,17 concentrazione del ligando saranno diluiti a 1 x tramite l'aggiunta di 1,5 μL della proteina bersaglio, VimRod. Questa concentrazione massima scelta deve essere almeno 20 volte il valore di KD stimato.
- Aggiungere il 8,5 μL di tampone Tubes2-16 utilizzando un nuovo puntale per ogni aliquota. Riutilizzare i puntali per pipette possono influenzare l'accuratezza (per consulenza accurata pipettaggio Vedi istruzioni). Trasferire 8,5 µ l di legante da 1 a Tube2 rilasciando lentamente la soluzione nel buffer di analisi senza generare bolle. Mescolare il buffer e ligando pipettando su e giù almeno 6 volte, senza generare bolle. Il PRD E nel tubo più concentrato era 1,5 mM e la VimRod con etichetta era presente ad una concentrazione finale di 50 nM.
- Trasferire 8,5 µ l di legante da Tube2 a Tube3 e mescolare con il tampone. Ripetere la diluizione seriale fino a quando tutti i tubi hanno avuto ligando aggiunto. Scartare 8.5 μL da Tube16 modo che tutti i tubi contengono 8,5 μL di ligand in una serie di diluizioni.
-
Preparazione dell'esperimento reazione vincolanti e MST
- Aggiungere 1,5 μL della proteina contrassegnati dell'obiettivo per ciascuna delle provette e mescolare delicatamente pipettando su e giù facendo attenzione a evitare bolle. Incubare per 15 min.
- Selezionare la modalità esperto per l'analisi di associazione e immettere i parametri per una serie di diluizioni seriali. Assicurarsi che sia impostato il controllo della temperatura a 25 ° C, il potere di eccitazione è impostato su 40% e il potere di MST di mezzo. Questi parametri devono essere ottimizzati per gli altri partner di associazione in fase di studio.
- Riempire i vasi capillari con le reazioni di associazione e posizionare nel cassetto del capillare. Caricare il vassoio nello strumento, attendere che la temperatura di riprendere il 25 ° C e avviare la misurazione.
-
Analisi dei dati
- Aprire il file di analisi di associazione del software di analisi di affinità. Rivedere le scansioni capillare; Livelli di fluorescenza non devono variare di oltre il 10% dalla media, nessuna aggregazione o adsorbimento dovrebbe essere rilevato come descritto nella sezione 3.6.
- Selezionare l'analisi di MST sul pannello destro e trascinare i dati di analisi nell'analisi impostato. Se esistono più esecuzioni della stessa analisi di associazione di destinazione-ligando in condizioni identiche possono essere unite facendo cadere nello stesso set. In alternativa, ogni esecuzione può essere eliminato in analisi in modo indipendente.
- Passare alla scheda Dose risposta adatta per rappresentare graficamente i dati. Scegli il modello KD se si prevede che un sito di legame singolo. Il modello di Hill è anche un'opzione per più siti di legame con comportamento cooperativo. Punti di valore erratico che non hanno superato il controllo di qualità possono essere rimosso dal fit in questa fase. Set di Unione con saggi multipli saranno mediati ed errori calcolati come la deviazione standard.
- Aprire la scheda finale e confrontare i risultati tra tutte le trame in un unico grafico. Recuperare i risultati di raccordo per ogni curva della tabella che viene generato. Esportare i dati o le curve componibile ad altri software di presentazione o analisi se lo si desidera.
Risultati
Il dominio E-PRD (residui 1822-2014 clonati nel pProEX-HTC) del gene umano envoplakin e il dominio di VimRod (residui 99-249 clonati in pET21a) di vimentin umano4 erano espressi con i tag His6 e purificato. Figura 6 e Figura 7 dimostrare i livelli di purezza di VimRod (18,8 kDa) ed E-PRD (21,8 kDa) ottenuti da questo metodo di purificazione della proteina. La rimozione di His6 tag dal costrutto E-PRD è essenziale per gli esperimenti di MST come la proteina di VimRod con l'etichetta utilizzando un colorante di associazione di tag His6 e qualsiasi E-PRD mantenendo il relativo tag di His6 possono competere per il legame della tintura. La seconda colonna IMAC dopo la scissione del tag con proteasi TEV rimuove la proteasi TEV, il tag spaccato e qualsiasi ApoAlert His6-E-PRD che è rimasto. Il passaggio finale di lucidatura della purificazione è la cromatografia di esclusione di formato. Pur essendo entrambe proteine di dimensioni simili, il VimRod eluisce dalla colonna a un 51 mL mentre il picco di eluizione E-PRD è centrato a 72 mL dove ci si aspetterebbe un monomero della proteina di queste dimensioni. L'apparente aumento di dimensioni del VimRod è probabilmente dovuto la sue caratteristiche come una lunga asta filamentosa a forma di proteina come analiticamente ultracentrifuga gli esperimenti hanno dimostrato che il VimRod è stato monomerico4. Rese più basse della proteina sono ottenute dalle colture coltivate in M9 rispetto a quelli da brodo ricco a causa di una quantità inferiore di cellule si producono in media minimi. La crescita iniziale di grandi colture starter per le preparazioni di M9 in TB permette il miglioramento delle rese di cella mantenendo nella misura di 15N etichettatura necessarie per gli esperimenti NMR.
15N -1H HSQC sono stati acquisiti per la wild-type e mutanti di R1914E di E-PRD in presenza o assenza di VimRod (Figura 8A-8D). Lo spettro di E-PRD in Figura 8A Mostra il numero previsto di picchi ben risolti, indicativi di una proteina correttamente piegata. In presenza di VimRod (Figura 8B) lo spettro mostra ad ampliare la vasta gamma e la scomparsa di picco, corrispondente a associazione tra E-PRD e VimRod. Questa associazione è perso dalla mutazione di R1914E come evidenziato dal confronto di Figura 8 e 8D. Piccolo cambiamento è osservata nello spettro con l'aggiunta di VimRod per il mutante di R1914E che indica una mancanza di associazione tra questo mutante E-PRD e VimRod. Le intensità di picco E-PRD in presenza/assenza di VimRod sono state confrontate e tracciate come le intensità di picco relativa in Figura 8E, che indica l'intervallo del picco ampliando nel complesso E-PRD. Il mutante di R1914E di E-PRD (non mostrato) mantenuto circa il 97% delle cime a 20% o intensità di picco più elevate in presenza di VimRod rispetto a circa il 20% per il tipo selvaggio (Figura 8E). Questo rappresenta una perdita del mutante di punto di funzione, con ulteriori mutanti avendo effetti intermedi anche essendo stato studiato4.
Per convalidare e quantificare l'associazione di analisi VimRod ed E-PRD MST utilizzando His6-VimRod marcati con colorante fluorescente rosso-tris-NTA come destinazione mista con la diminuzione della concentrazione del ligando E-PRD da 1,28 mM a 39,1 nM sono stati effettuati. Tre titolazioni di associazione sono stati effettuati e i risultati sono una media e illustrati nella Figura 9. I dati erano idonei con un modello standard di one-sito ligando e dato KD di 25,7 ± 2.1 μM. Valutazione dell'associazione tra VimRod ed E-PRD di risonanza plasmonica di superficie ha dato un simile valore KD 19.1 ± 1.3 μM4.

Figura 1: Screen Capture del Setup dell'esperimento NMR. La finestra mostrata viene utilizzata per impostare un esperimento standard per raccogliere un dataset HSQC. Esperimento i parametri vengono letti nella adiacente all'esperimento. L'esperimento ZGPR mostrato viene scelto come un esperimento iniziale per caricare i parametri standard e solvente protone dipendente. La finestra di titolo viene utilizzata per immettere dettagli sperimentali per scopi di conservazione. Per raccogliere lo spettro HSQC l'esperimento ZGPR viene sostituito con SFHMQC3GPPH. Clicca qui per visualizzare una versione più grande di questa figura.
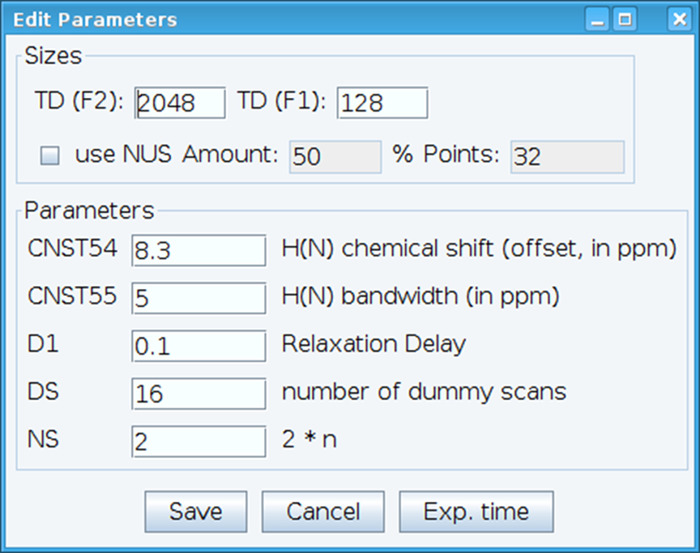
Figura 2: regolazione dei parametri sperimentali NMR. La finestra mostrata è utilizzata per l'immissione di parametri fondamentali per la sequenza di impulsi NMR al fine di ottimizzare il segnale. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: elaborazione dei dati NMR. Parametri utilizzati per l'elaborazione di ciascuna delle due dimensioni dello spettro NMR sono indicati, con le frecce che indica quelli che solitamente sono regolati. Clicca qui per visualizzare una versione più grande di questa figura.
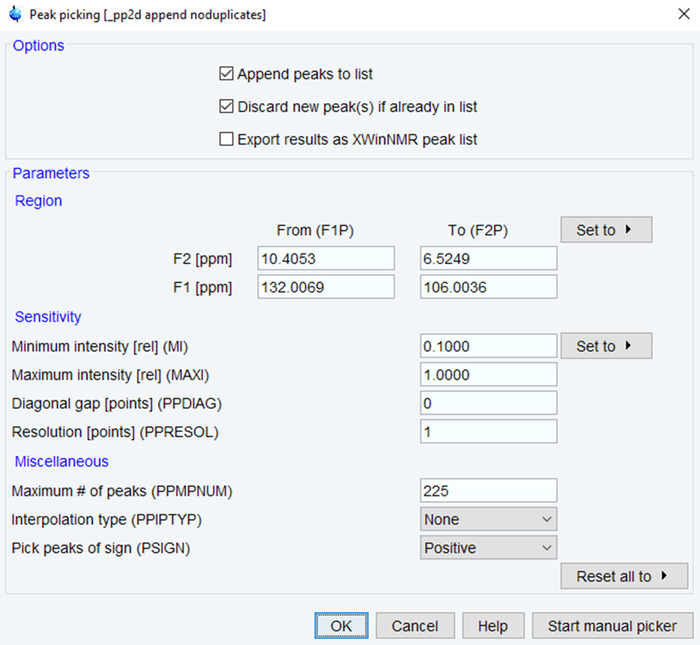
Figura 4: parametri per la raccolta di picco NMR. I parametri utilizzati per la raccolta di NMR picchi nello spettro NMR trasformato vengono visualizzati con valori tipici. Regolare la gamma ppm, intensità e numero di picchi per ottimizzare gli spettri. Clicca qui per visualizzare una versione più grande di questa figura.
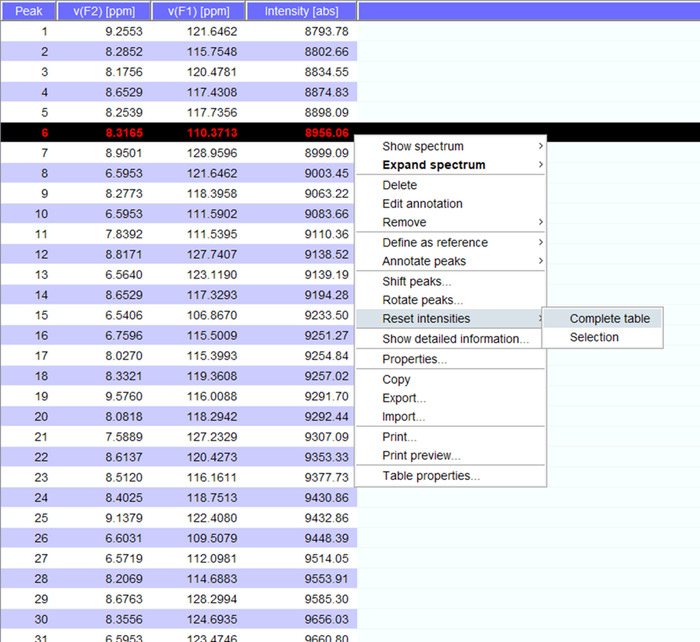
Figura 5: rappresentante Peaklist con intensità. Ogni picco che viene raccolto nello spettro NMR viene dato un numero e relativo 1H e 15N chimico sposta e intensità del segnale vengono visualizzati. Questo peaklist può quindi essere utilizzato per confrontare spettri ottenuti in presenza/assenza di un partner interagente. Clicca qui per visualizzare una versione più grande di questa figura.
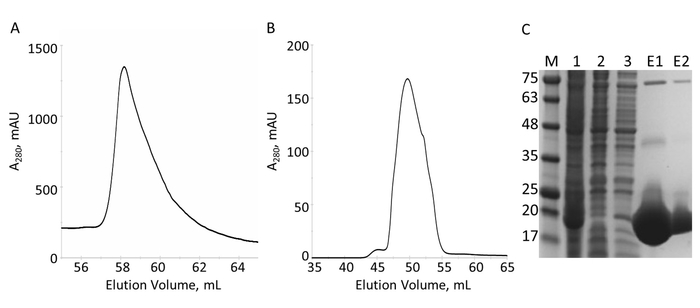
Figura 6: purificazione di VimRod His6-etichetta di IMAC e s. A. Il cromatogramma per l'eluizione dalla colonna IMAC Mostra un picco principale di VimRod. B. il cromatogramma per l'eluizione da colonna S Mostra un picco principale. C. SDS-PAGE delle frazioni raccolte nel corso di purificazione: standard MW con il MW indicato in kDa a sinistra del gel (M), lisato cellulare (1), IMAC flusso continuo (2), lavaggio (3), riunita eluizione (E1), riunito S eluizione (E2). Bande visibili alle più alti pesi molecolari in vicoli E1 ed E2 sono oligomeri di puro VimRod come confermato mediante western blot (dati non mostrati). Clicca qui per visualizzare una versione più grande di questa figura.
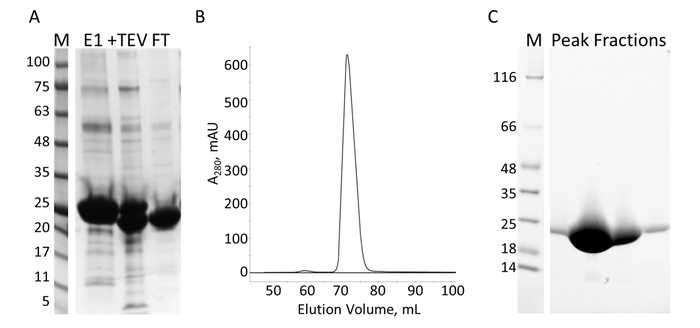
Figura 7: purificazione del E-PRD di IMAC e s. A. SDS-PAGE della purificazione IMAC mostrando gli standard di peso molecolare con il MW indicato in kDa a sinistra del gel (M) e l'eluito dalla prima colonna IMAC (E1), TEV il clivaggio di prodotti (+ TEV) e il flusso attraverso dalla seconda colonna IMAC (FT). B. il cromatografo dalla colonna S Mostra un picco principale. C. SDS-PAGE delle norme peso molecolare (M) e le frazioni da picco S. Clicca qui per visualizzare una versione più grande di questa figura.
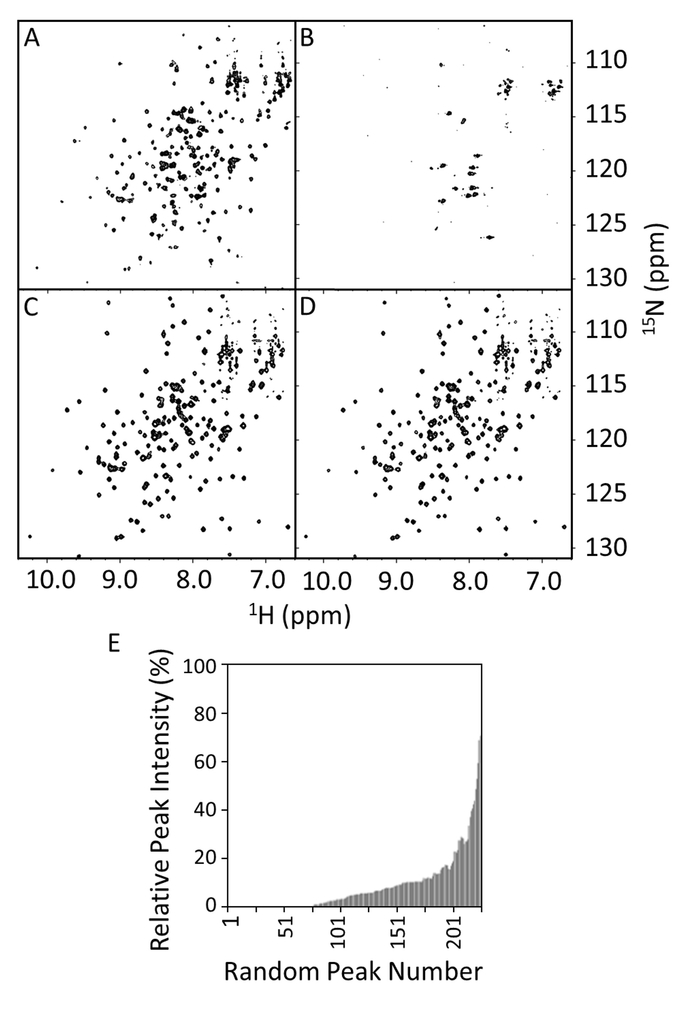
Figura 8: HSQC spettri di Wild-type e mutanti di R1914E di E-PRD in presenza e assenza di VimRod. Gli spettri HSQC Visualizza selvaggio-tipo E-PRD (100 µM) in 20 mM Tris-HCl, 150 mM NaCl, 1 millimetro DTT, pH 7 nell'assenza o nella presenza di 50 µM (A) VimRod (B). Pannelli C e D sono gli spettri HSQC del mutante R1914E (100 µm) nell'assenza o nella presenza di 50 µM VimRod, rispettivamente. Nel pannello E il picco di15N relativa 1H - intensità del E-PRD con o senza associazione di VimRod vengono visualizzati come una funzione del numero di picco, che è assegnato arbitrariamente e non basato sulla posizione di sequenza. Questi valori possono essere utilizzati per definire un valore soglia di significatività per riduzione di intensità di picco con l'aggiunta di un legante. Se le assegnazioni sono disponibili, i valori significativi spesso possono essere visto eseguire il mapping a una area di associazione. Clicca qui per visualizzare una versione più grande di questa figura.
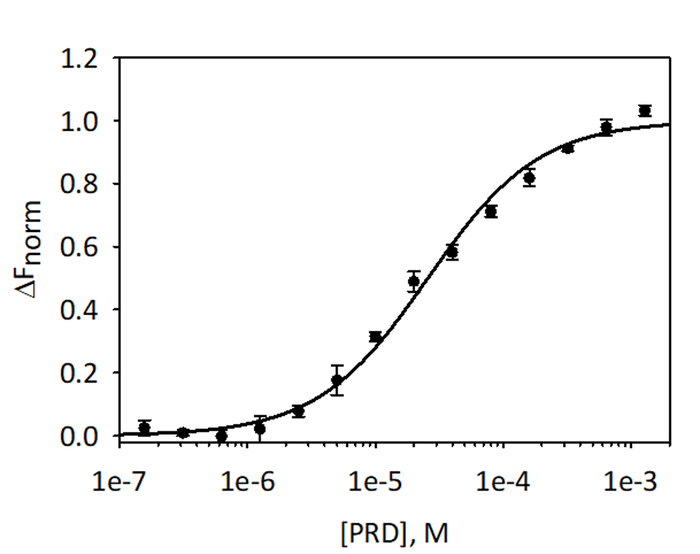
Figura 9: associazione di E-PRD a VimRod. E-PRD è stata diluita in una serie di diluizioni da 1,28 mM a 39,1 nM e incubati con VimRod con etichetta prima di eseguire analisi di MST. Dati da tre saggi indipendenti sono stati combinati. I dati erano idonei a un modello KD dando KD di 25,7 μM con una fiducia KD ± 2.1 μm. Clicca qui per visualizzare una versione più grande di questa figura.
| Reagente | Quantità |
| Fosfato di sodio bibasico (anidro) | 6,0 g |
| Fosfato di potassio monobasico (anidro) | 3,0 g |
| Cloruro di sodio | 0,5 g |
| H2O | Fino a 950 mL |
Tabella 1. M9 media per l'etichettatura isotopica.
| Reagente | Quantità |
| 15 NH4Cl | 1,0 g |
| Glucosio (o 13C-glucosio) | 2,0 g |
| 1 M MgSO4 | 2 mL |
| 50mm CaCl2 | 4 mL |
| 20 mg/mL della tiamina | 1,0 mL |
| 3 mM FeCl3 | 400 Μ l |
| Metallo Mix (tabella 3) | 500 Μ l |
| H2O | Fino a 50 mL |
Tabella 2. Miscela nutriente per il completamento della M9 media.
| Reagente | Quantità |
| 4 mM ZnSO4 | 323 mg |
| 1 mM MnSO4 | 75,5 mg |
| 4.7 mM H3BO3 | 145 mg |
| 0,7 mM CuSO4 | 55,9 mg |
| H2O | Fino a 500 mL |
Tabella 3. Supplemento di mescolare metalli per arricchire la miscela nutriente di MT.
| Campione | tampone A1 (µ l) di 1 mM E-PRD | tampone A (µ l) di 1mM VimRod | Buffer di un (µ l) | 200 µM DSS in D2O (µ l) | Tampone B2 (µ l) | Totale Volume (µ l) |
| E-PRD da solo | 50 | 0 | 50 | 50 | 350 | 500 |
| E-PRD + VimRod | 50 | 50 | 0 | 50 | 350 | 500 |
| 1 Tampone a: 20 mM Tris-HCl, 1 millimetro DTT, pH 7 | ||||||
| 2 Tampone b: 23 mM Tris-HCl, 1,14 mM DTT, pH 7 | ||||||
Tabella 4. Preparazione del campione NMR.
Discussione
Il 2D 15esperimento NMR N-risolto è uno dei più diffusi metodi per mostrare come due molecole interagiscono. È il metodo più ricco di informazioni che permette segnali di entrambi i partner di essere continuamente monitorati nel corso di un esperimento di titolazione in stato di soluzione. Anche se in genere qualitativa nel caso di grandi complessi, il metodo utilizzabile anche in casi favorevoli per misurare l'affinità di legame dove segnali NMR possono essere rintracciati in spettri ad alta risoluzione. Quando le assegnazioni possono essere fatte convenientemente, come nel caso di molte proteine sotto 20 kDa in dimensione, i siti di legame possono essere mappati. Saggi complementari quali MST forniscono informazioni quantitative sulle interazioni in soluzione e richiedono meno proteine negli Stati senza etichetta. Confronto di mutanti di associazione dei dati è utile per fornire controlli per garantire che le interazioni evidenziati dalla linea NMR ampliamento sono genuini e non artefatti di, ad esempio, le modifiche di aggregazione o viscosità.
Espressione della proteina
Semplificazione del processo di espressione riduce la quantità di produzione di proteine intensivo di manodopera. Parte di questo processo di ottimizzazione prevede l'individuazione di un appropriato ceppo di e. coli per l'espressione recombinant della proteina. Preferenza di ceppo dipende da elementi compresa la natura del vettore in uso e, più specificamente, il massimo della stabilità della proteina ricombinante è espresso7. Il rischio di degradazione della proteina eterologa da endogeno e. coli possono essere ridotto uso della proteasi proteasi carenti e. coli come il ceppo BL21. Per geni contenenti codoni rari, un ceppo ad esempio RIPL BL21-CodonPlus (DE3) può essere preferito. Questo ceppo unisce la natura carente di proteasi del ceppo BL21 copie aggiuntive endogeno del codone rara tRNAs per leucina, isoleucina, prolina e arginina. In alternativa, è possibile evitare codoni rari che possono compromettere la sovraespressione ordinando un costrutto codone-ottimizzato da una fonte commerciale. Molti ceppi di e. coli sono disponibili per l'espressione genica ricombinante, ciascuno ottimizzato per elusione di un particolare problema durante espressione7. Nel caso di questo studio, il ceppo carente di proteasi standard BL21 (DE3) prodotto quantità adeguate di proteine solubili per purificazione successiva e analisi.
Purificazione della proteina
Il protocollo di purificazione per una data proteina spesso è unico nel senso che ogni proteina rimane stabile e solubile in diverse condizioni quali temperatura, concentrazione di sale o pH. L'efficacia complessiva della purificazione mediante cromatografia di affinità è anche sensibile alla concentrazione di eluizione specie come imidazolo alle varie fasi durante il processo di purificazione. In questo lavoro, condizioni di buffer critico per IMAC erano pH per l'E-PRD e la concentrazione di imidazolo per il VimRod. Un pH di 7,5 era necessario per evitare la precipitazione del E-PRD seguendo eluizione iniziale dalla colonna IMAC. Per la purificazione di IMAC di VimRod, aumentando la concentrazione di imidazolo da 30 a 50 mM durante la fase di lavaggio della colonna è stato trovato per avere un miglioramento sostanza nella purezza delle frazioni eluizione finale. Per il passaggio di eluizione, anche aumentando la concentrazione di imidazolo da 250 a 350 mM è stato trovato per migliorare la resa dell'eluizione finale. I tentativi iniziali di eluire la proteina usando imidazolo 250 mM ha portato alla incompleta eluizione di VimRod come rivelato da una striscia di imidazolo 1m finale della colonna (dati non mostrati). Aumento della concentrazione di imidazolo e 350 mM per l'eluizione è stata sufficiente a recuperare tutta la proteina associata alla colonna. S può avere una duplice funzione perché agisce come un passaggio di lucidatura per purificazione della proteina durante l'esecuzione contemporaneamente cambio di buffer. Cambio di buffer è un passo fondamentale per l'analisi successiva associazione poiché rimuove l'imidazolo utilizzato per eluire la proteina His6-etichetta. Serve anche come un'opportunità per cambiare le condizioni come la concentrazione di sale o pH, che possono influenzare l'efficacia di determinate tecniche a valle o saggi. Proteina spostamento termico (PTS) può essere utilizzato per identificare il buffer ottimale per i dosaggi a valle, soprattutto per coloro che necessitano di proteina stabile per prolungati periodi di tempo a temperatura ambiente8,9.
Analisi di associazione
Proteina che viene preparata al momento è critica per analisi accurata obbligatorie, anche se congelata proteina può essere usato anche come i risultati vengono confrontati. Proteine filamentose come vimentin multimerize in un modo dipendente dalla sale e pH, e quindi la soluzione c'è bisogno di essere ottimizzato e oligomerico stato stimato da un metodo quale SEC10,11, dynamic light scattering condizione 12 o ultracentrifugazione analitica13,14,15. La spettroscopia NMR è adatta per la misurazione interazioni ligando di piccole proteine a risoluzione atomica. Tuttavia, quando una proteina interagisce con la molecola più grande, più lento barilatura deriva, e ciò si traduce in perdita di segnali, che può confermare l'associazione anche se esso non necessariamente consentire il mapping dei siti, che richiederebbero anche assegnazione di almeno di legame spina dorsale risonanze. In questo scenario, gli esperimenti NMR non consentono l'identificazione del sito di interazione. Quindi il sito diretto mutagenesi viene applicata per identificare i residui critici necessari per l'associazione. Tali mutanti pertanto non manifestano perdita di segnale. In questo protocollo, una forma mutante con una sostituzione alla posizione 1914 mantiene le intensità di picco in presenza di VimRod e conferma pertanto la rottura dell'interazione di E-PRD e VimRod. Assegnazione delle risonanze sidechain e backbone vorrei aggiungere valore a questo approccio, in particolare come la struttura per il gratuito E-PRD è stata risolta dalla cristallografia a raggi x4. Future applicazioni di RMN comprendono la caratterizzazione delle interazioni complesse fra le più grandi molecole e potranno beneficiare di magneti di campo ultra alto e l'uso di altri gruppi osservabile come 13C-etichetta e gruppi metile trifluoro come reporter.
MST ha una serie di vantaggi per lo studio di associazione interazioni16. I soci di associazione sono liberi in soluzione e non immobilizzati. Analisi della qualità dei campioni sono incorporata nel software con controllo qualità reporting dell'aggregazione, adsorbimento al capillari o insufficiente etichettatura fluorescente della molecola target. Piccole quantità di destinazione vengono in genere utilizzati, la concentrazione del target con etichetta è di solito tra 20-50 nM in un 10-20 μL volume/reazione. Questo protocollo utilizza volumi di reazione molto piccolo (10 μL) per massimizzare la concentrazione del ligando che possa essere raggiunti nelle titolazioni consentendo l'interazione di legame debole essere caratterizzato. Ciò richiede di pipettaggio accurata e la cura per evitare di introdurre bolle, mescolando ancora accuratamente. Miscelazione adeguata è fondamentale per misure di fluorescenza preciso, coerente lungo la serie di diluizioni seriali. La quantità di Tween-20 negli esperimenti MST è stato ridotto da standard 0.05-0.015% per ridurre la tendenza a creare bolle e migliorare la miscelazione.
Il colorante rosso-Tris-NTA fornisce un modo rapido, facile e conveniente per fluorescente etichettare qualsiasi proteina che ha una sua etichetta. L'etichettatura è effettivamente completo in soli 30 minuti ed è molto stretto in modo che non è necessaria alcuna procedura di rimozione tintura. Non sono modifiche ai residui dell'amminoacido nella proteina che potrebbe alterare le proprietà di legame del ligando. Un avvertimento è che solo la proteina di essere etichettato dovrebbe avere un tag His6. Ciò ha richiesto la scissione del tag dalla proteina legante, E-PRD e la rimozione del tag e ApoAlert E-PRD con un secondo passaggio di colonna IMAC. Se possibile, la proteina legante dovrebbe essere preparata senza l'uso di un suo tag. In alternativa, proteine possono essere etichettate in modo covalente con un fluoroforo attraverso ammina accoppiamento a residui di lisina o del tiolo di accoppiamento a residui di cisteina. Tuttavia, è necessario prestare attenzione quando l'utilizzo di tali sistemi dal collegamento covalente di un fluoroforo può influenzare interazioni elettrostatiche o polare associazione basandosi sui residui di lisina o cisteina. La quantificazione delle affinità tra la VimRod ed E-PRD da MST obbligatoria era insolitamente sensibile alla concentrazione di sale. Questo problema è stato attenuato da dialisi inizialmente la destinazione e il ligando in stesso lotto di tampone del saggio. Ciò nonostante, saturazione della curva associazione MST non poteva essere raggiunti quando si esegue il test di MST in presenza di 150 mM NaCl, a causa del comportamento complesso di VimRod. Dati affidabili e completi è stati ottenuti una volta che la concentrazione di NaCl è stata abbassata a 10 mM che permette il calcolo preciso della KD. Quindi, attenta ottimizzazione delle condizioni di soluzione e confronto con saggi complementari sono raccomandato per ottenere risultati affidabili. Inoltre, MST può essere usato per quantificare la dipendenza del sale per un'interazione specificata, quantificare stechiometrico proprietà delle interazioni della proteina, monitor il protein folding e sonda in enzima cinetica17.
Divulgazioni
Gli autori non divulgare conflitti di interesse.
Riconoscimenti
Questo progetto è stato supportato da NSERC RGPIN-2018-04994, Campus Alberta Innovation Program (RCP-12-002C) e Alberta Prion Research Institute / soluzioni di Alberta Innova Bio (201600018), assegnato a Mangano e Genome Canada e Fondazione canadese per Sovvenzioni di innovazione per la metabolomica Innovation Centre (TMIC) e NANUC.
Materiali
| Name | Company | Catalog Number | Comments |
| Monolith NT.115, includes control and analysis software | NanoTemper Technologies | MO-G008 | Instrument for microscal thermophoresis |
| His-Tag Labeling Kit RED-Tris-NTA | NanoTemper Technologies | MO-L008 | RED-tris-NTA dye for MST |
| Standard capillaries | NanoTemper Technologies | MO-K022 | capillaries for MST |
| HisTrap HP, 5 mL | GE Healthcare | 17524801 | IMAC column |
| HisPur Ni-NTA resin, 100 mL | Thermo Fisher Scientific | 88222 | IMAC resin |
| HiLoad 16/600 Superdex 75 pg | GE Healthcare | 28989333 | SEC column |
| TEV protease | Sigma Aldrich | T4455 | cleavage of his tag from E-PRD |
| BL21(DE3) Competent E. coli | New England Biolabs | C2527H | cells for protein expression |
| cOmplete Protease Inhibitor Cocktail Tablets EDTA-Free | Sigma Aldrich | 11873580001 | protease inhibitors for protein purification by IMAC |
| TCEP, Tris(2-carboxyethyl)phosphine hydrochloride | Sigma Aldrich | C4706 | reducing agent for protein purification |
| Reagents (HEPES, NaCl, etc) | Sigma Aldrich | various | Preparation of media and buffers |
| Ammonium chloride (15N, 99%) | Cambridge Isotope Laboratories | NLM-467 | isotope labelling for NMR |
| D2O, Deuterium Oxide (D, 99.8%) | Cambridge Isotope Laboratories | DLM-2259 | NMR sample preparation |
| DSS, Sodium 2,2-dimethyl-2-silapentane-5-sulfonate-D6 (D,98%) | Cambridge Isotope Laboratories | DLM-8206 | reference for NMR |
| Precision 5 mm NMR Tubes, 7” long | SJM/Deuterotubes | BOROECO-5-7 | NMR tubes |
| NMR spectrometer (14.1 Tesla) | Bruker | acquisition of NMR data | |
| TCI 5mm z-PFG cryogenic probe | Bruker | acquisition of NMR data | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Bruker TopSpin 4.0.1 | Bruker | processing of NMR data | |
| MO.Control | NanoTemper Technologies | included with Monlith NT.115 | |
| MO.Affinity Analysis | NanoTemper Technologies | included with Monlith NT.115 |
Riferimenti
- Vinogradova, O., Qin, J. NMR as a unique tool in assessment and complex determination of weak protein-protein interactions. Topics in Current Chemistry. 326, 35-45 (2012).
- McKercher, M. A., Wuttke, D. S. NMR Chemical Shift Mapping of SH2 Peptide Interactions. Methods in Molecular Biology. 1555, 269-290 (2017).
- Al-Jassar, C., Bikker, H., Overduin, M., Chidgey, M. Mechanistic Basis of Desmosome-Targeted Diseases. Journal of Molecular Biology. 425 (21), 4006-4022 (2013).
- Fogl, C., et al. Mechanism of intermediate filament recognition by plakin repeat domains revealed by envoplakin targeting of vimentin. Nature Communications. 7, (2016).
- Walker, J. M. . The Protein Protocols Handbook. , (2002).
- Wishart, D. S., et al. 1H, 13C and 15N chemical shift referencing in biomolecular NMR. J. Biomol. NMR. 6, 135-140 (1995).
- Casali, N. Escherichia coli Host Strains. E. coli Plasmid Vectors. , 27-48 (2003).
- Ericsson, U. B., Hallberg, B. M., DeTitta, G. T., Dekker, N., Nordlund, P. Thermofluor-based high-throughput stability optimization of proteins for structural studies. Analytical Biochemistry. 357 (2), 289-298 (2006).
- Kozak, S., et al. Optimization of protein samples for NMR using thermal shift assays. Journal of Biomolecular Nmr. 64, 281-289 (2016).
- Burgess, R. R. A brief practical review of size exclusion chromatography: Rules of thumb, limitations, and troubleshooting. Protein Expression and Purification. , (2018).
- Kunji, E. R. S., Harding, M., Butler, P. J. G., Akamine, P. Determination of the molecular mass and dimensions of membrane proteins by size exclusion chromatography. Methods. 46 (2), 62-72 (2008).
- Stetefeld, J., McKenna, S. A., Patel, T. R. Dynamic light scattering: a practical guide and applications in biomedical sciences. Biophysical Reviews. 8 (4), 409-427 (2016).
- Patel, T. R., Winzor, D. J., Scott, D. J. Analytical ultracentrifugation: A versatile tool for the characterisation of macromolecular complexes in solution. Methods. 95, 55-61 (2016).
- Pearson, J. Z., Cole, J. L., et al. Chapter One - Next-Generation AUC Adds a Spectral Dimension: Development of Multiwavelength Detectors for the Analytical Ultracentrifuge. Methods in Enzymology. 562, 1-26 (2015).
- Gorbet, G. E., Pearson, J. Z., Demeler, A. K., Cölfen, H., Demeler, B., Cole, J. L. Chapter Two - Next-Generation AUC: Analysis of Multiwavelength Analytical Ultracentrifugation Data. Methods in Enzymology. 562, 27-47 (2015).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nature Communications. 1, 100 (2010).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon