Method Article
Interações de proteínas globulares e filamentosas por espectroscopia de ressonância magnética Nuclear (NMR) e Thermophoresis de microescala (MST) de medição
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para a produção e purificação de proteínas que são rotulados com isótopos estáveis e posterior caracterização das interações da proteína-proteína usando espectroscopia de ressonância magnética Nuclear (RMN) e microescala Experimentos de Thermophoresis (MST).
Resumo
Proteínas filamentosas como vimentina fornecem organização dentro das células, fornecendo um andaime estrutural com sites que ligam proteínas contendo plakin repete. Aqui, um protocolo para a detecção e medição de tais interações é descrito usando o domínio globular plakin de envoplakin e a bobina helicoidal da vimentina. Isto fornece uma base para determinar se uma proteína liga vimentina (ou proteínas filamentosas semelhantes) e medição da afinidade da interação. A proteína globular de interesse é rotulada com 15N e titulada com proteína vimentina em solução. Um espectro de NMR bidimensional é adquirido para detectar interações observando alterações na forma de pico ou mudanças químicas e para elucidar os efeitos das condições de solução, incluindo os níveis de sal, que influenciam a estrutura quaternária vimentina. Se a proteína de interesse vincula o ligante filamentoso, a interação de ligação é quantificada pelo MST usando as proteínas purified. A abordagem é uma maneira simples para determinar se uma proteína de interesse vincula um filamento e avaliar como alterações, tais como mutações ou condições de solução, afetam a interação.
Introdução
Interações entre proteínas permitem a formação de máquinas moleculares que criam ordem dentro das células. As interações individuais muitas vezes são fracas, mas geralmente contribuem para multivalentes complexos que podem ser dinamicamente regulamentado e cooperativo. Ensaios sensíveis que fornecem resolução atômica e informações quantitativas sobre tais interações complexas são necessários para deduzir os mecanismos e projetar intervenções como droga-como moléculas. Espectroscopia RMN é um método eficiente para a obtenção de tais informações sobre as interações da proteína e também é usada para o rastreio rápido para ligantes, incluindo aqueles que se ligam fracamente1. Os métodos NMR usados podem ser categorizados em aqueles que são proteínas observar ou ligante observar. Este manuscrito usa a primeira abordagem, em que é adquirido um espectro de um isótopo estável-rotulado de proteína que é comparativamente pequena (geralmente sob 20 kDa) e o ligante sem rótulo é titulado. Isto permitir que os resíduos rotulados envolvidos na interação a serem mapeados em casos favoráveis. Uma vez as formas complexas, há mudanças na forma de seus sinais NMR e os ambientes químicos de resíduos de interação que se manifestam como alterações na mudança química. A extensão de tais mudanças se correlaciona com o grau de envolvimento destes grupos na interação. Perturbações de deslocamento químico (CSPs) podem ser medidas comparando uma série de espectros NMR da proteína coletados na ausência e presença de quantidades variáveis do ligante. Para maiores ligantes ou interações complexas, a mudança na forma de pico ou intensidade pode ser medida para deduzir as interações.
O experimento 2D mais comum usado para detectar interações ligante é a 15N-heteronuclear quântica única correlação (HSQC) experimento2. Isso requer que uma proteína uniformemente ser rotulado com 15N, que é normalmente conseguida expressá-los como versões de afinidade-tag em Escherichia coli culturas bacterianas crescidas em 15N-enriquecido de mídia. Vinculação é aparente quando os espectros HSQC coletados durante a titulação são sobrepostos, revelando mudanças de pico para um subconjunto dos resíduos envolvidos na formação da complexa. A interação pode ocorrer no regime de troca rápida, onde os sinais de estado livre e ligante saturada colapsam em uma população média de pico. Como alternativa, no caso de troca lenta entre os Estados, ambos os sinais são observados com integrais que representam suas quantidades relativas. Enquanto NMR lineshape análise pode ser usada para estimar as afinidades de ligação, em alguns casos, métodos tais como o MST também provaram ser convenientes e fornecem validação cruzada das interações genuínas.
O exemplo fornecido é de duas proteínas encontradas dentro desmossomas. Eles mediam as junções entre as superfícies da célula e o citoesqueleto e mediam multivalentes interações entre máquinas de adesão celular e filamentos intermediários para manter a integridade da pele e tecidos do coração e resistindo de forças de cisalhamento. Doenças podem resultar quando desmosomal proteínas tais como desmoplakin ou vimentina estão comprometidas por mutações ou auto-anticorpos, levando a desestabilização das junções célula-célula, e, portanto, suas interações são de importância crítica3. A base estrutural de ligação do ligante por proteínas de desmosomal pode ser caracterizada por espectroscopia de RMN, enquanto as interações podem ser quantificadas pelo MST. Métodos aqui foram usados para caracterizar as interações entre domínios repetição plakin (PRDs) que muitas vezes estão presentes como conjuntos em tandem que oferecem básicos sulcos e vimentina, um filamento intermediário que interage através de uma superfície ácida oferecida pela sua helicoidal pacote4. Estes complexos são formados na membrana da célula onde eles de ancoragem para os filamentos intermediários do citoesqueleto celular para desmossomas que conectam células adjacentes, formando assim uma rede de ligações adesivas que irradia ao longo de um tecido.
Protocolo
1. expressão da proteína recombinante
-
Expressão do PRD Envoplakin (E-PRD) e vimentina 99-249 (VimRod)
- Transforme células BL21(DE3) de Escherichia coli com o plasmídeo contendo o gene desejado. Espalhe as células em placas de ágar, contendo 100 ampicilina µ g/mL. Incube as placas a 37 ° C durante a noite.
- Escolher uma única colônia e inocular 20 mL de caldo fantástico (TB) contendo 100 ampicilina µ g/mL para selecionar para o plasmídeo. Cresce a cultura a 37 ° C, com agitação (180 rpm) durante a noite.
- Transferi a cultura inteira 20 mL para 1 L de TB contendo 50 ampicilina µ g/mL. Incubar a cultura, a 37 ° C, com agitação a 180 rpm até o OD600 = 0.6-0.8.
- Reduzir a temperatura de 18 ° C e induzir a expressão de proteínas adicionando isopropílico β-D-1-thiogalactopyranoside (IPTG) a uma concentração final de 1 mM. Continue a incubação a 18 ° C, com agitação a 160 rpm durante a noite para permitir a expressão da proteína.
- Recolher as células por centrifugação a cultura a 8.000 x g, durante 15 min. decantar e descartar o sobrenadante.
- Lave as células colhidas por resuspending o centrifugado em cerca de 40 mL de fosfato tamponado fisiológico (PBS: tampão de fosfato de 20 mM, pH 7,4, 120 mM de NaCl). Transfira a ressuspensão para um tubo de 50 mL. Centrifugar novamente 8.000 x g por 15 min.
- Decantar e descartar o sobrenadante. Imediatamente iniciar o protocolo de purificação ou congelar as pelotas de célula a-20 ° C para uso futuro.
-
Expressão da proteína desvendar rotulada
- Transformar células de Escherichia coli BL21(DE3) e preparar uma cultura de acionador de partida de 20 mL como em 1.1.1-1.1.2.
- Transferi a cultura inteira 20 mL para 1 L de enriquecido TB contendo uma adicional 4,0 g triptona, 5,0 g de NaCl e 100 µ g/mL ampicilina. Incubar a cultura, a 37 ° C, com agitação a 160 rpm até o OD600 = 1.6-1.9.
- Colheita da cultura de 1L por centrifugação a 8000 x g, durante 15 min. decantar e descartar o sobrenadante.
- Lave o centrifugado por resuspending suavemente em cerca de 40 mL de PBS e transfira a ressuspensão para um tubo de 50 mL.
- Centrifugar novamente a 8.000 x g, durante 15 min. decantar e descartar o sobrenadante.
- Ressuspender as células em 20 mL de M9 media mínimos (tabela 1) e a transferência para o restante da 950 mL de M9 media mínimos contendo 100 ampicilina µ g/mL.
- Adicione 50 mL de mistura de nutrientes de filtro esterilizado (tabelas 2 e 3).
- Aclimatar a cultura a 18 ° C por 30 min antes de adicionar uma concentração final de 1 mM de IPTG.
- Incube durante uma noite a 18 ° C, com agitação a 160 rpm.
- Colha as pilhas como na seção 1.1.4-1.1.6.
2. imobilizado afinidade Metal cromatografia (IMAC) purificação de VimRod e E-PRD
-
Purificação de VimRod de His6-tag
- Resuspenda o pellet de células em 5 mL/g de PBS contendo um inibidor de protease cocktail EDTA carente. Homogeneizar com 12 traços em um homogeneizador de homogenizacao para melhorar o lysis da pilha na etapa seguinte.
- No gelo, proceda à sonicação a suspensão de células em um pulso de 1 s em/1 s fora, amplitude de 80% para um total de 1,5 min. repetir o sonication adicionais duas vezes, agitando suavemente no gelo entre execuções para evitar o superaquecimento.
- Centrifugar a amostra a 75.000 x g, durante 45 min. decantar e filtrar o sobrenadante através de um filtro de seringa (0,45 µm).
- Equilibrar uma coluna IMAC de 5 mL com 5 volumes de coluna (CV) do buffer de vinculação (20 mM HEPES, pH 7,5, 500 mM de NaCl, imidazol 10 mM) com um caudal de 1 mL/min, usando um sistema de cromatografia líquida (FPLC) proteína rápida.
- Carrega o sobrenadante filtrado para a coluna em uma taxa de fluxo de 0,5 mL/min.
- Lave a coluna com 5 CV de lavar buffer (20 mM HEPES, pH 7,5, 500 mM de NaCl, imidazol 50mm) a uma taxa de fluxo de 1 mL/min.
- Eluir a proteína com 3 CV de tampão de eluição (20 mM HEPES, pH 7,5, 500 mM de NaCl, imidazol 350 mM) com um caudal de frações de 0,5 mL/min. coletar 1,5 mL. Se disponível, selecione o modo de eluição do up-flow para aumentar a concentração de proteína eluted.
- Identifica as frações a partir do cromatograma FPLC que contêm a proteína de interesse por SDS-PAGE e usam os métodos padrão para medir a concentração de proteína5.
- Piscina e concentrar-se as frações de eluição contendo as maiores quantidades de proteína, utilizando um dispositivo de ultrafiltração centrífuga (MWCO 3 kDa, 5ml) de 2 mL. Centrifugar a 21.000 x g para remover qualquer precipitado e passar por um filtro de 0,22 μm.
- Equilibrar uma coluna de cromatografia (S) de exclusão de tamanho 120ml com buffer de 2 CV de S (20 mM HEPES, 150 mM NaCl, pH 7,5, 0.5 mM TCEP) a uma taxa de fluxo de 1 mL/min, usando um FPLC.
- Injetar a proteína concentrada de 2.1.9 na coluna e eluir com 1 buffer de CV de S a uma taxa de fluxo de 0,5 mL/min, coletando frações de 1 mL.
- Identifica as frações que contêm a proteína de interesse como antes.
- Piscina as frações que contêm a proteína de montantes mais elevada.
- Armazenar a 4 ° C, para uso de curto prazo ou adicionar glicerol 20% e loja a-80 ° C em pequenas alíquotas.
-
Purificação de proteínas E-PRD com a Tag His6 removida
- Siga as etapas no 2.1.1-2.1.8 para purificar a proteína His6-com a tag E-PRD. As frações de pico do pool e determinar a concentração de proteína.
- Adicionar tabaco etch protease do vírus (TEV) (1mg/mL) 2 µ l/mg de proteína em pool. Transferir para diálise tubulação (6 kDa) e urinar no buffer de S durante a noite a 4 ° C. Esta etapa permite a clivagem da marca His6 e remoção de imidazol que interferirão com ligação à resina Ni-NTA na próxima etapa.
- Equilibrar a 5 mL de resina Ni-NTA em uma coluna de gravidade com 3 CV de S. Escorra o tampão excesso de resina.
- Despeje a proteína E-PRD clivada em resina e incubar durante 1 hora em uma plataforma oscilante para permitir que o uncleaved His6-com a tag E-PRD e a tag His6 clivada ligar. A protease do PEV é também His6-tag e irá vincular a resina. Coletar o fluxo através de, que contém o marca-free E-PRD. Lave a resina com buffer de 2 CV de S para garantir que todos o E-PRD é recuperado.
- Concentre-se E-PRD no fluxo através de 2 mL, usando um dispositivo de ultrafiltração centrífuga (MWCO 3 kDa, 5ml). Centrifugar a x 21 000 g para remover qualquer precipitado e passar por um filtro de 0,22 μm.
- Equilibrar uma 120 mL S coluna com 2 CV de S (20 mM HEPES, 150 mM de NaCl, pH 7,5, 0.5 mM TCEP para MST ou 20 mM Tris-HCl, 1 milímetro DTT, pH 7 para NMR) em uma taxa de fluxo de 1 mL/min, usando um FPLC.
- Injetar a proteína concentrada de E-PRD de 2.2.5 na coluna e eluir com 1 buffer de CV de S a uma taxa de fluxo de 0,5 mL/min, coletando frações de 1 mL.
- Identifica as frações que contêm a proteína de interesse como antes.
- Piscina as frações que contêm a proteína de montantes mais elevada.
- Armazenar a 4 ° C, para uso de curto prazo ou adicionar glicerol 20% e loja a-80 ° C em pequenas alíquotas.
3. NMR métodos
- Preparação da amostra NMR
- Purify 15selvagem-tipo N-rotulados ou proteína de R1914E E-PRD como descrito anteriormente, usando 20 mM Tris-HCl, 1 milímetro DTT, pH 7, como o buffer de S para etapa 2.2.6. Soluções estoque de proteínas geralmente variam de 0,3 a 1 mM, com volumes de cerca de 1 mL.
Nota: Pode ser concentrada de proteína > 100 µM usando um MWCO 3 kDa, dispositivo de ultrafiltração centrífuga de 5 mL para trazer a concentração em um intervalo adequado para a preparação da amostra. - Purifica uma amostra da proteína de VimRod sem rótulo usando 20 mM Tris-HCl, 1 milímetro DTT, pH 7, como o buffer de S para etapa 2.1.10.
- Em um volume final de 500 µ l, adicionar o selvagem-tipo ou mutante da proteína E-PRD para uma concentração final de 100µM, óxido de deutério (D.2O) a uma concentração final de 10% (v/v) e DSS (ácido 4,4-dimethyl-4-silapentane-1-sulfonic) para uma concentração final de 20 µM. Bring o volume até 500 µ l de amostra usando 20 mM Tris-HCl, 1 milímetro DTT, pH 7. Uma preparação de amostra representativa é descrita na tabela 4.
Nota: O 0 ppm ressonância de DSS é usada para calibrar as mudanças químicas 1H, bem como de referência indirecta do produto químico 15N muda da proteína6. D2O é usado para o sinal de bloqueio de deutério para manter o espectrômetro operando em um campo magnético de líquido constante. - Compõem uma segunda amostra de E-PRD, D2O e DSS como na etapa anterior e adicionar VimRod para uma concentração final de 50 µM antes de trazer o volume até 500 µ l.
- Transferi as amostras de 500 µ l para um ampla os tubos NMR de 5 mm para o experimento.
- Purify 15selvagem-tipo N-rotulados ou proteína de R1914E E-PRD como descrito anteriormente, usando 20 mM Tris-HCl, 1 milímetro DTT, pH 7, como o buffer de S para etapa 2.2.6. Soluções estoque de proteínas geralmente variam de 0,3 a 1 mM, com volumes de cerca de 1 mL.
- Instalação Experimental NMR
- Ligar o fluxo de ar com o comando eject "ej"; Isso irá abrir a amostra do ímã. Agora, colocar a amostra dentro de um girador em cima do ímã pela abertura e com o comando "ij". Espere até a amostra se instala no interior do íman antes de prosseguir.
- Criar um novo dataset usando o comando "edc" e carregar parâmetros de padrão 1H NMR, selecionando o experimento "ZGPR" (Figura 1). Preencha os campos nome, EXPNO (número do experimento) e PROCNO (número da pasta de dados processados). Selecione o solvente no campo "Set solvente" e clique em "Executar 'getprosol'" para ler o padrão probehead e parâmetros dependentes de solvente (prosol).
- Trave a amostra para o deuterado solvente, ou seja, D2O, usando o comando "trancar" e espere até que esteja terminado varrendo e alcança o bloqueio.
- Corrigi a frequência de ressonância do íman ajustando a amostra usando o comando de ajuste automático "atma". Monitore a curva de oscilação até que a sintonização automática completa.
- Calço o campo magnético usando TOPSHIM (comando "topshim"). Shims é o processo de ajustes de campo magnético para uniformizar o Sample. É boa prática para armazenar os valores de correção com o comando "wsh" e lê-los usando "rsh" antes topshim, se usando as amostras idênticas ou similares.
- Ajuste o ganho do receptor com o comando "rga" para alcançar o máximo sinal à relação de ruído.
- Colocar sobre o deslocamento de ressonância de água (o1) o centro do espectro e coloque o pulso de próton de 90 graus (p1) na alta potência usando "calibo1p1".
- Recolher o espectro de próton usando o zero ir processo com "pqe", que inclui a multiplicação exponencial ("em"), o decaimento livre de indução (FID) incorporando linha ampliando, "pés" e "zg" comando transformação de fourier da FID e "pk" para aplicar a fase correção.
- Aplica o correção automática de fase "apk" e a correção automática da linha de base "absn" usando o polinômio sem opção de integração.
- Crie um novo dataset (como em 3.2.2) para o experimento de SOFAST HMBC selecionando-se "SFHMQC3GPPH" no experimento.
- Cópia otimizada P1 e O1 do espectro de próton e preencher P1 dependentes pulsos usando o comando "getprosol 1H p1 plw1", onde p1 é o valor de P1 otimizado e plw1 é o nível de potência para P1.
- Otimizar a constante CNST54 para definir o deslocamento para o deslocamento químico amida e CNST55 para definir a largura de banda, a fim de abranger as regiões espectrais de interesse que permite que o ganho do receptor a ser otimizado (Figura 2). Para selecionar esses parâmetros, extrair o primeiro FID (decaimento livre da indução) do espectro bidimensional e procure o sinal observado para defini-los. Além disso, varia o atraso de relaxamento (D1), número de varreduras (NS) e fictícios scans (DS) para obter a sensibilidade do sinal aceitável com o comando "gs", que permite vai e digitalizar para monitorar a qualidade dos dados em tempo real.
- Grave os espectros usando Zero ir "zg".
- Processamento de dados NMR
- Definir os parâmetros de processamento para o tamanho da F2 direto (1H) e indireta F1 (15N) dimensões do espectro, usando "SI F2 = 2048, F1 = 512" com opcional predição linear na dimensão indireta (Figura 3).
- Selecione "QSINE" como a função de janela e digite um turno de sino de seno (SSB) de 2 para processar o espectro bidimensional.
- Digite o comando "xfb" para processar os dados em ambos os sentidos com a transformação de função e transformada de fourier de janela.
- Use o comando "apk2d" para realizar a correção automática de fase em ambas as direções. Se o processo automático não seja alcançado um nível satisfatório de correção de fase, extrair FIDs com o comando "rser", calcular valores de fase de processamento 1D e aplicá-los aos dados 2D.
- Corrigi a linha de base com a função de correção automática de linha de base "abs2" para dados 2D. Isto aplica-se uma função polinomial entre os valores de ppm definido nos parâmetros de processamento e produzirá um espectro 2D para posterior análise.
- Se você planeja executar processamento serial para comparação de dados de interação com outra molécula, armazenar os parâmetros de processamento com o comando "wpar" e recordá-los com "rpar". Deste modo que todos os conjuntos de dados serão processados com os mesmos parâmetros e variações não vai ser introduzido devido a diferenças de processamento.
- Análise de dados NMR
- Digite o comando "pp" para iniciar o processo de colheita de pico.
- Defina ppm intervalo e mínima intensidade/número máximo de picos com base em picos esperados (Figura 4). Clique no Okey e verificar os resultados por inspeção visual. Se necessário, execute novamente o processo até que os resultados são satisfatórios com base na qualidade de espectros.
- Gere um peaklist com o comando "pp".
Nota: Este peaklist contém informações de intensidade de altura/pico de dados por padrão e pode ser exportado para espectros subsequentes e pode ser lido por outros programas. - Observe as alterações nas intensidades pico ou movimento em turnos químicos em espectros HSQC de proteína que indicam a interação com outra molécula. Se a molécula interagindo é grande, esperamos reduções em intensidades de pico, juntamente com o desaparecimento de alguns picos.
- Importe a lista de pico para o próximo conjunto de dados clicando na guia "picos" e selecionar "importar" com um clique direito na janela de picos.
- Visualizar os picos ao longo do espectro e se necessário transferi-los para novas posições. Clique em "redefinir intensidades" para "tabela completa" para gerar um peaklist para o espectro com intensidades (Figura 5). Esta lista de pico vai transitar as informações de posição da lista de pico armazenado.
- Exporte as listas de pico de diferentes conjuntos de dados para uma planilha ou outro programa de matemático para a análise, selecionando a função "Exportar".
- Calcule a variação em intensidades de pico com a função de "pico de intensidade no complexo espectro/pico de intensidade no espectro de proteína" para cada pico. Valores podem ser convertidos à taxa de variação percentual por multiplicação de 100. Note-se que volumes de pico também são úteis, embora intensidades de pico são mais fáceis de medir para picos que estão posicionados próximos uns dos outros, como é geralmente o caso de proteínas com uma elevada densidade de picos relativamente amplas.
4. microescala Thermophoresis (MST)
-
Preparação do ligante E proteína-PRD
- Trocam o ligante em um buffer de MST compatível dializando até 800 μL de proteína em uma unidade de diálise mini 3.5 kDa suspensa em 1 L de 20 mM HEPES, pH 7,5, 10 mM de NaCl, agitando lentamente a 4 ° C durante a noite.
- Concentre o ligante usando uma unidade de ultrafiltração centrífuga (3 kDa MWCO) por centrifugação a 14 000 x g durante 10 min. a proteína concentrada de transferência para um tubo limpo.
- Centrifugue o ligante a 21.000 x g durante 10 minutos e transferir cuidadosamente o sobrenadante para um tubo novo para remover qualquer proteína precipitada. Determinar a concentração do ligante usando a absorbância em 280 nm e o coeficiente de extinção do ligante. Adicionar 10% Tween-20 para dar uma concentração final de 0,015%. Tween-20 é adicionado para o buffer de ensaio para evitar a adsorção aos capilares. O buffer de ensaio final que é usado para os experimentos de MST é 20 mM HEPES, pH 7,5, 10 mM de NaCl, de 0,015% Tween-20.
-
Preparação de VimRod de proteína alvo tingir-etiquetados
- Reconstitua o corante vermelho-tris-NTA adicionando 50 μL de 1X PBS-T fornecido com o vermelho-tris-NTA para dar uma concentração de 5 μM. Diluir 2 alíquotas μL em 200 μL de tubos e armazenar a-20 ° C.
- Dilua a proteína do alvo para 0,34 μM com buffer de ensaio. Adicionar 58 μL de alvo para uma alíquota de 2 μL da tintura vermelha-tris-NTA e incube por 30 min à temperatura ambiente. A concentração final de tingir-etiquetados alvo é 0,33 μM. Centrifugue o destino etiquetado em 21.000 x g durante 10 minutos e transferir cuidadosamente o sobrenadante para um tubo novo para remover qualquer precipitado. O corante vermelho-tris-NTA vincula-se a proteínas através da marca His6 e tem uma constante de dissociação de ligação (KD) na faixa nanomolar-sub. Efetivamente é 100% ligado à proteína alvo sem depuração adicional é necessária.
- Ligue o instrumento do MST e abra o Software de controle. Selecione a configuração de vermelho para o corante vermelho-tris-NTA. Ative o controle de temperatura de 25 ° C.
- Selecione o pré-teste para validar a rotulagem do alvo e verificar a agregação ou adsorção para as cubetas. Uma avaliação desses parâmetros é fornecida automaticamente.
- Mix 17 μL de tampão de ensaio e 3 μL da proteina alvo e mix por pipetagem. Preencha dois capilares padrão, mergulhando-os no destino diluído e elaboração de líquido para o centro do capilar. Coloque os capilares na bandeja e em instrumentos. Inicie a medição.
- Análise dos resultados à procura de um nível suficiente de fluorescência e sem sinais de adsorção (distorção na forma linha capilar) ou agregação (distorções no rastreamento de MST), que será sinalizada no software de análise. Se os resultados forem positivo mover para o ligante passos, se não um buffer diferente deve ser julgado ou a quantidade de Tween-20 pode ser aumentada a 0,05% ou superior.
-
Preparação da série E-PRD ligante dupla diluição
- Preparar uma série de diluição através da marcação de 16, 200 tubos μL de 1-16.
- Adicione 17 μL do ligante em 1,17 x concentração maior do que a concentração de ligante máximo desejada para Tube1. Este volume é duas vezes a quantidade necessária (8,5 μL) como uma alíquota então vai ser transferida para o próximo tubo em série. O volume final do ensaio é 10 μL então 8,5 μL de 1,17 concentração x de ligante será diluída a 1 x por adição de 1,5 μL da proteína alvo, VimRod. Esta concentração máxima escolhida deve ser pelo menos 20 vezes o valor de KD estimado.
- Adicione o 8.5 μL de tampão, ensaio de Tubes2-16 usando uma ponta de pipeta nova para cada alíquota. Reutilização de pontas de pipetas pode afetar a precisão (para aconselhamento sobre precisos pipetagem ver indicações do fabricante). Transferi 8,5 μL de ligante de Tube1 com Tube2 lentamente liberando a solução para o buffer de ensaio sem gerar bolhas. Misture o buffer e o ligante pipetando acima e para baixo pelo menos 6 vezes, novamente sem gerar bolhas. O E-PRD no tubo mais concentrado foi 1,5 mM e o rotulado VimRod estava presente em uma concentração final de 50 nM.
- Transferência de 8,5 μL de ligante de Tube2 para Tube3 e misture com o buffer de ensaio. Repita a diluição serial até que todos os tubos tiveram ligante adicionado. Descarte o 8.5 μL de Tube16 para que todos os tubos contêm 8,5 μL de ligante em uma série de diluições de duas vezes.
-
Preparação do experimento reação de ligação e o MST
- Cada um dos tubos adicionar 1,5 μL da proteína alvo etiquetados e misture suavemente pipetando acima e para baixo com cuidado para evitar bolhas. Incube durante 15 min.
- Selecione o Expert Mode para o ensaio de vinculação e entrar nos parâmetros para uma série de diluição serial. Certifique-se de controle de temperatura é definida a 25 ° C, o poder da excitação é definido para 40% e o poder do MST para médio. Esses parâmetros devem ser otimizados para outros parceiros de ligação a ser estudados.
- Encha os capilares com as reações de ligação e coloque na bandeja do capilar. Carregar a bandeja para o instrumento, espere até que a temperatura recuperar a 25 ° C e iniciar a medição.
-
Análise de dados
- Abra o arquivo de ensaio de ligação do software de análise de afinidade. Rever os exames capilares; Níveis de fluorescência não devem variar mais de 10% da média, nenhuma agregação ou adsorção deve ser detectada conforme descrito na seção 3.6.
- Selecione o MST a análise no painel direito e arraste os dados de ensaio para o conjunto de análise. Se houver várias execuções do ensaio de ligação do ligante-alvo mesmo sob condições idênticas podem ser mescladas, largando no mesmo conjunto. Alternativamente, cada execução pode ser descartada em análise independente.
- Mover para a guia de Dose resposta ajuste para os dados do gráfico. Escolha o modelo KD se espera-se um sítio de ligação único. O modelo de Hill é também uma opção para vários sites de ligação com o comportamento cooperativo. Pontos aberrante que não passe do controle de qualidade podem ser retirados o ajuste nesta fase. Vai ser uma média de Mesclar moda com vários ensaios e erros calculado como o desvio-padrão.
- Abra a guia final e comparar resultados entre todos os lotes em um único gráfico. Recupere resultados de ajuste para cada curva da tabela que é gerada. Exporte os dados ou curvas cabidas para outro software de apresentação ou análise se desejado.
Resultados
O domínio E-PRD (resíduos 1822-2014 clonados em pProEX-HTC) do gene da envoplakin humana e o domínio de VimRod (resíduos clonado em pET21a 99-249) de vimentina humana4 foram expressas com tags His6 e purificado. Figura 6 e Figura 7 demonstram os níveis de pureza de VimRod (18,8 kDa) e E-PRD (21,8 kDa) é obtido por este método de purificação de proteínas. A remoção do His6 marca de construção de E-PRD é essencial para as experiências do MST, como a proteína VimRod está identificada usando um corante de vinculação de marca His6 e qualquer PRD-E mantendo sua marca de His6 pode competir para ligação do corante. A segunda coluna IMAC após a clivagem da tag com protease TEV remove a protease do PEV, a tag clivada e qualquer uncleaved His6-E-PRD que permaneceu. A etapa de polimento final da purificação é cromatografia de exclusão. Apesar de ambas as proteínas ser de um tamanho similar, o VimRod elutes da coluna em um 51 mL, enquanto que o pico de eluição E-PRD é centrado em 72 mL onde se esperaria um monômero de proteína deste tamanho. O aparente aumento no tamanho do VimRod é provavelmente devido às suas características como uma vara longa filamentosa em forma de proteína como analiticamente experimentos se demonstraram que o VimRod era monomérico4. Rendimentos mais baixos de proteína são obtidos a partir das culturas cultivadas em M9 do que aqueles de caldo de carne rico devido a uma menor quantidade de células, sendo produzido na mídia mínima. O crescimento inicial das maiores fermentos para preparações M9 em TB permite a melhoria dos rendimentos de célula, mantendo a extensão de 15N rotulagem necessário para os experimentos de NMR.
O 15N -1H HSQCs foram adquiridas para o tipo selvagem e mutante R1914E de E-PRD em presença ou ausência de VimRod (Figura 8A-8D). O espectro de E-PRD na Figura 8A mostra o número esperado de picos bem resolvidos, indicativos de uma proteína devidamente dobrada. Na presença de VimRod (Figura 8B) o espectro mostra extensa linha ampliando e desaparecimento de pico, correspondente a vinculação entre o E-PRD e VimRod. Essa ligação é perdida por mutação de R1914E, como evidenciado pela comparação da Figura 8 e 8 D. Pequena mudança é observada no espectro mediante adição de VimRod para o mutante R1914E, indicando uma falta de ligação entre este E-PRD mutante e VimRod. As intensidades de pico E-PRD na presença/ausência de VimRod foram comparadas e plotadas como as intensidades de pico relativo na Figura 8E, que indica o intervalo de pico ampliação do complexo E-PRD. O mutante R1914E de E-PRD (não mostrado) retidos cerca de 97% dos picos em intensidades mais elevadas de pico ou de 20% na presença de VimRod em comparação com cerca de 20% para o tipo selvagem (Figura 8E). Este representa uma perda de mutante de ponto de função, com adicionais mutantes tendo efeitos intermediários também tendo sido estudado4.
Para validar e dosar a ligação da análise VimRod e E-PRD MST usando rotulado com corante vermelho-tris-NTA fluorescente como o destino de His6-VimRod misturado com diminuindo a concentração do ligante E-PRD de 1,28 mM para 39,1 nM foram realizados. Realizaram-se três titulações de vinculação e os resultados são em média e mostrados na Figura 9. Os dados foram se encaixam com um modelo padrão de vinculação de um sítio ligante e deram um KD 25,7 ± 2,1 μm. Avaliação da ligação entre VimRod e E-PRD por ressonância de plasmon de superfície deu um valor de KD semelhante de 19,1 ± 1,3 μM4.

Figura 1: tela de captura da instalação do experimento NMR. A janela mostrada é usada para configurar uma experiência padrão para coletar um conjunto de dados HSQC. Parâmetros do experimento são lidos no adjacente ao experimento. O experimento ZGPR mostrado é escolhido como um experimento inicial para carregar os parâmetros padrão e solvente próton dependente. A janela de título é usada para introduzir detalhes experimentais, para fins de manutenção de registros. Para coletar o espectro HSQC o experimento ZGPR é substituído por SFHMQC3GPPH. Clique aqui para ver uma versão maior desta figura.
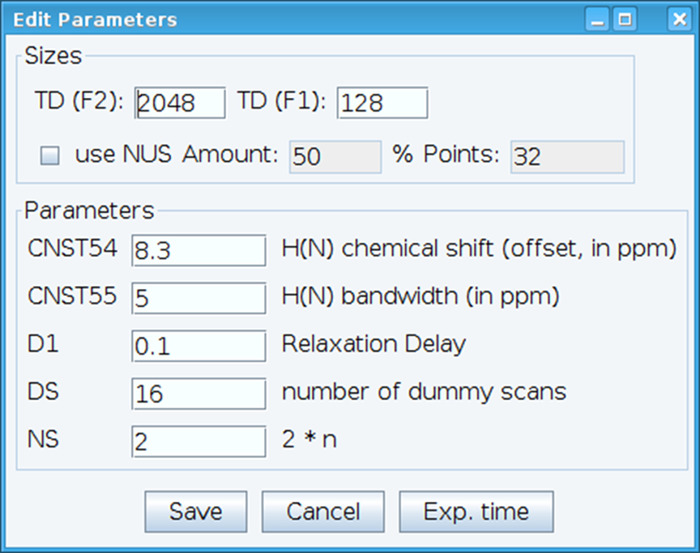
Figura 2: ajuste de parâmetros experimentais NMR. A janela mostrada é utilizada para inserir os parâmetros fundamentais para a sequência de pulso NMR para otimizar o sinal. Clique aqui para ver uma versão maior desta figura.

Figura 3: processamento de dados NMR. Parâmetros utilizados para o processamento de cada uma das duas dimensões do espectro NMR são mostrados, com setas indicando os que normalmente são ajustados. Clique aqui para ver uma versão maior desta figura.
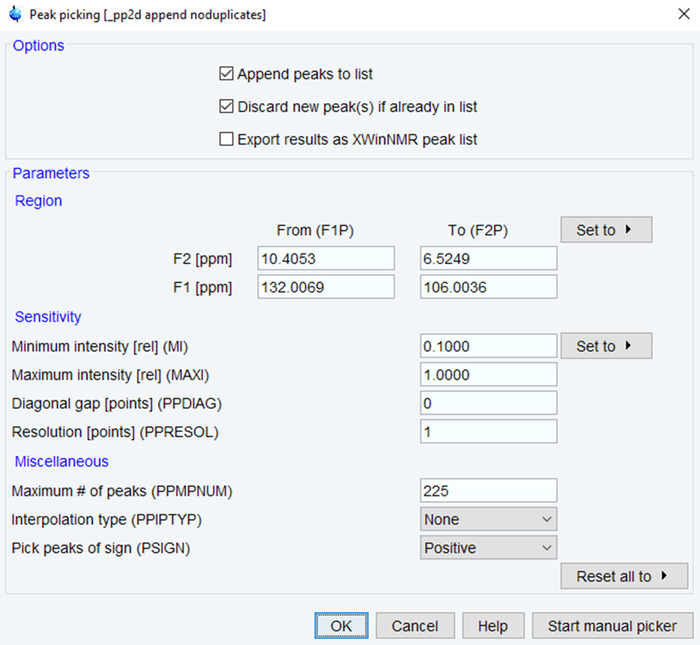
Figura 4: parâmetros para a colheita de pico NMR. Os parâmetros usados para escolher NMR picos no espectro NMR transformado são mostrados com valores típicos. Ajuste o intervalo de ppm, a intensidade e o número de picos para otimizar os espectros. Clique aqui para ver uma versão maior desta figura.
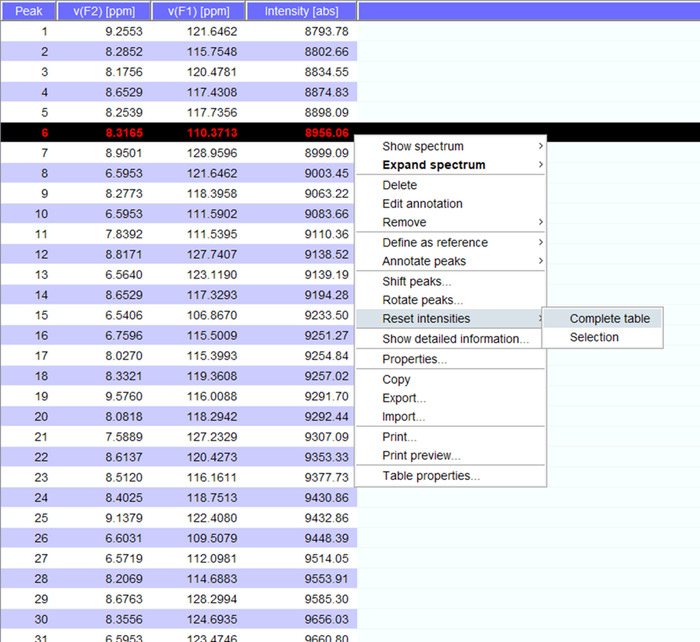
Figura 5: Peaklist representante com intensidades. Cada pico que é colhido no espectro NMR é dado um número e a 1H e 15N químico desloca e intensidade de sinal são exibidos. Este peaklist, então, pode ser usado para comparar os espectros obtidos na presença/ausência de um parceiro de interação. Clique aqui para ver uma versão maior desta figura.
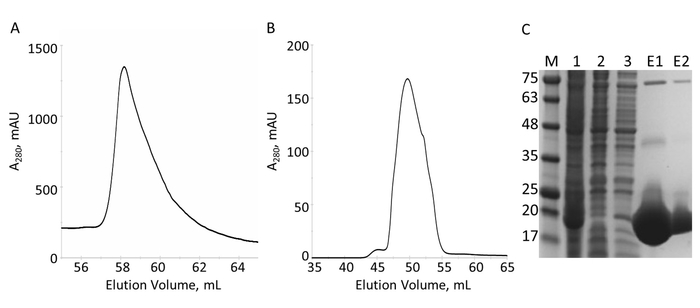
Figura 6: purificação de VimRod His6-marcados pelo IMAC e S. A. O cromatograma para a eluição da coluna IMAC mostra um grande pico de VimRod. B. o cromatograma para a eluição da coluna S mostra um pico maior. C. SDS-PAGE de fracções recolhidas ao longo de purificação: padrões de MW com o MW indicaram em kDa à esquerda do gel (M), lisado celular (1), IMAC de passagem (2), lavagem (3), em pool de eluição (E1), pool S eluição (E2). Bandas visíveis no maior peso molecular em vias E1 e E2 são oligômeros de pura VimRod confirmada pelo borrão ocidental (dados não mostrados). Clique aqui para ver uma versão maior desta figura.
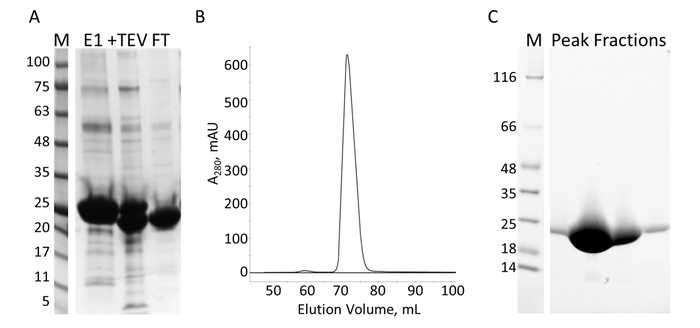
Figura 7: purificação de E-PRD de IMAC e S. A. SDS-PAGE da purificação IMAC mostrando os padrões de peso molecular com o MW indicado em kDa à esquerda do gel (M) e o eluído primeiro IMAC na coluna (E1), o PEV clivagem produtos (+ TEV) e o fluxo através da segunda coluna IMAC (FT). B. o cromatógrafo da coluna S mostra um pico maior. C. SDS-PAGE dos padrões de peso molecular (M) e as frações do pico de S. Clique aqui para ver uma versão maior desta figura.
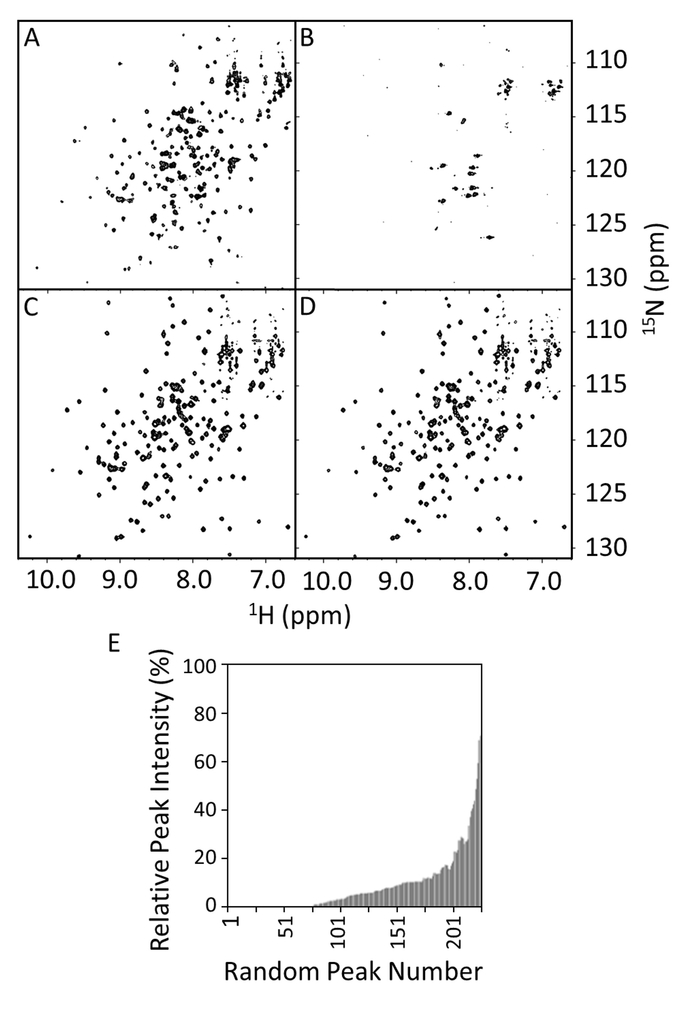
Figura 8: HSQC espectros do tipo selvagem e mutante R1914E de E-PRD na presença e ausência de VimRod. Os espectros HSQC mostram o selvagem-tipo E-PRD (100 µM) em 20 mM Tris-HCl, 150 mM de NaCl, 1 milímetro DTT, pH 7, na ausência (A) ou presença de 50 µM VimRod (B). Os painéis C e D são os espectros HSQC do mutante R1914E (100µM), na ausência ou presença de 50 µM VimRod, respectivamente. No painel E o pico de15N relativo 1H - intensidades do E-PRD com ou sem ligação de VimRod são mostradas como uma função do número de pico, o qual é atribuído arbitrariamente e não com base na posição de sequência. Esses valores podem ser usados para definir um limite de significância para a redução de intensidade de pico após a adição de um ligante. Se atribuições estão disponíveis, os valores significativos, muitas vezes podem ser vistos para mapear para uma área de vinculação. Clique aqui para ver uma versão maior desta figura.
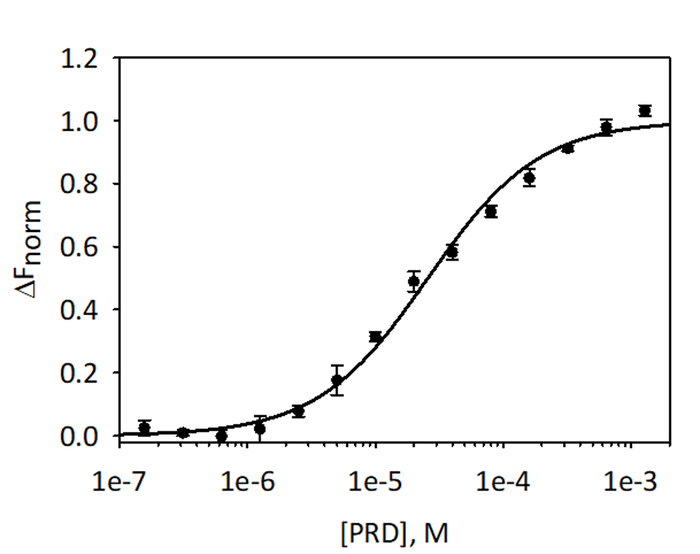
Figura 9: vinculação de E-PRD para VimRod. E-PRD foi diluído em uma série de diluições duplas de 1,28 mM para 39,1 nM e incubadas com rotulado VimRod antes de executar a análise de MST. Dados de três ensaios independentes foram combinados. Os dados eram próprios para um modelo KD dando um KD 25,7 μm com uma confiança de KD ± 2,1 μm. Clique aqui para ver uma versão maior desta figura.
| Reagente | Quantidade |
| Fosfato de sódio dibásico (anidro) | 6,0 g |
| Fosfato de potássio monobásico (forma anidra) | 3,0 g |
| Cloreto de sódio | 0,5 g |
| H2O | Até 950 mL |
Tabela 1. M9 mídia para etiquetar Isotopic.
| Reagente | Quantidade |
| 15 NH4Cl | 1,0 g |
| Glicose (ou 13C-glicose) | 2,0 g |
| 1 M MgSO4 | 2 mL |
| 50 mM CaCl2 | 4 mL |
| 20 mg/mL de tiamina | 1,0 mL |
| 3 mM FeCl3 | 400 Μ l |
| Metal Mix (tabela 3) | 500 Μ l |
| H2O | Até 50 mL |
Tabela 2. Mistura de nutrientes para suplementação de M9 media.
| Reagente | Quantidade |
| 4 mM ZnSO4 | 323 mg |
| 1 mM MnSO4 | 75,5 mg |
| 4,7 mM H3BO3 | 145 mg |
| 0,7 mM CuSO4 | 55,9 mg |
| H2O | Até 500 mL |
Tabela 3. Metal Mix suplemento para enriquecer a mistura de nutrientes de MT.
| Amostra | 1 mM E-PRD em tampão A1 (µ l) | 1mM VimRod no buffer A (µ l) | Reserva um (µ l) | 200 µM DSS na D2O (µ l) | Tampão B2 (µ l) | Volume total (µ l) |
| E-PRD sozinho | 50 | 0 | 50 | 50 | 350 | 500 |
| PRD-E + VimRod | 50 | 50 | 0 | 50 | 350 | 500 |
| 1 Buffer de r: 20 mM Tris-HCl, 1 milímetro DTT, pH 7 | ||||||
| 2 Tampão b: 23 mM Tris-HCl, 1,14 mM DTT, pH 7 | ||||||
Tabela 4. Preparação da amostra NMR.
Discussão
O 2D 15experiência NMR N-resolvido é um dos mais amplamente utilizados métodos para mostrar como duas moléculas interagem. É o método mais ricos em informação que permite que os sinais de ambos os parceiros a serem monitorados continuamente ao longo de uma experiência de titulação no estado de solução. Embora normalmente qualitativa no caso de grandes complexos, o método também pode ser usado em casos favoráveis para medir afinidades de ligação onde sinais NMR podem ser rastreados em espectros de alta resolução. Onde as atribuições podem ser feitas convenientemente, como no caso de muitas proteínas sob 20 kDa em tamanho, os sítios de ligação também podem ser mapeados. Ensaios complementares tais como MST fornecem informações quantitativas sobre interações em solução e requerem menos proteína nos Estados sem rótulo. Comparação de mutante de ligação de dados é útil para fornecer controles para garantir que interações evidenciadas pela linha NMR alargamento são genuínas e não artefatos de, por exemplo, alterações de agregação ou viscosidade.
Expressão da proteína
Agilizando o processo de expressão reduz a quantidade de produção de proteína intensivo de mão de obra. Parte deste processo de otimização envolve a identificação de uma tensão adequada de e. coli para a expressão recombinante da proteína. Preferência de tensão depende de elementos, incluindo a natureza do vetor em uso e, mais especificamente, a estabilidade final da proteína recombinante está sendo expresso7. O risco de degradação da proteína heteróloga por endógena Escherichia coli proteases podem ser reduzidos pelo uso de protease deficiente Escherichia coli como a estirpe BL21. Para genes contendo códons raros, uma estirpe como RIPL BL21-CodonPlus (DE3) pode ser preferida. Esta estirpe combina a natureza de protease deficiente da estirpe BL21 com cópias adicionais de endógenas de tRNAs códon rara de leucina, isoleucina, prolina e arginina. Alternativamente, códons raros que podem comprometer a superexpressão podem ser evitadas por encomendar uma construção códon otimizado de uma fonte comercial. Muitas cepas de e. coli estão disponíveis para a expressão do gene recombinante, cada um otimizado para evasão de um problema específico durante a expressão7. No caso deste estudo, a estirpe de padrão protease deficiente BL21(DE3) produzido quantidades adequadas de proteína solúvel para purificação subsequente e análise.
Purificação de proteínas
O protocolo de purificação para uma proteína dada frequentemente é único no sentido de que cada proteína permanece estável e solúvel em diferentes condições como temperatura, concentração de sal ou pH. A eficácia global da purificação por cromatografia de afinidade também é sensível à concentração de eluição espécies como imidazol em várias etapas durante o processo de purificação. Neste trabalho, condições de reserva crítica para IMAC foram pH para o E-PRD e concentração de imidazol para o VimRod. Um pH de 7,5 foi necessário para evitar a precipitação do PRD-E após eluição inicial da coluna IMAC. Para a purificação do IMAC de VimRod, aumentando a concentração de imidazole de 30 a 50 mM durante a etapa de lavagem da coluna foi encontrado para ter uma melhoria substancial na pureza das frações eluição final. Para a etapa de eluição, também aumentando a concentração de imidazole de 250 a 350 mM foi encontrado para melhorar o rendimento da eluição final. Tentativas iniciais para eluir proteína usando imidazol 250mm levaram a eluição incompleta de VimRod como revelado por uma tira de imidazol 1m final da coluna (dados não mostrados). Aumentando a concentração de imidazol para 350 mM para a eluição foi suficiente para recuperar toda a proteína associada à coluna. S pode servir a um propósito duplo porque actua como uma etapa de polimento para purificação de proteínas, enquanto simultaneamente, realizando troca de amortecedor. Troca de amortecedor é um passo crítico para análise posterior ligação desde que ele remove o imidazol usada para eluir a proteína His6-tag. Serve também como uma oportunidade para alterar condições tais como a concentração de sal ou pH, que pode afetar a eficácia de certas técnicas de jusante ou ensaios. Proteína térmico turno (PTS) pode ser usado para identificar buffers ideais para ensaios a jusante, especialmente para aqueles que exigem uma proteína estável para prolongados períodos de tempo em temperatura ambiente8,9.
Análise de ligação
Proteína que é preparada é fundamental para os ensaios de ligação exata, embora congelada proteína também pode ser usada desde que os resultados são comparados. Proteínas filamentosas como vimentina multimerize forma um sal e pH dependentes, e, portanto, a solução condição precisa ser otimizado e oligoméricos estado estimado por um método como SEC10,11, dinâmica de espalhamento de luz 12 ou ultracentrifugação analítica13,14,15. Espectroscopia RMN é adequada para medir as interações ligante de pequenas proteínas em resolução atômica. No entanto, quando uma proteína interage com a molécula maior, segue-se o tombo mais lento, e isso resulta em perda de sinais, que pode confirmar a vinculação, embora isso não necessariamente permitem mapeamento de binding sites, que também exigiria a atribuição pelo menos espinha dorsal ressonâncias. Neste cenário, experiências NMR não permitir a identificação do site da interação. Daí o local dirigido mutagênese é aplicado para identificar os resíduos críticos necessários para a ligação. Tais mutantes, portanto, não apresentam perda de sinal. Neste protocolo, uma forma mutante com uma substituição na posição 1914 retém as intensidades de pico na presença de VimRod e, por conseguinte, confirma o rompimento da interação do E-PRD e VimRod. Atribuição de espinha dorsal e sidechain ressonâncias gostaria de acrescentar valor a esta abordagem, particularmente como a estrutura para o livre E-PRD foi resolvida por cristalografia de raios x4. Futuras aplicações de NMR incluem caracterização de interações complexas entre moléculas maiores e beneficiarão de ultra altos ímãs de campo e o uso de outros grupos observáveis como 13C-etiquetadas e trifluoro metil grupos como repórteres.
MST tem uma série de vantagens para o estudo de interações de ligação16. Os parceiros de ligação são livres em solução e não imobilizado. Análise da qualidade das amostras é incorporado o software com controle de qualidade, relatórios de agregação, adsorção aos capilares ou rotulagem fluorescente insuficiente da molécula alvo. Pequenas quantidades do alvo são normalmente utilizadas, a concentração do alvo etiquetado é geralmente entre 20-50 nM em 10-20 μL volume/reação. Este protocolo utiliza volumes de reação muito pequeno (10 μL) para maximizar a concentração de ligante que pode ser alcançado nas titulações permitindo interações fraca ligação ser caracterizada. Isto necessita de pipetagem precisos e cuidado para evitar a introdução de bolhas enquanto ainda completamente a mistura. Mistura adequada é fundamental para a medição de fluorescência exata, consistente ao longo do conjunto de diluições em série. A quantidade de Tween-20 nas experiências de MST foi reduzida de um padrão de 0,05% a 0,015% para reduzir a tendência para criar bolhas e melhorar a mistura.
O corante vermelho-Tris-NTA fornece uma maneira rápida, fácil e conveniente de fluorescente rotular qualquer proteína que tem a sua marca. A rotulagem é efetivamente completo em apenas 30 minutos e é muito apertado para que nenhum procedimento de remoção do corante é necessário. Sem modificações são feitas para resíduos de aminoácidos na proteína que podem alterar as propriedades de ligação do ligante. Uma ressalva é que apenas a proteína a ser rotulado deve ter uma marca de His6. Isto exigiu a clivagem da marca desde a proteína ligante, E-PRD e a remoção da marca e E-PRD uncleaved com uma segunda etapa de coluna do IMAC. Se possível, a proteína ligante deve ser preparada sem o uso de uma sua marca. Alternativamente, as proteínas podem ser rotuladas covalentemente com um fluoróforo através de amina acoplamento de resíduos de lisina ou tiol acoplamento a resíduos de cisteína. No entanto, deve ter cuidado quando se utilizam esses sistemas desde a ligação covalente de um fluoróforo pode afetar as interações de ligação eletrostática ou polar confiando em resíduos de lisina ou a cisteína. A quantificação de vinculação afinidade entre o VimRod e E-PRD pelo MST foi extraordinariamente sensível à concentração de sal. Este problema foi atenuado pelo dializando inicialmente o alvo e ligante no mesmo lote do buffer de ensaio. Não obstante, saturação da curva de ligação do MST não poderia ser alcançada, ao realizar o ensaio de MST na presença de 150 mM de NaCl devido ao comportamento de complexo de VimRod. Confiáveis e completos de dados obteve-se uma vez que a concentração de NaCl foi reduzida a 10 mM, permitindo o cálculo exacto do KD. Portanto, cuidadosa otimização das condições de solução e comparação com ensaios complementares são recomendados para alcançar resultados robustos. Além disso, o MST pode ser utilizado para quantificar a dependência de sal para uma determinada interação, quantificar Propriedades estequiométricas de interações da proteína, monitor do enrolamento de proteínas e sonda em cinética de enzima17.
Divulgações
Os autores divulgar sem conflitos de interesse.
Agradecimentos
Este projecto foi apoiado pelo RGPIN NSERC-2018-04994, programa de inovação Campus Alberta (RCP-12-002 C) e Alberta prião Research Institute / Alberta inova Bio soluções (201600018), atribuído a Otávio e genoma Canadá e Fundação do Canadá para Subsídios de inovação atribuídos para o centro de inovação de Metabolomics (TMIC) e NANUC.
Materiais
| Name | Company | Catalog Number | Comments |
| Monolith NT.115, includes control and analysis software | NanoTemper Technologies | MO-G008 | Instrument for microscal thermophoresis |
| His-Tag Labeling Kit RED-Tris-NTA | NanoTemper Technologies | MO-L008 | RED-tris-NTA dye for MST |
| Standard capillaries | NanoTemper Technologies | MO-K022 | capillaries for MST |
| HisTrap HP, 5 mL | GE Healthcare | 17524801 | IMAC column |
| HisPur Ni-NTA resin, 100 mL | Thermo Fisher Scientific | 88222 | IMAC resin |
| HiLoad 16/600 Superdex 75 pg | GE Healthcare | 28989333 | SEC column |
| TEV protease | Sigma Aldrich | T4455 | cleavage of his tag from E-PRD |
| BL21(DE3) Competent E. coli | New England Biolabs | C2527H | cells for protein expression |
| cOmplete Protease Inhibitor Cocktail Tablets EDTA-Free | Sigma Aldrich | 11873580001 | protease inhibitors for protein purification by IMAC |
| TCEP, Tris(2-carboxyethyl)phosphine hydrochloride | Sigma Aldrich | C4706 | reducing agent for protein purification |
| Reagents (HEPES, NaCl, etc) | Sigma Aldrich | various | Preparation of media and buffers |
| Ammonium chloride (15N, 99%) | Cambridge Isotope Laboratories | NLM-467 | isotope labelling for NMR |
| D2O, Deuterium Oxide (D, 99.8%) | Cambridge Isotope Laboratories | DLM-2259 | NMR sample preparation |
| DSS, Sodium 2,2-dimethyl-2-silapentane-5-sulfonate-D6 (D,98%) | Cambridge Isotope Laboratories | DLM-8206 | reference for NMR |
| Precision 5 mm NMR Tubes, 7” long | SJM/Deuterotubes | BOROECO-5-7 | NMR tubes |
| NMR spectrometer (14.1 Tesla) | Bruker | acquisition of NMR data | |
| TCI 5mm z-PFG cryogenic probe | Bruker | acquisition of NMR data | |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Bruker TopSpin 4.0.1 | Bruker | processing of NMR data | |
| MO.Control | NanoTemper Technologies | included with Monlith NT.115 | |
| MO.Affinity Analysis | NanoTemper Technologies | included with Monlith NT.115 |
Referências
- Vinogradova, O., Qin, J. NMR as a unique tool in assessment and complex determination of weak protein-protein interactions. Topics in Current Chemistry. 326, 35-45 (2012).
- McKercher, M. A., Wuttke, D. S. NMR Chemical Shift Mapping of SH2 Peptide Interactions. Methods in Molecular Biology. 1555, 269-290 (2017).
- Al-Jassar, C., Bikker, H., Overduin, M., Chidgey, M. Mechanistic Basis of Desmosome-Targeted Diseases. Journal of Molecular Biology. 425 (21), 4006-4022 (2013).
- Fogl, C., et al. Mechanism of intermediate filament recognition by plakin repeat domains revealed by envoplakin targeting of vimentin. Nature Communications. 7, (2016).
- Walker, J. M. . The Protein Protocols Handbook. , (2002).
- Wishart, D. S., et al. 1H, 13C and 15N chemical shift referencing in biomolecular NMR. J. Biomol. NMR. 6, 135-140 (1995).
- Casali, N. Escherichia coli Host Strains. E. coli Plasmid Vectors. , 27-48 (2003).
- Ericsson, U. B., Hallberg, B. M., DeTitta, G. T., Dekker, N., Nordlund, P. Thermofluor-based high-throughput stability optimization of proteins for structural studies. Analytical Biochemistry. 357 (2), 289-298 (2006).
- Kozak, S., et al. Optimization of protein samples for NMR using thermal shift assays. Journal of Biomolecular Nmr. 64, 281-289 (2016).
- Burgess, R. R. A brief practical review of size exclusion chromatography: Rules of thumb, limitations, and troubleshooting. Protein Expression and Purification. , (2018).
- Kunji, E. R. S., Harding, M., Butler, P. J. G., Akamine, P. Determination of the molecular mass and dimensions of membrane proteins by size exclusion chromatography. Methods. 46 (2), 62-72 (2008).
- Stetefeld, J., McKenna, S. A., Patel, T. R. Dynamic light scattering: a practical guide and applications in biomedical sciences. Biophysical Reviews. 8 (4), 409-427 (2016).
- Patel, T. R., Winzor, D. J., Scott, D. J. Analytical ultracentrifugation: A versatile tool for the characterisation of macromolecular complexes in solution. Methods. 95, 55-61 (2016).
- Pearson, J. Z., Cole, J. L., et al. Chapter One - Next-Generation AUC Adds a Spectral Dimension: Development of Multiwavelength Detectors for the Analytical Ultracentrifuge. Methods in Enzymology. 562, 1-26 (2015).
- Gorbet, G. E., Pearson, J. Z., Demeler, A. K., Cölfen, H., Demeler, B., Cole, J. L. Chapter Two - Next-Generation AUC: Analysis of Multiwavelength Analytical Ultracentrifugation Data. Methods in Enzymology. 562, 27-47 (2015).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nature Communications. 1, 100 (2010).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados