Method Article
Uso de francotirador Cas9 para minimizar efectos Off-target de CRISPR Cas9 sin la pérdida de actividad en blanco mediante evolución dirigida
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí, presentamos un protocolo para optimizar CRISPR Cas9 para lograr una mayor especificidad sin la pérdida de actividad en el blanco. Utilizamos un enfoque de evolución dirigida llamado Sniper-pantalla para encontrar a un mutante Cas9 con las características deseadas. Sniper-Cas9 es compatible con truncado solo guía RNAs y entrega en un formato de ribonucleoproteína, conocidas estrategias para lograr mayor especificidad.
Resumen
El desarrollo de clusters regularmente otro corto palindrómico repeticiones (CRISPR)-proteína asociada 9 (Cas9) en modalidades terapéuticas requiere la evitación de sus efectos potencialmente nocivos de objetivo. Varios métodos han sido diseñados para reducir tales efectos. Aquí, presentamos un Escherichia coli-evolución dirigida basada en método llamado Sniper-pantalla para obtener una variante Cas9 con especificidad optimizado y conservan actividad en blanco, denominada Cas9 Sniper. Usar pantalla de francotirador, selección positiva y negativa puede realizarse de forma simultánea. La pantalla también se puede repetir con otras secuencias de RNA (sgRNA) solo guía para enriquecer de los éxitos positivos verdaderos. Usando el promotor dual CMV-PltetO1 expresar Cas9 variantes, el funcionamiento de la biblioteca combinada puede comprobarse rápidamente en células de mamíferos. También se describen los métodos para aumentar la especificidad de Cas9 Sniper. En primer lugar, el uso de sgRNAs truncada se ha demostrado previamente para aumentar especificidad Cas9. A diferencia de otros Cas9s ingeniería, francotirador Cas9 mantiene un nivel de tipo salvaje (WT) de la actividad en el blanco cuando se combina con sgRNAs truncado. En segundo lugar, la entrega de Sniper Cas9 en un formato de ribonucleoproteína (RNP) en lugar de un formato de plásmido es posible sin afectar a su actividad en el blanco.
Introducción
En este trabajo, nuestro objetivo es mejorar la especificidad de Cas9 combinando diferentes estrategias. Se han desarrollado diversos métodos para evitar los efectos off-target de CRISPR Cas9. Por ejemplo, sgRNAs truncado puede utilizarse para lograr mayor especificidad1. Además, el método de entrega Cas9 se puede cambiar de un formato de plásmido a un formato de RNP para obtener mayor especificidad2. Residuos de aminoácidos específicos de la Cas9 de Streptococcus pyogenes (SpCas9) proteína han sido modificados según el racional diseño descrito anteriormente3,4,5. Alternativamente, residuos de aminoácidos han sido alterados de forma aleatoria y se identificaron las variantes Cas9 la especificidad más alta usando una levadura6 o un7,de e. coli8 sistema.
Sin embargo, muchos grupos han informado de que variantes Cas9 diseñado utilizando el diseño para debilitar la interacción inespecífica entre Cas9 y el sustrato exposición baja en meta actividades7,8,9, 10 , 11 , 12. hemos desarrollado un e. coli-basado sistema de evolución dirigida, pantalla de francotirador, a la pantalla al azar mutagenized Cas9 variantes. Una e. coli sistema tiene ventajas sobre un sistema de levadura debido a las eficiencias de tiempo y mayor transformación más rápida duplicación de e. coli.
Selección negativa y positiva, basada en tres plásmidos diferentes y un gen de interés (GOI) integra en el genoma de e. coli , se utilizan en la pantalla de francotirador. Cas9 variantes se expresan en el sistema de doble-promotor CMV-PltetO1 de un plásmido número bajo-copia para que candidatos identificados en e. coli pueden ser probados en células de mamífero sin necesidad de subcloning. El GOI es introducido en el genoma de e. coli utilizando el sistema de transposón Tn7. El plásmido sgRNA, que contiene un origen de replicación de sensibles a la temperatura, expresa un sgRNA contra el GOI; sin embargo, los sgRNA y secuencias GOI no se combinan. Un sitio de destino perfectamente emparejados sgRNA existe un tercer plásmido que contiene el ccdB gene, que codifica un producto letal que envenena a la girasa. En este sistema, las células que expresan variantes Cas9 con actividades de alto objetivo se eliminan debido a roturas del doble-filamento (distritales) se introducen en el sitio no coinciden en la DNA genomic. Por otro lado, también se quitan células expresan variantes Cas9 con bajas actividades de en el blanco debido a la expresión del gen letal ccdB . El nivel de expresión de las variantes de Cas9 puede cambiarse alterando la concentración de anhydrotetracycline (ATC), que ajusta la fuerza de la selección.
Razonamos que localizar el sitio de destino sgRNA no coinciden en la DNA genomic y no en un plásmido aumentaría la sensibilidad del sistema. La ventaja de este enfoque es que hay solamente un sitio genómico, considerando que habría muchos plasmids, que contienen un sitio de destino, dentro de una sola célula de e. coli .
Usando este sistema, identificamos una variante Cas9, Sniper-Cas9, que muestra actividades en blanco a nivel de peso y reducido objetivo actividades respecto a WT Cas9. Sniper-Cas9 puede lograr aún mayores ratios de especificidad con sgRNAs truncado o entrega basadas en RNP en lugar de suministro basado en el plásmido.
Protocolo
1. integración de un humano GOI en la cepa BW25141 de Escherichia coli
-
Clonación de GOI
- Reacción en cadena de polimerasa (PCR) amplifican una longitud bp 500 de un humano GOI que contiene varios sitios de destino de candidato mediante los métodos estándar de PCR con cebadores que contienen sitios de restricción enzima NotI y XhoI.
Nota: En este experimento, el GOI era el gen humano EMX1 . - Digerir el producto PCR y el pgrg3613 vector con enzimas de restricción NotI y XhoI.
- Gel purifican los fragmentos deseados (500 bp y 12 kb).
- Ligar los fragmentos con ligasa de T4. Para hacer esto, mezcle 50 ng de pgrg36 digerido y 6 ng de parte movible de la PCR en un volumen de reacción de 20 μL que contiene 1 x de buffer ligasa y 0,5 enzima ligasa de U. Incubar durante una noche a temperatura ambiente (RT).
- Transformar el plásmido ligado en células competentes DH5 α. Crecen los transformantes resultante en placas de agar Luria caldo (LB) que contiene ampicilina (100 μg/mL) a 32 ° C e incubar durante una noche. Tomar una colonia y crecer en medio LB con ampicilina durante la noche. Aislar el plásmido de ADN de la e. coli, usando un comercialmente disponible miniprep Kit. Confirmar la inserción del fragmento GOI por Sanger secuenciación el plásmido obtenido el mini-plásmido preparación (mini-prep)13.
- Reacción en cadena de polimerasa (PCR) amplifican una longitud bp 500 de un humano GOI que contiene varios sitios de destino de candidato mediante los métodos estándar de PCR con cebadores que contienen sitios de restricción enzima NotI y XhoI.
-
Preparación de la BW25141 -GOI
- Transformar el plásmido obtenido pgrg36 -GOI en la cepa BW25141 de Escherichia coli . Es esencial utilizar una cepa de BW25141 para reducir al mínimo el número de colonias positivos falsos.
- Crecen las células transformadas en el búfer LB a 32 ° C durante la noche. GOI se inserta en el ADN genómico de la cepa de BW25141 (BW25141 -GOI). Retire el plásmido pgrg36 -GOI de la cepa BW25141 -GOI usando el protocolo estándar pgrg36 del13. Brevemente, diluir una colonia (aproximadamente 107-fold) y crecer en una placa LB a 42 ° C durante la noche. Raya las colonias en la placa de LB y crecer a 42 ° C durante la noche.
- Confirmar la correcta inserción de GOI por Colonia PCR utilizando los cebadores sugeridos en el protocolo de pgrg36: 5'-GATGCTGGTGGCGAAGCTGT-3 'y 5'-GATGACGGTTTGTCACATGGA-3'. El primer amplifica el sitio de inserción de DNA genómico y el tamaño del resultante del producto de PCR será 904 bp más el tamaño del inserto (500 bp en este caso).
- Preparar espectro de células BW25141 -GOI (un protocolo detallado se describe en pasos 3.2.6–3.2.10).
2. preparación de la biblioteca variante Cas9
-
Preparación de la biblioteca
- Transformar el vector Cas9 del7 en un comercial de e. coli mutator cepa (Tabla de materiales) y siga las instrucciones del fabricante para obtener una variante biblioteca (la biblioteca de Mutator).
- Realizar PCR errores en toda la secuencia Cas9 WT en el Cas9 de vectores, utilizando el kit de PCR propenso (Tabla de materiales).
Nota: El protocolo de la tarifa de error baja fue adoptado en el caso del francotirador Cas9 para evitar distorsionar la función original de la proteína. - Digerir el Cas9 vector con enzimas de restricción apropiadas. Gel de purificar el producto PCR (en el paso 2.1.2) y la columna vertebral digerida.
Nota: El tamaño del gen SpCas9 es aproximadamente 4,3 kb. XhoI y KpnI fueron elegidos para digerir pBLC SpCas9 vector que se utilizó en el caso del francotirador Cas9. - Montar el fragmento de la columna vertebral (de paso 2.1.3) y el inserto amplificado mediante PCR errores (en el paso 2.1.3) vía recombinación isotermo en vitro .
Nota: más de 500 ng de la columna vertebral es necesaria para obtener una concentración alta de biblioteca (error-prone PCR [EP]). Dos kits PCR errores diferentes fueron utilizados para preparar bibliotecas de EP en el caso del francotirador Cas9 (biblioteca EP I y II). - Purificar los productos de la Asamblea (del paso 2.1.4) usando un kit de purificación de ADN que permite la elución de bajo volumen (Tabla de materiales). Eluir con 6 μL de agua libre de nucleasas (NFW) y medir la concentración de ADN.
- Transformar más de 500 ng de Cas9 vector de biblioteca (para cada una de las tres bibliotecas) en 50 μL de células de e. coli de espectro (Tabla de materiales). Véase protocolo de electroporación en pasos 3.2.1-3.2.4. Para esta preparación de biblioteca, use 1 mL de medio SOC en lugar de 250 μL por 50 μL de células competentes.
- Hacer 1: 100, 1:1, 000 y las diluciones de 1: 10,000 de la mezcla que contiene las células recuperadas con medio SOC. Placa las células diluidas en placas de 100 mm LB agar suplidas con cloranfenicol (12,5 μg/mL). Las células restantes en un plato de2 245 milímetros de la placa. Incubar a 37 ° C durante la noche.
-
Cálculo de la complejidad de la biblioteca
- Fotografiar las placas de dilución a través de un sistema de documentación de gel o una cámara digital normal. Ejecute OpenCFU software14 y subir las fotografías de las placas de la dilución. Defina el área de conteo dentro de una placa y eliminar falsas colonias.
- Manualmente se multiplican al número de colonias por el factor de dilución para obtener el número original de transformantes. Convertir a estos números en forma logarítmica (base 10). Calcular el promedio para determinar la complejidad de la biblioteca.
- Cuando se obtiene el valor de complejidad deseado, se reúnen todas las colonias en la placa cuadrada de 245 mm (desde el paso 2.1.7) usando un esparcidor y 20 mL de LB suplementado con cloranfenicol. Hacer crecer las colonias se reunieron y purificar la biblioteca plásmido usando un kit comercial midiprep.
Nota: Cuanto mayor sea la complejidad de la biblioteca, mejor. Cuando Cas9 francotirador fue identificado, una diversidad de 3 x 106 fue alcanzada para cada biblioteca.
3. positivos y negativos de evolución Cas9
-
Selección de objetivos y construcción de plásmidos
- Seleccione una secuencia Diana sgRNA espaciador en el GOI. Sustituir uno o dos residuos en el nucleótido al azar para producir una secuencia no coinciden.
Nota: Sitio de destino humano EMX1 3 (GAGTCCGAGCAGAAGAAGAA con PAM GGG) fue utilizado en el caso del francotirador Cas9. Siguientes son las secuencias utilizadas: GAGTCCGAGCAGAAagAGAA, GAacCCGAGCAGAAGAAGAA, GAGTCCGAGCAGAgGAAGAA y GAGcCCGAGCAGAAGAAGAA. - Introduzca la secuencia no coinciden (ver paso 3.1.1) en el plásmido sgRNA usando oligonucleótidos estándar (oligo) clonación procedimientos7.
- Inserte la secuencia no coinciden con un PAM en el extremo 3' p11-encaje-wtx1 (Tabla de materiales) para construir el plásmido ccdB usando estándar oligo clonación procedimientos15.
- Seleccione una secuencia Diana sgRNA espaciador en el GOI. Sustituir uno o dos residuos en el nucleótido al azar para producir una secuencia no coinciden.
-
Preparación de células competentes Sniper-detección e. coli
- Descongelar las células de espectro BW25141 -GOI en hielo.
- Añadir 1 ng de plásmido ccdB y el plásmido sgRNA con doble desajustes en 50 μL de células descongeladas de BW25141 -GOI . Suavemente mezcle las células mediante pipeteo y colóquelos en una cubeta de electroporación prechilled 0,1 cm.
- Transformar la e. coli con la dos plásmidos mediante electroporación. Añadir 250 μL de medio SOC inmediatamente después de la electroporación. Pipetee suavemente la solución para mezclar las células y el medio. Transferir la mezcla a un tubo de microcentrífuga de 1,5 mL.
Nota: Para la eficacia máxima, ajustar la tensión en 1.80 kV y el tiempo de ejecución deben estar entre 4,8 ms y ms de 5,0. - Recuperar las células transformadas e incubar a 32 ° C por 1 h con agitación suave.
- Placa de 125 μL de las células recuperadas en una ampicilina (50 μg/mL) (condición de la cultura) de la placa de agar LB de kanamicina (25 μg/mL). Placa el resto de las células en una ampicilina/kanamicina/arabinosa (1,5 mg/mL) placa de agar LB (ccdB-expresar condición). Incubar a 32 ° C durante la noche.
- Comprobar la ausencia de sobrevivir colonias en ccdB-expresar placa de condición. Se reúnen las colonias de la placa de la condición de cultivo utilizando un separador y la cultura en 250 mL de medio de caldo super óptima (sollozo) suplementado con 50 μg/mL de ampicilina y 25 μg/mL de kanamicina a 32 ° C, con suave agitación.
- Cuando la densidad óptica a 600 nm (OD600) alcanza 0.4, enfríe el matraz en el hielo. Preparar agua desionizada prechilled y una solución de glicerol al 10% prechilled (esterilizar antes de uso).
- Centrifugar (a 4.000 x g durante 5 min a 4 ° C) las células y eliminar el sobrenadante. Añadir 200 mL de agua desionizada prechilled. Resuspender las células con una pipeta serológica de 10 mL. Repetir este paso 3 veces.
- Lavar las células con 50 mL de solución de glicerol 10% prechilled. Les centrifugar como antes (a 4.000 x g durante 5 min a 4 ° C).
- Deseche el sobrenadante y resuspender el precipitado en 300 μL de solución de glicerol del 10%. Hacer 50 alícuotas μL y congelar en nitrógeno líquido. Almacenar las células (examen de francotirador) a-80 ° C.
-
Examen de francotirador
- Transformar las células de proyección de francotirador (de paso 3.2.10) con 100 ng de la plásmidos variante Cas9 de cada biblioteca (de paso 2.2.3.See pasos 2.1.1 y 2.1.4). Siga los pasos de la electroporación se describe en pasos 3.2.1–3.2.3.
- Transferir 250 μL de las células a un tubo de microcentrífuga de 1,5 mL fresco. Añadir 250 pg de ATC para una concentración final de 10 ng/mL. Recupera las células que contienen ATC así como libres de ATC (ver paso 3.2.4 para el paso de recuperación).
- Placa de 25 μL de células libres de ATC recuperadas en una placa de agar cloranfenicol/kanamicina LB (condición no selectivo). Añadir ATC a las células que contienen el ATC recuperadas para una concentración final de 100 ng/mL en una placa LB de 245 mm. Inmediatamente la placa las células en una placa de agar kanamicina/cloranfenicol/arabinosa LB (condición selectiva). Incubar durante una noche a 32 ° C.
Nota: El tamaño y el número de la placa LB se determinan por el tamaño de la diversidad de las cubiertas de la proyección. En el caso de una placa de Petri con 20 mL de LB a 100 mm, añadir 2 μg de ATC. - Las placas de la fotografía. Contar el número de colonias viables usando software de OpenCFU14. (Ver paso 2.2.1) Asegúrese de que el número de colonias en la placa no selectivo es por lo menos 10 veces más grande que la diversidad de la biblioteca para cubrir todas las variantes.
- Calcular la frecuencia de supervivencia como sigue.
Frecuencia de supervivencia = el número de colonias en una placa selectiva / (el número de colonias en una placa no selectivo x 10) - Las colonias que han sobrevivido en las placas selectivas de todas las tres bibliotecas de la piscina. Incubar las colonias sobrevivientes en 250 mL de LB suplementado con cloranfenicol 12,5 de μg/mL a 42 ° C durante la noche. Aislar la malla Cas9 biblioteca ADN utilizando un kit de midiprep.
Nota: Este paso borra el plásmido sgRNA. - Repita el proceso de selección de 3.3.1–3.3.6 de pasos hasta que la frecuencia de supervivencia alcanza una meseta. Uso 10 ng de plásmido Cas9 seleccionado para la transformación y 10 ng/mL de ATC durante la recuperación. Mantener la concentración de ATC a 100 ng/mL para la condición de selectiva.
-
Arrastrando los pies y la segunda proyección
- Shuffle las variantes combinadas seleccionadas utilizando el siguiente protocolo de barajado de ADN. PCR amplifica el Cas9 inserto en el plásmido Cas9 utilizando cebadores que flanquean, 150 nucleótidos desde los límites de la inserción. Resumen 2 μg de producto PCR amplificado con DNasa I en 1 min a 37 ° C.
- Purificar fragmentos 70 – 200 bp de longitud usando electroforesis en gel de agarosa 2%. PCR se amplifican los fragmentos purificados. Uso del producto como plantilla para PCR amplifican el inserto Cas9 con adecuados primers flanqueando Cas9. Utilizar el producto final de PCR para construir una biblioteca de Cas9 como se describe en el paso 2.1.4.
- Preparar nuevas células Sniper-proyección (véase sección 3.2) con otro plásmido sgRNA no coinciden (ver paso 3.1.1). Rehacer el proceso de evaluación (secciones 3.2, 3.3) hasta los alcances de la tasa de supervivencia a la meseta. Uso 10 ng de plásmido Cas9 seleccionado para la transformación y ATC de 10 ng/mL durante la recuperación. Mantener la concentración de ATC en 10 ng/mL para la condición de selectiva.
-
Selección de plásmidos mutantes de evolucionado Cas9
- Después el último paso de la proyección, al azar escoger cien colonias de la placa selectiva y cultura en un medio LB que contiene cloranfenicol a 42 ° C durante la noche.
- Aislar la plásmidos mediante un procedimiento de miniprep y secuencia de Sanger el insertos con primers de secuencia dentro de Cas9.
- Seleccione las superior tres variantes más frecuentes para probar en líneas celulares humanas.
4. entrega de Cas9 como una RNP con sgRNA truncada
-
Selección del gene de destino utilizando Cas-OFFinder
- Elegir sitios de destino con Cas-OFFinder (http://www.rgenome.net/cas-offinder/). Seleccione el tipo de PAM apropiado para el tipo específico de Cas9 y el genoma de destino (humano, ratón, pez cebra, etcetera.). Llenar en la ficha secuencias de consulta elija la discrepancia de númeroy haga clic en el botón Enviar .
- Después de unos segundos, el en blanco (con un número de desajuste de '0') y sitios de destino aparezcan. En general, elegir los sitios de fuera de objetivo con desajustes de uno a tres.
-
Preparación de la plantilla de sgRNA
- Orden oligos crRNA y tracrRNA con las siguientes secuencias de plantilla, es decir, crRNA secuencia: 5'-TAATACGACTCACTATAGGNNNNNNNNNNNNNNNNNNNNGTTTTAGAGCTAGAA-3'; tracrRNA secuencia: 5'-AGCACCGACTCGGTGCCACTTTTTCAAGTTGATAACGGACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC-3'. Completar la secuencia de crRNA con la secuencia de destino obtenida en el paso 4.1.2 N20. Para diseñar el sgRNAs truncado, eliminar bases desde el extremo 5' para obtener secuencias sgRNA N18 y N19 y N17.
- Preparar la mezcla PCR para la amplificación de la secuencia de codificación sgRNA como sigue: combine 10 μL de 5 x buffer, 0.5 μL de crRNA oligo (100 pmol/μL), 0.5 μL de tracrRNA oligo (100 pmol/μL), 2.5 μL de dNTPs (10 mM), 0,5 μL de ADN polimerasa y 36 μL de NFW (tabla de materiales).
- Ampliar la plantilla con las siguientes condiciones, a saber, para la desnaturalización inicial: 1 min a 98 ° C; para la desnaturalización, recocido y extensión: 10 s a 98 ° C, 15 s a 54 ° C, 20 s a 72 ° C, durante 25 ciclos; para la extensión final: 5 min a 72 ° C.
- Analizar 2 μL de la plantilla amplificada DNA (de paso 4.2.3) en gel de agarosa al 2%. Purificar la plantilla utilizando un kit de potabilización de la polimerización en cadena.
Nota: El tamaño de la plantilla de DNA es 125 bp.
-
Síntesis de sgRNA
- Preparar la mezcla de reacción para la síntesis de sgRNA como sigue: combinar 8,5 μL de ADN de plantilla (del paso 4.2.3), 1 μL de UTP (25 mM), 1 μL de CTP (25 mM), 1 μL de GTP (25 mM), 1 μL de ATP (25 mM), 4.2 μL de MgCl2 (100 mM) , 4.5 μL de la T7 ARN polimerasa (50 U/μL), 3 μL de tampón de polimerasa del RNA de x T7 10, 1,2 μL de pirofosfatasa (0,5 U/μL), 0,75 μL de inhibidor de la Rnasa (40 U/μL) y 4.2 μL de NFW.
- Incubar la mezcla de reacción a 37 ° C durante la noche (al menos para 10 h). Añadir 0,5 μL de DNasa (2 U/μL) a la mezcla de reacción e incubar a 37 ° C durante 15 – 30 min purificar lo sgRNA usando un kit de purificación de RNA.
- Para reducir la toxicidad causada por una respuesta inmune innata por 5'-trifosfato en el sgRNA16, quitar el 5'-trifosfato de la guía RNAs con fosfatasa alcalina intestinal de ternera (CIP) como sigue: tratar 10 μg de in vitro- ARN transcrito con 250 U de CIP por 3 h a 37 ° C en presencia de inhibidor de la U de Rnasa 100. Purificar lo sgRNA CIP tratados usando un kit de purificación de RNA.
5. expresión de la proteína WT y Sniper Cas9 y purificación
-
Expresión de proteínas en e. coli
- Transforman plásmidos del animal doméstico18 codificación etiquetado su peso - y Sniper-Cas9 en la BL21 (DE3) e. coli cepa.
- Sembrar 50 mL de medio LB con kanamicina 50 de μg/mL con una colonia fresca que el plásmido de expresión pET-Cas9 y agitar noche (200 rpm) a 37 ° C (planas).
- Transferir 10 mL de la cultura durante la noche a 500 mL de medio LB fresco que contiene kanamicina 50 de μg/mL. Incubar el cultivo con agitación (200 rpm) a 37 ° C durante 2 h.
- Supervisar el OD600 hasta que el cultivo alcanza la fase de registro medio de crecimiento (OD600 ≈ 0.6 – 0.7).
- Inducir la expresión de la proteína WT o francotirador Cas9 con isopropílico β-D-1-tiogalactopiranósido (IPTG, a una concentración final de 0.25 nM). Incubar el cultivo a 18 ° C durante la noche.
-
Purificación de proteínas
- La cosecha de las células por centrifugación a 5.000 x g por 10 min a 4 ° C.
- Resuspender el precipitado en el tampón de lisis (50 mM NaH2PO4, 300 mM NaCl, 10 mM imidazol, 4 mM Ditiotreitol [TDT], 5 mM benzamidine, fluoruro de phenylmethylsulfonyl de 100 mM [PMSF], pH 8) a 20 mL por gramo de peso húmedo.
- Añadir PMSF, TDT y la lisozima a una concentración final de 1 mg/mL cada e incubar la mezcla en hielo durante 30 minutos.
- Someter a ultrasonidos las células en hielo. Pulse repetidamente para 10 s en W 200 – 300 con 10 s entre cada pulso, por un tiempo total de 20 minutos de enfriamiento.
- Centrifugue el lisado a 6.000 x g durante 30 min a 4 ° C.
- Quite el sobrenadante a un tubo nuevo. Añadir 1 mL de resina de agarosa su atar a 5 mL de despejó lisado y agite suavemente durante 1 h a 4 ° C.
- Carga de la mezcla de resina de agarosa lisado/su-Bind en una columna con una salida inferior tapado.
- Quitar la tapa inferior y recoge el fluir por columna.
- Lavar la columna 2 x con tampón de lavado (50 mM NaH2PO4, 300 mM de NaCl, imidazol de 20 mM, pH 8); recoger la fracción de lavado para el análisis de sodio dodecil sulfato-poliacrilamida gel electroforesis (SDS-PAGE).
- Eluir la proteína 10 x con 1 mL de elución del almacenador intermediario que recoge muestras de 50 mM (NaH2PO4, 300 mM de NaCl, imidazol de 250 mM, pH 8), para SDS-PAGE.
- Concentrado de la proteína Cas9 WT o francotirador eluídas con un filtro de columna 100 kDa. Almacenar las muestras en una solución de 10 mM Tris-HCl, 150 mM NaCl y 50% de glicerol a-80 ° C.
6. entrega de RNP
-
Transfección y preparación de las células para la entrega de la RNP
- Mantener las células HEK293T de Dulbecco modificación medio del águila (DMEM) suplementado con 10% suero bovino fetal (FBS) y antibióticos de 1% a 37 ° C con 5% CO2.
- Mezclar proteína WT o francotirador Cas9 (2 μg) con sgRNA (2 μg) e Incube por 10 min a temperatura ambiente para hacer complejos de RNP.
- Trypsinize y contar las células. Preparar 2 x 104 células por una reacción. Lavar las células con solución salina tamponada con fosfato (PBS) y centrifugadora. Aspirar el sobrenadante y resuspender el precipitado con buffer de electroporación.
- Complejos Electroporate RNP en las células con la siguiente configuración, es decir, 1.300 V, 30 ms y un pulso. Placa las células en una placa de 48 pozos llenan con 500 μL de DMEM suplementado con SBF y antibióticos (como se describe en el paso 6.1.1) justo después de la electroporación. Incubar a 37 ° C con 5% CO2.
- Aislar el ADN genómico con un kit de preparación gDNA, 48 h después de la transfección.
7. transfección de plásmidos codifican Cas9 Sniper y sgRNA
-
Construcción de un plásmido sgRNA
- Orden hacia adelante y reversa oligos con las secuencias siguientes de la plantilla, es decir, hacia adelante: 5'-CACCGNNNNNNNNNNNNNNNNNNNN-3'; Reverso: 5'-AAACNNNNNNNNNNNNNNNNNNNNC-3'. Reemplace el N20 con la secuencia de destino obtenida en el paso 4.1.2. Acortar la secuencia de destino para una longitud de N19, N18 o N17 para sintetizar sgRNA truncada.
- Cocer ambos oligos en 1 x buffer de T4 ADN ligasa.
- Digerir el vector con enzimas de restricción BsaI pRG2.
- Gel purifica el vector digerido (3.300 bp) utilizando un gel de agarosa al 0.8%.
- Ligar el oligo recocido y el fragmento purificado usando T4 ligasa a 37 ° C: mezcla 50 ng de pRG2 digerido y 1 ng de oligo recocido en un volumen de reacción de 20 μL. Incubar a temperatura ambiente durante 15 minutos.
- Transformar la mezcla de la ligadura en la cepa de DH5 alfa y crecer transformantes en placas de agar LB con ampicilina (100 μg/mL) a 37 ° C. Para confirmar la inserción de la oligo en el vector de la secuencia estándar.
-
Transfección de plásmidos codifican Cas9 Sniper y sgRNA
- Mantener las células HEK293T en DMEM suplementado con 10% FBS y 1% antibióticos a 37 ° C con 5% CO2. El día antes de la transfección, trypsinize y contar las células. Cuando se trabaja en una escala de pozo 48, células de placa de 1 x 105 por pozo en 250 μL de medio de cultivo completo. Las células deben ser 50% – 80% confluente en el día de la transfección.
- En la escala de 48 pozos, preparar 250 ng de plásmido p3s Cas9 y 250 ng de plásmido sgRNA de transfección, utilizando un reactivo de transfección basada en lípidos. Mezclar la plásmidos en 25 μL de suero libre-MEM.
- Diluir 1 μL de reactivo de transfección con 25 μL de suero libre-MEM. Incubar la mezcla a temperatura ambiente por 5 min combina las dos mezclas e incubar la solución resultante a temperatura ambiente por 20 min para formar plásmido lipofectamine complejos.
- Después de 20 min de incubación, añadir 50 μL de la solución que contiene los complejos de reactivo de transfección de plásmidos directamente a cada uno así que contiene células y mezclan suavemente, meciendo la placa hacia adelante y hacia atrás. Incube las células a 37 ° C en un incubador de CO2 para transfección de post de 48 – 72 h antes del ensayo para la expresión del transgén.
8. cálculo de las frecuencias de indel determinar las actividades en el destino y objetivo
-
Objetivo profunda secuencia para el análisis de objetivos y potenciales sitios de fuera de objetivo
- Aislar el ADN genómico de paso 6.1.5 o 7.2.4 con un kit de preparación gDNA. Generar bibliotecas de secuenciación profunda por amplificación por PCR de la gDNA con cebadores dirigidos a objetivos y objetivo.
- Usar primers de índice para etiquetar cada muestra. Tema combinado las bibliotecas para la secuencia final emparejado usando una máquina de secuenciación de próxima generación.
-
Análisis de secuenciación profunda utilizando analizador de Cas
- Analizar los datos de secuenciación profunda usando el analizador de la Cas evaluación herramienta17.
- Elija los archivos Fastq bajo las fichas de lectura 1 y lectura 2 (leer 1 = XX_SXX_L001_R1_001.fastq, lea 2 = XX_SXX_L001_R2_001.fastq).
- Llenar la ficha de Información básica y la ficha de Análisis de parámetros , haga clic en el botón Enviar .
Resultados
Después de realiza la pantalla de francotirador, el porcentaje de colonias de supervivencia puede calcularse dividiendo el número de colonias en la placa LB que contiene cloranfenicol, la kanamicina, la arabinosa y la ATC (CKAA) por el número de colonias en la placa LB que contiene cloranfenicol y kanamicina solamente (CK). Este porcentaje fue generalmente muy bajo cuando el pantalla de francotirador fue realizado con las bibliotecas de SpCas9. Golpes de verdad-positivas pueden ser enriquecidas repitiendo la pantalla con la piscina sobreviviente. En esta pantalla de francotirador representante, por ejemplo, una tasa de supervivencia de 100% se obtuvo después de la tercera pantalla (figura 1). Transfecciones con RNPs o Sniper-Cas9 plasmídico pueden hacerse para varios objetivos y el resultante en el blanco y actividades objetivo medidas por dirigidas secuenciación del amplicón (figura 2). A más objetivos, francotirador Cas9 muestra el mismo nivel de actividades en blanco y cocientes más altos de especificidad en comparación con el sgRNAs truncado pesos pueden utilizarse también para mejorar la especificidad (figura 3). Sin embargo, su uso se limita a sólo unos pocos objetivos, porque resultan en bajas actividades de en el blanco en comparación con sgRNAs de larga duración, en la mayoría de los casos. Por lo tanto, sgRNAs con diferentes longitudes (de 17 a 20 mers) debe ser probada y en blanco y objetivo las actividades deben medirse para optimizar la especificidad.

Figura 1: Representante Sniper-pantallas realizaron con diferentes bibliotecas de variantes aleatorias de Cas9. Mutator biblioteca, EP EP biblioteca II y me indican las bibliotecas utilizando kits comerciales diferentes. Primer, segundo y final indican el número de veces que la pantalla de enriquecimiento fue realizado7. Esta figura ha sido modificada de Lee et al.7. Haga clic aquí para ver una versión más grande de esta figura.
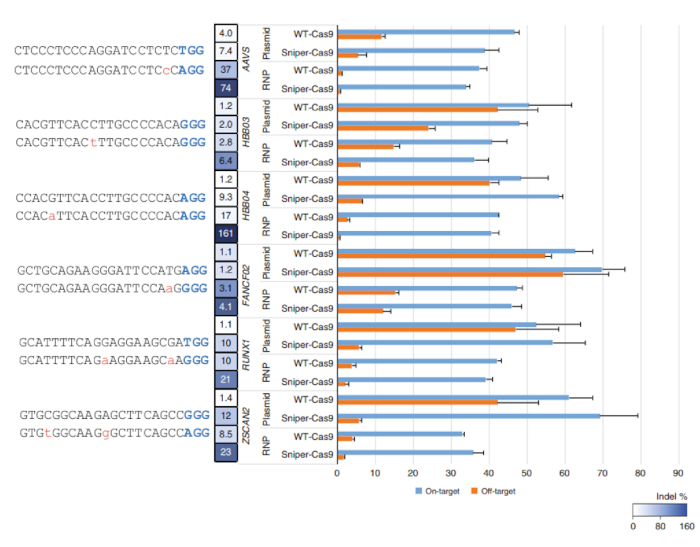
Figura 2: en blanco y objetivo actividades de WT-Cas9 o Sniper-Cas9 emparejado con una secuencia de guía 20-mer través de plásmido o RNP. Relaciones de especificidad se determinaron dividiendo la actividad en el destino por la actividad de objetivo. Barras de error indican SEM (n = 3)7. Esta figura ha sido modificada de Lee et al.7. Haga clic aquí para ver una versión más grande de esta figura.
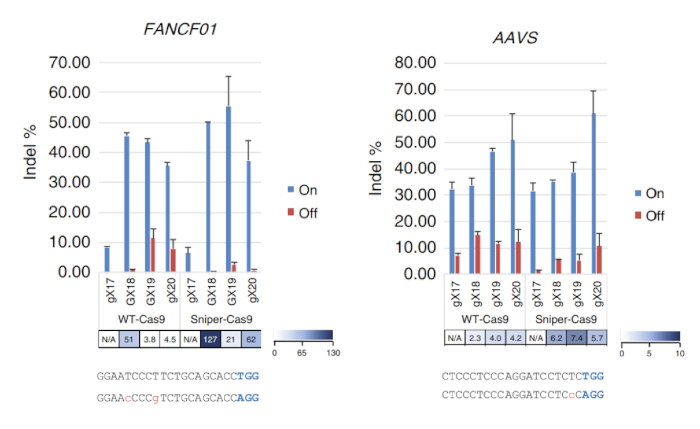
Figura 3: objetivos y actividades de francotirador-Cas9 comparado con WT-Cas9 obtenidos mediante sgRNAs con longitudes variables dirigidas a los sitios FANCF01 y aireación objetivo. Relaciones de especificidad se determinaron dividiendo indel frecuencias en sitios en blanco aquellos en los respectivos sitios de fuera de objetivo. sgRNAs con una guanina emparejada en la 5' terminal (GX18 o GX19) con una guanina no coinciden (gX17, gX18, gX19 o gX20) se indican. No se calcularon los ratios de especificidad cuando las actividades de en el blanco normalizadas < 70%7. Esta figura ha sido modificada de Lee et al.7. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Para aquellos que quieren evitar proyección engorrosos procedimientos para obtener Sniper Cas9, la proteína Cas9 Sniper y el plásmido de codificación están disponibles. Uso de estos materiales, la longitud óptima de lo sgRNA, que proporciona el cociente más alto de especificidad, debe ser determinado. Además, entrega de Sniper Cas9 y sgRNA en un formato de RNP es recomendable porque generalmente resulta en un cociente más alto de especificidad que entrega en forma de plásmido. A diferencia de francotirador-Cas9, otras variantes de Cas9 ingeniería no son compatibles con sgRNAs truncado6,7 o entrega en RNP forma8 (con la excepción de alta fidelidad-Cas9).
Para pantalla de francotirador, la selección de la secuencia es el paso más importante. Debe evitarse la selección de un sgRNA no coincidente que se asocia con actividad baja del escote hacia la secuencia GOI . Si no, Cas9 variantes con un nivel de especificidad WT no hienden la DNA genomic de e. coli con el sitio de destino no coinciden. Este efecto se traducirá en un gran número de colonias de fondo, poniendo en peligro todo el procedimiento de detección.
Porque e. coli tiene un rápido duplicando el tiempo y la eficiencia de transformación alta comparado con levadura, pantalla de francotirador es ventajoso en comparación con métodos de cribado basados en levadura. Además, debe ser más sensible que otras e. coliSniper-pantalla-basado en sistemas en que el sitio se realiza en un plásmido: hay una copia de la DNA genomic y, por tanto, sólo una copia del sitio no coinciden en nuestro sistema, pero un gran número de plásmidos en una sola célula de e. coli .
Las especificidades de otras endonucleasas de ADN que inducen distritales, como SaCas9 o Cpf1s, también pueden mejorarse mediante pantalla de francotirador. Por desgracia, pantalla de francotirador no puede utilizarse para aumentar la especificidad de los editores de base directamente, porque base editores no inducen distritales en la DNA genomic de e. coli. Como editores de base utilizan el nickase o muerta versión de Cas9 en la base de su sistema, las especificidades de los editores de base podrían aumentar mediante el uso de los éxitos obtenidos desde pantalla de francotirador.
Divulgaciones
ToolGen ha presentado una solicitud de patente (PCT/KR2017/006212) que cubre la pantalla de francotirador (estado: pendiente, inventor: Jungjoon K. Lee). Jungjoon K. Lee, Joonsun Lee, Minhee Jung y Euihwan Jeong son empleados de ToolGen, Inc.
Agradecimientos
Esta investigación fue apoyada por donaciones del Ministerio de ciencia y TIC de Corea (2017M3A9B4061406) y el nacional investigación Fundación de Corea (NRF) financiado por el gobierno coreano (MSIT) (grant números 2017M3A9B4061404 y 2018M3A9H3020844) para Jungjoon K. Lee. El plásmido pGRG36 de codificación fue un regalo de Nancy Craig (plásmido Addgene #16666) y p11-encaje-wtx1 fue un regalo de Huimin Zhao.
Materiales
| Name | Company | Catalog Number | Comments |
| Alkaline Phosphatase, Calf Intestinal (CIP) | NEB | M0290L | |
| Ampicillin Sodium Salt | Fisher Chemical | BP1760-25 | |
| Anhydrotetracycline hydrochloride | Sigma Aldrich | 37919 or 94664 | |
| Antibiotic Antimycotic solution | Welgene | LS203-01 | Media component |
| BamHI | Enzynomics | R003L | |
| Chloramphenicol | Sigma Aldrich | R4408 | |
| Diversify PCR Random Mutagenesis | Clontech | 630703 | Error-prone PCR kit |
| DNA-Shuffling Kit | Jena Bioscience | PP-103 | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Welgene | LM001-17 | Culture media |
| Endura Electrocompetent Cells (DUO) | Lucigen | 60242-2 | E. coli electrocompetent cells for library preparation |
| Fetal Bovine Serum (FBS) | Welgene | S101-01 | Media component |
| Gene Pulser II | Bio-Rad | E. coli electroporation equipment | |
| GeneMorph II Random Mutagenesis Kit | Agilent | 200550 | Error-prone PCR kit |
| genomic DNA prep kit | GenAll | 106-152 | gDNA-prep kit |
| Glycerol | BioShop | GLY001.1 | |
| GP/MP Cuvette, 0.1cm | Bio-Rad | BR165-2089 | E. coli electroporation equipment |
| HEK293T cell | ATCC | CRL-11268 | Human cell line |
| Kanamycin sulfate | Acros Organics | 450810500 | |
| L-(+)-Arabinose | Sigma Aldrich | A3256-25G | |
| LaboPass PCR Purification Kit | Cosmogenetech | CMR0112 | |
| LaboPass Plasmid Mini | Cosmogenetech | CMP0112 | Mini-prep kit |
| LB Agar Miller | Formedium | LMM0202 | |
| LB Broth Miller | Formedium | LMM0102 | |
| Lipofectamine | Invitrogen | 11668019 | Transfection reagent |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Gibson assembly (isothermal in vitro recombination) mixture |
| Neon Transfection System | Thermo | MPK5000 | RNP Electroporation equipment |
| Neon Transfection System 10 µL Kit | Thermo | MPK1096 | RNP Electroporation equipment |
| NucleoBond Xtra Midi EF | Macherey-Nagel | 740420.50 | Midi-prep kit |
| Oligo Clean & Concentrator | Zymo Research | D4061 | DNA purification kit that enables low-volume elution |
| Opti-MEM | Gibco | LS31985070 | Transfection media |
| p11-lacY-wtx1 | Addgene | #123945 | |
| pgrg36 | Addgene | #16666 | E. coli mutator strain |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Pyrophophatase | NEB | M2403L | |
| Ribonucleotide Solution Set | NEB | N0450L | |
| RNase inhibitor | NEB | M0314L | |
| T7 RNA polymerase | NEB | M0251L | |
| Turbo Dnase | Ambion | AM2238 | |
| XbaI | Enzynomics | R013L | |
| XL1-Red Competent Cells | Agilent | 200129 |
Referencias
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nature Biotechnology. 32 (3), 279-284 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Research. 24 (6), 1012-1019 (2014).
- Chen, J. S., et al. Enhanced proofreading governs CRISPR-Cas9 targeting accuracy. Nature. 550 (7676), 407-410 (2017).
- Kleinstiver, B. P., et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature. 529 (7587), 490-495 (2016).
- Slaymaker, I. M., et al. Rationally engineered Cas9 nucleases with improved specificity. Science. 351 (6268), 84-88 (2016).
- Casini, A., et al. A highly specific SpCas9 variant is identified by in vivo screening in yeast. Nature Biotechnology. 36 (3), 265-271 (2018).
- Lee, J. K., et al. Directed evolution of CRISPR-Cas9 to increase its specificity. Nature Communications. 9 (1), 3048(2018).
- Vakulskas, C. A., et al. A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nature Medicine. 24 (8), 1216-1224 (2018).
- Anderson, K. R., et al. CRISPR off-target analysis in genetically engineered rats and mice. Nature Methods. 15, 512-514 (2018).
- Kim, S., Bae, T., Hwang, J., Kim, J. S. Rescue of high-specificity Cas9 variants using sgRNAs with matched 5' nucleotides. Genome Biology. 18 (1), 218(2017).
- Kulcsar, P. I., et al. Crossing enhanced and high fidelity SpCas9 nucleases to optimize specificity and cleavage. Genome Biology. 18 (1), 190(2017).
- Zhang, D., et al. Perfectly matched 20-nucleotide guide RNA sequences enable robust genome editing using high-fidelity SpCas9 nucleases. Genome Biology. 18 (1), 191(2017).
- McKenzie, G. J., Craig, N. L. Fast, easy and efficient: site-specific insertion of transgenes into enterobacterial chromosomes using Tn7 without need for selection of the insertion event. BMC Microbiology. 6, 39(2006).
- Geissmann, Q. OpenCFU, a new free and open-source software to count cell colonies and other circular objects. PLoS One. 8 (2), e54072(2013).
- Chen, Z., Zhao, H. A highly sensitive selection method for directed evolution of homing endonucleases. Nucleic Acids Research. 33 (18), e154(2005).
- Kim, S., et al. CRISPR RNAs trigger innate immune responses in human cells. Genome Research. 28 (3), 367-373 (2018).
- Park, J., et al. Cas-Analyzer: an online tool for assessing genome editing results using NGS data. Bioinformatics. 33, 286-288 (2017).
- Studier, F. W., et al. Use of T7 RNA Polymerase to Direct Expression of Cloned Genes. Methods in Enzymology. 185, 60-89 (1990).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados