Method Article
Utilizzando il cecchino-Cas9 a minimizzare gli effetti di fuori bersaglio di CRISPR-Cas9 senza la perdita di attività On-target tramite Evolution diretto
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per ottimizzare CRISPR-Cas9 per ottenere una maggiore specificità senza la perdita di attività sul bersaglio. Utilizziamo un approccio diretto evoluzione chiamato Sniper-schermo per trovare un mutante Cas9 con le caratteristiche desiderate. Cecchino-Cas9 è compatibile con tronco singolo-guida RNAs e consegna in un formato della ribonucleoproteina, ben note strategie per conseguire maggiore specificità.
Abstract
Lo sviluppo di cluster regolarmente intercapedine ripetizioni brevi palindromi (CRISPR)-proteina associata 9 (Cas9) in modalità terapeutiche richiede l'elusione dei suoi effetti potenzialmente deleteri fuori bersaglio. Diversi metodi sono stati ideati per ridurre tali effetti. Qui, presentiamo un Escherichia coli-base diretta evoluzione metodo chiamato Sniper-schermo per ottenere una variante Cas9 con specificità ottimizzato e ha conservato l'attività sul bersaglio, chiamato Sniper-Cas9. Utilizzando il cecchino-schermo, selezione positiva e negativa possa essere eseguita contemporaneamente. Lo schermo può essere ripetuto anche con altre sequenze di RNA (sgRNA) singolo-guida per arricchire per i veri successi positivi. Utilizzando il promotore di CMV-PltetO1 dual per esprimere Cas9 varianti, le prestazioni della libreria pool possono essere controllate rapidamente in cellule di mammifero. Metodi per aumentare la specificità di cecchino-Cas9 inoltre sono descritti. In primo luogo, l'uso di sgRNAs troncato in precedenza ha dimostrato di aumentare la specificità di Cas9. A differenza di altri derivati dal Cas9s, Sniper-Cas9 mantiene un livello di (WT) di selvaggio-tipo di attività il bersaglio quando combinato con sgRNAs troncato. In secondo luogo, la consegna di cecchino-Cas9 in un formato di ribonucleoproteina (RNP) invece di un formato di plasmide è possibile senza intaccare la sua attività sul bersaglio.
Introduzione
In questa carta, puntiamo a migliorare la specificità della Cas9 combinando diverse strategie. Sono stati sviluppati diversi metodi per evitare gli effetti di fuori bersaglio di CRISPR-Cas9. Ad esempio, troncato sgRNAs può essere utilizzato per raggiungere maggiore specificità1. Inoltre, il metodo di consegna Cas9 può essere modificato da un formato di plasmide in un formato di RNP ottenere maggiore specificità2. Specifici residui amminoacidici del Cas9 di Streptococcus pyogenes (SpCas9) della proteina sono stati modificati secondo il razionale disegno descritto precedentemente3,4,5. In alternativa, residui dell'amminoacido sono stati alterati in modo casuale e le varianti di Cas9 con la più alta specificità sono state identificate utilizzando un lievito6 o un7,di e. coli8 sistema di screening.
Tuttavia, molti gruppi hanno segnalato che Cas9 varianti progettati utilizzando il design a debilitare l'interazione aspecifica tra Cas9 e il substrato espositivo basso on target attività7,8,9, 10 , 11 , 12. abbiamo sviluppato un e. coli-basato sistema evolution diretto, Sniper-schermo, allo schermo in modo casuale mutagenized Cas9 varianti. Un e. coli sistema di screening ha vantaggi rispetto ad un sistema di lievito a causa le efficienze di trasformazione tempo e superiore più veloce raddoppio di e. coli.
Positivi e negativi di selezione, basato su tre diversi plasmidi e un gene di interesse (GOI) integrato nel genoma di e. coli , sono utilizzati in Sniper-schermo. Cas9 varianti sono espressi sotto il sistema dual-promotore CMV-PltetO1 di un plasmide numero basso-copia, affinché i candidati identificati in e. coli possono essere testati in cellule di mammifero senza l'esigenza di subcloning. Il governo indiano è stato introdotto nel genoma di e. coli usando il sistema del trasposone Tn7. Il plasmide sgRNA, che contiene un termosensibile origine di replicazione, esprime un sgRNA targeting GOI; Tuttavia, il sgRNA e GOI sequenze non sono perfettamente abbinati. Esiste un sito di destinazione di sgRNA perfettamente su un terzo plasmide contenente il gene ccdB , che codifica per un prodotto letale che avvelena girasi. In questo sistema, le cellule che esprimono Cas9 varianti con alta attività fuori bersaglio vengono rimossi perché rotture del doppio filo (DSBs) vengono introdotti nel sito non corrispondente situato in DNA genomic. D'altra parte, le cellule che esprimono Cas9 varianti con bassa attività on target vengono rimossi anche a causa di espressione genica letale ccdB . Il livello di espressione delle varianti Cas9 può essere cambiato modificando la concentrazione di anidrotetraciclina (ATC), che regola la forza di selezione.
Abbiamo ragionato che l'individuazione del sito di destinazione non corrispondenti sgRNA nel DNA genomico, piuttosto che su un plasmide aumenterebbe la sensibilità del sistema. Il vantaggio di questo approccio è che c'è un solo sito genomico, considerando che ci sarebbero molti plasmidi, ciascuno contenente un sito di destinazione, all'interno di una singola cellula di Escherichia coli .
Utilizzando questo sistema, abbiamo identificato una variante Cas9, Sniper-Cas9, che Mostra attività sulla destinazione a livello di WT e ridotta attività fuori bersaglio rispetto al WT Cas9. Cecchino-Cas9 può raggiungere ancora più elevati rapporti di specificità utilizzando sgRNAs troncato o basati su RNP consegna anziché basata su plasmide consegna.
Protocollo
1. integrazione di un umano GOI nel ceppo di BW25141 e. coli
-
Clonazione del GOI
- Reazione a catena della polimerasi (PCR) amplificare una lunghezza di 500 bp di un umano GOI contenente vari siti di destinazione candidato tramite metodi standard di PCR con primer contenenti siti di enzima di restrizione NotI e XhoI.
Nota: In questo esperimento, il GOI è stato il gene umano EMX1 . - Digerire sia il prodotto PCR che il vettore di13 pgrg36 con enzimi di restrizione NotI e XhoI.
- Gel di purificare i frammenti desiderati (500 bp e 12 kb).
- Legare i frammenti insieme usando ligasi T4. Per fare questo, mescolare 50 ng di pgrg36 digerito e 6 ng di inserto PCR in un volume di reazione di 20 μL contenente tampone ligasi 1x e 0,5 U l'enzima ligasi. Incubare a temperatura ambiente (TA) per una notte.
- Trasformare il plasmide legato in cellule competenti DH5-α. Crescere la risultante trasformanti su piastre di agar di brodo (LB) di Luria contenente ampicillina (100 μg/mL) a 32 ° C ed incubare per una notte. Pick up una Colonia e crescere in media LB contenente ampicillina durante la notte. Isolare il DNA dal e. coli, utilizzando un miniprep disponibili in commercio Kit del plasmide. Confermare l'inserimento del frammento GOI di Sanger sequenziamento il plasmide ottenuto dal mini-plasmide preparazione (mini-prep)13.
- Reazione a catena della polimerasi (PCR) amplificare una lunghezza di 500 bp di un umano GOI contenente vari siti di destinazione candidato tramite metodi standard di PCR con primer contenenti siti di enzima di restrizione NotI e XhoI.
-
Preparazione del BW25141 -GOI
- Trasformare il plasmideGOI ottenuti pgrg36 - il ceppo di BW25141 e. coli . È essenziale utilizzare un ceppo di BW25141 al fine di ridurre al minimo il numero di falsi positivi colonie.
- Crescere le cellule trasformate nel buffer LB a 32 ° C durante la notte. GOI è inserito nel DNA genomic del ceppo BW25141 (BW25141 -GOI). Rimuovere il plasmideGOI pgrg36 - dal ceppo BW25141 -GOI utilizzando il protocollo standard pgrg3613. Brevemente, diluire una Colonia (circa 107-piegare) e crescere su una piastra di LB a 42 ° C durante la notte. Striscia di colonie sulla piastra LB e farle crescere a 42 ° C durante la notte.
- Confermare il corretto inserimento di GOI di PCR, utilizzando i primers suggeriti nel protocollo pgrg36 della Colonia: 5'-GATGCTGGTGGCGAAGCTGT-3 'e 5'-GATGACGGTTTGTCACATGGA-3'. Il primer amplifica il sito di inserzione di DNA genomico, e la dimensione del prodotto della PCR risultante sarà 904 bp più la dimensione dell'inserto (500 bp in questo caso).
- Preparare electrocompetent cellule BW25141 -GOI (un protocollo dettagliato è descritto nella procedura 3.2.6–3.2.10).
2. preparazione della biblioteca variante Cas9
-
Preparazione di biblioteca
- Trasformare il vettore Cas97 in un commerciale Escherichia coli mutator ceppo (Tabella materiali) e seguire le istruzioni del produttore per ottenere una variante biblioteca (la biblioteca di Mutator).
- Eseguire PCR errori sull'intera sequenza di WT Cas9 nel vettore Cas9, utilizzando un kit PCR errori (Tabella materiali).
Nota: Il protocollo di basso-errore tasso è stato adottato nel caso Sniper-Cas9 per evitare di interrompere la funzione originale della proteina. - Digerire il vettore Cas9 con enzimi di restrizione appropriati. Gel di purificare il prodotto di PCR (dal punto 2.1.2) e la spina dorsale digerita.
Nota: La dimensione del gene SpCas9 è circa 4,3 kb. XhoI e KpnI sono stati scelti per digerire vector pBLC-SpCas9 che è stato usato nel caso di cecchino-Cas9. - Assemblare il frammento di spina dorsale (dal punto 2.1.3) e l'inserto amplificato mediante PCR errori (dal punto 2.1.3) tramite ricombinazione isotermici in vitro .
Nota: più di 500 ng di backbone è necessaria per ottenere un'alta concentrazione di libreria (libreria di PCR [EP] errori). Kit PCR due errori diversi sono stati utilizzati per preparare le librerie EP nel caso Sniper-Cas9 (Biblioteca EP I e II). - Purificare i prodotti dall'assembly (dal punto 2.1.4) utilizzando un kit di purificazione di DNA che consente a basso volume di eluizione (Tabella materiali). Eluire con 6 μL di acqua priva di nucleasi (NFW) e misurare la concentrazione di DNA.
- Trasformare più di 500 ng di Cas9 libreria vettoriale (per ciascuna delle tre librerie) in 50 μL di cellule di e. coli electrocompetent (Tabella materiali). Vedi il protocollo di elettroporazione in passaggi 3.2.1-3.2.4. Per questa preparazione di libreria, è possibile utilizzare 1 mL di mezzo SOC anziché 250 µ l per 50 μL di cellule competenti.
- Fare 1: 100, 1:1, 000 e 1: 10.000 diluizioni della miscela contenente le celle recuperate con il mezzo di SOC. Piastra le cellule diluite su piastre di agar 100mm LB completati con cloramfenicolo (12,5 µ g/mL). Piastra le celle rimanenti su un piatto di2 245 mm. Incubare a 37 ° C durante la notte.
-
Calcolo della complessità di biblioteca
- Fotografare le piastre di diluizione tramite un sistema di documentazione del gel o una normale macchina fotografica digitale. Eseguire OpenCFU software14 e caricare le fotografie delle piastre di diluizione. Impostare l'area di conteggio all'interno di un piatto e rimuovere falsi colonie.
- Manualmente è possibile moltiplicare il numero delle colonie per il fattore di diluizione per ottenere il numero originale di trasformanti. Convertire questi numeri in forma logaritmica (base 10). Calcolare la media per determinare la complessità della biblioteca.
- Quando il valore di complessità desiderato è ottenuto, raccogliere tutte le colonie sulla piastra quadrata 245 mm (dal punto 2.1.7) utilizzando un divaricatore e 20 mL di LB completati con cloramfenicolo. Non crescono le colonie raccolte e purificare la libreria di plasmide utilizzando un kit commerciale midiprep.
Nota: Maggiore la complessità di libreria, il migliore. Quando è stato identificato da cecchino-Cas9, una diversità di 3 x 106 è stata realizzata per ogni libreria.
3. positivi e negativi di screening per l'evoluzione Cas9
-
Selezione dell'obiettivo e la costruzione del plasmide
- Selezionare una sequenza di destinazione sgRNA distanziatore in GOI. Sostituire uno o due residui nel nucleotide casuale per produrre una sequenza non corrispondente.
Nota: Sito di destinazione umana EMX1 3 (GAGTCCGAGCAGAAGAAGAA con GGG PAM) è stato utilizzato nel caso Sniper-Cas9. I followings sono le sequenze non corrispondenti utilizzate: GAGTCCGAGCAGAAagAGAA, GAacCCGAGCAGAAGAAGAA, GAGTCCGAGCAGAgGAAGAA e GAGcCCGAGCAGAAGAAGAA. - Inserire la sequenza non corrispondente (Vedi punto 3.1.1) nel plasmide sgRNA usando standard oligonucleotide (oligo) clonazione procedure7.
- Inserire la sequenza non corrispondente con un PAM all'estremità 3' p11-lacY-wtx1 (Tabella materiali) per costruire il plasmide ccdB utilizzando standard oligo clonazione procedure15.
- Selezionare una sequenza di destinazione sgRNA distanziatore in GOI. Sostituire uno o due residui nel nucleotide casuale per produrre una sequenza non corrispondente.
-
Preparazione di cellule competenti di cecchino-screening Escherichia coli
- Scongelare le cellule di electrocompetent BW25141 -GOI su ghiaccio.
- Aggiungere 1 ng ogni del plasmide ccdB e il plasmide sgRNA con doppia mismatch in 50 μL di cellule BW25141 -GOI scongelate. Delicatamente mescolare le cellule pipettando e spostarli in una cuvetta di elettroporazione prechilled 0,1 cm.
- Trasformare l' e. coli con i due plasmidi tramite elettroporazione. Aggiungere 250 μL di mezzo SOC immediatamente dopo l'elettroporazione. Pipettare delicatamente la soluzione per mescolare le cellule ed il mezzo. Trasferire il composto in una microcentrifuga da 1,5 mL.
Nota: Per la massima efficienza, impostare la tensione a 1.80 kV e il runtime deve essere compreso tra 4,8 ms e ms 5,0. - Recuperare le cellule trasformate e li Incubare a 32 ° C per 1 h con agitazione delicata.
- Piastra 125 μL delle cellule recuperate su un ampicillina (50 μg/mL) / piastra agar LB kanamicina (25 μg/mL) (condizione di cultura). Piastra le restanti celle su un ampicillina/kanamicina/arabinosio (1,5 mg/mL) piastra di agar LB (ccdB-esprimendo la condizione). Incubare a 32 ° C durante la notte.
- Verificare l'assenza di sopravvivere colonie su ccdB-esprimendo la piastra di condizione. Raccogliere colonie dalla piastra di condizione di cultura utilizzando un divaricatore e cultura li in 250 mL di brodo super ottimale (SOB) supplementato con 50 μg/mL di ampicillina e 25 μg/mL di kanamicina a 32 ° C, con delicata agitazione.
- Quando la densità ottica a 600 nm (OD600) raggiunge 0.4, raffreddare il pallone sul ghiaccio. Preparare una soluzione prechilled 10% glicerolo e prechilled acqua deionizzata (sterilizzare prima dell'uso).
- Centrifugare (a 4.000 x g per 5 min a 4 ° C) le cellule e scartare il surnatante. Aggiungere 200 mL di acqua deionizzata prechilled. Risospendere le cellule usando una pipetta sierologica di 10 mL. Ripetere questo passaggio x 3.
- Lavare le cellule con 50 mL di soluzione di glicerolo 10% prechilled. Centrifugare a loro come prima (a 4.000 x g per 5 min a 4 ° C).
- Eliminare il supernatante e risospendere il pellet in 300 μL di soluzione di 10% glicerolo. Fare 50 aliquote del μL e congelarli in azoto liquido. Conservare le cellule (cellule di cecchino-screening) a-80 ° C.
-
Cecchino-screening
- Trasformare le cellule Sniper-screening (dal punto 3.2.10) con 100 ng dei plasmidi varianti Cas9 da ogni biblioteca (dal passo di 2.2.3.See 2.1.1 e 2.1.4). La procedura di elettroporazione descritto nella procedura 3.2.1–3.2.3.
- Trasferire 250 μL delle cellule ad un tubo del microcentrifuge fresco 1,5 mL. Aggiungere 250 pg di ATC per rendere una concentrazione finale di 10 ng/mL. Recuperare le cellule contenenti ATC sia privo di ATC (Vedi punto 3.2.4 per la fase di recupero).
- Piastra 25 μL di cellule privo di ATC recuperate su una piastra di agar di cloramfenicolo/kanamicina LB (condizione non selettivi). Aggiungere ATC alle celle contenenti ATC recuperate per rendere una concentrazione finale di 100 ng/mL su un piatto LB 245 mm. Piastra immediatamente le cellule su una piastra di agar di cloramfenicolo/kanamicina/arabinosio LB (condizione selettiva). Incubare per una notte a 32 ° C.
Nota: Le dimensioni e il numero della piastra LB sono determinati dalle dimensioni della diversità delle coperture screening. Nel caso di un 100mm Petri con 20 mL di LB, aggiungere 2 μg di ATC. - Fotografare le piastre. Contare il numero di colonie vitali utilizzando OpenCFU software14. (Vedi punto 2.2.1) Assicurarsi che il numero di colonie sulla piastra non selettiva è almeno 10 volte più grandi rispetto alla diversità della libreria per coprire tutte le varianti.
- Calcolare la frequenza di sopravvivenza come segue.
Frequenza di sopravvivenza = il numero di colonie su una piastra selettiva / (il numero di colonie su una piastra non selettiva x 10) - Piscina le colonie che sono sopravvissuti sulle piastre selettive da tutte le tre librerie. Incubare le colonie sopravvissute in 250 mL di LB medium supplementato con 12,5 cloramfenicolo μg/mL a 42 ° C durante la notte. Isolare la libreria Cas9 schermata DNA utilizzando un kit di midiprep.
Nota: Questo passaggio consente di cancellare il plasmide sgRNA. - Ripetere il processo di screening da passaggi 3.3.1–3.3.6 fino a quando la frequenza di sopravvivenza raggiunge un plateau. Uso 10 ng del plasmide Cas9 selezionato per trasformazione e 10 ng/mL di ATC durante il recupero. Mantenere la concentrazione di ATC a 100 ng/mL per la condizione selettiva.
-
Mischiare e la seconda proiezione
- Rimescola le varianti pool selezionate utilizzando il seguente protocollo di DNA shuffling. PCR amplificano l'inserto Cas9 nel plasmide Cas9 utilizzando primers fiancheggianti, 150 nucleotidi dai confini di inserto. Digest 2 μg di prodotto PCR amplificato con dnasi I per 1 min a 37 ° C.
- Purificare i frammenti 70 – 200 bp di lunghezza mediante elettroforesi su gel di agarosio 2%. PCR amplificano i frammenti purificati. Utilizzare il prodotto come modello di PCR amplificano l'inserto Cas9 con appositi primer che fiancheggiano Cas9. Utilizzare il prodotto PCR finale per costruire una libreria di Cas9, come descritto al punto 2.1.4.
- Preparare nuove cellule Sniper-screening (Vedi sezione 3.2) con un altro non corrispondenti sgRNA plasmide (Vedi punto 3.1.1). Rifare il processo di screening (sezioni 3.2-3.3) fino a quando il tasso di sopravvivenza raggiunge un plateau. Uso 10 ng del plasmide Cas9 selezionato per trasformazione e 10 ng/mL ATC durante il recupero. Mantenere la concentrazione di ATC a 10 ng/mL per la condizione selettiva.
-
Selezione di evoluta Cas9 mutante plasmidi
- Dopo l'ultimo passaggio di screening, casualmente raccogliere cento colonie dalla piastra selettiva e cultura li in mezzo LB contenenti cloramfenicolo a 42 ° C durante la notte.
- Isolare i plasmidi con una procedura del miniprep e Sanger-sequenza gli inserti utilizzando primer di sequenziamento all'interno Cas9.
- Selezionare la top tre varianti più frequenti di testarli nelle linee cellulari umane.
4. consegna dei Cas9 come un RNP con sgRNA troncato
-
Selezione del gene target utilizzando Cas-OFFinder
- Scegliere siti di destinazione con Cas-OFFinder (http://www.rgenome.net/cas-offinder/). Selezionare il tipo di PAM appropriato per il tipo specifico di Cas9 e il genoma di destinazione (umano, mouse, zebrafish, ecc.). Compilare la scheda di sequenze di Query , scegliere la mancata corrispondenza del numeroe clicca sul pulsante Invia .
- Dopo pochi secondi, l'on target (con un numero di mancata corrispondenza di '0') e la volontà di fuori bersaglio siti vengono visualizzati. In generale, scegliere i siti fuori bersaglio con uno a tre mismatch.
-
Preparazione del modello sgRNA
- Ordinare i oligos crRNA e tracrRNA con le seguenti sequenze di modello, vale a dire crRNA sequenza: 5'-TAATACGACTCACTATAGGNNNNNNNNNNNNNNNNNNNNGTTTTAGAGCTAGAA-3'; sequenza di tracrRNA: 5'-AGCACCGACTCGGTGCCACTTTTTCAAGTTGATAACGGACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC-3'. Compilare N20 la sequenza di crRNA con la sequenza di destinazione ottenuta al punto 4.1.2. Per progettare sgRNAs troncato, è possibile rimuovere basi dall'estremità 5' di ottenere sequenze sgRNA N19, N18 o N17.
- Preparare la miscela PCR per l'amplificazione della sequenza codifica sgRNA come segue: combinare 10 μL di buffer 5x, 0,5 μL di oligo crRNA (100 pmol/μL), 0,5 μL di oligo tracrRNA (100 pmol/μL), 2,5 μL di dNTPs (10 mM), 0,5 µ l di DNA polimerasi e 36 μL di NFW (tabella materiali).
- Amplificare il modello utilizzando le seguenti condizioni, vale a dire, per la denaturazione iniziale: 1 min a 98 ° C; per la denaturazione, ricottura ed estensione: 10 s a 98 ° C, 15 s a 54 ° C, 20 s a 72 ° C, per 25 cicli; per l'estensione finale: 5 min a 72 ° C.
- Analizzare 2 μL di DNA (dal punto 4.2.3) del modello amplificato su un gel di agarosio al 2%. Purificare il modello utilizzando un kit di purificazione di PCR.
Nota: La dimensione del DNA della mascherina è 125 bp.
-
Sintesi di sgRNA
- Preparare la miscela di reazione per la sintesi di sgRNA come segue: combinare 8,5 µ l di DNA campione (dal punto 4.2.3), 1 μL di UTP (25 mM), 1 μL di CTP (25 mM), 1 μL di GTP (25 mM), 1 μL di ATP (25 mM), 4,2 μL di MgCl2 (100 mM) , 4,5 μL di T7 RNA polimerasi (50 U/μL), 3 μL di tampone di RNA polimerasi di x T7 10, 1,2 μL della pirofosfatasi (0,5 U/μL), 0.75 μL di inibitore di RNAsi (40 U/μL) e 4,2 μL di NFW.
- Incubare a 37 ° C, la miscela di reazione durante la notte (almeno per 10 h). Aggiungere 0,5 μL di dnasi (2 U/μL) alla miscela di reazione e incubare a 37 ° C per 15 – 30 min. purificare il sgRNA utilizzando un kit di purificazione di RNA.
- Per ridurre la tossicità causata da una risposta immunitaria innata innescata da 5'-trifosfato il sgRNA16, rimuovere la 5'-trifosfato della Guida RNAs con fosfatasi alcalina intestinale di vitello (CIP) come segue: trattare 10 µ g di in vitro- RNA trascritto con 250 U di CIP per 3 h a 37 ° C in presenza di inibitore di 100 U di RNAsi. Purificare il sgRNA CIP-trattati usando un kit di purificazione di RNA.
5. l'espressione della proteina WT e cecchino Cas9 e purificazione
-
Espressione della proteina in e. coli
- Trasformare i plasmidi pET18 codifica His-Tag WT - e Sniper-Cas9 nel BL21 (DE3) Escherichia coli ceppo.
- Inoculare 50 mL di mezzo LB contenente 50 kanamicina μg/mL con una colonia fresca che harboring il plasmide di espressione pET-Cas9 e agitare pernottamento (200 giri/min) a 37 ° C (coltura).
- Trasferire 10 mL di coltura durante la notte a 500 mL di terreno LB nuovo contenente 50 kanamicina μg/mL. Incubare la cultura con l'agitazione (200 giri/min) a 37 ° C per 2 h.
- Monitorare il OD600 fino a quando la cultura raggiunge la metà registro in fase di crescita (OD600 ≈ 0,6 – 0,7).
- Indurre l'espressione della proteina WT o Sniper Cas9 con isopropilico β-D-1-thiogalactopyranoside (IPTG, ad una concentrazione finale di 0.25 nM). Incubare la cultura a 18 ° C durante la notte.
-
Purificazione della proteina
- Raccogliere le cellule mediante centrifugazione a 5.000 x g per 10 min a 4 ° C.
- Risospendere il pellet in tampone di lisi (50 mM NaH2PO4, 300 mM NaCl, imidazolo di 10 mM, 4 mM dithiothreitol [DTT], 5 mM benzamidine, 100 mM phenylmethylsulfonyl fluoruro [PMSF], pH 8) a 20 mL per grammo di peso bagnato.
- Aggiungere PMSF, DTT e lisozima ad una concentrazione finale di 1 mg/mL e incubare la miscela sul ghiaccio per 30 min.
- Sonicare le cellule sul ghiaccio. Impulso ripetutamente per 10 s a 200 – 300 W con un 10 s raffreddamento periodo tra ogni impulso, per un tempo totale di 20 min.
- Centrifugare il lisato a 6.000 x g per 30 min a 4 ° C.
- Eliminare il surnatante in una nuova provetta. Aggiungere 1 mL di resina di agarosio His-Bind a 5 mL di deselezionata lisato e agitare delicatamente per 1 h a 4 ° C.
- Caricare la miscela di resina di agarosio lisato/His-Bind su una colonna con una presa inferiore rinforzata.
- Rimuovere il tappo di fondo e raccogliere il flusso-attraverso la colonna.
- Lavare la colonna 2 x con tampone di lavaggio (50 mM NaH2PO4, 300 mM NaCl, imidazolo di 20 mM, pH 8); raccogliere la frazione di lavaggio per l'analisi mediante elettroforesi su gel di sodio dodecil solfato-poliacrilammide (SDS-PAGE).
- Eluire la proteina 10 x con 1 mL di eluizione dell'amplificatore (50 millimetri NaH2PO4, 300 mM NaCl, imidazolo 250 mM, pH 8), raccolta campioni per SDS-PAGE.
- Concentrare la proteina eluiti WT - o Sniper-Cas9 utilizzando un filtro di colonna 100 kDa. Conservare i campioni in una soluzione di glicerolo al 10 mM Tris-HCl, 150 mM NaCl e 50% a-80 ° C.
6. consegna RNP
-
Transfezione e preparazione delle cellule per la consegna di RNP
- Mantenere HEK293T cellule in di Dulbecco modificate medium dell'Aquila (DMEM) supplementato con 10% siero bovino fetale (FBS) e gli antibiotici 1% a 37 ° C con 5% CO2.
- Mescolare proteina WT o Sniper Cas9 (2 μg) con sgRNA (2 μg) e incubare per 10 min a RT per rendere complessi RNP.
- Tripsinizzano e contare le celle. Preparare 2 x 104 celle per una reazione. Lavare le cellule con tampone fosfato salino (PBS) e centrifugare. Aspirare il supernatante e risospendere il sedimento con buffer di elettroporazione.
- Complessi di electroporate RNP nelle celle utilizzando le seguenti impostazioni, vale a dire 1.300 V, 30 ms e uno di impulso. Piastra le cellule su un piatto di 48-pozzo riempite con 500 μL di DMEM completate con FBS e antibiotici (come descritto al punto 6.1.1) subito dopo l'elettroporazione. Incubare a 37 ° C con 5% CO2.
- Isolare il DNA genomico con un kit di preparazione gDNA, 48 ore dopo la trasfezione.
7. transfezione di plasmidi codifica Sniper-Cas9 e sgRNA
-
Costruzione di un plasmide sgRNA
- Ordinare in avanti e invertire i oligos con le seguenti sequenze di modello, vale a dire in avanti: 5'-CACCGNNNNNNNNNNNNNNNNNNNN-3'; inversa: 5'-AAACNNNNNNNNNNNNNNNNNNNNC-3'. Sostituire N20 con la sequenza di destinazione ottenuta al punto 4.1.2. Accorciare la sequenza di destinazione per una lunghezza di N19, N18, N17 o sintetizzare sgRNA troncato.
- Tempri entrambi i oligos in 1 x T4 DNA ligasi tampone.
- Digerire il vettore pRG2 con enzima di restrizione BsaI.
- Gel di purificare il vettore digerito (3.300 bp) utilizzando un gel di agarosio 0.8%.
- Legare l'oligo ricotto e il frammento purificato utilizzando ligasi T4 a 37 ° c: mix 50 ng di pRG2 digerito e 1 ng di oligo ricotto in un volume di reazione di 20 μL. Incubare a temperatura ambiente per 15 min.
- Trasformare la miscela di legatura in ceppo DH5 alfa e crescere trasformanti su piastre di agar LB contenente ampicillina (100 μg/mL) a 37 ° C. Confermare l'inserimento di oligo nel vettore dall'ordinamento standard.
-
Transfezione di plasmidi codifica Sniper-Cas9 e sgRNA
- Mantenere le cellule HEK293T in DMEM completati con 10% FBS e 1% antibiotici a 37 ° C con 5% CO2. Il giorno prima di transfezione, tripsinizzano e contare le celle. Quando si lavora ad una scala di 48-Beh, piastra 1 x 105 celle per bene in 250 μL di medium di crescita completa. Le cellule dovrebbero essere 50% – 80% confluenti il giorno della transfezione.
- Alla scala 48-Beh, preparare 250 ng di plasmide p3s-Cas9 e 250 ng di plasmide sgRNA per la transfezione, utilizzando un reagente di transfezione basata sui lipidi. Mescolare i plasmidi in 25 μL di siero gratis-MEM.
- Diluire 1 μL di reagente di transfezione con 25 μL di siero gratis-MEM. Incubare la miscela a RT per 5 min, unire i due composti e incubare la soluzione risultante al RT per 20 min a formare plasmide-lipofectamine complessi.
- Dopo 20 minuti di incubazione, aggiungere 50 μL di soluzione contenente i complessi di reagente di transfezione plasmide direttamente a ciascun bene contenenti celle e mescolano delicatamente da dondolo avanti e indietro la piastra. Incubare le cellule a 37 ° C in un incubatore a CO2 per la transfezione di post di 48-72 h prima diluire per l'espressione del transgene.
8. calcolo delle frequenze di indel per determinare attività on target e fuori bersaglio
-
Mirate il sequenziamento profondo per l'analisi dei siti fuori bersaglio on target e potenziali
- Isolare il DNA di genomic dal punto 6.1.5 o 7.2.4 con un kit di preparazione gDNA. Generazione di librerie di sequenziamento profondo dall'amplificazione di PCR del gDNA con primer mira il bersaglio e fuori bersaglio.
- Utilizzare primer indice per etichettare ogni campione. Librerie in pool in base alla sequenziazione accoppiato-fine utilizzando una macchina di sequenziamento di nuova generazione.
-
Analisi di sequenziamento profondo utilizzando Cas-Analyzer
- Analizzare i dati di sequenziamento profondo utilizzando il tool di valutazione Cas-analizzatore17.
- Scegliere i file Fastq sotto le schede di lettura 1 e 2 di lettura (Read 1 = XX_SXX_L001_R1_001.fastq, leggi 2 = XX_SXX_L001_R2_001.fastq).
- Compila la scheda di Informazioni di base e la scheda Parametri di analisi fare clic sul pulsante Invia .
Risultati
Dopo Sniper-schermo viene eseguito, la percentuale delle colonie di sopravvivenza può essere calcolata dividendo il numero di colonie sulla piastra LB contenenti cloramfenicolo, kanamicina, arabinosio e ATC (CKAA) per il numero di colonie nella piastra LB contenente cloramfenicolo e kanamicina solo (CK). Questa percentuale era di solito molto bassa quando Sniper-schermo è stato effettuato con le librerie di SpCas9. True-positivi colpi possono essere arricchiti ripetendo lo schermo con la piscina superstita. In questo rappresentante Sniper-schermo, ad esempio, un tasso di sopravvivenza del 100% è stato ottenuto dopo la terza schermata (Figura 1). Transfezioni utilizzando RNP o plasmide-codificato Sniper-Cas9 possono essere fatto per vari obiettivi e la risultante sulla destinazione e attività fuori bersaglio misurata da mirati amplicone sequenziamento (Figura 2). Al più obiettivi, Sniper-Cas9 Mostra lo stesso livello di attività sul bersaglio e più alti rapporti di specificità rispetto alla sgRNAs WT Truncated utilizzabile anche per migliorare ulteriormente la specificità (Figura 3). Tuttavia, il loro uso è limitato a solo pochi obiettivi perché si traducono in attività a basso on target rispetto al full-length sgRNAs, nella maggior parte dei casi. Pertanto, sgRNAs con lunghezze diverse (da 17 a 20 mers) deve essere testato e attività on target sia fuori bersaglio deve essere misurata per ottimizzare la specificità.

Figura 1: rappresentante Sniper-schermi eseguita con diverse librerie di casuale varianti Cas9. Libreria di mutator, EP libreria ho e Biblioteca EP II indicano librerie realizzate con diversi kit commerciali. Primo, secondo e finale è necessario indicare il numero di volte che lo schermo di arricchimento è stato effettuato7. Questa figura è stata modificata da Lee et al.7. Clicca qui per visualizzare una versione più grande di questa figura.
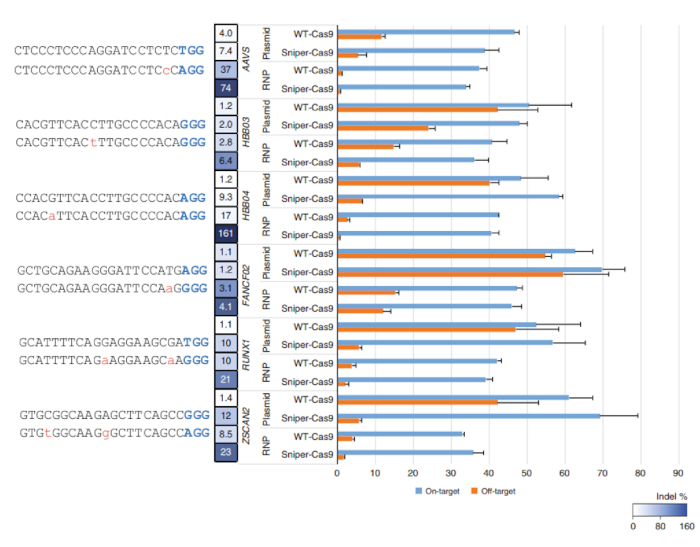
Figura 2: On-off obiettivo e attività di WT-Cas9 o Sniper-Cas9 accoppiato con una sequenza di guida 20-mer consegnata tramite plasmide o RNP. Rapporti di specificità sono stati determinati dividendo l'attività il bersaglio dall'attività fuori bersaglio. Barre di errore indicano SEM (n = 3)7. Questa figura è stata modificata da Lee et al.7. Clicca qui per visualizzare una versione più grande di questa figura.
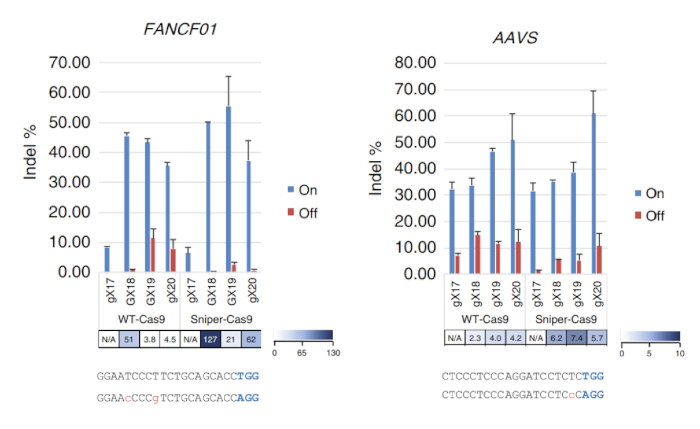
Figura 3: il bersaglio e attività di cecchino-Cas9 rispetto al WT-Cas9 ottenuti mediante sgRNAs con lunghezze variabili mira i siti FANCF01 e AAVS fuori bersaglio. Rapporti di specificità sono stati determinati dividendo le frequenze di indel siti sulla destinazione per quelli nei rispettivi siti fuori bersaglio. sgRNAs con una guanina abbinata all'estremità 5' (GX18 o GX19) e quelli con una guanina non corrispondente (gX17, gX18, gX19 o gX20) sono indicati. Rapporti di specificità non sono stati calcolati quando le attività di on target normalizzate erano < 70%7. Questa figura è stata modificata da Lee et al.7. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Per coloro che vogliono evitare procedure di screening ingombrante per ottenere Sniper-Cas9, la proteina di cecchino-Cas9 e il plasmide codifica sono disponibili. Utilizzando questi materiali, la lunghezza ottima del sgRNA, che fornendo il più alto rapporto di specificità, deve essere determinato. Inoltre, consegna di cecchino-Cas9 e sgRNA in un formato di RNP è consigliato perché di solito si traduce in un maggiore rapporto di specificità di consegna sotto forma di plasmide. A differenza di cecchino-Cas9, altre varianti di Cas9 derivati dal non sono compatibili con troncato sgRNAs6,7 o consegna in RNP modulo8 (ad eccezione di HiFi-Cas9).
Per Sniper-schermo, la selezione della sequenza non corrispondente è il passo più importante. Selezione di un sgRNA non corrispondente che è associato con attività di fenditura basso verso la sequenza GOI dovrebbe essere evitato. In caso contrario, Cas9 varianti con un livello WT di specificità non saranno fendere il DNA genomic di Escherichia coli con il sito di destinazione non corrispondenti. Questo effetto si tradurrà in un gran numero di colonie di sfondo, mettendo a repentaglio l'intero processo di vagliatura.
Perché Escherichia coli ha un veloce raddoppiando il tempo e l'efficienza di trasformazione alta rispetto al lievito, Sniper-schermo è vantaggioso rispetto ai metodi di screening basato su lievito. Inoltre, Sniper-schermo dovrebbe essere più sensibile di altri e. coli-sistemi in cui si svolge il sito non corrispondenti basati su un plasmide: c'è una copia del DNA genomic e così solo una copia del sito non corrispondente nel nostro sistema, ma un gran numero di plasmidi all'interno di una singola cellula di Escherichia coli .
La specificità di altri endonucleasi di DNA che inducono DSBs, ad esempio SaCas9 o Cpf1s, potrebbe anche essere migliorata utilizzando Sniper-schermo. Purtroppo, Sniper-schermo non utilizzabile per aumentare la specificità dei redattori base direttamente, perché base redattori non inducono DSBs nel DNA genomico di e. coli. Come editor di base utilizza il nickase o la versione morto di Cas9 nel nucleo del loro sistema, le specificità dei redattori di base potrebbero essere aumentate utilizzando i successi ottenuti da cecchino-schermo.
Divulgazioni
ToolGen ha depositato una domanda di brevetto (PCT/KR2017/006212) che copre Sniper-schermo (stato: in sospeso, inventore: Jungjoon K. Lee). Jungjoon K. Lee, Joonsun Lee, Minhee Jung ed Euihwan Jeong sono dipendenti di ToolGen, Inc.
Riconoscimenti
Questa ricerca è stata sostenuta da sovvenzioni dal Ministero della scienza e ICT di Corea (2017M3A9B4061406) e la nazionale Ricerca Fondazione della Corea (NRF) finanziata dal governo coreano (MSIT) (grant numeri 2017M3A9B4061404 e 2018M3A9H3020844) a Jungjoon K. Lee. Il plasmide codifica pGRG36 era un regalo da Nancy Craig (Addgene plasmide #16666) e p11-LacY-wtx1 era un regalo da Huimin Zhao.
Materiali
| Name | Company | Catalog Number | Comments |
| Alkaline Phosphatase, Calf Intestinal (CIP) | NEB | M0290L | |
| Ampicillin Sodium Salt | Fisher Chemical | BP1760-25 | |
| Anhydrotetracycline hydrochloride | Sigma Aldrich | 37919 or 94664 | |
| Antibiotic Antimycotic solution | Welgene | LS203-01 | Media component |
| BamHI | Enzynomics | R003L | |
| Chloramphenicol | Sigma Aldrich | R4408 | |
| Diversify PCR Random Mutagenesis | Clontech | 630703 | Error-prone PCR kit |
| DNA-Shuffling Kit | Jena Bioscience | PP-103 | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Welgene | LM001-17 | Culture media |
| Endura Electrocompetent Cells (DUO) | Lucigen | 60242-2 | E. coli electrocompetent cells for library preparation |
| Fetal Bovine Serum (FBS) | Welgene | S101-01 | Media component |
| Gene Pulser II | Bio-Rad | E. coli electroporation equipment | |
| GeneMorph II Random Mutagenesis Kit | Agilent | 200550 | Error-prone PCR kit |
| genomic DNA prep kit | GenAll | 106-152 | gDNA-prep kit |
| Glycerol | BioShop | GLY001.1 | |
| GP/MP Cuvette, 0.1cm | Bio-Rad | BR165-2089 | E. coli electroporation equipment |
| HEK293T cell | ATCC | CRL-11268 | Human cell line |
| Kanamycin sulfate | Acros Organics | 450810500 | |
| L-(+)-Arabinose | Sigma Aldrich | A3256-25G | |
| LaboPass PCR Purification Kit | Cosmogenetech | CMR0112 | |
| LaboPass Plasmid Mini | Cosmogenetech | CMP0112 | Mini-prep kit |
| LB Agar Miller | Formedium | LMM0202 | |
| LB Broth Miller | Formedium | LMM0102 | |
| Lipofectamine | Invitrogen | 11668019 | Transfection reagent |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Gibson assembly (isothermal in vitro recombination) mixture |
| Neon Transfection System | Thermo | MPK5000 | RNP Electroporation equipment |
| Neon Transfection System 10 µL Kit | Thermo | MPK1096 | RNP Electroporation equipment |
| NucleoBond Xtra Midi EF | Macherey-Nagel | 740420.50 | Midi-prep kit |
| Oligo Clean & Concentrator | Zymo Research | D4061 | DNA purification kit that enables low-volume elution |
| Opti-MEM | Gibco | LS31985070 | Transfection media |
| p11-lacY-wtx1 | Addgene | #123945 | |
| pgrg36 | Addgene | #16666 | E. coli mutator strain |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Pyrophophatase | NEB | M2403L | |
| Ribonucleotide Solution Set | NEB | N0450L | |
| RNase inhibitor | NEB | M0314L | |
| T7 RNA polymerase | NEB | M0251L | |
| Turbo Dnase | Ambion | AM2238 | |
| XbaI | Enzynomics | R013L | |
| XL1-Red Competent Cells | Agilent | 200129 |
Riferimenti
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nature Biotechnology. 32 (3), 279-284 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Research. 24 (6), 1012-1019 (2014).
- Chen, J. S., et al. Enhanced proofreading governs CRISPR-Cas9 targeting accuracy. Nature. 550 (7676), 407-410 (2017).
- Kleinstiver, B. P., et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature. 529 (7587), 490-495 (2016).
- Slaymaker, I. M., et al. Rationally engineered Cas9 nucleases with improved specificity. Science. 351 (6268), 84-88 (2016).
- Casini, A., et al. A highly specific SpCas9 variant is identified by in vivo screening in yeast. Nature Biotechnology. 36 (3), 265-271 (2018).
- Lee, J. K., et al. Directed evolution of CRISPR-Cas9 to increase its specificity. Nature Communications. 9 (1), 3048(2018).
- Vakulskas, C. A., et al. A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nature Medicine. 24 (8), 1216-1224 (2018).
- Anderson, K. R., et al. CRISPR off-target analysis in genetically engineered rats and mice. Nature Methods. 15, 512-514 (2018).
- Kim, S., Bae, T., Hwang, J., Kim, J. S. Rescue of high-specificity Cas9 variants using sgRNAs with matched 5' nucleotides. Genome Biology. 18 (1), 218(2017).
- Kulcsar, P. I., et al. Crossing enhanced and high fidelity SpCas9 nucleases to optimize specificity and cleavage. Genome Biology. 18 (1), 190(2017).
- Zhang, D., et al. Perfectly matched 20-nucleotide guide RNA sequences enable robust genome editing using high-fidelity SpCas9 nucleases. Genome Biology. 18 (1), 191(2017).
- McKenzie, G. J., Craig, N. L. Fast, easy and efficient: site-specific insertion of transgenes into enterobacterial chromosomes using Tn7 without need for selection of the insertion event. BMC Microbiology. 6, 39(2006).
- Geissmann, Q. OpenCFU, a new free and open-source software to count cell colonies and other circular objects. PLoS One. 8 (2), e54072(2013).
- Chen, Z., Zhao, H. A highly sensitive selection method for directed evolution of homing endonucleases. Nucleic Acids Research. 33 (18), e154(2005).
- Kim, S., et al. CRISPR RNAs trigger innate immune responses in human cells. Genome Research. 28 (3), 367-373 (2018).
- Park, J., et al. Cas-Analyzer: an online tool for assessing genome editing results using NGS data. Bioinformatics. 33, 286-288 (2017).
- Studier, F. W., et al. Use of T7 RNA Polymerase to Direct Expression of Cloned Genes. Methods in Enzymology. 185, 60-89 (1990).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon