Method Article
באמצעות צלף-Cas9 כדי למזער את ההשפעות את המטרה של CRISPR-Cas9 ללא אובדן פעילות--היעד באמצעות אבולוציה מכוונת
* These authors contributed equally
In This Article
Summary
כאן, אנו מציגים פרוטוקול כדי למטב את CRISPR-Cas9 כדי להשיג הם ברמת פירוט גבוהה ללא אובדן פעילות--היעד. אנו משתמשים בגישה אבולוציה מכוונת נקרא צלף מסך כדי למצוא. את המוטציה Cas9 עם התכונות הרצויות. צלף-Cas9 היא תואמת RNAs יחיד-מדריך קטום ו משלוח בתבנית ribonucleoprotein, ידועים אסטרטגיות להשגת specificities גבוהה יותר.
Abstract
ההתפתחות באשכולות interspaced בקביעות קצר palindromic חזרה (CRISPR)-חלבון המשויך 9 (Cas9) לתוך שיטות טיפוליות דורש ההתרחקות שלה מהשפעות חופש-יעד פוטנציאלי. מספר שיטות המציאו כדי לצמצם תופעות כאלה. כאן, אנו מציגים של Escherichia coli-שיטה המבוססת על אבולוציה מכוונת נקרא צלף-המסך כדי להשיג גרסה Cas9 עם ירידה לפרטים ממוטבת, נשמר ביעד פעילות, נקרא צלף-Cas9. באמצעות מסך צלף, חיוביות ושליליות הבחירה יכולה להתבצע בו זמנית. המסך יכול לחזור גם עם אחרים רצפי RNA (sgRNA) יחיד-מדריך להעשיר עבור הלהיטים חיובי אמיתי. באמצעות מקדם כפול על CMV-PltetO1 להביע את Cas9 וריאציות, הביצועים של הספרייה במאגר ניתן לבדוק במהירות בתאי יונקים. שיטות להגברת יחודיות של צלף-Cas9 מתוארים גם. ראשית, השימוש sgRNAs קטום שהוצג בעבר כדי להגדיל את Cas9 ירידה לפרטים. בניגוד Cas9s אחרים מהונדסים, צלף-Cas9 שומר על רמה (WT) פראי-סוג של פעילות--יעד בשילוב עם sgRNAs קטום. שנית, המסירה של צלף-Cas9 בתבנית ribonucleoprotein (RNP) במקום בתבנית פלסמיד אפשרי מבלי להשפיע על הפעילות על המטרה שלה.
Introduction
בנייר זה, אנו שואפים לשפר את ייחודה של Cas9 על ידי שילוב של אסטרטגיות שונות. פותחו שיטות שונות של הימנעות ההשפעות את המטרה של CRISPR-Cas9. לדוגמה, ניתן להשתמש sgRNAs קטום כדי להשיג ירידה לפרטים גבוה1. בנוסף, ניתן לשנות השיטה של Cas9 משלוח של תבנית פלסמיד לתבנית RNP כדי להשיג ירידה לפרטים גבוה יותר2. חומצת אמינו ספציפית שאריות Cas9 הפישחה ינפל תומימת סטרפטוקוקוס (SpCas9) חלבון שונו בהתאם הרציונלי עיצוב שתוארה לעיל4,3,5. לחלופין, שאריות חומצה אמינית שונתה באופן אקראי, גרסאות Cas9 עם יחודיות הגבוהה זוהו באמצעות שמרים6 או7, e. coli8 מערכת הסינון.
עם זאת, קבוצות רבות דיווחו כי גרסאות Cas9 הנדסה לאחור באמצעות העיצוב כדי להחליש את האינטראקציה לא ספציפית בין Cas9 את המצע בנספח פעילויות ביעד נמוך7,8,9, 10 , 11 , 12. פיתחנו של e. coli-מבוסס מערכת אבולוציה מכוונת, צלף מסך, למסך משתנים Cas9 mutagenized באופן אקראי. E. coli מערכת הסינון יש יתרונות על פני מערכת שמרים בגלל היעילות זמן והתמרת גבוה יותר מהר ההכפלה של e. coli.
בחירה שליליות וחיוביות, המבוסס על שלושה פלסמידים שונים ועל הגן עניין (גוי) משולב לתוך הגנום e. coli , משמשים מסך צלף. גרסאות Cas9 באים לידי ביטוי תחת מערכת כפולה-יזם CMV-PltetO1 של פלסמיד מספר עותק נמוך כך ניתן לבחון את המועמדים המזוהים ב- e. coli בתאי יונקים ללא צורך subcloning. גוי הוא הציג לתוך הגנום e. coli באמצעות מערכת transposon Tn7. פלסמיד sgRNA, אשר מכיל מקור רגיש לטמפרטורה של שכפול, מבטא את sgRNA של פילוח של גוי; עם זאת, sgRNA ואת גוי רצפים הם לא מתאימים. אתר יעד sgRNA להתאמה מושלמת קיים פלסמיד השלישי המכילים את הגן ccdB , שמקודד מוצר קטלני מרעילה gyrase. במערכת זו, תאים לבטא את Cas9 וריאציות עם פעילויות מחוץ-יעד גבוה מוסרים כי כפול-strand מעברי (DSBs) הם הציגו לתוך האתר לא תואמת ממוקם ב- DNA גנומי. מצד שני, התאים לבטא את Cas9 וריאציות עם פעילויות ביעד נמוך גם מוסרים בגלל התבטאות גנים קטלני ccdB . ניתן לשנות את רמת הביטוי של המשתנים Cas9 על ידי שינוי הריכוז של anhydrotetracycline (ATC), אשר מתאימה את כוח הבחירה.
אנחנו הסיק כי איתור אתר היעד sgRNA תואמים ב- DNA גנומי, ולא על פלסמיד יגביר את הרגישות של המערכת. היתרון של גישה זו הוא שיש אתר גנומית אחד בלבד, ואילו היו פלסמידים רבים, שכל אחד מהם מכיל אתר יעד, בתוך אחת תא החיידק .
באמצעות מערכת זו, זיהינו משתנה Cas9, צלף-Cas9, אשר מציג את פעילויות ביעד WT ברמת צמצמה את המטרה פעילויות בהשוואה WT Cas9. צלף-Cas9 ניתן להשיג יחס ירידה לפרטים גבוהה יותר על-ידי שימוש sgRNAs קטום או מבוסס-RNP משלוח במקום משלוח מבוסס פלסמיד.
Protocol
1. שילוב האדם גוי המתח BW25141 e. coli
-
שכפול של גוי
- תגובת שרשרת פולימראזית (PCR) להגביר את אורך bp 500 של האדם גוי המכיל אתרי היעד המועמד שונים באמצעות שיטות PCR סטנדרטי עם תחל המכיל אתרי אנזים הגבלה NotI ולא XhoI.
הערה: בניסוי זה, היה גוי הגן EMX1 האנושית. - לעכל את מוצר ה-PCR והן הווקטור13 pgrg36 עם אנזימי הגבלה NotI ו- XhoI.
- ג'ל לטהר את שברי הרצוי (500 bp ו 12 kb).
- מאתרים ומפסיקים את השברים יחד באמצעות ליגאז T4. כדי לעשות זאת, מערבבים 50 ng של pgrg36 מתעכל ו- 6 ng של PCR להוסיף נפח התגובה של 20 μL המכיל מאגר ליגאז 1 של x ו- 0.5 האנזים ליגאז U. דגירה בטמפרטורת החדר (RT) בן לילה.
- להפוך את פלסמיד מחוברים תאים המוסמכת DH5-α. לגדל את transformants שנוצר על צלחות אגר לוריא מרק (LB), המכיל אמפיצילין (100 μg/mL)-32 מעלות צלזיוס, דגירה בין לילה. לאסוף את מושבה, לגדל את זה בתקשורת LB המכיל אמפיצילין בן לילה. לבודד את פלסמיד הדנ א e. coli, באמצעות miniprep זמינים מסחרית של קיט. לאשר את הכניסה של השבר גוי על ידי סנגר רצף של פלסמיד המתקבל פלסמיד מיני הכנה (הכנה המצומצמת)13.
- תגובת שרשרת פולימראזית (PCR) להגביר את אורך bp 500 של האדם גוי המכיל אתרי היעד המועמד שונים באמצעות שיטות PCR סטנדרטי עם תחל המכיל אתרי אנזים הגבלה NotI ולא XhoI.
-
הכנת BW25141 -גוי
- להפוך את pgrg36 שהושג -גוי פלסמיד את המתח BW25141 e. coli . זה חיוני להשתמש זן BW25141 על מנת לצמצם את מספר מושבות חיובי כוזב.
- לגדל את התאים טרנספורמציה במאגר LB-32 מעלות צלזיוס בין לילה. גוי מוכנס לתוך ה-DNA גנומי של המתח BW25141 (BW25141 -גוי). הסר את pgrg36 -גוי פלסמיד את המתח BW25141 -גוי באמצעות פרוטוקול סטנדרטי pgrg3613. בקצרה, לדלל מושבה (כ 107-מקפלים) לגדל אותו בצלחת LB-42 ° C בין לילה. צובעות את המושבות על הצלחת ליברות, לגדל אותם ב 42 מעלות צלזיוס למשך הלילה.
- לאשר את הכניסה גוי הנכונה על ידי המושבה PCR, באמצעות תחל את הציע בפרוטוקול pgrg36: 5'-GATGCTGGTGGCGAAGCTGT-3 'ו 5'-GATGACGGTTTGTCACATGGA-3'. ומהצמיגים מגביר את האתר ההכנסה דנ א גנומי, ויהיה בגודל של ה-PCR ובמוצר 904 bp בנוסף לגודלם של הקדמי (500 bp במקרה זה).
- להכין electrocompetent תאים BW25141 -גוי (פרוטוקול מפורט המתואר 3.2.6–3.2.10 צעדים).
2. הכנה של הספרייה משתנה Cas9
-
ספריית הכנה
- להפוך את וקטור Cas97 בתוך מסחרית e. coli הנתונים זן (טבלה של חומרים), בצע את הוראות היצרן כדי להשיג ספרייה variant (הספרייה הנתונים).
- לבצע PCR לשגיאות על הרצף WT Cas9 שלם בוקטור Cas9, באמצעות ערכת ה-PCR לשגיאות (טבלה של חומרים).
הערה: פרוטוקול שיעור נמוך-שגיאה אומצה במקרה צלף-Cas9 כדי להימנע מהפרעה הפונקציה המקורית של החלבון. - לעכל את הווקטור Cas9 עם אנזימי הגבלה המתאים. ג'ל לטהר את המוצר PCR (מתוך שלב 2.1.2) ואת עמוד השדרה מעוכל.
הערה: הגודל של הגן SpCas9 היא בערך 4.3 kb. XhoI, KpnI נבחרו לעכל את וקטור pBLC-SpCas9 אשר שימש במקרה צלף-Cas9. - להרכיב על המקטע המרכזי (backbone) (מתוך שלב 2.1.3) וגם את תותב מוגבר באמצעות PCR לשגיאות (מתוך שלב 2.1.3) באמצעות איזותרמי במבחנה רקומבינציה.
הערה: יותר מ-500 ng חוט שדרה היא צריכה להשיג ריכוז גבוה של הספרייה (ספריית ה-PCR [EP] לשגיאות). שני קיטים שונים לשגיאות PCR שימשו להכנת EP ספריות במקרה צלף-Cas9 (ספריית EP I ו- II). - לטהר את המוצרים באמצעות ההרכבה (מתוך שלב 2.1.4) באמצעות ערכת טיהור DNA המאפשר בנפח נמוך • תנאי (טבלה של חומרים). Elute עם 6 μL של מים נטולי נוקלאז (בא ל) ומדידת הריכוז של ה-DNA.
- שינוי צורה יותר מ 500 ננוגרם של וקטור הספרייה Cas9 (עבור כל אחד של שלוש ספריות) לתוך μL 50 electrocompetent e. coli תאים (טבלה של חומרים). ראה פרוטוקול אלקטרופורציה שלבים 3.2.1-3.2.4. הכנה זו ספריה ', השתמש 1 מ"ל של מדיום SOC במקום μL 250 לכל 50 μL של תאים המוסמכת.
- להפוך מטריים, 1:1, 000, 1:10,000 דילולים של תערובת המכילות את התאים התאושש SOC בינונית. צלחת התאים מדולל על פלטות אגר 100 מ מ LB בתוספת כלורמפניקול (12.5 μg/mL). צלחת התאים הנותרים על צלחת2 245 מ מ. דגירה ב 37 מעלות צלזיוס למשך הלילה.
-
חישוב של ספריית מורכבות
- לצלם את הצלחות דילול באמצעות מערכת תיעוד ג'ל או מצלמה דיגיטלית רגילה. להפעיל את התוכנה OpenCFU14 ולהעלות את התמונות של הלוחות דילול. הגדר את אזור הספירה בתוך צלחת ולהסיר קולוניות כוזבים.
- באופן ידני להכפיל את מספר מושבות על ידי הגורם דילול כדי להשיג את המספר המקורי של transformants. להמיר את המספרים האלה לתוך טופס לוגריתמי (בסיס 10). לחשב את הממוצע כדי לקבוע את המורכבות של הספרייה.
- כאשר מתקבל ערך המורכבות הרצוי, מתאספים כל המושבות בצלחת 245 מ מ מרובע (מתוך שלב 2.1.7) באמצעות מפזר 20 מ ל LB בתוספת כלורמפניקול. לא לגדל את המושבות שנאספו ולטהר הספרייה פלסמיד באמצעות ערכת midiprep מסחרי.
הערה: גבוה יותר למורכבות ספריה, כן ייטב. כאשר מזוהה צלף-Cas9, מגוון של 3 x 106 הושג עבור כל ספריה.
3. חיוביים ושליליים הקרנת מתפתח Cas9
-
בחירת היעד ובנייה פלסמיד
- בחר יעד sgRNA מרווח רצף גוי. החלף את שאריות אחד או שניים נוקלאוטיד אקראי כדי לייצר רצף לא תואמת.
הערה: אתר היעד האנושי EMX1 3 (GAGTCCGAGCAGAAGAAGAA עם פאם GGG) שימש במקרה צלף-Cas9. להלן רצפי תואמים בשימוש: GAGTCCGAGCAGAAagAGAA, GAacCCGAGCAGAAGAAGAA, GAGTCCGAGCAGAgGAAGAA ו- GAGcCCGAGCAGAAGAAGAA. - הוסף את רצף לא תואמת (ראה שלב 3.1.1) לתוך פלסמיד sgRNA באמצעות oligonucleotide רגיל (oligo) שיבוט הליכים7.
- הכנס את רצף לא תואמת עם פאם בקצה 3' p11-תחרה-wtx1 (טבלה של חומרים) כדי לבנות את פלסמיד ccdB באמצעות הליכים שיבוט oligo תקן15.
- בחר יעד sgRNA מרווח רצף גוי. החלף את שאריות אחד או שניים נוקלאוטיד אקראי כדי לייצר רצף לא תואמת.
-
הכנה של תאים המוסמכת צלף-הקרנה e. coli
- הפשרת תאים electrocompetent BW25141 -גוי על קרח.
- להוסיף 1 ng של פלסמיד את ccdB , את פלסמיד sgRNA עם אי-התאמות כפול לתוך μL 50 של תאים BW25141 -גוי המופשרים. בעדינות לערבב את התאים באמצעות pipetting, להעביר אותם לתוך cuvette אלקטרופורציה prechilled ס מ 0.1.
- להפוך את החיידק עם פלסמידים 2 דרך אלקטרופורציה. הוסף μL 250 SOC בינוני מיד לאחר אלקטרופורציה. Pipette בעדינות את הפתרון לערבב את התאים ואת המדיום. להעביר את התערובת צינור microcentrifuge 1.5 mL.
הערה: עבור יעילות מקסימלית, להגדיר את המתח ב 1.80 kV ולאחר הריצה צריך להיות בין 4.8 ms ו- 5.0 ms. - לשחזר את התאים טרנספורמציה, דגירה אותם ב 32 ° C עבור h 1 ברעידות עדין.
- צלחת μL 125 של התאים התאושש לוקחת אמפיצילין (50 μg/mL) / צלחת אגר LB kanamycin (25 μg/mL) (תנאי תרבות). צלחת הנותרים תאים אמפיצילין/kanamycin/אראבינוז (1.5 mg/mL) על צלחת אגר ליברות (ccdB-מביע תנאי). דגירה-32 מעלות צלזיוס למשך הלילה.
- בדוק אם העדר לשרוד את המושבות ccdB-ביטוי תנאי צלחת. אסוף מושבות מהצלחת תנאי התרבות באמצעות מפזר תרבות אותם ב- 250 מ של מדיום מרק סופר אופטימלית (בכי) בתוספת 50 μg/mL של אמפיצילין, 25 μg/מ ל kanamycin ב 32 ° C, עדין רועדות.
- כאשר הצפיפות האופטית-600 nm (OD600) מגיע ל 0.4, מקררים את הבקבוק על קרח. להכין מים יונים prechilled ופתרון גליצרול 10% prechilled (לעקר לפני השימוש).
- Centrifuge (ב 4,000 x g למשך 5 דקות ב 4 ° C) התאים ולמחוק את תגובת שיקוע. להוסיף 200 מיליליטר מים יונים prechilled. Resuspend התאים באמצעות פיפטה סרולוגית 10 מ. חזור על שלב זה, 3 x.
- לשטוף את התאים עם 50 מ של prechilled 10% הפתרון גליצרול. Centrifuge אותם כמו קודם (ב 4000 x g למשך 5 דקות ב 4 ° C).
- למחוק את תגובת שיקוע, resuspend בגדר ב- 300 μL של 10% הפתרון גליצרול. 50 μL aliquots ולבצע להקפיא אותם בחנקן נוזלי. לאחסן את התאים (תאים ההקרנה-צלף) ב- 80 ° c
-
צלף-הקרנה
- להפוך את התאים צלף-הקרנה (מתוך שלב 3.2.10) עם 100 ננוגרם של פלסמידים variant Cas9 של כל ספריה (מתוך שלב 2.2.3.See שלבים 2.1.1 ו 2.1.4). בצע את השלבים אלקטרופורציה שמתואר שלבים 3.2.1–3.2.3.
- העברה 250 μL של התאים צינור microcentrifuge mL 1.5 טריים. הוסף עמוד 250 של ATC כדי להפוך ריכוז סופי של 10 ng/mL. שחזור תאים המכילים ATC והן ATC-חופשית (ראה שלב 3.2.4 לשלב השחזור).
- צלחת μL 25 התאושש תאים ללא ATC על צלחת אגר כלורמפניקול/kanamycin ליברות (מצב nonselective). להוסיף ATC התאים המכילים ATC התאושש כדי להפוך ריכוז סופי של 100 ננוגרם למ"ל על צלחת LB 245 מ מ. צלחת מיד את התאים על צלחת אגר LB כלורמפניקול/kanamycin/אראבינוז (תנאי סלקטיבי). דגירה בין לילה-32 מעלות צלזיוס.
הערה: הגודל ומספר הצלחת ליברות נקבעים לפי גודל המגוון של מכסה ההקרנה. במקרה 100 מ מ צלחת פטרי עם 20 מ ל LB, להוסיף 2 μg של ATC. - לצלם את הצלחות. לספור את מספר מושבות בת קיימא באמצעות תוכנת OpenCFU14. (ראה שלב 2.2.1) ודא כי המספר של המושבות בצלחת nonselective הוא לפחות 10 x גדול יותר המגוון של הספריה כדי לכסות את כל הווריאציות.
- לחשב את תדירות הישרדות כדלקמן.
הישרדות תדירות = מספר מושבות על צלחת סלקטיבית / (מספר מושבות על צלחת nonselective x 10) - בריכת המושבות ששרדו על לוחות סלקטיבי של כל שלוש הספריות. דגירה המושבות ששרד ב 250 מ של מדיום LB בתוספת 12.5 כלורמפניקול μg/mL ב 42 מעלות צלזיוס למשך הלילה. לבודד את הספריה Cas9 ממוסך DNA בעזרת ערכת midiprep.
הערה: שלב זה מנקה את פלסמיד sgRNA. - חזור על תהליך המיון של צעדים 3.3.1–3.3.6 עד התדר הישרדות מגיע מישור. שימוש 10 ng של פלסמיד Cas9 שנבחר לשינוי 10 ng/mL של ATC במהלך ההתאוששות. לשמור על ריכוז ATC-100 ננוגרם למ"ל עבור התנאי סלקטיבי.
-
ההקרנה השנייה ותעשה
- דשדוש על גרסאות במאגר שנבחר באמצעות פרוטוקול הבאה תגיע לעיקר-DNA. PCR להגביר את Cas9 להוסיף פלסמיד Cas9 באמצעות תחל איגוף נוקלאוטידים 150 מ לגבולות הוספה. 2 תקציר μg של מוצר ה-PCR מוגבר עם DNase אני עבור 1 דקות ב- 37 מעלות צלזיוס.
- לטהר bp שברי 200-70 אורך באמצעות 2% agarose בג'ל. PCR להגביר את השברים מטוהרים. שימוש במוצר כתבנית ל- PCR להגביר את תותב Cas9 עם תחל המתאים איגוף Cas9. השתמש המוצר הסופי PCR לבנות ספרייה Cas9 כפי שמתואר בשלב 2.1.4.
- להכין תאים צלף-הקרנה חדשים (ראו סעיף 3.2) עם פלסמיד sgRNA לא תואמת אחרת (ראה שלב 3.1.1). בצע שוב את תהליך המיון (סעיפים 3.2 – 3.3) עד מגיע שיעור הישרדות מישור. שימוש 10 ng של פלסמיד Cas9 שנבחר לשינוי 10 ננוגרם למ"ל ATC במהלך ההתאוששות. לשמור על ריכוז ATC-10 ng/mL עבור התנאי סלקטיבי.
-
מבחר של פלסמידים מוטציה מפותחת של Cas9
- לאחר השלב ההקרנה האחרונה, באופן אקראי לאסוף 100 מושבות מהצלחת סלקטיבית, תרבות אותם כלורמפניקול המכילות בינוני LB-42 ° C בלילה.
- לבודד את פלסמידים באמצעות הליך miniprep סנגר-רצף התוספות בעזרת רצף תחל בתוך Cas9.
- בחר שלושה המשתנים בתדירות הגבוהה ביותר כדי לבדוק אותם בשורות תאים אנושיים.
4. משלוח של Cas9 כמו RNP עם sgRNA קטום
-
בחירת גן היעד באמצעות רשויות אישורים-OFFinder
- לבחור אתרי היעד עם Cas-OFFinder (http://www.rgenome.net/cas-offinder/). בחר את סוג פאם המתאימים עבור סוג מסוים של Cas9 ואת הגנום היעד (אנושי, עכבר, דג זברה, וכו.). מילוי בכרטיסיה רצפים של שאילתה , לבחור את אי התאמה מספרולחץ על הלחצן Submit .
- לאחר כמה שניות, על המטרה (עם מספר אי התאמה של '0') ורצון את המטרה אתרים מופיעים. באופן כללי, בחר את האתרים את המטרה עם אחד עד שלושה הבדלים.
-
הכנת התבנית sgRNA
- סדר oligos crRNA ו- tracrRNA עם רצפי תבנית הבאים, כלומר crRNA רצף: 5'-TAATACGACTCACTATAGGNNNNNNNNNNNNNNNNNNNNGTTTTAGAGCTAGAA-3'; רצף tracrRNA: 5'-AGCACCGACTCGGTGCCACTTTTTCAAGTTGATAACGGACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC-3'. מלאו הרצף crRNA עם הרצף היעד שהושג בשלב 4.1.2 N20. כדי לעצב sgRNAs קטום, להסיר בסיסים מקצה 5' כדי לקבל רצפים sgRNA N19, N18 או N17.
- להכין לתערובת PCR הגברה של רצף קידוד sgRNA כדלקמן: לשלב 10 μL של 5 x מאגר, 0.5 μL של crRNA oligo (100 pmol/μL), μL 0.5 tracrRNA oligo (100 pmol/μL), μL 2.5 של dNTPs (10 מ מ כל אחד), 0.5 μL של ה-DNA פולימראז , וμl 36 של בא ל (טבלה של חומרים).
- להגביר את התבנית באמצעות התנאים הבאים, כלומר, עבור דנטורציה הראשונית: 1 דקות ב 98 ° C; עבור דנטורציה, חישול, סיומת: 10 s ב 98 ° C, 15 s ב 54 ° C, 20 s ב 72 מעלות צלזיוס, למשך 25 מחזורים; עבור הסיומת הסופי: 5 דקות-72 מעלות צלזיוס.
- לנתח את 2 μL של התבנית מוגבר דנ א (מתוך שלב 4.2.3) על 2% agarose ג'ל. לטהר את התבנית באמצעות ערכת טיהור PCR.
הערה: הגודל של תבנית ה-DNA הוא 125 bp.
-
סינתזה של sgRNA
- להכין לתערובת התגובה סינתזה sgRNA כדלקמן: לשלב μL 8.5 של תבנית ה-DNA (מתוך שלב 4.2.3), 1 μL של זוג שזור (25 מ מ), μL 1 של CTP (25 מ מ), μL 1 של GTP (25 מ מ), μL 1 של ATP (25 מ מ), μL 4.2 של MgCl2 (100 מ מ) , ΜL 4.5 של T7 RNA פולימראז (U 50/μL), μL 3 10 x T7 RNA פולימראז מאגר, 1.2 μL של pyrophosphatase (0.5 U μL), μL 0.75 של RNase מעכב (40 U μL) וμl 4.2 של בא ל.
- דגירה תערובת התגובה ב 37 ° C בלילה (לפחות עבור 10 h). להוסיף 0.5 μL של DNase (2 U μL) לתערובת התגובה דגירה ב 37 מעלות צלזיוס במשך 15-30 דקות Purify sgRNA באמצעות ערכת טיהור RNA.
- כדי להפחית רעילות הנגרמת על ידי תגובה חיסונית מולדת המופעלות על-ידי 5'-טריפוספט על ה sgRNA16, להסיר את 5'-טריפוספט המדריך RNAs עם עגל phosphatase אלקליין מעיים (CIP) כדלקמן: לטפל µg 10 של חוץ גופית ב- RNA משועתקים עם 250 U של CIP עבור h 3 ב 37 מעלות צלזיוס בנוכחות מעכב U של RNase 100. לטהר את sgRNA CIP שטופלו באמצעות ערכת טיהור RNA.
5. ביטוי חלבון Cas9 WT צלף וטיהור
-
ביטוי חלבון ב e. coli
- להפוך את חיית המחמד פלסמידים18 הקידוד שלו מתויג WT - ואני צלף-Cas9 לתוך BL21 המתח (DE3) e. coli .
- לחסן 50 מ ל LB בינוני המכיל 50 kanamycin μg/mL עם מושבה טריים מסתירים את חיית המחמד-Cas9 ביטוי פלסמיד ו- shake זה בין לילה (200 סל ד) ב 37 מעלות צלזיוס (preculture).
- העברת 10 מ"ל של תרבות לילה עד 500 מ"ל של מדיום LB טריים המכילים 50 kanamycin μg/mL. דגירה התרבות ברעידות (200 סל ד) ב 37 מעלות צלזיוס במשך שעתיים.
- נטר את יתר600 עד התרבות מגיע שלב אמצע יומן של צמיחה (OD600 ≈ 0.6-0.7).
- זירוז הביטוי של החלבון Cas9 WT או צלף עם איזופרופיל β-D-1-thiogalactopyranoside (IPTG, על ריכוז סופי של 0.25 ננומטר). דגירה התרבות ב 18 מעלות צלזיוס למשך הלילה.
-
חלבון טיהור
- לקצור את התאים על ידי צנטריפוגה ב x 5,000 g 10 דקות ב 4 º C.
- Resuspend בגדר במאגר של פירוק (50 מ מ NaH2PO4, 300 מ"מ NaCl, imidazole 10 מ מ, 4 מ מ dithiothreitol [DTT], benzamidine 5 מ מ, 100 מ מ phenylmethylsulfonyl פלואוריד [PMSF], pH 8) ב- 20 מ לכל משקל רטוב גרם.
- להוסיף PMSF DTT, ליזוזים ריכוז סופי של 1 מ"ג/מ"ל כל, דגירה התערובת בקירור למשך 30 דקות.
- Sonicate התאים על קרח. . הדופק שוב ושוב במשך 10 s ב 200 – 300 W עם 10 s קירור בתקופה שבין לכל פעימה, במשך הזמן הכולל של 20 דקות.
- Centrifuge את lysate ב x 6,000 g למשך 30 דקות ב 4 º C.
- הסר את תגובת שיקוע לרכבת התחתית טריים. להוסיף 1 מ"ל של שרף agarose שלו-Bind 5 מ של פינו lysate וללחוץ בעדינות על h 1-4 מעלות צלזיוס.
- לטעון את התערובת שרף Agarose lysate/שלו-Bind על עמודה עם שקע רצ'ט התחתון.
- הסירי את הפקק התחתון ולאסוף הזרימה עמודה של דרך.
- לשטוף את העמודה 2 x עם שטיפת מאגר (50 מ מ NaH2PO4, 300 מ"מ NaCl, 20 מ מ imidazole, pH 8); לאסוף את השבר שטיפת לניתוח על ידי נתרן dodecyl סולפט-לזיהוי בג'ל (מרחביות-עמוד).
- Elute החלבון 10 x עם 1 מ"ל של • תנאי מאגר (50 מ"מ. NaH2PO4, 300 מ"מ NaCl, 250 מ מ imidazole, pH 8), איסוף דגימות מרחביות-דף.
- לרכז את החלבון eluted WT - או צלף-Cas9 באמצעות מסנן עמודה kDa 100. לאחסן את הדגימות בפתרון של גליצרול טריס-HCl, 150 מ מ NaCl ו- 50% 10 מ מ ב- 80 ° c
6. RNP משלוח
-
תרביות תאים והכנה של תאים למסירה RNP
- לשמור על תאים HEK293T של Dulbecco שונה בינוני של הנשר (DMEM) בתוספת 10% סרום שור עוברית (FBS) ואנטיביוטיקה 1% ב 37 ° C עם 5% CO2.
- לערבב חלבונים WT - או צלף-Cas9 (2 μg) עם sgRNA (2 μg), תקופת דגירה של 10 דקות ב RT כדי להפוך RNP מתחמי.
- Trypsinize ולספור את התאים. להכין 2 x 10 תאים4 לכל תגובה אחת. לשטוף את התאים עם באגירה פוספט תמיסת מלח (PBS), צנטריפוגה. וארוקן את תגובת שיקוע, resuspend בגדר עם מאגר אלקטרופורציה.
- מתחמי Electroporate RNP לתוך התאים באמצעות ההגדרות הבאות, דהיינו 1,300 V, 30 ms, ואחד דופק. צלחת התאים על צלחת 48-ובכן מלא 500 μL של DMEM בתוספת FBS ואנטיביוטיקה (כפי שמתואר בשלב 6.1.1) מיד אחרי אלקטרופורציה. דגירה ב 37 ° C עם 5% CO2.
- לבודד את ה-DNA גנומי עם ערכת הכנה gDNA, 48 שעות לאחר תרביות תאים.
7. תרביות תאים של פלסמידים קידוד צלף-Cas9 ו sgRNA
-
בניית פלסמיד sgRNA
- סדר קדימה ולהפוך oligos עם רצפי תבנית הבאים, כלומר קדימה: 5'-CACCGNNNNNNNNNNNNNNNNNNNN-3'; הפוך: 5'-AAACNNNNNNNNNNNNNNNNNNNNC-3'. החלף רצף היעד שהושג בשלב 4.1.2 N20. לקצר את רצף המטרה לאורך של N19, N18 או N17 לסנתז sgRNA קטום.
- Anneal שתי oligos 1 x T4 DNA ליגאז מאגר.
- לעכל את הווקטור pRG2 עם אנזים הגבלה BsaI.
- ג'ל לטהר את הווקטור מעוכל (3,300 bp) באמצעות ג'ל agarose 0.8%.
- מאתרים ומפסיקים את oligo annealed, השבר מטוהרים באמצעות ליגאז T4-37 ° c: מערבבים 50 ng של pRG2 מתעכל ו- 1 ng של oligo annealed באמצעי אחסון התגובה של 20 μL. דגירה-RT למשך 15 דקות.
- להפוך את התערובת מצדו המתח אלפא DH5 ולגדול transformants על פלטות אגר LB המכיל אמפיצילין (100 μg/mL)-37 מעלות צלזיוס. לאשר החדרת oligo ב וקטור על ידי רצף רגיל.
-
תרביות תאים של פלסמידים קידוד צלף-Cas9 ו sgRNA
- לשמור על תאים HEK293T DMEM בתוספת 10% FBS ו 1% אנטיביוטיקה ב 37 ° C עם 5% CO2. יום לפני תרביות תאים, trypsinize, לספור את התאים. כאשר עובד בקנה מידה 48-. ובכן, הצלחת עונה 1 פרק 105 תאים לכל באר μL 250 של מדיום הגידול מלאה. התאים צריך להיות 50%-80% confluent ביום של תרביות תאים.
- את המשקל 48-ובכן, להכין 250 ng של פלסמיד p3s-Cas9, 250 ננוגרם של פלסמיד sgRNA עבור תרביות תאים, באמצעות ריאגנט ליפיד המבוסס על תרביות תאים. מערבבים את פלסמידים ב 25 μL של סרום חינם-מ.
- למהול 1 μL של תרביות תאים מגיב עם 25 μL של סרום חינם-מ. דגירה התערובת ב RT עבור 5 דק לשלב את שתי תערובות, דגירה הפתרון שיתקבל ב RT כעשרים דקות ל קומפלקסי פלסמיד-lipofectamine טופס.
- לאחר 20 דקות של דגירה, להוסיף 50 μL פתרון להכיל מתחמי ריאגנט פלסמיד-תקנים ישירות לכל אחד טוב תאים המכילים ומערבבים בעדינות במוזיקת את הצלחת הלוך ושוב. דגירה התאים ב- 37 מעלות צלזיוס חממה2 CO עבור תרביות תאים פוסט 48-72 שעות לפני assaying ביטוי transgene.
8. חישוב של תדרים ויאסין כדי לקבוע על-יעד ופעילות מחוץ-יעד
-
ממוקד רצף עמוק לניתוח של-יעד פוטנציאליים את המטרה ואתרים
- לבודד דנ א גנומי מהשלב 6.1.5 או 7.2.4 עם ערכת הכנה gDNA. ליצור רצף עמוק ספריות על ידי PCR הגברה של gDNA עם צבעי בסיס פילוח על-יעד, את המטרה.
- השתמש אינדקס תחל לתייג כל דגימה. ספריות במאגר הנושא כדי לזווג-קצה רצף בעזרת מכונה הדור הבא רצפי.
-
ניתוח רצף עמוק באמצעות רשויות אישורים-מנתח
- ניתוח נתונים רצף עמוק באמצעות כלי הערכה17Cas-מנתח.
- בחר את הקבצים Fastq מתחת ללשוניות קריאה 1 ו- 2 לקריאה (קריאה 1 = XX_SXX_L001_R1_001.fastq, קריאה 2 = XX_SXX_L001_R2_001.fastq).
- למלא את הכרטיסיה מידע בסיסי ולחץ על הכרטיסיה ניתוח הפרמטרים על הלחצן Submit .
תוצאות
אחרי מסך צלף מבוצע, האחוז של הישרדות מושבות יכול להיות מחושב על ידי חלוקת מספר מושבות בצלחת LB המכיל כלורמפניקול, kanamycin, אראבינוז ו- ATC (CKAA) על ידי מספר מושבות המכיל צלחת ליברות כלורמפניקול, kanamycin בלבד (CK). אחוז זה היה בדרך כלל נמוך מאוד, כאשר מסך צלף בוצעה עם הספריות של SpCas9. True-חיוביות להיטים יכול להעשיר חוזרת למסך עם הבריכה ששרדו. הצלף זה נציג-מסך, לדוגמה, שיעור הישרדות 100% הושג לאחר המסך השלישי (איור 1). Transfections באמצעות RNPs או פלסמיד מקודד צלף-Cas9 יכול להיעשות עבור מטרות שונות, וכתוצאה מכך בהמטרה, פעילויות את המטרה נמדדת ממוקד אמפליקון רצף (איור 2). לכל היותר מטרות, צלף-Cas9 מראה באותה הרמה של פעילויות--יעד, יחסי ירידה לפרטים גבוה יותר בהשוואה ל- sgRNAs WT. Truncated יכול לשמש גם כדי לשפר את ירידה לפרטים (איור 3). אולם, השימוש שלהם הוא מוגבל רק כמה מטרות כי הם תוצאה של פעילויות ביעד נמוך בהשוואה sgRNAs באורך מלא, ברוב המקרים. לכן, sgRNAs באורכים משתנים (מ- 17 - ל 20-מרס) חייב להיבדק, פעילויות ביעד והן את המטרה חייב להימדד למטב ירידה לפרטים.

איור 1: נציג צלף-מסכי הופיעה עם ספריות שונות של משתנים אקראיים Cas9. הנתונים ספריה, ספריה EP, ספריית EP השני לציין ספריות שנעשו באמצעות קיטים מסחריים שונים. הראשון, השני והאחרון מציינים מספר פעמים שהמסך העשרה היה תרגיל7. איור זה השתנה לי et al.7. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
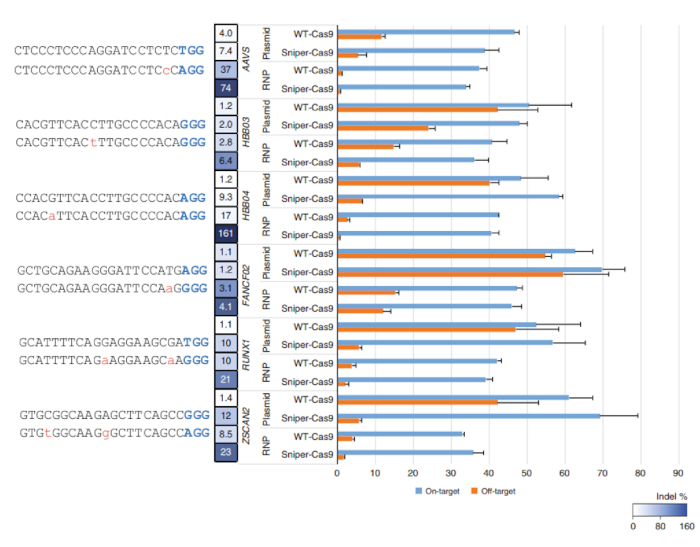
איור 2: ביעד ויעד חופש פעילות של WT-Cas9 או Cas9-צלף לזווג עם רצף מדריך 20-מר מועברת באמצעות פלסמיד או RNP. ירידה לפרטים יחסי נקבעו על-ידי חלוקת הפעילות על המטרה על ידי הפעילות את המטרה. קווי שגיאה לציין SEM (n = 3)7. איור זה השתנה לי et al.7. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
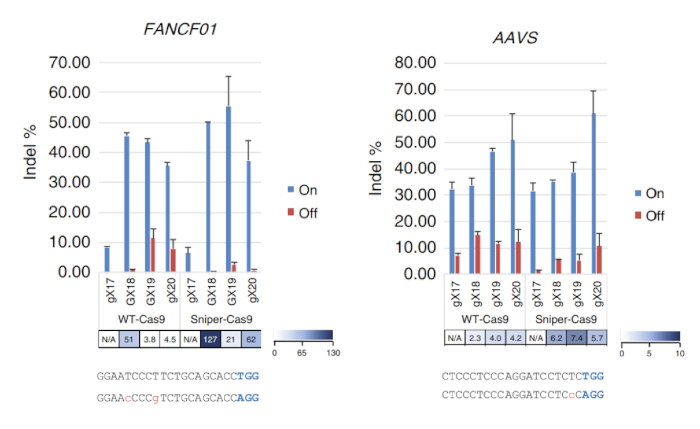
איור 3: על המטרה, פעילויות של צלף-Cas9 לעומת WT-Cas9 תוך שימוש sgRNAs באורכים משתנים מיקוד האתרים FANCF01, AAVS את המטרה. ירידה לפרטים יחסי נקבעו על-ידי חלוקת תדרים ויאסין באתרים-המטרה על ידי אלה באתרים מחוץ-יעד בהתאמה. sgRNAs עם גואנין מתאימים בקצהו 5' (GX18 או GX19) ואלה עם גואנין תואמים (gX17, gX18, gX19 או gX20) מסומנים. ירידה לפרטים יחסי לא חושבו כשהיית הפעילויות ביעד מנורמל < 70%7. איור זה השתנה לי et al.7. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
Discussion
לאלו שרוצים למנוע הליכי הסינון מסורבלת להשיג צלף-Cas9, החלבון צלף-Cas9 את פלסמיד קידוד זמינים. באמצעות חומרים אלה, האורך האופטימלי של sgRNA, כי מתן היחס הגבוה ביותר ירידה לפרטים, צריך להיות נחוש. בנוסף, משלוח של צלף-Cas9 ו- sgRNA בתבנית RNP מומלץ כי התוצאה היא בדרך כלל על יחס ירידה לפרטים גבוה יותר מאשר משלוח בתבנית פלסמיד. שלא כמו צלף-Cas9, גרסאות אחרות-Cas9 הנדסה אינם תואמים6,sgRNAs קטום7 או משלוח טופס RNP8 (למעט HiFi-Cas9).
עבור מסך צלף, הבחירה של הרצף תואמים הוא הצעד החשוב ביותר. יש להימנע מבחר sgRNA תואמים המשויך מחשוף נמוך פעילות לקראת הרצף גוי . אם לא, Cas9 גרסאות עם רמה WT היחודיות לא קליב ה-DNA גנומי עם אתר היעד תואמים e. coli . אפקט זה יביא מספר גדול של מושבות רקע, מסכנת את כל הקרנות בהליך.
כי החיידק יש מהיר הכפלת זמן ויעילות טרנספורמציה גבוהה לעומת שמרים, צלף מסך יתרון לעומת שיטות סינון שמבוסס שמרים. בנוסף, צלף מסך צריך להיות רגיש יותר מאשר אחרים, e. coli-שבו מתבצע באתר לא תואמת מערכות מבוססות על פלסמיד: יש עותק אחד של ה-DNA גנומי ובכך רק העתקה של האתר לא תואמת במערכת שלנו, אלא מספר גדול של פלסמידים יחיד בתוך תא החיידק .
Specificities של אחרים endonucleases DNA זה לגרום DSBs, כגון SaCas9 או Cpf1s, יכול להיות גם שיפור באמצעות מסך צלף. למרבה הצער, צלף מסך לא ניתן להשתמש כדי להגדיל את ייחודה של העורכים הבסיס ישירות, כי הבסיס עורכים אל תגרום / DSBs ב- DNA גנומי של e. coli. כפי עורכים הבסיס להשתמש nickase או מת גירסת Cas9 הליבה של המערכת שלהם, specificities של עורכים הבסיס יכול להיות מוגברת על-ידי שימוש הלהיטים המתקבל מסך צלף.
Disclosures
ToolGen הגיש בקשה לרישום פטנט (PCT/KR2017/006212) מכסה מסך צלפים (מצב: תלוי ועומד, ממציא: לי ק' Jungjoon). Jungjoon ק. לי, לי Joonsun, יונג Minhee ו- Euihwan ג'אונג עובדים של ToolGen, inc.
Acknowledgements
מחקר זה נתמך על ידי מענקים של משרד המדע ו- ICT של קוריאה (2017M3A9B4061406) את נבחרת מחקר קרן של קוריאה (NRF) ממומן על ידי ממשלת קוריאה (MSIT) (מענק מספרים 2017M3A9B4061404 ו- 2018M3A9H3020844) כדי Jungjoon ק' . לי. פלסמיד קידוד pGRG36 הייתה מתנת ננסי קרייג (Addgene פלסמיד #16666) והיה p11-תחרה-wtx1 מתנה מ- Huimin זאו.
Materials
| Name | Company | Catalog Number | Comments |
| Alkaline Phosphatase, Calf Intestinal (CIP) | NEB | M0290L | |
| Ampicillin Sodium Salt | Fisher Chemical | BP1760-25 | |
| Anhydrotetracycline hydrochloride | Sigma Aldrich | 37919 or 94664 | |
| Antibiotic Antimycotic solution | Welgene | LS203-01 | Media component |
| BamHI | Enzynomics | R003L | |
| Chloramphenicol | Sigma Aldrich | R4408 | |
| Diversify PCR Random Mutagenesis | Clontech | 630703 | Error-prone PCR kit |
| DNA-Shuffling Kit | Jena Bioscience | PP-103 | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Welgene | LM001-17 | Culture media |
| Endura Electrocompetent Cells (DUO) | Lucigen | 60242-2 | E. coli electrocompetent cells for library preparation |
| Fetal Bovine Serum (FBS) | Welgene | S101-01 | Media component |
| Gene Pulser II | Bio-Rad | E. coli electroporation equipment | |
| GeneMorph II Random Mutagenesis Kit | Agilent | 200550 | Error-prone PCR kit |
| genomic DNA prep kit | GenAll | 106-152 | gDNA-prep kit |
| Glycerol | BioShop | GLY001.1 | |
| GP/MP Cuvette, 0.1cm | Bio-Rad | BR165-2089 | E. coli electroporation equipment |
| HEK293T cell | ATCC | CRL-11268 | Human cell line |
| Kanamycin sulfate | Acros Organics | 450810500 | |
| L-(+)-Arabinose | Sigma Aldrich | A3256-25G | |
| LaboPass PCR Purification Kit | Cosmogenetech | CMR0112 | |
| LaboPass Plasmid Mini | Cosmogenetech | CMP0112 | Mini-prep kit |
| LB Agar Miller | Formedium | LMM0202 | |
| LB Broth Miller | Formedium | LMM0102 | |
| Lipofectamine | Invitrogen | 11668019 | Transfection reagent |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Gibson assembly (isothermal in vitro recombination) mixture |
| Neon Transfection System | Thermo | MPK5000 | RNP Electroporation equipment |
| Neon Transfection System 10 µL Kit | Thermo | MPK1096 | RNP Electroporation equipment |
| NucleoBond Xtra Midi EF | Macherey-Nagel | 740420.50 | Midi-prep kit |
| Oligo Clean & Concentrator | Zymo Research | D4061 | DNA purification kit that enables low-volume elution |
| Opti-MEM | Gibco | LS31985070 | Transfection media |
| p11-lacY-wtx1 | Addgene | #123945 | |
| pgrg36 | Addgene | #16666 | E. coli mutator strain |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Pyrophophatase | NEB | M2403L | |
| Ribonucleotide Solution Set | NEB | N0450L | |
| RNase inhibitor | NEB | M0314L | |
| T7 RNA polymerase | NEB | M0251L | |
| Turbo Dnase | Ambion | AM2238 | |
| XbaI | Enzynomics | R013L | |
| XL1-Red Competent Cells | Agilent | 200129 |
References
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nature Biotechnology. 32 (3), 279-284 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Research. 24 (6), 1012-1019 (2014).
- Chen, J. S., et al. Enhanced proofreading governs CRISPR-Cas9 targeting accuracy. Nature. 550 (7676), 407-410 (2017).
- Kleinstiver, B. P., et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature. 529 (7587), 490-495 (2016).
- Slaymaker, I. M., et al. Rationally engineered Cas9 nucleases with improved specificity. Science. 351 (6268), 84-88 (2016).
- Casini, A., et al. A highly specific SpCas9 variant is identified by in vivo screening in yeast. Nature Biotechnology. 36 (3), 265-271 (2018).
- Lee, J. K., et al. Directed evolution of CRISPR-Cas9 to increase its specificity. Nature Communications. 9 (1), 3048(2018).
- Vakulskas, C. A., et al. A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nature Medicine. 24 (8), 1216-1224 (2018).
- Anderson, K. R., et al. CRISPR off-target analysis in genetically engineered rats and mice. Nature Methods. 15, 512-514 (2018).
- Kim, S., Bae, T., Hwang, J., Kim, J. S. Rescue of high-specificity Cas9 variants using sgRNAs with matched 5' nucleotides. Genome Biology. 18 (1), 218(2017).
- Kulcsar, P. I., et al. Crossing enhanced and high fidelity SpCas9 nucleases to optimize specificity and cleavage. Genome Biology. 18 (1), 190(2017).
- Zhang, D., et al. Perfectly matched 20-nucleotide guide RNA sequences enable robust genome editing using high-fidelity SpCas9 nucleases. Genome Biology. 18 (1), 191(2017).
- McKenzie, G. J., Craig, N. L. Fast, easy and efficient: site-specific insertion of transgenes into enterobacterial chromosomes using Tn7 without need for selection of the insertion event. BMC Microbiology. 6, 39(2006).
- Geissmann, Q. OpenCFU, a new free and open-source software to count cell colonies and other circular objects. PLoS One. 8 (2), e54072(2013).
- Chen, Z., Zhao, H. A highly sensitive selection method for directed evolution of homing endonucleases. Nucleic Acids Research. 33 (18), e154(2005).
- Kim, S., et al. CRISPR RNAs trigger innate immune responses in human cells. Genome Research. 28 (3), 367-373 (2018).
- Park, J., et al. Cas-Analyzer: an online tool for assessing genome editing results using NGS data. Bioinformatics. 33, 286-288 (2017).
- Studier, F. W., et al. Use of T7 RNA Polymerase to Direct Expression of Cloned Genes. Methods in Enzymology. 185, 60-89 (1990).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved