Method Article
스나이퍼-Cas9를 사용 하 여 대상에서 감독된 진화를 통해 대상에 활동의 손실 없이 CRISPR Cas9의 영향을 최소화 하
요약
여기, 우리 CRISPR-Cas9-대상 활동의 손실 없이 더 높은 특이성을 달성 하기 위해 최적화 하는 프로토콜을 제시. 우리는 원하는 특성을 가진 돌연변이 Cas9를 찾을 수 라고 스나이퍼-화면 감독된 진화 접근을 사용 합니다. 스나이퍼-Cas9와 잘린된 단일 가이드 RNAs는 ribonucleoprotein 형태로 배달 높은 특이성을 달성 하기 위한 잘 알려진 전략 이다.
초록
클러스터 된 정기적으로 interspaced 짧은 구조 반복 (CRISPR)의 개발-관련된 단백질 9 (Cas9) 치료 modalities에 요구는 잠재적으로 해로운 오프 대상 효과의 회피. 이러한 효과 줄이기 위해 여러 가지 방법은 고안 되었습니다. 여기, 우리는 대장균을 제시-기반의 지시 진화 방법 스나이퍼-화면으로 최적화 된 Cas9 variant를 불리고 스나이퍼-Cas9 라는 대상에 활동을 유지. 스나이퍼-화면을 사용 하 여, 긍정적이 고 부정적인 선택을 수행할 수 있습니다 동시에. 화면도 다른 단일 가이드 RNA (sgRNA) 시퀀스 진정한 긍정적인 안타에 대 한 풍부 하 게 반복할 수 있습니다. Cas9 변종 표현 하 CMV PltetO1 듀얼 모터를 사용 하 여 풀링된 라이브러리의 성능은 포유류 세포에 신속 하 게 확인할 수 있습니다. 또한 스나이퍼-Cas9의 특이성을 증가 하는 방법은 설명 합니다. 첫째, 잘린된 sgRNAs 사용 하 여 이전 Cas9 특이성을 증가 표시 되었습니다. 다른 조작된 Cas9s와 달리 스나이퍼 Cas9 잘린된 sgRNAs와 결합 될 때에 대상 활동의 야생-타입 (WT) 수준을 유지 합니다. 둘째, 플라스 미드 형식 대신 ribonucleoprotein (RNP) 형태로 스나이퍼-Cas9의 배달 대상에 활동을 영향을 주지 않고 가능 하다.
서문
이 문서에서 우리는 다른 전략을 결합 하 여 Cas9의 특이성을 개선 하고자 합니다. CRISPR-Cas9의 대상 오프 효과 피하고 다양 한 방법 들이 개발 되었습니다. 예를 들어 잘린된 sgRNAs 높은 특이성1을 달성 하기 위해 사용할 수 있습니다. 또한, Cas9 배달 방법 높은 특이성2를 RNP 형식 플라스 미드 형식에서 변경할 수 있습니다. 연쇄 상 구 균 pyogenes Cas9의 특정 아미노산 잔류물 (SpCas9) 단백질에 따라 합리적인 수정 된 디자인3,,45설명한. 또한, 아미노산 잔류물 무작위 방식으로 변경 되었습니다 및 높은 특이성으로 Cas9 변종 대장균7,8 시스템 심사 또는 효 모6 을 사용 하 여 발견 했다.
그러나, 많은 그룹 보고 Cas9 변형 수 Cas9와 기판 전시 낮은 대상에 활동7,,89, 사이 일반적인 상호 작용을 디자인을 사용 하 여 설계 10 , 11 , 12. 우리는 대장균을 개발-감독된 진화 시스템, 스나이퍼-화면, 임의로 mutagenized Cas9 이체 화면을 기반으로. 시스템 심사는 E. 콜라이 대장균의 빠른 배로 시간과 더 높은 변환 효율성 때문에 효 모 시스템 장점이 있습니다.
부정적이 고 긍정적인 선택, 3 개의 다른 플라스 미드와 관심 (GOI)의 유전자를 대장균 게놈으로 통합에 따라 스나이퍼-화면에 사용 됩니다. Cas9 변종 대장균 에서 확인 된 후보자 subcloning 필요 없이 포유류 세포에서 테스트할 수 있도록 낮은 복사 번호 플라스 미드의 CMV PltetO1 듀얼 모터 시스템 아래 표시 됩니다. GOI Tn7 transposon 시스템을 사용 하 여 대장균 게놈으로 소개 된다. SgRNA 플라스 미드 복제의 온도 민감한 출처를 포함 하는 sgRNA GOI;을 대상으로 표현 그러나, sgRNA 및 GOI 시퀀스는 완벽 하 게 일치 되지. 완벽 하 게 일치 sgRNA 대상 사이트는 치명적인 제품 gyrase 독극물을 인코딩하는 ccdB 유전자를 포함 하는 세 번째 플라스 미드에 존재 합니다. 이 시스템에서 높은 오프 대상 활동 Cas9 이체를 표현 하는 세포 두 배 물가 틈 (DSBs)는 genomic DNA에 있는 일치 하지 않는 사이트에 도입 하기 때문에 제거 됩니다. 다른 한편으로, Cas9 변종 낮은 대상에 활동을 표현 하는 세포도 치명적인 ccdB 유전자 발현 때문에 제거 됩니다. Cas9 변종 식 수준 선택 힘을 조정 하는 anhydrotetracycline (ATC)의 농도 변경 하 여 변경할 수 있습니다.
우리는 그 시스템의 감도 증가 시킬 것입니다 보다는 플라스 미드는 genomic DNA에 일치 하지 않는 sgRNA 대상 사이트를 찾는 것을 권유. 이 방법의 장점은 하나의 게놈 사이트는 반면 각각 단일 내에서 대상 사이트를 포함 하는 많은 플라스 미드는 대장균 세포.
이 시스템을 사용 하 여, 우리는 Cas9 변종, 스나이퍼-Cas9, WT-대상에 활동을 표시 하 고 대상 오프 활동 WT Cas9에 비해 감소 확인. 스나이퍼-Cas9 잘린된 sgRNAs 또는 RNP 기반 배달 보다는 배달 플라스 미드-기반을 사용 하 여 더 높은 특이성 비율을 달성할 수 있다.
프로토콜
1. BW25141 E. 콜라이 긴장으로 인간의 GOI 의 통합
-
GOI 의 복제
- 연쇄 반응 (PCR)의 노트 및 XhoI 금지 효소 사이트를 포함 한 뇌관으로 표준 PCR 방법을 통해 다양 한 후보 대상 사이트를 포함 하는 인간의 GOI 500 bp 길이 증폭.
참고:이 실험에서 GOI 인간 EMX1 유전자를 했다. - PCR 제품 및 pgrg3613 벡터 노트 및 XhoI 금지 효소로 소화.
- 젤 정화 원하는 조각 (500 bp, 12 킬로바이트).
- T4 리가 사용 하 여 함께 조각을 선합니다 이렇게 하려면 50 혼합 소화 pgrg36 및 6의 ng ng 20 μ 1 x 리가 버퍼 및 0.5 U 리가 효소의 반응 볼륨에서 PCR 삽입의. 하룻밤 실 온 (RT)에서 품 어.
- 합자 플라스 미드 DH5 α 유능한 세포 변환. Luria 국물 (파운드) 한 천 배지 포함 하는 암 피 실린 (100 μ g/mL) 32 ° C에 결과 transformants 성장 하 고 밤새 품 어. 식민지를 선택 하 고 하룻밤 암 피 실린을 포함 하는 파운드 미디어에서 성장. 플라스 미드는 대장균, 상용 miniprep 키트를 사용 하 여에서 DNA를 분리. 생어 시퀀싱 미니 플라스 미드 준비 (소형 예비)13에서 얻은 플라스 미드에 의해 GOI 파편의 삽입을 확인 합니다.
- 연쇄 반응 (PCR)의 노트 및 XhoI 금지 효소 사이트를 포함 한 뇌관으로 표준 PCR 방법을 통해 다양 한 후보 대상 사이트를 포함 하는 인간의 GOI 500 bp 길이 증폭.
-
BW25141-GOI 의 준비
- BW25141 E. 콜라이 긴장으로 얻은 pgrg36-GOI 플라스 미드를 변환. BW25141 긴장을 사용 하 여 거짓 긍정적인 식민지의 수를 최소화 하기 위해 필수적 이다.
- 밤새 32 ° C에서 파운드 버퍼에서 변형 된 세포를 성장. GOI BW25141 스트레인 (BW25141-GOI)의 게놈 DNA에 삽입 됩니다. 13의 표준 pgrg36 프로토콜 사용 하 여 BW25141-GOI 긴장에서 pgrg36-GOI 플라스 미드를 제거 합니다. 간단히, 식민지를 희석 (약 107-배) 하룻밤 42 ° C에서 파운드 접시에 성장 하 고. 파운드에 식민지를 행진 하 고 하룻밤 42 ° C에서 그들을 성장.
- 식민지 PCR, pgrg36 프로토콜에서 제안 하는 뇌관을 사용 하 여 올바른 GOI 삽입 확인: 5'-GATGCTGGTGGCGAAGCTGT-3 '와 5'-GATGACGGTTTGTCACATGGA-3'. 뇌관 genomic DNA 삽입 사이트 증폭 되며 결과 PCR 제품의 크기 904 혈압 및 삽입의 크기 (500이 경우에서 bp).
- Electrocompetent BW25141-GOI 셀 (자세한 프로토콜 단계 3.2.6–3.2.10에서 설명)을 준비 합니다.
2입니다. Cas9 변형 라이브러리 준비
-
라이브러리 준비
- Cas9 벡터7 변환 상업 대장균 에 변경자 (mutator) (자료 테이블)을 변형 하 고 변형 라이브러리 (Mutator 라이브러리)를 제조업체의 지침을 따르십시오.
- 오류가 PCR 키트 (테이블의 재료)를 사용 하 여 Cas9 벡터에 전체 WT Cas9 시퀀스에 오류가 PCR을 수행 합니다.
참고: 낮은 에러율 프로토콜 단백질의 원래 기능을 방해 하지 않으려면 스나이퍼 Cas9 경우에 채택 되었다. - Cas9 벡터 적절 한 제한 효소로 다이제스트. 젤 (2.1.2 단계)에서 PCR 제품 및 소화 백본 정화.
참고: SpCas9 유전자의 크기는 약 4.3 kb. XhoI 및 KpnI 스나이퍼 Cas9 경우에 사용 된 pBLC SpCas9 벡터를 소화 하기 위해 선정 됐다. - 조립 (2.1.3 단계)에서 백본 조각 및 증폭 등온선 생체 외에서 재결합을 통해 (2.1.3 단계)에서 오류가 PCR를 사용 하 여 삽입.
참고: 500 이상 등뼈의 ng 라이브러리 (오류가 PCR [EP])의 높은 농도 얻기 위해 필요 하다. 두 개의 다른 오류가 PCR 키트 스나이퍼 Cas9 경우에서 EP 라이브러리를 준비 하는 데 사용 했다 (EP 도서관 I 및 II). - (에서 단계 2.1.4) 어셈블리에서 제품을 정화 낮은 볼륨 차입 (자료 테이블)을 가능 하 게 DNA 정제 키트를 사용 하 여. Nuclease 무료 물 (있)의 6 μ와 elute 고 DNA의 농도 측정 한다.
- 500 개 이상의 변환 electrocompetent 대장균 세포 (자료 테이블)의 50 μ로 (각 3 라이브러리)에 대 한 Cas9 라이브러리 벡터의 ng. 단계 3.2.1-3.2.4에서 electroporation 프로토콜을 참조 하십시오. 이 라이브러리 준비를 위해 유능한 세포의 50 μ 당 250 μ 대신 SOC 매체의 1 mL을 사용 합니다.
- 확인 1: 100, 1:1, 000, 및 SOC 매체 복구 된 셀을 포함 하는 혼합물의 1:10,000 희석. 페니 (12.5 μ g/mL)와 보충 100 mm 파운드 한 천 배지 희석된 셀 접시 245 m m2 접시에 나머지 셀 접시 37 ° C에서 밤새 품 어.
-
라이브러리 복잡도의 계산
- 젤 문서 시스템이 나 일반 디지털 카메라를 통해 희석 접시 사진. OpenCFU 소프트웨어14 를 실행 하 고 희석 판의 사진을 업로드. 접시 안에 세 영역을 설정 하 고 잘못 된 식민지를 제거 합니다.
- 수동으로 transformants의 원래 수를 희석 요인에 의해 식민지의 수를 곱하면. 로그 (밑이 10)에이 숫자를 변환 합니다. 라이브러리의 복잡성을 확인 하기 위해 평균을 계산 합니다.
- 복잡 한 원하는 값을 얻을 때 모든 식민지 245 m m 사각 접시에 (에서 수집 단계 2.1.7) 스프레더 및 보충 페니 파운드의 20 mL를 사용 하 여. 모여 식민지 성장 하지 고 정화 상업 midiprep 키트를 사용 하 여 플라스 미드 도서관.
참고: 라이브러리 복잡 한 더 높은, 더 나은. 스나이퍼-Cas9 확인 되었다, 3 x 106 의 각 라이브러리에 대 한 달성 되었다.
3. 긍정적이 고 부정적인 Cas9 발전에 대 한 심사
-
대상 선택 및 플라스 미드 건설
- GOI에 대상 sgRNA 공백 시퀀스를 선택 합니다. 일치 하지 않는 시퀀스를 임의의 뉴클레오티드에 하나 또는 두 개의 잔류물을 대체 합니다.
참고: 인간 EMX1 대상 사이트 3 (GGG pam GAGTCCGAGCAGAAGAAGAA)은 스나이퍼 Cas9 경우에 사용 되었다. 다음은 일치 하지 않는 시퀀스 사용: GAGTCCGAGCAGAAagAGAA, GAacCCGAGCAGAAGAAGAA, GAGTCCGAGCAGAgGAAGAA, 및 GAGcCCGAGCAGAAGAAGAA. - 일치 하지 않는 시퀀스를 삽입 (단계 3.1.1 참조) 표준 oligonucleotide (oligo) 절차7복제를 사용 하 여 sgRNA 플라스 미드로.
- P11-레이스-wtx1 (테이블의 재료) 표준 올리고 복제 절차15를 사용 하 여 ccdB 플라스 미드를 만드는 데에 3' 끝에 팸 일치 하지 않는 시퀀스를 삽입 합니다.
- GOI에 대상 sgRNA 공백 시퀀스를 선택 합니다. 일치 하지 않는 시퀀스를 임의의 뉴클레오티드에 하나 또는 두 개의 잔류물을 대체 합니다.
-
스나이퍼-상영 대장균 유능한 세포 준비
- BW25141-GOI electrocompetent 셀 얼음에 녹여
- 1 각 ccdB 플라스 미드 및 해 동 BW25141-GOI 셀 50 μ로 더블 불일치와 sgRNA 플라스 미드의 ng. 부드럽게 pipetting으로 세포를 혼합 하 고 prechilled 0.1 cm electroporation 베트로 이동.
- Electroporation 통해 두 개의 플라스 미드를 가진 대장균 을 변환. Electroporation 직후 SOC 매체의 250 μ를 추가 합니다. 부드럽게 세포와 매체 혼합 솔루션 플라스틱. Microcentrifuge 1.5 mL 튜브에 혼합물을 전송.
참고: 최대 효율성을 위해 설정 전압 1.80에서 kV와 런타임 4.8 ms와 5.0 ms 사이 있어야 한다. - 변형 된 세포를 복구 하 고 그들을 부드러운 동요와 1 h 32 ° C에서 품 어.
- 암 피 실린 (50 μ g/mL)에 복구 된 셀의 125 μ 접시 / 대 (25 μ g/mL) 파운드 한 천 격판덮개 (문화 조건). 접시에 남은 세포는 암 피 실린/대/arabinose (1.5 mg/mL)에 파운드 한 천 배지 (ccdB-상태를 표현). 32 ° C에서 밤새 품 어.
- CcdB에 식민지를 살아 나 기의 부재에 대 한 확인-조건 플레이트를 표현. 식민지 문화 조건 플레이트 분산기와 슈퍼 최적의 국물 (흐 느낌) 매체 50 μ g/mL 암 피 실린 및 25 μ g/mL에서 32 ° C 대의 보충의 250 mL에 흔들어 부드러운 문화를 사용 하 여에서 수집 합니다.
- 때 600 nm (OD600)에 도달, 0.4에서 광학 밀도 얼음에 플라스 크를 진정. Prechilled 이온된 수와 prechilled 10% 글리세롤 솔루션 준비 (사용 하기 전에 소독).
- 셀 (4000 x g 4 ° C에서 5 분)에서 centrifuge 고는 상쾌한 삭제. Prechilled 이온된 물 200 mL를 추가 합니다. 10 mL 혈 청 학적인 피 펫을 사용 하 여 셀 resuspend 3 x이이 단계를 반복 합니다.
- Prechilled 10% 글리세롤 솔루션 50 mL로 세포 세척. 이전 (4 ° C에서 5 분 동안 4000 x g )에서 원심 그들.
- 삭제는 상쾌한 고 10% 글리세롤 용액 300 μ에 펠 릿을 resuspend. 50 μ aliquots 하 고 액체 질소에 그들을 동결. 셀 (스나이퍼-상영)-80 ° c.에 저장
-
스나이퍼-상영
- 100 (에서 단계 3.2.10) 스나이퍼-상영 셀 변환 (단계 2.2.3.See 단계 2.1.1 2.1.4)에서 각 라이브러리에서 Cas9 변형 플라스 미드의 ng. 단계 3.2.1–3.2.3에 설명 된 electroporation 단계를 따릅니다.
- Microcentrifuge 신선한 1.5 mL 튜브에 세포의 250 μ를 전송 합니다. 10 ng/mL의 최종 농도 확인 하는 ATC의 250 페이지를 추가 합니다. (단계 3.2.4 복구 단계 참조) 모두 포함 하는 ATC와 ATC 무료 세포를 복구.
- 페니/대 파운드 한 천 배지 (비선택적 조건)에 복구 된 ATC 무료 셀의 25 μ 플레이트. ATC 245 m m 파운드 접시에 100 ng/mL의 최종 농도 만들기 위해 복구 된 ATC 포함 된 셀에 추가 합니다. 즉시 페니/대/arabinose 파운드 한 천 배지 (선택 조건)에 셀 접시. 32 ° c.에서 밤새 품 어
참고: 크기와 파운드 플레이트 심사 커버의 다양성의 크기에 의해 결정 됩니다. 100 mm 페 트리 접시 파운드의 20 mL의 경우 ATC의 2 μ g을 추가 합니다. - 사진 판. OpenCFU 소프트웨어14를 사용 하 여 가능한 식민지의 수를 계산 합니다. (2.2.1 단계 참조) 비선택적 접시에 식민지의 수 적어도 10 배 모든 이체를 라이브러리의 다양성 보다 더 큰 있는지 확인 합니다.
- 생존 주파수를 다음과 같이 계산 합니다.
생존 주파수 선택적 접시에 식민지의 수 = / (비선택적 접시 x 10에 식민지의 수) - 모든 3 개의 라이브러리에서 선택적 접시에 살아남은 식민지 풀. 42 ° C에서 12.5 μ g/mL 페니 하룻밤 보충 파운드 매체의 250 mL에 살아남은 식민지를 품 어. 스크린된 Cas9 라이브러리 midiprep 키트를 사용 하 여 DNA를 분리.
참고:이 단계는 sgRNA 플라스 미드를 지웁니다. - 생존 주파수 대 지에 도달할 때까지 단계 3.3.1–3.3.6에서 심사 과정을 반복 합니다. 변환 및 10 ng/mL ATC의 복구 하는 동안 선택한 Cas9 플라스 미드의 사용 10 ng. 선택 조건에 대 한 100 ng/mL에서 ATC 농도 유지 합니다.
-
더미와 두 번째 심사
- 다음 DNA 더미 프로토콜을 사용 하 여 선택한 풀링된 변종 셔플. PCR 증폭에 측면에 서 뇌관, 삽입 경계에서 150 뉴클레오티드를 사용 하 여 Cas9 플라스 미드 Cas9 삽입. 증폭 된 PCR 제품 DNase의 다이제스트 2 μ g 37 ° c.에 1 분 동안 내가
- 길이 2 %agarose 젤 전기 이동 법을 사용 하 여 조각 70-200 bp를 정화. PCR 증폭 순화 된 조각. PCR 템플릿으로 사용 제품 Cas9 측면 적절 한 뇌관으로 Cas9 삽입을 증폭. 사용 하 여 최종 PCR 제품 2.1.4 단계에서 설명한 대로 Cas9 라이브러리를 생성.
- 또 다른 일치 하지 않는 sgRNA 플라스 미드와 새로운 스나이퍼-상영 셀 (섹션 3.2 참조)를 준비 (단계 3.1.1 참조). 고원에는 생존 율에 도달할 때까지 (섹션 3.2-3.3) 심사 과정을 다시 실행 합니다. 변환 및 복구 하는 동안 10 ng/mL ATC에 대 한 선택 된 Cas9 플라스 미드의 사용 10 ng. 선택 조건에 대 한 10 ng/mL에서 ATC 농도 유지 합니다.
-
진화 Cas9 돌연변이 플라스 미드의 선택
- 마지막 심사 단계 후 임의로 선택적 접시에서 한 백 식민지를 선택 하 고 하룻밤 그들 42 ° C에서 포함 하는 페니 파운드 매체에 문화.
- 격리를 사용 하 여 miniprep 절차 및 생어 순서 Cas9에서 시퀀싱 뇌관을 사용 하 여 삽입 하는 플라스 미드.
- 인간의 세포 라인에 그들을 테스트 하려면 상위 3 가장 빈번한 변형을 선택한 다.
4. 잘린된 sgRNA와 RNP로 Cas9의 납품
-
Ca-OFFinder를 사용 하 여 대상 유전자 선택
- Cas-OFFinder (http://www.rgenome.net/cas-offinder/)와 대상 사이트를 선택 합니다. Cas9 및 대상 게놈의 특정 유형에 대 한 적절 한 PAM 종류 선택 (인간, 마우스, zebrafish, 등.). 쿼리 시퀀스 탭, 선택 하는 번호 불일치를 제출 버튼을 클릭 합니다.
- 후 몇 초, ('0'의 불일치 번호)와 대상 및 대상 오프 사이트 지 나타납니다. 일반적으로 1 ~ 3 불일치와 오프 대상 사이트를 선택 합니다.
-
SgRNA 서식 파일의 준비
- 다음 서식 파일 시퀀스, 즉 crRNA 시퀀스와 함께 crRNA 및 tracrRNA oligos 주문: 5'-TAATACGACTCACTATAGGNNNNNNNNNNNNNNNNNNNNGTTTTAGAGCTAGAA-3'; tracrRNA 순서: 5'-AGCACCGACTCGGTGCCACTTTTTCAAGTTGATAACGGACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC-3'. 네 20 단계 4.1.2에서에서 얻은 대상 시퀀스와 crRNA 시퀀스를 입력 합니다. 잘린된 sgRNAs를 디자인 하려면 N19, N18, 또는 N17 sgRNA 시퀀스를 5' 끝에서 기지를 제거 합니다.
- 다음과 같이 sgRNA 인코딩 시퀀스의 확대를 위한 PCR 혼합물 준비: 버퍼 x 5 10 μ, crRNA 올리고 (100 pmol/μ)의 0.5 μ, tracrRNA 올리고 (100 pmol/μ), 2.5 μ (10 mM 각) dNTPs의 DNA 중 합 효소의 0.5 μ의 0.5 μ 결합 그리고 있 (자료 테이블)의 36 μ.
- 초기 변성에 대 한 다음 조건, 즉,를 사용 하 여 템플릿을 증폭: 98 ° C에서 1 분 변성, 어 닐 링, 및 확장에 대 한: 10 s 98 ° C, 15에서 54 ° C, 20에서 s s 25 사이클; 72 ° C에서 최종 연장: 72 ° c.에서 5 분
- 2 μ 2 %agarose 젤에 증폭 된 템플릿 (4.2.3 단계)에서 DNA의 분석. 정화는 PCR 정화 키트를 사용 하 여 템플릿을.
참고: 서식 파일 DNA의 크기는 125 bp.
-
SgRNA의 합성
- 다음과 같이 sgRNA 합성 반응 혼합물 준비: 8.5 μ (4.2.3 단계)에서 서식 파일 DNA의 결합 UTP (25 m m), ctp (25 m m), GTP (25 m m), ATP (25 m m), MgCl2 (100 mM)의 4.2 μ의 1 μ의 1 μ 1 μ의 1 μ T7 RNA 중 합 효소 (50 U/μ)의 4.5 μ, 10 x T7 RNA 중 합 효소 버퍼의 3 μ, pyrophosphatase (0.5 U/μ)의 1.2 μ, RNase 억제제 (40 U/μ), 0.75 μ 그리고 있의 4.2 μ.
- 37 ° C에서 반응 혼합물을 밤새 품 어 (적어도 10 h에 대 한). 추가 DNase의 0.5 μ (2 U/μ) 반응 혼합물에와 15-30 분 Purify는 RNA 정화 키트를 사용 하 여 sgRNA에 대 한 37 ° C에서 품 어.
- 5'-3 인산 염 sgRNA16에 의해 트리거되는 타고 난 면역 반응에 의해 발생 하는 독성을 줄이기 위해는 5'-3 인산 염 종 아리 장 알칼리 성 인산 가수분해 효소 (CIP)와 RNAs 가이드에서 다음과 같이 제거: 10 µ g 체 외-의 치료 CIP 100 U의 RNase 억제제의 37 ° C에서 3 h의 U 250 하는 베낀된 RNA CIP 처리 sgRNA RNA 정화 키트를 사용 하 여 정화.
5. WT 및 스나이퍼 Cas9 단백질 표정과 정화
-
대장균 에서 단백질 표정
- 애완 동물 플라스 미드18 그의 태그 WT-와 스나이퍼-Cas9는 BL21으로 인코딩 변환 (DE3) E. 콜라이 긴장.
- 애완 동물-Cas9 식 플라스 미드 및 그것은 하룻밤 사이 37 ° C (preculture)에서 (200 rpm) 쉐이크 은닉 신선한 식민지와 50 μ g/mL 대를 포함 하는 파운드 매체의 50 mL 접종
- 야간 문화 10 mL 500 mL 50 μ g/mL 대를 포함 하는 신선한 파운드 매체의 전송. 2 h 37 ° C에서 (200 rpm)를 떨고 함께 문화를 품 어.
- 문화권의 성장 (OD600 ≈ 0.6-0.7) 중간 로그 단계에 도달할 때까지 세600 을 모니터링 합니다.
- 이소프로필 β-D-1-thiogalactopyranoside로 WT 또는 스나이퍼 Cas9 단백질의 표현을 유도 (0.25의 최종 농도에 IPTG nM). 밤새 18 ° C에서 문화를 품 어.
-
단백질 정화
- 4 ° c.에서 10 분 동안 5000 x g 에서 원심 분리 하 여 세포를 수확
- 그램 젖은 무게 당 20 mL에 lysis 버퍼 (50mm NaH2포4, 300 mM NaCl, 10mm 이미, 4 mM dithiothreitol [DTT], 5 m m benzamidine, 100 mM phenylmethylsulfonyl 불 [PMSF], pH 8)에 펠 릿을 resuspend.
- 1 mg/mL 각의 최종 농도에 PMSF, DTT, 그리고 lysozyme를 추가 하 고 30 분 동안 얼음에 혼합물을 품 어.
- 얼음에 셀 sonicate 펄스 반복 10 200-300 W 10에 s s 20 분의 총 시간에 대 한 각 펄스 사이의 기간을 냉각.
- 4 ° c.에 30 분 동안 6000 x g 에서 lysate 원심
- 상쾌한 신선한 튜브를 제거 합니다. 그의 바인드 agarose 수 지의 5 ml의 1 mL lysate 삭제 추가 하 고 1 시간 4 ° c.에 대 한 부드럽게 흔들어
- 출장된 하단 콘센트와 함께 열에 lysate/그의-바인딩 Agarose 수 지 혼합물을 로드 합니다.
- 하단 뚜껑을 제거 하 고 열 흐름 통해 수집 합니다.
- 2 열을 씻어 x 워시 버퍼 (50mm NaH2포4, 300 mM NaCl, 이미 20 m m, pH 8); 나트륨 라우릴 황산 polyacrylamide 젤 전기 이동 법 (SDS 페이지)에 의해 분석을 위해 세척 분수를 수집 합니다.
- 10 단백질을 elute 차입의 1 mL x SDS 페이지 (50mm NaH2포4, 300 mM NaCl, 이미 250 m m, pH 8) 수집 샘플 버퍼.
- 100 kDa 열 필터를 사용 하 여 eluted WT 또는 스나이퍼 Cas9 단백질을 집중 한다. -80 ° c.에서 10 mM Tris HCl, 150 mM NaCl, 그리고 50% 글리세롤의 솔루션에는 샘플을 저장
6. RNP 배달
-
Transfection 그리고 RNP 배달에 대 한 셀의 준비
- Dulbecco의 HEK293T 셀 수정이 글의 중간 (DMEM) 10% 태아 둔감 한 혈 청 (FBS)와 5% CO2와 37 ° C에서 1% 항생제 보완 유지 합니다.
- SgRNA (2 μ g)로 WT 또는 스나이퍼 Cas9 단백질 (2 μ g)를 혼합 하 고 RNP 단지 수 있도록 RT에서 10 분 동안 품 어.
- Trypsinize 및 셀. 한 반응 당 2 × 104 셀을 준비 합니다. 인산 염 버퍼 식 염 수 (PBS) 및 원심 분리기 세포를 씻어. 발음은 상쾌한 고 electroporation 버퍼와 펠 릿을 resuspend.
- 다음 설정을 사용 하 여 셀에 Electroporate RNP 단지, 즉 1300 V, 30 ms, 그리고 펄스. 플레이트는 electroporation 직후 FBS와 항생제 (6.1.1 단계에서 설명) 하는 대로 보충 DMEM의 500 μ 가득한 48-잘 접시에 셀. 5% CO2와 37 ° C에서 품 어.
- GDNA 준비 키트, transfection는 후 48 h 게놈 DNA를 격리 합니다.
7. 인코딩 스나이퍼-Cas9 및 sgRNA는 플라스 미드의 transfection
-
SgRNA 플라스 미드의 건설
- 앞으로 주문 하 고 반대로 oligos 다음 서식 파일 시퀀스, 즉 앞으로: 5'-CACCGNNNNNNNNNNNNNNNNNNNN-3'; 역: 5'-AAACNNNNNNNNNNNNNNNNNNNNC-3'. 4.1.2 단계에서 얻은 대상 시퀀스에 N20을 바꿉니다. 대상 시퀀스 N19, N18, 또는 N17 잘린된 sgRNA 합성의 길이를 단축.
- T4 DNA 리가 버퍼 x 1에서 모두 oligos anneal
- PRG2 벡터 BsaI 제한 효소로 다이제스트.
- 젤 정화 소화 벡터 (3300 bp) 0.8 %agarose 젤을 사용 하 여.
- 단련된 올리고 및 소화 pRG2의 37 ° c: 믹스 50 ng과 1에서 T4 리가 사용 하 여 순화 된 단편 선 20 μ의 반응 볼륨에서 단련 된 oligo의 ng. 15 분 동안 RT에서 품 어.
- 결 찰 혼합물 DH5 알파 변형으로 변환 하 고 transformants 암 피 실린 (100 μ g/mL) 37 ° c.를 포함 하는 파운드 한 천 배지에서 성장 표준 연속 벡터에 올리고의 삽입을 확인 합니다.
-
스나이퍼-Cas9 및 sgRNA를 인코딩 하는 플라스 미드의 transfection
- HEK293T 셀 DMEM 5% CO2와 37 ° C에서 10% 1 %FBS 항생제로 보완을 유지 합니다. Transfection, 전날 trypsinize 및 셀. 때 완전 한 성장 매체의 250 μ에 잘 당 플레이트 1 x 105 셀 48-잘 규모에서 작동 합니다. 세포는 transfection 당일 50%-80% 합칠 이어야 한다.
- 48-잘 규모 준비 250 p3s Cas9 플라스 미드 및 250의 ng ng transfection, 지질에 기초를 둔 transfection 시 약을 사용 하 여에 대 한 sgRNA 플라스 미드의. 혈 청 무료 MEM의 25 μ에 플라스 미드를 믹스.
- 혈 청 무료 MEM의 25 μ로 1 μ transfection 시 약의 희석. 두 섞어 5 분 결합에 대 한 실시간에 혼합물을 품 어와 양식 플라스 미드 lipofectamine 단지 20 분에 대 한 실시간에 결과 솔루션을 품 어.
- 보육의 20 분 후 50 μ 플라스 미드 transfection 시 약 단지 직접 각 포함 된 셀을 잘 그리고 접시와 락으로 부드럽게 혼합 포함 하는 솔루션을 추가 합니다. 48-72 h 게시물 transfection transgene 식 시 금 전에 대 한 공동2 인큐베이터에서 37 ° C에서 세포를 품 어.
8. indel 주파수의 계산에서 대상 및 대상 오프 활동을 결정 하
-
대상 및 잠재적인 대상 오프 사이트의 분석에 대 한 깊은 시퀀싱을 대상으로
- 6.1.5 또는 7.2.4 gDNA 준비 키트와 함께 단계에서 게놈 DNA를 격리 합니다. 뇌관에 대상 및 오프 대상 타겟팅으로 gDNA의 PCR 증폭에 의해 깊은 시퀀싱 라이브러리를 생성 합니다.
- 인덱스 프라이 머를 사용 하 여 각 샘플을. 다음-세대 시퀀싱 컴퓨터를 사용 하 여 쌍 간 연속을 풀링된 라이브러리를 주제.
-
Ca-분석기를 사용 하 여 깊은 시퀀싱 분석
- Cas-분석기 평가 도구17를 사용 하 여 깊은 시퀀싱 데이터를 분석 합니다.
- 읽기 1 과 읽기 2 탭 아래 Fastq 파일을 선택 (읽기 1 XX_SXX_L001_R1_001.fastq, 읽기 2 = XX_SXX_L001_R2_001.fastq =).
- 기본 정보 탭을 작성 하 고 분석 매개 변수 탭 제출 버튼을 클릭 합니다.
결과
스나이퍼-화면 수행 된 후 생존 식민지의 백분율 파운드 플레이트 포함에 식민지의 수에 의해 페니, 대, arabinose, ATC (CKAA)을 포함 하는 파운드에 식민지의 수를 분할 하 여 계산할 수 있다 페니와 대만 (CK)입니다. 이 비율은 일반적으로 매우 낮은 때 스나이퍼 화면 SpCas9의 라이브러리와 함께 수행 했다. True-긍정적인 안타 살아남은 풀 화면을 반복 하 여 농축 수 있습니다. 이 대표 스나이퍼-화면에서 예를 들어 100% 생존 율 얻은 후 세 번째 화면 (그림 1). Transfections RNPs 또는 플라스 미드로 인코딩된 스나이퍼-Cas9를 사용 하 여 다양 한 목표와 결과에 대상 할 수 있는 및 오프 대상 활동에 의해 측정 대상 amplicon 시퀀싱 (그림 2). 최대 및 대상, 스나이퍼 Cas9 보여준다에 대상 활동의 동일한 수준 WT. 끝을 자른 sgRNAs에 비해 더 높은 특이성 비 특이성 (그림 3)을 더욱 향상 하는 데 사용 또한 수 있습니다. 그러나, 그들의 사용 때문에 그들은 대부분의 경우에서 전체 길이 sgRNAs에 비해 낮은 대상에 활동 몇 가지 목표만 제한 됩니다. 따라서, 다양 한 길이 (에서 17-에 20-메)와 sgRNAs를 테스트 해야 합니다와 특이성을 최적화 하에 대상 및 대상 오프 활동을 측정 해야 합니다.

그림 1: 대표 스나이퍼-화면 임의의 Cas9 이체의 다른 라이브러리를 사용 하 여 수행. 변경자 (mutator) 도서관, EP 라이브러리 어 EP 라이브러리 II 다른 상업용 키트를 사용 하 여 만든 라이브러리를 나타냅니다. 첫 번째, 두 번째, 및 최종 농축 화면 횟수 수행7나타냅니다. 이 그림은 리 외.7에서 수정 되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
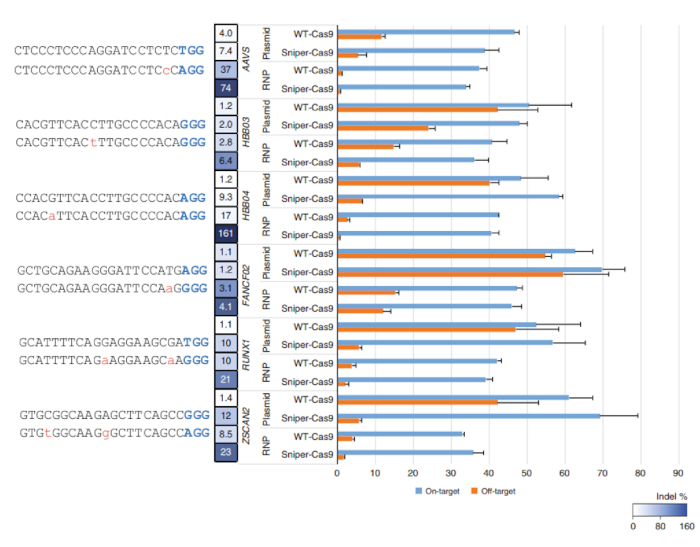
그림 2:-대상 및 대상 오프 활동 WT-Cas9 또는 스나이퍼-Cas9의 플라스 미드 또는 RNP 통해 전달 20-메 르 가이드 시퀀스와 짝. 특이성 비율 오프 대상 활동에 의해 대상에 작업을 분할 하 여 결정 했다. 오차 막대 표시 SEM (n = 3)7. 이 그림은 리 외.7에서 수정 되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
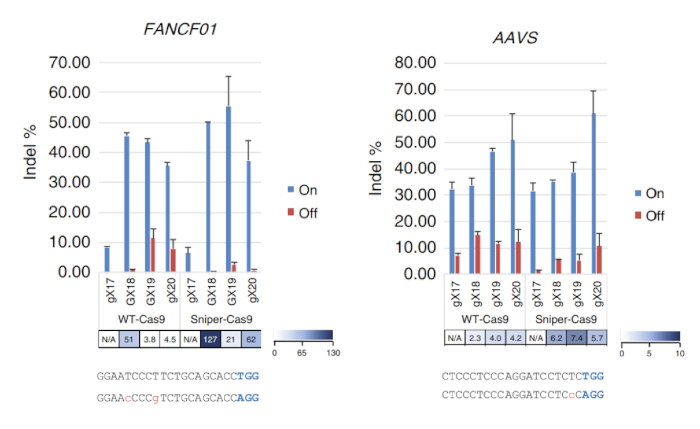
그림 3: 대상에 및 오프 대상의 스나이퍼-Cas9 WT-Cas9 FANCF01 및 AAVS 사이트를 대상으로 하는 가변 길이와 sgRNAs를 사용 하 여 얻은에 비해 활동. 특이성 비율 나누어 indel 주파수에 대상 사이트에서 각 대상 오프 사이트에서 그 결정 했다. 5' 종점 (GX18 또는 GX19)에 일치 구 아닌과 sgRNAs과 일치 하지 않는 구 아닌 (gX17, gX18, gX19, 또는 gX20)와 함께 표시 됩니다. 정규화 된 대상에 활동 했다 특이성 비율 계산 하지 < 707. 이 그림은 리 외.7에서 수정 되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
토론
스나이퍼-Cas9 단백질 및 인코딩 플라스 미드 스나이퍼-Cas9를 성가신 심사 절차를 피하려고 하는 사람들을 위해 사용할 수 있습니다. 높은 특이성 비율을 제공 하는 결정 한다 sgRNA의 최적의 길이 사용 하 여 이러한 자료. 또한, 스나이퍼-Cas9 및 sgRNA RNP 형태로 발생 하기 때문에 그것은 일반적으로 플라스 미드 형태로 배달 보다는 더 높은 특이성 비율에 좋습니다. 스나이퍼-Cas9, 달리 다른 조작된 Cas9 변종 잘린된 sgRNAs6,7 또는 예외 (고음질-Cas9) RNP 양식8 에서 배달와 호환 되지 않습니다.
스나이퍼-화면, 일치 하지 않는 시퀀스의 선택 가장 중요 한 단계입니다. GOI 시퀀스 향해 낮은 분열 활동에 연관 된 일치 하지 않는 sgRNA의 선택은 피해 야 한다. 그렇지 않다면, 특이성의 WT 수준 Cas9 변종 대장균 게놈 DNA 일치 하지 않는 대상 사이트를 쪼개 하지 것입니다. 이 효과 심사 절차 전체를 위태롭게 하는 배경 식민지의 많은 수에서 발생 합니다.
E. 콜라이 있기 때문에 빠른 시간 및 효 모, 스나이퍼-화면에 비해 높은 변환 효율을 두 배로 유리에 비해 효 모 기반 심사 방법. 또한, 스나이퍼 화면 다른 대장균보다 더 민감한 해야-는 플라스 미드에 일치 하지 않는 사이트를 수행 하는 시스템을 기반으로: genomic DNA의 1 개의 사본 및 따라서 단 하나 우리의 시스템에 많은 수의 일치 하지 않는 사이트의 복사본 플라스 미드는 단일 내에서 대장균 세포.
DSBs, Cpf1s, SaCas9 등을 유발 하는 다른 DNA endonucleases의 특이성도 스나이퍼-화면을 사용 하 여 향상 시킬 수 있습니다. 불행 하 게도, 스나이퍼 화면 기본 편집기 DSBs 대장균의 게놈 dna를 유도 하지 마십시오 때문에 직접, 기본 편집기의 특이성을 증가를 사용할 수 없습니다. Nickase 또는 Cas9의 그들의 시스템의 핵심에 죽은 버전을 사용 하는 기본 편집기, 스나이퍼 화면에서 얻은 안타를 사용 하 여 기본 편집기의 특이성을 증가 수 있습니다.
공개
ToolGen 스나이퍼 스크린을 다루는 특허 출원 (PCT/KR2017/006212)을 제기 했다 (상태: 보류, 발명가: Jungjoon K. Lee). Jungjoon 케이 리, Joonsun이, Minhee 정 Euihwan 정은 ToolGen, i n c.의 직원
감사의 말
이 연구는 과학 부와 한국 정보 통신 (2017M3A9B4061406) 국립 연구 재단의 한국 (NRF) 한국 정부 (MSIT) (보조금 번호 2017M3A9B4061404 및 2018M3A9H3020844) Jungjoon 공화국에 의해 자금에서 교부 금에 의해 지원 되었다 리입니다. PGRG36 인코딩 플라스 미드 낸시 크레이그 (Addgene 플라스 미드 #16666)에서 선물 이었고 p11-레이스-wtx1 후 이민 자오에서 선물 했다.
자료
| Name | Company | Catalog Number | Comments |
| Alkaline Phosphatase, Calf Intestinal (CIP) | NEB | M0290L | |
| Ampicillin Sodium Salt | Fisher Chemical | BP1760-25 | |
| Anhydrotetracycline hydrochloride | Sigma Aldrich | 37919 or 94664 | |
| Antibiotic Antimycotic solution | Welgene | LS203-01 | Media component |
| BamHI | Enzynomics | R003L | |
| Chloramphenicol | Sigma Aldrich | R4408 | |
| Diversify PCR Random Mutagenesis | Clontech | 630703 | Error-prone PCR kit |
| DNA-Shuffling Kit | Jena Bioscience | PP-103 | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Welgene | LM001-17 | Culture media |
| Endura Electrocompetent Cells (DUO) | Lucigen | 60242-2 | E. coli electrocompetent cells for library preparation |
| Fetal Bovine Serum (FBS) | Welgene | S101-01 | Media component |
| Gene Pulser II | Bio-Rad | E. coli electroporation equipment | |
| GeneMorph II Random Mutagenesis Kit | Agilent | 200550 | Error-prone PCR kit |
| genomic DNA prep kit | GenAll | 106-152 | gDNA-prep kit |
| Glycerol | BioShop | GLY001.1 | |
| GP/MP Cuvette, 0.1cm | Bio-Rad | BR165-2089 | E. coli electroporation equipment |
| HEK293T cell | ATCC | CRL-11268 | Human cell line |
| Kanamycin sulfate | Acros Organics | 450810500 | |
| L-(+)-Arabinose | Sigma Aldrich | A3256-25G | |
| LaboPass PCR Purification Kit | Cosmogenetech | CMR0112 | |
| LaboPass Plasmid Mini | Cosmogenetech | CMP0112 | Mini-prep kit |
| LB Agar Miller | Formedium | LMM0202 | |
| LB Broth Miller | Formedium | LMM0102 | |
| Lipofectamine | Invitrogen | 11668019 | Transfection reagent |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Gibson assembly (isothermal in vitro recombination) mixture |
| Neon Transfection System | Thermo | MPK5000 | RNP Electroporation equipment |
| Neon Transfection System 10 µL Kit | Thermo | MPK1096 | RNP Electroporation equipment |
| NucleoBond Xtra Midi EF | Macherey-Nagel | 740420.50 | Midi-prep kit |
| Oligo Clean & Concentrator | Zymo Research | D4061 | DNA purification kit that enables low-volume elution |
| Opti-MEM | Gibco | LS31985070 | Transfection media |
| p11-lacY-wtx1 | Addgene | #123945 | |
| pgrg36 | Addgene | #16666 | E. coli mutator strain |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Pyrophophatase | NEB | M2403L | |
| Ribonucleotide Solution Set | NEB | N0450L | |
| RNase inhibitor | NEB | M0314L | |
| T7 RNA polymerase | NEB | M0251L | |
| Turbo Dnase | Ambion | AM2238 | |
| XbaI | Enzynomics | R013L | |
| XL1-Red Competent Cells | Agilent | 200129 |
참고문헌
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nature Biotechnology. 32 (3), 279-284 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Research. 24 (6), 1012-1019 (2014).
- Chen, J. S., et al. Enhanced proofreading governs CRISPR-Cas9 targeting accuracy. Nature. 550 (7676), 407-410 (2017).
- Kleinstiver, B. P., et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature. 529 (7587), 490-495 (2016).
- Slaymaker, I. M., et al. Rationally engineered Cas9 nucleases with improved specificity. Science. 351 (6268), 84-88 (2016).
- Casini, A., et al. A highly specific SpCas9 variant is identified by in vivo screening in yeast. Nature Biotechnology. 36 (3), 265-271 (2018).
- Lee, J. K., et al. Directed evolution of CRISPR-Cas9 to increase its specificity. Nature Communications. 9 (1), 3048(2018).
- Vakulskas, C. A., et al. A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nature Medicine. 24 (8), 1216-1224 (2018).
- Anderson, K. R., et al. CRISPR off-target analysis in genetically engineered rats and mice. Nature Methods. 15, 512-514 (2018).
- Kim, S., Bae, T., Hwang, J., Kim, J. S. Rescue of high-specificity Cas9 variants using sgRNAs with matched 5' nucleotides. Genome Biology. 18 (1), 218(2017).
- Kulcsar, P. I., et al. Crossing enhanced and high fidelity SpCas9 nucleases to optimize specificity and cleavage. Genome Biology. 18 (1), 190(2017).
- Zhang, D., et al. Perfectly matched 20-nucleotide guide RNA sequences enable robust genome editing using high-fidelity SpCas9 nucleases. Genome Biology. 18 (1), 191(2017).
- McKenzie, G. J., Craig, N. L. Fast, easy and efficient: site-specific insertion of transgenes into enterobacterial chromosomes using Tn7 without need for selection of the insertion event. BMC Microbiology. 6, 39(2006).
- Geissmann, Q. OpenCFU, a new free and open-source software to count cell colonies and other circular objects. PLoS One. 8 (2), e54072(2013).
- Chen, Z., Zhao, H. A highly sensitive selection method for directed evolution of homing endonucleases. Nucleic Acids Research. 33 (18), e154(2005).
- Kim, S., et al. CRISPR RNAs trigger innate immune responses in human cells. Genome Research. 28 (3), 367-373 (2018).
- Park, J., et al. Cas-Analyzer: an online tool for assessing genome editing results using NGS data. Bioinformatics. 33, 286-288 (2017).
- Studier, F. W., et al. Use of T7 RNA Polymerase to Direct Expression of Cloned Genes. Methods in Enzymology. 185, 60-89 (1990).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유