Method Article
Usando o Sniper-Cas9 para minimizar efeitos fora do alvo de CRISPR-Cas9 sem perda de atividade no alvo através de evolução dirigida
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para otimizar CRISPR-Cas9 para alcançar uma maior especificidade sem perda de atividade no alvo. Usamos uma abordagem de evolução dirigida chamada Sniper-tela para encontrar um mutante Cas9 com as características desejadas. Franco-atirador-Cas9 é compatível com truncado RNAs single-guia e entrega em um formato ribonucleoprotein, conhecidas estratégias para atingir maiores especificidades.
Resumo
O desenvolvimento do cluster regularmente intercaladas curtas palíndromos repetições (CRISPR)-proteína associada 9 (Cas9) em modalidades terapêuticas exige a prevenção dos efeitos potencialmente deletérias fora do alvo. Vários métodos foram planejados para reduzir tais efeitos. Aqui, apresentamos uma Escherichia coli-evolução dirigida com base método chamado Sniper-tela para obter uma variante Cas9 com especificidade otimizada e retidos no alvo atividade, chamada Sniper-Cas9. Usando o Sniper-tela, positiva e negativa de seleção pode ser executada simultaneamente. A tela também pode ser repetida com outras sequências de RNA (sgRNA) single-guia para enriquecer para os verdadeiros hits positivos. Usando o promotor de CMV-PltetO1 duplo para expressar Cas9 variantes, o desempenho da biblioteca em pool pode ser rapidamente verificado em células de mamíferos. Também são descritos métodos para aumentar a especificidade do Sniper-Cas9. Primeiro, o uso de sgRNAs truncado anteriormente foi mostrado para aumentar a especificidade Cas9. Ao contrário de outros Cas9s engenharia, Sniper-Cas9 mantém um nível de (WT) do selvagem-tipo de atividade no alvo quando combinado com sgRNAs truncado. Em segundo lugar, a entrega de Sniper-Cas9 em um formato ribonucleoprotein (RNP) em vez de um formato de plasmídeo é possível sem afetar a sua actividade no alvo.
Introdução
Neste trabalho, pretendemos melhorar a especificidade das Cas9 combinando diferentes estratégias. Foram desenvolvidos vários métodos de evitar os efeitos fora do alvo de CRISPR-Cas9. Por exemplo, sgRNAs truncado pode ser usado para alcançar maior especificidade1. Além disso, o método de entrega de Cas9 pode ser alterado de um formato de plasmídeo para um formato de RNP para obter maior especificidade2. Resíduos de aminoácidos específicos de Cas9 o Streptococcus pyogenes (SpCas9) proteína foram modificados de acordo com o racional projeto descrito anteriormente3,4,5. Alternativamente, resíduos de aminoácidos foram alterados de forma aleatória e as variantes de Cas9 com a mais alta especificidade foram identificadas usando um fermento6 ou7, e. coli8 sistema de triagem.
No entanto, muitos grupos relataram que as variantes Cas9 projetado usando o projeto para debilitar a interação inespecífica entre Cas9 e o substrato exposição baixo no alvo de actividades7,8,9, 10 , 11 , 12. nós desenvolvemos uma Escherichia coli-sistema baseado em evolução dirigida, Sniper-tela, a tela aleatoriamente mutagenized Cas9 variantes. Uma Escherichia coli sistema de rastreio tem vantagens sobre um sistema de levedura devido a eficiência de transformação de tempo e maior mais rápido, dobrando de e. coli.
Seleção positiva e negativa, com base em três diferentes plasmídeos e um gene de interesse (GOI) integrado no genoma da e. coli , são usadas em Sniper-tela. Cas9 variantes são expressos no âmbito do sistema de dual-promotor CMV-PltetO1 de um plasmídeo número baixo-cópia, para que os candidatos identificados em e. coli podem ser testados em células de mamíferos, sem a necessidade de subcloning. O governo indiano é introduzida no genoma da e. coli usando o sistema de transposon Tn7. O plasmídeo sgRNA, que contém uma origem de replicação do sensível à temperatura, expressa uma sgRNA como alvo o governo indiano; no entanto, o sgRNA e o GOI sequências não são perfeitamente combinadas. Um site de destino perfeitamente sgRNA existe um terceiro plasmídeo contendo o gene que codifica um produto letal que envenena girase ccdB . Neste sistema, as células expressando Cas9 variantes com atividades de alto fora do alvo são removidas porque rupturas da dobro-Costa (DSBs) são introduzidas o site incompatível localizado no DNA genômico. Por outro lado, células expressando Cas9 variantes com baixas no alvo de atividades também são removidas por causa da expressão do gene letal ccdB . O nível de expressão das variantes Cas9 pode ser alterado, alterando a concentração de anhydrotetracycline (ATC), que ajusta a força da seleção.
Nós raciocinou que localizar o site de destino de sgRNA incompatíveis no DNA genômico, em vez de um plasmídeo aumentaria a sensibilidade do sistema. A vantagem dessa abordagem é que há apenas um site de genômico, Considerando que haveria muitos plasmídeos, cada um contendo um site de destino, dentro de uma única célula de e. coli .
Usando este sistema, identificamos uma variante Cas9, Sniper-Cas9, que mostra atividades de WT-nível no alvo e reduziu as atividades fora do alvo em comparação com WT Cas9. Franco-atirador-Cas9 pode atingir proporções de especificidade ainda maiores usando sgRNAs truncado ou entrega baseada na RNP, ao invés de entrega baseada em plasmídeo.
Protocolo
1. integração de um humano indiano para a BW25141 e. coli cepa
-
Clonagem de GOI
- Reação em cadeia da polimerase (PCR) amplificar um comprimento de bp 500 de um humano GOI contendo vários sites de destino do candidato através de métodos padrão PCR com primers contendo NotI e XhoI sites de enzima de restrição.
Nota: Neste experimento, o governo indiano foi o gene humano que EMX1 . - Digeri o produto do PCR e o vetor de13 pgrg36 com enzimas de restrição NotI e XhoI.
- Gel de purificar os fragmentos desejados (500 bp e 12 kb).
- Ligar os fragmentos juntos usando T4 ligase. Para fazer isso, misture 50 ng de pgrg36 digerido e 6 ng da inserção do PCR em um volume de reação de 20 μL contendo 1 buffer de ligase x e 0,5 enzima ligase de U. Incube a temperatura ambiente (RT) durante a noite.
- Transforme o plasmídeo ligado em células competentes DH5-α. Crescer o transformants resultante em placas de ágar Luria caldo (LB) contendo ampicilina (100 μg/mL) a 32 ° C e incubar durante uma noite. Pegar uma colônia e cultivá-la em LB mídia contendo ampicilina durante a noite. Isole o plasmídeo da e. coli, usando um disponível comercialmente miniprep Kit. Confirme a inserção do fragmento GOI por Sanger sequenciando o plasmídeo obtido do plasmídeo mini preparação (mini-preparação)13.
- Reação em cadeia da polimerase (PCR) amplificar um comprimento de bp 500 de um humano GOI contendo vários sites de destino do candidato através de métodos padrão PCR com primers contendo NotI e XhoI sites de enzima de restrição.
-
Preparação do BW25141 -GOI
- Transforme o plasmídeoGOI obtidos pgrg36 - BW25141 e. coli estirpe. É essencial usar uma tensão de BW25141 a fim de minimizar o número de colônias de positivos falsos.
- Crescem as células transformadas no buffer LB a 32 ° C durante a noite. GOI é inserido o DNA genômico da estirpe BW25141 (BW25141 -GOI). Remova o pgrg36 -GOI plasmídeo da estirpe BW25141 -GOI usando o protocolo padrão pgrg3613. Brevemente, diluir uma colônia (aproximadamente 107-Dobre) e cultivá-lo em um prato LB a 42 ° C durante a noite. Marcam as colônias na placa LB e cultivá-las a 42 ° C durante a noite.
- Confirme a inserção correta de GOI pela colônia PCR, utilizando primers sugeridos no protocolo pgrg36: 5'-GATGCTGGTGGCGAAGCTGT-3 'e 5'-GATGACGGTTTGTCACATGGA-3'. O primer amplifica o local de inserção do DNA genômico, e o tamanho do produto do PCR resultante será 904 bp, mais o tamanho da pastilha (500 bp neste caso).
- Prepare electrocompetent células de BW25141 -GOI (um protocolo detalhado é descrito em etapas 3.2.6–3.2.10).
2. preparação da biblioteca variante Cas9
-
Preparação de biblioteca
- Transformar o Cas9 vector7 em um comercial de e. coli modificador Coe (Tabela de materiais) e siga as instruções do fabricante para obter uma variante biblioteca (a biblioteca de modificador).
- Realize PCR propensa em WT Cas9 toda a sequência do vetor Cas9, usando um kit PCR propensa (Tabela de materiais).
Nota: O protocolo de taxa baixa-erro foi adotado no caso Sniper-Cas9 para não perturbar a função original da proteína. - Digeri o vetor Cas9 com enzimas de restrição apropriadas. Gel de purificar o produto do PCR (da etapa 2.1.2) e a espinha dorsal digerida.
Nota: O tamanho do gene SpCas9 é de cerca de 4,3 kb. XhoI e KpnI foram escolhidos para digerir o vetor de pBLC-SpCas9 que foi usado no caso do Sniper-Cas9. - Monte o fragmento de espinha dorsal (da etapa 2.1.3) e a inserção amplificado usando PCR propensa (da etapa 2.1.3) através de recombinação isotérmico em vitro .
Nota: mais de 500 ng da coluna vertebral é necessária para obter uma alta concentração de library (biblioteca de PCR [EP] propenso). Dois kits PCR propensa diferentes foram usados para preparar as bibliotecas de EP no caso Sniper-Cas9 (biblioteca EP I e II). - Purificar os produtos da Assembleia (da etapa 2.1.4) usando um kit de purificação de DNA que permite que o baixo volume de eluição (Tabela de materiais). Eluir com 6 μL de água livre de nuclease (NFW) e medir a concentração de DNA.
- Transformar a mais de 500 ng do vetor de biblioteca Cas9 (para cada uma das três bibliotecas) em 50 μL de células de Escherichia coli electrocompetent (Tabela de materiais). Consulte o protocolo de eletroporação em etapas 3.2.1-3.2.4. Para a preparação desta biblioteca, use 1 mL de meio SOC em vez de 250 μL por 50 μL de células competentes.
- Fazer 1: 100, 1:1, 000 e diluições de 1:10,000 da mistura que contém as células recuperadas com meio SOC. Placa das células diluídas em placas de ágar de 100mm LB suplementadas com cloranfenicol (12,5 μg/mL). As células restantes em uma placa de2 245 mm da placa. Incube a 37 ° C durante a noite.
-
Cálculo da complexidade de biblioteca
- Fotografe as placas de diluição através de um sistema de documentação do gel ou uma câmera digital comum. Executar o software de OpenCFU14 e carregar as fotografias das placas de diluição. Definir a área de contagem dentro de um prato e retire colônias falsas.
- Manualmente, multiplique o número de colônias pelo factor de diluição para obter o número original de transformants. Converta estes números em forma logarítmica (base 10). Calcule a média para determinar a complexidade da biblioteca.
- Quando o valor de complexidade desejada é obtido, reunir todas as colônias na placa quadrada de 245 mm (da etapa 2.1.7) usando um difusor e 20 mL de LB suplementado com cloranfenicol. Não crescem as colônias se reuniram e purificar a biblioteca do plasmídeo usando um kit de midiprep comercial.
Nota: Quanto maior a complexidade da biblioteca, melhor. Quando Sniper-Cas9 foi identificado, uma diversidade de 3 x 106 foi alcançada para cada biblioteca.
3. positivo e negativo de triagem para evoluir Cas9
-
Seleção de alvo e construção do plasmídeo
- Selecione uma sequência do alvo sgRNA espaçador no governo indiano. Substituir um ou dois resíduos no nucleotídeo aleatório para produzir uma sequência incompatível.
Nota: O site de destino humano EMX1 3 (GAGTCCGAGCAGAAGAAGAA com GGG PAM) foi utilizada no caso Sniper-Cas9. Os followings são as sequências incompatíveis usadas: GAGTCCGAGCAGAAagAGAA, GAacCCGAGCAGAAGAAGAA, GAGTCCGAGCAGAgGAAGAA e GAGcCCGAGCAGAAGAAGAA. - Inserir a sequência incompatível (ver passo 3.1.1) para o plasmídeo sgRNA usando o padrão do oligonucleotide (oligo) clonagem procedimentos7.
- Inserir a sequência incompatível com uma PAM na extremidade 3' p11-Landim-wtx1 (Tabela de materiais) para construir o plasmídeo ccdB usando padrão oligo clonagem procedimentos15.
- Selecione uma sequência do alvo sgRNA espaçador no governo indiano. Substituir um ou dois resíduos no nucleotídeo aleatório para produzir uma sequência incompatível.
-
Preparação de células competentes de Sniper-rastreio de Escherichia coli
- Descongele o BW25141 -GOI electrocompetent células no gelo.
- Adicionar 1 ng cada do plasmídeo ccdB e o plasmídeo sgRNA com duplos incompatibilidades em 50 μL de células de BW25141 -GOI descongeladas. Delicadamente, misture as células pipetando e movê-las para uma cubeta de eletroporação prechilled 0,1 cm.
- Transforme a Escherichia coli com o dois plasmídeo via eletroporação. Adicione 250 µ l de meio SOC imediatamente após eletroporação. Pipete suavemente a solução para misturar as células e o meio. Transfira a mistura para um tubo de microcentrifugadora de 1,5 mL.
Nota: Para máxima eficiência, definir a tensão em 1.80 kV e o tempo de execução devem ser entre ms 4,8 e 5,0 ms. - Recuperar as células transformadas e incube-os 32 ° c, durante 1 h com agitação suave.
- 125 μL de células de recuperados em uma ampicilina (50 μg/mL) da placa / placa de ágar-ágar LB canamicina (25 μg/mL) (condição de cultura). Placa restantes células em uma ampicilina/canamicina/arabinose (1,5 mg/mL), placa de ágar LB (ccdB-expressando a condição). Incube a 32 ° C durante a noite.
- Verifique a ausência de sobreviver colônias sobre o ccdB-expressando placa de condição. Reunir as colónias da placa da condição de cultura usando um difusor e cultura-los em 250 mL de meio de caldo super ideal (SOB) suplementado com 50 μg/mL de ampicilina e 25 μg/mL de canamicina a 32 ° C, com suave tremer.
- Quando a densidade óptica em 600 nm (OD600) atinge 0,4, chill o balão no gelo. Preparar uma solução de prechilled de 10% de glicerol e água prechilled (esterilizar antes do uso).
- Centrifugar (a 4.000 x g por 5 min a 4 ° C), as células e descartar o sobrenadante. Adicione 200 mL de água desionizada prechilled. Ressuspender as células usando uma pipeta sorológica 10ml. Repita este passo 3 x.
- Lave as células com 50 mL de solução de glicerol 10% prechilled. Centrifugá-los como antes (a 4.000 x g por 5 min a 4 ° C).
- Desprezar o sobrenadante e ressuspender o precipitado em 300 μL de solução de glicerol 10%. Faça 50 alíquotas μL e congelá-los em nitrogênio líquido. Armazenar as células (células de triagem de Sniper) a-80 ° C.
-
Triagem de franco-atirador
- Transformar as células de triagem de Sniper (da etapa 3.2.10) com 100 ng dos Plasmideos variantes Cas9 de cada biblioteca (da etapa 2.2.3.See etapas 2.1.1 e 2.1.4). Siga os passos de eletroporação, descritos em etapas 3.2.1–3.2.3.
- Transferi 250 μL de células para um tubo de microcentrifugadora de 1,5 mL. Adicione 250 pg de ATC tornar-se uma concentração final de 10 ng/mL. Recupere células tanto contendo ATC e livre de ATC (consulte a etapa 3.2.4 para a etapa de recuperação).
- Placa 25 μL de células de ATC-free recuperadas em uma placa de ágar de cloranfenicol/canamicina LB (condição não seletivo). Adicione o ATC para as células que contêm ATC recuperadas tornar-se uma concentração final de 100 ng/mL em uma placa LB 245 mm. Imediatamente as células numa placa de ágar LB arabinose/cloranfenicol/canamicina (condição seletiva) da placa. Incubar durante uma noite a 32 ° C.
Nota: O tamanho e o número da placa de LB são determinadas pelo tamanho da diversidade das tampas triagem. No caso de uma placa de Petri com 20 mL de LB a 100 mm, adicione 2 μg de ATC. - Fotografe as placas. Conte o número de colônias viáveis usando software de OpenCFU14. (Consulte a etapa 2.2.1) Certifique-se que o número de colônias na placa não seletivo é pelo menos 10x maior do que a diversidade da biblioteca para cobrir todas as variantes.
- Calcule a frequência de sobrevivência como segue.
Frequência de sobrevivência = número de colônias em uma placa seletiva / (o número de colônias em um prato não seletivo x 10) - As colônias que sobreviveram nas placas seletivas de todas as três bibliotecas da piscina. Incube as colônias sobreviventes em 250 mL de LB suplementado com 12,5 cloranfenicol μg/mL a 42 ° C durante a noite. Isole a biblioteca Cas9 selecionada DNA usando um kit de midiprep.
Nota: Este passo limpa o plasmídeo sgRNA. - Repita o processo de seleção de 3.3.1–3.3.6 passos até a frequência de sobrevivência atinge um platô. Uso 10 ng do plasmídeo Cas9 selecionado para transformação e 10 ng/mL de ATC durante a recuperação. Manter a concentração de ATC em 100 ng/mL para a condição de seletiva.
-
Baralhar e a segunda triagem
- Embaralhe as variantes em pool selecionadas usando o seguinte protocolo de DNA-baralhar. PCR amplificar a inserção Cas9 no plasmídeo Cas9 usando as primeiras demão flanqueando, 150 nucleotídeos da divisa inserir. Digest 2 μg de produto PCR amplificado com DNase I para 1 min a 37 ° C.
- Purifica fragmentos 70-200 bp de comprimento usando a eletroforese em gel de agarose 2%. PCR amplificar os fragmentos purificados. Uso o produto como um modelo para PCR amplificar a inserção Cas9 com primers adequados flanqueando Cas9. Use o produto final do PCR para construir uma biblioteca Cas9 conforme descrito na etapa 2.1.4.
- Preparar novas células de triagem de Sniper (ver secção 3.2) com outra do plasmídeo sgRNA incompatíveis (ver passo 3.1.1). Refazer o processo de seleção (seções 3.2-3.3) até a taxa de sobrevivência atinge um platô. Uso 10 ng do plasmídeo Cas9 selecionado para transformação e 10 ng/mL ATC durante a recuperação. Manter a concentração de ATC em 10 ng/mL para a condição de seletiva.
-
Seleção de plasmídeos de mutante evoluídos Cas9
- Após a última etapa de triagem, aleatoriamente escolher 100 colônias da chapa seletiva e cultura-los no meio de LB contendo cloranfenicol a 42 ° C durante a noite.
- Isole o plasmídeo usando um procedimento de miniprep e Sanger-sequência as inserções usando primeiras demão de sequenciamento dentro Cas9.
- Selecione os top três variantes mais frequentes para testá-los em linhas de células humanas.
4. entrega de Cas9 como um RNP com sgRNA truncado
-
Seleção de gene alvo usando Cas-OFFinder
- Escolha os sites de destino com Cas-OFFinder (http://www.rgenome.net/cas-offinder/). Selecione o tipo de PAM apropriado para o tipo específico de Cas9 e genoma alvo (humano, rato, peixe-zebra, etc.). Preencha a guia de sequências de consulta , escolher o número de incompatibilidadee clique no botão Enviar .
- Após alguns segundos, o em-alvo (com um número de incompatibilidade de '0') e sites de fora do alvo aparecem. Em geral, escolha os sites fora do alvo com um a três incompatibilidades.
-
Preparação do modelo sgRNA
- Encomendar os oligos crRNA e tracrRNA com as seguintes sequências de modelo, ou seja, crRNA sequência: 5'-TAATACGACTCACTATAGGNNNNNNNNNNNNNNNNNNNNGTTTTAGAGCTAGAA-3'; sequência de tracrRNA: 5'-AGCACCGACTCGGTGCCACTTTTTCAAGTTGATAACGGACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC-3'. Preencha a sequência de crRNA com a sequência alvo obtida na etapa 4.1.2 N20. Para a concepção de sgRNAs truncado, remova bases da extremidade 5' para obter sequências de sgRNA N19, N18 ou N17.
- Prepare a mistura PCR para a amplificação da sequência sgRNA-codificação da seguinte forma: combinar 10 μL de 5 x tampão, 0,5 μL de oligo crRNA (100 pmol/μL), 0,5 μL de oligo tracrRNA (100 pmol/μL), 2,5 µ l de dNTPs (10 mM cada), 0,5 μL de DNA polimerase e 36 μL de NFW (tabela de materiais).
- Amplificar o modelo usando as seguintes condições, ou seja, para a desnaturação inicial: 1 min a 98 ° C; para a desnaturação, recozimento e extensão: 10 s a 98 ° C, 15 s 54 ° c, 20 s a 72 ° C, durante 25 ciclos; para a extensão final: 5 min a 72 ° C.
- Analise 2 μL do modelo amplificado DNA (da etapa 4.2.3) em um gel de agarose 2%. Purifica o modelo usando um kit de purificação de PCR.
Nota: O tamanho do modelo de DNA é 125 bp.
-
Síntese de sgRNA
- Prepare a mistura de reação para a síntese de sgRNA da seguinte forma: combinar 8,5 μL de DNA de modelo (da etapa 4.2.3), 1 μL de UTP (25 mM), 1 μL de CTP (25 mM), 1 μL de GTP (25 mM), 1 μL de ATP (25 mM), 4.2 μL de MgCl2 (100 mM) , 4,5 μL de T7 RNA polimerase (50 U/μL), 3 μL de tampão de RNA polimerase de x T7 10, 1,2 μL de pyrophosphatase (0.5 U/μL), 0,75 μL de inibidor de RNase (40 U/μL) e 4.2 μL de NFW.
- Incubar a mistura a 37 ° C durante a noite (pelo menos para 10 h). Adicionar 0,5 μL de DNase (2 U/μL) a mistura de reacção e incubar a 37 ° C por 15 a 30 min.. Purify o sgRNA usando um kit de purificação de RNA.
- Para reduzir a toxicidade causada por uma resposta imune inata, desencadeada pelo 5'-trifosfato em sgRNA16, remover a 5'-trifosfato de guia de RNAs com bezerro intestinal da fosfatase alcalina (CIP), como segue: tratar a 10 µ g em vitro- transcrito de RNA com 250 U da CIP por 3 h a 37 ° C, na presença de inibidor de 100 U de RNase. Purifica o CIP-tratados sgRNA usando um kit de purificação de RNA.
5. expressão de proteínas de WT - e Sniper-Cas9 e purificação
-
Expressão da proteína em e. coli
- Transformar os plasmídeos de estimação18 codificação com sua tag WT - e Sniper-Cas9 no BL21 (DE3) Escherichia coli cepa.
- Inocule 50 mL de meio LB contendo 50 canamicina μg/mL com uma colônia fresca, abrigando o plasmídeo de expressão do animal de estimação-Cas9 e agitar durante a noite (200 rpm) a 37 ° C (preculture).
- Transferi 10 mL da cultura durante a noite para 500 mL de meio LB fresco contendo 50 canamicina μg/mL. Incube a cultura com agitação (200 rpm) a 37 ° C por 2 h.
- Monitore o OD600 até que a cultura atinja a fase de registro médio de crescimento (OD600 ≈ 0,6-0,7).
- Induzir a expressão da proteína WT - ou Sniper-Cas9 com isopropílico β-D-1-thiogalactopyranoside (IPTG, em uma concentração final de 0,25 nM). Incube a cultura a 18 ° C durante a noite.
-
Purificação de proteínas
- Recolher as células por centrifugação a 5.000 x g durante 10 minutos a 4 ° C.
- Resuspenda o pellet na Lise (50mm NaH2PO4, 300 mM de NaCl, imidazol 10 mM, 4 mM ditiotreitol [TDT], 5mm benzamidina, 100mm phenylmethylsulfonyl de flúor [PMSF], pH 8) a 20 mL por grama de peso úmido.
- Adicionar PMSF, TDT e lisozima, para uma concentração final de 1 mg/mL cada e incubar a mistura no gelo por 30 min.
- Proceda à sonicação as células no gelo. Pulso repetidamente para 10 s em 200 – 300 W com um 10 s refrigerando o período entre cada pulso, para um tempo total de 20 min.
- Centrifugue o lisado a 6.000 x g durante 30 min a 4 ° C.
- Remova o sobrenadante para um tubo de fresco. Adicionar 1 mL de resina de agarose His-Bind a 5 mL de desmarcada lisado e agitar suavemente durante 1 h a 4 ° C.
- Coloque a mistura de resina de Agarose lisado/His-Bind em uma coluna com uma tomada de fundo tampado.
- Retire a tampa de baixo e recolher a coluna de passagem.
- Lavar a coluna 2 x com tampão de lavagem (50mm NaH2PO4, 300 mM de NaCl, imidazol de 20 mM, pH 8); colete a fração de lavagem para análise por eletroforese em gel polyacrylamide do sulfato dodecyl de sódio (SDS-PAGE).
- Eluir a proteína 10 x 1ml de eluição do buffer coleta amostras de 50 mM (NaH2PO4, 300 mM de NaCl, imidazol 250 mM, pH 8), para SDS-PAGE.
- Concentre a proteína eluted WT - ou Sniper-Cas9 usando um filtro de coluna 100 kDa. Armazenar as amostras em uma solução de glicerol a 10 mM Tris-HCl, NaCl de 150 mM e 50% a-80 ° C.
6. entrega RNP
-
Transfection e preparação de células para a entrega da RNP
- Manter HEK293T células de Dulbecco modificado suplementado do Eagle (DMEM) com 10% de soro fetal bovino (FBS) e antibióticos de 1% a 37 ° C com 5% de CO2.
- Misturar proteína WT - ou Sniper-Cas9 (2 μg) com sgRNA (2 μg) e incube por 10 min a RT tornar complexos da RNP.
- Trypsinize e contagem das células. Prepare 2 x 104 células por uma reação. Lave as células com tampão fosfato salino (PBS) e centrifugar. Aspirar o sobrenadante e ressuspender o precipitado com eletroporação buffer.
- Complexos electroporate RNP para as células com as seguintes configurações, nomeadamente de 1.300 V, 30 ms e um pulso. As células em uma placa de 48 preenchidas com 500 μL de DMEM suplementado com FBS e antibióticos (conforme descrito na etapa 6.1.1) logo após a eletroporação de placa. Incube a 37 ° C com 5% de CO2.
- Isole o DNA genômico com um kit de preparação gDNA, 48 h após o transfeccao.
7. transfection de plasmídeos codificação Sniper-Cas9 e sgRNA
-
Construção de um plasmídeo sgRNA
- Ordem para a frente e reverter oligos com as seguintes sequências de modelo, ou seja, para a frente: 5'-CACCGNNNNNNNNNNNNNNNNNNNN-3'; reverso: 5'-AAACNNNNNNNNNNNNNNNNNNNNC-3'. Substitua a sequência alvo obtida na etapa 4.1.2 N20. Encurte a sequência de destino para um comprimento de N19, N18 ou N17 para sintetizar sgRNA truncado.
- Recoze a ambos os oligos em T4 DNA ligase tampão 1x.
- Digeri o vetor de pRG2 com BsaI enzima de restrição.
- Gel de purificar o vetor digerido (3.300 bp) usando um gel de agarose 0,8%.
- Ligate oligo recozido e o fragmento purificado utilizando T4 ligase em 37 ° c: mistura 50 ng de pRG2 digerido e 1 ng de oligo recozido em um volume de reação de 20 μL. Incube a RT por 15 min.
- Transformar a mistura de ligadura para a cepa alfa DH5 e crescer transformants em placas de ágar LB contendo ampicilina (100 μg/mL) a 37 ° C. Confirme a inserção do oligo do vetor por sequenciamento padrão.
-
Transfection de plasmídeos codificação Sniper-Cas9 e sgRNA
- Manter HEK293T células em DMEM suplementadas com antibióticos FBS e 1% de 10% a 37 ° C com 5% de CO2. O dia antes de transfeccao, trypsinize e contar as células. Quando se trabalha em uma escala de 48-bem, células de5 placa 1 x 10 por bem em 250 μL de meio de cultura completo. As células devem ser confluente 50 – 80% no dia do transfection.
- Na escala de 48-bem, preparar 250 ng de plasmídeo p3s-Cas9 e 250 ng do plasmídeo sgRNA para transfeccao, usando um reagente de transfeccao baseada em lipídios. Misture os plasmideos em 25 μL de soro livre-MEM.
- Dilua 1 µ l de reagente de transfeccao com 25 μL de soro livre-MEM. Incubar a mistura a RT 5 min. Combine as duas misturas e incubar a solução resultante no RT por 20 min para formar complexos de plasmídeo-lipofectamine.
- Depois de 20 min. de incubação, adicione 50 μL de solução contendo os complexos de reagente de transfeccao Plasmideo diretamente a cada um bem contendo células e misturam delicadamente agite-a placa e para trás. Incube as células a 37 ° C numa incubadora de CO2 para transfeccao de post de 48-72 h antes do ensaio para a expressão do transgene.
8. cálculo das frequências indel para determinar atividades no alvo e o alvo
-
Alvo de profundo sequenciamento para a análise de sites de fora do alvo no alvo e potenciais
- Isole o DNA genômico da etapa 6.1.5 ou 7.2.4 com um kit de preparação gDNA. Gere bibliotecas de sequenciamento profunda por amplificação por PCR do gDNA com primers mirando no alvo e o alvo.
- Use as primeiras demão índice para rotular cada amostra. Bibliotecas em pool de assunto para sequenciamento emparelhado-final usando uma máquina de sequenciamento de nova geração.
-
Análise profunda de sequenciamento usando analisador de Cas
- Analise dados de sequenciamento profunda usando o de ferramenta de avaliação de Cas-analisador17.
- Escolha os arquivos de Fastq sob as guias de leitura 1 e 2 de leitura (Read 1 = XX_SXX_L001_R1_001.fastq, leitura 2 = XX_SXX_L001_R2_001.fastq).
- Preencha a guia de Informação básica e na guia Parâmetros de análise clique no botão Enviar .
Resultados
Depois Sniper-tela é executada, a porcentagem de colônias de sobrevivência pode ser calculada dividindo-se o número de colônias na placa LB contendo cloranfenicol, canamicina, arabinose e ATC (CKAA) pelo número de colônias em placa contendo LB cloranfenicol e canamicina apenas (CK). Esta percentagem era geralmente muito baixa quando Sniper-tela foi realizada com as bibliotecas de SpCas9. Sucessos de verdadeiro-positivo podem ser enriquecidos, repetindo a tela com o pool de sobrevivente. Nesta representante Sniper-tela, por exemplo, uma taxa de sobrevivência de 100% foi obtida após a terceira tela (Figura 1). Transfections usando RNPs ou codificado em plasmídeo Sniper-Cas9 podem ser feitos para várias metas e o resultante no alvo e atividades fora do alvo, medidas pelo alvo amplicon sequenciamento (Figura 2). No máximo, alvos, Sniper-Cas9 mostra o mesmo nível de atividades no alvo e mais elevados rácios de especificidade em comparação com o sgRNAs truncado de peso também podem ser usados para melhorar ainda mais a especificidade (Figura 3). No entanto, seu uso é limitado a apenas alguns alvos, porque eles resultam em baixas atividades no alvo, em comparação com o longa-metragem sgRNAs, na maioria dos casos. Portanto, sgRNAs com diferentes comprimentos (de 17 - a 20-mers) deve ser testado e atividades tanto no alvo e o alvo devem ser medidas para otimizar a especificidade.

Figura 1: representante Sniper-telas realizadas com diferentes bibliotecas de variantes de Cas9 aleatórias. Biblioteca de modificador, biblioteca EP eu e biblioteca EP II indicar bibliotecas feitas usando kits comerciais diferentes. Primeiro, segundo e final indicam o número de vezes que a tela de enriquecimento foi realizada7. Esta figura foi modificada de Lee et al.7. Clique aqui para ver uma versão maior desta figura.
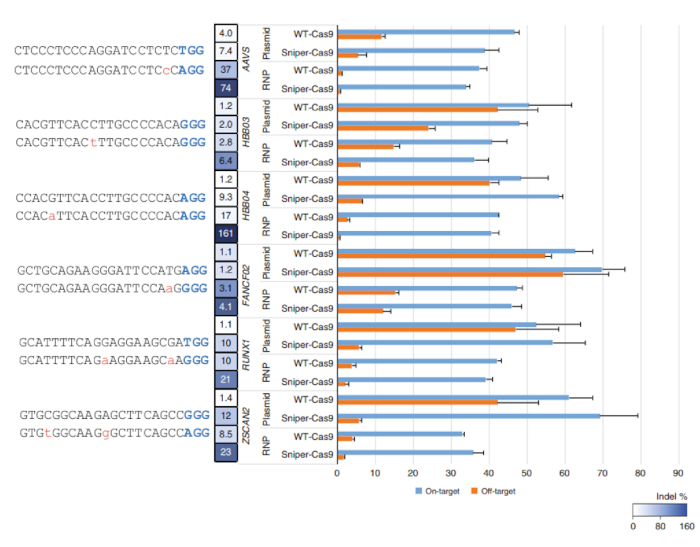
Figura 2: no alvo e o alvo atividades de WT-Cas9 ou Sniper-Cas9 emparelhado com uma sequência de 20-mer guia entregada via plasmídeo ou RNP. Rácios de especificidade foram determinados dividindo-se a atividade no alvo pela atividade fora do alvo. Barras de erro indicam SEM (n = 3)7. Esta figura foi modificada de Lee et al.7. Clique aqui para ver uma versão maior desta figura.
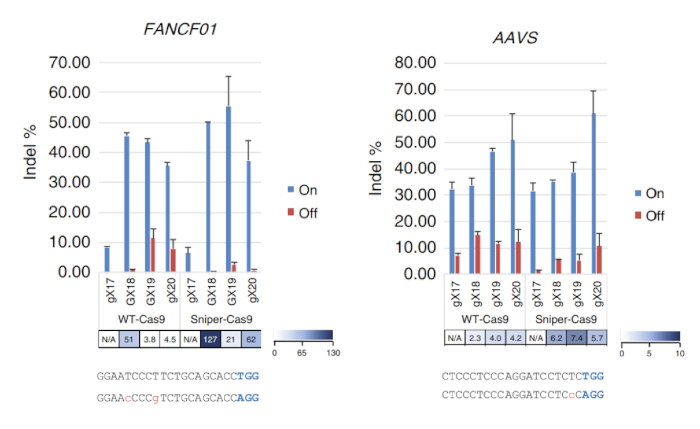
Figura 3: no alvo e fora-alvo atividades de Sniper-Cas9 comparado ao WT-Cas9 obtidos usando sgRNAs com comprimentos variáveis como alvo os sites FANCF01 e AAVS. Rácios de especificidade foram determinados dividindo indel frequências em sites no alvo por aqueles nos respectivos sites fora do alvo. sgRNAs com uma guanina correspondente no 5' terminus (GX18 ou GX19) e aqueles com uma guanina incompatível (gX17, gX18, gX19 ou gX20) são indicados. Rácios de especificidade não foram calculados quando as atividades no alvo normalizadas foram < 70%7. Esta figura foi modificada de Lee et al.7. Clique aqui para ver uma versão maior desta figura.
Discussão
Para aqueles que querem evitar procedimentos de triagem pesado para obter Sniper-Cas9, a proteína do Sniper-Cas9 e o plasmídeo de codificação estão disponíveis. Usando estes materiais, o comprimento ideal do sgRNA, que fornece a maior relação de especificidade, deve ser determinado. Além disso, entrega de Sniper-Cas9 e sgRNA em um formato de RNP é recomendada porque geralmente resulta em uma relação de especificidade maior do que a entrega em um formato de plasmídeo. Ao contrário de Sniper-Cas9, outras variantes Cas9 engenharia não são compatíveis com truncado sgRNAs6,7 ou entrega na RNP formulário8 (com excepção de HiFi-Cas9).
Para Sniper-tela, a seleção da sequência incompatível é o passo mais importante. Seleção de um sgRNA incompatível que está associado com a atividade de decote baixo em direção a sequência GOI deve ser evitada. Se não, Cas9 variantes com um nível WT de especificidade não irão decompor o DNA genômico de Escherichia coli com o site de destino incompatíveis. Este efeito irá resultar em um grande número de colônias de fundo, pondo em risco todo o processo de triagem.
Porque a e. coli tem um rápido dobrar o tempo e a eficiência de transformação de alta em relação ao fermento, Sniper-tela é vantajosa em comparação com métodos de triagem baseada em levedura. Além disso, o Sniper-tela deve ser mais sensível do que outras e. coli-baseado sistemas em que o site não correspondente é carregado em um plasmídeo: há uma cópia do DNA genômico e, portanto, apenas uma cópia do site incompatível no nosso sistema, mas um grande número de plasmídeos dentro de uma única célula de e. coli .
As especificidades de outras endonucleases DNA que induzem DSBs, tais como SaCas9 ou Cpf1s, também poderiam ser melhoradas usando Sniper-tela. Infelizmente, Sniper-tela não pode ser usado para aumentar a especificidade dos editores base diretamente, porque a base editores não induzem DSBs no DNA genômico de e. coli. Como base editores usam o nickase ou morto versão de Cas9 no núcleo do seu sistema, as especificidades dos editores de base poderiam ser aumentadas usando os sucessos obtidos de Sniper-tela.
Divulgações
ToolGen entrou com um pedido de patente (PCT/KR2017/006212) cobrindo Sniper-tela (status: pendente, inventor: Jungjoon K. Lee). Jungjoon K. Lee, Joonsun Lee, Minhee Jung e Euihwan Jeong são empregados da ToolGen, Inc.
Agradecimentos
Esta pesquisa foi apoiada por concessões do Ministério da ciência e das TIC da Coreia (2017M3A9B4061406) e o nacional Research Foundation da Coreia (NRF) financiado pelo governo coreano (MSIT) (concessão números 2017M3A9B4061404 e 2018M3A9H3020844) para Jungjoon K. Lee. O plasmídeo codificação pGRG36 foi um presente de Nancy Craig (plasmídeo Addgene #16666) e p11-LacY-wtx1 foi um presente do Huimin Zhao.
Materiais
| Name | Company | Catalog Number | Comments |
| Alkaline Phosphatase, Calf Intestinal (CIP) | NEB | M0290L | |
| Ampicillin Sodium Salt | Fisher Chemical | BP1760-25 | |
| Anhydrotetracycline hydrochloride | Sigma Aldrich | 37919 or 94664 | |
| Antibiotic Antimycotic solution | Welgene | LS203-01 | Media component |
| BamHI | Enzynomics | R003L | |
| Chloramphenicol | Sigma Aldrich | R4408 | |
| Diversify PCR Random Mutagenesis | Clontech | 630703 | Error-prone PCR kit |
| DNA-Shuffling Kit | Jena Bioscience | PP-103 | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Welgene | LM001-17 | Culture media |
| Endura Electrocompetent Cells (DUO) | Lucigen | 60242-2 | E. coli electrocompetent cells for library preparation |
| Fetal Bovine Serum (FBS) | Welgene | S101-01 | Media component |
| Gene Pulser II | Bio-Rad | E. coli electroporation equipment | |
| GeneMorph II Random Mutagenesis Kit | Agilent | 200550 | Error-prone PCR kit |
| genomic DNA prep kit | GenAll | 106-152 | gDNA-prep kit |
| Glycerol | BioShop | GLY001.1 | |
| GP/MP Cuvette, 0.1cm | Bio-Rad | BR165-2089 | E. coli electroporation equipment |
| HEK293T cell | ATCC | CRL-11268 | Human cell line |
| Kanamycin sulfate | Acros Organics | 450810500 | |
| L-(+)-Arabinose | Sigma Aldrich | A3256-25G | |
| LaboPass PCR Purification Kit | Cosmogenetech | CMR0112 | |
| LaboPass Plasmid Mini | Cosmogenetech | CMP0112 | Mini-prep kit |
| LB Agar Miller | Formedium | LMM0202 | |
| LB Broth Miller | Formedium | LMM0102 | |
| Lipofectamine | Invitrogen | 11668019 | Transfection reagent |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Gibson assembly (isothermal in vitro recombination) mixture |
| Neon Transfection System | Thermo | MPK5000 | RNP Electroporation equipment |
| Neon Transfection System 10 µL Kit | Thermo | MPK1096 | RNP Electroporation equipment |
| NucleoBond Xtra Midi EF | Macherey-Nagel | 740420.50 | Midi-prep kit |
| Oligo Clean & Concentrator | Zymo Research | D4061 | DNA purification kit that enables low-volume elution |
| Opti-MEM | Gibco | LS31985070 | Transfection media |
| p11-lacY-wtx1 | Addgene | #123945 | |
| pgrg36 | Addgene | #16666 | E. coli mutator strain |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530L | |
| Pyrophophatase | NEB | M2403L | |
| Ribonucleotide Solution Set | NEB | N0450L | |
| RNase inhibitor | NEB | M0314L | |
| T7 RNA polymerase | NEB | M0251L | |
| Turbo Dnase | Ambion | AM2238 | |
| XbaI | Enzynomics | R013L | |
| XL1-Red Competent Cells | Agilent | 200129 |
Referências
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nature Biotechnology. 32 (3), 279-284 (2014).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Research. 24 (6), 1012-1019 (2014).
- Chen, J. S., et al. Enhanced proofreading governs CRISPR-Cas9 targeting accuracy. Nature. 550 (7676), 407-410 (2017).
- Kleinstiver, B. P., et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects. Nature. 529 (7587), 490-495 (2016).
- Slaymaker, I. M., et al. Rationally engineered Cas9 nucleases with improved specificity. Science. 351 (6268), 84-88 (2016).
- Casini, A., et al. A highly specific SpCas9 variant is identified by in vivo screening in yeast. Nature Biotechnology. 36 (3), 265-271 (2018).
- Lee, J. K., et al. Directed evolution of CRISPR-Cas9 to increase its specificity. Nature Communications. 9 (1), 3048 (2018).
- Vakulskas, C. A., et al. A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nature Medicine. 24 (8), 1216-1224 (2018).
- Anderson, K. R., et al. CRISPR off-target analysis in genetically engineered rats and mice. Nature Methods. 15, 512-514 (2018).
- Kim, S., Bae, T., Hwang, J., Kim, J. S. Rescue of high-specificity Cas9 variants using sgRNAs with matched 5' nucleotides. Genome Biology. 18 (1), 218 (2017).
- Kulcsar, P. I., et al. Crossing enhanced and high fidelity SpCas9 nucleases to optimize specificity and cleavage. Genome Biology. 18 (1), 190 (2017).
- Zhang, D., et al. Perfectly matched 20-nucleotide guide RNA sequences enable robust genome editing using high-fidelity SpCas9 nucleases. Genome Biology. 18 (1), 191 (2017).
- McKenzie, G. J., Craig, N. L. Fast, easy and efficient: site-specific insertion of transgenes into enterobacterial chromosomes using Tn7 without need for selection of the insertion event. BMC Microbiology. 6, 39 (2006).
- Geissmann, Q. OpenCFU, a new free and open-source software to count cell colonies and other circular objects. PLoS One. 8 (2), e54072 (2013).
- Chen, Z., Zhao, H. A highly sensitive selection method for directed evolution of homing endonucleases. Nucleic Acids Research. 33 (18), e154 (2005).
- Kim, S., et al. CRISPR RNAs trigger innate immune responses in human cells. Genome Research. 28 (3), 367-373 (2018).
- Park, J., et al. Cas-Analyzer: an online tool for assessing genome editing results using NGS data. Bioinformatics. 33, 286-288 (2017).
- Studier, F. W., et al. Use of T7 RNA Polymerase to Direct Expression of Cloned Genes. Methods in Enzymology. 185, 60-89 (1990).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados