Method Article
Etiquetado metabólico extremadamente rápido y específico de ARN In Vivo con 4-Thiouracil (Ers4tU)
En este artículo
Resumen
El uso de uracilo tiolado para purificar sensible y específicamente el ARN recién transcrito de la levadura Saccharomyces cerevisiae.
Resumen
El análogo de nucleótidos, 4-tiouracilo (4tU), es fácilmente tomado por las células e incorporado en el ARN a medida que se transcribe in vivo, permitiendo el aislamiento del ARN producido durante un breve período de etiquetado. Esto se hace mediante la unión de una mitad de biotina al grupo tio incorporado y purificación de afinidad, utilizando cuentas recubiertas de estreptavidina. Lograr un buen rendimiento de ARN puro y recién sintetizado que está libre de ARN preexistente hace posible tiempos de etiquetado más cortos y permite una mayor resolución temporal en estudios cinéticos. Este es un protocolo para la purificación muy específica y de alto rendimiento del ARN recién sintetizado. El protocolo presentado aquí describe cómo se extrae el ARN de la levadura Saccharomyces cerevisiae. Sin embargo, el protocolo para la purificación del ARN tiolado del ARN total debe ser eficaz utilizando ARN de cualquier organismo una vez que se ha extraído de las células. El ARN purificado es adecuado para el análisis por muchas técnicas ampliamente utilizadas, tales como transcriptasa inversa-qPCR, RNA-seq y SLAM-seq. La especificidad, sensibilidad y flexibilidad de esta técnica permiten una visión sin precedentes sobre el metabolismo del ARN.
Introducción
El ARN tiene una naturaleza dinámica; poco después de que se produce mucho ARN se procesa y degrada rápidamente. Actualmente, la mayoría de los estudios de metabolismo del ARN analizan el ARN celular total, que en su mayoría se procesa completamente y a nivel de estado estacionario. Este nivel depende del equilibrio entre las tasas de transcripción, maduración post-transcripción y degradación. El análisis de los procesos que conducen al equilibrio en estado estacionario requiere técnicas especializadas para capturar especies de ARN de muy corta duración.
El etiquetado metabólico del ARN con análogos de nucleótidos como 4-tiouracilo (4tU) o 4-tiouridina (4sU) (ver Duffy et al.1 para una excelente revisión), ofrece la capacidad de aislar ARN nacientes con tio-etiquetados y sus intermedios de procesamiento. Sin embargo, los protocolos publicadosimplican tiempos de etiquetado de varios minutos 2,3, lo que es lento en relación con la tasa de producción de muchas transcripciones. Tarda en transcribir el gen de la levadura promedio, por lo que etiquetar el ARN de levadura durante menos de un minuto puede considerarse extremadamente corto. El protocolo de 4 tiouracilos extremadamente rápido y específico (ers4tU) maximiza la relación señal-ruido maximizando la incorporación de 4tUy minimizando la recuperación del ARN preexistente sin etiquetar, haciendo que los tiempos de etiquetado muy cortos sean posibles 4.
La base modificada por tio debe importarse a las células rápidamente y en cantidad suficiente para etiquetar eficientemente el ARN recién sintetizado (nsRNA). Para promover esto, las células se cultivan en medio libre de uracilo, y la expresión de una permeasa adecuada ayuda a aumentar la ingesta de 4tU o 4sU (ver Tabla 1 para obtener una lista de plásmidos que llevan genes permeasasa adecuados y Figura Suplementaria 1). La solubilidad de 4tU en hidróxido sódico evita la necesidad de disolventes orgánicos tóxicos requeridos por otros análogos de nucleótidos. Desafortunadamente, se ha observado que el cultivo en cultivos en crecimiento durante largos períodos con nucleósidos modificados por tio a concentraciones superiores a 50 m interrumpe los ribosomas5. Sin embargo, la concentración (10 m) utilizada aquí, y los tiempos de etiquetado extremadamente cortos, minimizan los efectos nocivos5 (Figura1a),mientras que todavía producen suficiente ARN para el análisis.
Esta técnica se puede combinar con un agotamiento rápido y específico mediado por auxina de una proteína diana6,7 (Figura2), denominada el protocolo "a partir de LA AID 4U", en el que el estradiol regulaba la expresión de la auxina sistema de degron inducible (AID) se combina con el etiquetado 4tU. Con el enfoque de AID 4U, se puede agotar una proteína diana y controlar estrechamente el efecto sobre el metabolismo del ARN (Figura2). El momento es crítico; es aconsejable ver el vídeo adjunto y prestar mucha atención a la Figura 2 y su forma animada (consulte la Figura complementaria 2).
El procesamiento y la degradación del ARN deben detenerse extremadamente rápidamente para una resolución de tiempo precisa. Esto se logra utilizando metanol a baja temperatura, que fija el contenido celular muy rápidamente y degrada la membrana celular preservando el contenido de ácido nucleico8. La extracción del ARN debe ser eficiente y no dañar el ARN. La lisis mecánica es eficaz en ausencia de agentes chaotrópicos (a menudo estos contienen grupos tio, por lo que debe evitarse). Se prefiere la precipitación de cloruro de litio del ARN, ya que los ARNA se precipitan de manera menos eficiente. los tRNA se transcriben rápidamente yse tiolan naturalmente 9, por lo que la eliminación de los tRNA reduce la competencia por el reactivo de biotinylación. Si los ARN pequeños y altamente estructurados son de interés, se recomiendan métodos de precipitación de ARN a base de alcohol.
Para recuperar el ARN tiolado, la biotina se une covalentemente a través de los grupos tio incorporados en el ARN con 4tU. Se recomienda el uso de biotina modificada, que se une a través de un enlace de disulfuro escleable (p. ej., HPDP-biotina (N-[6-(Biotinamido)hexyl]-3'-(2o-pyridyldithio)propionamida, ) o MTS-biotina (metane thiosulfonate)) ya que permite la liberación del ARN mediante la adición de un agente reductor. El ARN biotinylated es la afinidad purificada en estreptavidina acoplada a cuentas magnéticas. Este protocolo es similar a otros enumerados anteriormente10, pero se ha optimizado intensivamente para reducir el fondo.
Existen dos tipos de experimentos de etiquetado tiol que se pueden realizar, el etiquetado continuo y discontinuo. Cada uno tiene sus propias ventajas. En el etiquetado continuo, el 4tU se añade al cultivo y las muestras tomadas a intervalos regulares. Este tipo de experimento muestra cómo se procesa el ARN y cómo cambian los niveles con el tiempo. Algunos ejemplos son la comparación de mutantes con experimentos de tipo salvaje y un experimento de persecución de pulsos. Los experimentos que se muestran en la Figura 3b,c son de este tipo. Para el etiquetado discontinuo se induce un cambio en el sistema y se supervisa el ARN. Una vez que el cambio ha sido inducido, la cultura debe dividirse en varias subculturas, y en momentos específicos, cada una es etiquetada por tio durante un breve período. Un ejemplo es el AID 4U que se muestra en la Figura 27. Este tipo de experimento es particularmente útil para monitorear el efecto de un cambio metabólico en el procesamiento de ARN (ver Figura 3d).
En la Figura 4 y la Figura 5se presenta una representación gráfica de un experimento de etiquetado tio, y está disponible una hoja de cálculo que simplifica en gran medida el rendimiento del protocolo (consulte 4tU experiment template.xlsx). Además de esto, la información complementaria contiene una extensa guía de solución de problemas. Para el protocolo AID 4U que integra el etiquetado 4tU con el protocolo de agotamiento de la auxina, véase la Figura 2 y la Figura Suplementaria2. Para obtener el protocolo detallado de agotamiento de AID, consulte Barrass etal.
Protocolo
1. Crecimiento y etiquetado tio
NOTA: El tiempo para completar esta sección del protocolo es muy variable, dependiendo de la tasa de crecimiento celular. Permita 1 h para preparar las soluciones y equipos antes del tio-etiquetado y 30 min post-etiquetado para procesar muestras.
- Asegúrese de que la cepa S. cerevisiae contieneun plásmido que codifica una permeasa (Tabla 1) para impulsar la importación de 4tU en la celda.
NOTA: Sin un importador, es poco probable que el etiquetado de menos de 2 minutos tenga éxito11 (véase la figura complementaria 1). La incorporación de 4tU es más eficiente si el crecimiento es medio sin uracilo, por lo que la cepa debe ser URA3+; varios de los plásmidos de la Tabla 1 llevan URA3 como marcador. Si este protocolo se va a combinar con el agotamiento de AID7, se requieren modificaciones de deformación unitaria adicionales. - Preparar el medio libre de uracilo YMM añadiendo 6,9 g de base de nitrógeno de levadura sin aminoácidos, 20 g de glucosa y 1,92 g de SCSM de una sola entrega (Tablade materiales)a 1 L de agua. Autoclave o filtro esterilizar el medio de crecimiento antes de su uso.
NOTA: La esterilización por filtro se prefiere como complejos de péptidos/azúcar producidos por la autoclave coprecipitada con las células del metanol utilizado en la recolección de muestras. - Cultivar levadura en medio libre de uracilo YMM a una densidad óptica a 600 nm (OD600) de 0.6 a 0.8. Asegúrese de que la referencia cultural está en crecimiento en la fase de registro y ha sido durante al menos dos duplicaciones. Normalmente se recomienda un crecimiento a 30 oC, pero se pueden utilizar otras temperaturas, por ejemplo, para cepas sensibles a la temperatura.
NOTA: Dependiendo de la tensión, las condiciones de crecimiento y el rendimiento del ARN, se necesitará un volumen de muestra de aproximadamente 30 ml. Esta cantidad se asumirá a lo largo del protocolo. 30 ml de cultivo es lo más que cabe en un tubo centrífugo de 50 ml con 20 ml de metanol, por lo que es un volumen conveniente para iniciar la optimización. Considere la posibilidad de utilizar más volumen de muestra para los primeros puntos de tiempo para aumentar la recuperación de ARN, hasta 2000 ml se ha utilizado para células de crecimiento más lento en tiempos de etiquetado realmente cortos (<1 min). - Enfríe unos 50 ml de H2O sobre hielo. Para cada muestra, agregue 200 ml de perlas de zirconia a un tubo de tapa de tornillo de 2 ml y enfríe en hielo. También poner 20 ml de metanol (CAUTION), en tubos centrífugos de 50 ml, y colocar sobre hielo seco (CAUTION). El metanol debe ser 1/3 a 2/3 el volumen de la muestra.
ADVERTENCIA: El metanol es tóxico por inhalación, contacto y consumo. Dispensar grandes volúmenes en una campana de humos, y usar dos pares de guantes, como el metanol puede penetrar guantes de laboratorio de nitrilo. El metanol es altamente inflamable, manténgase alejado de todas las fuentes de ignición.
NOTA: Como el hielo seco puede causar quemaduras por frío en contacto y produce gas asfixiante, use guantes cuando manipule y use en un espacio bien ventilado.
NOTA: Añadir las perlas en este punto es más fácil que después de que se haya añadido la muestra, ya que el tubo está seco y al girar hacia abajo el pellet celular las perlas también se giran claramente de la rosca del tubo ahorrando algún tiempo. Además, esto permite que las perlas se enfríen antes de agregar la muestra. - Si se va a añadir un pico de S. pombe al cultivo (en lugar de más tarde), descongelar una alícuota de células de S. pombe tioladas en hielo y vórtice a fondo, al menos 30 s, luego añadir al cultivo. Si se prepara de acuerdo con las instrucciones a continuación, una alícuota S. pombe es suficiente para 400 ml de cultivo (suficiente para doce muestras de 30 ml más un poco para permitir errores en la manipulación). Si se utiliza más o menos cultivo, ajuste el volumen de S. pombe añadido a la referencia cultural.
- Crecer 1 L de cultivo de S. pombe a OD600 a 0.8 exactamente como se describe en el protocolo para S. cerevisiae.
- Tio-etiqueta como paso 1.7, pero por 10 min.
- Arreglar todo el cultivo usando 400 ml de metanol sobre hielo seco, esencialmente como se describe en el paso 1.9.
- Reventar las células por centrifugación a 3000 x g durante 3 min.
- Deseche el sobrenadante y vuelva a suspender el pellet celular en 3,3 ml de H2O.
- Dividir en alícuotas de 80 ol cada una. Conservar a -80oC.
- Utilice toda una alícuota para un cultivo de 400 ml o 10 ml por muestra de 30 ml.
NOTA: Reduzca el volumen de pico a 1/10 si realiza RNAseq. No reutilice alícuotas; desechar cualquier pico no utilizado. Este pico es útil para normalizar y comparar resultados entre puntos de tiempo y experimentos.
- Para el etiquetado discontinuo, induzca la perturbación metabólica requerida (por ejemplo, condiciones de crecimiento, inducción o agotamiento de genes como la AID7 (Figura2 y Figura Suplementaria2) y, a continuación, divida el cultivo. Asegúrese de que todos los matraces y medios estén a la temperatura requerida y, si es posible, airee el medio antes de añadir el cultivo.
- Añadir 4tU al cultivo a una concentración de 10mM y mezclar vigorosamente (1 /10.000 del volumen de cultivo de 100 mM 4tU disuelto en 1 M NaOH). Etiqueta tiol para 15 s a 5 min.
NOTA: Treinta segundos es un buen punto de partida. El etiquetado tio durante menos de 20 s da resultados más variables debido a las dificultades para manipular el cultivo bajo presión de tiempo. Sin embargo, el etiquetado durante más de 1 min reduce la resolución temporal de la técnica. - Si se va a realizar un experimento de persecución; permitir el tio-etiquetado para 20-30 s y luego perseguir añadiendo 1/200 volumen de cultivo de 1 M de uridina (no tiolada), a una concentración final de 5 mM.
NOTA: La uridina es preferible al uracilo para la persecución, ya que la uridina es más soluble en agua, lo que permite añadir un volumen más pequeño al cultivo y por lo tanto hay menos perturbación en el crecimiento de las células. - Tome muestras de cultivo a intervalos regulares (al menos 15 s), hasta el final del curso del tiempo. Los intervalos de muestreo más cortos que esto son difíciles de realizar de forma fiable. Agregue la muestra al metanol sobre hielo seco preparado en el paso 1.4. Para mayor comodidad, añada 30 ml de cultivo a un tubo de 50 ml que contenga 20 ml de metanol.
NOTA: El dióxido de carbono se disuelve en el metanol cuando está frío; esto sale de la solución en la adición de la muestra y espumas vigorosamente al mezclar- resultando en la pérdida de la muestra. Para evitar esto, enfríe el metanol a <-70 oC en un tubo herméticamente sellado hasta que esté cerca del tiempo que se necesita, luego transfiera al hielo seco. - Selle el tubo y mezcle bien agitando. Coloque las muestras sobre hielo. Compruebe que ninguna de las muestras se haya congelado; si es así, caliente suavemente en la mano, invirtiendo constantemente. Esto se hace mejor en la mano, ya que la temperatura de la muestra se puede evaluar, siempre debe sentirfrío. Colocar en hielo. Esto no es un punto de pausa; una vez que toda la muestra es fluida proceda al siguiente paso.
- Gire a 3000 x g durante 2 min (a 4 oC si es posible) para peletizar las células. Vierta el líquido y resuspenda el pellet en al menos 1 ml de agua helada pipeteando suavemente hacia arriba y hacia abajo.
NOTA: El metanol residual en la muestra de pellets ayuda a la resuspensión. - Transfiera a tubos de tapa de tornillo de 2 ml según lo preparado en el paso 1.4. Gire brevemente (por ejemplo, 10 s de tiempo total) a >13.000 x g para volver a pelañizar las células, colocar de nuevo sobre hielo y eliminar el líquido.
NOTA: El pellet celular se puede almacenar a -70 a -80 oC durante varios meses.
2. Preparación del ARN total
NOTA: El tiempo de finalización es de 90 min.
- Utilice soluciones tratadas con piracarbonato dietílico (DEPC) para proteger el ARN de la degradación. Alícuota las soluciones utilizando puntas de pipeta de filtro y guantes de uso en todo momento.
- A una solución añadir 1/1000 volumen de DEPC y mezclar por agitación vigorosa.
- Dejar actuar a temperatura ambiente (RT) durante 24 h y luego autoclave.
- Las soluciones con grupos de aminas (como tris) no se pueden tratar con DEPC. Aliquot el polvo y almacenar especialmente para el trabajo de ARN. Utilice previamente DEPC tratado H2O para hacer la solución.
NOTA: Como el grupo tio en el ARN es fotoactivable, minimice la exposición a la luz UV a partir de este momento. El almacenamiento debe estar en la oscuridad y la incubación se realiza mejor en una máquina pcR con una tapa.
- Si se va a añadir un pico de S. pombe al pellet celular en lugar de al cultivo (no haga ambas cosas), agréguela ahora. Descongelar una alícuota de células de S. pombe tioladas sobre hielo y vórtice a fondo, al menos 30 s, antes de añadir al pellet.
NOTA: Si se prepara de acuerdo con los pasos 1.5.1-1.5.7, se requiere 10 l de S. pombe alícuota para un pellet derivado de 30 ml de cultivo. - Antes de poner se pone la tapa, gire muy brevemente durante 1 x 2 s para asegurarse de que no hay perlas de circonio atrapados entre la tapa y el tubo, lo que puede causar que la muestra y el fenol se escapen del tubo.
- Resuspenda las células en un tampón EDTA de acetato de acetato (AE) de 400 ml (50 mM pH de acetato de sodio 5,3, 10 mM EDTA pH 8.0), vórtina vigorosamente. Añadir 40 s de 10% (p/v) de dodecilo sulfato sódico (SDS). No vórtice, ya que SDS se espumará.
- Si se va a utilizar el protocolo AID 4U, tome 40 ml de la suspensión celular para el análisis proteico7. Añadir 40 s l de AE para hacer que el volumen vuelva a 400 s.
- Añadir 800 l de fenol (CAUTION) a pH y vórtice bajos durante 10 s.
ADVERTENCIA: El fenol es tóxico y corrosivo por inhalación y contacto. Realice siempre procedimientos que impliquen fenol en una campana de humos y use dos pares de guantes. - Lisiar las células en un homogeneizador (por ejemplo, Tabla de materiales)para tres ráfagas de 2 minutos en el ajuste de potencia más bajo. Deje las muestras en hielo durante 2 minutos entre pulsos de homogeneización.
NOTA: Optimice las condiciones si utiliza otros homogeneizadores. Un temblor insuficiente dará lugar a rendimientos deficientes, mientras que el temblor excesivo resulta en un rendimiento aparente mayor, determinado por la absorbancia a 260 nm (A260), pero el ARN puede degradarse. Se prefiere un homogeneizador, pero se puede utilizar la purificación de ARN de fenolcaliente 12. - Colocar la muestra de lysed sobre hielo seco durante 5 min, hasta que se solidifique, esto reduce el arrastre de ADN genómico al ARN. No se congele durante demasiado tiempo, ya que la muestra no se descongelará. Girar 5 min en microfúctea a >13,000 x g en RT; no se sienta tentado a hacer esto a 4 oC, ya que la mezcla de muestra/fenol permanecerá sólida durante todo el giro si se realiza a baja temperatura.
NOTA: Si la muestra sigue congelada al final del giro, vuelva a girar durante otros 5 minutos hasta que la muestra se haya descongelado por completo. - Extracto de fenol/cloroformo y luego extracto de cloroformo con un volumen igual (aproximadamente 600 l) de fenol:cloroformo 5:1 y luego cloroformo (CAUTION). Transfiera la fase superior a otro tubo que contenga fenol:cloroformo 5:1 o cloroformo. Vórtice, luego girar durante 5 minutos en un microfúpelo a RT. A continuación, transfiera la fase superior a un nuevo tubo de 1,5 ml.
ADVERTENCIA: El cloroformo es tóxico por inhalación y contacto. Realice siempre procedimientos que impliquen cloroformo en una campana de humos y use dos pares de guantes. - Añadir un volumen de tercera a la mitad (aproximadamente 300 l) de 10 M LiCl, y mezclar para precipitar el ARN. La muestra debe ir turbia inmediatamente, pero dejar la pena durante al menos 10 minutos sobre hielo o a 4 oC (no almacenar por debajo de -20 oC, ya que se congelará), o hasta que el precipitado flocula.
- Gire durante 5 min a >13.000 x g en un microfófugo. Retire el líquido, vuelva a girar brevemente y retire las escorias. Lavar el pellet con 300 a 500 ml de etanol al 70 %, girar brevemente y eliminar el etanol restante.
NOTA: Durante estos lavados mantener el pellet en el mismo lado del tubo que el primer giro, de esta manera el pellet no se moverá y se romperá; si rompe parte del ARN podría perderse accidentalmente.
NOTA: No seque el pellet; mientras la mayor parte del líquido se haya extraído, no interferirá con los pasos posteriores. El ARN también se puede almacenar en esta etapa a -20 oC durante unos meses o de -70 a -80 oC para el almacenamiento a largo plazo. - Vuelva a disolver el pellet de ARN en 90 oL de TE pH 7,0 (10 mM Tris HCl pH 7,0, 1 mM EDTA pH 8.0) calentando a 65 oC con agitación, ya que el pellet de ARN puede ser difícil de volver a disolver. Esto debe ser por no más de 5 minutos ya que el ARN se degrada a temperaturas más altas. Compruebe si hay solubilización completa del ARN y luego transfiera a un tubo de 0,2 ml. Pipetear la muestra hacia arriba y hacia abajo; no debe haber "bultos", y el líquido debe subir y caer suavemente en la punta. Esta solución es viscosa por lo que el movimiento final de pipeteo debe ser lento.
NOTA: El ARN se puede almacenar a -20 oC en la oscuridad en esta etapa; esto también puede ser beneficioso para la solubilidad del ARN.
3. Biotinylación
NOTA: El tiempo de finalización es de 60 min. Los siguientes pasos se realizan convenientemente en una tira de tubos con tapas integrales, ya que tienen menos tendencia a abrirse en el vórtice que las tiras con tapas separadas.
- Biotinylate añadiendo 10 l (volumen final de1/10) de una solución de biotina HPDP de 5 mM (MTS-biotina se puede utilizar exactamente de la misma manera que HPDP-biotina), al ARN y mezclar a fondo. Precalentar el ARN durante no más de unos segundos a 65 oC antes de añadir la biotina. Incubar a 65oC durante 15 min a un máximo de 30 min en la oscuridad.
NOTA: Este calentamiento es necesario ya que algunos lotes HPDP se precipitan a RT en la muestra de ARN. Un bloque PCR con tapa calentada es ideal para esto. - Preparar un pequeño volumen de resina, columna de exclusión de tamaño (Tablade materiales)para excluir la biotina no incorporada. Retire la etiqueta inferior de la columna y afloje la tapa, colóquela en un tubo centrífugo de 2 ml. Gire a 1500 x g durante 1 min para vaciar el búfer Añadir 0,3 ml de TE suavemente a la parte superior de la columna y girar de nuevo. Repita el lavado y gire dos veces más para un total de 3 lavados. Finalmente transfiera la columna lavada a un tubo fresco de 1,5 ml.
- Una vez completada la incubación de la muestra (paso 3.1), agregue la muestra a la parte superior de la columna. Gire a 1500 x g durante 2 min. La muestra de ARN biotinilado está ahora en la parte inferior del tubo.
NOTA: Un giro de 1 min no es suficiente para eluir toda la muestra. - Añadir un volumen de tercero a medio (aproximadamente 40 l) de 10 M LiCl, mezclar para volver a precipitar el ARN como paso 2.10. La muestra debe quedar turbia inmediatamente, pero dejar durante al menos 5 minutos sobre hielo o a 4 oC o hasta que el precipitado flocula; no almacenar por debajo de -20 oC, ya que se congelará. Centrifugar la muestra durante 5 min a >13.000 x g en un microfófugo.
- Lavar con 80% de etanol, 1 h girando. Siga el procedimiento descrito en el paso 2.11 para extraer la mayor cantidad posible de líquido.
NOTA: Como la biotina HPDP es muy soluble en etanol al 80%, este es un paso de purificación adicional. - Repita el lavado de etanol del 80% para eliminar la mayor cantidad posible de biotina no incorporada.
NOTA: El ARN también se puede almacenar en esta etapa a -20 oC en la oscuridad.
4. Purificación del ARN recién sintetizado
NOTA: El tiempo de finalización es de 2 h.
- Vuelva a disolver el ARN en 200 ml de H2O tratada con DEPC (se puede utilizar la incubación de 65 oC, similar al procedimiento del paso 2.12).
- Mida la concentración de ARN aA 260 utilizando un espectrofotómetro; la muestra puede tener que diluirse 1/10 para obtenerla dentro del rango lineal del espectrofotómetro. Vortex esta dilución durante al menos 10 s para asegurar que el ARN viscoso se disuelva uniformemente.
NOTA: La eficiencia de la biotinylación puede ser evaluada por el punto13 si es necesario. - Añadir cantidades iguales de ARN a un tubo nuevo y hacer hasta 200 l en H2O tratado con DEPC. Utilice toda la muestra con la concentración de ARN más baja y utilice un volumen adecuado de las otras muestras para tener una cantidad similar de ARN para cada una.
NOTA: La plantilla de experimento de hoja de cálculo 4tU.xlsx tiene un formulario para ayudar a este cálculo. - Cuando la muestra esté en RT, añada 25 ml de búfer de 10 x NaTM (0,1 M Tris HCl pH 7,0, 2 M NaCl, 250 mM MgCl2), 25 ml de 1 M de NaPi pH 6,8 (0,5 M NaH2PO4 0,5 M Na2 HPO4) , y 2,5 l de 10% SDS. Mezclar a fondo y girar suavemente (<30 s; aproximadamente 100 x g).
NOTA: Para evitar la precipitación de la SDS y las sales, las muestras deben mantenerse en RT durante los siguientes procedimientos hasta el paso 4.13. - Haga el búfer de cordón que contiene 1 búfer NaTM, 0,1 M NaPi y 0,1% de SDS, 2 ml por muestra. Agregue primero la cantidad necesaria de H2O y el SDS. Esto debe ser fresco cada vez como un precipitado se forma después de 24 h.
NOTA: Para evitar la formación de precipitados, el tampón de cuentas debe mantenerse en RT durante los siguientes procedimientos. No tratamiento DEPC o autoclave. - Añadir 50 l de perlas de estreptavidina a un tubo de baja retención de 1,5 ml. Coloque el tubo en el bastidor magnético, espere a que las perlas se asienten y luego retire el líquido
- Lave las cuentas de estreptavidina.
- Añadir 200 l de tampón de perlas y vórtice hasta que el pellet de cordón se resuspende por completo. Por lo general, 3 x 5 s es todo lo que se requiere. Para los lavados antes de que se agregue la muestra de ARN es suficiente girar los tubos para que las perlas viajen a través del tubo hacia el otro lado. A continuación, vuelva a girar los tubos hacia el lado original para que las cuentas vuelvan a viajar a través del tubo.
- Gire el tubo a baja velocidad (aproximadamente 100 x g) para un máximo de 5 s para girar hacia abajo el fluido, pero no las perlas.
- Colocar en el bastidor magnético para permitir que las cuentas sean capturadas por el imán.
- Retire el líquido por aspiración para un pequeño número de muestras; verter el líquido si muchas muestras.
NOTA: Con un gran número de muestras, eliminar todo el líquido puramente por aspiración puede ser problemático, ya que las perlas de la primera muestra se pueden secar antes de que finalice la última muestra, esto aumenta el fondo. El lavado se puede acelerar vertiendo el líquido de todas las muestras a la vez mientras está en el imán. Se deben dejar un poco más en el imán antes de verter y la pequeña cantidad de líquido que queda tiene que ser aspirado lejos, pero, en general, significa menos tiempo en el imán y sin líquido. De esta manera, es posible hacer 24 o más extracciones rápidamente.
- Bloque con tampón de perlas de 200 l, glucógeno de 10 l 20 mg/ml y arnm de 2,5 ml a 5 mg/ml, extremo giratorio de 20 minutos sobre el extremo a velocidad moderada a RT. La rotación es mantener las cuentas en suspensión. Una vez que el bloqueo se haya completado, retire el líquido como pasos 4.7.2-4.7.4 y lávelo de nuevo, como los pasos de la sección 4.7.
- Vuelva a suspender las perlas de la muestra. Incubar a RT con rotación durante 30 min.
- Durante la incubación, prepare un tubo fresco de 1,5 ml para cada muestra. Añadir 1/10 volumen (aproximadamente 10 l) de 3 M de pH de acetato de sodio 5,3 y 20 g de glucógeno, y girar a aproximadamente 100 x g durante 3 s. Almacenar en un bastidor hasta que sea necesario.
- Retire el ARN no enlazado de las cuentas, como pasos 4.7.2-4.7.4. El ARN sin ataduras se puede perseverar en un tubo fresco, pero las sales y sDS hacen que sea muy difícil de purificar. A continuación, lave las perlas, como sección 4.7 con vórtice, para un mínimo de 3 a un máximo de 5 veces.
- Después del lavado final tenga especial cuidado de aspirar todo el líquido; volver a cada tubo y aspirar las escorias del tampón una vez más.
- Para eluir el ARN, añadir 50 ml de mercaptoetanol recién preparado de 0,7 M a las perlas (1/20 dilución de la solución de stock suministrada comercialmente). Vórtice y gire brevemente, como pasos 4.7.1 y 4.7.2. Coloque la suspensión en el bastidor magnético y pipetee la solución que contiene EL ARN en el tubo centrífugo de 1,5 ml preparado en el paso 4.10.
- Eluir una vez más como paso 4.13 para recuperar el ARN residual de las cuentas y añadir la muestra eluida al tubo que contiene la primera elución de estas cuentas.
- Retire las perlas residuales del ARN eluyéjado colocando la muestra de nuevo en el bastidor magnético y transfiriendo el fluido a un tubo de centrífuga de 0,5 ml de unión baja y fresco.
- Mezcle la muestra y luego precipite el nsRNA añadiendo 2,5x volúmenes (280 l) de etanol y mezcle una vez más. Dejar actuar durante 1 h hasta pasar la noche a -20oC. Girar en una centrífuga pre-enfriada (4 oC) durante 20 minutos a la velocidad máxima (al menos 13.000 x g).
- Lavar bien con 200 ml de etanol al 70% a -20 oC. Como el éME residual inhibirá las aplicaciones aguas abajo, gire en cada paso para eliminar la mayor cantidad posible de escoria; al final, la muestra no debe oler a "ME".
- Re-disolver en 10-20 l de TE tratado con DEPC 1x con el equivalente a 0,005 l de inhibidor de la rnalosa.
NOTA: Todas las etapas posteriores deben realizarse sobre hielo. - Mida la concentración y pureza del ARN.
- Mida el A260 y el A225 en un espectrofotómetro de bajo volumen de muestra.
NOTA: Un máximo de absorbancia cercano a 225 nm proviene de un contaminante inevitable de las perlas. En ausencia de ARN, la señal del contaminante disminuye a 35% a 260 nm. Por lo tanto, la cantidad real de ARN se aproxima por la fórmula: (A260-(A225*0.35))*40 ng/L. - Alternativamente, analice la muestra en un sistema de electroforesis microfluídica como un bioanalizador.
NOTA: Este análisis es preferible al uso de un espectrofotómetro, ya que se puede evaluar la integridad del ARN, el contaminante no interfiere con la cuantificación y se requiere menos muestra.
- Mida el A260 y el A225 en un espectrofotómetro de bajo volumen de muestra.
- Analice el nsRNA.
NOTA: Por ejemplo, los ARN específicos se pueden cuantificar mediante técnicas qPCR de transcriptasa inversa estándar. El ARN preparado de esta manera es compatible con la preparación de la biblioteca para el ARN-seq.
Resultados
Los rendimientos típicos para nsRNA recuperados usando este protocolo ers4tU se muestran en la Figura 1b, esto ha sido producido por un bioanalizador y el seguimiento muestra el rendimiento del ARN frente al tamaño (nucleótidos [nt]). Tenga en cuenta, tanto en la traza del bioanalizador como en el gráfico de inserción, que la recuperación de ARN desde el punto de tiempo 0 es una porción muy pequeña recuperada de puntos de tiempo más largos - aproximadamente 0,3 g de ARN recuperado de aproximadamente 109 células en comparación con más del doble de después de sólo 30 s de etiquetado (0,8 g de nsRNA) del mismo número de células. La recuperación del ARN a 15 s es más variable, ya que las pequeñas diferencias en la realización del muestreo tienen un efecto proporcionalmente mayor en la recuperación del ARN. En la traza del bioanalizador, los precursores de rRNA se pueden ver como un pico cercano a 1000 nt y un doblete de picos a 1700-1800 nt. La abundancia de estos intermedios aumenta a medida que continúa la tiolación.
El etiquetado tio se utilizó para cuantificar el empalme de la transcripción ACT1 (Figura 3). Se realizó tiolación y se monitorizaron muestras tomadas a intervalos de 15 s desde el inicio del tioetiquetado y el procesamiento del ARN ACT1 (Figura 3a,b). Como se puede ver, se genera pre-mRNA (por transcripción), y lariats (por el primer paso de empalme de pre-mRNA), incluso después de sólo 15 s de etiquetado. Después de unos 45 s a 1 min, las cantidades de lariats y pre-mRNA alcanzan el equilibrio con la mayor cantidad de estas especies de ARN que se crean por transcripción como se procesan por empalme.
Para producir los datos mostrados en la Figura 3c, la cepa se pulsaba con 4tU durante 25 s y luego se perseguía con uridina. La generación de pre-mRNA y lariats alcanza un máximo de 1 minuto. Esto se compara bien con la Figura 3b; el máximo que se alcanza después de 45 s para alcanzar el equilibrio más los 25 s del etiquetado. Después del pico, los niveles disminuyen a medida que los ARN etiquetados por tio se persiguen a través del proceso de empalme.
La Figura 3d muestra el agotamiento de un factor de empalme proteico y su efecto sobre el metabolismo del ARN, utilizando el sistema AID 4U6,7. Aquí, Prp16p se reduce de niveles fisiológicos cercanos al 5% de este nivel después de 25 minutos de agotamiento. Prp16p es un factor de empalme esencial para el segundo paso de empalme15. Los lariats se eliminan durante el segundo paso de empalme (Figura3a),pero aquí aumentan por encima del nivel de pre-mRNA como Prp16 se convierte en limitante. En momentos de agotamiento posteriores, otros factores se vuelven limitadores debido a los efectos secundarios, por lo que los niveles de lariat disminuyen, y los niveles de pre-arNM aumentan. El nivel de ARNm empalmado disminuye.
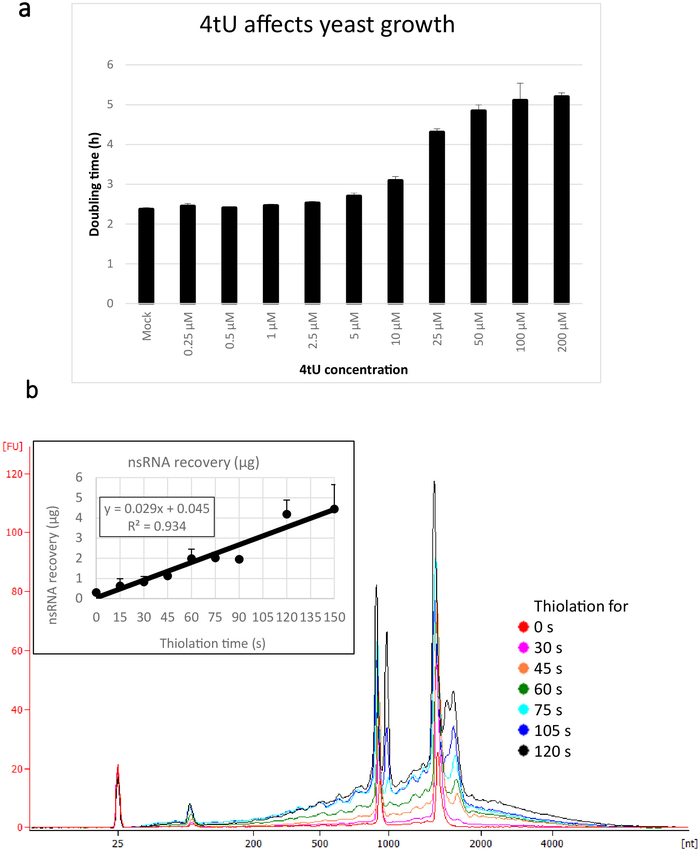
Figura 1: Crecimiento en 4tU y recuperación de ARN. (a ) 4-tiouracilo afecta el crecimiento. El aumento de la concentración de 4tU en el medio de crecimiento de abandono de YMM sin uracilo aumenta el tiempo de duplicación de S. cerevisiae (BY4741) que lleva el plásmido p4Fui-PEST. El crecimiento de cuatro cultivos de réplica sin resultado fue monitoreado a 30oC en un Tecan Infinite Pro 200. Todos los cultivos estaban en fase log a lo largo, con OD600 entre 0.1 y 0.6. Mock es una cultura de control con una cantidad equivalente de NaOH agregada, que por sí misma no cambia la tasa de crecimiento. Este gráfico demuestra que el etiquetado tio es un compromiso entre el etiquetado rápido y el daño a la célula. Las barras de error son un error estándar de 4 réplicas. (b) el rendimiento del nsRNA aumenta linealmente a partir de aproximadamente 15 s de etiquetado. La figura principal muestra los rastros bioanalizadores de purificado, nsRNA de 0 (no tiolado) a 2 min después de la adición de 4tU a intervalos de 15 s. Tenga en cuenta que la muestra de 15 s no se muestra, ya que era indistinguible de la muestra sin etiquetar. Los dos grandes picos corresponden a ARN ribosomales (RNN). Los precursores de rRNA e intermedios son visibles como varios picos con mayor peso molecular que los ARNm maduros. La recuperación de estos precursores e intermedios aumenta con el tiempo. Se muestran los resultados de un experimento representativo. El gráfico de inserción muestra la recuperación de nsRNA con incubación creciente con 4tU. El rendimiento de nsRNA aumenta con el aumento del tiempo de crecimiento con 4tU. La recuperación es notablemente lineal (R2 a 0,934) a lo largo de la escala temporal de este experimento y muestra un ligero aumento sobre el fondo, incluso a 15 s de etiquetado con 4tU, aunque no se distingue de la muestra sin etiquetar por ojo del bioanalizador Rastro. Las barras de error muestran un error estándar para tres réplicas biológicas. Haga clic aquí para ver una versión más grande de esta figura.
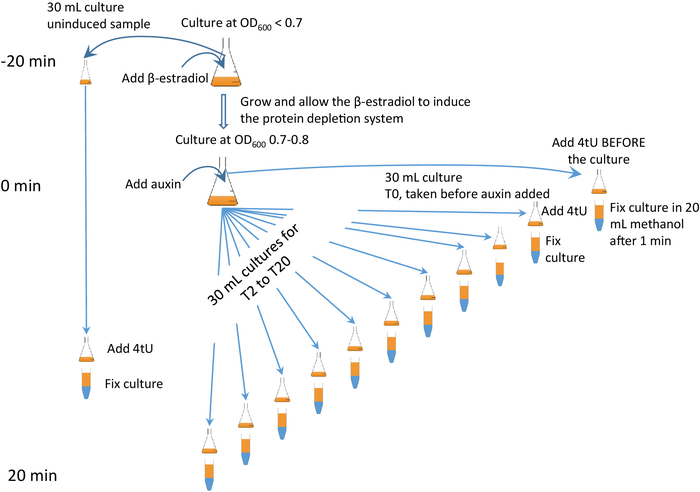
Figura 2: protocolo gráfico AID 4U-est AID 4U. Un resumen gráfico del protocolo del protocolo AID 4U. El sistema de descontento inducible de auxina (AID) que, a su vez, agota una proteína diana etiquetada CON AID*, consulte Barrass et al.7 para obtener un protocolo detallado. En este caso, la expresión del sistema de degron se inicia 25 minutos antes de que comience la degradación de la proteína y la tiolación en cada punto de tiempo es de 1 min. Aparece una versión animada en la figura complementaria 2. Haga clic aquí para ver una versión más grande de esta figura.
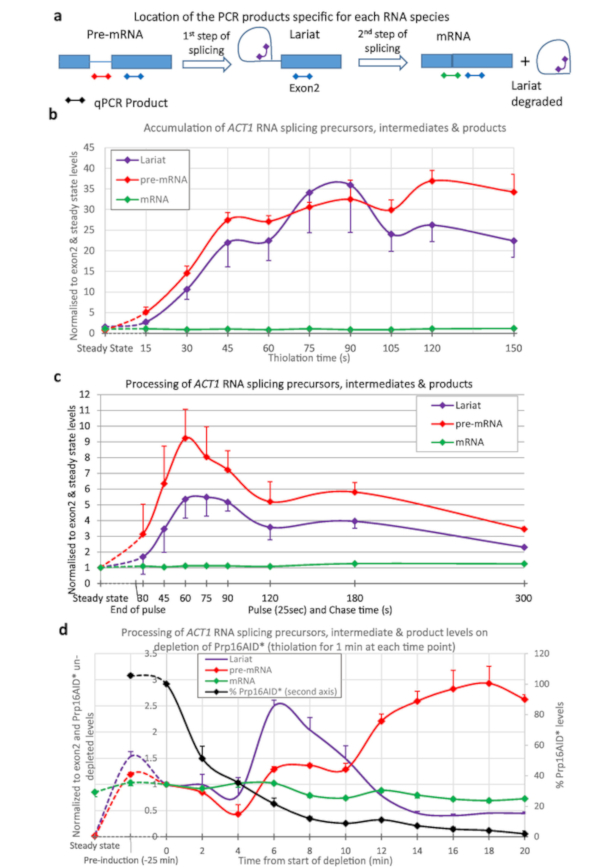
Figura 3: Precursores e intermedios del empalme de ARN ACT1. El empalme de las transcripciones pre-mRNA ACT1 fue monitoreado por transcripción inversa cuantitativa PCR16. Los niveles de precursor ACT1 (pre-mRNA), lariat-exon2 intermedio (Lariat) y producto empalmado (mRNA) se muestran normalizados frente al nivel de ACT1 Exon2 y los niveles de estado estacionario de estos ARN. (a) Ubicación de los productos qPCR en la transcripción act1. Esquema de las ubicaciones de los productos qPCR utilizados para analizar los niveles de precursores, intermedios y productos de la reacción de empalme de las transcripciones ACT1 16. Los exons están representados por cajas, intron como una línea y los productos qPCR como líneas con diamantes en ambos extremos, el color coincide con los utilizados en los gráficos. El PCR pre-mRNA es específico para pre-mRNA y no cualquier intermedio de empalme ya que este producto cruza el punto de bifurcación que se interrumpe después del primer paso de empalme. Lariat PCR detectará el producto del primer paso de empalme y el lariat extirpado producido después del segundo. El MRNA PCR es específico para el producto de empalme, ARNm. Los resultados del PCR exón (presente en todos los precursores, intermedios y productos, excepto el lariat extirpado) no se muestran en los gráficos, ya que se utilizó para normalizar los datos y, por lo tanto, siempre es igual a 1. (b) Tiolar continuo. La cantidad de pre-mRNA aumenta con el tiempo a medida que 4tU se incorpora por transcripción y, después de un breve retraso, el empalme lo convierte en productos intermedios y empalmados lariat-exon2. Los niveles de estas especies pre-mRNA y lariat son detectables sobre el fondo después de tan solo 15 s de crecimiento con 4tU y alcanzan un máximo después de aproximadamente 45 s de etiquetado continuo con 4tU, momento en el que su producción se equilibra por conversión a empalmada ARNm y/o degradación. Los valores se normalizan a su estado estacionario (punto más a la izquierda del gráfico) y los niveles de exón 2 para mostrar su apariencia y procesamiento en comparación con la transcripción del exón 2. Como el empalme de ARN de ACT1 es en gran parte co-transcripcional4,17 mRNA empalmado rápidamente se convierte en la especie más abundante, su nivel es similar al del exón 2. Error estándar de tres réplicas biológicas, cada una ensayada por triplicado. (c) Pulso/persecución. Pulso de tiolación de 25 segundos seguido de persecución con uridina. En comparación con los niveles de estado constante de estos ARN (punto izquierdo), inicialmente son muy abundantes en la piscina recién sintetizada. Los niveles disminuyen gradualmente a medida que se procesan en ARNm (o degradado), acercándose a niveles muy similares a los niveles de estado estacionario en 5 min. Error estándar de tres réplicas biológicas, cada una ensayada por triplicado. (d) nsRNA y agotamiento de proteínas. Empalme de transcripciones pre-mRNA ACT1 supervisadas por PCR de transcripción inversa cuantitativa como en el panel(a ) tras el agotamiento de la proteína Prp16 utilizando el sistema de degron auxin como se describe en la Figura 2. Los niveles de proteína Prp16 también se muestran en el gráfico trazado contra el segundo eje Y como porcentaje de los niveles antes del agotamiento de la auxina. Prp16 es un componente vital del espliceosoma, particularmente importante para el segundo paso de empalme que se muestra en el panel(a ), después de lo cual los lariats se degradan. Cuando este paso se convierte en lariats limitantes se acumulan inicialmente. En momentos posteriores, el empalme falla por completo, los lariats ya no se producen y los niveles de pre-mRNA aumentan. Las barras de error son un error estándar de tres réplicas biológicas, cada una de las que se ensayo en triplicado. Haga clic aquí para ver una versión más grande de esta figura.
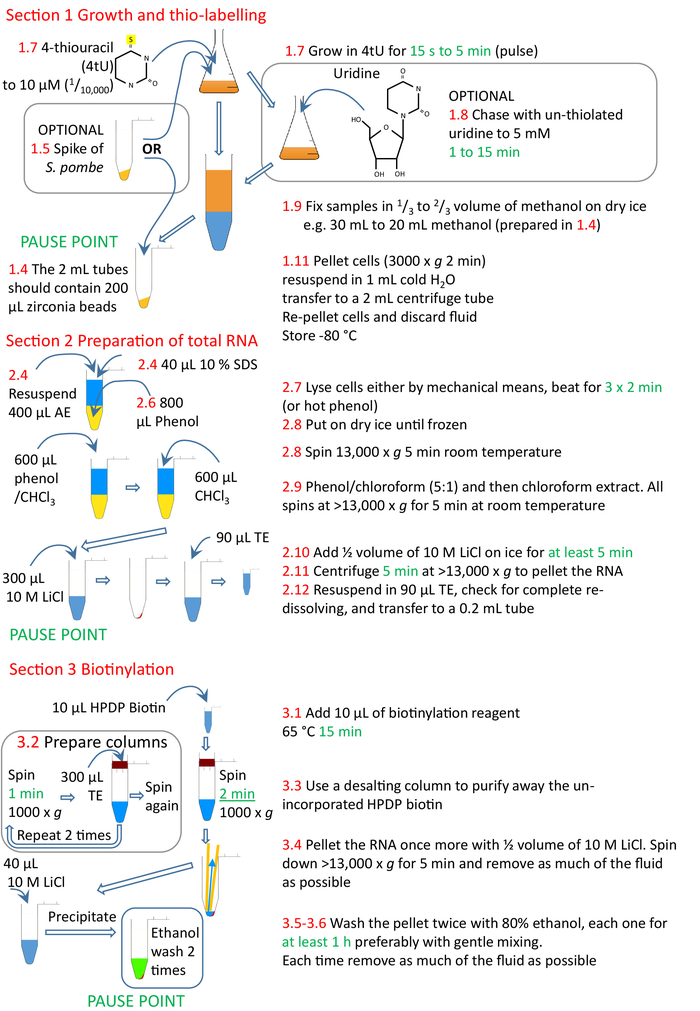
Figura 4: Resumen gráfico de las secciones de protocolo 1 a 3. Las células son tioladas con 4tU y se les permite crecer para incorporar el nucleótido modificado en el ARN. Se puede añadir un pico de s. pombe tilado para permitir la normalización a través de puntos de tiempo y experimentos. El pulso de 4tU se puede perseguir usando uridina no tiolada. El etiquetado puede realizarse continuamente a partir de la adición de 4tU o de un cambio en las condiciones de crecimiento, la división de cultivos y 4tU añadido a los cultivos en momentos cada vez mayores a partir del cambio de condición de crecimiento, pero cada etiquetado sólo por un breve tiempo. Las células se recogen, y el ARN se prepara de las células, preferiblemente utilizando un homogeneizador y métodos basados en fenol. El ARN se biotintila y luego el ARN biotinilado purificado a partir de biotina no incorporada utilizando una columna de exclusión de tamaño. El nsRNA ya está listo para su purificación con perlas de estreptavidina (sección 4, Figura5). Los números en rojo corresponden a los números de paso del protocolo. Haga clic aquí para ver una versión más grande de esta figura.
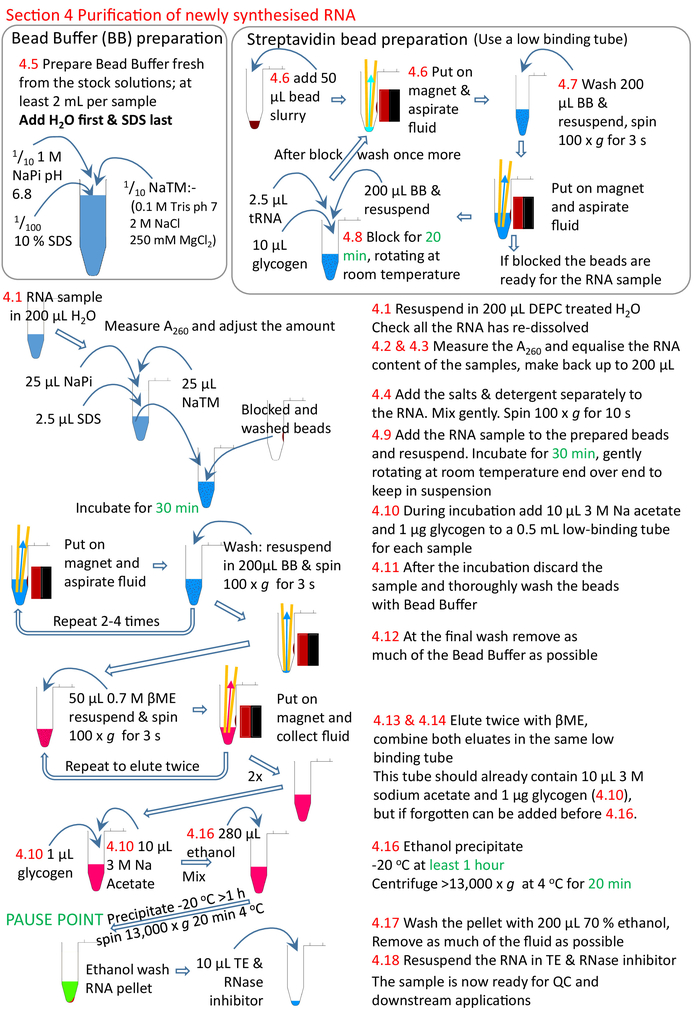
Figura 5: Resumen gráfico de la sección 4 del protocolo. Siguiendo con las secciones 1 a 3 (Figura4),las perlas de estreptavidina se bloquean y la muestra de ARN biotintilado se añade a las perlas preparadas. El ARN biotintida se une a las perlas de estreptavidina y el ARN no biotinilado eliminado y lavado. El ARN biotinylated se eluye de las cuentas usando éME y se precipita listo para más investigación. Los números en rojo corresponden a los números de paso del protocolo. Haga clic aquí para ver una versión más grande de esta figura.
Figura suplementaria 1: Mejora de la recuperación del ARNN de las células de levadura con y sin copias adicionales del importador a los 1 y 3 minutos de etiquetado tio. Tenga en cuenta que Fui1 es el propio promotor de la levadura expresado a partir de un plásmido de 2 m. La copia genómica de este gen está presente en ambas cepas. Haga clic aquí para descargar este archivo.
Figura suplementaria 2: Versión animada del protocolo gráfico AID 4U-est AID 4U. Haga clic aquí para descargar este archivo.
Archivo suplementario 1: 4tU_experiment_template.xltx. Haga clic aquí para descargar este archivo.
| Nombre del plásmido | Importador/permease | Marcador | Comentario | |
| p4Fui | S. cerevisiae Fui1 | URA3 | Fui1 importa Uracil y Uridina, por lo que es ideal para experimentos de pulso/persecución. | |
| pAT2 | S. cerevisiae Fui1 | LEU2 | ||
| p4Fui--PEST | S. cerevisiae Fui1 | URA3 | El motivo PEST de Fui1 ha sido desactivado, por lo que el permeasano no se degrada cuando hay suficiente uracilo intracelular para las necesidades de la célula. Funciona bien en experimentos de etiquetado y mejora el rendimiento de pulso/persecución. | |
| p4Fur | S. cerevisiae Fur4 | URA3 | Uracil permeasa | |
| YEpEBI311 | H. Sapiens ENT1 | LEU2 | Miller et al.11. También contiene un gen de la timidas quinasa de timidina HSV. | |
| (transportador de nucleósidos equilibrativos) | ||||
| Todos los plásmidos se basan en 2 m. Todos los plásmidos p4 y pAT se basan en la serie pRS16 de plásmidos. FUI1 y FUR4 se expresan a partir de sus propios promotores endógenos. | ||||
Tabla 1: Plásmidos utilizados con este protocolo.
Discusión
Este artículo presenta un protocolo para el etiquetado 4tU extremadamente rápido y específico, para la recuperación del ARN naciente y recién sintetizado de S. cerevisiae después de tan solo 15 s de etiquetado, con muy baja contaminación por ARN sin etiquetar.
El usuario siempre debe tener cuidado de mantener la integridad del ARN mediante el uso de temperaturas frías y reactivos tratados con DEPC. La purificación de perlas de streptavidina es generalmente confiable; sin embargo, el búfer de cuentas es difícil de manejar; debe hacerse recién, con sus componentes añadidos en el orden correcto, y no refrigerados o autoclavedos. Las fallas comunes incluyen que el ARN se disuelva incompletamente después de los pasos de precipitación, y por lo tanto no se biotinyla o se pierde de otra manera durante los pasos de procesamiento. Hay una amplia ayuda para la solución de problemas en el material complementario.
Hay algunas limitaciones a tener en cuenta en ers4tU. Uno ya se menciona es que 4tU ralentiza el crecimiento de la levadura (Figura1a). Aparte de los ARN osnicidas endógenasmente9, sólo los ARN que se han transcrito durante el período de etiquetado pueden ser purificados por este método. Las polimerasas que se detienen en genes durante todo el tiempo de tiolación no producirán transcripciones tioladas que puedan purificarse, aunque se pueden recuperar transcripciones que están parcialmente etiquetadas debido a que las polimerasas entran o salen de un estado de pausa durante la tiolación. Las cepas que transcriben mal, ya sea debido a mutaciones o condiciones de crecimiento, producen poco nsRNA, aunque las técnicas utilizadas aquí sin embargo mejorarán la recuperación de nsRNA en comparación con otros métodos. Pueden ser necesarios tiempos más largos y mayores volúmenes de cultivo en estas cepas y condiciones. Tenga en cuenta que el uracilo es una buena fuente de nitrógeno y por lo que este método debe ser probado antes de ser utilizado para estudios que implican inanición de nitrógeno.
El protocolo ers4tU es particularmente útil para el análisis de ARN de corta duración, muchos de los cuales se degradan tan rápidamente que no se pueden identificar sin paralizar la maquinaria de degradación. Algunos ejemplos son las transcripciones inestables crípticas (SUPitas)4y las transcripciones cortas producidas por la terminación prematura o la pausa proximal del promotor18 y la transcripción antisentido "ascendente" de un promotor (PROMPT)19. Los intermedios producidos durante el procesamiento de especies estables de ARN también son transitorios, pero pueden enriquecerse utilizando la transcripción ers4tU4. Por lo tanto, el protocolo ers4tU es excepcional al permitir que las especies de ARN altamente transitorias sean analizadas y capturadas en condiciones casi fisiológicas, lo que es una gran ventaja sobre otros métodos. Esta técnica se ha utilizado para estudiar la transcripción y la cinética de procesamiento de ARN aguas abajo en mutantes de ARN polimerasa que se alargan más rápido o más lento de lo normal20.
La tiolación también es compatible con RNA-seq y SLAM-seq21,permitiendo que todo el ARN producido en un plazo de tiempo muy corto se caracterizó con exquisito detalle.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por la financiación de Wellcome a JB [104648]. El trabajo en el Wellcome Centre for Cell Biology está respaldado por la financiación básica de Wellcome [092076]. Los autores reconocen a los miembros del laboratorio por su ayuda: Bella Maudlin, Emanuela Sani, Susanna De Lucas-Arias y Shiney George. Los autores también quieren dar las gracias a Patrick Cramer por el plásmido YEpEBI31111.
Materiales
| Name | Company | Catalog Number | Comments |
| β-mercaptoethanol (βME) | Sigma-Aldrich | M3148 | CAUTION toxic. Stock solutions are aproximatly 14 M, make at 1/20 dilution for use |
| Chloroform | Sigma-Aldrich | 25668 | CAUTION toxic |
| Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758 | add 1/1000 volume to a solution, leave at room temperature for 24 h, then autoclave |
| DMF (N,N-dimethylformamide) | Sigma-Aldrich | 227056 | CAUTION toxic |
| EDTA | Sigma-Aldrich | 3609 | Make 0.5 M and pH to 8.0 with sodium hydroxide |
| Ethanol | Sigma-Aldrich | 29221 | |
| EZ-link HPDP Biotin | Thermo scientific | 21341 | Store protected from light. Disolve all the vial contents in 22.7 mL DMF (to make a 4 mM stock solution). Store away from water, in the dark & at -20 °C. Check the solution before using, as some batches of HPDP precipitate in storage; heat at 42 °C to resuspend. |
| Glucose | Fisher Scientific | G/0500/60 | |
| Glycogen [20 mg/mL] | Sigma-Aldrich | 10901393001 | Store at -20 °C |
| Immobilised TCEP Disulfide Reducing Gel | Thermo Scientific | 77712 | Optional |
| LiCl | Sigma-Aldrich | 793620 | 10 M solution. CAUTION: this gets very hot as is dissolves and can even boil at greater than 100 oC, add the LiCl crystals to the water slowly. |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 63033 | 1 M solution. CAUTION: this gets very hot as is dissolves and can even boil at greater than 100 oC, add the MgCl2 crystals to the water slowly. |
| Methanol | Fisher Scientific | M/4000/PC17 | CAUTION Toxic and flammable |
| NaH2PO4 | Sigma-Aldrich | S3139 | Make 1 M solutions of each and mix in equal amount to obtain a solution of the appropriate pH |
| Na2HPO4 | Sigma-Aldrich | S3264 | |
| NaCl | Sigma-Aldrich | S9888-M | 5 M solution |
| Phenol, low pH. | Sigma-Aldrich | P4682 | Store in the dark at 4 °C. CAUTION toxic |
| Phenol Chloroform 5:1 (125:24:1) low pH. | Sigma-Aldrich | P1944 | Store in the dark at 4 °C. CAUTION toxic |
| Pierce Spin Columns | Thermo Scientific | 69702 | Optional |
| SCSM single drop-out –ura | Formedium | DSCS101 | |
| Sodium Acetate | Sigma-Aldrich | 32318-M | Make a 3 M solution and pH to 5.3 with acetic acid |
| Sodium hydroxide | Sigma-Aldrich | 795429 | CAUTION corrosive |
| SDS (Sodium dodecyl sulfate) | Sigma-Aldrich | 436143 | CAUTION irritant, do not inhale |
| Streptavidin Magnetic beads | NEB | 1420S | Store at 4°C |
| SUPERase-In, RNase inhibitor | Life technologies | AM2696 | Store at -20°C |
| Thiolated Schizosaccharomyces pombe for spike | See section 1.7 of the protocol | ||
| 4-thiouracil (4tU) | ACROS ORGANICS | 359930010 | Store in the dark. Make 100 mM Stock in 1M NaOH, store solutions at -20°C. |
| Tris base | Sigma-Aldrich | 93362 | 1 M solutions at various pH |
| tRNA | Sigma-Aldrich | 10109541001 | 5mg/ml, store at -20°C |
| Uridine | Sigma-Aldrich | U3750 | Make 1 M solution in H2O. Split into 2 mL aliquots and store at -20 C. |
| Yeast nitrogen base without amino acids with amonium sulphate | Formedium | CYN0410 | |
| Zeba Columns 0.5ml | Thermo Scientific | 89882 | Store at 4 °C |
| Zirconia beads | Thistle Scientific | 110791052 | |
| Equipment and Consumables | |||
| Beadbeater | Biospec | 112011EUR | Other homogenisers can be used; the correct conditions for each homogeniser and strain must be established. |
| Bioanalyser (Agilent) or similar to assess RNA quality. If this is not important a spectrophotometer is useful to quantify the RNA. | |||
| Centrifuge: capable of spinning cultures at 4 °C and at least 3000 g. Pre-chill if possible. | |||
| Centrifuge: capable of spinning up to 2 mL tubes at variable speeds upto 13,000 g and down to 1000 g | |||
| Magnetic rack for separating the beads from the sample. The one used in the paper is 3D printed, available from Thingiverse (thing:3562952). Comercially available racks exist | |||
| PCR machine with a heated lid that will allow incubation in the dark. | |||
| Rotating wheel to rotate 1.5 mL tubes end over end | |||
| Shaking heating block (such as Eppendorf Thermomixer) is recomended | |||
| Tubes, centrifuge, Low retention, RNase free 0.5mL | Eppendorf | H179467N | |
| Tubes, centrifuge, Low retention, RNase free 1.5mL | Ambion | AM12350 | |
| Tubes, centrifuge, 50 mL | Sarstedt | 62.547.004 | Other centrifuge tubes are not gas proof allowing CO2 to disolve in the methanol, this comes out of solution vigorously on adding warm culture, leading to sample loss |
| Tubes, centrifuge, 15 mL | Sarstedt | 62.554.001 | |
| Tubes, 2 mL, screw cap | Greiner | 723361 | |
| Tubes 0.2 mL strip of 8 with integral lids | Brand | 781332 |
Referencias
- Duffy, E. E., Schofield, J. A., Simon, M. D. Gaining insight into transcriptome-wide RNA population dynamics through the chemistry of 4-thiouridine. Wiley Interdisciplinary Reviews: RNA. 10 (1), e1513(2018).
- Windhager, L., et al. Ultrashort and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Research. 22, 2031-2042 (2012).
- Baptista, T., Devys, D. Saccharomyces cerevisiae Metabolic Labeling with 4-thiouracil and the Quantification of Newly Synthesized mRNA As a Proxy for RNA Polymerase II Activity. Journal of Visualized Experiments. (140), e57982(2018).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282(2015).
- Burger, K., et al. 4-thiouridine inhibits rRNA synthesis and causes a nucleolar stress response. RNA Biology. 10, 1623-1630 (2013).
- Mendoza-Ochoa, G. I., et al. A fast and tuneable auxin-inducible degron for depletion of target proteins in budding yeast. Yeast (Chichester England). 36 (1), 75-81 (2018).
- Barrass, J. D., Mendoza-Ochoa, G. I., Maudlin, I. E., Sani, E., Beggs, J. D. Tuning degradation to achieve specific and efficient protein depletion. Journal of Visualized Experiments. , (2019).
- Hobro, A. J., Smith, N. I. An evaluation of fixation methods: Spatial and compositional cellular changes observed by Raman imaging. Vibrational Spectroscopy. 91, 31-45 (2017).
- Gustilo, E. M., Vendeix, F. A. P., Agris, P. F. tRNA's Modifications Bring Order to Gene Expression. Current Opinion in Microbiology. 11, 134-140 (2008).
- Dolken, L., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14, 1959-1972 (2008).
- Miller, C., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Molecular Systems Biology. 7, 458(2011).
- Schmitt, M. E., Brown, T. A., Trumpower, B. L. A rapid and simple method for preparation of RNA from Saccharomyces cerevisiae. Nucleic Acids Research. 18, 3091-3092 (1990).
- Rädle, B., et al. Metabolic Labeling of Newly Transcribed RNA for High Resolution Gene Expression Profiling of RNA Synthesis, Processing and Decay in Cell Culture. Journal of Visualized Experiments. (78), e50195(2013).
- Herzog, V. A., et al. Thiol-linked alkylation of RNA to assess expression dynamics. Nature Methods. 14, 1198-1204 (2017).
- Ohrt, T., et al. Molecular dissection of step 2 catalysis of yeast pre-mRNA splicing investigated in a purified system. RNA. 19, 902-915 (2013).
- Alexander, R. D., et al. RiboSys, a high-resolution, quantitative approach to measure the in vivo kinetics of pre-mRNA splicing and 3′-end processing in Saccharomyces cerevisiae. RNA. 16, 2570-2580 (2010).
- Wallace, E. W. J., Beggs, J. D. Extremely fast and incredibly close: cotranscriptional splicing in budding yeast. RNA. 23, 601-610 (2017).
- Adelman, K., Lis, J. T. Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nature Reviews Genetics. 13, 720-731 (2012).
- Preker, P., et al. RNA Exosome Depletion Reveals Transcription Upstream of Active Human Promoters. Science. 322, 1851-1854 (2008).
- Aslanzadeh, V., Huang, Y., Sanguinetti, G., Beggs, J. D. Transcription rate strongly affects splicing fidelity and cotranscriptionality in budding yeast. Genome Research. 28, 203-213 (2018).
- Schofield, J. A., Duffy, E. E., Kiefer, L., Sullivan, M. C., Simon, M. D. TimeLapse-seq: adding a temporal dimension to RNA sequencing through nucleoside recoding. Nature Methods. 15, 221-225 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados