Method Article
Etichettatura metabolica estremamente rapida e specifica dell'RNA In Vivo con 4-Thiouracil (Ers4tU)
In questo articolo
Riepilogo
L'uso di uracil tiolato per purificare in modo sensibile e specifico l'RNA appena trascritto dal lievito Saccharomyces cerevisiae.
Abstract
L'analogo nucleotide, 4-thiouracil (4tU), è prontamente assorbito dalle cellule e incorporato nell'RNA come viene trascritto in vivo, consentendo l'isolamento dell'RNA prodotto durante un breve periodo di etichettatura. Questo viene fatto attaccando una moiety biotina al gruppo thio incorporato e purificando l'affinità, utilizzando perline rivestite di streptavidin. Raggiungere un buon rendimento di RNA puro, appena sintetizzato, privo di RNA preesistente, rende possibili tempi di etichettatura più brevi e consente una maggiore risoluzione temporale negli studi cinetici. Questo è un protocollo per la purificazione molto specifica e ad alto rendimento dell'RNA appena sintetizzato. Il protocollo qui presentato descrive come l'RNA viene estratto dal lievito Saccharomyces cerevisiae. Tuttavia, il protocollo per la purificazione dell'RNA tiolato dall'RNA totale dovrebbe essere efficace usando l'RNA da qualsiasi organismo una volta che è stato estratto dalle cellule. L'RNA purificato è adatto per l'analisi da parte di molte tecniche ampiamente utilizzate, come la trascrizione inversa-qPCR, RNA-seq e SLAM-seq. La specificità, la sensibilità e la flessibilità di questa tecnica consentono una conoscenza senza precedenti del metabolismo dell'RNA.
Introduzione
L'RNA ha una natura dinamica; subito dopo la produzione, molto RNA viene rapidamente elaborato e degradato. Attualmente, la maggior parte degli studi sul metabolismo dell'RNA analizza l'RNA cellulare totale, che è per lo più completamente elaborato e a livello di stato costante. Questo livello dipende dall'equilibrio tra i tassi di trascrizione, la maturazione post-trascrizione e la degradazione. L'analisi dei processi che portano all'equilibrio dello stato costante richiede tecniche specializzate per catturare specie di RNA di breve durata.
L'etichettatura metabolica dell'RNA con analoghi nucleotidici come 4-thiouracil (4tU) o 4-thiouridine (4sU) (vedi Duffy et al.1 per una recensione eccellente), offre la possibilità di isolare gli RNA nascenti e i loro intermedi di elaborazione. Tuttavia, i protocolli pubblicati comportano tempi di etichettatura di diversi minuti2,3, che è lento rispetto al tasso di produzione di molte trascrizioni. Ci vuole nell'ordine di un minuto per trascrivere il gene medio del lievito, quindi l'etichettatura dell'RNA di lievito per meno di un minuto può essere considerata estremamente breve. Il protocollo 4 tiouracil estremamente rapido e specifico (ers4tU) massimizza il rapporto segnale-rumore massimizzando l'incorporazione 4tU e riducendo al minimo il recupero di RNA preesistente senza etichetta rendendo possibili tempi di etichettatura molto brevi4.
La base tio-modificata deve essere importata nelle cellule rapidamente e in quantità sufficiente per etichettare in modo efficiente l'RNA appena sintetizzato (nsRNA). Per promuovere questo, le cellule sono coltivate in mezzo privo di uracil, e l'espressione di una permeasi appropriata aiuta ad aumentare l'assorbimento di 4tU o 4sU (vedi tabella 1 per un elenco di plasmidi che portano geni di permeasi adatti e Figura supplementare 1). La solubilità di 4tU nell'idrossido di sodio evita la necessità di solventi organici tossici richiesti da altri analoghi nucleotidi. Purtroppo, è stato osservato che le colture in crescita per lunghi periodi con nucleosides thio-modificati a concentrazioni superiori a 50 m sono state osservate interrompere i ribosomi5. Tuttavia, la concentrazione (10 M) utilizzata in questo caso, e i tempi di etichettatura estremamente brevi, riducono al minimo gli effetti deleteri5 (Figura1a), pur producendo RNA sufficiente per l'analisi.
Questa tecnica può essere combinata con un rapido e specifico esaurimento mediato dall'auxina di una proteina bersaglio6,7 ( Figura2), indicato come il protocollo "z-est AID 4U", in cui l'espressione regolata estradiolo dell'auxina sistema di degron inducibile (AID) è combinato con l'etichettatura 4tU. Con l'approccio di 8U, una proteina bersaglio può essere esaurita e l'effetto sul metabolismo dell'RNA attentamente monitorato (Figura2). La tempistica è fondamentale; si consiglia di visualizzare il video di accompagnamento e prestare molta attenzione alla figura 2 e la sua forma animata (vedere Figura supplementare 2).
L'elaborazione e la degradazione dell'RNA devono essere interrotte molto rapidamente per una risoluzione precisa del tempo. Questo si ottiene utilizzando il metanolo a bassa temperatura, che fissa il contenuto cellulare molto rapidamente e degrada la membrana cellulare preservando il contenuto di acido nucleico8. L'estrazione dell'RNA deve essere efficiente e non danneggiare l'RNA. La lisi meccanica è efficace in assenza di agenti caotropici (spesso questi contengono gruppi di thio, quindi dovrebbe essere evitato). È preferibile la precipitazione del cloruro di litio dell'RNA, in quanto i tRNA sono precipitati in modo meno efficiente. I tRNA sono rapidamente trascritti e naturalmente tiolati9, quindi la rimozione dei tRNA riduce la concorrenza per il reagente di biotinylazione. Se sono interessanti RNA piccoli e altamente strutturati, si raccomandano metodi di precipitazione dell'RNA a base di alcol.
Per recuperare l'RNA tiolato, la biotina è attaccata covalentmente attraverso i gruppi di thio incorporati nell'RNA con 4tU. L'uso di biotina modificata, che si attacca tramite un'obbligazione disulfide scissione (ad esempio, HPDP-biotin (N-[6-(Biotinamido)hexyl]-3 in quanto consente il rilascio dell'RNA aggiungendo un agente di riduzione. L'RNA biotinylato è purificato su streptavidina accoppiato a perline magnetiche. Questo protocollo è simile ad altri elencati in precedenza10, ma è stato intensamente ottimizzato per ridurre lo sfondo.
Ci sono due tipi di esperimento di thiol-labelling che possono essere eseguiti, l'etichettatura continua e discontinua. Ognuno ha i suoi vantaggi. Nell'etichettatura continua, il 4tU viene aggiunto alla coltura e ai campioni prelevati a intervalli regolari. Questo tipo di esperimento mostra come viene elaborato l'RNA e come i livelli cambiano nel tempo. Gli esempi includono il confronto di mutanti con esperimenti di tipo selvaggio e un esperimento di inseguimento a impulsi. Gli esperimenti illustrati nella figura 3b.c sono di questo tipo. Per l'etichettatura discontinua un cambiamento viene indotto nel sistema e nell'RNA monitorato. Una volta che il cambiamento è stato indotto la cultura deve essere divisa in diverse sotto-culture, e in momenti specifici, ognuno è poi thio-etichettato per un breve periodo. Un esempio è lo stato mostrato nella figura 27. Questo tipo di esperimento è particolarmente utile per monitorare l'effetto di un cambiamento metabolico sull'elaborazione dell'RNA (vedi Figura 3d).
Una rappresentazione grafica di un esperimento di etichette tio è presentata nella figura 4 e Figura 5e un foglio di calcolo che semplifica notevolmente le prestazioni del protocollo è disponibile (vedere modello di esperimento 4tU.xlsx). Oltre a questo, le informazioni supplementari contengono un'ampia guida alla risoluzione dei problemi. Per il protocollo di z-est AID 4U che integra l'etichettatura 4tU con il protocollo di deplezione dell'auxina, vedere Figura 2 e Figura supplementare 2. Vedere Barrass et al.7 per il protocollo dettagliato di esaurimento dell'AID.
Protocollo
1. Crescita ed etichettatura
NOTA: il tempo per il completamento di questa sezione del protocollo è altamente variabile, a seconda del tasso di crescita delle cellule. Lasciare 1 h per preparare le soluzioni e le attrezzature prima dell'etichettatura e 30 min di post-etichettatura per elaborare i campioni.
- Assicurarsi che il ceppo S. cerevisiae contenga una codifica plasmide di una permeasi (Tabella 1) per aumentare l'importazione di 4tU nella cella.
NOTA: Senza un importatore, è improbabile che l'etichettatura per meno di 2 min abbia successo11 (vedere la figura supplementare 1). L'incorporazione di 4tU è più efficiente se la crescita è in media senza uracile, quindi il ceppo deve essere URA3; molti dei plasmichi nella tabella 1 portano URA3 come marcatore. Se questo protocollo deve essere combinato con l'esaurimentodi7, sono necessarie ulteriori modifiche di deformazione. - Preparare il mezzo yMM esente da uracil aggiungendo 6,9 g di base di azoto di lievito senza aminoacidi, 20 g di glucosio e 1,92 g di SCSM single drop-out- ura (Table of Materials) a 1 L di acqua. Autoclave o filtro sterilizzare il mezzo di crescita prima dell'uso.
NOTA: la sterilizzazione del filtro è preferita come complessi peptidi/zucchero prodotti dalla co-precipitazione autoclavante con le cellule nel metanolo utilizzate nella raccolta dei campioni. - Coltiva il lievito in YMM senza uracil a una densità ottica a 600 nm (OD600) di 0,6-0,8. Assicurarsi che le impostazioni cultura siano in fase di crescita del log e siano state per almeno due raddoppi. La crescita è normalmente raccomandata a 30 gradi centigradi, ma possono essere utilizzate altre temperature, ad esempio, per i ceppi sensibili alla temperatura.
NOTA: A seconda della tensione, delle condizioni di crescita e della resa dell'RNA, saranno necessari circa 30 mL di volume del campione. Questo importo verrà assunto in tutto il protocollo. 30 mL di coltura è il massimo che si inserisce in un tubo di centrifuga 50 mL con 20 mL di metanolo, quindi è un volume conveniente per avviare l'ottimizzazione. Considerare l'utilizzo di più volume campione per i punti di tempo iniziale per aumentare il recupero dell'RNA, fino a 2000 mL è stato utilizzato per le cellule a crescita più lenta in tempi di etichettatura molto brevi (<1 min). - Raffreddare circa 50 mL di H2O sul ghiaccio. Per ogni campione, aggiungere 200 l. di perline di zirconia a un tubo a vite da 2 mL e raffreddare sul ghiaccio. Mettete anche 20 mL di metanolo (CAUTION), in tubi di centrifugazione da 50 mL, e mettete sul ghiaccio secco (CAUTION). Il metanolo deve essere 1/3 a 2/3 il volume del campione.
AMMONIMento: Il metanolo è tossico per inalazione, contatto e consumo. Distribuisci grandi volumi in un cofano a fumi e indossa due paia di guanti, poiché il metanolo può penetrare i guanti da laboratorio nitrile. Il metanolo è altamente infiammabile, tenere lontano da tutte le fonti di accensione.
NOTA: Poiché il ghiaccio secco può causare ustioni fredde a contatto e produce gas asfissiante, utilizzare i guanti durante la manipolazione e l'uso in uno spazio ben ventilato.
NOTA: L'aggiunta delle perline a questo punto è più facile che dopo che il campione è stato aggiunto come il tubo è asciutto e quando filatura lungo il pellet cella le perline sono anche filati fuori del filo tubo risparmiando un po 'di tempo. Inoltre, questo permette alle perline di raffreddarsi prima che il campione venga aggiunto. - Se un picco S. pombe deve essere aggiunto alla coltura (piuttosto che più tardi), scongelare un scongelato di cellule S. pombe tiolate sul ghiaccio e vortice accuratamente, almeno 30 s, quindi aggiungere alla coltura. Se preparato secondo le istruzioni riportate di seguito, un S. pombe aliquot è sufficiente per 400 mL di coltura (abbastanza per dodici campioni da 30 mL più un po 'per consentire errori nella gestione). Se si utilizzano più o meno impostazioni cultura, regolare il volume di S. pombe aggiunto alla cultura.
- Crescere 1 L di s. pombe coltura a OD600 a 0.8 esattamente come descritto nel protocollo per S. cerevisiae.
- Thio-label come passo 1.7, ma per 10 min.
- Correggere tutta la coltura utilizzando 400 mL di metanolo sul ghiaccio secco, essenzialmente come descritto al punto 1.9.
- Pellet le cellule per centrifugazione a 3000 x g per 3 min.
- Scartare il supernatante e risospendere il pellet cellulare in 3,3 mL di H2O.
- Suddivise in aliquote di 80 gradi l ciascuna. Conservare a -80 gradi centigradi.
- Utilizzare tutto uno aliquoto per la coltura 400 mL o 10 l per 30 mL di campione.
NOTA: ridurre il volume di picco a 1/10 se si esegue RNAseq. Non riutilizzare aliquote; scartare qualsiasi picco inutilizzato. Questo picco è utile per normalizzare e confrontare i risultati tra punti temporali ed esperimenti.
- Per l'etichettatura discontinua, indurre la perturbazione metabolica richiesta (ad esempio, condizioni di crescita, induzione o esaurimento genico, come ad esempio l'AID7 di z-est (Figura2 e Figura supplementare 2), quindi dividere la coltura. Assicurarsi che tutti i flaconi e i supporti siano alla temperatura richiesta e, se possibile, aerare il mezzo prima di aggiungere la coltura.
- Aggiungere la 4tU alla coltura a una concentrazione di 10 mM e mescolare vigorosamente (1/10.000 del volume di coltura di 100 mM 4tU sciolto in 1 M NaOH). Thiol-label per 15 s a 5 min.
NOTA: Trenta secondi è un buon punto di partenza. L'etichettatura a meno di 20 s dà più risultati variabili a causa delle difficoltà di manipolazione della coltura sotto pressione temporale. Tuttavia, l'etichettatura per più di 1 min riduce la risoluzione temporale della tecnica. - Se deve essere eseguito un esperimento di inseguimento; consentire l'etichettatura per 20-30 s poi inseguire aggiungendo 1/200 volume di coltura di 1 M di uridina (non tiolata), ad una concentrazione finale di 5 mM.
NOTA: L'uridina è preferibile all'uracil per la caccia, in quanto l'uridina è più solubile in acqua permettendo di aggiungere un volume più piccolo alla coltura e quindi c'è meno disturbo alla crescita delle cellule. - Prelevare campioni di cultura a intervalli regolari (almeno 15 s), fino alla fine del corso temporale. Gli intervalli di campionamento più brevi di questo sono difficili da eseguire in modo affidabile. Aggiungere il campione al metanolo sul ghiaccio secco preparato al punto 1.4. Per comodità, aggiungere 30 mL di coltura a un tubo da 50 mL contenente 20 mL di metanolo.
NOTA: L'anidride carbonica si scioglie nel metanolo quando è freddo; questo viene fuori soluzione con l'aggiunta del campione e schiume vigorosamente al momento della miscelazione, con conseguente perdita del campione. Per evitare ciò, raffreddare il metanolo a <-70 gradi centigradi in un tubo ben sigillato fino a quando è vicino al momento necessario, quindi trasferire sul ghiaccio secco. - Sigillare il tubo e mescolare accuratamente agitando. Mettere i campioni sul ghiaccio. Verificare che nessuno dei campioni sia congelato; in tal caso, delicatamente caldo in mano, invertendo costantemente. Questo è meglio fare in mano come la temperatura del campione può essere valutato, dovrebbe sempre sentire freddo. Mettere sul ghiaccio. Questo non è un punto di pausa; una volta che tutto il campione è fluido procedere alla fase successiva.
- Girare a 3000 x g per 2 min (a 4 gradi centigradi se possibile) per pelletare le cellule. Versare il liquido e risospendere il pellet in almeno 1 mL di acqua ghiacciata con il tubo dolcemente su e giù.
NOTA: Il metanolo residuo nel pellet campione aiuta a una sospensione. - Trasferire a 2 mL tubi del tappo a vite come preparato al passo 1.4. Girare brevemente (ad esempio, 10 s tempo totale) a >13,000 x g per re-pellet le cellule, rimettere sul ghiaccio e rimuovere il liquido.
NOTA: Il pellet cellulare può essere conservato a -70 a -80 gradi centigradi per diversi mesi.
2. Preparazione dell'RNA totale
NOTA: il tempo per il completamento è di 90 min.
- Utilizzare soluzioni trattate DEPC (Diethyl pyrocarbonato) per proteggere l'RNA dalla degradazione. Le soluzioni Aliquot che utilizzano sempre punte filtrante pipette e guanti da indossare.
- A una soluzione aggiungere 1/1000 volume di DEPC e mescolare da agitazione vigorosa.
- Lasciare a temperatura ambiente (RT) per 24 h, quindi autoclave.
- Le soluzioni con gruppi di ammine (come tris) non possono essere trattate DEPC. Aliquota la polvere e conservare appositamente per il lavoro di RNA. Utilizzare precedentemente DEPC trattato H2O per rendere la soluzione.
NOTA: Poiché il tiogruppo sull'RNA è fotoattivabile, ridurre al minimo l'esposizione alla luce UV da questo punto in poi. L'immagazzinamento deve essere al buio e l'incubazione è meglio fare in una macchina PCR con un coperchio.
- Se un picco S. pombe deve essere aggiunto al pellet cellulare piuttosto che alla coltura (non fare entrambe le cose), aggiungerlo ora. Scongelare accuratamente una aliquota di cellule S. pombe tiolate sul ghiaccio e sul vortice, almeno 30 s, prima di aggiungerle al pellet.
NOTA: Se preparato secondo i passi 1.5.1,5,7, è richiesto 10 -L di S. pombe per un pellet derivato da 30 mL di coltura. - Prima di indossare il tappo, girare brevemente per 1/2 s per assicurarsi che non ci siano perline di zirconia intrappolate tra il tappo e il tubo, il che può causare la fuoriuscita di campioni e fenoli dal tubo.
- Risospendere le cellule in 400 , l of acetato EDTA (AE) tampone (50 mM di acetato di sodio pH 5,3, 10 mM EDTA pH 8.0), vorticando vigorosamente. Aggiungete 40 L del 10% (w/v) di solfato di sodio (SDS). Non vortice, come SDS sarà schiuma.
- Se si vuole utilizzare il protocollo AID 4U, prendere 40 l-L della sospensione cellulare per l'analisi delle proteine7. Aggiungere 40 L di AE per riportare il volume fino a 400.
- Aggiungere 800 ll di fenolo (CAUTION) a basso pH e vortice per 10 s.
AMMONIEnte: Il fenolo è tossico e corrosivo per inalazione e contatto. Eseguire sempre procedure che coinvolgono il fenolo in una cappa di fumi e indossare due paia di guanti. - Le cellule vengono filate in un omogeneizzatore (ad esempio, Tabella deimateriali) per tre raffiche di 2 min all'impostazione di potenza più bassa. Lasciare i campioni sul ghiaccio per 2 min tra impulsi di omogeneizzazione.
NOTA: Ottimizzare le condizioni se si utilizzano altri omogeneizzatori. Un agitazione insufficiente comporterà scarse rese, mentre un'eccessiva agitazione si traduce in un rendimento apparente mente più elevato, come determinato dall'assorbimento a 260 nm (A260), ma l'RNA può essere degradato. È preferibile un omogeneizzatore, ma è possibile utilizzare la purificazione dell'RNA fenolo caldo12. - Posizionare il campione lisciviato sul ghiaccio secco per 5 min, fino a quando non si solidifica, questo riduce il dna genomico che trasporta nell'RNA. Non congelare troppo a lungo poiché il campione non si scongela. Spin 5 min in microfuge a >13,000 x g a RT; non essere tentati di farlo a 4 gradi centigradi, poiché la miscela campione/fenolo rimarrà solida per tutta la rotazione se eseguita a bassa temperatura.
NOTA: Se il campione è ancora congelato alla fine della rotazione, rigirare per altri 5 min fino a quando il campione non si è completamente scongelato. - Estratto di fenolo/cloroformio poi estratto di cloroformio con un volume uguale (circa 600 l) di fenolo:cloroformio 5:1 poi cloroformio (CAUTION). Trasferire la fase superiore in un altro tubo contenente fenolo:cloroformio 5:1 o cloroformio. Vortice, poi girare per 5 min in un microfuge a RT. Trasferire quindi la fase superiore in un nuovo tubo da 1,5 mL.
AVVISO: il cloroformio è tossico per inalazione e contatto. Eseguire sempre procedure che coinvolgono il cloroformio in una cappa di fumi e indossare due paia di guanti. - Aggiungere da un terzo alla metà del volume (circa 300 gradi) di 10 M LiCl e mescolare per far precipitare l'RNA. Il campione deve andare immediatamente torbido, ma lasciare per almeno 10 min sul ghiaccio o a 4 gradi centigradi (non conservare al di sotto di -20 gradi centigradi in quanto si congela), o fino a quando il precipitato flocculates.
- Girare per 5 min a >13,000 x g in un microfuge. Rimuovere il fluido, rigirare brevemente e rimuovere la feccia. Lavare il pellet con 300-500 -L del 70% di etanolo, girare brevemente e rimuovere l'etanolo rimanente.
NOTA: Durante questi lavamenti mantenere il pellet sullo stesso lato del tubo come il primo giro, in questo modo il pellet non si muove e si rompe; se si rompe parte dell'RNA potrebbe essere perso accidentalmente.
NOTA: Non asciugare il pellet; finché la maggior parte del fluido è stato rimosso non interferirà con i passaggi successivi. L'RNA può anche essere conservato in questa fase a -20 gradi centigradi per alcuni mesi o da -70 a -80 gradi centigradi per lo stoccaggio a lungo termine. - Sciogliere il pellet di RNA in 90 gradi l di TE pH 7.0 (10 mM Tris HCl pH 7.0, 1 mM EDTA pH 8.0) riscaldando a 65 gradi centigradi con lo scuotimento dell'RNA, poiché il pellet di RNA può essere difficile da riscioglionare. Questo deve essere per non più di 5 min come RNA si degrada a temperature più elevate. Verificare la solubilità completa dell'RNA e trasferirla in un tubo da 0,2 mL. Pipette il campione su e giù; non ci dovrebbero essere "grumi", e il fluido dovrebbe salire e cadere senza intoppi nella punta. Questa soluzione è viscosa, quindi il movimento finale del pipettaggio dovrebbe essere lento.
NOTA: L'RNA può essere conservato a -20 gradi centigradi al buio in questa fase; questo può anche essere utile per la solubilità dell'RNA.
3. Biotinylation
NOTA: il tempo per il completamento è di 60 min. I seguenti passaggi sono convenientemente fatti in una striscia di tubi con tappi integrali in quanto hanno meno tendenza ad aprirsi sul vortice rispetto alle strisce con tappi separati.
- Biotinylate con l'aggiunta di 10 -L (1/10 volume finale) di una soluzione HPDP-biotina da 5 mM (MTS-biotina può essere utilizzata esattamente nello stesso modo dell'HPDP-biotin), all'RNA e mescolare accuratamente. Preriscaldare l'RNA per non più di pochi secondi a 65 gradi centigradi prima di aggiungere la biotina. Incubare a 65 gradi centigradi per 15 min fino a un massimo di 30 min al buio.
NOTA: Questo riscaldamento è necessario in quanto alcuni lotti HPDP precipitano a RT nel campione di RNA. Un blocco PCR con un coperchio riscaldato è l'ideale per questo. - Preparare un piccolo volume di resina, colonna di esclusione delle dimensioni (Tabella dei materiali) per escludere la biotina non incorporata. Rimuovere l'etichetta inferiore della colonna e allentare il tappo, posizionarlo in un tubo di centrifuga di 2 mL. Ruotare a 1500 x g per 1 min per svuotare il buffer Aggiungere 0,3 mL di TE delicatamente nella parte superiore della colonna e girare di nuovo. Ripetere il lavaggio e girare altre due volte per un totale di 3 lavaggi. Trasferire infine la colonna lavata in un tubo fresco da 1,5 mL.
- Una volta completato l'incubazione del campione (passaggio 3.1), aggiungere l'esempio nella parte superiore della colonna. Girare a 1500 x g per 2 min. Il campione di RNA biotinylato si trova ora nella parte inferiore del tubo.
NOTA: Un giro di 1 min non è sufficiente per eluire l'intero campione. - Aggiungere da un terzo alla metà del volume (circa 40 o l) di 10 M LiCl, mescolare per far precipitare nuovamente l'RNA come passo 2.10. Il campione deve andare immediatamente nuvoloso, ma lasciare per almeno 5 min sul ghiaccio o a 4 gradi centigradi o fino a quando il precipitato flocculates; non conservare al di sotto di -20 gradi centigradi in quanto si fermo. Centrifugare il campione per 5 min a >13,000 x g in un microfuge.
- Lavare con 80% di etanolo, rotante 1 h. Seguire la procedura nel passaggio 2.11 per rimuovere la maggior parte del fluido possibile.
NOTA: Poiché l'HPDP-biotina è molto solubile nell'80% di etanolo, questo è un ulteriore passo di purificazione. - Ripetere il lavaggio dell'etanolo dell'80% per rimuovere quanta più biotina non incorporata possibile.
NOTA: L'RNA può anche essere conservato in questa fase a -20 gradi centigradi al buio.
4. Purificazione dell'RNA appena sintetizzato
NOTA: il tempo per il completamento è di 2 h.
- È possibile utilizzare il re-scioglimento dell'RNA in 200 gradi l di incubazione H2O trattata con DEPC, simile alla procedura descritta al punto 2.12.
- Misurare la concentrazione di RNA a A260 utilizzando uno spettrofotometro; il campione potrebbe dover essere diluito 1/10 per ottenerlo all'interno della gamma lineare dello spettrofotometro. Vortice questa diluizione per almeno 10 s per garantire che l'RNA viscoso sia disciolto uniformemente.
NOTA: L'efficienza della biotinylazione può essere valutata con la macchia del punto13, se necessario. - Aggiungere la stessa quantità di RNA a un tubo fresco e fare fino a 200 L in DEPC trattati H2O. Utilizzare tutto il campione con la più bassa concentrazione di RNA e utilizzare un volume appropriato degli altri campioni per avere una quantità simile di RNA per ciascuno.
NOTA: il foglio di calcolo 4tU experiment template.xlsx ha un modulo per facilitare questo calcolo. - Quando il campione è a RT, aggiungere 25 l di 10 x buffer NaTM (0,1 M Tris HCl pH 7.0, 2 M NaCl, 250 mM MgCl2), 25 L di 1 M NaPi pH 6,8 (0,5 M NaH2PO4 0,5 M Na2HPO4) e 2,5 ldi del 10% di SDS. Mescolare accuratamente e ruotare delicatamente (<30 s; circa 100 x g).
NOTA: Per evitare la precipitazione della SDS e dei sali, i campioni devono essere conservati in RT durante le seguenti procedure fino al passaggio 4.13. - Rendere il buffer di perline contenente 1x buffer NaTM, 0,1 M NaPi e 0,1% SDS, 2 mL per campione. Aggiungere prima la quantità richiesta di H2O e la SDS per ultima. Questo deve essere reso fresco ogni volta come un precipitato si forma dopo 24 h.
NOTA: Per evitare la formazione di precipitati, il tampone di perline deve essere mantenuto in RT durante le seguenti procedure. Non DEPC trattare o autoclave. - Aggiungere 50 - L di perline streptavidine ad un tubo a bassa ritenzione 1,5 mL. Posizionare il tubo sul rack magnetico, attendere che le perline si stabilizzino e quindi rimuovere il fluido
- Lavare le perline streptavidin.
- Aggiungere 200 l di tampone e vortice fino a quando il pellet di perline non viene completamente risospeso. Di solito è tutto ciò che è necessario. Per i fusti prima dell'aggiunta del campione di RNA è sufficiente girare i tubi in modo che le perline attraversino il tubo verso l'altro lato. Quindi girare i tubi di nuovo al lato originale in modo che le perline viaggiano attraverso il tubo ancora una volta.
- Girare il tubo a bassa velocità (circa 100 x g) per un massimo di 5 s per girare verso il basso il fluido, ma non le perline.
- Posizionare nel rack magnetico per consentire alle perline di essere catturate dal magnete.
- Rimuovere il fluido per aspirazione per un piccolo numero di campioni; versare il liquido se molti campioni.
NOTA: Con un gran numero di campioni, la rimozione di tutto il fluido puramente per aspirazione può essere problematica, in quanto le perline nel primo campione possono essere essiccate prima che l'ultimo campione sia finito, questo aumenta lo sfondo. Il lavaggio può essere accelerato versando il fluido da tutti i campioni in una sola volta mentre sul magnete. Dovrebbero essere lasciati un po 'più a lungo sul magnete prima di versare e la piccola quantità di liquido che rimane deve essere aspirato via, ma, nel complesso, significa meno tempo sul magnete e senza fluido. In questo modo, è possibile effettuare rapidamente 24 o più estrazioni.
- Blocco con buffer di 200, 10 l 20 mg/mL di glicogeno e 2,5 mg/mL di tRNA, 20 min rotante fine oltre alla fine a velocità moderata a RT. La rotazione è quella di mantenere le perline in sospensione. Una volta completato il blocco, rimuovere il fluido come passaggi 4.7.2-4.7.4 e lavare di nuovo, come i passaggi nella sezione 4.7.
- Risospendere le perline nel campione. Incubare a RT con rotazione per 30 min.
- Durante l'incubazione, preparare un tubo fresco da 1,5 mL per ogni campione. Aggiungete 1/10 di volume (circa 10 g) di 3 M di acetato di sodio pH 5,3 e 20 g di glicogeno, e girare a circa 100 x g per 3 s. Conservare in un rack fino a quando necessario.
- Rimuovere l'RNA non legato dalle perline, come passi 4.7.2-4.7.4. L'RNA non legato può essere perseverato in un tubo fresco, ma i sali e sDS lo rendono molto difficile da purificare. Quindi lavare le perline, come sezione 4.7 con vortice, per un minimo di 3 a un massimo di 5 volte.
- Dopo il lavaggio finale fare particolare attenzione ad aspirare tutto il liquido; tornare ad ogni tubo e aspirare la feccia del buffer ancora una volta.
- Per eluire l'RNA, aggiungere alle perline di diluizione della soluzione di stock fornita commercialmente da0,7 M. Vortice e girare brevemente, come passi 4.7.1 e 4.7.2. Collocare il liquame nel rack magnetico e il tubo l'RNA contenente la soluzione nel tubo di centrifugadi da 1,5 ml preparato al punto 4.10.
- Elutare ancora una volta come passo 4.13 per recuperare l'RNA residuo dalle perline e aggiungere il campione eluito al tubo contenente la prima eluizione da queste perline.
- Rimuovere le perle residue dall'RNA eluito riponendo il campione nel rack magnetico e trasferendo il fluido a un tubo di centrifugatura fresco e a bassa legatura di 0,5 mL.
- Mescolare il campione e poi far precipitare l'nsRNA aggiungendo 2,5x volumi di etanolo e mescolare ancora una volta. Lasciare agire per 1 h per una notte a -20 gradi centigradi. Ruotare in una centrifuga pre-raffreddata (4 gradi centigradi) per 20 min alla velocità massima (almeno 13.000 x g).
- Lavare accuratamente con 200 gradi l di 70% di etanolo a -20 gradi centigradi. Poiché il residuo di ME inibirà le applicazioni a valle, girare ad ogni passo per rimuovere la maggior parte della feccia possibile; alla fine il campione non deve prestare odore di .ME.
- Ri-dissolversi in 10-20 litri di DEPC-treated TE con l'equivalente di 0.005 -L RNase inibitore.
NOTA: tutte le fasi successive devono essere eseguite sul ghiaccio. - Misurare la concentrazione di RNA e la purezza.
- Misurare gli A260 e A225 in uno spettrofotometro a basso volume di campione.
NOTA: un massimo di assorbimento vicino a 225 nm proviene da un contaminabile proveniente dalle perline. In assenza di RNA il segnale proveniente dal contaminante diminuisce al 35% a 260 nm. Pertanto, la quantità effettiva di RNA è approssimata dalla formula: (A260-(A2250,35)) - In alternativa, analizzare il campione su un sistema di elettroforesi microfluidica come un bioanalisi.
NOTA: Questa analisi è preferibile all'uso di uno spettrofotometro in quanto è possibile valutare l'integrità dell'RNA, il contaminante non interferisce con la quantificazione ed è richiesto meno campione.
- Misurare gli A260 e A225 in uno spettrofotometro a basso volume di campione.
- Analizzare l'NSRNA.
NOTA: Ad esempio, gli RNA specifici possono essere quantificati con tecniche standard di trascrizione inversa qPCR. L'RNA preparato in questo modo è compatibile con la preparazione della libreria per l'RNA-seq. La rimozione del rRNA non è necessaria per l'etichettatura di tempi inferiori a 5 min.
Risultati
Le rese tipiche per nsRNA recuperate utilizzando questo protocollo ers4tU sono mostrate nella Figura 1b, questo è stato prodotto da un bioanalizzatore e la traccia mostra la resa di RNA contro dimensione (nucleotidi [nt]). Si noti, sia nel grafico della traccia bio-analizzata che in quella a incisione, il recupero dell'RNA dal punto di tempo 0 è una porzione molto piccola di quella recuperata da periodi di tempo più lunghi - circa 0,3 g di RNA recuperata da circa 109 cellule rispetto a oltre il doppio dopo soli 30 s di etichettatura (0,8 g di nsRNA) dallo stesso numero di cellule. Il recupero di RNA a 15 s è più variabile in quanto piccole differenze nell'esecuzione del campionamento hanno un effetto proporzionalmente maggiore sul recupero dell'RNA. Nella traccia bio-analizzatore, i precursori del rRNA possono essere visti come un picco vicino a 1000 nt e un doppietto di picchi a 1700-1800 nt. L'abbondanza di questi intermedi aumenta man mano che la tiola continua.
L'etichettatura Thio è stata utilizzata per quantificare lo splicing della trascrizione ACT1 (Figura 3). È stata eseguita la lite e sono stati prelevati campioni prelevati a intervalli di 15 s dall'inizio dell'etichettatura e dall'elaborazione dell'RNA ACT1 monitorato (Figura 3a,b). Come si può vedere, il pre-mRNA viene generato (per trascrizione) e le lariats (dal primo passo dello splicing dal pre-mRNA), anche dopo solo 15 s di etichettatura. Dopo circa 45 s a 1 min, le quantità di lariats e pre-mRNA raggiungono l'equilibrio con la maggior parte di queste specie di RNA create dalla trascrizione come vengono elaborate via dalla splicing.
Per produrre i dati mostrati in Figura 3c il ceppo è stato pulsato con 4tU per 25 s e poi inseguito con uridina. La generazione di pre-mRNA e lariati raggiunge un massimo a 1 minuto. Questo si confronta bene con la figura 3b; il massimo raggiunto dopo 45 s per raggiungere l'equilibrio più le 25 s dell'etichettatura. Dopo il picco, i livelli diminuiscono con gli RNA tio-etichettati vengono inseguiti attraverso il processo di giunzione.
La figura 3d mostra l'esaurimento di un fattore di giunzione delle proteine e il suo effetto sul metabolismo dell'RNA, utilizzando il sistema AID 4U6,7. Qui, Prp16p è ridotto da livelli quasi fisiologici al 5% di questo livello dopo 25 min di esaurimento. Prp16p è un fattore di giunzione essenziale per la seconda fase di giunzione15. I lariati vengono rimossi durante la seconda fase della giunzione (Figura 3a), ma qui aumentano al di sopra del livello di pre-mRNA come Prp16 diventa limitante. In tempi di esaurimento successivi, altri fattori diventano limitanti a causa di effetti secondari, in modo che i livelli di diminuzione lariat e i livelli di pre-mRNA aumentino. Il livello di mRNA spliced diminuisce.
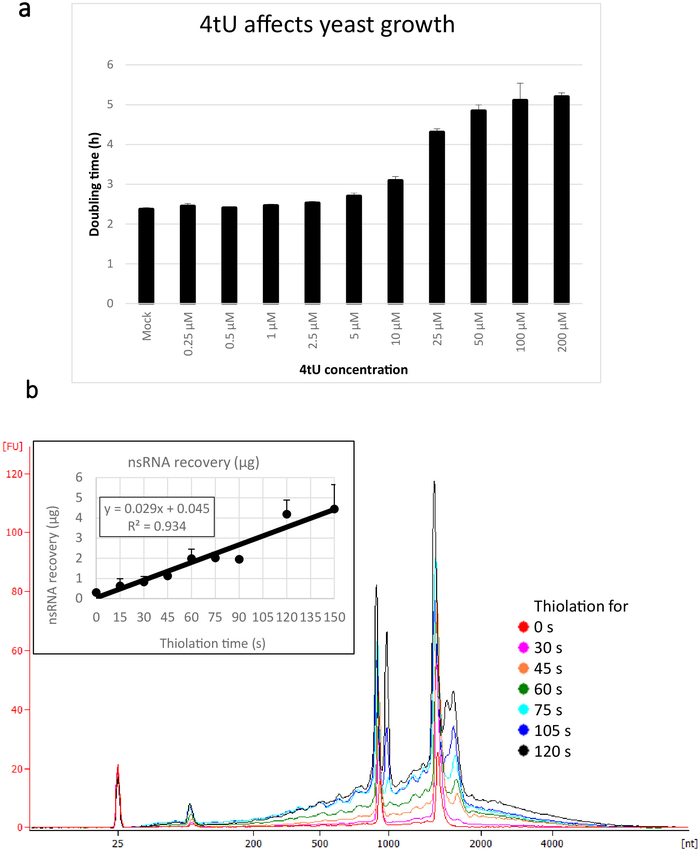
Figura 1: Crescita nel 4TU e nel recupero dell'RNA. (a) 4-thiouracil influisce sulla crescita. Aumentando la concentrazione di 4tU nel mezzo di crescita drop-out YMM senza uracil aumenta il tempo di raddoppio di S. cerevisiae (BY4741) portando il p4FuiPUp plasmide. La crescita di quattro colture di replica è stata monitorata a 30 gradi centigradi in un Tecan Infinite Pro 200. Tutte le impostazioni cultura erano in fase di log in tutto, con OD600 tra 0.1 e 0.6. Mock è una cultura di controllo con una quantità equivalente di NaOH aggiunto, che di per sé non cambia il tasso di crescita. Questo grafico dimostra che l'etichettatura tio è un compromesso tra l'etichettatura rapida e i danni alla cellula. Le barre di errore sono un errore standard di 4 repliche. (b) la resa di nsRNA aumenta linearmente da circa 15 s di etichettatura. La figura principale mostra le tracce bioanalizzate di nsRNA purificato da 0 (non tiolato) a 2 min dopo l'aggiunta di 4tU a intervalli di 15 s. Si noti che il campione 15 s non viene visualizzato, in quanto non è stato distinguibile dal campione senza etichetta. I due grandi picchi corrispondono agli RNA ribosomici (rRNA). I precursori di rRNA e gli intermedi sono visibili in quanto diversi picchi con un peso molecolare maggiore rispetto ai rRNA maturi. Il recupero di questi precursori e intermedi aumenta con il tempo. Vengono mostrati i risultati di un esperimento rappresentativo. Il grafico a instacono mostra il recupero di nsRNA con incubazione crescente con 4tU. La resa di nsRNA aumenta con l'aumento del tempo di crescita con 4tU. Il recupero è straordinariamente lineare (R2 - 0,934) durante la scala cronologica di questo esperimento e mostra un leggero aumento rispetto allo sfondo anche a 15 s etichettatura con 4tU anche se non distinguibile dal campione senza etichettato a occhio dal bio/analizzatore trovare. Le barre di errore mostrano un errore standard per tre repliche biologiche. Fare clic qui per visualizzare una versione più grande di questa figura.
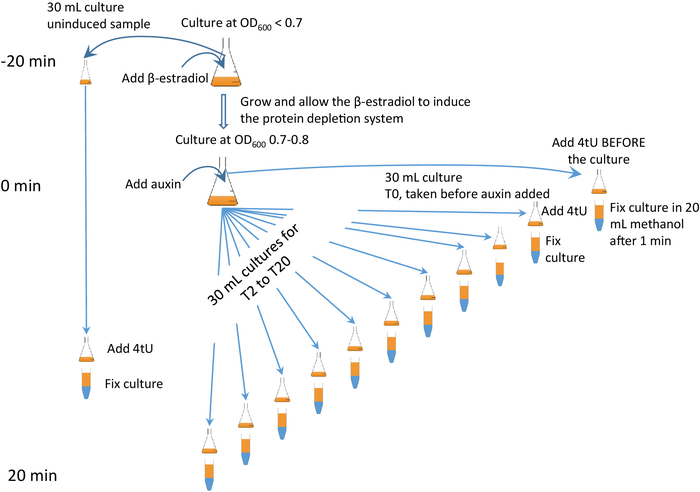
Figura 2: Protocollo grafico 8U 4U 4U AID 4U. Una sintesi grafica del protocollo del protocollo DI zest AID 4U. Il sistema di degron auxina (AID) che a sua volta esaurisce una proteina bersaglio marcata con AID, si riferisce a Barrass et al.7 per un protocollo dettagliato. In questo caso, l'espressione del sistema di degron viene avviata 25 minuti prima dell'inizio della degradazione delle proteine e la diiolazione in ogni punto temporale è per 1 min. Una versione animata viene visualizzata nella Figura 2 supplementare. Fare clic qui per visualizzare una versione più grande di questa figura.
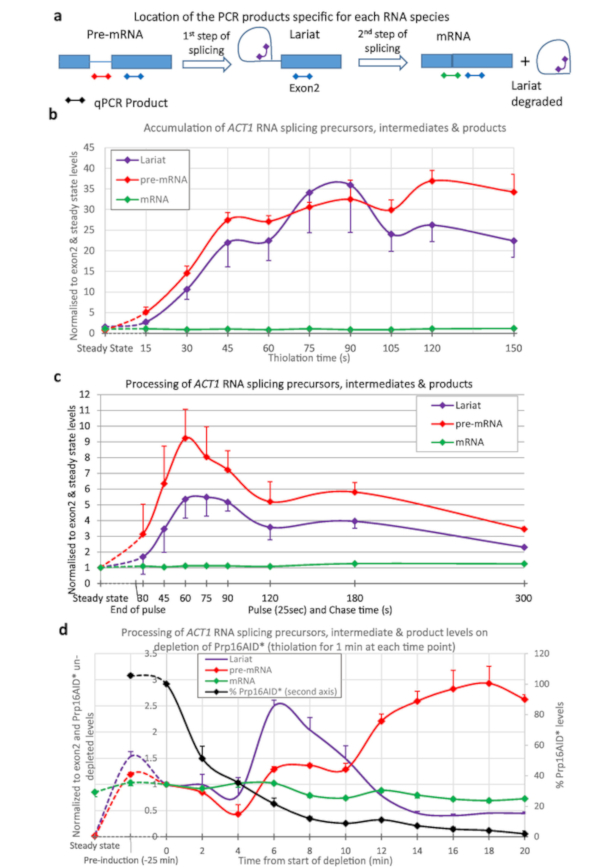
Figura 3: Precursori e intermedi dello splicing dell'RNA ACT1. L'aglicing delle trascrizioni pre-mRNA ACT1 è stato monitorato dalla trascrizione inversa quantitativa PCR16. I livelli di precursore di ACT1 (pre-mRNA), lariat-exon2 intermedio (Lariat) e prodotto spliced (mRNA) sono mostrati normalizzati rispetto al livello di ACT1 Exon2 e livelli di stato costante di questi RNA. (a) Posizione dei prodotti qPCR nella trascrizione ACT1. Schematico delle posizioni dei prodotti qPCR utilizzati per testare i livelli di precursori, intermedi e prodotti della reazione di giunzione delle trascrizioni ACT1 16. Gli esoni sono rappresentati da scatole, intron come una linea e i prodotti qPCR come linee con diamanti a entrambe le estremità, il colore corrisponde a quelli utilizzati nei grafici. La PCR pre-mRNA è specifica per il pre-mRNA e non per eventuali intermedi dello splicing in quanto questo prodotto attraversa il punto di diramazione che viene interrotto dopo il primo passaggio di giunzione. Lariat PCR rileverà il prodotto della prima fase di giunzione e la lariat eccitata prodotta dopo la seconda. L'mRNA PCR è specifico per il prodotto della giunzione, mRNA. I risultati della PCR esone (presenti in tutti i precursori, intermedi e prodotti, ad eccezione del lariato asforsato) non sono mostrati nei grafici in quanto questi sono stati utilizzati per normalizzare i dati ed è quindi sempre uguale a 1. (b) Tiolabelling continuo. La quantità di pre-mRNA aumenta con il tempo come 4tU è incorporata dalla trascrizione e, dopo un breve ritardo, lo splicing lo converte in prodotti intermedi e spliced lariat-els. I livelli di queste specie pre-mRNA e lariat sono rilevabili sopra lo sfondo dopo appena 15 s di crescita con 4tU e raggiungono un massimo dopo circa 45 s di etichettatura continua con 4tU, a quel punto la loro produzione è bilanciata dalla conversione in giunzione mRNA e/o degradazione. I valori vengono normalizzati allo stato stazionario (punto più a sinistra del grafico) e i livelli di exon 2 per mostrarne l'aspetto e l'elaborazione rispetto alla trascrizione dell'esone 2. Poiché lo splicing di RNA di ACT1 è in gran parte co-trascrizione4,17 mRNA spliced diventa rapidamente la specie più abbondante, il suo livello è simile a quello di exon 2. Errore standard di tre repliche biologiche, ognuna saggiata in triplice copia. (c) Impulso/inseguimento. Thiolation impulso di 25 secondi seguito da inseguimento con uridina. Rispetto ai livelli di stato costante di questi RNA (punto più a sinistra), sono inizialmente molto abbondanti nel pool appena sintetizzato. I livelli diminuiscono gradualmente man mano che vengono trasformati in mRNA (o degradati), avvicinandosi a livelli molto simili a quelli dello stato costante di 5 min. (d) nsRNA e esaurimento proteico. Splicing di trascrizioni pre-mRNA ACT1 monitorate dalla trascrizione inversa quantitativaPCR come nel pannello (a ) dopo l'esaurimento della proteina Prp16 utilizzando il sistema di degron di auxina come descritto nella Figura 2. I livelli di proteina Prp16 vengono visualizzati anche nel grafico tracciato rispetto al secondo asse Y come percentuale dei livelli precedenti all'esaurimento dell'auxina. La Prp16 è una componente vitale dello spliceosome, particolarmente importante perla seconda fase di giunzione mostrata nel pannello (a ), dopo di che i lariati vengono degradati. Quando questo passaggio diventa lariats limitante si accumulano inizialmente. In momenti successivi lo splicing fallisce completamente, i lariati non vengono più prodotti e i livelli pre-mRNA aumentano. Le barre di errore sono un errore standard di tre repliche biologiche, ognuna delle quali è stata saggiata in triplice copia. Fare clic qui per visualizzare una versione più grande di questa figura.
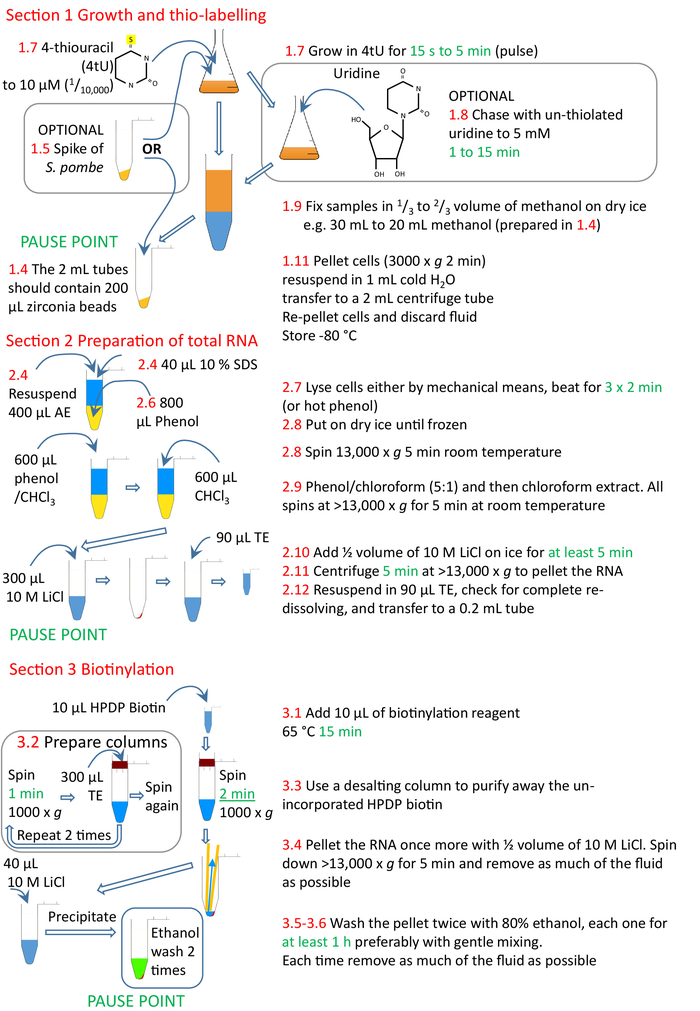
Figura 4: Riepilogo grafico delle sezioni di protocollo da 1 a 3. Le cellule sono tiolate con 4tU e hanno permesso di crescere per incorporare il nucleotide modificato nell'RNA. È possibile aggiungere un picco S. pombe tiolato per consentire la normalizzazione attraverso punti temporali ed esperimenti. L'impulso di 4tU può essere inseguito utilizzando uridina non thiolated. L'etichettatura può essere eseguita continuamente dall'aggiunta di 4tU o da un cambiamento alle condizioni di crescita, la divisione culturale e 4tU viene aggiunta alle culture in tempi crescenti a partire dal cambiamento della condizione di crescita, ma ogni etichettatura solo per un breve periodo di tempo. Le cellule vengono raccolte e l'RNA preparato dalle cellule, preferibilmente utilizzando un omogeneizzatore e metodi a base di fenolo. L'RNA è biotinylato e quindi l'RNA biotinylato purificato dalla biotina non incorporata utilizzando una colonna di esclusione delle dimensioni. Il nsRNA è ora pronto per la purificazione con perline di streptavidina (sezione 4, Figura 5). I numeri in rosso corrispondono ai numeri di passo nel protocollo. Fare clic qui per visualizzare una versione più grande di questa figura.
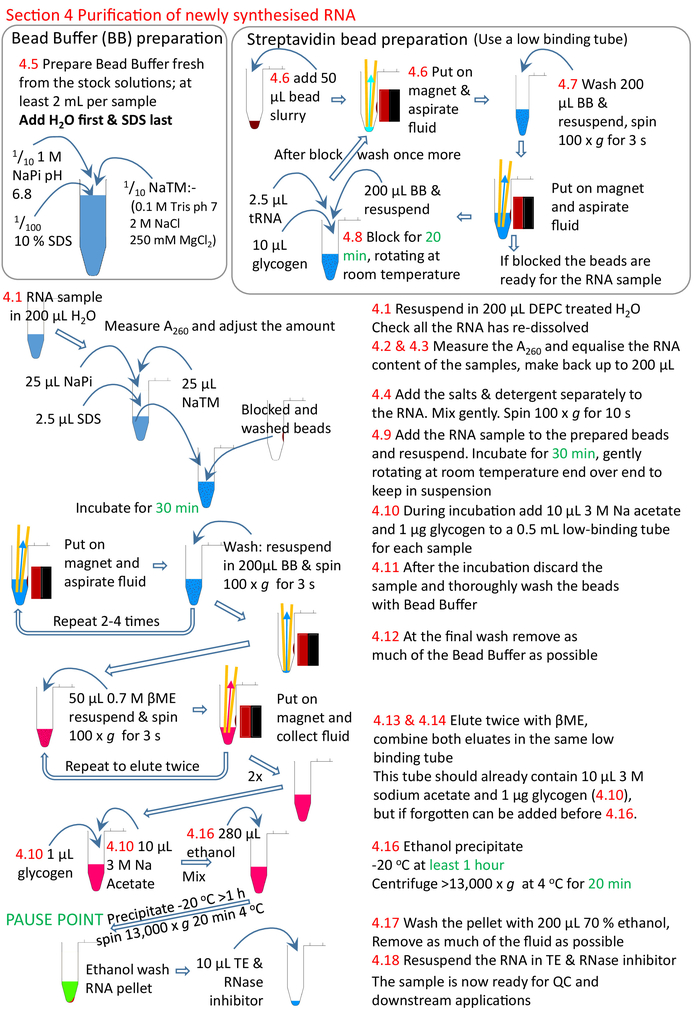
Figura 5: Riepilogo grafico della sezione 4 del protocollo. A seguito delle sezioni da 1 a 3 (Figura 4), le perle di streptavidina vengono bloccate e il campione di RNA biotinylato viene aggiunto alle perline preparate. L'RNA biotinylato si lega alle perle di streptavidina e all'RNA non biotinylated rimosso e lavato. L'RNA biotinylato viene eluito dalle perline utilizzando il file .ME e precipitato pronto per ulteriori ricerche. I numeri in rosso corrispondono ai numeri di passo nel protocollo. Fare clic qui per visualizzare una versione più grande di questa figura.
Figura supplementare 1: Miglioramento del recupero di nsRNA da cellule di lievito con e senza copie aggiuntive dell'importatore a 1 e 3 minuti di tio-etichettatura. Si noti che Fui1 è il promotore del lievito espresso da un plsmide di 2 m. La copia genomica di questo gene è presente in entrambi questi ceppi. Fare clic qui per scaricare questo file.
Supplementare Figura 2: Versione animata del protocollo grafico AID 4U 4U 4U. Fare clic qui per scaricare questo file.
File supplementare 1: 4tU_experiment_template.xltx. Fare clic qui per scaricare questo file.
| Nome Plasmid | Importatore/permeasi | pennarello | commento | |
| p4Fui | S. cerevisiae Fui1 | URA3 | Fui1 importa Uracil e Uridina, rendendolo ideale per esperimenti pulse/chase. | |
| pAT2 | S. cerevisiae Fui1 | LEU2 | ||
| p4Fui -- PEST | S. cerevisiae Fui1 | URA3 | Il motivo PEST di Fui1 è stato disattivato, quindi la permeasi non è degradata quando c'è sufficiente uracile intracellulare per le esigenze della cellula. Funziona bene negli esperimenti di etichettatura e migliora le prestazioni di impulso/inseguimento. | |
| p4Fur | S. cerevisiae Pelliccia4 | URA3 | Uracil permeasi | |
| YEpEBI311 | H. Sapiens ENT1 | LEU2 | Miller etal. Contiene anche un gene della chinasi della timina. | |
| (trasportatore nucleoside equilibrato) | ||||
| Tutti i plasmidi sono a base di 2 m. Tutti i plasmidi p4 e pAT sono basati sul pRS16 serie di plasmidi. FUI1 e FUR4 sono espressi dai propri promotori endogeni. | ||||
Tabella 1: Plasmidi utilizzati con questo protocollo.
Discussione
Questo articolo presenta un protocollo per l'etichettatura 4tU estremamente rapida e specifica, per il recupero dell'RNA nascente e sintetizzato da S. cerevisiae dopo appena 15 s di etichettatura, con contaminazione molto bassa da RNA senza etichetta.
L'utente deve sempre fare attenzione a mantenere l'integrità dell'RNA mediante l'uso di temperature fredde e reagenti trattati con DEPC. La purificazione del tallone Streptavidin è generalmente affidabile; tuttavia, il buffer di perline è difficile da gestire; deve essere fatto fresco, con i suoi componenti aggiunti nel giusto ordine, e non refrigerati o autoclaved. Le carenze più comuni includono l'incompleto dissolvimento dell'RNA dopo le fasi di precipitazione, e quindi non essendo biotinylato o altrimenti perso durante le fasi di lavorazione. C'è un ampio aiuto per la risoluzione dei problemi nel materiale supplementare.
Ci sono alcune limitazioni di cui essere a conoscenza in ers4tU. Uno già menzionato è che 4tU rallenta la crescita del lievito (Figura 1a). A parte gli RNA endogenamente tiolati9, solo gli RNA che sono stati trascritti durante il periodo di etichettatura possono essere purificati con questo metodo. Le polimerasi si sono fermate sui geni per tutto il tempo di tiolazione non produrranno trascrizioni tiolate che possono essere purificate, anche se le trascrizioni che sono parzialmente etichettate a causa di polimerasi che entrano o lasciano uno stato di pausa durante la diiolazione possono essere recuperate. I ceppi che trascrivono male, a causa di mutazioni o condizioni di crescita, producono poco nsRNA, anche se le tecniche qui utilizzate miglioreranno comunque il recupero di nsRNA rispetto ad altri metodi. In questi ceppi e condizioni possono essere necessari tempi più lunghi e volumi di coltura più elevati. Si noti che l'uracile è una buona fonte di azoto e quindi questo metodo dovrebbe essere sperimentato prima di essere utilizzato per studi che coinvolgono la fame di azoto.
Il protocollo ers4tU è particolarmente utile per l'analisi degli RNA di breve durata, molti dei quali sono così rapidamente degradati che non possono essere identificati senza paralizzare i meccanismi di degradazione. Gli esempi includono trascrizioni criptiche instabili (CUT)4e brevi trascrizioni prodotte dalla terminazione prematura o promotrice che si ferma18 e la trascrizione antisenso "a monte" da un promoter (PROMPT)19. Anche gli intermedi prodotti durante la lavorazione di specie di RNA stabili sono transitori, ma possono essere arricchiti utilizzando la trascrizione 4tU4. Il protocollo ers4tU è quindi eccezionale nel permettere alle specie di RNA altamente transitorie di essere analizzate e catturate in condizioni quasi fisiologiche, il che è un enorme vantaggio rispetto ad altri metodi. Questa tecnica è stata utilizzata per studiare la trascrizione e le cineci di elaborazione dell'RNA a valle nei mutanti di rna polimerasi che si allungano più velocemente o più lentamente del normale20.
Thiolation è anche compatibile con RNA-seq e SLAM-seq21, permettendo a tutto l'RNA prodotto in un tempo molto breve di essere caratterizzato in dettaglio squisito.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dal finanziamento Wellcome per JB [104648]. Il lavoro nel Wellcome Centre for Cell Biology è supportato dal finanziamento del nucleo del Wellcome [092076]. Gli autori riconoscono i membri del laboratorio per il loro aiuto: Bella Maudlin, Emanuela Sani, Susanna De Lucas-Arias e Shiney George. Gli autori ringraziano anche Patrick Cramer per il plasmide YEpEBI31111.
Materiali
| Name | Company | Catalog Number | Comments |
| β-mercaptoethanol (βME) | Sigma-Aldrich | M3148 | CAUTION toxic. Stock solutions are aproximatly 14 M, make at 1/20 dilution for use |
| Chloroform | Sigma-Aldrich | 25668 | CAUTION toxic |
| Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758 | add 1/1000 volume to a solution, leave at room temperature for 24 h, then autoclave |
| DMF (N,N-dimethylformamide) | Sigma-Aldrich | 227056 | CAUTION toxic |
| EDTA | Sigma-Aldrich | 3609 | Make 0.5 M and pH to 8.0 with sodium hydroxide |
| Ethanol | Sigma-Aldrich | 29221 | |
| EZ-link HPDP Biotin | Thermo scientific | 21341 | Store protected from light. Disolve all the vial contents in 22.7 mL DMF (to make a 4 mM stock solution). Store away from water, in the dark & at -20 °C. Check the solution before using, as some batches of HPDP precipitate in storage; heat at 42 °C to resuspend. |
| Glucose | Fisher Scientific | G/0500/60 | |
| Glycogen [20 mg/mL] | Sigma-Aldrich | 10901393001 | Store at -20 °C |
| Immobilised TCEP Disulfide Reducing Gel | Thermo Scientific | 77712 | Optional |
| LiCl | Sigma-Aldrich | 793620 | 10 M solution. CAUTION: this gets very hot as is dissolves and can even boil at greater than 100 oC, add the LiCl crystals to the water slowly. |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 63033 | 1 M solution. CAUTION: this gets very hot as is dissolves and can even boil at greater than 100 oC, add the MgCl2 crystals to the water slowly. |
| Methanol | Fisher Scientific | M/4000/PC17 | CAUTION Toxic and flammable |
| NaH2PO4 | Sigma-Aldrich | S3139 | Make 1 M solutions of each and mix in equal amount to obtain a solution of the appropriate pH |
| Na2HPO4 | Sigma-Aldrich | S3264 | |
| NaCl | Sigma-Aldrich | S9888-M | 5 M solution |
| Phenol, low pH. | Sigma-Aldrich | P4682 | Store in the dark at 4 °C. CAUTION toxic |
| Phenol Chloroform 5:1 (125:24:1) low pH. | Sigma-Aldrich | P1944 | Store in the dark at 4 °C. CAUTION toxic |
| Pierce Spin Columns | Thermo Scientific | 69702 | Optional |
| SCSM single drop-out –ura | Formedium | DSCS101 | |
| Sodium Acetate | Sigma-Aldrich | 32318-M | Make a 3 M solution and pH to 5.3 with acetic acid |
| Sodium hydroxide | Sigma-Aldrich | 795429 | CAUTION corrosive |
| SDS (Sodium dodecyl sulfate) | Sigma-Aldrich | 436143 | CAUTION irritant, do not inhale |
| Streptavidin Magnetic beads | NEB | 1420S | Store at 4°C |
| SUPERase-In, RNase inhibitor | Life technologies | AM2696 | Store at -20°C |
| Thiolated Schizosaccharomyces pombe for spike | See section 1.7 of the protocol | ||
| 4-thiouracil (4tU) | ACROS ORGANICS | 359930010 | Store in the dark. Make 100 mM Stock in 1M NaOH, store solutions at -20°C. |
| Tris base | Sigma-Aldrich | 93362 | 1 M solutions at various pH |
| tRNA | Sigma-Aldrich | 10109541001 | 5mg/ml, store at -20°C |
| Uridine | Sigma-Aldrich | U3750 | Make 1 M solution in H2O. Split into 2 mL aliquots and store at -20 C. |
| Yeast nitrogen base without amino acids with amonium sulphate | Formedium | CYN0410 | |
| Zeba Columns 0.5ml | Thermo Scientific | 89882 | Store at 4 °C |
| Zirconia beads | Thistle Scientific | 110791052 | |
| Equipment and Consumables | |||
| Beadbeater | Biospec | 112011EUR | Other homogenisers can be used; the correct conditions for each homogeniser and strain must be established. |
| Bioanalyser (Agilent) or similar to assess RNA quality. If this is not important a spectrophotometer is useful to quantify the RNA. | |||
| Centrifuge: capable of spinning cultures at 4 °C and at least 3000 g. Pre-chill if possible. | |||
| Centrifuge: capable of spinning up to 2 mL tubes at variable speeds upto 13,000 g and down to 1000 g | |||
| Magnetic rack for separating the beads from the sample. The one used in the paper is 3D printed, available from Thingiverse (thing:3562952). Comercially available racks exist | |||
| PCR machine with a heated lid that will allow incubation in the dark. | |||
| Rotating wheel to rotate 1.5 mL tubes end over end | |||
| Shaking heating block (such as Eppendorf Thermomixer) is recomended | |||
| Tubes, centrifuge, Low retention, RNase free 0.5mL | Eppendorf | H179467N | |
| Tubes, centrifuge, Low retention, RNase free 1.5mL | Ambion | AM12350 | |
| Tubes, centrifuge, 50 mL | Sarstedt | 62.547.004 | Other centrifuge tubes are not gas proof allowing CO2 to disolve in the methanol, this comes out of solution vigorously on adding warm culture, leading to sample loss |
| Tubes, centrifuge, 15 mL | Sarstedt | 62.554.001 | |
| Tubes, 2 mL, screw cap | Greiner | 723361 | |
| Tubes 0.2 mL strip of 8 with integral lids | Brand | 781332 |
Riferimenti
- Duffy, E. E., Schofield, J. A., Simon, M. D. Gaining insight into transcriptome-wide RNA population dynamics through the chemistry of 4-thiouridine. Wiley Interdisciplinary Reviews: RNA. 10 (1), e1513(2018).
- Windhager, L., et al. Ultrashort and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Research. 22, 2031-2042 (2012).
- Baptista, T., Devys, D. Saccharomyces cerevisiae Metabolic Labeling with 4-thiouracil and the Quantification of Newly Synthesized mRNA As a Proxy for RNA Polymerase II Activity. Journal of Visualized Experiments. (140), e57982(2018).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282(2015).
- Burger, K., et al. 4-thiouridine inhibits rRNA synthesis and causes a nucleolar stress response. RNA Biology. 10, 1623-1630 (2013).
- Mendoza-Ochoa, G. I., et al. A fast and tuneable auxin-inducible degron for depletion of target proteins in budding yeast. Yeast (Chichester England). 36 (1), 75-81 (2018).
- Barrass, J. D., Mendoza-Ochoa, G. I., Maudlin, I. E., Sani, E., Beggs, J. D. Tuning degradation to achieve specific and efficient protein depletion. Journal of Visualized Experiments. , (2019).
- Hobro, A. J., Smith, N. I. An evaluation of fixation methods: Spatial and compositional cellular changes observed by Raman imaging. Vibrational Spectroscopy. 91, 31-45 (2017).
- Gustilo, E. M., Vendeix, F. A. P., Agris, P. F. tRNA's Modifications Bring Order to Gene Expression. Current Opinion in Microbiology. 11, 134-140 (2008).
- Dolken, L., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14, 1959-1972 (2008).
- Miller, C., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Molecular Systems Biology. 7, 458(2011).
- Schmitt, M. E., Brown, T. A., Trumpower, B. L. A rapid and simple method for preparation of RNA from Saccharomyces cerevisiae. Nucleic Acids Research. 18, 3091-3092 (1990).
- Rädle, B., et al. Metabolic Labeling of Newly Transcribed RNA for High Resolution Gene Expression Profiling of RNA Synthesis, Processing and Decay in Cell Culture. Journal of Visualized Experiments. (78), e50195(2013).
- Herzog, V. A., et al. Thiol-linked alkylation of RNA to assess expression dynamics. Nature Methods. 14, 1198-1204 (2017).
- Ohrt, T., et al. Molecular dissection of step 2 catalysis of yeast pre-mRNA splicing investigated in a purified system. RNA. 19, 902-915 (2013).
- Alexander, R. D., et al. RiboSys, a high-resolution, quantitative approach to measure the in vivo kinetics of pre-mRNA splicing and 3′-end processing in Saccharomyces cerevisiae. RNA. 16, 2570-2580 (2010).
- Wallace, E. W. J., Beggs, J. D. Extremely fast and incredibly close: cotranscriptional splicing in budding yeast. RNA. 23, 601-610 (2017).
- Adelman, K., Lis, J. T. Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nature Reviews Genetics. 13, 720-731 (2012).
- Preker, P., et al. RNA Exosome Depletion Reveals Transcription Upstream of Active Human Promoters. Science. 322, 1851-1854 (2008).
- Aslanzadeh, V., Huang, Y., Sanguinetti, G., Beggs, J. D. Transcription rate strongly affects splicing fidelity and cotranscriptionality in budding yeast. Genome Research. 28, 203-213 (2018).
- Schofield, J. A., Duffy, E. E., Kiefer, L., Sullivan, M. C., Simon, M. D. TimeLapse-seq: adding a temporal dimension to RNA sequencing through nucleoside recoding. Nature Methods. 15, 221-225 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon