Method Article
Rotulagem metabólica extremamente rápida e específica do RNA in vivo com 4-Thiouracil (Ers4tU)
Neste Artigo
Resumo
O uso do uracil swcnts a purificar sensitivamente e especificamente o RNA recentemente transcrito do fermento Saccharomyces cerevisiae.
Resumo
O análogo do nucleotide, 4-thiouracil (4tU), é tomado prontamente pelas pilhas e incorporado no RNA porque é transcrito in vivo, permitindo o isolamento do RNA produzido durante um breve período de rotulagem. Isto é feito anexando um metade do biotina ao grupo incorporado do Thio e à afinidade que purifying, usando grânulos revestidos estreptavidina. Conseguir um bom rendimento do RNA puro, recentemente sintetizado que é livre do RNA pre-existing faz uns tempos mais curtos da rotulagem possível e permite a definição temporal aumentada em estudos cinéticos. Este é um protocolo para a purificação muito específica, elevada do rendimento do RNA recentemente sintetizado. O protocolo aqui apresentado descreve como o RNA é extraído da levedura Saccharomyces cerevisiae. Entretanto, o protocolo para a purificação do RNA swcnts do RNA total deve ser eficaz usando o RNA de todo o organismo uma vez que estêve extraído das pilhas. O RNA purificado é adequado para análise por muitas técnicas amplamente utilizadas, tais como transcriptase reversa-qPCR, RNA-Seq e SLAM-Seq. A especificidade, a sensibilidade e a flexibilidade desta técnica permitem introspecções inigualáveis no metabolismo do RNA.
Introdução
O RNA tem uma natureza dinâmica; logo após ele é produzido muito RNA é rapidamente processado e degradado. Atualmente, a maioria de estudos do metabolismo do RNA analisam o RNA celular total, que é processado na maior parte inteiramente e no nível do estado estacionário. Este nível depende do equilíbrio entre as taxas de transcrição, maturação pós-transcricional e degradação. A análise dos processos que levam ao equilíbrio do estado estacionário requer técnicas especializadas para captar espécies de RNA de vida muito curta.
A rotulagem metabólica de RNA com análogos de nucleotídeo, como 4-thiouracil (4tU) ou 4-tiouridina (4sU) (ver Duffy et al.1 para uma excelente revisão), oferece a capacidade de isolar os RNAs nascentes rotulados por Thio e seus intermediários de processamento. No entanto, os protocolos publicados envolvem tempos de rotulagem de vários minutos2,3, queé lento em relação à taxa de produção de muitas transcrições. Toma na ordem de um minuto para transduir o gene médio do fermento, assim que etiquetar o RNA do fermento para menos de um minuto pode ser considerado extremamente curto. O protocolo de 4 tiouracila extremamente rápido e específico (ers4tU) maximiza a relação sinal/ruído maximizando a incorporação de 4tu e minimizando a recuperação de RNA não rotulado, pré-existente, tornando os tempos de rotulagem muito curtos possíveis4.
A base Thio-modificada deve ser importada nas pilhas ràpida e na quantidade suficiente para etiquetar eficientemente o RNA recentemente sintetizado (nsRNA). Para promovê-lo, as células são cultivadas em meio livre de uracilo e a expressão de uma Permease adequada ajuda a impulsionar a captação de 4tU ou 4sU (ver tabela 1 para uma lista de plasmíos que carregam genes de Permease adequados e complementar Figura 1). a solubilidade do 4o em hidróxido de sódio evita a necessidade de solventes orgânicos tóxicos exigidos por outros análogos de nucleotídeo. Infelizmente, culturas crescentes por longos períodos com nucleosídeos Thio-modificados em concentrações superiores a 50 μM têm sido observadas para interromper os ribossomas5. No entanto, a concentração (10 μM) utilizada aqui, e os tempos de rotulagem extremamente curtos, minimizam os efeitos deletérios5 (Figura 1a), enquanto ainda produzindo RNA suficiente para análise.
Esta técnica pode ser combinada com a depleção rápida e específica de uma proteína alvo, mediada por auxina6,7( Figura 2), referida como o protocolo "β-est Aid 4U", no qual o β-estradiol regulou a expressão da auxina o sistema induzível degron (Aid) é combinado com a rotulagem 4tu. Com a abordagem β-est AID 4U, uma proteína alvo pode ser esgotada e o efeito sobre o metabolismo do RNA é monitorado de perto (Figura 2). O timing é crítico; é aconselhável ver o vídeo que acompanha e prestar muita atenção à Figura 2 e sua forma animada (ver Figura complementar 2).
O processamento e a degradação do RNA devem ser parados extremamente ràpida para a definição exata do tempo. Isto é conseguido usando metanol em baixa temperatura, que corrige o conteúdo da célula muito rapidamente e degrada a membrana celular, preservando o teor de ácido nucleico8. A extração do RNA deve ser eficiente e não danificar o RNA. O lysis mecânico é eficaz na ausência de agentes agente (frequentemente estes contêm grupos do Thio, assim que deve ser evitado). A precipitação do cloreto de lítio do RNA é preferida, porque os tRNAs são precipitados menos eficientemente. tRNAs são transcritas rapidamente e naturalmente swcnts9, assim que removendo tRNAs reduz a competição para o reagente do biotinilação. Se os RNAs pequenos, altamente estruturados são do interesse, os métodos álcool-baseados da precipitação do RNA são recomendados.
Para recuperar o RNA swcnts, a biotina é unida covalentemente através dos grupos do Thio incorporados no RNA com 4tu. O uso de biotina modificada, que atribui através de uma ligação dissulfeto ligante (por exemplo, hpdp-biotina (N-[6-(biotinamido)hexyl]-3 ́-(2 ́-pyridyldithio)propionamide,) ou MTS-biotina (metano thiosulfonate)) recomenda-se como permite a liberação do RNA pela adição de um agente de diminuição. O RNA biotinylated é afinidade purified no estreptavidina acoplado aos grânulos magnéticos. Este protocolo é semelhante a outros listados anteriormente10 , mas foi intensivamente otimizado para reduzir o fundo.
Existem dois tipos de experimento de rotulagem de tiol que podem ser realizados, rotulagem contínua e descontínua. Cada um tem suas próprias vantagens. Na rotulagem contínua o 4tU é adicionado à cultura e amostras colhidas em intervalos regulares. Este tipo de experimento mostra como o RNA é processado e como os níveis mudam ao longo do tempo. Exemplos incluem comparação de mutantes com experimentos de tipo selvagem e um experimento de perseguição por impulsos. Os experimentos mostrados na Figura 3B, c são desse tipo. Para a rotulagem descontínua uma mudança é induzida no sistema e no RNA monitorado. Uma vez que a mudança foi induzida a cultura deve ser dividida em várias subculturas, e em momentos específicos, cada um é então Thio-rotulado por um breve período. Um exemplo é β-est AID 4U mostrado na Figura 27. Este tipo de experimento é particularmente útil para monitorar o efeito de uma alteração metabólica no processamento do RNA (ver Figura 3D).
Uma representação gráfica de um experimento de rotulagem de Thio é apresentada na Figura 4 e na Figura 5, e uma planilha que simplifica muito o desempenho do protocolo está disponível (consulte modelo de experimento 4tu. xlsx). Assim como este a informação suplementar contem um guia de Troubleshooting extensivo. Para o protocolo β-est AID 4U que integra a rotulagem 4tU com o protocolo de depleção de auxina, ver Figura 2 e Figura complementar 2. Ver Barrass et al.7 para o protocolo detalhado de DEPLEÇÃO de auxílios.
Protocolo
1. crescimento e rotulagem de Thio
Nota: a hora para a conclusão desta seção do protocolo é altamente variável, dependendo da taxa de crescimento da pilha. Permitir que 1 h Prepare as soluções e equipamentos antes da rotulagem e 30 min pós-rotulagem para processar amostras.
- Assegure-se de que a cepa S. cerevisiae contenha um plasmídeo que codifica uma Permease (tabela 1) para impulsionar a importação de 4tu para a célula.
Nota: sem um importador, é improvável que a rotulagem por menos de 2 min seja bem-sucedida11 (ver Figura 1 suplementar). a incorporação de 4tU é mais eficiente se o crescimento estiver em meio sem uracil, de modo que a cepa deve ser URA3+; diversos dos plasmídeos na tabela 1 carreg URA3 como o marcador. Se este protocolo for combinado com a depleção de ajuda β-est7, são necessárias modificações de deformação adicionais. - Prepare o meio livre de uracilo YMM adicionando 6,9 g de base de nitrogênio de levedura sem aminoácidos, 20 g de glicose e 1,92 g de SCSM única gota-saída − ura (tabela de materiais) a 1 L de água. Autoclave ou filtro esterilizar o meio de crescimento antes de usar.
Nota: a esterilização do filtro é preferida como peptídeos/complexos de açúcar produzidos por autoclavagem de coprecipitado com as células do metanol utilizados na coleta de amostras. - Cresça o fermento no meio uracil-livre de YMM a uma densidade ótica em 600 nanômetro (OD600) de 0.6 − 0.8. Assegure-se de que a cultura esteja no crescimento da fase do registro e tenha sido pelo menos duas duagações. O crescimento a 30 ° c é normalmente recomendado, mas outras temperaturas podem ser usadas, por exemplo, para cepas sensíveis à temperatura.
Nota: dependendo da estirpe, condições de crescimento e rendimento de RNA, serão necessários aproximadamente 30 mL de volume de amostra. Este montante será assumido em todo o protocolo. 30 mL de cultura é o mais que vai caber em um tubo de centrífuga 50 mL com 20 mL de metanol, por isso é um volume conveniente para iniciar a otimização. Considere o uso de mais volume de amostra para os pontos de tempo precoce para aumentar a recuperação de RNA, até 2000 mL tem sido usado para células em crescimento mais lento em tempos de rotulagem muito curtos (< 1 min). - Chill cerca de 50 mL de H2O no gelo. Para cada amostra, adicione 200 μL de grânulos de zircônia a um tubo de tampão de rosca de 2 mL e relaxe no gelo. Coloque também 20 mL de metanol (precaução), em tubos de centrifugação de 50 mL, e coloque no gelo seco (precaução). O metanol deve ser 1/3 a 2/3 o volume da amostra.
PRECAUÇÃO: o metanol é tóxico por inalação, contacto e consumo. Dispense grandes volumes em uma capa de fumaça, e usar dois pares de luvas, como metanol pode penetrar luvas de laboratório de nitrilo. Metanol é altamente inflamável, manter afastado de todas as fontes de ignição.
Nota: como o gelo seco pode causar queimaduras frias no contato e produz o gás asphixiant, luvas do uso ao segurar e usar-se em um espaço bem ventilado.
Nota: adicionando as contas neste ponto é mais fácil do que depois que a amostra foi adicionada como o tubo está seco e ao girar para baixo a pelota da pilha as esferas são giradas igualmente desobstruídas da linha do tubo que conservam alguma hora. Adicionalmente, isto permite que os grânulos esfriem antes que a amostra esteja adicionada. - Se um ponto s. pombe deve ser adicionado à cultura (em vez de mais tarde), descongelar uma alíquota de células de s. pombe swcnts no gelo e Vortex completamente, pelo menos 30 s, em seguida, adicionar à cultura. Se preparado de acordo com as instruções abaixo, uma alíquota S. pombe é suficiente para 400 ml de cultura (suficiente para 12 30 ml amostras mais um pouco para permitir erros no manuseio). Se mais ou menos cultura for usada, ajuste o volume de S. pombe adicionado à cultura.
- Cresça 1 L de cultura de s. pombe para OD600 a 0,8 exatamente como descrito no protocolo para S. cerevisiae.
- Thio-Label como passo 1,7, mas por 10 min.
- Corrija toda a cultura usando 400 mL de metanol em gelo seco, essencialmente conforme descrito na etapa 1,9.
- Pellet as células por centrifugação em 3000 x g por 3 min.
- Descarte o sobrenadante e ressuscitem o pellet celular em 3,3 mL de H2o.
- Divida em alíquotas de 80 μL cada. Conservar a-80 ° c.
- Utilize toda uma alíquota para 400 mL de cultura ou 10 μL por amostra de 30 mL.
Nota: Reduza o volume de pico para 1/10 se executar rnaseq. Não reutilizar alíquotas; descartar qualquer espiga não utilizada. Esse pico é útil para normalizar e comparar resultados entre pontos de tempo e experimentos.
- Para a rotulagem descontínua, induzir a perturbação metabólica necessária (por exemplo, condições de crescimento, indução gênica ou depleção como β-est AID7 (Figura 2 e Figura 2 complementar), em seguida, dividir a cultura. Assegure-se de que todas as garrafas e suportes estejam à temperatura desejada e, se possível, arejar o meio antes de adicionar a cultura.
- Adicione 4tU à cultura a uma concentração de 10 μmM e misture vigorosamente (1/10.000 do volume de cultura de 100 mm 4Tu dissolvido em 1 M NaOH). Tiol-etiqueta para 15 s a 5 min.
Nota: trinta segundos é um bom ponto de partida. A rotulagem de Thio para menos de 20 s dá resultados mais variáveis devido às dificuldades que manipulam a cultura a pressão de tempo. No entanto, a rotulagem por mais de 1 min reduz a resolução temporal da técnica. - Se um experimento de perseguição deve ser executado; permitir a etiquetagem por 20 − 30 s, em seguida, perseguir adicionando 1/200 volume de cultura de 1 M de uridina (não thiolated), a uma concentração final de 5 mm.
Nota: uridina é preferível ao uracil para a perseguição, porque o uridina é mais solúvel em água permitindo que um volume menor seja adicionado à cultura e assim que há menos distúrbio ao crescimento das pilhas. - Tome amostras de cultura em intervalos regulares (pelo menos 15 s), até o final do curso do tempo. Intervalos de amostragem mais curtos do que isso são difíceis de executar de forma confiável. Adicione a amostra ao metanol em gelo seco preparado na etapa 1,4. Para maior comodidade, adicione 30 mL de cultura a um tubo de 50 mL contendo 20 mL de metanol.
Nota: o dióxido de carbono dissolve-se no metanol quando o frio; isto sai da solução na adição da amostra e espumas vigorosamente em cima da mistura ― tendo por resultado a perda da amostra. Para evitar isso, Chill o metanol para <-70 ° c em um tubo firmemente selado até perto do tempo que é necessário, em seguida, transferir para gelo seco. - Sele o tubo e misture completamente agitando. Coloque as amostras no gelo. Verifique se nenhuma das amostras congelou; Se assim for, suavemente quente na mão, invertendo constantemente. Isto é feito melhor na mão como a temperatura da amostra pode ser avaliada, ele deve sempre sentir frio. Coloque no gelo. Este não é um ponto de pausa; uma vez que toda a amostra é fluido prossiga para o próximo passo.
- Girar em 3000 x g por 2 min (a 4 ° c, se possível) para pellet as células. Despeje o líquido e ressuscite o pellet em pelo menos 1 mL de gelo-água fria, delicadamente pipetando para cima e para baixo.
Nota: o metanol residual na amostra da pelota auxilia a ressuscição. - Transfira para os tubos de 2 mL de tampão de rosca, conforme preparado no passo 1,4. Gire brevemente (por exemplo, 10 s de tempo total) em > 13000 x g para re-pellet as células, coloque de volta no gelo e remover o líquido.
Nota: a pelota da pilha pode ser armazenada em-70 a-80 ° c por diversos meses.
2. preparação do RNA total
Nota: o tempo de conclusão é 90 min.
- Use soluções tratadas com pirocarbonato de dietilo (DEPC) para proteger o RNA da degradação. Aliquot as soluções usando pontas da pipeta do filtro e luvas do desgaste em todas as vezes.
- Para uma solução Adicionar 1/1000 volume de DEPC e misture por agitação vigorosa.
- Deixar em temperatura ambiente (RT) para 24 h, em seguida, autoclave.
- Soluções com grupos de amina (como Tris) não podem ser tratadas com DEPC. Aliquot o pó e armazene especialmente para o trabalho do RNA. Use anteriormente DEPC tratados H2o para fazer a solução.
Nota: como o Thio-grupo no RNA é photoactivatable, minimize a exposição à luz UV deste ponto sobre. Armazenamento deve ser no escuro e incubação é melhor feito em uma máquina de PCR com uma tampa.
- Se um ponto de S. pombe deve ser adicionado à pelota da pilha um pouco do que a cultura (não faça ambos), adicione-o agora. Descongelar uma alíquota de células de S. pombe swcnts no gelo e Vortex completamente, pelo menos 30 S, antes de adicionar à pelota.
Nota: se preparado de acordo com as etapas 1.5.1 − 1.5.7, 10 μL de S. pombe alíquota é exigido para um pellet derivado de 30 ml de cultura. - Antes de põr sobre a tampa, gire muito momentaneamente para 1 − 2 s para assegurar-se de que nenhuma grânulos do Zirconia esteja prendido entre a tampa e o tubo, que pode fazer com que a amostra e o phenol escapem do tubo.
- Ressuscita as células em 400 μL de acetato de EDTA (AE) tampão (50 mM acetato de sódio pH 5,3, 10 mM EDTA pH 8,0), por vortexing vigorosamente. Adicionar 40 μL de 10% (p/v) de dodecilo sulfato de sódio (SDS). Não vórtice, como SDS vai espuma.
- Se o protocolo β-est AID 4U deve ser utilizado, tomar 40 μL da suspensão celular para análise proteica7. Adicione 40 μL de AE para fazer o volume voltar até 400 μL.
- Adicionar 800 μL de fenol (precaução) em pH baixo e Vortex por 10 s.
Cuidado: o fenol é tóxico e corrosivo por inalação e contato. Realize sempre procedimentos envolvendo fenol em uma capa de fumaça e use dois pares de luvas. - Lyse as células em um homogeneizador (por exemplo, tabela de materiais) para três rajadas de 2 min na configuração de energia mais baixa. Deixe as amostras no gelo por 2 min entre os pulsos de homogeneização.
Nota: Otimize as condições se utilizar outros Homogeneizadores. A agitação insuficiente resultará em rendimentos pobres, enquanto a agitação excessiva resulta em um rendimento mais elevado aparente, conforme determinado pela absorvência a 260 nm (A260), mas o RNA pode ser degradado. Um homogeneizador é preferível, mas A purificação de RNA de fenol quente12 pode ser usada. - Coloc a amostra lisada no gelo seco por 5 minutos, até que solidifies, esta reduz o ADN genomic carreg sobre no RNA. Não congele por muito tempo como a amostra não descongelar. Spin 5 min em microcentrífuga a > 13000 x g em RT; Não seja tentado fazer isto em 4 ° c, porque a mistura da amostra/fenol permanecerá contínua durante todo a rotação se executada na baixa temperatura.
Nota: se a amostra ainda estiver congelada no final da rotação, gire novamente por mais 5 min até que a amostra tenha sido completamente desbloqueada. - Extrato de fenol/clorofórmio, em seguida, extrato de clorofórmio com um volume igual (aproximadamente 600 μL) de fenol: clorofórmio 5:1 então clorofórmio (precaução). Transfira a fase superior para outro tubo contendo fenol: clorofórmio 5:1 ou clorofórmio. Vortex, em seguida, girar por 5 min em um microcentrífuga at RT. Em seguida, transfira a fase superior para um novo tubo de 1,5 mL.
Atenção: o clorofórmio é tóxico por inalação e contacto. Realize sempre procedimentos envolvendo clorofórmio em uma capa de fumaça e use dois pares de luvas. - Adicionar um terço ao meio volume (aproximadamente 300 μL) de 10 M de LiCl, e misture para precipizar o RNA. A amostra deve ir nublado imediatamente, mas deixar por pelo menos 10 min no gelo ou a 4 ° c (não armazene abaixo de-20 ° c como ele vai congelar), ou até que o precipitado floculates.
- Gire por 5 min em > 13000 x g em um microfuge. Retire o fluido, regire brevemente e retire os resíduos. Lave a pelota com 300 − 500 μL de 70% de etanol, gire brevemente e remova o etanol restante.
Nota: durante estas lavas manter o pellet no mesmo lado do tubo como o primeiro giro, desta forma a pelota não vai se mover e quebrar; Se quebrar algum do RNA poderia ser perdido acidentalmente.
Nota: não seque o pellet; desde que a maior parte do fluido tenha sido removida, ela não interferirá nas etapas subsequentes. O RNA pode igualmente ser armazenado nesta fase em-20 ° c por alguns meses ou-70 a-80 ° c para o armazenamento a longo prazo. - Re-dissolver o pellet de RNA em 90 μL de TE pH 7,0 (10 mM Tris HCl pH 7,0, 1 mM EDTA pH 8,0) por aquecimento a 65 ° c com agitação como a pelota do RNA pode ser difícil de re-dissolver. Isto deve ser para não mais de 5 minutos como o RNA degrada em umas temperaturas mais elevadas. Verifique a solubilização total de RNA e depois transfira para um tubo de 0,2 mL. Pipeta a amostra para cima e para baixo; Não deve haver "caroços", e o fluido deve subir e cair suavemente na ponta. Esta solução é viscosa de modo que o movimento de pipetagem final deve ser lento.
Nota: o RNA pode ser armazenado a-20 ° c no escuro nesta fase; Isto pode igualmente ser benéfico à solubilidade do RNA.
3. biotinilação
Nota: o tempo de conclusão é 60 min. As seguintes etapas são feitas convenientemente em uma tira dos tubos com tampões integrais porque têm menos tendência a abrir em vortexing do que tiras com tampões separados.
- Biotinylate adicionando 10 μL (1/10 volume final) de uma solução do hpdp-biotina de 5 milímetros (a MTS-biotina pode ser usada exatamente da mesma maneira que hpdp-biotina), ao RNA e misturar-se completamente. Pré-aqueça o RNA por não mais do que alguns segundos em 65 ° c antes de adicionar a biotina. Incubar a 65 ° c por 15 min a um máximo de 30 min no escuro.
Nota: Este aquecimento é exigido porque alguns lotes de HPDP precipitam em RT na amostra do RNA. Um bloco do PCR com uma tampa heated é ideal para este. - Prepare um pequeno volume de resina, coluna de exclusão de tamanho (tabela de materiais) para excluir a biotina não incorporada. Retire a etiqueta inferior da coluna e afrouxe a tampa, coloque-a num tubo de centrifugação de 2 mL. Gire em 1500 x g por 1 minuto para nivelar para fora o amortecedor adiciona 0,3 ml de te delicadamente à parte superior da coluna e gira outra vez. Repita a lavagem e gire duas vezes mais para um total de 3 lavagens. Finalmente transfira a coluna lavada para um tubo de 1,5 mL fresco.
- Depois que a incubação da amostra (etapa 3,1) estiver concluída, adicione a amostra à parte superior da coluna. Girar em 1500 x g por 2 min. A amostra de RNA biotinylated está agora na parte inferior do tubo.
Nota: uma rotação de 1 min não é suficiente para eluir a amostra inteira. - Adicione um terço ao meio volume (aproximadamente 40 μL) de 10 M de LiCl, misture para re-precipitate o RNA como a etapa 2,10. A amostra deve ir nublado imediatamente, mas deixar por pelo menos 5 min no gelo ou a 4 ° c ou até o precipitado floculados; Não armazene abaixo de-20 ° c porque congelará. Centrifugue a amostra durante 5 min a > 13000 x g num microfuge.
- Lave com 80% de etanol, ≤ 1 h girando. Siga o procedimento na etapa 2,11 para remover tanto do fluido quanto possível.
Nota: como HPDP-biotina é muito solúvel em 80% etanol, esta é uma etapa adicional de purificação. - Repita a lavagem do etanol 80% para remover tanto a biotina un-incorporada como possível.
Nota: o RNA também pode ser armazenado nesta fase a-20 ° c no escuro.
4. purificação do RNA recém-sintetizado
Nota: o tempo de conclusão é 2 h.
- Redissolver o RNA em 200 μL de H2O tratado com DEPC (a incubação de 65 ° c pode ser utilizada, semelhante ao procedimento na etapa 2,12).
- Meça a concentração do RNA em A260 usando um espectrofotômetro; a amostra pode ter que ser diluída 1/10 para obtê-lo dentro da faixa linear do Espectrofotômetro. Vórtice esta diluição para pelo menos 10 s para garantir que o RNA viscoso é uniformemente dissolvido.
Nota: a eficiência da biotinilação pode ser avaliada por dot blot13 se necessário. - Adicione quantidades iguais de RNA a um tubo fresco e faça até 200 μL em H2o do DEPC-treated use toda a amostra com a mais baixa concentração do RNA e use um volume apropriado das outras amostras para ter uma quantidade similar de RNA para cada um.
Nota: a folha de cálculo 4Tu experimento template. xlsx tem um formulário para ajudar este cálculo. - Quando a amostra está em RT, adicionar 25 μL de 10 x tampão NaTM (0,1 M Tris HCl pH 7,0, 2 M NaCl, 250 mM MgCl2), 25 μL de 1 m Napi pH 6,8 (0,5 m NaH2po4 0,5 m na2HPO4) e 2,5 μL de SDS de 10%. Misture cuidadosamente e Gire suavemente (< 30 s; aproximadamente 100 x g).
Nota: para evitar a precipitação da SDS e sais, as amostras devem ser mantidas em RT durante os seguintes procedimentos até a etapa 4,13. - Faça o tampão do grânulo que contem o amortecedor de 1x NaTM, 0,1 M NaPi, e 0,1% SDS, 2 mL por a amostra. Adicione a quantidade necessária de H2o primeiro e a última SDS. Isso deve ser feito fresco cada vez como um precipitado formas após 24 h.
Nota: para evitar a formação de precipitados, o tampão de talão deve ser mantido na RT durante os seguintes procedimentos. Não DEPC tratar ou autoclave. - Adicionar 50 μL de grânulos de streptavidina a um tubo de 1,5 mL de baixa retenção. Coloque o tubo na cremalheira magnética, aguarde que as contas se instalem e, em seguida, retire o fluido
- Lave os grânulos de estreptavidina.
- Adicionar 200 μL de tampão de talão e vórtice até que a pelota do talão seja totalmente ressopalizada. Geralmente 3 − 5 s é tudo o que é necessário. Para lavagens antes que a amostra do RNA esteja adicionada é suficiente girar os tubos redondos assim que os grânulos viajam através do tubo ao outro lado. Em seguida, vire os tubos de volta para o lado original para que as contas de viajar através do tubo mais uma vez.
- Gire o tubo em baixa velocidade (aproximadamente 100 x g) para um máximo de 5 s para girar para baixo o fluido, mas não os grânulos.
- Coloc na cremalheira magnética para permitir que os grânulos sejam capturados pelo ímã.
- Retire o fluido por aspiração para um pequeno número de amostras; Despeje o líquido se muitas amostras.
Nota: com um grande número de amostras, removendo todo o fluido puramente por aspiração pode ser problemático, como os grânulos na primeira amostra pode ser secado antes da última amostra é concluída, isso aumenta o fundo. A lavagem pode ser acelerada derramando fora o líquido de todas as amostras imediatamente enquanto no ímã. Eles devem ser deixados um pouco mais no ímã antes de derramar e a pequena quantidade de fluido que permanece tem de ser aspirado, mas, no geral, significa menos tempo no ímã e sem fluido. Desta forma, é possível fazer 24 ou mais extrações rapidamente.
- Bloco com 200 μL de tampão de talão, 10 μL de 20 mg/mL de glicogênio e 2,5 μL de 5 mg/mL de tRNA, 20 min de rotação final na extremidade a uma velocidade moderada em RT. A rotação é manter as contas em suspensão. Uma vez que o bloqueio está completo remova o líquido como etapas 4.7.2-4.7.4 e lave-o outra vez, como as etapas na seção 4,7.
- Ressuscitem os grânulos na amostra. Incubar em RT com rotação por 30 min.
- Durante a incubação, prepare um tubo fresco de 1,5 mL para cada amostra. Adicione 1/10 de volume (aproximadamente 10 μL) de acetato de sódio a 3 M pH 5,3 e 20 μg de glicogênio, e gire a aproximadamente 100 x g por 3 s. Armazene em um rack até que seja necessário.
- Remova o RNA não acoplado dos grânulos, como etapas 4.7.2-4.7.4. O RNA não acoplado pode ser perseverado em um tubo fresco, mas os sais e SDS tornam muito difícil de purificar. Em seguida, lave os grânulos, como seção 4,7 com vortexing, para um mínimo de 3 a um máximo de 5 vezes.
- Após a lavagem final Tome especial cuidado para aspirar todo o líquido; voltar a cada tubo e aspirar os resíduos do tampão mais uma vez.
- Para Eluir o RNA, adicione 50 μL de 0,7 M β-mercaptoethanol recentemente preparado (βme) aos grânulos (1/20 diluição da solução de estoque fornecida comercialmente). Vortex e girar brevemente, como os passos 4.7.1 e 4.7.2. Coloque a pasta no rack magnético e pipetar a solução contendo RNA no tubo de centrífuga 1,5 mL preparado na etapa 4,10.
- Elute uma vez mais como etapa 4,13 para recuperar o RNA residual dos grânulos e para adicionar a amostra eluída ao tubo que contem a primeira eluição destes grânulos.
- Retire os grânulos residuais do RNA eluída colocando a amostra de volta no rack magnético e transferindo o fluido para um tubo de centrífuga de 0,5 ml de ligação fresca e baixa.
- Misture a amostra e, em seguida, precipeie o nsRNA adicionando volumes de 2,5 x (280 μL) de etanol e misture mais uma vez. Deixe por 1 h para pernoite a-20 ° c. Gire em um centrifugador pre-refrigerado (4 ° c) por 20 minutos na velocidade máxima (pelo menos 13.000 x g).
- Lave cuidadosamente com 200 μL de 70% de etanol a-20 ° c. Porque o βme residual inibirá aplicações a jusante, gire em cada etapa para remover tanto quanto dos escória como possível; na extremidade a amostra não deve cheirar de βME.
- Redissolver em 10 − 20 μL de 1x TE tratado com DEPC com o equivalente a 0, 5 μL de inibidor de RNase.
Nota: todos os estágios subsequentes devem ser realizados no gelo. - Meça a concentração e a pureza do RNA.
- Meça o a260 e a225 em um Espectrofotômetro de baixo volume de amostra.
Nota: um máximo de absorvância perto de λ = 225 nm é proveniente de um contaminante inevitável dos grânulos. Na ausência de RNA o sinal do contaminante declina a 35% em λ = 260 nm. Portanto, a quantidade real de RNA é aproximada pela fórmula: (a260-(a225* 0.35)) * 40 ng/μl. - Alternativamente, analise a amostra em um sistema da electroforese do Microfluidics tal como um Bioanalyzer.
Nota: esta análise é preferível ao uso de um espectrofotômetro como a integridade do RNA pode ser avaliada, o contaminante não interfere com a quantitação e menos amostra é necessária.
- Meça o a260 e a225 em um Espectrofotômetro de baixo volume de amostra.
- Analise o nsRNA.
Nota: por exemplo, as RNAs específicas podem ser quantificadas por técnicas de transcriptase reversa padrão do qPCR. O RNA preparado desta maneira é compatível com a preparação da biblioteca para RNA-Seq. a remoção de rRNA não é necessária para tempos de rotulagem inferiores a 5 min. recodificação SLAMseq14 também pode ser realizada neste RNA.
Resultados
Os rendimentos típicos para o nsRNA recuperado usando este protocolo ers4tU são indicados na Figura 1b, este foi produzido por um Bioanalyzer e o traço mostra a produção do RNA contra o tamanho (nucleotides [NT]). Note, no traço do Bioanalyzer e no gráfico do Inset, que a recuperação do RNA do ponto de tempo 0 é uma parcela muito pequena daquela recuperada dos pontos mais longos do tempo-aproximadamente 0,3 μg do RNA recuperado de aproximadamente 109 pilhas comparado com o excesso duas vezes tanto apenas 30 s de rotulagem (0,8 μg de nsRNA) do mesmo número de células. A recuperação do RNA em 15 s é mais variável porque as diferenças pequenas em executar a amostragem têm um efeito proporcionalmente maior na recuperação do RNA. No traço do Bioanalyzer, os precursores do rRNA podem ser vistos como um pico perto de 1000 NT e um Doublet dos picos em 1700 − 1800 NT. A abundância desses intermediários aumenta à medida que a tiolação continua.
A rotulagem de Thio foi utilizada para quantificar a emenda do transcrito Act1 (Figura 3). A tiolação foi realizada e as amostras colhidas em intervalos de 15 s desde o início da rotulagem e o processamento do RNA Act1 monitorado (Figura 3a, b). Como pode ser visto, o pré-mRNA é gerado (por transcrição), e lariats (pelo primeiro passo de splicing de pré-mRNA), mesmo depois de apenas 15 s de rotulagem. Após aproximadamente 45 s a 1 minuto, as quantidades de lariats e pre-mRNA alcangam o equilíbrio com tanto quanto destas espécies do RNA que estão sendo criadas pela transcrição como são processadas afastado pela emenda.
Para produzir os dados mostrados na Figura 3C a cepa foi pulsada com 4tu para 25 s e, em seguida, perseguido com uridina. A geração de pré-mRNA e lariats atinge um máximo de 1 minuto. Isso se compara bem com a Figura 3B; o máximo que está sendo conseguido após 45 s para alcangar o equilíbrio mais os 25 s da rotulagem. Após o pico, os níveis de declínio como o Thio-rotulados RNAs são perseguidos através do processo de emenda.
A Figura 3D mostra a depleção de um fator de splicing protéico e seu efeito sobre o metabolismo do RNA, utilizando o sistema β-est Aid 4U6,7. Aqui, o Prp16p é reduzido de níveis fisiológicos próximos para 5% deste nível após 25 min de depleção. Prp16p é um fator de emenda essencial para a segunda etapa de emenda15. Os lariats são removidos durante a segunda etapa da emenda (Figura 3a), mas aqui aumentam acima do nível de pre-mRNA como Prp16 torna-se limitando. Em tempos de depleção posteriores, outros fatores tornam-se limitantes devido a efeitos secundários, de modo que os níveis de Lariat diminuem, e os níveis de pré-mRNA aumentam. O nível de mRNA emendados declina.
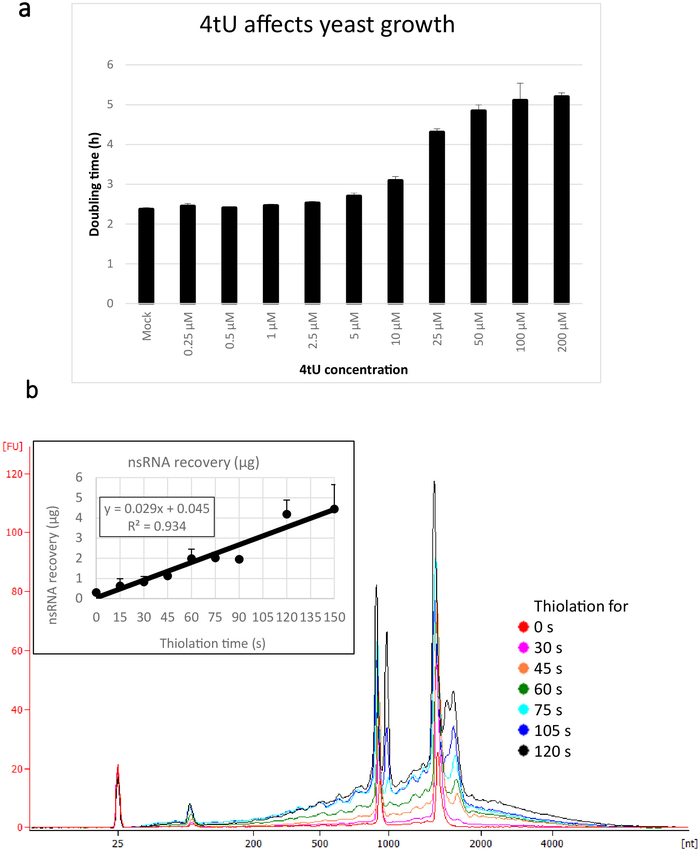
Figura 1: crescimento na recuperação de 4tU e RNA. (a) 4-tiouracil afecta o crescimento. O aumento da concentração de 4tU no meio de crescimento drop-out de YMM sem uracil aumenta o tempo de duplicação de S. cerevisiae (BY4741) carregando o plasmídeo p4FuiΔPEST. O crescimento de quatro culturas replicadas foi monitorado a 30 ° c em um Tecan Infinite pro 200. Todas as culturas estavam em fase de log por toda parte, com OD600 entre 0,1 e 0,6. Mock é uma cultura de controle com uma quantidade equivalente de NaOH adicionado, que não muda por si só a taxa de crescimento. Este gráfico demonstra que a rotulagem é um compromisso entre a rotulagem rápida e os danos à célula. As barras de erro são erro padrão de 4 repetições. b) a produção de nsrna aumenta linearmente de cerca de 15 s de rotulagem. A figura principal mostra os traços do bioanalisador de purificados, nsRNA de 0 (não tiolated) a 2 minutos após a adição de 4tU em intervalos de 15 s. Observe que a amostra de 15 s não é mostrada, pois era indistinguível da amostra não rotulada. Os dois grandes picos correspondem a RNAs ribossômicas (rRNAs). Os precursores e intermediários do rRNA são visíveis como vários picos em maior peso molecular do que rRNAs maduros. A recuperação desses precursores e intermediários aumenta com o tempo. Os resultados de um experimento representativo são mostrados. O gráfico Inset mostra a recuperação de nsRNA com o aumento da incubação com 4tU. O rendimento de nsRNA aumenta com o aumento do tempo de crescimento com 4tU. A recuperação é notavelmente linear (R2 = 0,934) ao longo da escala temporal deste experimento e mostra um ligeiro aumento sobre o fundo, mesmo a 15 s rotulagem com 4tu, embora não distinguível da amostra não rotulada por olho do bioanalisador Rastreamento. As barras de erro mostram erro padrão para três replicações biológicas. Por favor clique aqui para ver uma versão maior desta figura.
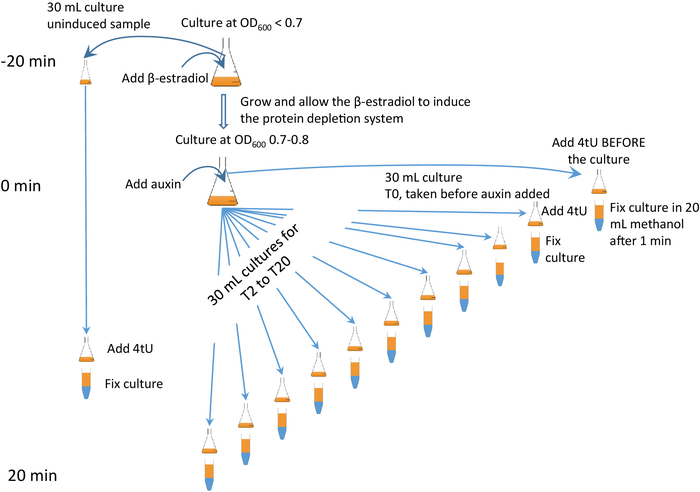
Figura 2: β-est Aid 4U β-est Aid 4U protocolo gráfico. Um resumo gráfico do protocolo do protocolo β-est AID 4U. β-estradiol (β-est) promove a expressão do sistema de auxina induzível degron (Aid) que por sua vez esgota um auxílio * Tagged proteína alvo, referem-se a Barrass et al.7 para um protocolo detalhado. Neste caso, a expressão do sistema degron é iniciada 25 min antes do início da degradação da proteína e a tiolação em cada ponto de tempo é de 1 min. as amostras são tomadas antes da indução e a cada 2 minutos durante a depleção. Uma versão animada aparece na Figura complementar 2. Por favor clique aqui para ver uma versão maior desta figura.
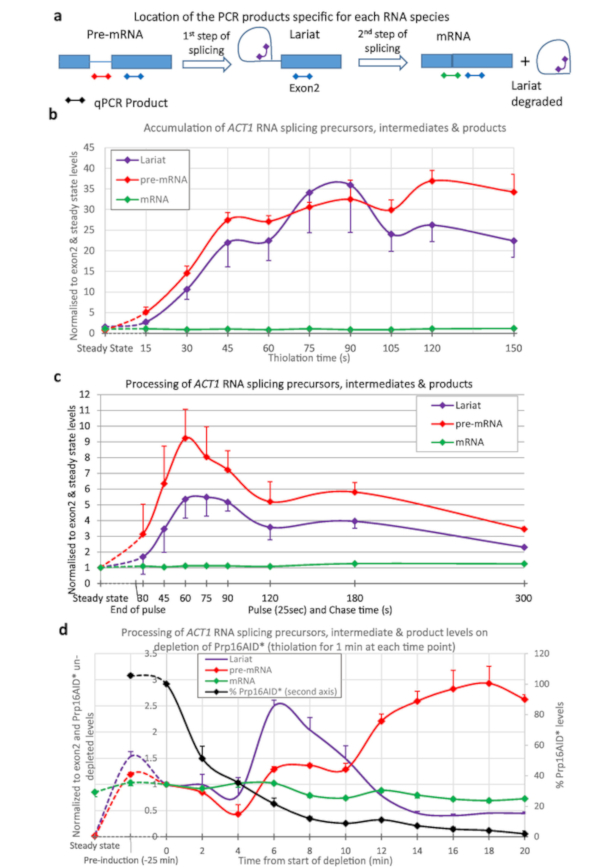
Figura 3: precursores e intermediários de Act1 RNA splicing. A emenda de Act1 transcritos do pre-mRNA foi monitorada pela transcrição reversa quantitativa PCR16. Os níveis do precursor Act1 (pre-mRNA), o Lariat-exon2 intermediário (Lariat) e o produto Emenido (mRNA) são mostrados normalizados de encontro ao nível de Act1 exon2 e de níveis de estado estáveis destes RNAs. (a) localização de produtos qPCR na transcrição Act1. Esquemático dos locais dos produtos qPCR utilizados para o ensaio dos níveis de precursores, intermediários e produtos da reação de emenda das transcrições do Act1 16. Os exons são representados por caixas, Intron como uma linha e os produtos de qPCR como linhas com diamantes em uma ou outra extremidade, a cor combina aqueles usados nos gráficos. O pre-mRNA PCR é específico para pre-mRNA e não todos os intermediários da emenda porque este produto cruza o ponto do ramo que é interrompido após a primeira etapa do splicing. O PCR de Lariat detectará o produto da primeira etapa da emenda e do Lariat extirpado produzido após o segundo. O PCR de mRNA é específico para o produto da emenda, mRNA. Os resultados do PCR do exon (presente em todos os precursores, intermediários e produtos, exceto o Lariat extirpado) não são mostrados nos gráficos porque este foi usado para normalizar os dados e é conseqüentemente sempre igual a 1. (b) tiolabelling contínuo. A quantidade de pré-mRNA aumenta com o tempo como 4tU é incorporado pela transcrição e, após um curto atraso, splicing converte-lo para Lariat-exon2 produtos intermediários e emendados. Os níveis destas espécies de pré-mRNA e de Lariat são detectáveis acima do fundo após tão pouco quanto 15 s do crescimento com 4tu e alcangam um máximo após aproximadamente 45 s da rotulagem contínua com 4tu, em que ponto sua produção é balançada pela conversão ao emendados mRNA e/ou degradação. Os valores são normalizados para o seu estado estacionário (ponto mais à esquerda do gráfico), e exon 2 níveis para mostrar a sua aparência e processamento em comparação com a transcrição do exon 2. Como a emenda do RNA de Act1 é pela maior parte co-transcriptional4,17 o mRNA emendados torna-se ràpida a espécie a mais abundante, seu nível é similar àquele do exon 2. Erro padrão de três repetições biológicas, cada uma ensaiada em triplicado. (c) Pulse/Chase. Pulso de tiolação de 25 segundos seguido de perseguição com uridina. Comparado aos níveis de estado estacionário destas RNAs (esquerda-mais ponto), são inicialmente muito abundantes no pool recentemente sintetizado. Os níveis diminuem gradualmente à medida que são processados em mRNA (ou degradados), aproximando-se de níveis muito semelhantes aos níveis de estado estacionário por 5 min. erro padrão de três repetições biológicas, cada um analisado em triplicado. (d) nsrna e depleção de proteínas. A emenda de transcrições do pre-mRNA de Act1 monitorou pelo PCR quantitativo da transcrição reversa como no painel (a) em cima do esgotamento da proteína Prp16 usando o sistema do degron do auxina como descrito em Figura 2. Os níveis de proteína Prp16 também são exibidos no gráfico plotado contra o segundo eixo Y como porcentagem de níveis antes da depleção de auxina. Prp16 é um componente vital do spliceosome, particularmente importante para a segunda etapa da emenda mostrada no painel (a), depois do qual os lariats são degradados. Quando esta etapa se torna limitar lariats acumular inicialmente. Em pontos de tempo posteriores splicing falha completamente, lariats já não são produzidos e níveis de pré-mRNA subir. As barras de erro são erro padrão de três repetições biológicas, cada uma ensaiada em triplicado. Por favor clique aqui para ver uma versão maior desta figura.
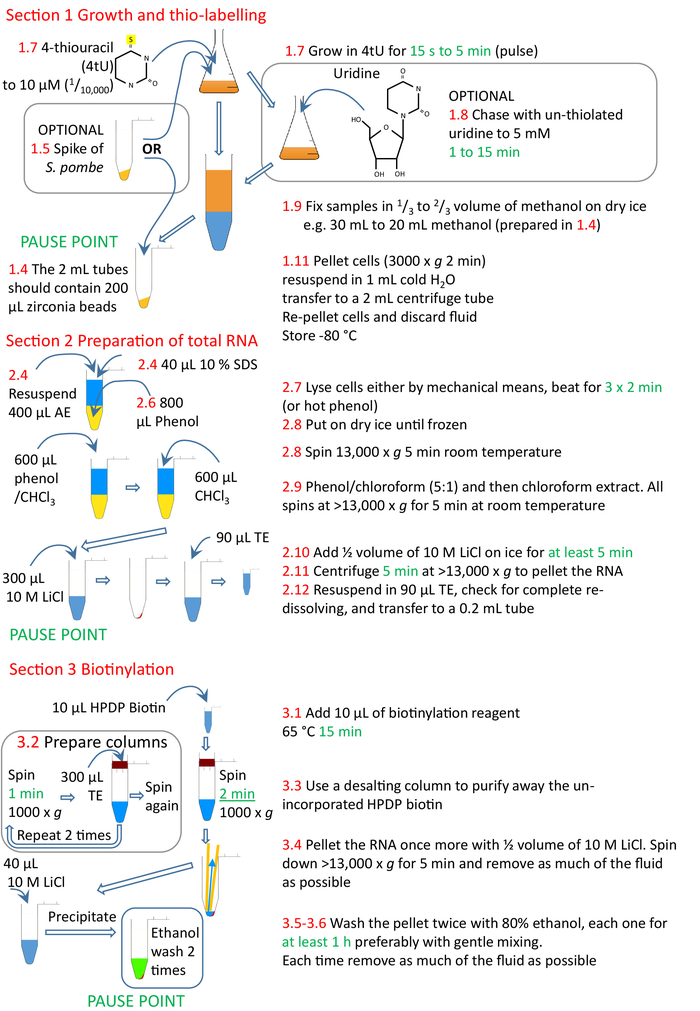
Figura 4: Resumo gráfico das secções de protocolo 1 a 3. As células são tioladas com 4tU e podem crescer para incorporar o nucleotídeo modificado no RNA. Um pico de S. pombe swcnts pode ser adicionado para permitir a normalização através dos pontos e dos experimentos do tempo. O pulso de 4tU pode ser perseguido usando o uridine un-thiolated. A rotulagem pode ser executada continuamente da adição 4tU ou de uma mudança às condições de crescimento, a separação da cultura e 4tU adicionados às culturas em épocas crescentes da mudança da condição de crescimento, mas cada rotulagem somente por um tempo breve. As células são coletadas, e RNA preparados a partir das células, de preferência usando um homogeneizador e métodos baseados em fenol. O RNA é biotinylated e então o RNA biotinylated purificado da biotina região usando uma coluna da exclusão do tamanho. O nsRNA está agora pronto para a purificação com grânulos de estreptavidina (seção 4, Figura 5). Os números em vermelho correspondem aos números de etapa no protocolo. Por favor clique aqui para ver uma versão maior desta figura.
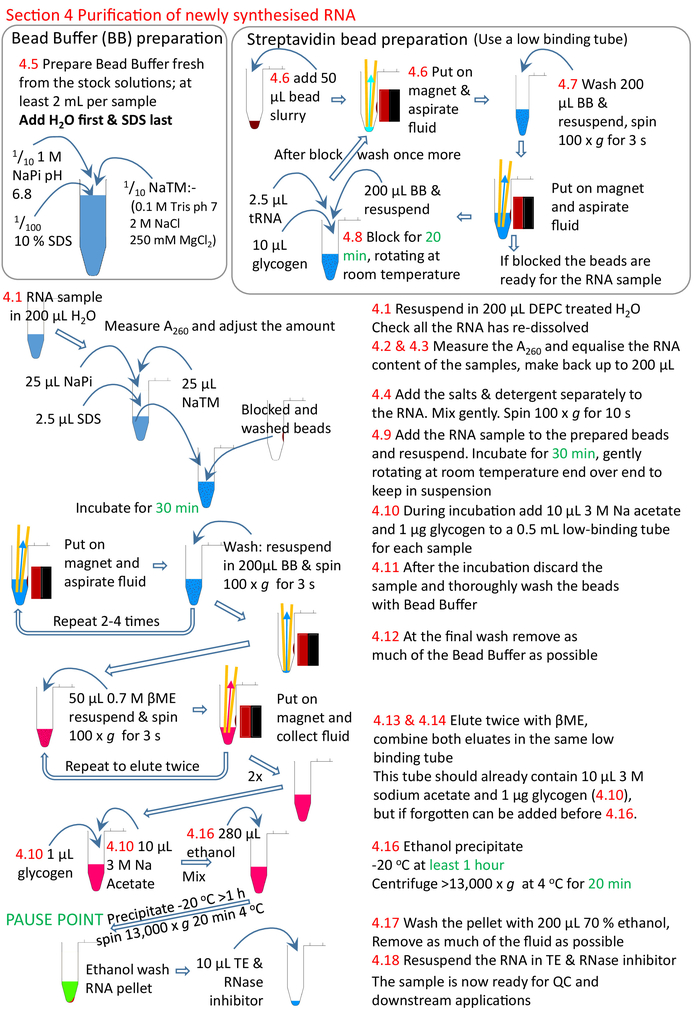
Figura 5: Resumo gráfico da seção de protocolo 4. Na sequência das secções 1 a 3 (Figura 4), os grânulos de estreptavidina são bloqueados e a amostra de RNA biotinilado adicionada aos grânulos preparados. O RNA biotinylated liga-se aos grânulos do estreptavidina e ao RNA un-biotinylated removido e lavado. O RNA biotinylated é eluída dos grânulos usando βme e precipitado pronto para uma pesquisa mais adicional. Os números em vermelho correspondem aos números de etapa no protocolo. Por favor clique aqui para ver uma versão maior desta figura.
Figura complementar 1: melhoria da recuperação de nsRNA a partir de células de levedura com e sem cópias adicionais do importador a 1 e 3 minutos de rotulagem de Thio. Note que Fui1 é o próprio promotor do fermento expresso a partir de um plasmídeo de 2 μm. A cópia genomic deste gene está atual em ambas estas tensões. Por favor, clique aqui para baixar este arquivo.
Figura complementar 2: versão animada do protocolo gráfico de ajuda 4U β-est Aid 4U de β-est. Por favor, clique aqui para baixar este arquivo.
Arquivo complementar 1:4tU_experiment_template. xltx. Por favor, clique aqui para baixar este arquivo.
| Nome plasmídeo | Importador/Permease | Marcador | Comentário | |
| p4Fui | S. cerevisiae Fui1 | URA3 | Fui1 importações uracil e uridine, tornando-o ideal para pulso/perseguição experimentos. | |
| pAT2 | S. cerevisiae Fui1 | LEU2 | ||
| p4Fui-ΔPEST | S. cerevisiae Fui1 | URA3 | O motivo PEST de Fui1 foi desativado, de modo que a Permease não é degradada quando há uracil intracelular suficiente para as necessidades da célula. Funciona bem em experimentos de rotulagem e melhora o desempenho de pulso/perseguição. | |
| p4Fur | S. cerevisiae Fur4 | URA3 | Permease de uracil | |
| YEpEBI311 | H. sapiens ENT1 | LEU2 | Miller et al.11. Igualmente contem um gene da quinase do timidina de HSV. | |
| (transportador de nucleosídeo equilibrativo) | ||||
| Todos os plasmímids são 2 μm baseados. Todos os plasmíos e pAT P4 baseiam-se na série pRS16 de plasmís. FUI1 e FUR4 são expressos a partir de seus próprios promotores endógenos. | ||||
Tabela 1: Plasmís utilizados com este protocolo.
Discussão
Este artigo apresenta um protocolo para a rotulagem 4tu extremamente rápida e específica, para a recuperação do RNA nascent, recentemente sintetizado de s. cerevisiae após tão pouco quanto 15 S da rotulagem, com contaminação muito baixa pelo RNA sem rótulo.
O usuário deve sempre tomar o cuidado para manter a integridade do RNA pelo uso de temperaturas frias e de reagentes DEPC-tratados. A purificação do grânulo de streptavidin é geralmente de confiança; no entanto, o buffer de talão é difícil de manusear; Ele deve ser feito recentemente, com seus componentes adicionados na ordem certa, e não refrigerados ou esterilizados. As falhas comuns incluem o RNA que está sendo dissolvido incompleta após as etapas da precipitação, e assim que sendo não biotinylated ou perdido de outra maneira durante as etapas de processamento. Há uma extensa ajuda de solução de problemas no material complementar.
Há algumas limitações a estar cientes em ers4tU. Um já mencionado é que 4tU retarda o crescimento do fermento (Figura 1a). Para além das RNAs9endogenamente tioladas, apenas as RNAs transcritas durante o período de rotulagem podem ser purificadas por este método. Polimerases pausadas em genes durante todo o tempo de de não produzirá transcritos swcnts que podem ser purificados, embora os transcritos que são rotulados parcialmente devido às polimerases que entram ou que saem de um estado pausado durante o de possam ser recuperados. Cepas que transproduzem mal, seja por causa da mutação ou condições de crescimento, produzir nsRNA pouco, embora as técnicas utilizadas aqui, no entanto, melhorar a recuperação de nsRNA em comparação com outros métodos. Os tempos mais longos e os volumes de cultura aumentados podem ser necessários nestas tensões e circunstâncias. Note-se que o uracil é uma boa fonte de nitrogênio e assim este método deve ser trialed antes de ser utilizado para estudos envolvendo a fome de nitrogênio.
O protocolo ers4tU é particularmente útil para análise de RNAs de curta duração, muitos dos quais são tão rapidamente degradados que não podem ser identificados sem prejudicar a maquinaria de degradação. Exemplos incluem transcrições crípticas instáveis (cortes)4, e transcrições curtas produzidas por terminação prematura ou promotor proximal pausando18 e transcrição antisense "upstream" de um promotor (PROMPTs)19. Os intermediários produzidos durante o processamento de espécies estáveis de RNA também são transitórios, mas podem ser enriquecidos usando ers4tU transcrição4. O protocolo ers4tU é conseqüentemente excepcional em permitir que as espécies altamente transientes do RNA sejam analisadas e capturadas circunstâncias physiological próximas, que é uma vantagem enorme sobre outros métodos. Esta técnica tem sido usada para estudar a transcrição e a cinética de processamento de RNA a jusante em mutantes de RNA polimerase que alongam mais rápido ou mais lento que o normal20.
A tiolação também é compatível com RNA-Seq e SLAM-Seq21, permitindo que todos os RNA produzidos dentro de uma janela de tempo muito curto para ser caracterizado em detalhes requintados.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado por Wellcome financiamento para JB [104648]. O trabalho no centro Wellcome para a biologia de pilha é apoiado pelo financiamento do núcleo de Wellcome [092076]. Os autores reconhecem os membros do laboratório por sua ajuda: Bella Maudlin, Emanuela Sani, Susanna de Lucas-Arias e shiney George. Os autores também gostariam de agradecer a Patrick Cramer pelo plasmídeo YEpEBI31111.
Materiais
| Name | Company | Catalog Number | Comments |
| β-mercaptoethanol (βME) | Sigma-Aldrich | M3148 | CAUTION toxic. Stock solutions are aproximatly 14 M, make at 1/20 dilution for use |
| Chloroform | Sigma-Aldrich | 25668 | CAUTION toxic |
| Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758 | add 1/1000 volume to a solution, leave at room temperature for 24 h, then autoclave |
| DMF (N,N-dimethylformamide) | Sigma-Aldrich | 227056 | CAUTION toxic |
| EDTA | Sigma-Aldrich | 3609 | Make 0.5 M and pH to 8.0 with sodium hydroxide |
| Ethanol | Sigma-Aldrich | 29221 | |
| EZ-link HPDP Biotin | Thermo scientific | 21341 | Store protected from light. Disolve all the vial contents in 22.7 mL DMF (to make a 4 mM stock solution). Store away from water, in the dark & at -20 °C. Check the solution before using, as some batches of HPDP precipitate in storage; heat at 42 °C to resuspend. |
| Glucose | Fisher Scientific | G/0500/60 | |
| Glycogen [20 mg/mL] | Sigma-Aldrich | 10901393001 | Store at -20 °C |
| Immobilised TCEP Disulfide Reducing Gel | Thermo Scientific | 77712 | Optional |
| LiCl | Sigma-Aldrich | 793620 | 10 M solution. CAUTION: this gets very hot as is dissolves and can even boil at greater than 100 oC, add the LiCl crystals to the water slowly. |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 63033 | 1 M solution. CAUTION: this gets very hot as is dissolves and can even boil at greater than 100 oC, add the MgCl2 crystals to the water slowly. |
| Methanol | Fisher Scientific | M/4000/PC17 | CAUTION Toxic and flammable |
| NaH2PO4 | Sigma-Aldrich | S3139 | Make 1 M solutions of each and mix in equal amount to obtain a solution of the appropriate pH |
| Na2HPO4 | Sigma-Aldrich | S3264 | |
| NaCl | Sigma-Aldrich | S9888-M | 5 M solution |
| Phenol, low pH. | Sigma-Aldrich | P4682 | Store in the dark at 4 °C. CAUTION toxic |
| Phenol Chloroform 5:1 (125:24:1) low pH. | Sigma-Aldrich | P1944 | Store in the dark at 4 °C. CAUTION toxic |
| Pierce Spin Columns | Thermo Scientific | 69702 | Optional |
| SCSM single drop-out –ura | Formedium | DSCS101 | |
| Sodium Acetate | Sigma-Aldrich | 32318-M | Make a 3 M solution and pH to 5.3 with acetic acid |
| Sodium hydroxide | Sigma-Aldrich | 795429 | CAUTION corrosive |
| SDS (Sodium dodecyl sulfate) | Sigma-Aldrich | 436143 | CAUTION irritant, do not inhale |
| Streptavidin Magnetic beads | NEB | 1420S | Store at 4°C |
| SUPERase-In, RNase inhibitor | Life technologies | AM2696 | Store at -20°C |
| Thiolated Schizosaccharomyces pombe for spike | See section 1.7 of the protocol | ||
| 4-thiouracil (4tU) | ACROS ORGANICS | 359930010 | Store in the dark. Make 100 mM Stock in 1M NaOH, store solutions at -20°C. |
| Tris base | Sigma-Aldrich | 93362 | 1 M solutions at various pH |
| tRNA | Sigma-Aldrich | 10109541001 | 5mg/ml, store at -20°C |
| Uridine | Sigma-Aldrich | U3750 | Make 1 M solution in H2O. Split into 2 mL aliquots and store at -20 C. |
| Yeast nitrogen base without amino acids with amonium sulphate | Formedium | CYN0410 | |
| Zeba Columns 0.5ml | Thermo Scientific | 89882 | Store at 4 °C |
| Zirconia beads | Thistle Scientific | 110791052 | |
| Equipment and Consumables | |||
| Beadbeater | Biospec | 112011EUR | Other homogenisers can be used; the correct conditions for each homogeniser and strain must be established. |
| Bioanalyser (Agilent) or similar to assess RNA quality. If this is not important a spectrophotometer is useful to quantify the RNA. | |||
| Centrifuge: capable of spinning cultures at 4 °C and at least 3000 g. Pre-chill if possible. | |||
| Centrifuge: capable of spinning up to 2 mL tubes at variable speeds upto 13,000 g and down to 1000 g | |||
| Magnetic rack for separating the beads from the sample. The one used in the paper is 3D printed, available from Thingiverse (thing:3562952). Comercially available racks exist | |||
| PCR machine with a heated lid that will allow incubation in the dark. | |||
| Rotating wheel to rotate 1.5 mL tubes end over end | |||
| Shaking heating block (such as Eppendorf Thermomixer) is recomended | |||
| Tubes, centrifuge, Low retention, RNase free 0.5mL | Eppendorf | H179467N | |
| Tubes, centrifuge, Low retention, RNase free 1.5mL | Ambion | AM12350 | |
| Tubes, centrifuge, 50 mL | Sarstedt | 62.547.004 | Other centrifuge tubes are not gas proof allowing CO2 to disolve in the methanol, this comes out of solution vigorously on adding warm culture, leading to sample loss |
| Tubes, centrifuge, 15 mL | Sarstedt | 62.554.001 | |
| Tubes, 2 mL, screw cap | Greiner | 723361 | |
| Tubes 0.2 mL strip of 8 with integral lids | Brand | 781332 |
Referências
- Duffy, E. E., Schofield, J. A., Simon, M. D. Gaining insight into transcriptome-wide RNA population dynamics through the chemistry of 4-thiouridine. Wiley Interdisciplinary Reviews: RNA. 10 (1), e1513 (2018).
- Windhager, L., et al. Ultrashort and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Research. 22, 2031-2042 (2012).
- Baptista, T., Devys, D. Saccharomyces cerevisiae Metabolic Labeling with 4-thiouracil and the Quantification of Newly Synthesized mRNA As a Proxy for RNA Polymerase II Activity. Journal of Visualized Experiments. (140), e57982 (2018).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282 (2015).
- Burger, K., et al. 4-thiouridine inhibits rRNA synthesis and causes a nucleolar stress response. RNA Biology. 10, 1623-1630 (2013).
- Mendoza-Ochoa, G. I., et al. A fast and tuneable auxin-inducible degron for depletion of target proteins in budding yeast. Yeast (Chichester England). 36 (1), 75-81 (2018).
- Barrass, J. D., Mendoza-Ochoa, G. I., Maudlin, I. E., Sani, E., Beggs, J. D. Tuning degradation to achieve specific and efficient protein depletion. Journal of Visualized Experiments. , (2019).
- Hobro, A. J., Smith, N. I. An evaluation of fixation methods: Spatial and compositional cellular changes observed by Raman imaging. Vibrational Spectroscopy. 91, 31-45 (2017).
- Gustilo, E. M., Vendeix, F. A. P., Agris, P. F. tRNA's Modifications Bring Order to Gene Expression. Current Opinion in Microbiology. 11, 134-140 (2008).
- Dolken, L., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14, 1959-1972 (2008).
- Miller, C., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Molecular Systems Biology. 7, 458 (2011).
- Schmitt, M. E., Brown, T. A., Trumpower, B. L. A rapid and simple method for preparation of RNA from Saccharomyces cerevisiae. Nucleic Acids Research. 18, 3091-3092 (1990).
- Rädle, B., et al. Metabolic Labeling of Newly Transcribed RNA for High Resolution Gene Expression Profiling of RNA Synthesis, Processing and Decay in Cell Culture. Journal of Visualized Experiments. (78), e50195 (2013).
- Herzog, V. A., et al. Thiol-linked alkylation of RNA to assess expression dynamics. Nature Methods. 14, 1198-1204 (2017).
- Ohrt, T., et al. Molecular dissection of step 2 catalysis of yeast pre-mRNA splicing investigated in a purified system. RNA. 19, 902-915 (2013).
- Alexander, R. D., et al. RiboSys, a high-resolution, quantitative approach to measure the in vivo kinetics of pre-mRNA splicing and 3′-end processing in Saccharomyces cerevisiae. RNA. 16, 2570-2580 (2010).
- Wallace, E. W. J., Beggs, J. D. Extremely fast and incredibly close: cotranscriptional splicing in budding yeast. RNA. 23, 601-610 (2017).
- Adelman, K., Lis, J. T. Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nature Reviews Genetics. 13, 720-731 (2012).
- Preker, P., et al. RNA Exosome Depletion Reveals Transcription Upstream of Active Human Promoters. Science. 322, 1851-1854 (2008).
- Aslanzadeh, V., Huang, Y., Sanguinetti, G., Beggs, J. D. Transcription rate strongly affects splicing fidelity and cotranscriptionality in budding yeast. Genome Research. 28, 203-213 (2018).
- Schofield, J. A., Duffy, E. E., Kiefer, L., Sullivan, M. C., Simon, M. D. TimeLapse-seq: adding a temporal dimension to RNA sequencing through nucleoside recoding. Nature Methods. 15, 221-225 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados