Method Article
Extrem schnelle und spezifische metabolische Kennzeichnung von RNA In Vivo mit 4-Thiouracil (Ers4tU)
In diesem Artikel
Zusammenfassung
Die Verwendung von thioliertem Uracil zur empfindlichen und gezielten Reinigung neu transkribierter RNA aus der Hefe Saccharomyces cerevisiae.
Zusammenfassung
Das Nukleotid-Analogon 4-Thiouracil (4tU) wird leicht von Zellen aufgenommen und in die RNA aufgenommen, da es in vivo transkribiert wird, was die Isolierung der RNA ermöglicht, die während einer kurzen Phase der Etikettierung produziert wird. Dies geschieht durch Anbringen eines Biotin-Moiety summieren an der eingebauten Thio-Gruppe und Affinität reinigung, mit Streptavidin beschichteten Perlen. Das Erreichen einer guten Ausbeute an reiner, neu synthetisierter RNA, die frei von bereits vorhandener RNA ist, ermöglicht kürzere Etikettierzeiten und ermöglicht eine erhöhte zeitliche Auflösung in kinetischen Studien. Dies ist ein Protokoll zur sehr spezifischen, hochtragreichen Reinigung neu synthetisierter RNA. Das hier vorgestellte Protokoll beschreibt, wie RNA aus der Hefe Saccharomyces cerevisiae extrahiert wird. Das Protokoll zur Reinigung der thiolierten RNA aus der gesamten RNA sollte jedoch mit HILFE von RNA aus jedem Organismus wirksam sein, sobald sie aus den Zellen extrahiert wurde. Die gereinigte RNA eignet sich für die Analyse durch viele weit verbreitete Techniken, wie Reverse Transcriptase-qPCR, RNA-seq und SLAM-seq. Die Spezifität, Empfindlichkeit und Flexibilität dieser Technik ermöglichen unvergleichliche Einblicke in den RNA-Stoffwechsel.
Einleitung
RNA hat eine dynamische Natur; kurz nach der Produktion wird viel RNA schnell verarbeitet und abgebaut. Derzeit analysieren die meisten Studien des RNA-Stoffwechsels die gesamte zelluläre RNA, die meist vollständig verarbeitet ist und auf stationärer Ebene ist. Diese Höhe hängt vom Gleichgewicht zwischen den Transkriptionsraten, der posttranskriptionellen Reifung und dem Abbau ab. Die Analyse der Prozesse, die zum stabilen Zustandsgleichgewicht führen, erfordert spezielle Techniken, um sehr kurzlebige RNA-Arten zu erfassen.
Die metabolische Kennzeichnung von RNA mit Nukleotid-Analoga wie 4-Thiouracil (4tU) oder 4-Thiouridin (4sU) (siehe Duffy et al.1 für eine ausgezeichnete Bewertung) bietet die Möglichkeit, thio-markierte nascente RNAs und ihre Verarbeitungszwischenprodukte zu isolieren. Die veröffentlichten Protokolle beinhalten jedoch Kennzeichnungszeiten von mehreren Minuten2,3, was im Verhältnis zur Produktionsrate vieler Transkripte langsam ist. Es dauert in der Größenordnung von einer Minute, um das durchschnittliche Hefegen zu transkribieren, so dass die Kennzeichnung von Hefe-RNA für weniger als eine Minute als extrem kurz angesehen werden kann. Das extrem schnelle und spezifische 4 Thiouracil-Protokoll (ers4tU) maximiert das Signal-Rausch-Verhältnis, indem es die 4tU-Integration maximiert und die Rückgewinnung von unbeschrifteter, bereits vorhandener RNA minimiert, was sehr kurze Kennzeichnungszeiten ermöglicht4.
Die thiomodifizierte Basis muss schnell und in ausreichender Menge in die Zellen importiert werden, um die neu synthetisierte RNA (nsRNA) effizient zu kennzeichnen. Um dies zu fördern, werden Zellen in urcil-freiem Medium angebaut, und die Expression einer geeigneten Permease trägt dazu bei, die Aufnahme von 4tU oder 4sU zu steigern (siehe Tabelle 1 für eine Liste von Plasmiden, die geeignete Permeasegene und ergänzende Abbildung 1tragen). Die Löslichkeit von 4tU in Natriumhydroxid vermeidet die Notwendigkeit toxischer organischer Lösungsmittel, die von anderen Nukleotidanaloga benötigt werden. Leider wurden Wachstumskulturen über lange Zeiträume mit thiomodifizierten Nukleosiden bei Konzentrationen von mehr als 50 m beobachtet, um Ribosomen zu stören5. Die hier verwendete Konzentration (10 m) und die extrem kurzen Etikettierzeiten minimieren jedoch schädliche Effekte5 (Abbildung 1a), während dennoch genügend RNA für die Analyse zur Folge erhalten wird.
Diese Technik kann mit einer schnellen und spezifischen auxin-vermittelten Erschöpfung eines Zielproteins6,7 ( Abbildung2) kombiniert werden, das als "-est AID 4U"-Protokoll bezeichnet wird, in dem die induzierbares Degron-System (AID) wird mit 4tU-Kennzeichnung kombiniert. Mit dem Ansatz des '-est AID 4U kann ein Zielprotein aufgebraucht und die Wirkung auf den RNA-Stoffwechsel genau überwacht werden (Abbildung 2). Das Timing ist entscheidend; Es ist ratsam, das begleitende Video anzusehen und auf Abbildung 2 und seine animierte Form aufmerksam zu sein (siehe Ergänzende Abbildung 2).
Die Verarbeitung und Degradation von RNA muss extrem schnell gestoppt werden, um eine genaue Zeitauflösung zu ermöglichen. Dies wird mit Methanol bei niedriger Temperatur erreicht, das den Zellinhalt sehr schnell fixiert und die Zellmembran herabsetzt, während der Nukleinsäuregehalt8erhalten bleibt. Die RNA-Extraktion sollte effizient sein und die RNA nicht beschädigen. Mechanische Lyse ist wirksam in Abwesenheit von chaotropen Mitteln (oft enthalten diese Thio-Gruppen, so sollte vermieden werden). Lithiumchlorid-Ausfällung von RNA wird bevorzugt, da tRNAs weniger effizient gefällt werden. tRNAs werden schnell transkribiert und natürlich thioliert9, so dass die Entfernung von tRNAs die Konkurrenz um das Biotinylierungsreagenz reduziert. Wenn kleine, stark strukturierte RNAs von Interesse sind, werden alkoholbasierte RNA-Fällmethoden empfohlen.
Um die thiolierte RNA wiederherzustellen, wird Biotin kovalent über die Thio-Gruppen, die in die RNA mit 4tU integriert sind, angehängt. Die Verwendung von modifiziertem Biotin, das über eine skleavable Disulfidbindung (z.B. HPDP-Biotin (N-[6-(Biotinamido)hexyl]-3 -(2'pyridyldithio)propionamid) oder MTS-Biotin (Methan thiosulfonat)) anheftet, wird empfohlen. da es die Freisetzung der RNA durch Zugabe eines Reduktionsmittels ermöglicht. Die biotinylierte RNA ist Affinität gereinigt auf Streptavidin gekoppelt mit magnetischen Perlen. Dieses Protokoll ähnelt anderen zuvor10 aufgeführten, wurde aber intensiv optimiert, um den Hintergrund zu reduzieren.
Es gibt zwei Arten von Thiol-Etikettierungsexperimenten, die durchgeführt werden können, kontinuierliche und diskontinuierliche Etikettierung. Jeder hat seine eigenen Vorteile. Bei der kontinuierlichen Etikettierung wird der 4tU der Kultur zugesetzt und in regelmäßigen Abständen entnommene Proben entnommen. Diese Art von Experiment zeigt, wie die RNA verarbeitet wird und wie sich die Pegel im Laufe der Zeit ändern. Beispiele hierfür sind der Vergleich von Mutanten mit Wildtypexperimenten und ein Puls-Chase-Experiment. Die in Abbildung 3b,c gezeigten Experimente sind von diesem Typ. Für die diskontinuierliche Kennzeichnung wird eine Veränderung in das System induziert und die RNA überwacht. Sobald die Veränderung induziert wurde, muss die Kultur in mehrere Subkulturen aufgeteilt werden, und zu bestimmten Zeiten wird jede kulturisch für einen kurzen Zeitraum mit Thio-Label versehen. Ein Beispiel ist die in Abbildung 27dargestellte '-est AID 4U . Diese Art von Experiment ist besonders nützlich, um die Auswirkungen einer metabolischen Veränderung auf die RNA-Verarbeitung zu überwachen (siehe Abbildung 3d).
Eine grafische Darstellung eines Thio-Label-Experiments ist in Abbildung 4 und Abbildung 5dargestellt, und eine Tabelle, die die Leistung des Protokolls erheblich vereinfacht, ist verfügbar (siehe 4tU experiment template.xlsx). Darüber hinaus enthält die ergänzende Information einen umfangreichen Leitfaden zur Fehlerbehebung. Für das Protokoll "'-est AID 4U', das die 4tU-Kennzeichnung mit dem Auxin-Depletion-Protokoll integriert, siehe Abbildung 2 und ergänzende Abbildung 2. Siehe Barrass et al.7 für das detaillierte AID-Erschöpfungsprotokoll.
Protokoll
1. Wachstum und Thio-Kennzeichnung
HINWEIS: Die Zeit für den Abschluss dieses Abschnitts des Protokolls ist je nach Zellwachstumsrate sehr variabel. Erlauben Sie 1 h, die Lösungen und Geräte vor der Thio-Kennzeichnung vorzubereiten und 30 min nach der Kennzeichnung, um Proben zu verarbeiten.
- Stellen Sie sicher, dass der Stamm S. cerevisiae ein Plasmid enthält, das eine Permease kodiert (Tabelle 1), um den 4tU-Import in die Zelle zu steigern.
ANMERKUNG: Ohne einen Importeur ist es unwahrscheinlich, dass die Etikettierung für weniger als 2 minuten erfolgreich ist11 (siehe Ergänzende Abbildung 1). 4tU-Einbau ist effizienter, wenn das Wachstum im Medium ohne Uracil ist, so dass die Sorte URA3+; mehrere der Plasmide in Tabelle 1 tragen URA3 als Marker. Wenn dieses Protokoll mit der Erschöpfung7von '-est AID kombiniert werden soll, sind zusätzliche Dehnungsänderungen erforderlich. - Bereiten Sie YMM uracil-freies Medium vor, indem Sie 6,9 g Hefestickstoffbasis ohne Aminosäuren, 20 g Glukoseund 1,92 g SCSM-Einzelaussetzer (Materialtabelle) zu 1 L Wasser hinzufügen. Autoklav oder Filter sterilisieren das Wachstumsmedium vor Gebrauch.
HINWEIS: Die Filtersterilisation wird als Peptid-/Zuckerkomplex bevorzugt, die durch Autoklavieren mit den Zellen in dem bei der Probenentnahme verwendeten Methanol hergestellt werden. - Wachsen Sie Hefe in YMM urcil-freies Medium zu einer optischen Dichte bei 600 nm (OD600) von 0,6 -0,8. Stellen Sie sicher, dass sich die Kultur im Wachstum der Protokollphase befindet und sich mindestens zwei Verdoppelungen beläuft. Normalerweise wird ein Wachstum bei 30 °C empfohlen, aber es können andere Temperaturen beispielsweise für temperaturempfindliche Stämme verwendet werden.
HINWEIS: Je nach Stamm, Wachstumsbedingungen und RNA-Ausbeute werden ca. 30 ml Probenvolumen benötigt. Dieser Betrag wird während des gesamten Protokolls angenommen. 30 ml Kultur ist das beste, das in ein 50 ml Zentrifugenrohr mit 20 ml Methanol passt, so ist ein bequemes Volumen, um die Optimierung zu starten. Erwägen Sie die Verwendung von mehr Probenvolumen für frühe Zeitpunkte, um die RNA-Wiederherstellung zu erhöhen, bis zu 2000 ml wurden für langsamer wachsende Zellen zu wirklich kurzen Etikettierungszeiten verwendet (<1 min). - Ca. 50 ml H2O auf Eis abkühlen lassen. Für jede Probe 200 L Zirkonia-Perlen zu einem 2 ml Schraubverschlussrohr geben und auf Eis abkühlen lassen. Auch 20 ml Methanol (VORSICHT) in 50 ml Zentrifugenrohre geben und auf Trockeneis legen (VORSICHT). Das Methanol sollte 1/3 bis 2/3 das Volumen der Probe sein.
VORSICHT: Methanol ist durch Einatmen, Kontakt und Konsum giftig. Geben Sie große Mengen in einer Dunstabzugshaube und tragen Sie zwei Paar Handschuhe, da Methanol in Nitril-Laborhandschuhe eindringen kann. Methanol ist leicht entzündlich, halten Sie sich von allen Zündquellen fern.
HINWEIS: Da Trockeneis bei Kontakt zu kalten Verbrennungen führen kann und Asphixiantgas erzeugt, verwenden Sie Handschuhe bei der Handhabung und verwendung in einem gut belüfteten Raum.
HINWEIS: Das Hinzufügen der Perlen an dieser Stelle ist einfacher als nach dem Hinzufügen der Probe, da das Rohr trocken ist und beim Abspinnen des Zellpellets werden die Perlen auch aus dem Rohrfaden gesponnen, was einige Zeit spart. Darüber hinaus können die Perlen abkühlen, bevor die Probe hinzugefügt wird. - Wenn eine S. Pombe-Spitze der Kultur hinzugefügt werden soll (und nicht später), tauen Sie ein Aliquot von thiolierten S. Pombe-Zellen auf Eis und Wirbel gründlich, mindestens 30 s, dann fügen Sie der Kultur hinzu. Wenn nach den nachstehenden Anweisungen vorbereitet, reicht ein S. pombe aliquot für 400 ml Kultur aus (genug für zwölf 30 ml Proben plus ein wenig, um Fehler in der Handhabung zu ermöglichen). Wenn mehr oder weniger Kultur verwendet wird, passen Sie die Lautstärke von S. pombe an, die der Kultur hinzugefügt wird.
- Wachsen Sie 1 L der S. Pombe-Kultur auf OD600 bis 0.8 genau wie im Protokoll für S. cerevisiaebeschrieben.
- Thio-Label als Schritt 1.7, aber für 10 min.
- Beheben Sie die gesamte Kultur mit 400 ml Methanol auf Trockeneis, im Wesentlichen wie in Schritt 1.9 beschrieben.
- Pellet die Zellen durch Zentrifugation bei 3000 x g für 3 min.
- Entsorgen Sie den Überstand und setzen Sie das Zellpellet in 3,3 ml h2O wieder auf.
- Aufgeteilt in Aliquots von je 80 L. Bei -80 °C lagern.
- Verwenden Sie alle ein Aliquot für 400 ml Kultur oder 10 l pro 30 ml Probe.
HINWEIS: Reduzieren Sie das Volumen der Spitze auf 1/10, wenn RNAseq durchführen. Aliquots nicht wiederverwenden; nicht verwendete Spitzen zu verwerfen. Diese Spitze ist nützlich, um Ergebnisse über Zeitpunkte und Experimente hinweg zu normalisieren und zu vergleichen.
- Bei der diskontinuierlichen Etikettierung die erforderliche metabolische Störung (z. B. Wachstumsbedingungen, Geninduktion oder Erschöpfung wie z.B. '-est AID7 (Abbildung 2 und Ergänzende Abbildung2) induzieren und dann die Kultur aufteilen. Stellen Sie sicher, dass alle Kolben und Medien die erforderliche Temperatur haben, und befeuchten Sie das Medium, wenn möglich, bevor Sie die Kultur hinzufügen.
- 4tU zu einer Konzentration von 10 mM hinzufügen und kräftig mischen (1/10.000 des Kulturvolumens von 100 mM 4tU in 1 M NaOH aufgelöst). Thiol-Label für 15 s bis 5 min.
HINWEIS: Dreißig Sekunden sind ein guter Ausgangspunkt. Die Thio-Kennzeichnung für weniger als 20 s liefert aufgrund von Schwierigkeiten bei der Manipulation der Kultur unter Zeitdruck variablere Ergebnisse. Die Etikettierung für mehr als 1 min reduziert jedoch die zeitliche Auflösung der Technik. - Wenn ein Verfolgungsexperiment durchgeführt werden soll; Thio-Etikettierung für 20 bis 30 s zulassen und dann jagen, indem 1/200 Kulturvolumen von 1 M Uridin (nicht thioliert) zu einer Endkonzentration von 5 mM hinzugefügt werden.
HINWEIS: Uridin ist vorzuziehen, um uracil für die Jagd, wie Uridin ist mehr wasserlöslich so dass ein kleineres Volumen, um die Kultur hinzugefügt werden und so gibt es weniger Störungen des Wachstums der Zellen. - Nehmen Sie in regelmäßigen Abständen (mindestens 15 s) Kulturproben bis zum Ende des Zeitkurses. Probenahmeintervalle, die kürzer sind, sind schwierig, zuverlässig durchzuführen. Fügen Sie die Probe auf trockenem Eis in Schritt 1.4 zubereitete Methanol hinzu. Fügen Sie 30 ml Kultur in ein 50 ml-Rohr mit 20 ml Methanol ein.
HINWEIS: Kohlendioxid löst sich im Methanol auf, wenn es kalt ist; dies kommt aus lösungsweise nach Zugabe der Probe und schäumt kräftig beim Mischen, was zu Probenverlust führt. Um dies zu vermeiden, kühlen Sie das Methanol auf <-70 °C in einem dicht verschlossenen Rohr, bis es in der Nähe der Zeit benötigt wird, dann in Trockeneis übertragen. - Versiegeln Sie das Rohr und mischen Sie gründlich durch Schütteln. Legen Sie die Proben auf Eis. Überprüfen Sie, ob keine der Proben eingefroren ist. wenn ja, sanft in der Hand warm, ständig invertieren. Dies geschieht am besten in der Hand, da die Temperatur der Probe beurteilt werden kann, sollte es sich immer kalt anfühlen. Auf Eis legen. Dies ist kein Pausenpunkt; sobald die gesamte Probe flüssig ist, fahren Sie mit dem nächsten Schritt fort.
- Drehen Sie bei 3000 x g für 2 min (bei 4 °C, wenn möglich), um die Zellen zu pellet. Gießen Sie die Flüssigkeit ab und setzen Sie das Pellet in mindestens 1 ml eiskaltem Wasser wieder auf, indem Sie sanft nach oben und unten pfeifen.
HINWEIS: Das Restmethanol im Probenpellet hilft zur Resuspension. - In Schritt 1.4 auf 2 ml Schraubkappenrohre übertragen. Drehen Sie kurz (z.B. 10 s Gesamtzeit) bei >13.000 x g, um die Zellen neu zu pellet, wieder auf Eis zu legen und Flüssigkeit zu entfernen.
HINWEIS: Das Zellpellet kann mehrere Monate bei -70 bis -80 °C gelagert werden.
2. Vorbereitung der gesamten RNA
HINWEIS: Die Zeit für die Fertigstellung beträgt 90 min.
- Verwenden Sie diethylpyrocarbonat (DEPC)-behandelte Lösungen, um die RNA vor Degradation zu schützen. Aliquot die Lösungen mit Filter Pipettenspitzen und tragen Handschuhe zu jeder Zeit.
- Zu einer Lösung fügen Sie 1/1000 Volumen von DEPC und mischen durch kräftiges Schütteln.
- Lassen Sie bei Raumtemperatur (RT) für 24 h, dann Autoklav.
- Lösungen mit Amingruppen (z. B. Tris) können nicht DEPC behandelt werden. Aliquot das Pulver und speichern sie speziell für RNA-Arbeiten. Verwenden Sie zuvor DEPC behandelt E22O, um die Lösung zu machen.
HINWEIS: Da die Thio-Gruppe auf der RNA photoaktivierbar ist, minimieren Sie die Exposition gegenüber UV-Licht von diesem Zeitpunkt an. Die Lagerung sollte im Dunkeln sein und die Inkubation erfolgt am besten in einer PCR-Maschine mit Deckel.
- Wenn dem Zellpellet eine S. Pombe-Spitze hinzugefügt werden soll, anstatt der Kultur (nicht beides), fügen Sie sie jetzt hinzu. Tauen Sie ein Aliquot von thiolierten S. Pombe-Zellen auf Eis und Wirbel gründlich, mindestens 30 s, bevor Sie das Pellet hinzufügen.
HINWEIS: Bei Der Vorbereitung nach den Schritten 1.5.1-1.5.7 sind 10 l S. pombe aliquot für ein Pellet erforderlich, das aus 30 ml Kultur gewonnen wurde. - Vor dem Anziehen der Kappe, drehen Sie sehr kurz für 1-2 s, um sicherzustellen, dass keine Zirkonia Perlen zwischen der Kappe und dem Rohr gefangen sind, was dazu führen kann, dass Probe und Phenol aus dem Rohr auslaufen.
- Setzen Sie die Zellen in 400 l Acetat-EDTA(AE)-Puffer (50 mM Natriumacetat pH 5,3, 10 mM EDTA pH 8,0) durch kräftiges Wirbeln wieder aus. Fügen Sie 40 l von 10% (w/v) Natriumdodecylsulfat (SDS) hinzu. Wirbel nicht, da SDS schäumt.
- Wenn das Protokoll "'-est AID 4U" verwendet werden soll, nehmen Sie 40 l der Zellsuspension für die Proteinanalyse7. Fügen Sie 40 L AE hinzu, um das Volumen wieder auf bis zu 400 l zu erhöhen.
- Fügen Sie bei niedrigem pH-Wert 800 l Phenol (VORSICHT) und Wirbel für 10 s hinzu.
VORSICHT: Phenol ist giftig und ätzend durch Einatmen und Kontakt. Führen Sie immer Verfahren mit Phenol in einer Dunstabzugshaube durch und tragen Sie zwei Paar Handschuhe. - Lyse die Zellen in einem Homogenisator (z.B. Tabelle der Materialien) für drei 2-min Bursts bei der niedrigsten Leistungseinstellung. Lassen Sie die Proben 2 min zwischen den Homogenisierungsimpulsen auf Eis.
HINWEIS: Optimieren Sie die Bedingungen, wenn Sie andere Homogenisatoren verwenden. Unzureichendes Schütteln führt zu schlechten Erträgen, während übermäßiges Schütteln zu einer offensichtlich höheren Ausbeute führt, wie durch Absorption bei 260 nm (A260) bestimmt, aber die RNA kann abgebaut werden. Ein Homogenisator wird bevorzugt, aber heiße Phenol-RNA-Reinigung12 kann verwendet werden. - Legen Sie die lysierte Probe für 5 min auf Trockeneis, bis sie sich verfestigt, reduziert dies die Genom-DNA-Übertragung in die RNA. Nicht zu lange einfrieren, da die Probe nicht auftaut. Spin 5 min in Mikrofuge bei >13.000 x g bei RT; Seien Sie nicht versucht, dies bei 4 °C zu tun, da die Proben-/Phenolmischung während der gesamten Drehung fest bleibt, wenn sie bei niedriger Temperatur durchgeführt wird.
HINWEIS: Wenn die Probe am Ende der Drehung noch eingefroren ist, drehen Sie sich für weitere 5 min, bis die Probe vollständig aufgetaut ist. - Phenol/Chlorform-Extrakt dann Chloroform-Extrakt mit einem gleichen Volumen (ca. 600 l) Phenol:Chloroform 5:1 dann Chloroform (VORSICHT). Die Oberphase in eine andere Röhre geben, die Phenol enthält: Chloroform 5:1 oder Chloroform. Wirbel, dann drehen für 5 min in einer Mikrofuge bei RT. Dann die Oberste Phase auf ein neues 1,5 ml Rohr übertragen.
VORSICHT: Chloroform ist durch Einatmen und Kontakt toxisch. Führen Sie immer Verfahren mit Chloroform in einer Dunstabzugshaube durch und tragen Sie zwei Paar Handschuhe. - Fügen Sie ein Drittel zu einem halben Volumen (ca. 300 l) von 10 M LiCl hinzu und mischen Sie sie, um die RNA auszufällen. Die Probe sollte sofort trüb werden, aber mindestens 10 min auf Eis oder bei 4 °C lassen (nicht unter -20 °C lagern, da sie einfrieren wird), oder bis der Niederschlag flockt.
- Drehen Sie für 5 min bei >13.000 x g in einer Mikrofuge. Entfernen Sie die Flüssigkeit, drehen Sie kurz neu und entfernen Sie die Dregs. Pellet mit 300 bis 500 l Ethanol waschen, kurz drehen und restliches Ethanol entfernen.
ANMERKUNG: Während dieser Wähen halten Sie das Pellet auf der gleichen Seite des Rohres wie die erste Drehung, auf diese Weise wird das Pellet nicht bewegen und brechen; wenn es bricht, könnte ein Teil der RNA versehentlich verloren gehen.
HINWEIS: Trocknen Sie das Pellet nicht; solange der größte Teil der Flüssigkeit entfernt wurde, wird es die nachfolgenden Schritte nicht stören. Die RNA kann in dieser Phase auch einige Monate bei -20 °C oder -70 bis -80 °C für eine Langzeitlagerung gelagert werden. - Lösen Sie das RNA-Pellet in 90 l TE pH 7.0 (10 mM Tris HCl pH 7.0, 1 mM EDTA pH 8.0) durch Erhitzen bei 65 °C mit Schütteln wieder auf, da das RNA-Pellet schwer wieder aufzulösen sein kann. Dies darf nicht mehr als 5 min sein, da die RNA bei höheren Temperaturen abgebaut wird. Überprüfen Sie die vollständige RNA-Löslichkeit und übertragen Sie sie dann in ein 0,2 ml-Rohr. Pipette die Probe nach oben und unten; es sollte keine "Klumpen" geben, und die Flüssigkeit sollte in der Spitze aufsteigen und sanft fallen. Diese Lösung ist zähflüssig, so dass die letzte Pipettierbewegung langsam sein sollte.
HINWEIS: Die RNA kann in diesem Stadium bei -20 °C im Dunkeln gespeichert werden; dies kann auch vorteilhaft für die RNA-Löslichkeit sein.
3. Biotinylierung
HINWEIS: Die Zeit für die Fertigstellung beträgt 60 min. Die folgenden Schritte werden bequem in einem Streifen von Rohren mit integrierten Kappen durchgeführt, da sie weniger Tendenz haben, sich auf Wirbeln zu öffnen, als Streifen mit separaten Kappen.
- Biotinylat durch Zugabe von10 l (1 /10 Endvolumen) einer 5 mM HPDP-Biotin-Lösung (MTS-Biotin kann genau wie HPDP-Biotin verwendet werden) in die RNA einfügt und gründlich mischen. Die RNA vordemererererern die RNA für nicht mehr als ein paar Sekunden bei 65 °C, bevor das Biotin hinzugefügt wird. Bei 65 °C für 15 min bis maximal 30 min im Dunkeln inkubieren.
HINWEIS: Diese Erwärmung ist erforderlich, da einige HPDP-Chargen bei RT in der RNA-Probe ausfallen. Ein PCR-Block mit beheiztem Deckel ist dafür ideal. - Bereiten Sie ein kleines Harzvolumen, Größenausschlussspalte (Materialtabelle) vor, um das nicht inkorporierte Biotin auszuschließen. Entfernen Sie das untere Tag der Säule und lösen Sie die Kappe, legen Sie sie in ein 2 ml Zentrifugenrohr. Drehen Sie bei 1500 x g für 1 min, um den Puffer zu spülen Fügen Sie 0,3 ml VON TE sanft an den oberen Rand der Säule und drehen Sie wieder. Wiederholen Sie das Waschen und drehen Sie zweimal für insgesamt 3 Wäschen. Schließlich die gewaschene Säule in ein frisches 1,5 ml Rohr übertragen.
- Sobald die Probeinkubation (Schritt 3.1) abgeschlossen ist, fügen Sie das Beispiel am oberen Rand der Spalte hinzu. Drehen Sie bei 1500 x g für 2 min. Die biotinylierte RNA-Probe befindet sich nun im Boden des Rohres.
HINWEIS: Eine Drehung von 1 min reicht nicht aus, um die gesamte Probe zu entwirn. - Fügen Sie ein Drittel zu einem halben Volumen (ca. 40 l) von 10 M LiCl hinzu, mischen Sie, um die RNA als Schritt 2.10 neu auszustoßen. Die Probe sollte sofort trüb werden, aber mindestens 5 min auf Eis oder bei 4 °C oder bis der Niederschlag sprikisiert; nicht unter -20 °C lagern, da es einfrieren wird. Zentrifuge die Probe für 5 min bei >13.000 x g in einer Mikrofuge.
- Waschen Sie mit 80% Ethanol, 1 h rotierend. Befolgen Sie das Verfahren in Schritt 2.11, um so viel Flüssigkeit wie möglich zu entfernen.
HINWEIS: Da HPDP-Biotin in 80% Ethanol sehr löslich ist, ist dies ein zusätzlicher Reinigungsschritt. - Wiederholen Sie die 80% Ethanolwäsche, um so viel nicht eingebautes Biotin wie möglich zu entfernen.
HINWEIS: Die RNA kann in diesem Stadium auch bei -20 °C im Dunkeln gespeichert werden.
4. Reinigung der neu synthetisierten RNA
HINWEIS: Die Zeit für die Fertigstellung beträgt 2 h.
- Lösen Sie die RNA in 200 l DEPC-behandelten H2O(65 °C Inkubation kann verwendet werden, ähnlich wie das Verfahren in Schritt 2.12).
- Messung der RNA-Konzentration bei A260 mit einem Spektralphotometer; Die Probe muss möglicherweise 1/10 verdünnt werden, um sie innerhalb des linearen Bereichs des Spektralphotometers zu erhalten. Wirbel diese Verdünnung für mindestens 10 s, um sicherzustellen, dass die viskose RNA gleichmäßig gelöst wird.
HINWEIS: Die Wirksamkeit der Biotinylierung kann bei Bedarf mit Punktblot13 beurteilt werden. - Fügen Sie gleiche Mengen an RNA zu einem frischen Röhrchen hinzuund machen Sie bis zu 200 l in DEPC-behandeltem H2 O. Verwenden Sie die gesamte Probe mit der niedrigsten RNA-Konzentration und verwenden Sie ein geeignetes Volumen der anderen Proben, um jeweils eine ähnliche RNA-Menge zu haben.
HINWEIS: Die Tabelle 4tU experiment template.xlsx verfügt über ein Formular, das diese Berechnung unterstützt. - Wenn die Probe bei RT ist, fügen Sie 25 l von 10 x NaTM Puffer (0,1 M Tris HCl pH 7,0, 2 M NaCl, 250 mM MgCl2), 25 l von 1 M NaPi pH 6,8 (0,5 M NaH2PO4 0,5 M Na2HPO4) und 2,5 l von 10% SDS. Gründlich mischen und sanft drehen (<30 s; ca. 100 x g).
HINWEIS: Um eine Ausfällung der SDS und der Salze zu vermeiden, müssen die Proben während der folgenden Verfahren bis Schritt 4.13 bei RT aufbewahrt werden. - Erstellen Sie den Perlenpuffer mit 1x NaTM-Puffer, 0,1 M NaPi und 0,1% SDS, 2 ml pro Probe. Fügen Sie zuerst die erforderliche Menge von H2O und das SDS zuletzt hinzu. Diese muss jedes Mal als Niederschlagsform nach 24 h frisch gemacht werden.
HINWEIS: Um die Bildung von Ausscheidungen zu vermeiden, muss der Perlenpuffer während der folgenden Verfahren bei RT gehalten werden. DePC nicht behandeln oder Autoklaven. - Fügen Sie 50 l Streptavidinperlen zu einem 1,5 ml-Rohr mit niedriger Retention hinzu. Legen Sie das Rohr auf das Magnetgestell, warten Sie, bis sich die Perlen absetzen, und entfernen Sie dann die Flüssigkeit
- Waschen Sie die Streptavidin Perlen.
- Fügen Sie 200 L Perlenpuffer und Wirbel hinzu, bis das Perlenpellet vollständig wieder freigegeben ist. In der Regel ist 3 x 5 s alles, was erforderlich ist. Für Washes, bevor die RNA-Probe hinzugefügt wird, reicht es aus, die Röhren umzudrehen, damit die Perlen über die Röhre auf die andere Seite wandern. Dann drehen Sie die Rohre zurück auf die ursprüngliche Seite, so dass die Perlen wieder über die Röhre wandern.
- Drehen Sie das Rohr bei niedriger Geschwindigkeit (ca. 100 x g) für maximal 5 s, um die Flüssigkeit herunterzudrehen, aber nicht die Perlen.
- Legen Sie in das magnetische Rack, damit die Perlen vom Magneten erfasst werden können.
- Entfernen Sie die Flüssigkeit durch Aspiration für eine kleine Anzahl von Proben; die Flüssigkeit abgießen, wenn viele Proben.
HINWEIS: Bei einer großen Anzahl von Proben kann das Entfernen der gesamten Flüssigkeit rein durch Aspiration problematisch sein, da die Perlen in der ersten Probe ausgetrocknet werden können, bevor die letzte Probe fertig ist, dies erhöht den Hintergrund. Das Waschen kann beschleunigt werden, indem die Flüssigkeit aus allen Proben auf einmal auf dem Magneten abgegossen wird. Sie sollten vor dem Gießen etwas länger auf dem Magneten gelassen werden und die kleine Menge an Flüssigkeit, die übrig bleibt, muss abgesaugt werden, aber insgesamt bedeutet es weniger Zeit auf dem Magneten und ohne Flüssigkeit. Auf diese Weise ist es möglich, 24 oder mehr Extraktionen schnell zu tun.
- Block mit 200 l Perlenpuffer, 10 l 20 mg/ml Glykogen und 2,5 l 5 mg/ml tRNA, 20 min rotierendes Ende über Ende bei moderater Geschwindigkeit bei RT. Die Rotation besteht darin, die Perlen in Suspension zu halten. Sobald die Blockierung vollständig ist, entfernen Sie die Flüssigkeit in den Schritten 4.7.2-4.7.4 und waschen Sie sie erneut, wie die Schritte in Abschnitt 4.7.
- Setzen Sie die Perlen in der Probe wieder aus. Inkubieren bei RT mit Drehung für 30 min.
- Während der Inkubation ein frisches 1,5 ml Rohr für jede Probe vorbereiten. Fügen Sie 1/10 Volumen (ca. 10 l) von 3 M Natriumacetat pH 5,3 und 20 g Glykogen hinzu und drehen Sie bei ca. 100 x g für 3 s. In einem Rack lagern, bis es erforderlich ist.
- Entfernen Sie die ungebundene RNA aus den Perlen, wie die Schritte 4.7.2-4.7.4. Die ungebundene RNA kann in einem frischen Rohr ausgeharrt werden, aber die Salze und SDS machen es sehr schwierig zu reinigen. Dann waschen Sie die Perlen, wie Abschnitt 4.7 mit Wirbeln, für ein Minimum von 3 bis maximal 5 Mal.
- Nach der letzten Wäsche besondere Sorgfalt auf die gesamte Flüssigkeit zu aspirieren; zurück zu jedem Rohr und die Dregs des Puffers wieder ansaugen.
- Um die RNA zu entbluten, fügen Sie den Perlen 50 l frisch zubereitetes 0,7 M-Mercaptoethanol (ME) hinzu(1/20 Verdünnung der kommerziell gelieferten Stofflösung). Wirbel und drehen Kurz, wie die Schritte 4.7.1 und 4.7.2. Legen Sie die Gülle in das magnetische Rack und pipettedien Sie die RNA-haltige Lösung in das in Schritt 4.10 hergestellte 1,5 ml Zentrifugenrohr.
- Elute noch einmal als Schritt 4.13, um Die restliche RNA aus den Perlen zurückzugewinnen und die eluierte Probe in das Rohr zu geben, das die erste Elution aus diesen Perlen enthält.
- Entfernen Sie Restperlen aus der eluierten RNA, indem Sie die Probe wieder in das magnetische Rack legen und die Flüssigkeit in ein frisches, niedrig bindendes 0,5 ml Zentrifugenrohr übertragen.
- Mischen Sie die Probe und fällen Sie dann die nsRNA, indem Sie 2,5x Volumina (280 l) Ethanol hinzufügen und noch einmal mischen. 1 h bis über Nacht bei -20 °C lassen. Drehen Sie in einer vorgekühlten Zentrifuge (4 °C) für 20 min bei der maximalen Geschwindigkeit (mindestens 13.000 x g).
- Gründlich mit 200 l 70% Ethanol bei -20 °C waschen. Da die Rest-ME nachgelagerte Anwendungen hemmt, drehen Sie sich bei jedem Schritt, um so viel wie möglich von den Dregs zu entfernen; am Ende sollte die Probe nicht nach ME riechen.
- Lösen Sie sich in 10 bis 20 l DEPC-behandeltem 1x TE mit dem Äquivalent von 0,005 l RNase-Inhibitor auf.
HINWEIS: Alle nachfolgenden Stufen sollten auf Eis durchgeführt werden. - Messen Sie die RNA-Konzentration und -Reinheit.
- Messen Sie das A260 und A225 in einem Spektralphotometer mit geringer Probenvolumen.
ANMERKUNG: Ein Absorptionsmaximum nahe 225 nm stammt aus einem unvermeidbaren Schadstoff aus den Perlen. In Ermangelung von RNA sinkt das Signal des Schadstoffs auf 35% bei n = 260 nm. Daher wird die tatsächliche RNA-Menge durch die Formel angenähert: (A260-(A225*0.35)*40 ng/'L. - Alternativ können Sie die Probe auf einem mikrofluidischen Elektrophoresesystem wie einem Bioanalyzer analysieren.
HINWEIS: Diese Analyse ist der Verwendung eines Spektralphotometers vorzuziehen, da die RNA-Integrität beurteilt werden kann, der Schadstoff die Quantifizierung nicht beeinträchtigt und weniger Proben erforderlich sind.
- Messen Sie das A260 und A225 in einem Spektralphotometer mit geringer Probenvolumen.
- Analysieren Sie die nsRNA.
HINWEIS: Beispielsweise können bestimmte RNAs durch Standard-Reverse-Transkriptase-qPCR-Techniken quantifiziert werden. Die auf diese Weise hergestellte RNA ist mit der Bibliotheksvorbereitung für RNA-seq kompatibel. Die Entfernung von rRNA ist für die Kennzeichnungszeiten von weniger als 5 min nicht erforderlich.
Ergebnisse
Typische Erträge für nsRNA, die mit diesem ers4tU-Protokoll zurückgewonnen werden, sind in Abbildung 1bdargestellt, dies wurde von einem Bioanalyzer erzeugt und die Spur zeigt die Ausbeute von RNA versus Größe (Nukleotide [nt]). Beachten Sie sowohl in der Bioanalyse-Spur als auch im Einfügediagramm, dass die RNA-Wiederherstellung vom Zeitpunkt 0 ein sehr kleiner Teil dessen ist, der sich aus längeren Zeitpunkten erholt hat - etwa 0,3 g RNA, die aus etwa 109 Zellen zurückgewonnen wurde, verglichen mit mehr als doppelt so viel nach nur 30 s Etikettierung (0,8 g nsRNA) aus der gleichen Anzahl von Zellen. Die RNA-Wiederherstellung bei 15 s ist variabler, da kleine Unterschiede bei der Durchführung der Probenahme einen proportional größeren Effekt auf die RNA-Wiederherstellung haben. In der Bioanalyse-Spur können rRNA-Vorläufer als ein Peak nahe 1000 nt und ein Doublet von Spitzen bei 1700-1800 nt gesehen werden. Die Fülle dieser Zwischenprodukte nimmt zu, wenn die Thiolation anhält.
Thio-Kennzeichnung wurde verwendet, um die Spleißung des ACT1-Transkripts zu quantifizieren (Abbildung 3). Die Thiolation wurde durchgeführt und Proben in 15 s Abständen ab Beginn der Thio-Kennzeichnung und der Verarbeitung von ACT1-RNA überwacht (Abbildung 3a,b). Wie man sieht, wird Pre-mRNA (durch Transkription) und Lariate (durch den ersten Schritt des Spleißens von pre-mRNA) erzeugt, auch nach nur 15 s Etikettierung. Nach ca. 45 s bis 1 min erreichen die Mengen an Lariaten und Pre-mRNA das Gleichgewicht, wobei so viele dieser RNA-Arten durch Transkription erzeugt werden, wie durch Spleißen verarbeitet werden.
Um die in Abbildung 3c dargestellten Daten zu erzeugen, wurde die Sorte mit 4tU für 25 s gepulst und dann mit Uridin gejagt. Die Erzeugung von Pre-mRNA und Lariats erreicht maximal 1 Minute. Dies ist im Vergleich zu Abbildung 3bgut vergleichbar; das Maximum, das nach 45 s erreicht wird, um das Gleichgewicht zu erreichen, zuzüglich der 25 s der Etikettierung. Nach dem Höhepunkt sinken die Pegel, wenn die thio-markierten RNAs durch den Spleißprozess gejagt werden.
Abbildung 3d zeigt die Erschöpfung eines Proteinsplegierfaktors und seine Wirkung auf den RNA-Stoffwechsel, mit dem System6,7. Hier wird Prp16p nach 25 min Erschöpfung von nahezu physiologischen Werten auf 5% dieses Niveaus reduziert. Prp16p ist ein wesentlicher Spleißfaktor für den zweiten Schritt des Spleißens15. Lariate werden während des zweiten Schritts des Spleißens entfernt (Abbildung 3a), aber hier erhöhen sie sich über das Niveau der Pre-mRNA, wenn Prp16 begrenzt wird. In späteren Erschöpfungszeiten werden andere Faktoren aufgrund von Sekundäreffekten begrenzt, so dass der Lariatspiegel abnimmt und der Pre-mRNA-Spiegel steigt. Der Gehalt an gespleißter mRNA sinkt.
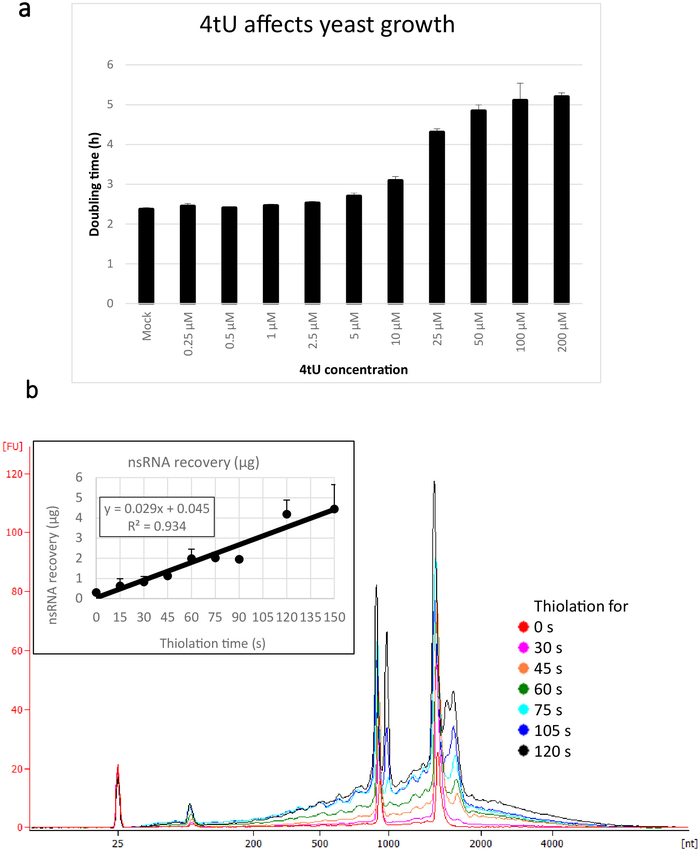
Abbildung 1: Wachstum bei der 4tU- und RNA-Wiederherstellung. (a) 4-Thiouracil wirkt sich auf das Wachstum aus. Die Erhöhung der Konzentration von 4tU im YMM-Drop-out-Wachstumsmedium ohne Uracil erhöht die Verdoppelungszeit von S. cerevisiae (BY4741) mit dem p4Fui-PEST-Plasmid. Das Wachstum von vier Replizienkulturen wurde bei 30 °C in einem Tecan Infinite Pro 200 überwacht. Alle Kulturen befanden sich durchweg in der Protokollphase, wobei OD600 zwischen 0,1 und 0,6 lagen. Mock ist eine Kontrollkultur mit einer entsprechenden Menge an NaOH hinzugefügt, die an sich nicht die Wachstumsrate ändert. Diese Grafik zeigt, dass die Kennzeichnung zwischen schneller Kennzeichnung und Beschädigung der Zelle einen Kompromiss ist. Fehlerleisten sind Standardfehler von 4 Replikationen. (b) die nsRNA-Ausbeute steigt linear ab etwa 15 s der Etikettierung. Die Hauptfigur zeigt die Bioanalysespuren von gereinigten, nsRNA von 0 (nicht thioliert) bis 2 min nach Zugabe von 4tU in 15 s Intervallen. Beachten Sie, dass die 15 s-Probe nicht angezeigt wird, da sie nicht von der nicht gekennzeichneten Probe zu unterscheiden war. Die beiden großen Spitzen entsprechen ribosomalen RNAs (rRNAs). Die rRNA-Vorstufen und Zwischenprodukte sind als mehrere Spitzen mit einem höheren Molekulargewicht als reife rRNAs sichtbar. Die Wiederherstellung dieser Vorstufen und Zwischenprodukte nimmt mit der Zeit zu. Gezeigt werden Ergebnisse aus einem repräsentativen Experiment. Das Einsetdiagramm zeigt die Erholung von nsRNA mit zunehmender Inkubation mit 4tU. Die Ausbeute von nsRNA steigt mit zunehmender Wachstumszeit mit 4tU. Die Erholung ist während des gesamten Zeitrahmens dieses Experiments bemerkenswert linear (R2 = 0,934) und zeigt einen leichten Anstieg gegenüber dem Hintergrund, selbst bei 15 s Etikettierung mit 4tU, obwohl sie nicht von der nicht gekennzeichneten Probe per Auge vom Bioanalyzer unterschieden werden kann. Spur. Fehlerbalken zeigen Standardfehler für drei biologische Replikationen an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
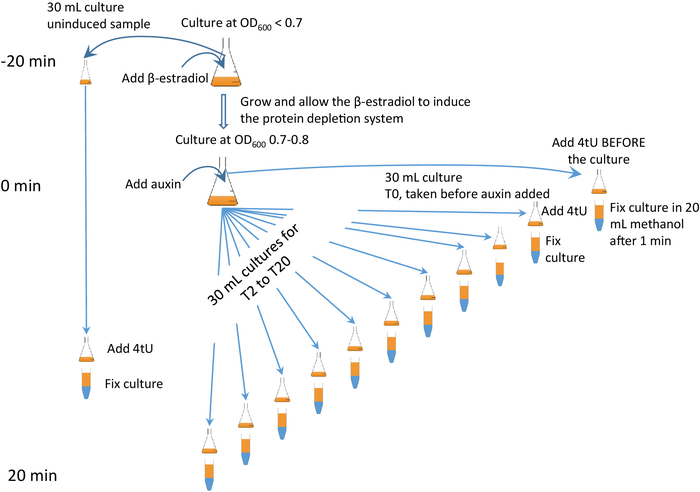
Abbildung 2: grafisches Protokoll von é-est AID 4U . Eine grafische Zusammenfassung des Protokolls des Protokolls von '-est AID 4U. Die Expression des auxin induzierbaren Degron-Systems (AID), das wiederum ein AID*-markiertes Zielprotein ausschöpft, fördert die Expression des auxin induzierbaren Degron-Systems (AID), das wiederum ein AID*-markiertes Zielprotein aush. In diesem Fall wird die Degron-Systemexpression 25 min eingeleitet, bevor der Proteinabbau beginnt und die Thiolation zu jedem Zeitpunkt 1 min. ist. Proben werden vor der Induktion und alle 2 Minuten während der Erschöpfung entnommen. Eine animierte Version wird in der Ergänzenden Abbildung 2angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
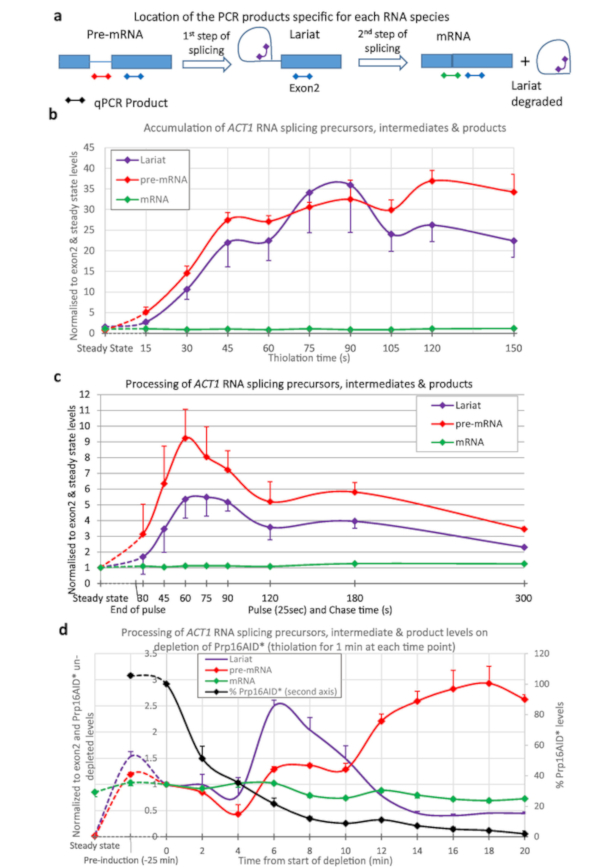
Abbildung 3: Vorläufer und Zwischenprodukte der ACT1-RNA-Spleißung. Das Spleißen von ACT1 Pre-mRNA-Transkripten wurde durch quantitative Reverse-Transkription PCR16überwacht. Die Konzentrationen von ACT1-Vorläufer (pre-mRNA), Zwischenlariat-Exon2 (Lariat) und spliciertem Produkt (mRNA) werden normalisiert gegen das Niveau von ACT1 Exon2 und den stationären Zustand dieser RNAs dargestellt. (a) Position der qPCR-Produkte auf dem ACT1-Transkript. Schematische Lage der qPCR-Produkte, die verwendet werden, um die Gehalte an Vorprodukten, Zwischenprodukten und Produkten der Spleißreaktion der ACT1-Transkripte16zu asseraten. Exons werden durch Felder, Intron als Linie und die qPCR-Produkte als Linien mit Diamanten an beiden Enden dargestellt, wobei die Farbe mit der farbe übereinstimmt, die in den Diagrammen verwendet wird. Die Pre-mRNA PCR ist spezifisch für pre-mRNA und keine Zwischenprodukte des Spleißens, da dieses Produkt den Astpunkt kreuzt, der nach dem ersten Schritt des Spleißens gestört wird. Lariat PCR erkennt das Produkt des ersten Schritts des Spleißens und das verbrauchsteuerne Lariat, das nach dem zweiten hergestellt wird. Die mRNA PCR ist spezifisch für das Produkt spleißen, mRNA. Die Ergebnisse der Exon-PCR (in allen Vorstufen, Zwischenprodukten und Erzeugnissen, mit Ausnahme des ausgeschnittenen Lariats) sind in den Schaubildern nicht dargestellt, da diese zur Normalisierung der Daten verwendet wurde und daher immer gleich 1 ist. (b) Kontinuierliche Thiolabelling. Die Menge der Pre-mRNA erhöht sich mit der Zeit, da 4tU durch Transkription aufgenommen wird und nach einer kurzen Verzögerung in Lariat-Exon2-Zwischenprodukte und Spleißprodukte umgewandelt wird. Die Konzentrationen dieser Prä-mRNA- und Lariat-Arten sind über dem Hintergrund nach nur 15 s Wachstum mit 4tU nachweisbar und erreichen ein Maximum nach etwa 45 s kontinuierlicher Kennzeichnung mit 4tU, wobei ihre Produktion durch Umwandlung in gespleißte mRNA und/oder Abbau. Die Werte werden auf ihren stabilen Zustand normalisiert (links-der Punkt des Graphen), und exon 2 Ebenen, um ihr Aussehen und ihre Verarbeitung im Vergleich zur Transkription von Exon 2 anzuzeigen. Da die RNA-Spleißung von ACT1 weitgehend ko-transkriptionsiv4ist, wird17 spleißte mRNA schnell zur am häufigsten vorkommenden Art, ihr Gehalt ähnelt dem von Exon 2. Standardfehler von drei biologischen Repliken, die jeweils in Dreifachausführung geprüft werden. (c) Puls/Jagd. Thiolationspuls von 25 Sekunden gefolgt von Verfolgungsjagd mit Uridin. Im Vergleich zu den stationären Niveaus dieser RNAs (links-punkt), Sie sind zunächst sehr reichlich in der neu synthetisierten Pool. Die Werte sinken allmählich, wenn sie zu mRNA verarbeitet (oder degradiert) werden, und nähern sich dem stationären Niveau um 5 min. Standardfehler von drei biologischen Replikationen, die jeweils in Dreifacharbeit untersucht werden. (d) nsRNA und Proteinmangel. Spleißen von ACT1 Pre-mRNA-Transkripten, die durch quantitative Reverse-Transkription PCR wie im Panel (a) nach Erschöpfung des Prp16-Proteins unter Verwendung des Auxin-Degron-Systems überwacht werden, wie in Abbildung 2beschrieben. Die Prp16-Proteinspiegel werden auch in der Grafik angezeigt, die gegen die zweite Y-Achse als Prozentsatz der Werte vor der Auxin-Erschöpfung dargestellt wird. Prp16 ist ein wichtiger Bestandteil des Spleceooms, besonders wichtig für den zweiten Schritt der Spleißung in Panel (a), nach dem Lariate abgebaut werden. Wenn dieser Schritt wird begrenzende Lariate anhäufen sich zunächst. Zu späteren Zeiten fällt das Spleißen vollständig aus, Lariate werden nicht mehr produziert und die Pre-mRNA-Spiegel steigen. Fehlerbalken sind Standardfehler von drei biologischen Replikationen, die jeweils in Dreifachausführung untersucht werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
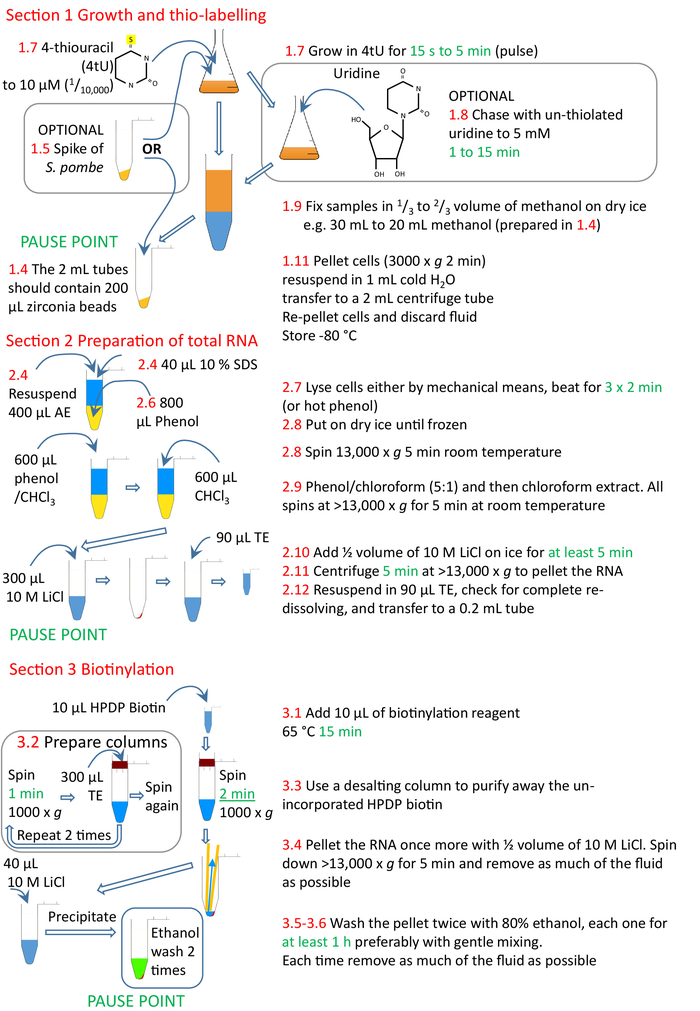
Abbildung 4: Grafische Zusammenfassung der Protokollabschnitte 1 bis 3. Die Zellen werden mit 4tU thioliert und dürfen wachsen, um das modifizierte Nukleotid in die RNA zu integrieren. Eine thiolierte S. Pombe-Spitze kann hinzugefügt werden, um eine Normalisierung über Zeitpunkte und Experimente zu ermöglichen. Der Puls von 4tU kann mit unthioliertem Uridin gejagt werden. Die Etikettierung kann entweder kontinuierlich von 4tU-Zugabe oder von einer Änderung zu Wachstumsbedingungen durchgeführt werden, die Kultur spaltet und 4tU zu Beginn der Wachstumsbedingung ändern, aber jede Kennzeichnung nur für eine kurze Zeit. Die Zellen werden gesammelt und RNA aus den Zellen hergestellt, vorzugsweise mit einem Homogenisator und phenolbasierten Methoden. Die RNA wird biotinyliert und dann die biotinylierte RNA aus nicht inkorporiertem Biotin mit einer Größenausschlussspalte gereinigt. Die nsRNA ist nun bereit für die Reinigung mit Streptavidinperlen (Abschnitt 4, Abbildung 5). Zahlen in Rot entsprechen den Schrittnummern im Protokoll. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
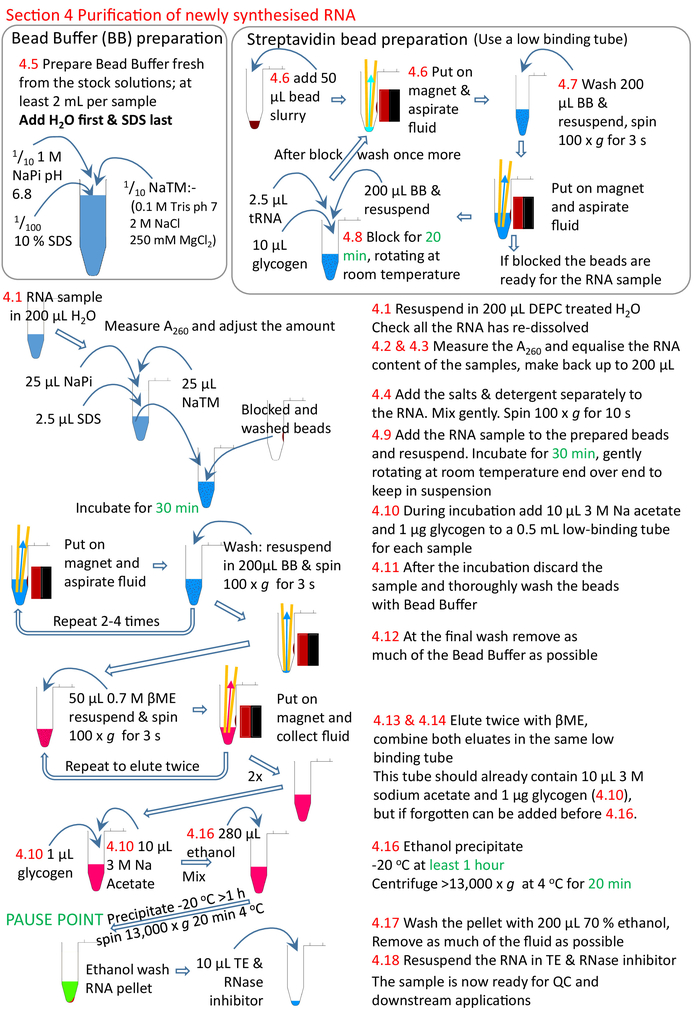
Abbildung 5: Grafische Zusammenfassung des Protokollabschnitts 4. Nach den Abschnitten 1 bis 3 (Abbildung4) werden die Streptavidinperlen blockiert und die biotinylierte RNA-Probe den vorbereiteten Perlen zugesetzt. Die biotinylierte RNA bindet an die Streptavidinperlen und die un-biotinylierte RNA entfernt und gewaschen. Die biotinylierte RNA wird aus den Perlen mit Hilfe von "ME" eluiert und für weitere Forschungsarbeiten ausgefällt. Zahlen in Rot entsprechen den Schrittnummern im Protokoll. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: Verbesserung der nsRNA-Rückgewinnung aus Hefezellen mit und ohne zusätzliche Kopien des Importeurs bei 1 und 3 Minuten Thio-Kennzeichnung. Beachten Sie, dass Fui1 der eigene Promotor der Hefe ist, der aus einem 2 m Plasmid ausgedrückt wird. Die genomische Kopie dieses Gens ist in beiden Stämmen vorhanden. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 2: Animierte Version des grafischen Protokolls von '-est AID 4U '-est AID 4U. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 1: 4tU_experiment_template.xltx. Bitte klicken Sie hier, um diese Datei herunterzuladen.
| PlasmidName | Importeur/Permease | anschreiber | bemerkung | |
| p4Fui | S. cerevisiae Fui1 | URA3 | Fui1 importiert Uracil und Uridin, was es ideal für Puls-/Chase-Experimente macht. | |
| pAT2 | S. cerevisiae Fui1 | LEU2 | ||
| p4Fui-PEST | S. cerevisiae Fui1 | URA3 | Das PEST-Motiv von Fui1 wurde deaktiviert, so dass die Permease nicht abgebaut wird, wenn es genügend intrazelluläre Uracil für die Bedürfnisse der Zelle gibt. Funktioniert gut bei der Kennzeichnung von Experimenten und verbessert die Puls-/Chase-Leistung. | |
| p4Fur | S. cerevisiae Fur4 | URA3 | Uracil permease | |
| YEpEBI311 | H. Sapiens ENT1 | LEU2 | Miller et al.11. Enthält auch ein HSV-Thymidin-Kinase-Gen. | |
| (gleichgewichtiver Nukleosidtransporter) | ||||
| Alle Plasmide sind 2 m basiert. Alle p4-Plasmide und pAT basieren auf der Serie pRS16 von Plasmiden. FUI1 und FUR4 werden von ihren eigenen, endogenen Projektträgern ausgedrückt. | ||||
Tabelle 1: Plasmide, die mit diesem Protokoll verwendet werden.
Diskussion
Dieser Artikel stellt ein Protokoll für eine extrem schnelle und spezifische 4tU-Kennzeichnung zur Rückgewinnung von aufkeimender, neu synthetisierter RNA aus S. cerevisiae nach nur 15 s Etikettierung dar, mit sehr geringer Kontamination durch unbeschriftete RNA.
Der Anwender sollte immer darauf achten, die Integrität der RNA durch die Verwendung von kalten Temperaturen und DEPC-behandelten Reagenzien zu erhalten. Streptavidin Perle Reinigung ist in der Regel zuverlässig; Der Perlenpuffer ist jedoch schwer zu handhaben. es muss frisch gemacht werden, mit seinen Komponenten in der richtigen Reihenfolge hinzugefügt, und nicht gekühlt oder autoklaviert. Häufige Mängel sind die unvollständige Auflösung der RNA nach den Fällungsschritten und daher entweder nicht biotinyliert oder anderweitig während der Verarbeitungsschritte verloren. Im Ergänzungsmaterial gibt es umfangreiche Hilfe zur Fehlerbehebung.
Es gibt einige Einschränkungen, die in ers4tU zu beachten sind. Eine bereits erwähnte ist, dass 4tU verlangsamt das Wachstum der Hefe (Abbildung 1a). Abgesehen von endogen thiolierten RNAs9können mit diesem Verfahren nur RNAs gereinigt werden, die während des Etikettierungszeitraums transkribiert wurden. Polymerasen, die während der Thiolationszeit an Genen angehalten wurden, erzeugen keine thiolierten Transkripte, die gereinigt werden können, obwohl Transkripte, die teilweise aufgrund von Polymerasen gekennzeichnet sind, die während der Thiolation in einen angehaltenen Zustand eintreten oder einen angehaltenen Zustand verlassen, wiederhergestellt werden können. Stämme, die schlecht transkribieren, entweder aufgrund von Mutation oder Wachstumsbedingungen, produzieren wenig nsRNA, obwohl die hier verwendeten Techniken dennoch die Erholung von nsRNA im Vergleich zu anderen Methoden verbessern werden. Längere Zeiten und erhöhte Kulturvolumina können in diesen Stämmen und Bedingungen notwendig sein. Beachten Sie, dass Uracil eine gute Stickstoffquelle ist und daher diese Methode vor der Verwendung für Studien mit Stickstoffhunger erprobt werden sollte.
Das ers4tU-Protokoll ist besonders nützlich für die Analyse kurzlebiger RNAs, von denen viele so schnell abgebaut werden, dass sie nicht identifiziert werden können, ohne die Abbaumaschinerie zu lähmen. Beispiele sind kryptische unstable Transkripte (CUTs)4, und kurze Transkripte durch vorzeitige Beendigung oder Promoter proximal Pausierung18 und Antisense Transkription "upstream" von einem Promoter (PROMPTs)19. Die bei der Verarbeitung stabiler RNA-Arten produzierten Zwischenprodukte sind ebenfalls vorübergehend, können aber mit ers4tU-Transkription4angereichert werden. Das ers4tU-Protokoll ist daher außergewöhnlich, wenn es darum geht, hochtransiente RNA-Arten unter nahezu physiologischen Bedingungen zu analysieren und zu erfassen, was einen großen Vorteil gegenüber anderen Methoden darstellt. Diese Technik wurde verwendet, um Transkription und nachgeschaltete RNA-Verarbeitung Kinetik in RNA-Polymerase-Mutanten zu studieren, die schneller oder langsamer als normale20erlälten.
Thiolation ist auch kompatibel mit RNA-seq und SLAM-seq21, so dass alle RNA produziert innerhalb eines sehr kurzen Zeitfensters in exquisiten Details charakterisiert werden.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde durch Wellcome-Mittel an JB [104648] unterstützt. Die Arbeit im Wellcome Centre for Cell Biology wird durch Wellcome-Kernförderung unterstützt [092076]. Die Autoren würdigen mitglieder des Labors für ihre Hilfe: Bella Maudlin, Emanuela Sani, Susanna De Lucas-Arias und Shiney George. Die Autoren danken auch Patrick Cramer für das Plasmid YEpEBI31111.
Materialien
| Name | Company | Catalog Number | Comments |
| β-mercaptoethanol (βME) | Sigma-Aldrich | M3148 | CAUTION toxic. Stock solutions are aproximatly 14 M, make at 1/20 dilution for use |
| Chloroform | Sigma-Aldrich | 25668 | CAUTION toxic |
| Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758 | add 1/1000 volume to a solution, leave at room temperature for 24 h, then autoclave |
| DMF (N,N-dimethylformamide) | Sigma-Aldrich | 227056 | CAUTION toxic |
| EDTA | Sigma-Aldrich | 3609 | Make 0.5 M and pH to 8.0 with sodium hydroxide |
| Ethanol | Sigma-Aldrich | 29221 | |
| EZ-link HPDP Biotin | Thermo scientific | 21341 | Store protected from light. Disolve all the vial contents in 22.7 mL DMF (to make a 4 mM stock solution). Store away from water, in the dark & at -20 °C. Check the solution before using, as some batches of HPDP precipitate in storage; heat at 42 °C to resuspend. |
| Glucose | Fisher Scientific | G/0500/60 | |
| Glycogen [20 mg/mL] | Sigma-Aldrich | 10901393001 | Store at -20 °C |
| Immobilised TCEP Disulfide Reducing Gel | Thermo Scientific | 77712 | Optional |
| LiCl | Sigma-Aldrich | 793620 | 10 M solution. CAUTION: this gets very hot as is dissolves and can even boil at greater than 100 oC, add the LiCl crystals to the water slowly. |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 63033 | 1 M solution. CAUTION: this gets very hot as is dissolves and can even boil at greater than 100 oC, add the MgCl2 crystals to the water slowly. |
| Methanol | Fisher Scientific | M/4000/PC17 | CAUTION Toxic and flammable |
| NaH2PO4 | Sigma-Aldrich | S3139 | Make 1 M solutions of each and mix in equal amount to obtain a solution of the appropriate pH |
| Na2HPO4 | Sigma-Aldrich | S3264 | |
| NaCl | Sigma-Aldrich | S9888-M | 5 M solution |
| Phenol, low pH. | Sigma-Aldrich | P4682 | Store in the dark at 4 °C. CAUTION toxic |
| Phenol Chloroform 5:1 (125:24:1) low pH. | Sigma-Aldrich | P1944 | Store in the dark at 4 °C. CAUTION toxic |
| Pierce Spin Columns | Thermo Scientific | 69702 | Optional |
| SCSM single drop-out –ura | Formedium | DSCS101 | |
| Sodium Acetate | Sigma-Aldrich | 32318-M | Make a 3 M solution and pH to 5.3 with acetic acid |
| Sodium hydroxide | Sigma-Aldrich | 795429 | CAUTION corrosive |
| SDS (Sodium dodecyl sulfate) | Sigma-Aldrich | 436143 | CAUTION irritant, do not inhale |
| Streptavidin Magnetic beads | NEB | 1420S | Store at 4°C |
| SUPERase-In, RNase inhibitor | Life technologies | AM2696 | Store at -20°C |
| Thiolated Schizosaccharomyces pombe for spike | See section 1.7 of the protocol | ||
| 4-thiouracil (4tU) | ACROS ORGANICS | 359930010 | Store in the dark. Make 100 mM Stock in 1M NaOH, store solutions at -20°C. |
| Tris base | Sigma-Aldrich | 93362 | 1 M solutions at various pH |
| tRNA | Sigma-Aldrich | 10109541001 | 5mg/ml, store at -20°C |
| Uridine | Sigma-Aldrich | U3750 | Make 1 M solution in H2O. Split into 2 mL aliquots and store at -20 C. |
| Yeast nitrogen base without amino acids with amonium sulphate | Formedium | CYN0410 | |
| Zeba Columns 0.5ml | Thermo Scientific | 89882 | Store at 4 °C |
| Zirconia beads | Thistle Scientific | 110791052 | |
| Equipment and Consumables | |||
| Beadbeater | Biospec | 112011EUR | Other homogenisers can be used; the correct conditions for each homogeniser and strain must be established. |
| Bioanalyser (Agilent) or similar to assess RNA quality. If this is not important a spectrophotometer is useful to quantify the RNA. | |||
| Centrifuge: capable of spinning cultures at 4 °C and at least 3000 g. Pre-chill if possible. | |||
| Centrifuge: capable of spinning up to 2 mL tubes at variable speeds upto 13,000 g and down to 1000 g | |||
| Magnetic rack for separating the beads from the sample. The one used in the paper is 3D printed, available from Thingiverse (thing:3562952). Comercially available racks exist | |||
| PCR machine with a heated lid that will allow incubation in the dark. | |||
| Rotating wheel to rotate 1.5 mL tubes end over end | |||
| Shaking heating block (such as Eppendorf Thermomixer) is recomended | |||
| Tubes, centrifuge, Low retention, RNase free 0.5mL | Eppendorf | H179467N | |
| Tubes, centrifuge, Low retention, RNase free 1.5mL | Ambion | AM12350 | |
| Tubes, centrifuge, 50 mL | Sarstedt | 62.547.004 | Other centrifuge tubes are not gas proof allowing CO2 to disolve in the methanol, this comes out of solution vigorously on adding warm culture, leading to sample loss |
| Tubes, centrifuge, 15 mL | Sarstedt | 62.554.001 | |
| Tubes, 2 mL, screw cap | Greiner | 723361 | |
| Tubes 0.2 mL strip of 8 with integral lids | Brand | 781332 |
Referenzen
- Duffy, E. E., Schofield, J. A., Simon, M. D. Gaining insight into transcriptome-wide RNA population dynamics through the chemistry of 4-thiouridine. Wiley Interdisciplinary Reviews: RNA. 10 (1), e1513(2018).
- Windhager, L., et al. Ultrashort and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Research. 22, 2031-2042 (2012).
- Baptista, T., Devys, D. Saccharomyces cerevisiae Metabolic Labeling with 4-thiouracil and the Quantification of Newly Synthesized mRNA As a Proxy for RNA Polymerase II Activity. Journal of Visualized Experiments. (140), e57982(2018).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282(2015).
- Burger, K., et al. 4-thiouridine inhibits rRNA synthesis and causes a nucleolar stress response. RNA Biology. 10, 1623-1630 (2013).
- Mendoza-Ochoa, G. I., et al. A fast and tuneable auxin-inducible degron for depletion of target proteins in budding yeast. Yeast (Chichester England). 36 (1), 75-81 (2018).
- Barrass, J. D., Mendoza-Ochoa, G. I., Maudlin, I. E., Sani, E., Beggs, J. D. Tuning degradation to achieve specific and efficient protein depletion. Journal of Visualized Experiments. , (2019).
- Hobro, A. J., Smith, N. I. An evaluation of fixation methods: Spatial and compositional cellular changes observed by Raman imaging. Vibrational Spectroscopy. 91, 31-45 (2017).
- Gustilo, E. M., Vendeix, F. A. P., Agris, P. F. tRNA's Modifications Bring Order to Gene Expression. Current Opinion in Microbiology. 11, 134-140 (2008).
- Dolken, L., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14, 1959-1972 (2008).
- Miller, C., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Molecular Systems Biology. 7, 458(2011).
- Schmitt, M. E., Brown, T. A., Trumpower, B. L. A rapid and simple method for preparation of RNA from Saccharomyces cerevisiae. Nucleic Acids Research. 18, 3091-3092 (1990).
- Rädle, B., et al. Metabolic Labeling of Newly Transcribed RNA for High Resolution Gene Expression Profiling of RNA Synthesis, Processing and Decay in Cell Culture. Journal of Visualized Experiments. (78), e50195(2013).
- Herzog, V. A., et al. Thiol-linked alkylation of RNA to assess expression dynamics. Nature Methods. 14, 1198-1204 (2017).
- Ohrt, T., et al. Molecular dissection of step 2 catalysis of yeast pre-mRNA splicing investigated in a purified system. RNA. 19, 902-915 (2013).
- Alexander, R. D., et al. RiboSys, a high-resolution, quantitative approach to measure the in vivo kinetics of pre-mRNA splicing and 3′-end processing in Saccharomyces cerevisiae. RNA. 16, 2570-2580 (2010).
- Wallace, E. W. J., Beggs, J. D. Extremely fast and incredibly close: cotranscriptional splicing in budding yeast. RNA. 23, 601-610 (2017).
- Adelman, K., Lis, J. T. Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nature Reviews Genetics. 13, 720-731 (2012).
- Preker, P., et al. RNA Exosome Depletion Reveals Transcription Upstream of Active Human Promoters. Science. 322, 1851-1854 (2008).
- Aslanzadeh, V., Huang, Y., Sanguinetti, G., Beggs, J. D. Transcription rate strongly affects splicing fidelity and cotranscriptionality in budding yeast. Genome Research. 28, 203-213 (2018).
- Schofield, J. A., Duffy, E. E., Kiefer, L., Sullivan, M. C., Simon, M. D. TimeLapse-seq: adding a temporal dimension to RNA sequencing through nucleoside recoding. Nature Methods. 15, 221-225 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten