Method Article
4-チウラシル(Ers4tU)を使用したビボにおけるRNAの非常に迅速かつ特異的な代謝標識
要約
酵母サッカロマイセスセレビシエから新たに転写されたRNAを敏感かつ特異的に精製するためにチオレートされたウラシルを使用する。
要約
ヌクレオチド類似体である4-thiouracil(4tU)は、細胞によって容易に取り込まれ、生体内で転写される際にRNAに組み込まれ、ラベリングの短い期間中に産生されるRNAの単離を可能にする。これは、組み込まれたチオ群にビオチンモイエティを付着させ、親和性精製を、ストレプトアビジン被覆ビーズを用いて行う。既存のRNAを含まない純粋で新しく合成されたRNAの良好な収率を達成することで、ラベリング時間を短縮し、運動学的研究における時間分解能の増加を可能にします。これは、新たに合成されたRNAの非常に特異的で高収率の精製のためのプロトコルである。ここで提示されるプロトコルは、酵母サッカロマイセスセレビシエからRNAを抽出する方法を説明する。しかし、全RNAからチオ化RNAを精製するためのプロトコルは、細胞から抽出された後、任意の生物からのRNAを使用して有効であるべきである。精製されたRNAは、逆転写酵素-qPCR、RNA-seqおよびSLAM-seqなどの多くの広く使用されている技術による分析に適しています。この技術の特異性、感受性および柔軟性はRNA代謝に比類のない洞察を可能にする。
概要
RNA は動的な性質を持っています。それは多くのRNAが急速に処理され、分解された後すぐに生成されます。現在、RNA代謝のほとんどの研究は、主に完全に処理され、安定した状態レベルで、総細胞RNAを分析します。このレベルは、転写後の成熟と劣化の速度のバランスに依存します。定常状態平衡につながるプロセスの分析には、非常に短命なRNA種を捕捉するための専門的な技術が必要です。
4-チオウラシル(4tU)または4-チウリジン(4sU)などのヌクレオチド類似体を有するRNAの代謝標識(優れたレビューについてはDuffy et al.1を参照)は、チオ標識付きの新生RNAとその処理中間体を単離する能力を提供する。しかし、公開されたプロトコルは、数分2、3のラベリング時間を含み、これは多くのトランスクリプトの生産速度に比べて遅いです。平均酵母遺伝子を転写するのに1分の順序で行われるので、酵母RNAを1分未満の標識は非常に短いと考えることができます。非常に迅速かつ特定の4 thiouracilプロトコル(ers4tU)は、4tUの組み込みを最大化し、ラベルなしの既存のRNAの回収を最小限に抑えることで、ノイズ比に信号を最大化し、非常に短いラベリング時間を可能にします4。
チオ修飾塩基は、新たに合成されたRNA(nsRNA)を効率的に標識するために、迅速かつ十分な量で細胞内に輸入されなければならない。これを促進するために、細胞はウラシルフリー培地で増殖し、適切なパーマーゼの発現は4tUまたは4sUの取り込みを高めるのに役立ちます(適切なパーマーゼ遺伝子および補足図1を運ぶプラスミドのリストについては、表1を参照)。4tUの水酸化ナトリウムの溶解度は、他のヌクレオチド類似体に必要な有毒な有機溶媒の必要性を回避します。残念ながら、50μMを超える濃度でチオ修飾ヌクレオシドを用いて長期間培養を増殖させることが観察され、リボソーム5を破壊することが観察されている。しかしながら、ここで用いる濃度(10μM)は、極めて短い標識時間と、有害な影響を最小限に抑えながら(図1a)、分析に十分なRNAを得ている。
この技術は、標的タンパク質6、7(図2)の迅速かつ特異的なアウクシン媒介破壊と組み合わせることができ、そのβ-エストラジオールがオーキシンの発現を調節する「β-est AID 4U」プロトコルと呼ばれる。誘導性デグロン(AID)システムは4tUラベリングと組み合わされます。β-est AID 4Uアプローチでは、標的タンパク質が枯渇し、RNA代謝に及ぼす影響を注意深くモニタリングすることができる(図2)。タイミングは重要です。付属のビデオを表示し、図 2とそのアニメーション形式に細心の注意を払うことをお勧めします (補足図 2を参照)。
正確な時間分解のためには、RNAの処理と劣化を非常に迅速に停止する必要があります。これは、低温でメタノールを使用して達成され、これは非常に迅速に細胞内容を固定し、核酸含有量8を維持しながら細胞膜を分解する。RNA抽出は効率的で、RNA を損傷しないようにする必要があります。機械的なライシスは、カオトロピック剤の不在で有効である(多くの場合、これらはチオ基を含むので、避けるべきである)。RNAの塩化リチウム沈殿が好ましく、tRNAの方が効率よく沈殿しがちである。tRNAは急速に転写され、自然にチオレートされる9なので、tRNAを除去すると、バイオチリン化試薬の競合が減少します。小さく、高度に構造化されたRNAが関心がある場合は、アルコールベースのRNA沈殿方法が推奨されます。
チオ化RNAを回収するために、ビオチンは4tUを有するRNAに組み込まれたチオ基を介して共生的に付着する。硬化性ジスルフィド結合を介して付着する修飾ビオチンの使用(例えば、HPDP-ビオチン(例えば、HPDP-biotin)-3/3(2-pイリジルdイチオ)pロピオナミド、またはMTS-ビオチン(メタンチオスルフォネート)が推奨される還元剤を添加してRNAの放出を可能にする。生体化RNAは、磁気ビーズに結合したストレプトアビジン上で精製された親和性である。このプロトコルは、前述の10に記載されている他のプロトコルに似ていますが、バックグラウンドを減らすために集中的に最適化されています。
チオールラベリング実験には、連続ラベリングと不連続ラベリングの2種類があります。それぞれに独自の利点があります。連続ラベリングでは、4tUが一定の間隔で採取された培養物およびサンプルに添加される。このタイプの実験は、RNAの処理方法と、時間の経過とともにレベルがどのように変化するかを示しています。例としては、変異体と野生型実験とパルスチェイス実験の比較が挙まれる。図3b,cに示す実験は、このタイプのである。不連続ラベリングの場合、変更はシステムに誘導され、RNA はモニターされます。変更が誘導されると、カルチャはいくつかのサブカルチャに分割され、特定の時間に、各カルチャは短時間でティオラベル付けされます。1 つの例は、図 27に示す β-est AID 4U です。この種の実験は、RNA処理に対する代謝変化の影響をモニタリングするのに特に有用である(図3d参照)。
チオラベリング実験のグラフィカルな表現を図4と図5に示し、プロトコルのパフォーマンスを大幅に簡素化するスプレッドシートを用意しています(4tU実験テンプレート.xlsxを参照)。補足情報には、広範なトラブルシューティング ガイドが含まれています。4tU ラベリングと auxin 枯渇プロトコルを統合する β-est AID 4U プロトコルについては、図 2および補足図 2を参照してください。詳細なAID枯渇プロトコルについては、Barrassら7を参照してください。
プロトコル
1. 成長とチオラベリング
注: プロトコルのこのセクションの完了に必要な時間は、細胞の増殖速度に応じて非常に変動します。チオラベリングの前にソリューションと機器を1時間、サンプルを処理するために30分のポストラベリングを行います。
- S.セレビシエ株にパーマゼ(表1)をコードするプラスミドが含まれていることを確認し、セルへの4tUのインポートを高める。
注: 輸入者がいなければ、2 分未満のラベリングが成功する可能性は低くなります (補足図 1を参照)。4tUの組み込みは、成長がウラシルなしで媒体である場合より効率的ですので、株はURA3+でなければなりません。表1のプラスミドのいくつかはURA3をマーカーとして運ぶ。このプロトコルをβ-est AID枯渇7と組み合わせるには、追加の歪み修飾が必要である。 - アミノ酸を含まない酵母窒素塩基6.9g、ブドウ糖20g、SCSM単一ドロップアウトウラ(材料表)1Lを1Lの水に加えてYMMウラシルフリー培地を調製します。オートクレーブまたはフィルタは、使用前に成長培地を殺菌します。
注:フィルター殺菌は、サンプル採取に使用されるメタノール中の細胞と共沈殿するオートクレーブによって生成されるペプチド/糖複合体として好ましい。 - 酵母をYMMウラシルフリー培地で0.6~0.8の600nm(OD600)の光学密度に成長させる。カルチャがログ フェーズの増加にあり、少なくとも 2 倍になっていることを確認します。通常、30°Cでの成長が推奨されますが、他の温度は、例えば、温度感受性株のために使用することができます。
注:株、成長条件およびRNA収率に応じて、約30 mLサンプル容積が必要になります。この量はプロトコル全体で想定されます。培養の30 mLは、メタノールの20 mLで50 mL遠心チューブに収まる最も多いので、最適化を開始するのに便利なボリュームです。RNA回収を増加させるために早期の時点でより多くのサンプル体積を使用することを検討し、最大2000 mLは、本当に短い標識時間(<1分)で遅い成長細胞に使用されています。 - H2Oの約50mLを氷の上で冷やします。各サンプルについて、2mLスクリューキャップチューブに200μLのジルコニアビーズを加え、氷の上で冷やします。また、20 mLのメタノール(注意)を50mL遠心管に入れ、ドライアイス(注意)の上に置きます。メタノールは、試料の体積を1/3~2/3にする必要があります。
注意:メタノールは吸入、接触および消費によって有毒である。ヒュームフードに大量のボリュームを分配し、メタノールがニトリル実験室の手袋を貫通することができるので、手袋の2組を着用してください。メタノールは非常に可燃性であり、点火のすべてのソースから遠ざけます。
注:ドライアイスは接触時に冷たい火傷を引き起こし、非酸化ガスを発生させる可能性がありますので、換気の良い空間で取り扱い、使用する場合は手袋を使用してください。
注:この時点でビーズを追加すると、チューブが乾燥し、セルペレットを回転させるときに、ビーズがチューブスレッドからスピンアウトされるよりも、サンプルを追加した後よりも簡単です。さらに、これはサンプルが加えられる前にビーズを冷却することを可能にする。 - S.ポンベスパイクを(後でではなく)培養に加える場合は、氷と渦上のチオレートS.ポンベ細胞のアリコートを完全に解凍し、少なくとも30s、培養物に加えます。以下の指示に従って調製した場合、1つのS.ポンベアリコートは400 mLの培養物に対して十分です(12個の30mLサンプルに加えて、取り扱いのエラーを許容するのに十分です)。多かれ少なかれ培養を使用する場合は、カルチャに追加されたS.ポンベの体積を調整します。
- S.cerevisiaeのプロトコルに記載されているように正確にOD600に0.8にS.ポンベ培養の1 Lを成長させる。
- ティオラベルはステップ1.7として、しかし10分間。
- ドライアイス上の400 mLのメタノールを使用して、すべての培養物を修正します( 基本的に、ステップ1.9に記載されているように)。
- 3000 x gで遠心分離によって細胞を3分間ペレットする。
- 上清を廃棄し、H2Oの3.3mLでセルペレットを再懸濁する。
- それぞれ80μLのアリコートに分割します。-80 °C.で保存します。
- 400 mL培養用のアリコート 1 つまたは 30 mL サンプルあたり 10 μL を使用します。
注: RNAseqを実行する場合は、スパイクの音量を 1/10に減らしてください。アリコートを再利用しないでください。未使用のスパイクを破棄します。このスパイクは、時間ポイントと実験の間で結果を正規化して比較するのに役立ちます。
- 不連続標識の場合、必要な代謝摂動(例えば、成長条件、β-est AID 7(図2および補足図2)などの遺伝子誘導または枯渇)を誘導し、次いで培養物を分割する。すべてのフラスコとメディアが必要な温度であることを確認し、可能であれば培養を加える前に培地を通気します。
- 10 μmMの濃度に4tUを加え、激しく混合する(100mM 4tUの培養量の1/10,000を1M NaOHに溶解)。チオールラベルを15~5分
注:30秒は良い出発点です。20s未満のチオラベリングは、時間の圧力下で培養を操作するのが難しいため、より可変的な結果を与えます。ただし、1 分を超えるラベリングは、手法の時間的分解能を低下させます。 - チェイス実験を行う場合。20-30 sのチオラベリングを許可し、その後、1Mウリジン(チオレートされていない)の1/200培養量を5mMの最終濃度に加えることによって追跡する。
注:ウリジンは、より多くの水溶性が培養に加えられるので、細胞の増殖に少ない乱れがあるので、ウリジンは、追跡のためのウラシルよりも好ましいです。 - 時間コースの最後まで、一定の間隔(少なくとも15秒)で培養のサンプルを採取してください。これより短いサンプリング間隔を確実に実行することは困難です。ステップ1.4で調製したドライアイス上のメタノールにサンプルを加えます。便宜上、20 mLのメタノールを含む50mLチューブに30mLの培養を加えます。
注:二酸化炭素は寒いときにメタノールに溶解します。これは、サンプルを添加すると溶液から出て、混合時に激しく泡を出し、サンプル損失をもたらす。これを避けるために、必要な時間に近づくまで密閉されたチューブでメタノールを<-70 °Cに冷やし、ドライアイスに移します。 - チューブを密閉し、振って十分に混ぜます。サンプルを氷の上に置く。どのサンプルも凍結されていないことを確認します。もしそうなら、手で穏やかに暖かく、絶えず反転する。これは、サンプルの温度を評価することができ、それは常に寒さを感じる必要がありますので、手で行うのが最善です。氷の上に置けこれは一時停止ポイントではありません。すべてのサンプルが流体になったら、次のステップに進みます。
- 3000 x gで2分間(可能な場合は4°C)で回転させ、細胞をペレットします。液体を注ぎ、ペレットを少なくとも1mLの氷冷水でゆっくりと上下にピペッティングします。
注:サンプルペレット中の残留メタノールは、再懸濁液を補助する。 - ステップ1.4で準備された2 mLねじキャップチューブに移します。13,000 x gで短時間(例えば、合計10回)スピンして細胞を再ペレットし、氷の上に戻し、液体を取り除きます。
注:細胞ペレットは数ヶ月間-70から-80 °Cで貯えることができる。
2. 総RNAの調製
注:完成時間は90分です。
- ジエチルパイロカーボネート(DEPC)処理溶液を使用して、RNAを分解から保護します。フィルターピペットの先端を使用してソリューションをアリコートし、常に手袋を着用してください。
- 溶液にDEPCの1/1000ボリュームを追加し、激しい振盪によって混合する。
- 室温(RT)で24時間放置し、オートクレーブを使用します。
- アミン基(トリスなど)を持つ溶液は、DEPC処理できません。アリコート粉末とRNA作業のために特別に保存します。溶液を作るために、以前にDEPC処理されたH2 Oを使用してください。
注:RNA上のチオグループは光電化可能なため、この時点から紫外線への曝露を最小限に抑えます。ストレージは暗闇の中にあるべきであり、インキュベーションはふたが付いているPCR機械で行われるのが最善である。
- S.ポンベスパイクを培養ではなくセルペレットに追加する場合(両方を行わない)、今すぐ追加します。ペレットに添加する前に、氷と渦上のチオレートS.ポンベ細胞のアリコートを完全に30sで解凍する。
注:ステップ1.5.1−1.5.7に従って調製した場合、30mLの培養物に由来する1つのペレットに対して10μLのS.ポンベアリコートが必要です。 - キャップを置く前に、1−2sのために非常に短時間スピンして、ジルコニアビーズがキャップとチューブの間に閉じ込められており、サンプルとフェノールがチューブから漏れる可能性があることを確認します。
- アセテートEDTA(AE)バッファーの400μL(50mM酢酸ナトリウムpH 5.3、10mM EDTA pH 8.0)で細胞を再ステージングし、激しく渦を酸化する。10%(w/v)のドデシル硫酸ナトリウム(SDS)の40 μLを加えます。SDSが発泡するように、渦をしないでください。
- β-est AID 4U プロトコルを使用する場合は、タンパク質分析7の細胞懸濁液の 40 μL を取ります。AEを40μL加算して、体積を400μLまで戻します。
- 低pHでフェノール(注意)の800 μLを追加し、10sの渦。
注意:フェノールは、吸入と接触によって有毒かつ腐食性です。常にヒュームフードにフェノールを含む手順を実行し、手袋の2ペアを着用してください。 - 最も低い電力設定で3つの2分間のバーストのために、ホモジナイザー(例えば、材料の表)で細胞をlyseします。均質化のパルスの間に2分間氷の上にサンプルを残します。
注: 他のホモジナイザーを使用する場合は、条件を最適化します。不十分な揺れは収率が悪くなるのに対し、過度の揺れは260nm(A260)での吸光度によって決定される明らかに高い収率をもたらすが、RNAは分解され得る。ホモジナイザーが好ましいが、ホットフェノールRNA精製12を用いることができる。 - ライズしたサンプルをドライアイスに5分間置き、固化するまで、RNAに持ち越されるゲノムDNAを減少させます。サンプルが解凍されない限り、あまりにも長く凍結しないでください。RTで13,000 x gでマイクロフュージでスピン5分;サンプル/フェノールミックスは、低温で行われた場合、スピン全体を通して固体のままになりますので、4 °Cでこれを行うように誘惑しないでください。
注:サンプルがスピンの終わりに凍結している場合は、サンプルが完全に解凍されるまで、さらに5分間再スピンします。 - フェノール/クロロホルム抽出物は、フェノールの等しい体積(約600μL)を持つクロロホルム抽出物:クロロホルム5:1、クロロホルム(注意)。フェノール:クロロホルム5:1またはクロロホルムを含む別のチューブに上相を移します。渦は、RTでマイクロフュージで5分間回転します。その後、新しい1.5 mLチューブにトップフェーズを転送します。
注意:クロロホルムは吸入と接触によって有毒です。常にヒュームフードにクロロホルムを含む手順を実行し、手袋の2ペアを着用してください。 - 10M LiClの3分の1から半分の体積(約300μL)を加え、混合してRNAを沈殿させる。サンプルはすぐに曇りに行く必要がありますが、氷の上または4 °C(凍結するように-20 °C以下に保存しないでください)、または沈殿物が凝集するまで、少なくとも10分間放置する必要があります。
- マイクロフュージで13,000 x gで5分間スピンします。流体を取り外し、短時間再スピンし、ドレッグを取り外します。70%エタノールの300-500 μLでペレットを洗浄し、短時間スピンし、残りのエタノールを除去します。
注:これらの洗い流しの間、最初のスピンと同じ側にペレットを保つ、このようにペレットが移動し、壊れはありません。それが壊れた場合、RNAの一部が誤って失われる可能性があります。
注:ペレットを乾燥させないようにしてください。流体のほとんどが取り除かれる限り、後続のステップに干渉しません。RNAはまた、この段階で-20 °Cで数ヶ月間、または-70〜-80°Cで長期保存することができます。 - RNAペレットを90μLのTE pH 7.0(10mMトリスHCl pH 7.0、1mM EDTA pH 8.0)で再溶解し、RNAペレットとして振盪を起こすと再溶解が困難な場合があります。これは、より高い温度でRNAが劣化するにつれて5分以下でなければなりません。完全なRNA可溶化を確認し、0.2 mLチューブに移します。サンプルを上下にピペット。「しこり」が存在するべきではない、と流体は先端にスムーズに上昇し、落ちる必要があります。このソリューションは粘性なので、最終的なピペッティングモーションは遅くなります。
注:RNAは、この段階で暗闇の中で-20 °Cで保存することができます。これはまた、RNA溶解性に有益であることができる。
3. バイオチニル化
注:完成時間は60分です。次の手順は、別々のキャップを持つストリップよりも渦で開く傾向が低いので、一体型キャップ付きチューブのストリップで便利に行われます。
- 5mM HPDP-ビオチン溶液(MTS-biotin)の10 μL(1/10最終容積)をRNAに添加し、完全に混合してバイオチリン化する。ビオチンを加える前に、65°Cで数秒以内にRNAを予熱します。暗闇の中で最大30分間15分間65°Cでインキュベートします。
注:この加熱は、一部のHPDPバッチがRNAサンプル内のRTで沈殿するため必要です。加熱されたふたが付いているPCRブロックはこれに理想的である。 - 小さな樹脂量、サイズの除外カラム(材料の表)を準備し、未組み込みのビオチンを除外します。カラムの下部タグを取り外し、キャップを緩め、2 mL遠心管に入れます。1500 x gで 1 分間スピンして、バッファーをフラッシュする TE の 0.3 mL を列の上部にそっと追加し、再度スピンします。洗浄を繰り返し、合計3回の洗浄のためにもう2回回転させます。最後に、洗浄されたカラムを新鮮な1.5 mLチューブに移します。
- サンプルインキュベーション(ステップ3.1)が完了したら、サンプルを列の上部に追加します。1500 x gで2 分間スピンします。生体化したRNAサンプルは、チューブの底部に入っています。
注:1分のスピンでは、サンプル全体を溶出させるには十分ではありません。 - 10M LiClの3分の1から半分の体積(約40μL)を加え、混合してステップ2.10としてRNAを再沈殿させる。サンプルはすぐに曇りに行く必要がありますが、氷の上または4 °Cで、または沈殿物が凝集するまで少なくとも5分間放置する必要があります。凍結しますので-20°C以下に保管しないでください。マイクロフュージで13,000 x gで5分間サンプルを遠心分離します。
- 80%エタノール、≤1時間回転で洗浄します。ステップ 2.11 の手順に従って、できるだけ多くの流体を取り除きます。
注:HPDP-ビオチンは80%エタノールに非常に可溶性なので、これは追加の精製工程です。 - 80%エタノール洗浄を繰り返し、できるだけ多くの未組み込みビオチンを除去します。
注:RNAはまた、暗闇の中で-20 °Cでこの段階で保存することができます。
4. 新たに合成されたRNAの精製
注:完了の時間は2時間です。
- DEPC処理H2Oの200μLでRNAを再溶解する(65°Cインキュベーションは、ステップ2.12の手順と同様に使用することができる)。
- 分光光度計を用いてA260のRNA濃度を測定する。試料は、分光光度計の線形範囲内に収めるために1/10を希釈する必要があります。粘性RNAが均等に溶解することを確実にするために、少なくとも10sのためにこの希釈を渦。
注:バイオチニル化の効率は、必要に応じてドットブロット13によって評価することができる。 - 新鮮なチューブに同量のRNAを追加し、DEPC処理されたH2 Oで最大200 μLを作ります。
注: スプレッドシート4tU 実験テンプレート.xlsxには、この計算を支援するフォームがあります。 - サンプルが RT の場合は、10 x NaTM バッファーの 25 μL を追加します (0.1 M トリス HCl pH 7.0、2 M NaCl、250 mM MgCl 2)、25 μL の 1 M NaPi pH 6.8 (0.5 M NaH2PO4 0.5 M Na2 HPO4)10% SDS の 2.5 μL。よく混ぜて軽く回転させます(<30 s;約100 x g)。
注:SDSおよび塩の沈殿を避けるために、サンプルはステップ4.13まで次の手順を通してRTで保たれなければならない。 - 1x NaTM バッファ、0.1 M NaPi、および 0.1% SDS、サンプルあたり 2 mL を含むビーズ バッファを作成します。最初に H2O の必要量を追加し、最後に SDS を追加します。これは、24時間後に沈殿物の形態として毎回新鮮にする必要があります。
注: 沈殿物の形成を避けるために、ビードバッファは以下の手順を通じてRTに保持する必要があります。DEPC は、治療やオートクレーブを使用しないでください。 - 低保持1.5 mLチューブに50 μLのストレプトアビジンビーズを追加します。チューブを磁気ラックに置き、ビーズが落ち着くのを待ってから流体を取り外します。
- ストレプトアビジンビーズを洗います。
- ビーズペレットが完全に再懸濁されるまで、ビーズバッファと渦の200 μLを追加します。通常は 3−5 s が必要です。RNAサンプルを添加する前に洗い物をする場合は、ビーズがチューブを横切って反対側に移動するようにチューブを回すので十分です。次に、チューブを元の側に戻して、ビーズが再びチューブを横切るようにします。
- チューブを低速(約100 x g)で回転し、最大5sで流体をスピンダウンしますが、ビーズは回転しません。
- 磁気ラックに入れ、ビーズを磁石で捕捉できるようにします。
- 少数のサンプルの吸引によって液体を取り除く;多くのサンプルの場合は液体を注ぎます。
注:多数のサンプルでは、最初のサンプルのビーズが最後のサンプルが終了する前に乾燥する可能性があるため、純粋に吸引によってすべての流体を除去することは問題になる可能性があります。洗浄は磁石の上で一度にすべてのサンプルから液体を注ぐことによって促進することができる。彼らは注ぐ前に磁石にもう少し長く残す必要があり、残っている流体の少量は離れて吸引する必要がありますが、全体的に、それは磁石上と流体なしでより少ない時間を意味します。このようにして、24以上の抽出を迅速に行うことができます。
- 200 μLビーズバッファー、10 μL 20 mg/mL グリコーゲン、および 2.5 μL 5 mg/mL tRNA を備えたブロック、RT で適度な速度でエンドを 20 分回転します。回転は、ビーズをサスペンションに保つことです。ブロッキングが完了したら、ステップ4.7.2−4.7.4として流体を取り外し、セクション4.7のステップとして再び洗浄します。
- サンプル内のビーズを再中断します。30分間回転してRTでインキュベートします。
- インキュベーション中に、各サンプルに新鮮な1.5 mLチューブを準備します。3MアセテートpH 5.3および20 μgのアセテートナトリウムの1/10体積(約10μL)を加え、必要になるまでラック内に約100xgで3sの貯蔵で回転させる。
- ステップ 4.7.2−4.7.4に従って、バインドされていない RNA をビーズから取り外します。非結合RNAは新鮮なチューブで耐えることができますが、塩とSDSは精製を非常に困難にします。その後、断面4.7として、最低3~最大5回、ビーズを洗います。
- 最終的な洗浄の後、すべての液体を吸引するために特別な注意を払います。各チューブに戻り、バッファのドレッグをもう一度吸引します。
- RNAを溶出させるには、ビーズに50μLの新たに調製した0.7M β-メルカプトエタノール(βME)をビーズ(市販のストック溶液の1/20希釈)を加える。渦とスピンは、ステップ 4.7.1 および 4.7.2 として短時間です。スラリーを磁気ラックに入れ、ステップ4.10で調製した1.5 mL遠心管にRNA含有溶液をピペットします。
- ステップ4.13としてもう一度Eluteを加えてビーズから残留RNAを回収し、これらのビーズからの最初の溶出を含むチューブに溶出したサンプルを添加する。
- サンプルを磁気ラックに戻し、流体を新鮮で低結合の0.5 mL遠心分離管に移すことによって、ELUTED RNAから残留ビーズを取り除きます。
- サンプルを混合し、エタノールの2.5xボリューム(280 μL)を追加してnsRNAを沈殿させ、もう一度混ぜます。-20 °Cで一晩1時間放置します。最大速度(少なくとも13,000 x g)で20分間、あらかじめ冷やされた遠心分離機(4°C)でスピンします。
- -20°Cで70%エタノールの200 μLで十分に洗浄します。残留βMEは下流のアプリケーションを阻害するように、可能な限り多くのドレッグを除去するために、すべてのステップでスピンします。最後に、サンプルはβMEのにおいをかぐべきではありません。
- 0.005 μL RNase阻害剤に相当するDEPC処理1x TEの10-20 μLで再溶解する。
注:後続のすべてのステージは氷上で実行する必要があります。 - RNA濃度と純度を測定します。
- A260と A225を低サンプル体積分光光度計で測定します。
注:λ = 225 nm付近の吸光度最大値は、ビーズからの避けられない汚染物質から得られます。RNAがない場合、汚染物質からのシグナルはλ =260 nmで35%まで低下します。したがって、実際のRNA量は式(A 260-(A225*0.35))*40 ng/μLで近似されます。 - あるいは、バイオアナライザなどの微小流体電気泳動システム上でサンプルを分析する。
注:この分析は、RNAの完全性を評価することができるように分光光度計を使用する方が好ましく、汚染物質は定量を妨げなく、より少ないサンプルが必要とされる。
- A260と A225を低サンプル体積分光光度計で測定します。
- nsRNA を分析します。
注:例えば、特定のRNAは、標準的な逆転写酵素qPCR技術によって定量することができる。このように調製されたRNAは、RNA-seqに対するライブラリー調製と互換性があり、rRNAの除去は5分未満のラベリング時間には必要ありません。
結果
このers4tUプロトコルを用いて回収されたnsRNAの典型的な収率は図1bに表示され、これはバイオアナリサータによって生成され、トレースはRNA対サイズ(ヌクレオチド[nt])の収率を示す。なお、バイオアナライザトレースとインセットグラフの両方において、タイムポイント0からのRNA回収は、より長い時点から回収されたもののごく小さな部分であり、約10 9μgのRNAが約109μgと比較して2倍以上に増加した。同じ数の細胞からのラベリングのわずか30s(nsRNAの0.8 μg)の後。15sでのRNA回収は、サンプリングを行う際の小さな違いがRNA回収に比例して大きな影響を及ぼすため、より可変的である。バイオアナリサータトレースでは、rRNA前駆体は1000nt付近のピークと1700−1800 ntのピークの二重として見ることができます。これらの中間体の豊富さは、チオレーションが続くにつれて増加します。
チオラベリングは、ACT1トランスクリプトのスプライシングを定量するために使用されました(図3)。チオレーションを行い、チオ標識の開始から15秒間隔で採取した試料及びACT1 RNAの処理を監視した(図3a,b)。ごく一見、プレmRNAは(転写によって)生成され、ラリアット(プレmRNAからのスプライシングの最初のステップによって)、わずか15sのラベリングの後でさえも。約45~1分後、ラリアットとプリmRNAの量は平衡に達し、これらのRNA種の多くはスプライシングによって処理されるのと同じくらい転写によって作成される。
図3cに示すデータを生成するために、株を25sの4tUでパルスし、次いでウリジンで追いかけた。プレmRNAおよびラリアットの生成は1分で最大に達する。これは図 3bとよく比較されます。45s後に達成される最大値は、平衡に加えてラベリングの25sに達する。ピーク後、チオラベル付きRNAがスプライシングプロセスを通じて追いかけるにつれて、レベルは低下します。
図3dは、β-est AID 4U系6、7を用いて、タンパク質スプライシング因子の枯渇およびRNA代謝への影響を示す。ここで、Prp16pは、枯渇の25分後にこのレベルの近い生理学的レベルから5%に減少する。Prp16p は、スプライシング15の 2 番目のステップに不可欠なスプライシング係数です。ラリアットはスプライシングの2番目のステップ(図3a)の間に除去されるが、ここではPrp16が制限されるにつれて、前mRNAのレベルを上回る。その後の枯渇時には、他の要因が二次的な影響によって制限され、ラリアットのレベルが低下し、mRNA前のレベルが上昇する。スプライスされたmRNAのレベルは低下する。
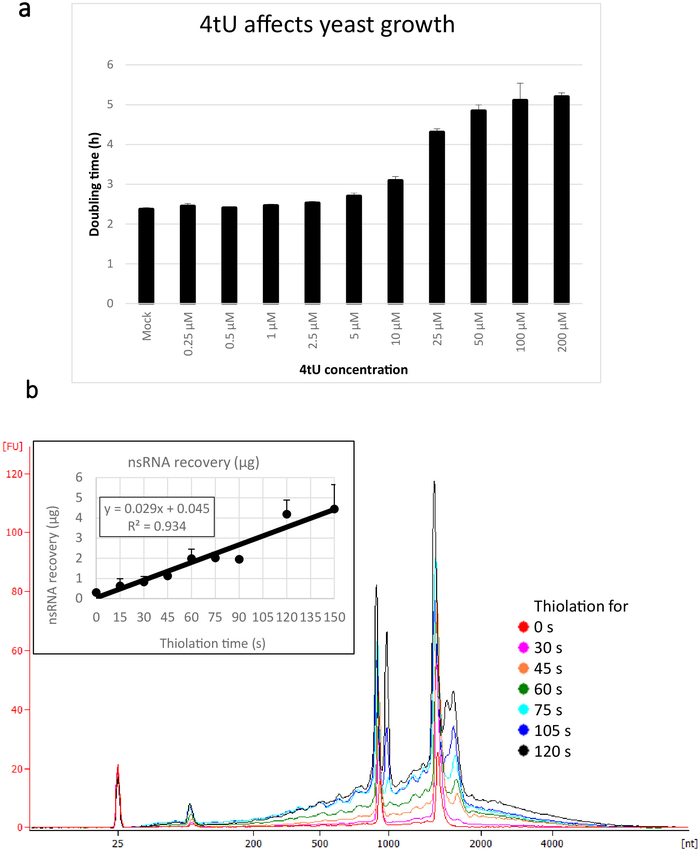
図1:4tUおよびRNA回収における成長(a) 4-thiouracilは成長に影響を与える。ウラシルを含まないYMMドロップアウト成長培地中の4tU濃度を増加させると、p4FuiΔプラスミドを運ぶS.セレビシエ(BY4741)の倍増時間が増加する。4つの複製培養の成長をテカン無限プロ200で30°Cでモニタリングした。すべてのカルチャはログ フェーズ全体にあり、OD600は 0.1 ~ 0.6 でした。モックは、同等の量のNaOHを追加したコントロールカルチャであり、それ自体は成長率を変更しません。このグラフは、チオラベリングが急速な標識と細胞への損傷との間で妥協していることを示しています。誤差余数は、4 反復の標準誤差です。(b) nsRNA収率は約15sのラベリングから直線的に増加する。主な図は、精製されたバイオアナライザの痕跡を示し、nsRNAを0(チオレートされていない)から15秒間隔で4tUを添加した後2分までを示す。15sサンプルはラベル付けされていないサンプルと区別がつかないため、表示されないことに注意してください。2つの大きなピークはリボソームRNA(rRNA)に対応しています。rRNA前駆体および中間体は、成熟したrRNAよりも大きな分子量で複数のピークとして見える。これらの前駆体および中間体の回復は時間とともに増加する。1つの代表的な実験の結果が示されている。インセットグラフは、4tUでインキュベーションを増加させるnsRNAの回復を示す。nsRNAの収率は、4tUで成長の時間が増加するにつれて増加する。この実験のタイムスケール全体を通して回復は著しく直線的(R2 = 0.934)であり、バイオアナライザからの目でラベル付けされていないサンプルと区別できないにもかかわらず、4tUで15sラベリングでもバックグラウンド上のわずかな増加を示しています。トレース。誤差バーは、3つの生物学的反復の標準誤差を示します。この図のより大きなバージョンを表示するには、ここをクリックしてください。
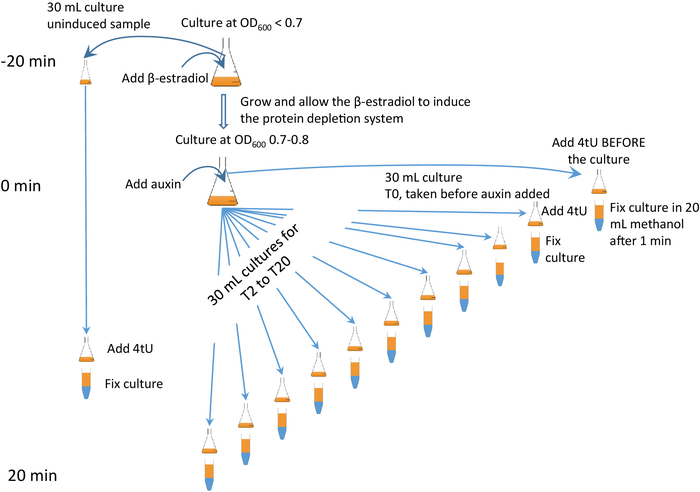
図2:βエストAID 4Uβ-est AID 4Uグラフィカルプロトコル。β-est AID 4U プロトコルのプロトコルのグラフィカルな要約。β-エストラジオール(β-est)は、AID*タグ付き標的タンパク質を枯渇させる補助誘導性デグロン(AID)系の発現を促進し、詳細なプロトコルについてはBarrass et al.7を参照してください。この場合、デグロン系発現はタンパク質分解が始まる25分前に開始され、各時点でのチオレーションは1分間です。補足図 2にアニメーション版が表示されます。この図のより大きなバージョンを表示するには、ここをクリックしてください。
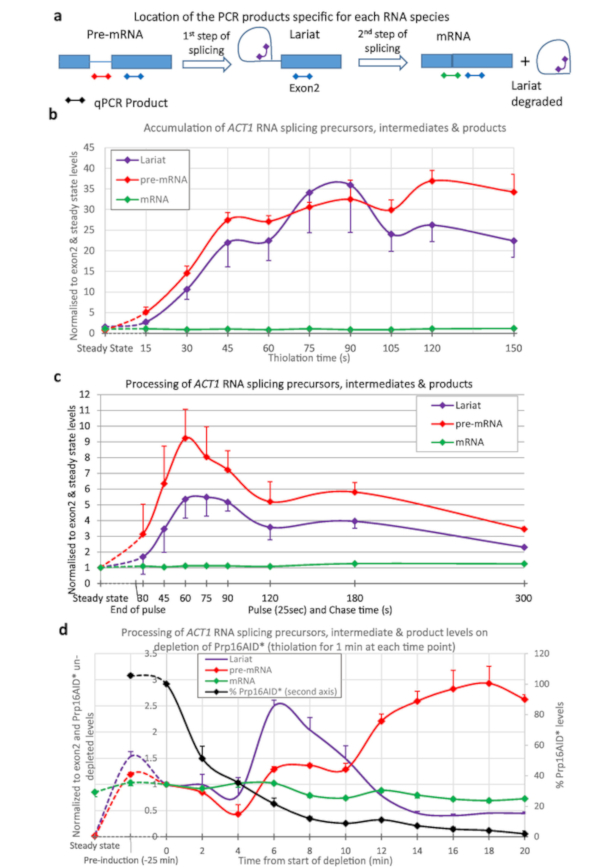
図3:ACT1 RNAスプライシングの前駆体および中間体。 ACT1前mRNA転写物のスプライシングは、定量的逆転転写PCR16によってモニターした。ACT1前駆体(前mRNA)、中間ラリアットエキソン2(ラリアット)およびスプライス産物(mRNA)のレベルは、ACT1 Exon2のレベルおよびこれらのRNAの定常状態レベルに対して正規化されたことが示されている。(a) ACT1トランスクリプト上のqPCR製品の場所。ACT1転写産物16のスプライシング反応の前駆体、中間体および産物のレベルをアッセイするために使用されるqPCR産物の位置の概略図。エクソンは箱で表され、ラインとしてイントロン、qPCR製品は両端にダイヤモンドを持つラインとして表され、色はグラフで使用されるものと一致します。プレmRNA PCRは、プリmRNAに特異的であり、この製品がスプライシングの最初のステップの後に中断される分岐点を横切るため、スプライシングの任意の中間体ではありません。ラリアットPCRは、スプライシングの第1段階の産物と、第2の後に産生される切除ラリアットを検出する。mRNA PCRは、スプライシング、mRNAの積に特異的である。エクソンPCR(取除きラリアットを除くすべての前駆体、中間体および製品に存在する)からの結果は、データを正規化するために使用されたため、常に1に等しいため、グラフには表示されません。(b) 連続チオラベリング。4tUが転写によって組み込まれると時間とともにmRNAの量が増加し、短い遅延の後、スプライシングはそれをlariat-exon2中間およびスプライスされた製品に変換する。これらの前mRNAおよびラリアット種のレベルは、4tUで成長のわずか15sの後にバックグラウンドの上に検出され、4tUとの連続的な標識の約45s後に最大に達し、その時点でその生産はスプライスへの変換によってバランスが取れていますmRNAおよび/または分解。値は定常状態(グラフの左端)に正規化され、exon 2レベルはエキソン2の転写と比較してその外観と処理を示す。ACT1のRNAスプライシングは、主に共写性4、17スプライスmRNAが急速に最も豊富な種となり、そのレベルはエキソン2のものと同様である。3つの生物学的反復の標準誤差は、それぞれが三量体でアッセイされる。(c) パルス/チェイス。25秒のチオレーションパルスに続き、ウリジンで追いかける。これらのRNAの定常状態レベル(左端点)と比較して、新しく合成されたプールでは、最初は非常に豊富です。それらはmRNA(または分解)に処理されるにつれて徐々に低下し、5分の定常状態レベルに非常に類似したレベルに近づき、3つの生物学的反復の標準誤差、それぞれが三極化でアッセイされる。(d) nsRNAおよびタンパク質枯渇。図2に記載されているオーキシンデグロン系を用いたPrp16タンパク質の枯渇時のパネル(a)のように定量的逆転転写PCRによってモニターされたACT1プレmRNA転写物のスプライシング。 Prp16タンパク質レベルは、オーキシン枯渇前のレベルのパーセンテージとして第2のY軸に対してプロットされたグラフにも表示されます。Prp16は、パネル(a)に示すスプライシングの第2段階において特に重要なスプリセオソームの重要な構成要素であり、その後ラリアットが分解される。このステップが制限になると、ラリアットは最初に蓄積します。後でスプライシングが完全に失敗し、ラリアットはもはや産生されなくなり、mRNA前のレベルは上昇する。誤差バーは、3つの生物学的反復の標準的な誤差であり、それぞれが三量体でアッセイされる。この図のより大きなバージョンを表示するには、ここをクリックしてください。
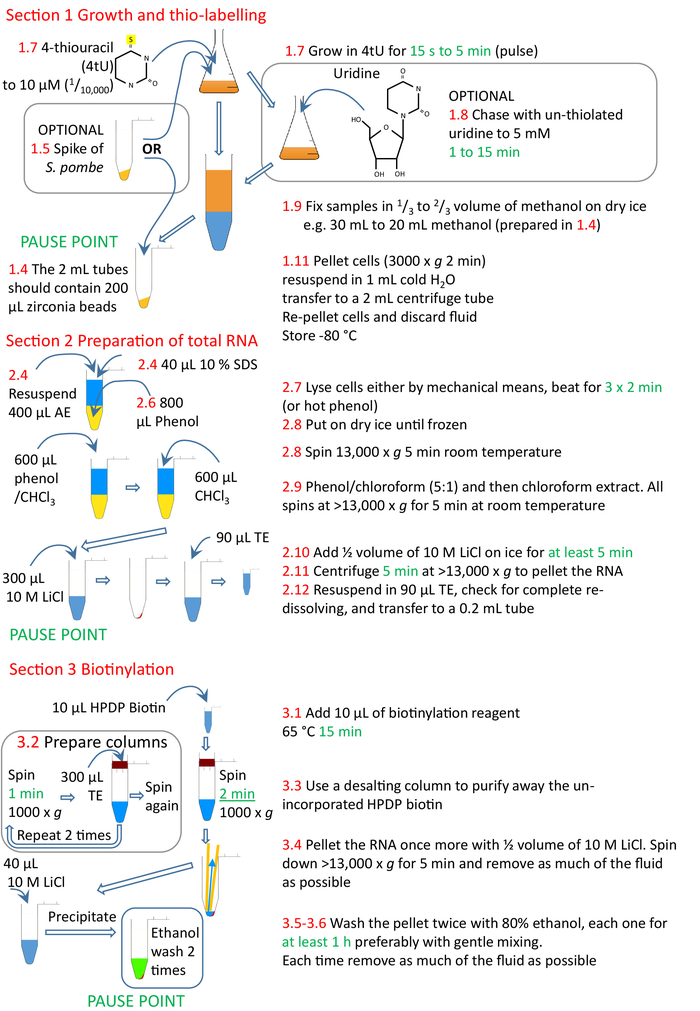
図 4: プロトコル セクション 1 から 3 のグラフィカルな概要。細胞を4tUでチオレートし、改変ヌクレオチドをRNAに組み込むために増殖させた。チオレートされたS.ポンベスパイクを追加して、時間ポイントと実験を通じて正規化することができます。4tUのパルスは、非チオレートウリジンを使用して追うことができる。ラベリングは、4tU添加から継続的に行うことができるか、または成長条件の変化から、培養分割および4tUは成長条件の変化から増加する時間に培養物に加えられるが、各ラベリングは短時間のみである。細胞を採取し、細胞から調製したRNAを、ホモジナイザーおよびフェノール系の方法を用いることに好ましい。RNAはビオチン化され、次いで、サイズ排除カラムを用いて非組み込みビオチンから精製されたビオチン化RNAを精製する。nsRNAは、ストレプトアビジンビーズで精製する準備ができました(セクション4、図5)。赤色の数字は、プロトコルのステップ番号に対応します。この図のより大きなバージョンを表示するには、ここをクリックしてください。
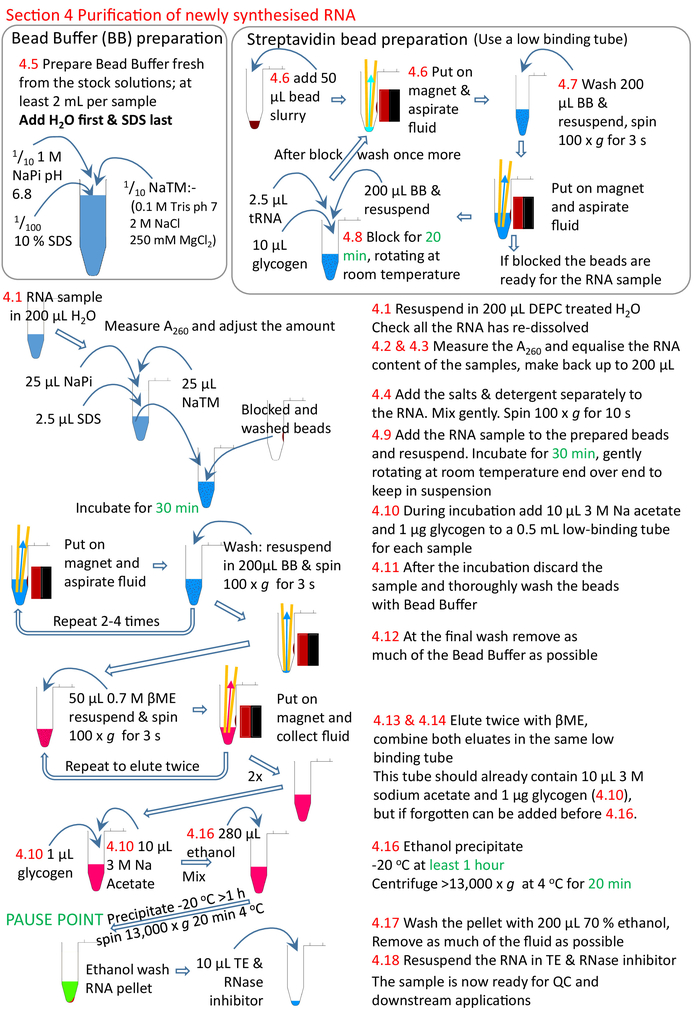
図 5: プロトコル セクション 4 のグラフィカルな概要。セクション1から3(図4)に続いて、ストレプトアビジンビーズがブロックされ、ビオチン化されたRNAサンプルが調製されたビーズに添加される。生体化RNAは、ストレプトアビジンビーズと非ビオチン化RNAを除去して洗浄して結合する。ビオチン化RNAはβMEを用いてビーズから照出され、さらなる研究の準備が整った。赤色の数字は、プロトコルのステップ番号に対応します。この図のより大きなバージョンを表示するには、ここをクリックしてください。
補足図1:チオ標識の1分および3分間の輸入者の追加コピーの有無にかかわらず酵母細胞からのnsRNA回収の改善。なお、Fui1は2μmプラスミドから発現した酵母自身のプロモーターである。この遺伝子のゲノムコピーは、これらの株の両方に存在する。このファイルをダウンロードするには、ここをクリックしてください。
補足図2:βエストAID 4Uβ-est AID 4Uグラフィカルプロトコルのアニメーション版。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 1: 4tU_実験_テンプレート.xltx. このファイルをダウンロードするには、ここをクリックしてください。
| プラスミド名 | 輸入者/パーマイーズ | マーカー | コメント | |
| p4フイ | S.セレビシエ・フイ1 | ウラ3 | Fui1はウラシルとウリジンを輸入し、パルス/チェイス実験に最適です。 | |
| pAT2 | S.セレビシエ・フイ1 | ルー2 | ||
| p4フイ-ΔPEST | S.セレビシエ・フイ1 | ウラ3 | Fui1のPESTモチーフは非活性化されているので、細胞のニーズに十分な細胞内ウラシルがある場合、パーマシーは分解されません。ラベリング実験でうまく機能し、パルス/チェイス性能を向上させます。 | |
| p4ファー | S. セレビシエ・ファー4 | ウラ3 | ウラシルパーマーゼ | |
| イェペビ311 | H. サピエンス ENT1 | ルー2 | ミラーら11.また、HSVチミジンキナーゼ遺伝子も含まれています。 | |
| (平衡核サイドトランスポーター) | ||||
| すべてのプラスミドは2 μmベースである。すべてのp4プラスミドおよびpATはプラスミドのpRS16シリーズに基づいている。FUI1およびFUR4は、独自の内因性プロモーターから発現される。 | ||||
表1:このプロトコルで使用されるプラスミド。
ディスカッション
この記事では、非常に迅速かつ特異的な4tUラベリングのためのプロトコルを提示し、新生の回復のために、わずか15sのラベリング後にS.セレビシエから新たに合成されたRNAを、標識されていないRNAによる非常に低い汚染と共に提供する。
ユーザーは、常に低温およびDEPC処理試薬を使用してRNAの完全性を維持するように注意する必要があります。ストレプトアビジンビーズ精製は一般的に信頼できます。ただし、ビード バッファは処理が困難です。それは、そのコンポーネントが正しい順序で追加され、冷やされたりオートクレーブされていない、新鮮に作られる必要があります。一般的な障害には、RNAが沈殿ステップの後に不完全に溶解され、生分解されないか、処理ステップ中に失われるものが含まれます。補足資料には広範なトラブルシューティングのヘルプがあります。
ers4tU では注意が必要な制限があります。すでに述べたものの1つは、4tUが酵母の成長を遅らせることです(図1a)。内因性チオレートされたRNA9とは別に、ラベリング期間中に転写されたRNAのみがこの方法によって精製することができる。チオレーション時間を通じて遺伝子に一時停止したポリメラーサスは、精製可能なチオレート転写物を生成しませんが、チオレーション中にポリメラーが入ったり、一時停止状態を残したりして部分的に標識された転写物は回収できます。変異または増殖条件のために転写が不十分な株は、少しnsRNAを産生するが、ここで使用される技術は、他の方法と比較してnsRNAの回復を改善する。これらの菌株および条件においては、より長い時間および増加した培養量が必要であり、必要な場合がある。ウラシルは窒素の良い供給源であることに注意してくださいので、この方法は窒素飢餓を含む研究に使用される前に試されるべきです。
ers4tUプロトコルは、短命RNAの分析に特に有用であり、その多くは急速に劣化しているため、劣化機構を損なうことなく同定することができません。例としては、不可解な不安定転写物(CUTs)4、およびプロモーター(PROMPTs)19からの早期終了またはプロモーター近位休止18およびアンチセンス転写「上流」によって産生される短い転写物が挙げられる。安定したRNA種の処理中に産生される中間体も一過性であるが、ers4tU転写4を用いて濃縮することができる。したがって、ers4tUプロトコルは、非常に一過性のRNA種を分析し、他の方法に対して大きな利点である近くの生理学的条件下で捕捉することを可能にする上で例外的である。この技術は、通常の20よりも速くまたは遅く伸びるRNAポリメラーゼ変異体における転写および下流RNA処理動態を研究するために使用されてきた。
チオレーションはRNA-seqおよびSLAM-seq21とも互換性があり、非常に短い時間枠内で生産されるすべてのRNAが絶妙な細部で特徴づけられることを可能にする。
開示事項
著者は何も開示していない。
謝辞
この作品は、ウェルカムがJB[104648]に資金を提供して支援されました。細胞生物学のためのウェルカムセンターでの仕事はウェルカムコア資金[092076]によってサポートされています。著者らは、ベラ・モードリン、エマニュエラ・サニ、スザンナ・デ・ルーカス・アリアス、シャイニー・ジョージの助けを研究室のメンバーに認めています。著者はまた、プラスミドYEpEBI31111のためにパトリック・クラマーに感謝したいと思います.
資料
| Name | Company | Catalog Number | Comments |
| β-mercaptoethanol (βME) | Sigma-Aldrich | M3148 | CAUTION toxic. Stock solutions are aproximatly 14 M, make at 1/20 dilution for use |
| Chloroform | Sigma-Aldrich | 25668 | CAUTION toxic |
| Diethyl pyrocarbonate (DEPC) | Sigma-Aldrich | D5758 | add 1/1000 volume to a solution, leave at room temperature for 24 h, then autoclave |
| DMF (N,N-dimethylformamide) | Sigma-Aldrich | 227056 | CAUTION toxic |
| EDTA | Sigma-Aldrich | 3609 | Make 0.5 M and pH to 8.0 with sodium hydroxide |
| Ethanol | Sigma-Aldrich | 29221 | |
| EZ-link HPDP Biotin | Thermo scientific | 21341 | Store protected from light. Disolve all the vial contents in 22.7 mL DMF (to make a 4 mM stock solution). Store away from water, in the dark & at -20 °C. Check the solution before using, as some batches of HPDP precipitate in storage; heat at 42 °C to resuspend. |
| Glucose | Fisher Scientific | G/0500/60 | |
| Glycogen [20 mg/mL] | Sigma-Aldrich | 10901393001 | Store at -20 °C |
| Immobilised TCEP Disulfide Reducing Gel | Thermo Scientific | 77712 | Optional |
| LiCl | Sigma-Aldrich | 793620 | 10 M solution. CAUTION: this gets very hot as is dissolves and can even boil at greater than 100 oC, add the LiCl crystals to the water slowly. |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 63033 | 1 M solution. CAUTION: this gets very hot as is dissolves and can even boil at greater than 100 oC, add the MgCl2 crystals to the water slowly. |
| Methanol | Fisher Scientific | M/4000/PC17 | CAUTION Toxic and flammable |
| NaH2PO4 | Sigma-Aldrich | S3139 | Make 1 M solutions of each and mix in equal amount to obtain a solution of the appropriate pH |
| Na2HPO4 | Sigma-Aldrich | S3264 | |
| NaCl | Sigma-Aldrich | S9888-M | 5 M solution |
| Phenol, low pH. | Sigma-Aldrich | P4682 | Store in the dark at 4 °C. CAUTION toxic |
| Phenol Chloroform 5:1 (125:24:1) low pH. | Sigma-Aldrich | P1944 | Store in the dark at 4 °C. CAUTION toxic |
| Pierce Spin Columns | Thermo Scientific | 69702 | Optional |
| SCSM single drop-out –ura | Formedium | DSCS101 | |
| Sodium Acetate | Sigma-Aldrich | 32318-M | Make a 3 M solution and pH to 5.3 with acetic acid |
| Sodium hydroxide | Sigma-Aldrich | 795429 | CAUTION corrosive |
| SDS (Sodium dodecyl sulfate) | Sigma-Aldrich | 436143 | CAUTION irritant, do not inhale |
| Streptavidin Magnetic beads | NEB | 1420S | Store at 4°C |
| SUPERase-In, RNase inhibitor | Life technologies | AM2696 | Store at -20°C |
| Thiolated Schizosaccharomyces pombe for spike | See section 1.7 of the protocol | ||
| 4-thiouracil (4tU) | ACROS ORGANICS | 359930010 | Store in the dark. Make 100 mM Stock in 1M NaOH, store solutions at -20°C. |
| Tris base | Sigma-Aldrich | 93362 | 1 M solutions at various pH |
| tRNA | Sigma-Aldrich | 10109541001 | 5mg/ml, store at -20°C |
| Uridine | Sigma-Aldrich | U3750 | Make 1 M solution in H2O. Split into 2 mL aliquots and store at -20 C. |
| Yeast nitrogen base without amino acids with amonium sulphate | Formedium | CYN0410 | |
| Zeba Columns 0.5ml | Thermo Scientific | 89882 | Store at 4 °C |
| Zirconia beads | Thistle Scientific | 110791052 | |
| Equipment and Consumables | |||
| Beadbeater | Biospec | 112011EUR | Other homogenisers can be used; the correct conditions for each homogeniser and strain must be established. |
| Bioanalyser (Agilent) or similar to assess RNA quality. If this is not important a spectrophotometer is useful to quantify the RNA. | |||
| Centrifuge: capable of spinning cultures at 4 °C and at least 3000 g. Pre-chill if possible. | |||
| Centrifuge: capable of spinning up to 2 mL tubes at variable speeds upto 13,000 g and down to 1000 g | |||
| Magnetic rack for separating the beads from the sample. The one used in the paper is 3D printed, available from Thingiverse (thing:3562952). Comercially available racks exist | |||
| PCR machine with a heated lid that will allow incubation in the dark. | |||
| Rotating wheel to rotate 1.5 mL tubes end over end | |||
| Shaking heating block (such as Eppendorf Thermomixer) is recomended | |||
| Tubes, centrifuge, Low retention, RNase free 0.5mL | Eppendorf | H179467N | |
| Tubes, centrifuge, Low retention, RNase free 1.5mL | Ambion | AM12350 | |
| Tubes, centrifuge, 50 mL | Sarstedt | 62.547.004 | Other centrifuge tubes are not gas proof allowing CO2 to disolve in the methanol, this comes out of solution vigorously on adding warm culture, leading to sample loss |
| Tubes, centrifuge, 15 mL | Sarstedt | 62.554.001 | |
| Tubes, 2 mL, screw cap | Greiner | 723361 | |
| Tubes 0.2 mL strip of 8 with integral lids | Brand | 781332 |
参考文献
- Duffy, E. E., Schofield, J. A., Simon, M. D. Gaining insight into transcriptome-wide RNA population dynamics through the chemistry of 4-thiouridine. Wiley Interdisciplinary Reviews: RNA. 10 (1), e1513(2018).
- Windhager, L., et al. Ultrashort and progressive 4sU-tagging reveals key characteristics of RNA processing at nucleotide resolution. Genome Research. 22, 2031-2042 (2012).
- Baptista, T., Devys, D. Saccharomyces cerevisiae Metabolic Labeling with 4-thiouracil and the Quantification of Newly Synthesized mRNA As a Proxy for RNA Polymerase II Activity. Journal of Visualized Experiments. (140), e57982(2018).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282(2015).
- Burger, K., et al. 4-thiouridine inhibits rRNA synthesis and causes a nucleolar stress response. RNA Biology. 10, 1623-1630 (2013).
- Mendoza-Ochoa, G. I., et al. A fast and tuneable auxin-inducible degron for depletion of target proteins in budding yeast. Yeast (Chichester England). 36 (1), 75-81 (2018).
- Barrass, J. D., Mendoza-Ochoa, G. I., Maudlin, I. E., Sani, E., Beggs, J. D. Tuning degradation to achieve specific and efficient protein depletion. Journal of Visualized Experiments. , (2019).
- Hobro, A. J., Smith, N. I. An evaluation of fixation methods: Spatial and compositional cellular changes observed by Raman imaging. Vibrational Spectroscopy. 91, 31-45 (2017).
- Gustilo, E. M., Vendeix, F. A. P., Agris, P. F. tRNA's Modifications Bring Order to Gene Expression. Current Opinion in Microbiology. 11, 134-140 (2008).
- Dolken, L., et al. High-resolution gene expression profiling for simultaneous kinetic parameter analysis of RNA synthesis and decay. RNA. 14, 1959-1972 (2008).
- Miller, C., et al. Dynamic transcriptome analysis measures rates of mRNA synthesis and decay in yeast. Molecular Systems Biology. 7, 458(2011).
- Schmitt, M. E., Brown, T. A., Trumpower, B. L. A rapid and simple method for preparation of RNA from Saccharomyces cerevisiae. Nucleic Acids Research. 18, 3091-3092 (1990).
- Rädle, B., et al. Metabolic Labeling of Newly Transcribed RNA for High Resolution Gene Expression Profiling of RNA Synthesis, Processing and Decay in Cell Culture. Journal of Visualized Experiments. (78), e50195(2013).
- Herzog, V. A., et al. Thiol-linked alkylation of RNA to assess expression dynamics. Nature Methods. 14, 1198-1204 (2017).
- Ohrt, T., et al. Molecular dissection of step 2 catalysis of yeast pre-mRNA splicing investigated in a purified system. RNA. 19, 902-915 (2013).
- Alexander, R. D., et al. RiboSys, a high-resolution, quantitative approach to measure the in vivo kinetics of pre-mRNA splicing and 3′-end processing in Saccharomyces cerevisiae. RNA. 16, 2570-2580 (2010).
- Wallace, E. W. J., Beggs, J. D. Extremely fast and incredibly close: cotranscriptional splicing in budding yeast. RNA. 23, 601-610 (2017).
- Adelman, K., Lis, J. T. Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nature Reviews Genetics. 13, 720-731 (2012).
- Preker, P., et al. RNA Exosome Depletion Reveals Transcription Upstream of Active Human Promoters. Science. 322, 1851-1854 (2008).
- Aslanzadeh, V., Huang, Y., Sanguinetti, G., Beggs, J. D. Transcription rate strongly affects splicing fidelity and cotranscriptionality in budding yeast. Genome Research. 28, 203-213 (2018).
- Schofield, J. A., Duffy, E. E., Kiefer, L., Sullivan, M. C., Simon, M. D. TimeLapse-seq: adding a temporal dimension to RNA sequencing through nucleoside recoding. Nature Methods. 15, 221-225 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved