Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Ros imágenes de células vivas durante el desarrollo neuronal
En este artículo
Resumen
Este protocolo describe el uso de un peróxido de hidrógeno genéticamente codificado (H2O2)-biosensoren neuronas de pez cebra cultivadas y larvas para evaluar las funciones fisiológicas de señalización de H 2 O2durante el desarrollo del sistema nervioso. Se puede aplicar a diferentes tipos de células y modificar con tratamientos experimentales para estudiar especies reactivas de oxígeno (ROS) en el desarrollo general.
Resumen
Las especies reactivas de oxígeno (ROS) son moléculas de señalización bien establecidas, que son importantes en el desarrollo normal, la homeostasis y la fisiología. Entre los diferentes ROS, el peróxido de hidrógeno(H2O2)se caracteriza mejor con respecto a los roles en la señalización celular. H2O2 se ha implicado durante el desarrollo en varias especies. Por ejemplo, se ha detectado un aumento transitorio deH2O2 en embriones de pez cebra durante los primeros días después de la fertilización. Además, el agotamiento de una importante fuente celular de H2O2, la NADPH oxidasa (NOX), deteriora el desarrollo del sistema nervioso, como la diferenciación, el crecimiento axonal y la orientación de las células ganglionares de la retina (RGCs) tanto in vivo como in vitro. Aquí, se describe un método para la obtención de imágenes intracelulares H2O2 niveles en las neuronas de pez cebra cultivadas y larvas enteras durante el desarrollo utilizando el genéticamente codificado H2O2-biosensor específico, roGFP2-Orp1. Esta sonda puede expresarse de forma transitoria o estable en larvas de pez cebra. Además, la lectura ratiométrica disminuye la probabilidad de detectar artefactos debido a la expresión génica diferencial o efectos de volumen. En primer lugar, se demuestra cómo aislar y cultivar RGCs derivados de embriones de pez cebra que expresan transitoriamente roGFP2-Orp1. Luego, utilizamos larvas enteras para monitorear los niveles deH2O2 a nivel de tejido. El sensor ha sido validado por la adición deH2O2. Además, esta metodología podría utilizarse para medir los niveles deH2O2 en tipos de células y tejidos específicos mediante la generación de animales transgénicos con expresión de biosensores específicos de tejidos. Como el pez cebra facilita las manipulaciones genéticas y de desarrollo, el enfoque demostrado aquí podría servir como una tubería para probar el papel de H2O2 durante el desarrollo embrionario neuronal y general en vertebrados.
Introducción
La señalización reactiva de especies de oxígeno (ROS) regula el desarrollo y el funcionamiento del sistema nervioso1. Una fuente celular importante de ROS son las NADPH oxidasas (NOX), que son proteínas transmembrana que generan superóxido y peróxido de hidrógeno(H2O2)2. Las enzimas NOX se encuentran en todo el sistema nervioso central (SNC), y el ROS derivado de NOX contribuye al desarrollo neuronal3,4,5,6. Se ha demostrado que el mantenimiento y la diferenciación de las células madre neurales, el establecimiento de la polaridad neuronal, la excrecencia de la neurita y la plasticidad sináptica requieren niveles adecuados de ROS7,8,9,10,11. Por otro lado, la producción incontrolada de ROS por los NOXes contribuye a trastornos neurodegenerativos como la enfermedad de Alzheimer, la esclerosis múltiple y la lesión cerebral traumática12,13,14. Por lo tanto, la producción de ROS fisiológicamente relevante es fundamental para mantener condiciones saludables.
El desarrollo de biosensores codificados genéticamente facilitó enormemente la detección de ROS celular. Una ventaja importante de los biosensores codificados genéticamente es el aumento de la resolución temporal y espacial de la señal ROS, ya que estos sensores se pueden dirigir específicamente a ubicaciones distintas. GFP sensible a redox (roGFP) es un tipo de estos biosensores ROS. La variante roGFP2-Orp1 detecta específicamenteH2O2 a través de su dominio Orp1, que es una proteína de la familia glutatión peroxiredoxina de la levadura15,16. La oxidación de la proteína Orp1 se transfiere a roGFP2 para alterar su conformación (Figura 1A). La sonda exhibe dos picos de excitación cercanos a 405 nm y 480 nm, y un solo pico de emisión a 515 nm. Tras la oxidación, la intensidad de fluorescencia alrededor de los picos de excitación cambia: mientras que la excitación de 405 nm aumenta, la excitación de 480 nm disminuye. Por lo tanto, roGFP2-Orp1 es un biosensor ratiométrico, y los niveles deH2O2se detectan por la relación de intensidades de fluorescencia excitadas en dos longitudes de onda diferentes (Figura 1B). En general, roGFP2-Orp1 es una herramienta versátil para imágenes ROS que se puede utilizar de manera eficiente in vivo.
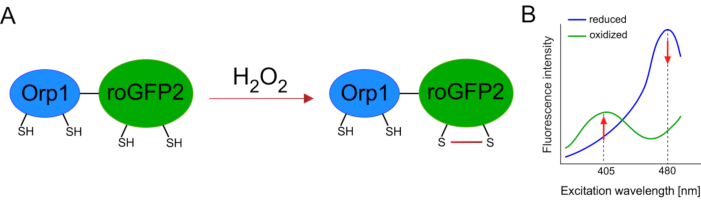
Figura 1:Representación esquemática y espectros de excitación de roGFP2-Orp1. (A)La transferencia de oxidantes se produce entre Orp1 y roGFP2 en respuesta aH2O2,dando lugar a cambios conformacionales en roGFP2. (B) Los espectros de excitación del roGFP2-Orp1 exhiben dos picos de excitación a 405 nm y 480 nm y un pico de emisión única a 515 nm. Tras la oxidación porH2O2, la excitación de 405 nm aumenta mientras que la excitación de 480 nm disminuye. Esto da lugar a una lectura ratiometric para la presencia deH2O2. La cifra ha sido modificada de Bilan y Belousov (2017)16 y Morgan et al. (2011)25. Haga clic aquí para ver una versión más amplia de esta figura.
El sistema modelo Danio rerio (pez cebra) tiene varias ventajas para aplicar biosensores codificados genéticamente. La transparencia óptica de los embriones y larvas permite la obtención de imágenes in vivo no invasivas. Se están desarrollando nuevas herramientas de imagen para lograr una mayor resolución y una penetración más profunda17. Además, existen herramientas establecidas para la manipulación genética (expresión ectópica de ARNm, transgénesis de Tol2, etc.) y la edición del genoma (TALENs, CRISPR/Cas9, etc.), que promueve la generación de animales transgénicos18. A medida que los embriones de pez cebra se desarrollan fuera de la madre, este sistema permite aún más el acceso y la manipulación de los embriones. Por ejemplo, las inyecciones en etapa unicelular y los tratamientos farmacológicos se pueden hacer fácilmente.
Aquí, utilizamos pez cebra para expresar transitoriamente el H2O2-biosensores específicos roGFP2-Orp1 mediante la inyección de ARNm transcrito in vitro. Estos embriones se pueden utilizar tanto para la obtención de imágenes in vitro de neuronas cultivadas como para la obtención de imágenes in vivo (Figura 2). Se describe un protocolo para la disección y chapado de células ganglionares de la retina (RGCs) de embriones de pez cebra seguido de la evaluación de H2O2- niveles en neuronas cultivadas. Entonces, presentamos un método para la proyección de imagen in vivo de roGFP2-Orp1-expresando embriones y larvas usando microscopia confocal. Este enfoque no sólo permite determinar los niveles fisiológicos deH2O2-sino también los cambios potenciales que ocurren en diferentes etapas o condiciones de desarrollo. En general, este sistema proporciona un método confiable para detectarH2O2 en células vivas y animales para estudiar el papel deH2O2 en el desarrollo, la salud y la enfermedad.
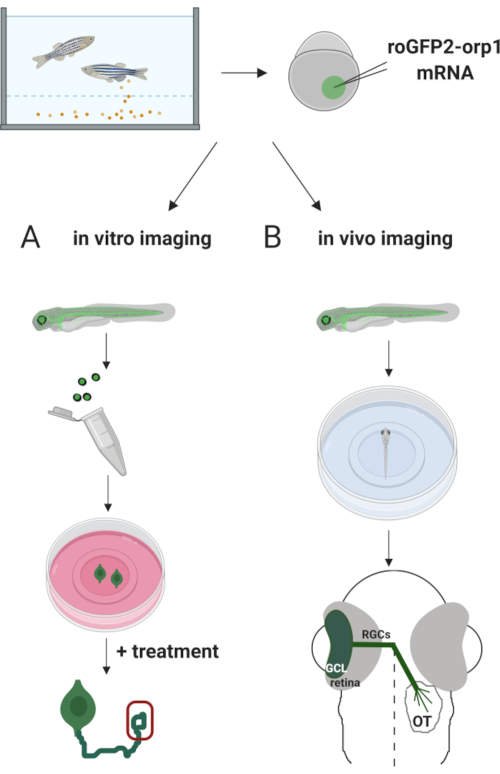
Figura 2. Esquema del enfoque experimental. Brevemente, después de la recolección de embriones, roGFP2-Orp1 ARNm se inyecta en la yema de los embriones de pez cebra en etapa unicelular. Los embriones en desarrollo se pueden utilizar tanto para(A) imágenes in vitro como(B) in vivo. (A)Los embriones GFP-positivos se utilizan para diseccionar retinas para la colección de RGC en 34 hpf. Los RGCs disociados se platean en cubrebocas recubiertas de PDL/laminina en medios ZFCM (+). La proyección de imagen del cono del crecimiento se puede conducir mientras que RGCs extiende sus axones después de 6-24 h de la galjanoplastia. Las células pueden ser sometidas a diferentes tratamientos para medir los posibles cambios en los niveles deH2O2.- Aquí, medimos H2O2-niveles en los conos de crecimiento de RGCs (rojo). (B)Los embriones positivos de GFP se utilizan para la obtención de imágenes in vivo. A la edad deseada, los embriones pueden anestesiarse y montarse en platos con fondo de vidrio de 35 mm para obtener imágenes confocales. Aquí, los embriones se montan ventralmente para la proyección de imagen retiniana. Schematic muestra el desarrollo de la retina en el pez cebra. Los RGCs forman la capa de la célula del ganglio (GCL), que es la capa más interna en retina. Los axones de RGC se convierten en nervio óptico para cruzar midline, formando quiasma óptico. Entonces, los axones de RGC crecen dorsalmente para hacer sinapsis en el tectum óptico en el mesencéfano. Haga clic aquí para ver una versión más amplia de esta figura.
Protocolo
Todos los experimentos con animales fueron revisados éticamente y aprobados por el Comité de Cuidado y Uso de Animales de Purdue (PACUC), siguiendo las directrices de los NIH con el protocolo 2006002050 aprobado el 24/07/2020.
1. Preparación de soluciones
- Medios E2 (1x)
- Prepare soluciones de 100x E2A (500 mL), 500x E2B (100 mL) y 500x E2C (100 mL) combinando todos los componentes que se muestran en la Tabla 1. Soluciones de autoclave E2A, E2B y E2C. Conservar a 4 °C.
- Para medios 1x E2: Combine 5 mL de 100x E2A, 1 mL de 500x E2B y 1 mL de 500x E2C. Llevar el volumen a 500 mL con agua estéril. Ajuste el pH a 7.0-7.5.
- Prepare alícuotas de 50 mL de 1x almacén de medios E2 a -20 °C para almacenamiento a largo plazo. Sin embargo, las precipitaciones pueden ocurrir al descongelarse. Asegúrese de que la precipitación esté completamente disuelta antes de usar la solución común.
| solución | componente | importe | concentración |
| 100X E2A (500mL) | |||
| NaCl | 43,8 g | 1500 milímetros | |
| Kcl | 1,88 g | 50 mM | |
| MgSO4 | 6 g | 100 mM | |
| KH2PO4 | 1,03 g | 15 mM | |
| Na2HPO4 | 0,34 g | 5 mM | |
| 500X E2B (100 mL) | |||
| CaCl2 | 5,5 g | 500 mM | |
| 500X E2C (100 mL) | |||
| NaHCO3 | 3 g | 350 milímetros | |
| 1X E2 (500 mL) | |||
| 100X E2A | 5 mL | 1X | |
| 500X E2B | 1 mL | 1X | |
| E2C de 500X | 1 mL | 1X |
Tabla 1: Componentes de 1x medios E2 para el cultivo celular de pez cebra.
- Medios E3 (1x)
- Disuelva los componentes en agua estéril de 1 L como se muestra en la Tabla 2 para hacer 100x stock. Diluir el stock en agua estéril para hacer 1x E3 media.
- Añadir 0,2% de azul de metileno. Para 20 mL de 1x medio E3, añadir 40 μL de azul de metileno.
- Haga otro lote sin azul de metileno para obtener imágenes fluorescentes.
| componente | Importe (g) | Concentración en stock 100X (mM) |
| NaCl | 29.22 | 500 |
| Kcl | 1.26 | 17 |
| CaCl2 2H2O | 4.85 | 33 |
| MgSO4 7H2O | 8.13 | 33 |
Tabla 2: Componentes de medios 100x E3 para el mantenimiento de embriones de pez cebra.
- Solución de 80x solución salina
- Combinar todos los componentes que se muestran en el Cuadro 3. Añadir agua para hacer 100 mL de solución. Mezclar hasta que se disuelvan todos los componentes. Guarde la solución a 4 °C.
| componente | Importe (g) | Concentración en stock (mM) |
| glucosa | 1.44 | 80 |
| Piruvato de sodio | 0.44 | 40 |
| CaCl2 2H2O | 0.148 | 10 |
| HEPES | 6.1 | 256 |
Tabla 3: Componentes de la solución salina 80x para medios de cultivo celular de pez cebra.
- Medio de cultivo celular de pez cebra (ZFCM+)
- Combine todos los componentes que se muestran en la Tabla 4 para fabricar medios de 250 mL. Ajuste el pH a 7.5. Filtrar soportes que utilizan filtros de 0,22 μm y almacenarlos a 4 °C.
| componente | Importe (mL) | Volumen % |
| Medio L-15 (con rojo fenol) | 212.75 | 85.1 |
| Suero fetal bovino (FBS) | 5 | 2 |
| Penicilina/Estreptomicina | 1 | 0.4 |
| Solución salina 80X | 3.125 | 1.25 |
| Agua | 28.125 | 11.25 |
Tabla 4: Componentes del medio de cultivo celular de pez cebra con suero y antibióticos.
- Medio de cultivo celular de pez cebra para la obtención de imágenes (ZFCM-)
- Combine todos los componentes que se muestran en la Tabla 5 para fabricar medios de 250 mL. Ajuste el pH a 7.5. Soporte de filtro que utiliza un filtro de 0,22 μm.
- Realice alícuotas de un solo uso (lotes de 50 mL) para evitar la contaminación. Mantener a 4 °C.
| componente | Importe (mL) | Volumen % |
| Medio L-15 (sin rojo fenol) | 212.75 | 85.1 |
| Solución salina 80X | 3.125 | 1.25 |
| Agua | 34.125 | 13.65 |
Tabla 5: Componentes del medio de cultivo celular de pez cebra sin suero y antibióticos para la obtención de imágenes in vitro.
- Moldes de inyección
- Disuelva la agarosa al 1,5% en medios E3. Verter ~ 25 mL de agarosa en una placa de Petri de 100 x 15 mm.
- Establezca el molde en la parte superior de la agarosa con un ángulo de 45 ° con respecto a la superficie, y deje que flote sobre la agarosa con una cámara lenta. Esto ayudará a evitar burbujas. Deje que la agarosa se enfríe y solidifique en la parte superior del banco.
- Una vez completamente solidificado, retire el molde lentamente para evitar la rotura de la agarosa. Agregue medios E3 frescos, agregue película de parafina alrededor del plato para evitar derrames y guárdelo a 4 °C ( Figura 3).
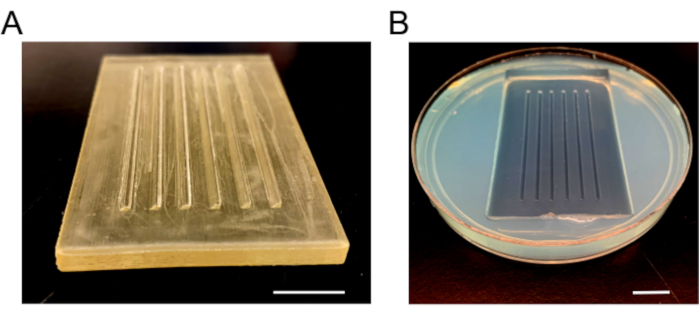
Figura 3:Imágenes de molde de inyección. (A) El molde de plástico que se utiliza para hacer placas de inyección. El molde tiene seis rampas, una de 90° y otra de 45° bisela para mantener los embriones en su lugar. (B) La placa de inyección después de que la agarosa se solidifique y se retire el molde. Barras de escala = 1 cm. Haga clic aquí para ver una versión más grande de esta figura.
2. Preparación e inyección de arnm roGFP2-Orp1
NOTA: la construcción roGFP2-Orp1 se obtuvo del Dr. Tobias Dick, DKFZ, Alemania. Fue sub-clonado en el vector pCS2+ en el Laboratorio del Dr. Qing Deng, Universidad de Purdue. Para prevenir la degradación por RNase, se deben tomar varias precauciones. Los reactivos y tubos libres de ARNasa deben usarse en todo momento, se deben usar guantes para todos los pasos y, alternativamente, los materiales y las superficies se pueden limpiar con un agente de limpieza para la eliminación de RNasa.
- Linealice los 3-10 μg del vector pCS2+/roGFP2-Orp1 con NotI.
- Purificar el plásmido linealizado con un kit de limpieza por PCR.
- In vitro transcribir el ARNm roGFP2-Orp1 con un kit de transcripción in vitro según las instrucciones proporcionadas por el fabricante.
- Ensamblaje de reacción de transcripción capsular
- Coloque la ARN polimerasa y el ADN linealizado/purificado sobre el hielo. Tampón de reacción vortex 10x y NTP/CAP 2x hasta que estén completamente en solución. Guarde NTP/CAP en hielo, pero mantenga el tampón a temperatura ambiente (RT) mientras ensambla la reacción. Toque y gire todos los reactivos antes de abrir los tubos para evitar la contaminación.
- Configure la reacción de síntesis de ARN en el orden indicado a continuación en RT en un tubo centrífugo de 0,5 mL libre de ARNasa. El volumen final de la reacción es de 20 μL. La configuración de la reacción se muestra en el Cuadro 6.
- Agregue 10 μL de mezcla de ribonucleótidos, 2x NTP/CAP y agua libre de nucleasa, si es necesario, al tubo. Añadir 2 μL 10X tampón de reacción. Añadir 1-1,5 μg de ADN lineal (hasta 6 μL). Si es necesario, agregue agua sin nucleasa para que el volumen de reacción de 20 μL sea de 20 μL.
- Cierre el tubo, el vórtice brevemente y el microfugio touch-spin. Añadir 2 μL de mezcla de enzimas 10x SP6. Cierre con un clic con el dedo y toque-spin en un microfuge.
- Colocar en 37 °C durante 2-2,5 h (puede ir hasta 18 h).
NOTA: En el experimento presentado durante la noche se realizó una incubación de 16-18 h a 37 °C para obtener los mejores resultados. - Agregue 1 μL de DNasa para eliminar la plantilla de ADN, haga clic con el dedo, toque el giro e incube a 37 °C durante 15 min.
- Ensamblaje de reacción de transcripción capsular
| Reactivo | Volumen (μL) | Cantidad en reacción |
| 2X NTP/CAP | 10 | 1X |
| Búfer de reacción 10X | 2 | 1X |
| Plantilla DE ADN | Hasta 6 | 1-1,5 μg |
| Agua libre de nucleasa | Añadir para hacer 20 μL | |
| Mezcla de enzimas 10X SP6 | 2 | 1X |
Tabla 6: Configuración de la reacción para la transcripción in vitro del ARNm roGFP2-Orp1.
- Recuperación de ARN
- Añadir 25 μL de cloruro de litio (LiCl) suministrado en el kit de transcripción in vitro. Colocar a -20 °C en un congelador sin heladas durante al menos 30 min.
- Girar durante 25 min a la velocidad máxima en una centrífuga de sobremesa a 4 °C. Retire y deseche el sobrenadante con cuidado, para no molestar a la pastilla. Añadir 25 μL de etanol frío al 75% y girar durante 5 min a 4 °C.
- Retirar con cuidado y desechar el sobrenadante. Deje secar al aire pellet durante al menos 5 min en RT. No dejar secar. Añadir 12 μL de tampón Tris-EDTA (TE) libre de nucleasa (pH 7.0) y mantener la muestra en hielo.
- Mida la concentración de ARN con un espectrofotómetro. Se suele obtener 0,5- 1 μg/μL.
- Preparar 100 ng/μL de ARNm en solución de color rojo fenol (0,5% de rojo fenol en la solución salina tamponada con fosfato de Dulbecco -DPBS) y alícuota para un solo uso (3-5 μL). Guarde las alícuotas de ARNm a - 80 °C.
- Microinyección de ARNm
- El día de la inyección, utilice una de las alícuotas de ARNm y siga el protocolo de inyección de embriones de pez cebra para inyectar 1 nL del ARNm en los embriones en etapa unicelular a través de su yema19. A continuación se proporciona una breve descripción.
- Criar peces adultos y recoger embriones como se describió anteriormente20.
- Tire de las agujas de microinyección con el tirador de pipetas. Corte la punta de las agujas con las fuerzas para crear una abertura de punta de 10 μm.
- Alinear embriones en un molde de inyección que se describió en el paso 1.6.
- Inyecte 1 nL del ARNm en rojo fenol con una pipeta de microinyección de vidrio.
- Recoger embriones y mantenerlos en medios E3.
- Mantenga los embriones en la incubadora de 27 °C en medios E3 hasta que se logre la etapa de desarrollo deseada. Los embriones inyectados se pueden utilizar tanto para imágenes in vitro (sección 3 y sección 4) como in vivo (sección 5). Los embriones que expresan roGFP2-Orp1 pueden ser preseleccionados antes de los experimentos con un microscopio de disección regular equipado con luz fluorescente y un conjunto de filtros azules / verdes.
3. Cultivo celular de ganglios primarios de la retina derivados de embriones de pez cebra
Nota : este protocolo está adaptado de un método publicado previamente 21. Realice los pasos 3.1 y 3.2 en una campana de flujo laminar.
- Preparación de cubrebocas
- Prepare 4-6 placas de cultivo en cada experimento. Utilice cubrebocas limpiadas con ácido (22 x 22 mm cuadrados; 0.16-0.19 mm de espesor) que se almacenan en etanol 100%.
- Retire una cubierta del contenedor de almacenamiento mediante el uso de autocarórceps y llama para eliminar el etanol residual.
- Seque al aire el cubrebocas por completo colocándolo en un ángulo dentro de un plato de cultivo de 35 mm.
- Prepare la solución de trabajo de poli-D-lisina (PDL) (1x) diluyendo 10x caldo (5 mg/mL) en agua estéril.
- Aplique 100 μL de 0,5 mg/mL de PDL en el centro de cada cubrebocas y evite la dispersión de la solución a los bordes.
- Incubar la PDL en cubrebocas durante 20-30 min a temperatura ambiente (RT). Asegúrese de que la PDL no se seque.
- Lave la PDL con 0,5 mL de agua estéril tres veces. Dejar que las placas se sequen por completo.
- Preparar la solución de trabajo de laminina (1x) diluyendo 50x caldo (1 mg/mL) en 1x PBS.
- Aplique 100 μL de 20 μg/mL de laminina en PBS al centro de cada cubrebocas y evite la propagación de la solución a los bordes.
- Incubar las placas a 37 °C en una incubadora humidificada durante 2-6 h. Evite el secado de la solución de laminina.
- Disección embrionaria y galjanoplastia de CRG
- Preparar y etiquetar cuatro platos de cultivo de tejidos de 35 mm y rellenar con 4 mL de: etanol al 70%, "E2 media 1", "E2 media 2", "E2 media 3" el día de la disección. Mantenga los platos en la nevera hasta las disecciones.
- Cuando los embriones de pez cebra son 34 horas después de la fertilización (hpf), tomar los platos de cultivo recubiertos con laminina fuera de la incubadora y lavar las cubiertas tres veces con 0,5 mL de 1x PBS.
- Después del lavado final, agregue 4 mL de medios ZFCM(+) a cada plato de cultivo y evite secar el plato.
- Recupere los platos de cultivo preparados del paso 3.2.1. Déjalos equilibrar a RT.
- Llene 4-6 tubos de PCR con 15 μL de soporte ZFCM(+). Se necesita un tubo para preparar RGCs de 4 ojos para ser plateados en una cubierta.
- Recupere embriones de pez cebra de la incubadora y sumerja los embriones en un plato de cultivo de tejidos de 35 mm que contenga etanol al 70% durante 5-10 s para esterilizar.
- Usando una pipeta de transferencia, transfiera embriones a la antena parabólica E2 Media 1 que contiene medios E2 estériles para lavar el exceso de etanol.
- Transfiera los embriones al plato E2 Media 2 y retire sus coriones con pórceps afilados.
- Transferir embriones a la placa final E2 Media 3 para realizar disecciones.
- Usando un par de pórceps finos, diseccione las retinas como se describió anteriormente 22.
- Coloque y sostenga los embriones anteriores a su yema con uno de los pórceps y retire la cola posterior al saco vitelino con las otras fuerzas.
- Agarra el cuello con las medidas de subapas y típate la cabeza para exponer el cerebro y los ojos a los medios E2. Evite cortar el saco vitelino.
- Con la punta de las fórceps finas, gire suavemente los ojos fuera de la cabeza, mientras que sostiene el tejido craneal hacia abajo con las segundas fórceps. Mantenga los ojos aislados de los restos de tejido adyacentes.
- Transfiera cuatro ojos a uno de los tubos previamente preparados que contienen ZFCM(+).
- Valore suavemente hacia arriba y hacia abajo con la pipeta P20 y una punta amarilla unas 45 veces para disociar las células. Evite las burbujas de aire.
- Transfiera el ZFCM(+) con las células disociadas al centro del coverslip. Repita los pasos 10-12 para cubrir los cubrebocas adicionales.
- Mantenga los cultivos en la sobremesa a 22 °C en un estante de espuma de poliestireno para absorber las vibraciones.
- Realice la proyección de imagen 6-24 h después de la galjanoplastia.
NOTA: Utilice pipeta de transferencia para translocar embriones a diferentes platos de cultivo. Cambie la pipeta de cada solución para evitar el transporte de etanol (Pasos 6-8).
4. Imágenes ros in vitro de neuronas RGC cultivadas
- En el día de la proyección de imagen (típicamente 6-24 h después de la galjanoplastia de la célula), compruebe las células bajo microscopio para validar el crecimiento de los axones de RGC.
- Para las imágenes de células vivas, transfiera los cubrebocas del plato de cultivo a una cámara de imágenes de células vivas. En este caso se utilizó una cámara abierta a medida, que se ha descrito anteriormente23.
- Configure el microscopio para la toma de imágenes. Utilice un microscopio invertido equipado con un objetivo de contraste de interferencia diferencial (DIC), un filtro rojo de paso largo OG590 y una cámara EM-CCD.
- Antes de la creación de imágenes, sustituya el medio ZFCM(+) por ZFCM(-).
- Una vez que las células se colocan con un objetivo de 10x, adquiera imágenes a un aumento de 60x utilizando un objetivo de inmersión de aceite de NA alto. Utilice un aumento adicional de 1,5x.
- En primer lugar, adquirir imágenes DIC. A continuación, imagen roGFP2-Orp1 utilizando un conjunto de filtros adecuado. Excitar roGFP2-Orp1 con filtros de excitación 405/20 y 480/30 nm secuencialmente y adquirir imágenes con filtro de emisión de 535/30 nm después de que la luz de emisión haya pasado el espejo dicroico 505DCXR.
- Después de tomar el primer conjunto de imágenes, intercambie medios con medios que contengan diferentes soluciones de tratamiento. Los medios deben cambiarse cada 30 minutos de imágenes para evitar cambios en el pH y la osmolaridad.
5. Imágenes ros in vivo de embriones en desarrollo
- Para la proyección de imagen in vivo, mantenga los embriones en los medios E3 que contienen 0.003% Phenylthiourea (PTU) sin el azul de metileno de 22-24 hpf. Intercambiar medios y eliminar embriones muertos a diario.
- A la edad deseada, anestesiar los embriones en 0.016% tricaine. Monte embriones anestesiados en agarosa de baja fusión al 1% en platos de cultivo con fondo de vidrio de 35 mm. Los embriones se pueden orientar dorsal, ventral o lateralmente, dependiendo de la región de interés para la proyección de imagen.
- Después de que la agarosa se solidifique, llene los platos con medios E3 sin azul de metileno / tricaína al 0.016%.
- Configure el microscopio para la toma de imágenes. Utilice un microscopio confocal de barrido láser invertido. Alternativamente, use un microscopio confocal vertical equipado con una lente de inmersión en agua para obtener imágenes de embriones montados en la parte superior de una gota de agarosa.
- Excitar roGFP2-Orp1 con filtros de excitación de 405 nm y 488 nm secuencialmente y adquirir las imágenes correspondientes con filtros de emisión en el rango de 515-535 nm.
- Adquirir z-stacks con 5 μm de espesor de sección a través de la parte deseada de los embriones. Los embriones se pueden conservar para obtener imágenes en etapas posteriores del desarrollo.
- Después de la toma de imágenes, retire los embriones de la agarosa con pórceps finos y manténgalo en la incubadora hasta la edad deseada en medios libres de azul de metileno con PTU.
6. Análisis y procesamiento de imágenes
- Medición de los niveles de H2O2 basada en valores de relación 405/480
- Utilice un software adecuado para el análisis de imágenes. El software ImageJ se utilizó aquí para el análisis y procesamiento de imágenes.
- Abra imágenes DIC, 405/535 y 480/535 en el software ImageJ arrastrando los archivos o haciendo clic en | Abrir. Si aún no lo ha hecho, convierta las imágenes a 32 bits haciendo clic en | Tipo | .. . . de 32 bits
- Defina la región de interés (ROI) con la herramienta de mano libre desde la barra de control (cuerpo celular, cono de crecimiento, retina, etc.). Abra el administrador de ROI haciendo clic en Analizar | Herramientas | Gestor de ROI. Haga clic en Agregar en la pestaña Administrador de ROI para agregar el ROI definido.
- Dibuje una región cercana al ROI y agréguele como ROI en segundo plano. Mida los valores medios de fondo seleccionando el ROI y haciendo clic en Medir en la pestaña administrador de ROI.
- Tenga en cuenta los valores de intensidad promedio de la medición. Reste el valor de fondo promedio de las imágenes fluorescentes haciendo clic en Procesar | | matemáticas Restar. Realice este paso para las imágenes 405/535 y 480/535.
- Agregue el valor de "1" a la imagen fluorescente 480/535 para eliminar los valores "0" haciendo clic en Procesar | | matemáticas Agregar función antes del cálculo de la relación.
- Haga clic en procesar | Calculadora de imágenes | Función Dividir en ImageJ para dividir la imagen 405/535 por la imagen 480/535 píxel por píxel. Seleccione la imagen 405/535 para dividir por la imagen 480/535. Seleccione una imagen de salida de 32 bits.
- Aplique el ROI a la imagen de proporción haciendo clic primero en la imagen de proporción y luego en el ROI en la pestaña Administrador de ROI.
- Mida los valores de relación promedio de 405/535 imagen a 480/535 imagen haciendo clic en Medir en la pestaña Administrador de ROI.
- Realice los pasos 6.1.2-6.1.9 para que se realicen tantas muestras como sea posible para realizar el análisis estadístico adecuado.
- Visualización de la imagen de relación
NOTA: Este procedimiento consiste en restar el fondo fuera de la muestra y aplicar una tabla de busca de color a la imagen.- Una vez que la imagen de proporción se ha creado en ImageJ en el paso 6.1.7., cree una imagen negra de 32 bits haciendo clic en Archivo | Nueva | Imagen.
- Aplique el ROI para el que desea mostrar los niveles H2O2 a la nueva imagen haciendo clic primero en la nueva imagen y luego en el ROI desde la pestaña del administrador de ROI.
- Cree una máscara haciendo clic en Editar | | de selección Crear máscara.
- Divida la imagen de la máscara por 255 para ajustar el valor del ROI a "1" y los valores de fondo serán "0". Haga clic en procesar | | matemáticas Dividir y escribir 255.
- Multiplique la máscara con la imagen de relación haciendo clic en Procesar | Calculadora de imágenes | Función multiplicar. Esto dará como resultado una imagen de relación de escala de grises que muestra solo el ROI.
- Cambie la tabla de buscar a "Fuego" haciendo clic en | de imagen Buscar tablas | Fuego.
NOTA: Se puede aplicar un factor de multiplicación a todas las imágenes para una mejor visualización de la relación haciendo clic en Procesar | | de imagen Multiplique. - Convierta la imagen de proporción a 8 bits haciendo clic en | Tipo | 8 bits.
- Agregue la barra de calibración haciendo clic en Analizar | Herramientas | Barra de calibración.
Resultados
Los RGCs de pez cebra cultivados extienden axones dentro de 1d. En la Figura 4A se muestra una imagen representativa de la relación 405/480 del biosensor H2O2. El cuerpo celular, el axón y los conos de crecimiento son claramente visibles en las neuronas individuales. Estas neuronas pueden ser sometidas a diferentes tratamientos a lo largo del tiempo para monitorizar los cambios deH2O2. Anteriormente encontramos que la adición de 100 μM H2...
Discusión
Hay varios pasos críticos que necesitan atención a lo largo de este protocolo. Creemos que teniendo en cuenta estos puntos mejorará el flujo experimental. Para el cultivo primario de RGC, la esterilidad de la ZFCM (-) es muy importante, ya que este medio de cultivo no contiene antibióticos y la contaminación puede ocurrir antes o durante la toma de imágenes. Para evitarlo, aconsejamos preparar y utilizar ZFCM(-) sólo dentro de un gabinete de bioseguridad y hacer medios ZFCM(-) frescos regularmente (Paso 1.5). Adem...
Divulgaciones
Los autores declaran que no tienen ningún conflicto de intereses.
Agradecimientos
Este trabajo fue apoyado por los Institutos Nacionales de Salud (Subvención R01NS117701), la Fundación Nacional de Ciencias (Subvención 1146944-IOS), el Fondo de Investigación de la Médula Espinal Traumática y lesiones cerebrales de Indiana (Subvención 20000289), la Fundación de Investigación Purdue (Subvención 209911) y la Oficina del Vicepresidente Ejecutivo de Investigación y Asociaciones de la Universidad de Purdue (Subvención 210362). Agradecemos al Dr. Cory J. Weaver y Haley Roeder por establecer el protocolo de cultivo de RGC de pez cebra. Agradecemos a Haley Roeder, además, por proporcionar los datos de la Figura 4. Agradecemos a Leah Biasi y Kenny Nguyen por la ayuda con la cultura RGC. Agradecemos a Gentry Lee por editar el texto. Agradecemos al Dr. Tobias Dick por proporcionar roGFP2-Orp1 y al Dr. Qing Deng para el vector pCS2+ que contiene roGFP2-Orp1. La figura 2 se crea con Biorender.com.
Materiales
| Name | Company | Catalog Number | Comments |
| 35-mm culture dish | Sarstedt | 83-3900 | |
| 35-mm glass bottom dish | MatTek | P35G-1.5-10-C | |
| Agarose | Fisher Scientific | BP160-500 | |
| Borosilicate Glass Capillary Tubes | Sutter/Fisher Scientific | NC9029378 | |
| Calcium Chloride Dihydrate | Fisher Scientific | C79-500 | |
| Cover glass | Corning | 2850-22 | |
| Disposable Petri Dishes (100 x 15 mm) | VWR | 25384-094 | |
| Fetal Bovine Serum | ThermoFisher Scientific | 26140087 | |
| Glucose | Sigma Aldich | G7528 | |
| HEPES | Sigma Aldich | H4034 | |
| Injection Mold | Adaptive Science Tools | TU-1 | |
| Inverted Microscope | Nikon | TE2000 | |
| Laminin | ThermoFisher Scientific | 23017-015 | |
| Laser Scanning Confocal Microscopy | Zeiss | 710 | |
| Leibovitz's L-15 Medium with phenol red | Gibco/Fisher Scientific | 11-415-064 | |
| Leibovitz's L-15 Medium without phenol red | Gibco/Fisher Scientific | 21-083-027 | |
| Low melting agarose | Promega | V2111 | |
| mMessage mMachine SP6 Transcription Kit | Invitrogen | AM1340 | |
| NotI | New England Biolabs | R0189S | |
| PBS | Hyclone/Fisher Scientific | SH3025601 | |
| Penicillin/streptomycin | ThermoFisher Scientific | 15140122 | |
| Phenol Red | Sigma Aldich | P0290 | |
| Phenylthiourea (PTU) | Sigma Aldich | P7629 | |
| Pneumatic PicoPump | World Precision Instruments | PV820 | |
| Poly-D-Lysine (PDL) | Sigma Aldich | P7280 | |
| QiaQUICK PCR Purification Kit | QIAGEN | 28104 | |
| Recombinant mouse slit2 | R&D Systems | 5444-SL-050 | |
| Sodium Pyruvate | Sigma Aldich | P5280 | |
| Steritop 0.22 µm filter | Millipore | S2GPT05RE | |
| TE Buffer | Ambion | AM9860 | |
| Tricaine Methanesulfonate | Sigma Aldich | E10521 | |
| Vertical Pipette Puller | David Kopf Instruments | 700C |
Referencias
- Bórquez, D. A., et al. Dissecting the role of redox signaling in neuronal development. Journal of Neurochemistry. 137 (4), 506-517 (2016).
- Bedard, K., Krause, K. -. H. The NOX family of ROS-generating NADPH oxidases: Physiology and pathophysiology. Physiological Reviews. 87 (1), 245-313 (2007).
- Weaver, C. J., Leung, Y. F., Suter, D. M. Expression dynamics of NADPH oxidases during early zebrafish development. Journal of Comparative Neurology. 524 (10), 2130-2141 (2016).
- Terzi, A., Suter, D. M. The role of NADPH oxidases in neuronal development. Free Radical Biology and Medicine. 154, 33-47 (2020).
- Infanger, D. W., Sharma, R. V., Davisson, R. L. NADPH oxidases of the brain: Distribution, regulation, and function. Antioxidants & Redox Signaling. 8 (9-10), 1583-1596 (2006).
- Coyoy, A., Olguin-Albuerne, M., Martinez-Briseno, P., Moran, J. Role of reactive oxygen species and NADPH-oxidase in the development of rat cerebellum. Neurochemistry International. 62 (7), 998-1011 (2013).
- Le Belle, J. E., et al. Proliferative neural stem cells have high endogenous ROS levels that regulate self-renewal and neurogenesis in a PI3K/Akt-dependant manner. Cell Stem Cell. 8 (1), 59-71 (2011).
- Nayernia, Z., et al. Decreased neural precursor cell pool in NADPH oxidase 2-deficiency: from mouse brain to neural differentiation of patient derived iPSC. Redox Biology. 13, 82-93 (2017).
- Wilson, C., Nunez, M. T., González-Billault, C. Contribution of NADPH oxidase to the establishment of hippocampal neuronal polarity in culture. Journal of Cell Science. 128 (16), 2989-2995 (2015).
- Munnamalai, V., et al. Bidirectional interactions between Nox2-type NADPH oxidase and the F-actin cytoskeleton in neuronal growth cones. Journal of Neurochemistry. 130 (4), 526-540 (2014).
- Kishida, K. T., et al. Synaptic plasticity deficits and mild memory impairments in mouse models of chronic granulomatous disease. Molecular and Cellular Biology. 26 (15), 5908-5920 (2006).
- Ravelli, K. G., et al. Nox2-dependent neuroinflammation in an EAE model of multiple sclerosis. Translational Neuroscience. 10 (1), 1-9 (2019).
- Park, L., et al. Nox2-derived radicals contribute to neurovascular and behavioral dysfunction in mice overexpressing the amyloid precursor protein. Proceedings of the National Academy of Sciences. 105 (4), 1347-1352 (2008).
- Schiavone, S., Neri, M., Trabace, L., Turillazzi, E. The NADPH oxidase NOX2 mediates loss of parvalbumin interneurons in traumatic brain injury: Human autoptic immunohistochemical evidence. Scientific Reports. 7 (1), 8752 (2017).
- Gutscher, M., et al. Proximity-based protein thiol oxidation by H2O2-scavenging peroxidases. Journal of Biological Chemistry. 284 (46), 31532-31540 (2009).
- Bilan, D. S., Belousov, V. V. New tools for redox biology: from imaging to manipulation. Free Radical Biology and Medicine. 109, 167-188 (2016).
- Abu-Siniyeh, A., Al-Zyoud, W. Highlights on selected microscopy techniques to study zebrafish developmental biology. Laboratory Animal Research. 36 (1), 12 (2020).
- Sassen, W. A., Koster, R. W. A molecular toolbox for genetic manipulation of zebrafish. Advances in Genomics and Genetics. 5, 151-163 (2015).
- Rosen, J. N., Sweeney, M. F., Mably, J. D. Microinjection of zebrafish embryos to analyze gene function. Journal of Visualized Experiments. (25), e1115 (2009).
- Avdesh, A., et al. Regular care and maintenance of a Zebrafish (Danio rerio) laboratory: An introduction. Journal of Visualized Experiments. (69), e4196 (2012).
- Chen, Z., et al. Primary neuron culture for nerve growth and axon guidance studies in Zebrafish (Danio rerio). PLoS One. 8 (3), 57539 (2013).
- Zhang, L., Leung, Y. F. Microdissection of zebrafish embryonic eye tissues. Journal of Visualized Experiments. (40), e2028 (2010).
- Suter, D. M. Live cell imaging of neuronal growth cone motility and duidance in vitro. Cell Migration: Methods in Molecular Biology. , 65-86 (2011).
- Weaver, C. J., et al. nox2/cybb deficiency affects zebrafish retinotectal connectivity. Journal of Neuroscience. 38 (26), 5854-5871 (2018).
- Morgan, B., Sobotta, M. C., Dick, T. P. Measuring EGSH and H2O2 with roGFP2-based redox probes. Free Radical Biology and Medicine. 51, 1943-1951 (2011).
- Li, Z., et al. Phenylthiourea specifically reduces zebrafish eye size. PLoS One. 7 (6), 40132 (2012).
- Ermakova, Y. G., et al. Red fluorescent genetically encoded indicator for intracellular hydrogen peroxide. Nature Communications. 5, 5222 (2014).
- Oparka, M., et al. Quantifying ROS levels using CM-H2DCFDA and HyPer. Methods. 109, 3-11 (2016).
- Dickinson, B. C., Peltier, J., Stone, D., Schaffer, D. V., Chang, C. J. Nox2 redox signaling maintains essential cell populations in the brain. Nature Chemical Biology. 7 (2), 106-112 (2011).
- Cannon, M. B., Remington, S. J. Redox-sensitive green fluorescent protein: Probes for dynamic intracellular redox responses. A review. Methods in Molecular Biology. 476, 51-65 (2009).
- Meyer, A. J., Dick, T. P. Fluorescent protein-based redox probes. Antioxidants & Redox Signaling. 13 (5), 621-650 (2010).
- Panieri, E., Millia, C., Santoro, M. M. Real-time quantification of subcellular H2O2 and glutathione redox potential in living cardiovascular tissues. Free Radical Biology and Medicine. 109, 189-200 (2017).
- Breus, O., Dickmeis, T. Genetically encoded thiol redox-sensors in the zebrafish model: Lessons for embryonic development and regeneration. Biological Chemistry. , (2020).
- Morgan, B., et al. Real-time monitoring of basal H2O2 levels with peroxiredoxin-based probes. Nature Chemical Biology. 12 (6), 437-443 (2016).
- Terzi, A., Roeder, H., Weaver, C. J., Suter, D. M. Neuronal NADPH oxidase 2 regulates growth cone guidance downstream of slit2/robo2. Developmental Neurobiology. , (2020).
- Bilan, D. S., Belousov, V. V. HyPer family probes: State of the art. Antioxidants & Redox Signaling. 24 (13), 731-751 (2016).
- Ermankova, Y. G., et al. SypHer3s: a genetically encoded fluorescent ratiometric probe with enhanced brightness and an improved dynamic range. Chemistry Communications. 54 (23), 2898-2901 (2018).
- Pak, V. V., et al. Ultrasensitive genetically encoded indicator for hydrogen peroxide identifies roles for the oxidant in cell migration and mitochondrial function. Cell Metabolism. 31 (3), 642-653 (2020).
- Kwan, K. M., et al. The Tol2kit: A multisite gateway-based construction Kit for Tol2 transposon transgenesis constructs. Developmental Dynamics. 236 (11), 3088-3099 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados