Method Article
Análisis de la restricción SAMHD1 por citometría de flujo en células mieloides humanas U937
En este artículo
Resumen
Aquí se describe un método establecido para determinar el grado de restricción del VIH-1 por la proteína inhibidora celular SAMHD1. Las células U937 del linaje mieloide humano se transducen con un vector de expresión SAMHD1 que coexpresa YFP, se diferencian y luego se desafían con VIH-RFP. El nivel de restricción se determina mediante análisis de citometría de flujo.
Resumen
La proteína 1 estéril que contiene el dominio α-motivo/histidina-aspartato (SAMHD1) inhibe la replicación del VIH-1 en células mieloides quiescentes. Las células U937 son ampliamente utilizadas como un sistema celular conveniente para analizar la actividad de SAMHD1 debido a un bajo nivel de expresión de ARN SAMHD1, lo que lleva a una expresión de proteínas endógenas indetectables. Basándose en ensayos similares desarrollados en el laboratorio de Stoye para caracterizar otros factores de restricción retroviral, el laboratorio Bishop desarrolló un ensayo de restricción de dos colores para analizar SAMHD1 en células U937. Las partículas similares al virus de la leucemia murina que expresan SAMHD1, junto con YFP expresada desde un IRES, se utilizan para transducir células U937. Las células se tratan con acetato de miristato de forbol para inducir la diferenciación a un fenotipo quiescente. Después de la diferenciación, las células se infectan con partículas similares al virus VIH-1 que expresan un reportero fluorescente. Después de 48 h, las células se cosechan y se analizan por citometría de flujo. La proporción de células infectadas por el VIH en la población que expresa SAMHD1 se compara con la de las células de control interno que carecen de SAMHD1. Esta comparación revela una relación de restricción. La expresión de SAMHD1 conduce a una reducción de cinco veces en la infección por VIH, lo que corresponde a una relación de restricción de 0,2. Nuestra reciente sustitución de RFP por la GFP original como el gen reportero para la infección por VIH ha facilitado el análisis de citometría de flujo.
Este ensayo se ha utilizado con éxito para caracterizar el efecto de las sustituciones de aminoácidos sobre la restricción de SAMHD1 mediante la transducción con virus que codifican proteínas SAMHD1 alteradas, derivadas de la mutagénesis dirigida al sitio del vector de expresión. Por ejemplo, las sustituciones de sitios catalíticos HD206-7AA muestran un fenotipo de restricción de 1, lo que indica una pérdida de actividad de restricción. Igualmente, se puede determinar la susceptibilidad de diferentes virus probadores. El ensayo se puede adaptar aún más para incorporar el efecto del estado de diferenciación, el estado metabólico y los modificadores de SAMHD1 para comprender mejor la relación entre SAMHD1, el estado metabólico celular y la restricción viral.
Introducción
La proteína estéril que contiene el dominio α-motivo/histidina-aspartato (SAMHD1) impide la replicación del virus de la inmunodeficiencia humana tipo 1 (VIH-1) en células quiescentes del linaje mieloide. SAMHD1 bloquea la replicación a través de su actividad enzimática como una trifosfohidrolasa dNTP, lo que resulta en una disminución de los niveles de dNTP intracelulares. Como resultado, el VIH-1 no puede realizar el proceso de transcripción inversa de manera eficiente. Se ha avanzado mucho en los pocos años transcurridos desde esta observación inicial, particularmente con respecto a la contribución de dominios y aminoácidos específicos a la actividad antiviral de SAMHD1. Estas ideas se han realizado utilizando ensayos bioquímicos, así como sistemas celulares que imitan el entorno mieloide quiescente fisiológicamente relevante. Las células U9371 son ampliamente utilizadas como un sistema de células mieloides conveniente para analizar la actividad de SAMHD1. Esto se debe a la falta de expresión endógena de SAMHD1, que se cree que se debe a los bajos niveles de ARN en comparación con las células que expresan SAMHD1 (un área de investigación en curso). Aquí, el protocolo describe la transducción transitoria de células U937 con partículas similares a virus que coexpresan SAMHD1 y un reportero de proteína fluorescente amarilla (YFP) para examinar el mecanismo de restricción SAMHD1 del VIH-1. Tales ensayos transitorios de citometría de flujo de dos colores para examinar las restricciones retrovirales se desarrollaron por primera vez en el laboratorio de Stoye2 y desde entonces se han adaptado para investigar otros factores de restricción, incluidas las proteínas del motivo tripartito (TRIM)3. Inspirado por estos ensayos, el laboratorio Bishop desarrolló un ensayo de dos colores para analizar la restricción de SAMHD1 en células U937.
El flujo de trabajo para el experimento se muestra en la figura 1. Las partículas similares al virus de la leucemia murina (MLV) que contienen un mensaje bicistrónico que codifica SAMHD1 junto con YFP expresado desde un IRES se utilizan para transducir células U937. Luego, las células se tratan con acetato de miristato de forbol (PMA) para inducir la diferenciación a un fenotipo quiescente. Las células se infectan con partículas similares al virus VIH-1 que expresan proteína fluorescente roja (RFP). Después de 48 h, las células se cosechan y se analizan mediante citometría de flujo de dos colores. Este ensayo también se utiliza con YFP y proteína fluorescente verde (GFP), que requiere menos combinaciones de filtros estándar para el análisis de citometría de flujo. El uso de VIH-RFP permite una compensación más directa y, por lo tanto, el ensayo es más fácilmente accesible para los usuarios de citometría de flujo menos experimentados y se puede lograr con la mayoría de los citómetros.
Durante el análisis, la proporción de células infectadas por el VIH se compara entre las células que expresan SAMHD1 y las que carecen de SAMHD1 dentro del mismo pocillo de células. Esto permite un control interno en el tubo de citometría de pozo/flujo, que es una característica clave. La comparación de los niveles de infección en células transducidas y no transducidas revela una relación de restricción. Una proporción de 1,0 indica que el factor transducido no tiene ningún efecto sobre la infectividad. La expresión de SAMHD1 de tipo salvaje conduce a una reducción de cinco veces en la infección por VIH en este ensayo, lo que corresponde a una relación de restricción de 0,2. Si bien este efecto es modesto en comparación con factores de restricción más clásicos como TRIM5, el efecto es, sin embargo, reproducible y permite la clasificación de expresores SAMHD1 modificados en aquellos que se restringen de manera equivalente a la proteína de tipo salvaje, aquellos que no restringen y aquellos con un fenotipo intermedio.
Este ensayo se ha utilizado con éxito para caracterizar el dominio SAMHD1 y los fenotipos de restricción de los mutantes de aminoácidos mediante la transducción con virus que codifican secuencias SAMHD1 mutantes. Por ejemplo, las sustituciones de sitios catalíticos HD206-7AA no se restringen en este ensayo. Igualmente, se puede determinar la susceptibilidad de diferentes virus probadores. Por ejemplo, los mutantes de la transcriptasa inversa del VIH-1 son más susceptibles a la restricción mediada por SAMHD1 (por ejemplo, 151V4). Este protocolo describe los detalles de la producción de partículas similares a virus (VLP), la transducción de U937 con vectores de expresión SAMHD1, la infección con VIH portadora de un reportero fluorescente y el posterior análisis por citometría de flujo. En este artículo se describe qué datos esperar y cómo evitar resultados subóptimos. Finalmente, se describen los usos alternativos para el ensayo, además de examinar diferentes variantes de SAMHD1 para comprender la interacción entre SAMHD1, los niveles de dNTP y la infección viral más a fondo. Dado el papel de SAMHD1 en el centro del metabolismo celular, con vínculos adicionales con el cáncer, esta sigue siendo un área de intenso interés.
Protocolo
Este protocolo no contiene ningún estudio con animales o participantes humanos realizado por ninguno de los autores. Todos los pasos de este protocolo se realizaron siguiendo las directrices y códigos de prácticas de las instituciones donde se llevaron a cabo.
1. Transfección de células 293T para la producción de VLP (tanto vectores de expresión SAMHD1 como partículas de VIH testers)
NOTA: Los contribuyentes más significativos a la producción exitosa de partículas de virus son la salud de las células 293T productoras, la confluencia en la transfección y el momento de la cosecha. Los laboratorios generalmente tendrán sus reactivos de transfección preferidos y el protocolo para la producción de partículas retrovirales. El siguiente protocolo produce partículas infecciosas de buena calidad, pero protocolos equivalentes también serían adecuados para esta aplicación. Para mantener células 293T de buena calidad, pase al menos tres veces por semana para mantener una tasa de crecimiento constante. Las células deben sembrarse en la fase logarítmica de crecimiento para una transfección eficiente.
- Día 1: Siembre el número requerido de platos de 10 cm con una división de 1/4 de un plato casi confluente de células 293T para producir alrededor del 60% de confluencia al día siguiente (aproximadamente 5 x 105 células / ml). Es posible que sea necesario sembrar varias densidades para lograr una densidad adecuada para la transfección al día siguiente cuando se trabaja con un nuevo material celular.
- Día 2 (mañana): Verifique la confluencia de aproximadamente el 60% mediante microscopía y reemplace suavemente los medios en las células con 10 ml de medio de águila modificado (DMEM) fresco de Dulbecco.
- Día 2 (tarde, al menos 4 h después del cambio de medios): Efecto para producción VLP.
- Constituir la mezcla de dilución de ADN que contiene 3 μg de plásmido expresor gag-pol , plásmido reportero impulsado por repetición terminal larga (LTR) y plásmido expresor de la proteína G del virus de la estomatitis vesicular (VSV-G) (total 9 μg, ver NOTA) en 600 μL de DMEM libre de suero. Vórtice y centrífuga brevemente (15 s, >500 x g).
- Añadir 20 μL de reactivo de transfección (ver Tabla de materiales), vórtice (no centrifugar) e incubar a 20 °C durante 15-20 min. Agregue la mezcla de transfección gota a gota al plato, agite suavemente para mezclar y regrese a la incubadora.
NOTA: Para MLV VLP que expresa SAMHD1 use KB4 5,6 (plásmido expresor gag-pol), pLGatewaySAMHD1IeYFP1 (plásmido reportero impulsado por LTR) y pczVSV-G7. Para HIV-RFP VLP, use p8.91 8,9 (plásmido expresor gag-pol), SCRPSY10 (plásmido reportero impulsado por LTR) y pczVSV-G. Las alternativas se discuten en la Tabla de materiales. Es posible que sea necesario optimizar las proporciones de plásmidos para diferentes sistemas de plásmido/transfección.
- Día 3 (tarde en la mañana): Retire el medio cuidadosamente de las células mediante pipeteo, lave con 5 ml de DMEM fresco y reemplácelo con 5 ml de DMEM fresco.
- Día 4 (mañana): Cosechar el virus recogiendo el sobrenadante de las células y reemplazarlo con 5 ml de DMEM fresco. Pasar el sobrenadante que contiene VLP a través de un filtro de 0,45 nm, alícuota y transferir a -70 °C para su almacenamiento. Asegúrese de que las alícuotas sean de un tamaño adecuado para los experimentos planificados (250 μL como sugerencia), ya que los ciclos repetidos de congelación-descongelación darán lugar a una pérdida del título viral.
- Día 5 (mañana): Cosechar el virus como en 1.5 pero desechar las placas después de recoger el sobrenadante.
2. Titulación de nuevas VLP antes de su uso
- Día 1: Semilla 293T a 2 x 105 células por pocillo de una placa de 24 pocillos - 4 pocillos por virus a valorar, incluyendo un virus de infectividad conocida para cada columna vertebral. Optimice la densidad de siembra para un stock celular determinado.
- Día 2: Observe la placa para verificar la confluencia (idealmente ~ 70%). Descongele el sobrenadante que contiene el virus de los pasos 1.5 y 1.6. Agregue 0, 10, 25 o 100 μL a cada pocillo.
- Día 4 (Mañana): Cosecha de las células para su análisis por citometría de flujo.
- Retire el medio mediante un pipeteo o aspiración cuidadosos. Lave las células suavemente con 250 μL de solución salina tamponada con fosfato (PBS). Retire las células de la placa pipeteando hacia arriba y hacia abajo con 250 μL de PBS y transfiéralas a un tubo de microcentrífuga. Añadir 250 μL de paraformaldehído al 4% (llevando la concentración final al 2%).
PRECAUCIÓN: El paraformaldehído es tóxico. No debe mezclarse con desinfectantes a base de cloro. - Granular las células por centrifugación a 500 x g durante 5 min, desechar el sobrenadante y resuspender el pellet celular en 200 μL de PBS o tampón de citometría de flujo alternativo. Analice la RFP o el YFP, según corresponda, mediante citometría de flujo. Normalizar la infectividad de un stock de virus determinado con los valores de un stock de infectividad conocida.
NOTA: VLP también se puede normalizar mediante el ensayo inmunoabsorbente ligado a enzimas p24 (ELISA); sin embargo, esto no es necesariamente un buen indicador de la infectividad de VLP, especialmente cuando las existencias de VLP se preparan en diferentes transfecciones. Los métodos publicados de dosis infecciosa mediana de cultivo tisular o multiplicidad de infección (TCID50/MOI)11 también pueden proporcionar cuantificación relativa, aunque estos no están bien establecidos para MLV. La fluorescencia relativa proporciona una alternativa rentable a los métodos comerciales ELISA de transcriptasa inversa.
- Retire el medio mediante un pipeteo o aspiración cuidadosos. Lave las células suavemente con 250 μL de solución salina tamponada con fosfato (PBS). Retire las células de la placa pipeteando hacia arriba y hacia abajo con 250 μL de PBS y transfiéralas a un tubo de microcentrífuga. Añadir 250 μL de paraformaldehído al 4% (llevando la concentración final al 2%).
3. Transducción de U937 con VLP expresando SAMHD1 y YFP
NOTA: Los pasos 3-6 constituyen el experimento de restricción. Los días están contados para facilitar la planificación.
- Día 1 (tarde): Descongelar VLP para transducción incluyendo MLV-Wild-type SAMHD1-YFP (control positivo), MLV-SAMHD1(HD206-7AA)-YFP (control negativo) y variante MLV-SAMHD1-YFP (muestras experimentales). Diluir la VLP normalizada a un volumen final de 500 μL con medios Roswell Park Memorial Institute 1640 (RPMI) en un tubo de microfuga, agregar 0,5 μL de polibrono (10 mg/ml) y mezclar con el movimiento.
- Si la VLP no se puede ajustar de antemano, use 100 μL en primera instancia. Utilice una proporción máxima de 1:1 de VLP a RPMI para limitar la toxicidad inducida por VLP. Apunte a aproximadamente un 30% de transducción.
- Alícuota 5 x 105 células U937 en un tubo de microfuga estéril para cada condición de transducción. Granular por centrifugación a 500 x g durante 5 min, y retirar el sobrenadante (incluir dos tubos adicionales como controles no transducidos para citometría de flujo). Resuspender el pellet celular en 500 μL de VLP diluido (o RPMI para controles no transducidos) y transferir a un pocillo de una placa de 24 pocillos.
- Espinocular en una centrífuga de sobremesa a 800 x g durante 90 min a 20 °C. Añadir 1 mL de RPMI a 37 °C al pocillo y dejar recuperar en una incubadora a 37 °C durante 3 días.
4. Diferenciación del U937 transducido
- Día 4 (mañana): Vuelva a suspender las células pipeteando suavemente y transfiera 350 μL a cada uno de los 4 pocillos de una placa de 12 pocillos. Añadir 700 μL de 37 °C RPMI que contiene 150 nM PMA a cada pocillo dando una concentración final de 100 nM y dejar diferenciar durante 3 días.
NOTA: El PMA se compra en forma de polvo y debe resuspenderse a 1 mM en DMSO y almacenarse en la oscuridad. Se debe preparar un material de trabajo de 100 μM diluyendo 1/10 del material de 1 mM con DMSO.
5. Infección de U937 diferenciado, que expresa SAMHD1 con VIH-RFP
- Día 7: Observar las células por microscopía. Asegúrese de que las células sean adherentes. Aspire los medios de todos los pocillos, agregue 1 ml de RPMI al pocillo 1 de cada conjunto de 4, lo que permite un control no transducido y no infectado, así como controles de un solo color. Agregue 0.5 ml de RPMI que contiene aproximadamente 10 μL de VIH-1-RFP (aproximadamente 10 ng p24 como guía) a todos los demás pocillos. Apunte a aproximadamente el 50% de infección.
- Día 8 (mañana): Agregue 0.5 mL de RPMI a cada uno de los pocillos infectados.
6. Análisis de citometría de flujo
NOTA: Es una buena práctica incluir una tinción de muertos vivos en las tuberías de citometría de flujo. Sin embargo, si las células U937 son de buena calidad, este paso puede omitirse sin consecuencias negativas para el análisis posterior. El pipeteo suave de las células tripsinizadas debería resultar fácilmente en una suspensión de una sola célula.
- Día 10 (mañana): Aspirar los medios de los pozos y lavar una vez con PBS. Añadir 300 μL de enzima de disociación celular (o tripsina) durante 5-10 min. Agregue 300 μL adicionales de PBS, resuspenda completamente asegurándose de que todas las células se hayan desprendido de la placa y transfiera a tubos de citometría de flujo.
- Añadir 300 μL de paraformaldehído al 4% (llevando la concentración final al 2%). Granular las células por centrifugación a 500 x g durante 5 min, desechar el sobrenadante y resuspender el pellet celular en 200 μL de PBS o tampón de citometría de flujo alternativo. Analizar por citometría de flujo.
NOTA: Los pasos a continuación son para un analizador Fortessa, pero se puede lograr un análisis equivalente utilizando cualquier analizador capaz de leer RFP y fluorescencia YFP. Consulte al soporte de citometría de flujo local u otro investigador experimentado antes de configurar cualquier análisis de citometría de flujo. Cuando es necesario examinar muchas muestras al mismo tiempo, puede ser apropiado un muestreador de alto rendimiento. En este caso, se recomienda un mayor número y volumen de celda.
7. Análisis de citometría de flujo
- Encienda el citómetro 10 minutos antes de lo necesario y encienda la computadora. Verifique que los residuos estén vacíos y que el tanque de la vaina esté lleno.
- Inicie sesión en la máquina y abra el software de análisis.
- Mueva el brazo hacia un lado, retire el tubo de agua, ajuste el caudal a Alto y presione Prime. Espere a que se apague la luz y repita tres veces más.
- Vuelva a poner el agua y corra a fuego alto durante 3 minutos y luego cambie a Bajo y presione Standby hasta proceder a la adquisición.
- Mientras tanto, configure el experimento dentro del software:
- Seleccione Experimento/Nuevo experimento (nombre según las directrices locales). En el inspector, elimine los fluorocromos no necesarios, dejando Blue Laser 530/30 y Yellow Laser 610/20 o las combinaciones recomendadas de láser y filtro para YFP y RFP para la máquina.
- Haga clic derecho en el experimento y elija Configuración del citómetro / Configuración de la aplicación / Crear hoja de trabajo. En la hoja de cálculo, cree una ranura de puntos de área de dispersión directa (FSC-A)/área de dispersión lateral (FSC-A) e histograma YFP, histograma RFP y ranura de punto GFP/YFP haciendo clic en el icono correspondiente y cambiando los ejes haciendo clic en la etiqueta del eje.
- Haga clic derecho de nuevo en Experimento y agregue Nuevo espécimen. Cambie el nombre según corresponda. Abra la muestra haciendo clic en el signo más para revelar un nuevo tubo.
- Abra el panel de adquisición desde View, cargue el control no infectado y no transducido y pulse Adquirir. Ajuste los voltajes FSC y SSC en el tablero para colocar las celdas en el cuadrante inferior izquierdo (sin celdas en los ejes). Puerta alrededor de esta población P1. Haga clic derecho en otros gráficos y seleccione mostrar P1 para eliminar los residuos del análisis posterior.
- Cargue el control de un solo color para YFP (solo SAMHD1 de tipo salvaje) y adquiera. Ajuste el voltaje YFP en el tablero para que las celdas más fluorescentes estén dentro del rango del detector (no en el borde del eje). Repita el procedimiento para el control RFP de un solo color.
- Ejecute la compensación automática utilizando los controles no transducidos, no infectados y de un solo color.
- Seleccione Experimento > Configuración de compensación > Crear controles de compensación en el menú para cambiar a una hoja de cálculo normal. Seleccione la hoja de trabajo normal sin teñir, presione Ejecutar y adquiera para el control sin teñir.
- Dibuje una puerta alrededor de las celdas intactas (P1) y registre. Retire el tubo y ponga la máquina en modo de espera. Haga clic derecho en P1, haga clic en Aplicar a todos los controles de compensación.
- Cambie a la hoja de cálculo normal de RFP y presione Ejecutar. Grabe el control RFP de un solo color y ponga la máquina en modo de espera. El software debe seleccionar automáticamente la población positiva.
- Repita el procedimiento para el control de color único YFP. Seleccione Experimento > Configuración de compensación > Calcular compensación > Enlace y guardar.
- Cambie a la hoja de cálculo global. Adquirir las células transducidas con SAMHD1 de tipo salvaje, infectadas con VIH-RFP y comprobar que se pueden ver cuatro poblaciones distintas en las esquinas de los cuadrantes que están alineadas vertical y horizontalmente para YFP y RFP, respectivamente. Retire el tubo y pulse Standby.
- Vuelva a cargar la muestra no transducida y no infectada, pulse Ejecutar y Registrar 30.000 eventos con P1 como puerta de parada. Repita el procedimiento para las muestras restantes (incluidos otros controles). Cambie el nombre de las muestras mientras se ejecuta o post hoc. Una vez exportados, no se puede cambiar el nombre de los archivos.
- Exporte los datos como archivos .fcs y limpie los fluidos de acuerdo con las instrucciones locales.
8. Análisis de datos
NOTA: Los pasos siguientes corresponden al análisis en FlowJo v10; Sin embargo, pasos equivalentes se pueden realizar fácilmente en otro software de análisis.
- Abra el software y arrastre todos los archivos .fcs al tablero. Seleccione los controles de compensación y arrástrelos a la subcarpeta de compensación. Abra el archivo para el tubo no transducido y no infectado haciendo doble clic, lo que mostrará la gráfica FSC-A / SSC-A.
- Seleccione la herramienta poligonal y la puerta en la población de celdas intactas, excluyendo los residuos en la esquina inferior izquierda. Asigne a estas celdas el nombre. Arrastre esta puerta a toda la población de tubos en la barra Todas las muestras . Desplácese por cada muestra individual usando los botones de flecha horizontal para asegurarse de que la puerta sea apropiada para cada tubo.
- Haga doble clic en la población de celdas para la muestra no transducida y no infectada y ajuste los ejes a FSC-Height (H) vs FSC-Area (A). Seleccione la única población grande utilizando una puerta rectangular para excluir dobletes. El software sugerirá automáticamente nombrar estas celdas individuales.
- Arrastre esta puerta a todas las poblaciones de Cells y, de nuevo, compruebe que la puerta de acceso es adecuada para todas las muestras. Seleccione la población de células individuales para la muestra no infectada y no transducida, cambie los ejes a láser azul compensado (y , YFP) frente al láser amarillo compensado (x, RFP). Pulse T en el eje y seleccione Escala biexponencial para x e y.
- Seleccione la herramienta Cuadrante y haga clic en el extremo superior derecho de la población de celdas negativas. Aplique esta activación preliminar a todas las poblaciones de células individuales arrastrando a la barra Todas las muestras . Cuando las puertas del cuadrante no separan las poblaciones satisfactoriamente, puede ser necesario bloquear cuadrantes individuales utilizando la herramienta polígono, como en la Figura 2.
- Desplácese hasta la muestra de solo SAMHD1 de tipo salvaje (solo YFP) y verifique que la activación entre la no transducida (YFP negativa) y la positiva YFP sea correcta. Puede ser útil cambiar a la vista de contorno para discriminar las células YFP negativas de las tenues. Si las celdas YFP + ve superiores están ampliamente extendidas, ajuste la puerta vertical superior a la derecha.
- Desplácese por todas las muestras y verifique la puerta con respecto a la positividad de YFP. Utilice la mejor división entre YFP negativo y positivo que sea válida para todas las muestras.
NOTA: La compensación se habrá calculado en función del nivel de expresión YFP SAMHD1 de tipo salvaje. Si los niveles de infección son muy diferentes entre las diferentes células transducidas SAMHD1-YFP, es posible que sea necesario ajustar la compensación. Por lo tanto, es preferible que YFP VLP se normalice antes de su uso. - Desplácese hasta el tubo infectado con VIH-RFP no transducido y verifique si la puerta de acceso entre RFP negativa no infectada y RFP positiva infectada es correcta. Ajuste según corresponda, utilizando la vista de contorno si es necesario y desplácese por las muestras restantes para verificar que la puerta de acceso sea válida para todas las muestras.
- Cada muestra requiere dobles negativos (Q4: abajo a la izquierda, sin transducir, sin infectar), YFP positivo (Q1: arriba a la izquierda, SAMHD1 expresando, no infectado), RFP positivo (Q3: abajo a la derecha, sin transducir, infectado por VIH) y doble positivo (Q2: arriba a la derecha, SAMHD1 que expresa VIH infectado). Abra el editor de tablas haciendo clic en el icono y arrastre las cuatro puertas del cuadrante al tablero. Seleccione Exportar Excel y haga clic en Crear tabla. Guarde la hoja de cálculo generada de acuerdo con las convenciones de nomenclatura de archivos locales.
- Para exportar representaciones de parcelas y estrategias de compuerta, haga clic en el icono Editor de diseño . Seleccione cada población y arrástrela al editor con los ejes, el etiquetado y el espaciado necesarios.
- Para crear un diseño con los mismos gráficos que se muestran para todas las muestras, seleccione una columna y pulse Lote. Presione Escalar a ancho y luego Evitar saltos de página. Guardar en el formato de archivo requerido.
- En la hoja de cálculo exportada, calcule la relación de restricción generando columnas de porcentaje RFP de YFP-ves (RFP+ve / (dobles negativos + RFP+ve)) y porcentaje de RFP de YFP+ves (doble positivo / (doble positivo + YFP+ve)) y luego una columna final de (%RFP de YFP+ve / %RFP de YFP-ve), consulte la Figura 1. Promedie los datos replicados para cada construcción SAMHD1 y calcule si los valores de restricción para las variantes SAMHD1 probadas son significativamente diferentes de los de tipo salvaje y / o 206-7AA utilizando el software de análisis de datos adecuado.
Resultados
Los resultados del análisis anterior deberían arrojar una relación de restricción de 0.25 o menor para el control positivo SAMHD1 de tipo salvaje y 1.0 para el control negativo. Si estos dos controles de calidad son válidos, considere la significación estadística de los resultados. Las variantes de SAMHD1 que no muestran diferencias significativas con respecto al tipo salvaje, por lo tanto, llevan sustituciones que no afectan la restricción de SAMHD1 en este contexto. Aquellos significativamente diferentes del tipo salvaje muestran una restricción deteriorada. Si estos no son significativamente diferentes del control negativo, entonces carecen de la capacidad de restringir en este contexto (Figura 4 paneles izquierdos).
Si el valor de restricción SAMHD1 de tipo salvaje es mayor que 0,3, los resultados de los virus de prueba pueden ser indicativos, pero no se puede confiar en ellos. La restricción ineficaz por proteína de tipo salvaje puede resultar del uso de células U937 demasiado temprano después de la recuperación de la reconstitución (dentro de 2 semanas), o cuando son demasiado viejas (>2-3 meses). Es posible que sea necesario determinar empíricamente estos parámetros para un stock celular dado. Por lo general, cuanto más bajo es el paso, más confiablemente se diferencian las células y, por lo tanto, proporcionan el entorno apropiado para la restricción SAMHD1. El nivel de infección inadecuado con SAMHD1-YFP o VIH-RFP también puede dar lugar a dificultades tanto con la compensación como con la determinación posterior de la relación de restricción. Un ejemplo se ilustra en los paneles de la derecha de la Figura 4.
Si el control negativo se desvía de 1.0 (fuera del rango de 0.9-1.2), esto puede indicar un problema con el análisis, ya sea la proporción de células infectadas, la estrategia de activación o la salud de las células que afectan el ensayo. Consulte las NOTAS anteriores.
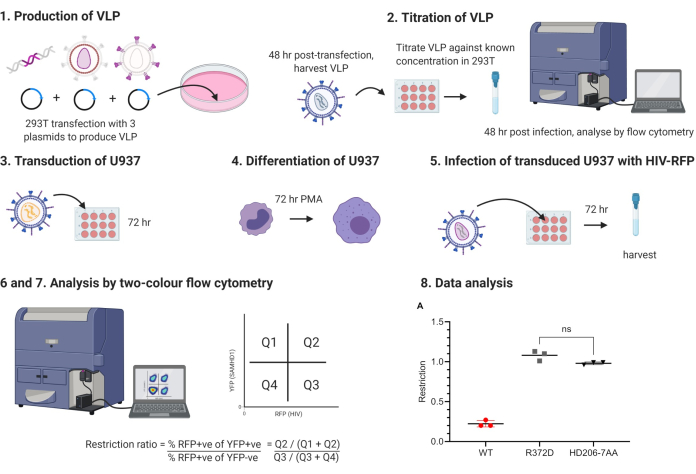
Figura 1: Protocolo de esquema esquemático. VLP, partículas similares a virus, PMA, acetato de forbol miristato. Las etapas numeradas corresponden a etapas en protocolo. Figura producida utilizando BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
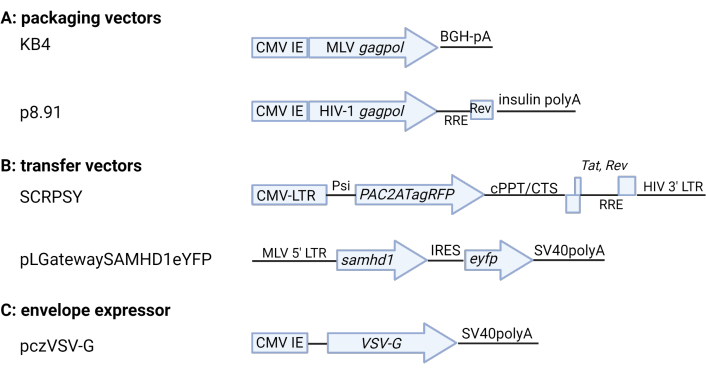
Figura 2: Esquema de plásmidos retrovirales . (A) Vectores de empaquetamiento (B) Vectores de transferencia (C) Expresor de envoltura VSV-G. Se muestran los elementos clave de codificación y regulación. Para obtener más detalles, consulte la Tabla de materiales. CMV IE: promotor inmediato-temprano del citomegalovirus, BGH: hormona de crecimiento bovino, pA: polyA, RRE: elemento de respuesta Rev, CMV-LTR: promotor compuesto de LTR CMV-VIH-1, Psi: señal de empaquetamiento del VIH-1, cPPT/CTS: tracto central de polipurinas/secuencia de terminación central, SV40: virus vacuolante simio 40. Figura producida utilizando BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
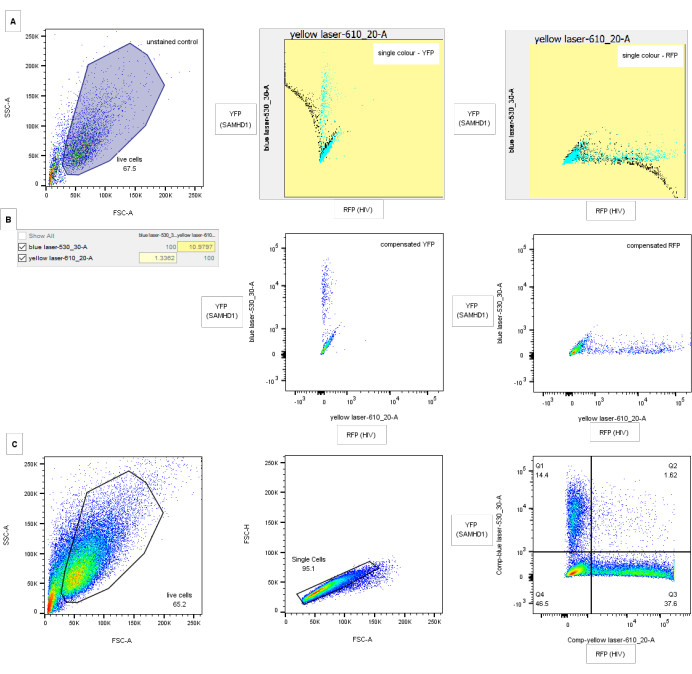
Figura 3: Estrategia de gating para el análisis de citometría de flujo. (A) Controles de compensación: el panel izquierdo muestra la activación FSC-A/SSC-A en las células intactas para el control no transducido y no infectado. Los paneles central y derecho muestran capturas de pantalla de controles de compensación de un solo color para YFP (medio) y RFP (derecha) con datos no compensados en negro y compensados en azul. (B) Matriz de compensación correspondiente y parcelas para los controles de compensación anteriores. (C) Estrategia de acceso. Los desechos se eliminan mediante el análisis de todas las células mediante FSC-A / SSC-A (panel izquierdo). Los dobletes se excluyen mediante la puerta en altura FSC versus área (panel central). El panel derecho muestra un ejemplo de restricción del VIH-1 por SAMHD1 de tipo salvaje. Los ejes muestran fluorescencia láser azul y amarilla compensada correspondiente a las células YFP (SAMHD1) y RFP (VIH) positivas, respectivamente. Las puertas de cuadrante se dibujan a través de la comparación de controles negativos y de un solo color para RFP e YFP. Los números indican el porcentaje de la población parental. Los valores de compensación para YFP con GFP serán mucho más altos, pero es posible discriminar utilizando conjuntos de filtros apropiados. Haga clic aquí para ver una versión más grande de esta figura.
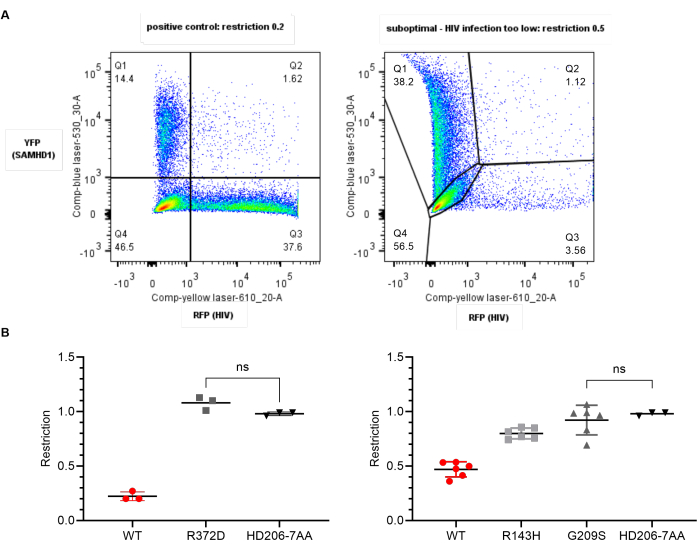
Figura 4: Resultados esperados: datos ideales versus subóptimos. (A) Gráficos representativos de YFP/RFP para datos óptimos (izquierda) y subóptimos (derecha). Los números indican el porcentaje de la población parental. En el panel derecho, la infección por el VIH es demasiado baja, lo que crea dificultades con la compensación y la contratación. La relación de restricción es más alta de lo esperado en 0.5. (B) Gráficos de la relación de restricción para variantes de SAMHD1 con respecto al tipo salvaje (WT, rojo) o el control negativo (HD206-7AA, negro) generado utilizando software estadístico. Cada punto representa un valor de replicación. Se muestran la media y la desviación estándar. Las pruebas t pareadas entre cada grupo fueron significativas en todos los casos esperados donde se mostraron. Izquierda: Datos ideales - WT muestra la restricción esperada de aproximadamente 0.2, negativa muestra 1.0. La variante R372D (gris) es significativamente diferente de WT pero no significativa del control negativo y, por lo tanto, ha perdido la capacidad de restringir. Derecha: Datos subóptimos. Aquí, el negativo se comporta como se esperaba, pero en las seis réplicas el WT solo muestra una relación de restricción de 0.5, debido a la baja tasa de infección. La baja varianza dentro de los grupos significa que R143H muestra un fenotipo intermedio estadísticamente diferente del WT y el negativo, mientras que G209S no restringe; Sin embargo, esto debe repetirse con células frescas ya que el control positivo no ha dado la relación de restricción esperada. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Como se discutió en las notas anteriores, los aspectos críticos del protocolo se centran en retener el fenotipo celular correcto para la producción de VLP y para crear el entorno apropiado para observar la restricción de SAMHD1. En primer lugar, el análisis preciso de la restricción por citometría de flujo de dos colores se basa en lograr proporciones apropiadas de células transducidas e infectadas para que haya suficientes células en cada área de la gráfica para el análisis. Como tal, el investigador debe aspirar a un MOI de menos de 1 (ver Protocolo). Las tasas de transducción e infección que son demasiado altas o demasiado bajas conducen a problemas con el análisis debido a la falta de células no infectadas o doblemente infectadas. Del mismo modo, una tasa de transducción similar para los vectores SAMHD1 que se compararán es clave para permitir que se aplique la misma matriz de compensación y compuerta a todo el experimento, aumentando la confianza en el análisis posterior.
En segundo lugar, la edad de las células U937 utilizadas es un factor clave para diferenciarse con éxito. La diferenciación exitosa puede ser monitoreada a través de la observación de la adherencia, una acidificación reducida de los medios a lo largo del tiempo después de la diferenciación y una restricción adecuada por el control positivo de tipo salvaje en el ensayo. La diferenciación de hasta 5 días ha sido exitosa.
Una de las ventajas clave de este ensayo es su flexibilidad. Una variedad de versiones modificadas de SAMHD1 (dominios, aminoácidos, secuencias de especies) se pueden probar a través de la simple mutagénesis dirigida al sitio del vector o la clonación. Igualmente, se pueden explorar variantes del virus tester. El ensayo se ha utilizado previamente para demostrar que los mutantes de la transcriptasa inversa del VIH-1 con capacidad reducida para unirse a dNTP son más sensibles a la restricción SAMHD1. El mismo principio se puede utilizar para probar la restricción de otros virus por SAMHD1. Además, se pueden utilizar condiciones de diferenciación variables o manipulación artificial de las concentraciones intracelulares de dNTP para investigar aún más la interacción entre las condiciones intracelulares y la actividad de SAMHD1, un área de investigación activa en el laboratorio de Bishop. También se ha descrito la interacción de SAMHD1 con varios inhibidores nucleósidos de la transcriptasa inversa, y se ha demostrado el efecto de SAMHD1 utilizando este ensayo modificado para su uso en células primarias12,13,14,15.
La adaptación del protocolo para su uso en células primarias supera la limitación planteada por el examen de fenotipos metabólicos en células transformadas. Sin embargo, el formato existente permite un protocolo conveniente y menos desafiante técnicamente para el análisis de alto rendimiento, especialmente con el uso de un citómetro de flujo con un muestreador de alto rendimiento. Al adaptar el protocolo, se debe considerar la susceptibilidad de las células no transducidas internamente a los efectos espectadores (por ejemplo, señalización inmune).
Al igual que con muchos aspectos de la biología celular, es esencial que tales ensayos se utilicen como parte de un enfoque holístico para comprender un fenómeno determinado. El uso paralelo de ensayos bioquímicos para la actividad SAMHD116, la cuantificación de grupos intracelulares de dNTP y la observación de fenotipos mutantes SAMHD1 en humanos, ex vivo y en modelos animales (llevados a cabo por laboratorios de todo el mundo, algunos descritos en este número) son clave para la comprensión evolutiva de esta proteína compleja.
Divulgaciones
A los efectos del acceso abierto, los autores han aplicado una licencia de derechos de autor pública CC BY a cualquier versión de manuscrito aceptado por el autor que surja de esta presentación. Las opiniones expresadas son las del autor (es) y no necesariamente las del NIHR o el Departamento de Salud y Asistencia Social.
Agradecimientos
Este trabajo fue apoyado por el Instituto Francis Crick, que recibe su financiación principal de Cancer Research UK (FC001042 y FC001162), el Consejo de Investigación Médica del Reino Unido (FC001042 y FC001162) y el Wellcome Trust (FC001042 y FC001162) y por un Premio Wellcome Trust Investigator a JPS (108012 / Z / 15 / Z). El trabajo en la Universidad de Cambridge fue cofinanciado por el NIHR Cambridge Biomedical Research Centre, The Evelyn Trust (16/21), The Rosetrees Trust (M590) y el Consejo de Investigación Médica del Reino Unido (MR / S009752 / 1). Este último premio financiado por el Reino Unido forma parte del programa EDCTP2 apoyado por la Unión Europea.
Materiales
| Name | Company | Catalog Number | Comments |
| 293T cells | ATCC | CRL-3216 | |
| 0.45 µm syringe filters | Sartorius | FCT122 | |
| 10 cm dishes | Corning | 430167 | virus preps can be scaled up through transfection in higher capacity plates/flasks - see transfection reagent guidance |
| 12 well plates | Greiner | 665180 | |
| 24 well plates | Greiner | 662160 | |
| BD LSRFortessa cell analyzer | BD Bioscience | 649225 | alternative analysers can be used as long as they can read RFP and YFP fluorescence |
| DMEM | Sigma | D6429 | ATCC recommends Dulbecco's Modified Eagle's Medium (DMEM) is modified to contain 4 mM L-glutamine, 4500 mg/L glucose, 1 mM sodium pyruvate, and 1500 mg/L sodium bicarbonate. |
| DMSO | Fisher | BP-231-100 | |
| fetal bovine serum | Gibco | 10500064 | |
| Flowjo | BD Bioscience | - | alternative analysis software can be used |
| light microscope | - | - | Any bright field light microscope |
| p8.91 | - | - | HIV GagPol expressor (PMID: 8602510) |
| paraformaldehyde (4 % in PBS) | Alfa Aesar | J61889 | |
| PBS | Sigma | D8537 | |
| Penicillin-Streptomycin (10,000 U/mL) | ThermoFisher | 15140122 | |
| phorbol 12-myristate 13-acetate (PMA) | Sigma | P8139 | |
| polybrene | Sigma | TR-1003 | |
| pKB4 | - | - | Murine leukaemia Virus GagPol expressor (PMID: 23035841) |
| pLGatewaySAMHD1eYFP | - | - | MLV packaging plasmid encoding wild-type human sequence. Details and cloning procedures Arnold et al 2015 (Ref 1). |
| pLGatewaySAMHD1HD206-7AAeY FP | - | - | As above, encodes amino acid substitutions at crucial active sites residues |
| Prism | Graphpad | - | https://www.graphpad.com/scientific-software/prism/ Other data representation software are also suitable |
| pVSV-G | - | - | VSV-G expressor (https://www.addgene.org/138479/), alternative: pMD2.G (https://www.addgene.org/12259/) |
| RPMI | ThermoFisher | 21875034 | |
| screw-cap tubes | StarLab | E1415-2231 | |
| SCRPSY | - | - | HIV packaging plasmid encoding RFP, Sam Wilson, https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5026698/ |
| stripettes 10 mL | Corning | 10084450 | |
| stripettes 5 mL | Corning | 10420201 | |
| TrypLE express | Gibco | 12604021 | Trypsin will also be suitable |
| Turbofect | ThermoFisher | R0531 | alternative transfection reagents will also work well |
| U937 cells | ATCC | CRL-1593.2 |
Referencias
- Sundström, C., Nilsson, K. Establishment and characterization of a human histiocytic lymphoma cell line (U-937). International Journal of Cancer. 17 (5), 565-577 (1976).
- Bock, M., Bishop, K. N., Towers, G., Stoye, J. P. Use of a transient assay for studying the genetic determinants of Fv1 restriction. Journal of Virology. 74 (16), 7422-7430 (2000).
- Yap, M. W., Nisole, S., Lynch, C., Stoye, J. P. Trim5alpha protein restricts both HIV-1 and murine leukemia virus. Proceedings of the National Academy of Sciences of the United States of America. 101 (29), 10786-10791 (2004).
- Arnold, L. H., et al. Phospho-dependent regulation of SAMHD1 oligomerisation couples catalysis and restriction. PLoS Pathogens. 11 (10), 1005194 (2015).
- Groom, H. C. T., et al. Absence of xenotropic murine leukaemia virus-related virus in UK patients with chronic fatigue syndrome. Retrovirology. 7, 10 (2010).
- Mothes, W., Boerger, A. L., Narayan, S., Cunningham, J. M., Young, J. A. Retroviral entry mediated by receptor priming and low pH triggering of an envelope glycoprotein. Cell. 103 (4), 679-689 (2000).
- Pietschmann, T., et al. Foamy virus capsids require the cognate envelope protein for particle export. Journal of Virology. 73 (4), 2613-2621 (1999).
- Naldini, L., et al. In vivo gene delivery and stable transduction of nondividing cells by a lentiviral vector. Science. 272 (5259), 263-267 (1996).
- Bainbridge, J. W., et al. In vivo gene transfer to the mouse eye using an HIV-based lentiviral vector; efficient long-term transduction of corneal endothelium and retinal pigment epithelium. Gene Therapy. 8 (21), 1665-1668 (2001).
- Kane, M., et al. Identification of interferon-stimulated genes with antiretroviral activity. Cell Host and Microbe. 20 (3), 392-405 (2016).
- Gao, Y., et al. Calculating HIV-1 infectious titre using a virtual TCID50 method. Methods in Molecular Biology. 485, 27-35 (2009).
- Ordonez, P., et al. SAMHD1 enhances nucleoside-analogue efficacy against HIV-1 in myeloid cells. Scientific Reports. 7, 42824 (2017).
- Ballana, E., et al. SAMHD1 specifically affects the antiviral potency of thymidine analog HIV reverse transcriptase inhibitors. Antimicrobial Agents and Chemotherapy. 58 (8), 4804-4813 (2014).
- Huber, A. D., et al. SAMHD1 has differential impact on the efficacies of HIV nucleoside reverse transcriptase inhibitors. Antimicrobial Agents and Chemotherapy. 58 (8), 4915-4919 (2014).
- Amie, S. M., et al. Anti-HIV host factor SAMHD1 regulates viral sensitivity to nucleoside reverse transcriptase inhibitors via modulation of cellular deoxyribonucleoside triphosphate (dNTP) levels. The Journal of Biological Chemistry. 288 (28), 20683-20691 (2013).
- Arnold, L. H., Kunzelmann, S., Webb, M. R., Taylor, I. A. A continuous enzyme-coupled assay for triphosphohydrolase activity of HIV-1 restriction factor SAMHD1. Antimicrobial Agents and Chemotherapy. 59 (1), 186-192 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados