Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Experimentos de RMN de alta presión para detectar estados conformacionales de baja altitud de proteínas
En este artículo
Resumen
Proporcionamos una descripción detallada de los pasos necesarios para ensamblar una celda de alta presión, configurar y registrar experimentos de RMN de alta presión y, finalmente, analizar tanto la intensidad máxima como los cambios de cambio químico bajo presión. Estos experimentos pueden proporcionar información valiosa sobre las vías de plegamiento y la estabilidad estructural de las proteínas.
Resumen
La alta presión es un método de perturbación bien conocido que se puede utilizar para desestabilizar las proteínas globulares y disociar los complejos de proteínas de manera reversible. La presión hidrostática impulsa los equilibrios termodinámicos hacia el estado (s) con el volumen molar más bajo. El aumento de la presión ofrece, por lo tanto, la oportunidad de ajustar finamente la estabilidad de las proteínas globulares y los equilibrios de oligomerización de los complejos de proteínas. Los experimentos de RMN de alta presión permiten una caracterización detallada de los factores que gobiernan la estabilidad de las proteínas globulares, sus mecanismos de plegamiento y los mecanismos de oligomerización mediante la combinación de la capacidad de ajuste de estabilidad fina de la perturbación de la presión y la resolución del sitio ofrecida por la espectroscopia de RMN de solución. Aquí presentamos un protocolo para sondear la estabilidad de plegamiento local de una proteína a través de un conjunto de experimentos 2D 1H-15N registrados desde 1 bar hasta 2.5 kbar. Los pasos necesarios para la adquisición y el análisis de tales experimentos se ilustran con datos adquiridos en el dominio RRM2 de hnRNPA1.
Introducción
Durante mucho tiempo se ha reconocido que los estados conformacionales de mayor energía y escasamente poblados de proteínas y complejos de proteínas desempeñan un papel clave en muchas vías biológicas1,2,3. Gracias a experimentos basados en secuencias de pulsos Carr-Purcell-Meiboom-Gill (CPMG)4,Chemical Exchange Saturation Transfer (CEST)5y dark-state exchange saturation transfer (DEST)6 (entre otras), la espectroscopia de RMN en solución ha surgido como un método de elección para caracterizar estados conformacionales transitorios7. Junto con estos experimentos, se pueden introducir perturbaciones como la temperatura, el pH o los desnaturalizantes químicos para aumentar la población relativa de subestados conformacionales de mayor energía. Del mismo modo, los equilibrios de proteínas también pueden ser perturbados mediante la aplicación de alta presión hidrostática. Dependiendo de la magnitud del cambio de volumen asociado con los cambios conformacionales correspondientes, un aumento de la presión de unos pocos cientos a unos pocos miles de bares puede estabilizar significativamente un estado de energía más alto o hacer que una proteína se despliegue completamente8,9,10. Los espectros de RMN de proteínas suelen mostrar dos tipos de cambios con presión hidrostática: (i) cambios de cambio químico y (ii) cambios de intensidad máxima. Los cambios químicos reflejan cambios en la interfaz proteína superficie-agua y/o compresión local de la estructura de la proteína en una escala de tiempo rápida (en relación con la escala de tiempo de RMN)11. Los cruces que exhiben grandes cambios químicos no lineales la dependencia de la presión pueden indicar la presencia de estados conformacionales de mayor energía12,13. Por otro lado, los cambios de intensidad máxima apuntan a transiciones conformacionales importantes en una escala de tiempo lenta, como cambios en las poblaciones de estados plegados / desplegados. La presencia de intermedios plegables o estados de energía superior se puede detectar a partir de grandes variaciones en la magnitud del cambio de volumen al desplegarse medido para diferentes residuos de una proteína dada14,15,16,17. Según nuestra experiencia, incluso las proteínas pequeñas que generalmente se clasifican como carpetas de dos estados exhiben respuestas no uniformes a la presión, lo que proporciona información útil sobre su estabilidad de plegamiento local. Aquí se describe un protocolo para la adquisición y análisis de la intensidad máxima de la amida y la dependencia de la presión de los cambios químicos de 1H, utilizando como proteína modelo el motivo de reconocimiento de ARN aislado 2 (RRM2) de la ribonucleoproteína nuclear heterogénea A1 (hnRNPA1).
Access restricted. Please log in or start a trial to view this content.
Protocolo
NOTA: El protocolo descrito aquí requiere (i) una bomba y celda de alta presión con un tubo de zirconia templado en aluminio de 2,5 kbar18,(ii) el software SPARKY19 para el análisis de los espectros de RMN, y (iii) un software de ajuste de curvas.
1. Preparación de la muestra, montaje de la celda de alta presión y puesta en preparación de los experimentos.
- Elección del tampón: Utilice la misma mezcla de tampones aniónicos y catiónicos, como fosfato y Tris20,21.
NOTA: El pKa de los tampones aniónicos como el fosfato y el MES se asocia con un volumen de reacción sustancial (es decir, la diferencia en los volúmenes molales parciales del ácido y los productos ionizados). Por lo tanto, el pH de tales tampones puede verse afectado significativamente por un cambio de presión (~ 0.25-0.5 unidad de pH / kbar). - Asegúrese de que el volumen de muestra requerido sea similar al de un tubo de RMN estándar de 3 mm de diámetro (~300μL).
- Introduzca la muestra etiquetada con 15N con una pipeta de vidrio en el tubo de zirconia. Asegúrese de que la muestra se atraque en la parte inferior del tubo. Completo con 200 μL de aceite mineral para evitar que la muestra se mezcle con el líquido de transmisión (por ejemplo, agua). Llene el resto del tubo con líquido de transmisión.
- Coloque una junta tórica de un solo uso en la parte superior del tubo de zirconia y deslice el tubo hacia la base(Figura 1A,B). Luego, conecte el tubo a la línea de sudación de alta presión y apriete la base a la celda primero a mano. Luego, aplique 14.7 Nm de torque para evitar fugas a menor presión(Figura 1C,D).
- Para comprobar la integridad del conjunto de la celda de presión, presurice el tubo hasta 300 bar fuera del espectrómetro utilizando el soporte de la celda y el recipiente de contención. Espere 15 minutos, restablezca la presión a 1 bar y verifique si hay fugas con una toallita limpia y sin pelusa.
- Inserte el tubo no presurizado en el espectrómetro guiando cuidadosamente la línea de sujeción. Deslice el tubo en el espectrómetro hasta alcanzar la posición sentada de la muestra (Figura 1E).
- Bloquea, shim, combina y sintoniza los canales 1H y 15N como de costumbre.
NOTA: Los shims para tubos de zirconia con clasificación de alta presión son muy diferentes de los tubos de RMN estándar. Se recomienda guardar los shims optimizados para su uso futuro. - Configure un 1H-15N-HSQC o TROSY-HSQC y registre un experimento de referencia en condiciones atmosféricas (1bar).
2. Registro de experimentos de RMN de alta presión
- Aumente gradualmente la presión de 1 bar a 2,5 kbar con incrementos de 500 bar para probar la estabilidad general de la proteína. Ajuste la velocidad de la bomba de presión de forma predeterminada en ~ 18 bar / s. Si no se conocen las tasas precisas de plegado/despliegue, deje que la muestra se equilibre 15-20 min después de cada incremento de 500 bares. Registre un espectro a 2,5 kbar.
- Disminuya gradualmente la presión a 1 bar con pasos de 500 bar para probar la reversibilidad de la perturbación de presión. Registrar otro espectro en condiciones atmosféricas y comparar los cambios químicos y las intensidades máximas con los del espectro de referencia previamente registrado en las mismas condiciones.
NOTA: Si los cruces nativos son más intensos después de la carrera de presión, puede indicar que pequeños agregados presentes en solución a presión atmosférica pueden haberse disociado y replechado adecuadamente. Por otro lado, una pérdida en la intensidad o cambios químicos significativos sugieren que la proteína puede experimentar un mal plegamiento no reversible en condiciones de alta presión. - Graba una serie de experimentos 2D desde 1 bar hasta 2,5 kbar cada 500 bar. Se recomienda registrar experimentos adicionales cerca del punto de inflexión de la transición de plegado/despliegue para mejorar la precisión del ajuste.
3. Análisis de los cambios de intensidad máxima
- Procese todos los espectros y transfiera la asignación de la columna vertebral del espectro de referencia a 1 bar al espectro registrado a 500 bar, y luego transfiera la asignación de 500 bar a 1 kbar y así sucesivamente.
NOTA: Debido a que la presión induce un desplazamiento no uniforme de 1H y 15N cambios químicos, no se limite a copiar la asignación de la columna vertebral de un espectro a otro. Ajústelo manualmente. - En el menú Sparky, haga clic en Peak > Peak List (lt). En la ventana Lista de picos, haga clic en Opciones y seleccione la opción para mostrar tanto Frecuencias (ppm) como Altura de datos. Guarde la lista obtenida para cada espectro.
- En un software de ajuste de curvas, copie la identidad de cruce y los valores de intensidad máxima para tener los valores de presión (en bar) como la variable del eje X y la intensidad como la variable del eje Y.
- Si se observa un despliegue completo o casi completo (>80%), ajuste los perfiles de intensidad máxima individuales para extraer la energía libre y el cambio de volumen al desplegarse, respectivamente, utilizando un modelo simple de dos estados:
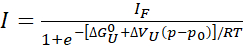 Eq. 1
Eq. 1
Donde, "I" es la intensidad observada de un diacruces a una presión dada p e IF es la intensidad del mismo diapasón en un estado completamente plegado. R es la constante del gas, T es la temperatura absoluta, ΔGU0 la diferencia de energía libre de Gibbs estándar entre los estados desplegado y plegado a presión atmosférica p0 (1 bar), y ΔVu el cambio de volumen al desplegarse. Con la presión p en bar y la temperatura T en kelvin, R = 1.987 cal/K, ΔGU0 está en cal/mol, y ΔVU está en cal/mol/bar. Multiplique los valores de ΔVU obtenidos del ajuste por 41,84 para convertirlos en mL/mol. Los valores de ΔVU están típicamente en el rango de -50 a -150 mL/mol para proteínas globulares22. Aquí, todos los parámetros se expresan con respecto a la reacción de despliegue, pero se pueden convertir fácilmente en parámetros de reacción de plegamiento (ΔGU0 = -ΔGF0 y ΔVU = -ΔVF).
4. Análisis de los cambios químicos en los cambios
- Organice las columnas en el software para que los puntos de presión como variables y los desplazamientos químicos de 1H extraídos de las listas Sparky como el eje Y.
- Ajuste la dependencia de la presión de los cambios químicos de 1H a una ecuación cuadrática simple:
δ(p) = δ0(p0) + B1(p-p0) + B2 (p-p0)2 Eq. 2
Donde, δ(p) es el desplazamiento químico medido de 1H de un cruce de una diafonía a una presión dada p y δ0(p0) los desplazamientos químicos de 1H del mismo cruce en el espectro de referencia registrado a 1 bar. B1 y B2 representan los parámetros de primer y segundo orden expresados en ppm/bar y ppm/bar2,respectivamente.
Access restricted. Please log in or start a trial to view this content.
Resultados
El protocolo aquí descrito se utilizó para sondear la dependencia de la presión de RRM2, el segundo motivo de reconocimiento de ARN de hnRNPA1 (residuos 95-106), que se despliega casi por completo dentro del rango de 2,5 kbar (>90%). 1 Los espectrosH-15N se recogieron a 1 bar, 500 bar, 750 bar, 1 kbar, 1.5 kbar, 2 kbar y 2.5 kbar(Figura 2). Dado que ninguno de los cruces nativos era visible por encima del nivel de ruido a 2,5 kbar, a todos los residuos correspondient...
Access restricted. Please log in or start a trial to view this content.
Discusión
Este estudio detalla un protocolo implementado para sondear las respuestas estructurales y termodinámicas de las proteínas a la perturbación de la presión. Los experimentos de alta presión registrados aquí en RRM2 demuestran que se pueden encontrar grandes variaciones en los valores de ΔVU, indicativos de un despliegue no totalmente cooperativo, en una proteína de dominio único relativamente pequeña. Una imagen similar surge del análisis de los cambios de cambio químico de 1H bajo presi?...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Todos los autores han leído y aprobado el manuscrito. Declaran que no hay conflictos de intereses.
Agradecimientos
Este trabajo fue apoyado por fondos del Roy J. Carver Charitable Trust a Julien Roche. Agradecemos a J. D. Levengood y B. S. Tolbert por proporcionar amablemente la muestra RRM2.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Bruker Nmr Cell 2.5 Kbar | Daedalus Innovations LLC | NMRCELL-B | |
| Sparky3 | University of California San Francisco, CA | N/A | |
| Xtreme-60 Syringe pump | Daedalus Innovations LLC | XTREME-60 |
Referencias
- Alderson, R. T., Kay, L. E. Unveiling invisible protein states with NMR spectroscopy. Current Opinion in Structural Biology. 60, 39-49 (2020).
- Korzhnev, D. M., Kay, L. E. probing invisible, low-populated states of protein molecules by relaxation dispersion NMR spectroscopy: An application to protein folding. Accounts of Chemical Research. 41, 442-451 (2008).
- Loria, P. J., Berlow, R. B., Watt, E. D. Characterization of enzyme motions by solution NMR relaxation dispersion. Accounts of Chemical Research. 41, 214-221 (2008).
- Ishima, R. CPMG relaxation dispersion. Methods in Molecular Biology. 1084, 29-49 (2014).
- Longo, D. L., et al. Chemical exchange saturation transfer (CEST): an efficient tool for detecting molecular information on proteins' behaviour. Analyst. 39, 2687-2690 (2014).
- Fawzi, N. L., Ying, J., Torchia, D. A., Clore, M. G. Probing exchange kinetics and atomic resolution dynamics in high-molecular-weight complexes using dark-state exchange saturation transfer NMR spectroscopy. Nature Protocols. 7, 1523-1533 (2012).
- Anthis, N. J., Clore, M. G. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48, 35-116 (2015).
- Roche, J., et al. Cavities determine the pressure unfolding of proteins. Proceedings of the National Academy of Sciences of the United States of America. 109, 6945-6950 (2012).
- Chen, C. R., Makhatadze, G. I. Molecular determinant of the effects of hydrostatic pressure on protein folding stability. Nature Communications. 8, 14561(2017).
- Roche, J., Royer, C. A. Lessons from pressure denaturation of proteins. Journal of the Royal Society Interface. 15, 20180244(2018).
- Xu, X., Gagné, D., Aramini, J. M., Gardner, K. H. Volume and compressibility differences between protein conformations revealed by high-pressure NMR. Biophysical Journal. 120, 924-935 (2021).
- Akasaka, K., Li, H. Low-lying excited states of proteins revealed from non-linear pressure shifts in 1H and 15N NMR. Biochemistry. 40, 8665-8671 (2001).
- Akasaka, K. Probing conformational fluctuation of proteins by pressure perturbation. Chemical Reviews. 106, 1814-1835 (2006).
- Kitahara, R., Yokoyama, S., Akasaka, K. NMR snapshots of a fluctuating protein structure: ubiquitin at 30 bar-3 kbar. Journal of Molecular Biology. 347, 277-285 (2005).
- Roche, J., et al. remodeling of the folding free energy landscape of Staphylococcal nuclease by cavity-creating mutations. Biochemistry. 51, 9535-9546 (2012).
- Nucci, N. V., Fuglestad, B., Athanasoula, E. A., Wand, J. A. Role of cavities and hydration in the pressure unfolding of T4 lysozyme. Proceedings of the National Academy of Sciences of the United States of America. 111, 13846-13851 (2014).
- Maeno, A., et al. Cavity as a source of conformational fluctuation and high-energy state: High-pressure NMR study of a cavity-enlarged mutant of T4 lysozyme. Biophysical Journal. 108, 133-145 (2015).
- Peterson, R. W., Wand, J. A. Self-contained high-pressure cell, apparatus, and procedure for the preparation of encapsulated proteins dissolved in low viscosity fluids for nuclear magnetic resonance spectroscopy. Review of Scientific Instruments. 76, 094101(2005).
- Goddard, T. D., Kneller, D. G. Sparky 3. , University of California San Francisco. San Francisco, CA. (2010).
- Caro, J. A., Wand, J. A. Practical aspects of high-pressure NMR spectroscopy and its applications in protein biophysics and structural biology. Methods. 148, 67-80 (2018).
- Kitamura, T., Itoh, J. Reaction volume of protonic ionization for buffering agents. Prediction of pressure dependence of pH and pOH. Journal of Solution Chemistry. 16, 715-725 (1987).
- Royer, C. A. Revisiting volume changes in pressure-induced protein unfolding. Biochimica et Biophysica Acta. 1595, 201-209 (2002).
- Erlach, M. B., et al. Relationship between nonliner pressure-induced chemical shift changes and thermodynamic parameters. Journal of Physical Chemistry B. 118, 5681-5690 (2014).
- de Oliveira, G. A. P., Silva, J. L. A hypothesis to reconcile the physical and chemical unfolding of proteins. Proceedings of the National Academy of Sciences of The United States of America. 112, 2775-2784 (2015).
- Nguyen, L. M., Roche, J. High-pressure NMR techniques for the study of protein dynamics, folding and aggregation. Journal of Magnetic Resonance. 277, 179-185 (2017).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados