È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Esperimenti NMR ad alta pressione per il rilevamento di stati conformazionali a bassa pressione delle proteine
In questo articolo
Riepilogo
Forniamo una descrizione dettagliata dei passaggi necessari per assemblare una cella ad alta pressione, impostare e registrare esperimenti NMR ad alta pressione e infine analizzare sia l'intensità di picco che i cambiamenti di spostamento chimico sotto pressione. Questi esperimenti possono fornire preziose informazioni sui percorsi di ripiegamento e sulla stabilità strutturale delle proteine.
Abstract
L'alta pressione è un metodo di perturbazione ben noto che può essere utilizzato per destabilizzare le proteine globulari e dissociare i complessi proteici in modo reversibile. La pressione idrostatica guida gli equilibri termodinamici verso lo stato o gli stati con il volume molare inferiore. L'aumento della pressione offre, quindi, l'opportunità di sintonizzare finemente la stabilità delle proteine globulari e gli equilibri di oligomerizzazione dei complessi proteici. Gli esperimenti NMR ad alta pressione consentono una caratterizzazione dettagliata dei fattori che governano la stabilità delle proteine globulari, i loro meccanismi di ripiegamento e i meccanismi di oligomerizzazione combinando la capacità di sintonizzazione della stabilità fine della perturbazione della pressione e la risoluzione del sito offerta dalla spettroscopia NMR della soluzione. Qui presentiamo un protocollo per sondare la stabilità di ripiegamento locale di una proteina tramite una serie di esperimenti 2D 1H-15N registrati da 1 bar a 2,5 kbar. I passaggi necessari per l'acquisizione e l'analisi di tali esperimenti sono illustrati con i dati acquisiti sul dominio RRM2 di hnRNPA1.
Introduzione
È stato a lungo riconosciuto che gli stati conformazionali a più alta energia e scarsamente popolati di proteine e complessi proteici svolgono un ruolo chiave in molte vie biologiche1,2,3. Grazie a esperimenti basati su Carr-Purcell-Meiboom-Gill (CPMG)4,Chemical Exchange Saturation Transfer (CEST)5e dark-state exchange saturation transfer (DEST) 6 sequenze di impulsi (tra gli altri), la spettroscopia NMR insoluzione è emersa come metodo di scelta per caratterizzare gli stati conformazionali transitori7. Insieme a questi esperimenti, possono essere introdotte perturbazioni come temperatura, pH o denaturanti chimici per aumentare la popolazione relativa di sottostati conformazionali a energia più elevata. Allo stesso modo, gli equilibri proteici possono anche essere perturbati applicando un'alta pressione idrostatica. A seconda dell'entità della variazione di volume associata ai corrispondenti cambiamenti conformazionali, un aumento della pressione da poche centinaia a poche migliaia di bar può stabilizzare significativamente uno stato energetico più elevato o causare il completo dispiegamento di una proteina8,9,10. Gli spettri NMR delle proteine mostrano tipicamente due tipi di cambiamenti con la pressione idrostatica: (i) cambiamenti di spostamento chimico e (ii) cambiamenti di intensità di picco. I cambiamenti di spostamento chimico riflettono i cambiamenti nell'interfaccia proteina superficie-acqua e/o la compressione locale della struttura proteica su una scala temporale rapida (rispetto alla scala temporale NMR)11. Crosspeaks che esibisce grandi spostamenti chimici non lineari la dipendenza dalla pressione può indicare la presenza di stati conformazionali di energia superiore12,13. D'altra parte, i cambiamenti di intensità di picco indicano importanti transizioni conformazionali su una scala temporale lenta, come i cambiamenti nelle popolazioni statali piegate / dispiegate. La presenza di intermedi ripieganti o stati energetici superiori può essere rilevata da grandi variazioni nell'entità della variazione di volume allo spiegamento misurata per diversi residui di una data proteina14,15,16,17. Sulla base della nostra esperienza, anche le piccole proteine che sono tipicamente classificate come cartelle a due stati mostrano risposte non uniformi alla pressione, il che fornisce informazioni utili sulla loro stabilità di ripiegamento locale. Qui è descritto un protocollo per l'acquisizione e l'analisi dell'intensità di picco dell'ammide e della dipendenza dalla pressione degli spostamenti chimici di 1H, utilizzando come proteina modello il motivo di riconoscimento dell'RNA isolato 2 (RRM2) della ribonucleoproteina nucleare eterogenea A1 (hnRNPA1).
Access restricted. Please log in or start a trial to view this content.
Protocollo
NOTA: Il protocollo qui descritto richiede (i) una pompa e una cella ad alta pressione con un tubo di zirconia in alluminio resistente da 2,5 kbar18,(ii) il software SPARKY19 per l'analisi degli spettri NMR e (iii) un software di raccordo a curva.
1. Preparazione del campione, assemblaggio della cella ad alta pressione e impostazione degli esperimenti.
- Scelta del tampone: Utilizzare una miscela uguale di tamponi anionici e cationici, come fosfato e Tris20,21.
NOTA: Il pKa di tamponi anionici come fosfato e MES è associato a un volume di reazione sostanziale (cioè la differenza nei volumi molali parziali dell'acido e dei prodotti ionizzati). Il pH di tali tamponi può quindi essere significativamente influenzato da un cambiamento di pressione (~ 0,25-0,5 unità di pH / kbar). - Assicurarsi che il volume del campione richiesto sia simile a quello di un tubo NMR standard di 3 mm di diametro (~ 300μL).
- Introdurre il campione con etichetta 15N con una pipetta di vetro nel tubo di zirconia. Assicurarsi che il campione si sieda nella parte inferiore del tubo. Completo di 200 μL di olio minerale per evitare che il campione si mescoli con il liquido di trasmissione (ad esempio, acqua). Riempire il resto del tubo con liquido di trasmissione.
- Mettere un O-ring monouso sopra il tubo di zirconia e far scorrere il tubo nella base (Figura 1A,B). Quindi, collegare il tubo alla linea di collegamento ad alta pressione e stringere la base alla cella prima a mano. Quindi, applicare 14,7 Nm di coppia per evitare perdite a pressione inferiore (Figura 1C, D).
- Per verificare l'integrità del gruppo della cella di pressione, pressurizzare il tubo fino a 300 bar all'esterno dello spettrometro utilizzando il supporto cellulare e il contenitore di contenimento. Attendere 15 minuti, ripristinare la pressione a 1 bar e verificare la disponibilità di perdite con una salvietta pulita senza lanugine.
- Inserire il tubo non pressurizzato nello spettrometro guidando attentamente la linea di legame. Far scorrere il tubo nello spettrometro fino a raggiungere la posizione seduta del campione (Figura 1E).
- Blocca, shim, abbina e sintonizza i canali 1H e 15N come al solito.
NOTA: gli spessori per tubi in zirconia ad alta pressione sono molto diversi dai tubi NMR standard. Si consiglia di salvare gli spessori ottimizzati per un uso futuro. - Impostare un 1H-15N-HSQC o TROSY-HSQC e registrare un esperimento di riferimento in condizioni atmosferiche (1bar).
2. Registrazione di esperimenti NMR ad alta pressione
- Aumentare gradualmente la pressione da 1 bar a 2,5 kbar con incrementi di 500 bar per testare la stabilità complessiva della proteina. Impostare la velocità della pompa a pressione per impostazione predefinita a ~ 18 bar / s. Se le velocità precise di piegatura/ dispiegamento non sono note, lasciare che il campione equilibra 15-20 minuti dopo ogni incremento di 500 bar. Registrare uno spettro a 2,5 kbar.
- Ridurre gradualmente la pressione a 1 bar con passi di 500 bar per testare la reversibilità della perturbazione della pressione. Registrare un altro spettro in condizioni atmosferiche e confrontare gli spostamenti chimici e le intensità di picco con quello dello spettro di riferimento precedentemente registrato nelle stesse condizioni.
NOTA: Se i crosspeak nativi sono più intensi dopo la corsa di pressione, può indicare che piccoli aggregati presenti in soluzione a pressione atmosferica potrebbero essersi dissociati e ripiegati correttamente. D'altra parte, una perdita di intensità o cambiamenti significativi dello spostamento chimico suggeriscono che la proteina può sperimentare un misfolding non reversibile in condizioni di alta pressione. - Registra una serie di esperimenti 2D da 1 bar a 2,5 kbar ogni 500 bar. Si consiglia di registrare ulteriori esperimenti vicino al punto di inflessione della transizione di piegatura / dispiegamento per migliorare la precisione della vestibilità.
3. Analisi delle variazioni di intensità di picco
- Elaborare tutti gli spettri e trasferire l'assegnazione della spina dorsale dallo spettro di riferimento a 1 bar allo spettro registrato a 500 bar, quindi trasferire l'assegnazione da 500 bar a 1 kbar e così via.
NOTA: poiché la pressione induce uno spostamento non uniforme di 1H e 15N di spostamenti chimici, non copiare semplicemente l'assegnazione della spina dorsale da uno spettro all'altro. Regolalo manualmente. - Nel menu Sparky, fare clic su Peak > Peak List (lt). Nella finestra Elenco picchi, fare clic su Opzioni e selezionare l'opzione per visualizzare sia frequenze (ppm) che Altezza dati. Salva l'elenco ottenuto per ogni spettro.
- In un software di adattamento della curva, copiare l'identità crosspeak e i valori di intensità di picco per avere i valori di pressione (in bar) come variabile dell'asse X e l'intensità come variabile dell'asse Y.
- Se si osserva uno svolgimento completo o quasi completo (>80%), adattarsi ai singoli profili di intensità di picco per estrarre l'energia libera e la variazione di volume allo svolgimento, rispettivamente, utilizzando un semplice modello a due stati:
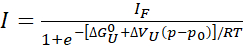 Eq. 1
Eq. 1
Dove, "I" è l'intensità osservata di un crosspeak a una data pressione p e IF è l'intensità dello stesso crosspeak in uno stato completamente piegato. R è la costante del gas, T è la temperatura assoluta, ΔGU0 la differenza standard di energia libera di Gibbs tra gli stati dispiegati e piegati alla pressione atmosferica p0 (1 bar) e ΔVu la variazione di volume allo spiegamento. Con la pressione p in bar e la temperatura T in kelvin, R = 1.987 cal/K, ΔGU0 è in cal/mol e ΔVU è in cal/mol/bar. Moltiplicare i valori di ΔVU ottenuti dall'adattamento per 41,84 per convertirlo in mL/mol. I valori di ΔVU sono tipicamente nell'intervallo da -50 a -150 ml/mol per le proteine globulari22. Qui, tutti i parametri sono espressi per quanto riguarda la reazione di dispiegamento ma possono essere facilmente convertiti in parametri di reazione di piegatura(ΔG U0 = -ΔGF0 e ΔVU = -ΔVF).
4. Analisi dei cambiamenti di spostamento chimico
- Disporre le colonne nel software in modo da avere punti di pressione variabili e gli spostamenti chimici 1H estratti dagli elenchi Sparky come asse Y.
- Adatta la dipendenza dalla pressione degli spostamenti chimici di 1H a una semplice equazione quadratica:
δ(p) = δ0(p0) + B1(p-p0) + B2 (p-p0)2 Eq. 2
Quando, δ(p) è lo spostamento chimico misurato di 1H di un crosspeak ad una data pressione p e δ0(p0) gli spostamenti chimici 1H dello stesso crosspeak nello spettro di riferimento registrati a 1 bar. B1 e B2 rappresentano i parametri del primo e del secondo ordine espressi rispettivamente in ppm/bar e ppm/bar2.
Access restricted. Please log in or start a trial to view this content.
Risultati
Il protocollo qui descritto è stato utilizzato per sondare la dipendenza dalla pressione di RRM2, il secondo motivo di riconoscimento dell'RNA di hnRNPA1 (residui 95-106), che è quasi completamente dispiegato all'interno dell'intervallo di 2,5 kbar (>90%). 1 Gli spettriH-15N sono stati raccolti a 1 bar, 500 bar, 750 bar, 1 kbar, 1,5 kbar, 2 kbar e 2,5 kbar (Figura 2). Poiché nessuno dei crosspeak nativi era visibile al di sopra del livello di rumore a 2,5 kbar, a tut...
Access restricted. Please log in or start a trial to view this content.
Discussione
Questo studio descrive in dettaglio un protocollo implementato per sondare le risposte strutturali e termodinamiche delle proteine alla perturbazione della pressione. Gli esperimenti ad alta pressione registrati qui su RRM2 dimostrano che grandi variazioni nei valori di ΔVU, indicative di uno sviluppo non completamente cooperativo, possono essere trovate in una proteina a dominio singolo relativamente piccola. Un quadro simile emerge dall'analisi dei cambiamenti di spostamento chimico di 1H sotto p...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Tutti gli autori hanno letto e approvato il manoscritto. Non dichiarano conflitti di interesse.
Riconoscimenti
Questo lavoro è stato sostenuto da fondi dal Roy J. Carver Charitable Trust a Julien Roche. Ringraziamo J. D. Levengood e B. S. Tolbert per aver gentilmente fornito il campione RRM2.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Bruker Nmr Cell 2.5 Kbar | Daedalus Innovations LLC | NMRCELL-B | |
| Sparky3 | University of California San Francisco, CA | N/A | |
| Xtreme-60 Syringe pump | Daedalus Innovations LLC | XTREME-60 |
Riferimenti
- Alderson, R. T., Kay, L. E. Unveiling invisible protein states with NMR spectroscopy. Current Opinion in Structural Biology. 60, 39-49 (2020).
- Korzhnev, D. M., Kay, L. E. probing invisible, low-populated states of protein molecules by relaxation dispersion NMR spectroscopy: An application to protein folding. Accounts of Chemical Research. 41, 442-451 (2008).
- Loria, P. J., Berlow, R. B., Watt, E. D. Characterization of enzyme motions by solution NMR relaxation dispersion. Accounts of Chemical Research. 41, 214-221 (2008).
- Ishima, R. CPMG relaxation dispersion. Methods in Molecular Biology. 1084, 29-49 (2014).
- Longo, D. L., et al. Chemical exchange saturation transfer (CEST): an efficient tool for detecting molecular information on proteins' behaviour. Analyst. 39, 2687-2690 (2014).
- Fawzi, N. L., Ying, J., Torchia, D. A., Clore, M. G. Probing exchange kinetics and atomic resolution dynamics in high-molecular-weight complexes using dark-state exchange saturation transfer NMR spectroscopy. Nature Protocols. 7, 1523-1533 (2012).
- Anthis, N. J., Clore, M. G. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48, 35-116 (2015).
- Roche, J., et al. Cavities determine the pressure unfolding of proteins. Proceedings of the National Academy of Sciences of the United States of America. 109, 6945-6950 (2012).
- Chen, C. R., Makhatadze, G. I. Molecular determinant of the effects of hydrostatic pressure on protein folding stability. Nature Communications. 8, 14561(2017).
- Roche, J., Royer, C. A. Lessons from pressure denaturation of proteins. Journal of the Royal Society Interface. 15, 20180244(2018).
- Xu, X., Gagné, D., Aramini, J. M., Gardner, K. H. Volume and compressibility differences between protein conformations revealed by high-pressure NMR. Biophysical Journal. 120, 924-935 (2021).
- Akasaka, K., Li, H. Low-lying excited states of proteins revealed from non-linear pressure shifts in 1H and 15N NMR. Biochemistry. 40, 8665-8671 (2001).
- Akasaka, K. Probing conformational fluctuation of proteins by pressure perturbation. Chemical Reviews. 106, 1814-1835 (2006).
- Kitahara, R., Yokoyama, S., Akasaka, K. NMR snapshots of a fluctuating protein structure: ubiquitin at 30 bar-3 kbar. Journal of Molecular Biology. 347, 277-285 (2005).
- Roche, J., et al. remodeling of the folding free energy landscape of Staphylococcal nuclease by cavity-creating mutations. Biochemistry. 51, 9535-9546 (2012).
- Nucci, N. V., Fuglestad, B., Athanasoula, E. A., Wand, J. A. Role of cavities and hydration in the pressure unfolding of T4 lysozyme. Proceedings of the National Academy of Sciences of the United States of America. 111, 13846-13851 (2014).
- Maeno, A., et al. Cavity as a source of conformational fluctuation and high-energy state: High-pressure NMR study of a cavity-enlarged mutant of T4 lysozyme. Biophysical Journal. 108, 133-145 (2015).
- Peterson, R. W., Wand, J. A. Self-contained high-pressure cell, apparatus, and procedure for the preparation of encapsulated proteins dissolved in low viscosity fluids for nuclear magnetic resonance spectroscopy. Review of Scientific Instruments. 76, 094101(2005).
- Goddard, T. D., Kneller, D. G. Sparky 3. , University of California San Francisco. San Francisco, CA. (2010).
- Caro, J. A., Wand, J. A. Practical aspects of high-pressure NMR spectroscopy and its applications in protein biophysics and structural biology. Methods. 148, 67-80 (2018).
- Kitamura, T., Itoh, J. Reaction volume of protonic ionization for buffering agents. Prediction of pressure dependence of pH and pOH. Journal of Solution Chemistry. 16, 715-725 (1987).
- Royer, C. A. Revisiting volume changes in pressure-induced protein unfolding. Biochimica et Biophysica Acta. 1595, 201-209 (2002).
- Erlach, M. B., et al. Relationship between nonliner pressure-induced chemical shift changes and thermodynamic parameters. Journal of Physical Chemistry B. 118, 5681-5690 (2014).
- de Oliveira, G. A. P., Silva, J. L. A hypothesis to reconcile the physical and chemical unfolding of proteins. Proceedings of the National Academy of Sciences of The United States of America. 112, 2775-2784 (2015).
- Nguyen, L. M., Roche, J. High-pressure NMR techniques for the study of protein dynamics, folding and aggregation. Journal of Magnetic Resonance. 277, 179-185 (2017).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon