É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Experimentos de RMN de alta pressão para detectar estados conformais de baixa mentira de proteína
Neste Artigo
Resumo
Fornecemos uma descrição detalhada das etapas necessárias para montar uma célula de alta pressão, configurar e registrar experimentos de RMN de alta pressão, e finalmente analisar tanto a intensidade máxima quanto as mudanças de mudança química sob pressão. Esses experimentos podem fornecer informações valiosas sobre as vias dobráveis e a estabilidade estrutural das proteínas.
Resumo
A alta pressão é um método de perturbação bem conhecido que pode ser usado para desestabilizar proteínas globulares e dissociar complexos proteicos de forma reversível. A pressão hidrostática impulsiona o equilíbrio termodinâmico em direção ao estado com o menor volume molar. O aumento da pressão oferece, portanto, as oportunidades de afinar a estabilidade das proteínas globulares e o equilíbrio da oligomerização dos complexos proteicos. Experimentos de RMN de alta pressão permitem uma caracterização detalhada dos fatores que regem a estabilidade das proteínas globulares, seus mecanismos de dobramento e mecanismos de oligomerização, combinando a fina capacidade de ajuste de estabilidade da perturbação da pressão e a resolução do local oferecida pela solução espectroscopia NMR. Aqui apresentamos um protocolo para sondar a estabilidade de dobramento local de uma proteína através de um conjunto de experimentos 2D 1H-15N registrados de 1 bar a 2,5 kbar. As etapas necessárias para a aquisição e análise de tais experimentos são ilustradas com dados adquiridos no domínio RRM2 do hnRNPA1.
Introdução
Há muito se reconhece que estados conformacionais de maior energia e pouco povoados de proteínas e complexos proteicos desempenham um papel fundamental em muitas vias biológicas1,2,3. Graças a experimentos baseados em Carr-Purcell-Meiboom-Gill (CPMG)4, Transferência de Saturação de Câmbio Químico (CEST)5, e transferência de saturação de câmbio de estado escuro (DEST)6 sequências de pulso (entre outras), a solução de espectroscopia NMR surgiu como um método de escolha para caracterizar estados conformacionais transitórios7. Junto com esses experimentos, perturbações como temperatura, pH ou desnaturantes químicos podem ser introduzidas para aumentar a população relativa de subestados conformais de maior energia. Da mesma forma, o equilíbrio proteico também pode ser perturbado pela aplicação de alta pressão hidrostática. Dependendo da magnitude da mudança de volume associada às mudanças conformais correspondentes, um aumento da pressão em algumas centenas para alguns milhares de barras pode estabilizar significativamente um estado de energia mais elevado ou fazer com que uma proteína se desdobre completamente8,9,10. Os espectros de NMR proteicas normalmente apresentam dois tipos de alterações com pressão hidrostática: (i) alterações químicas de mudança e (ii) alterações de intensidade de pico. As alterações de mudança química refletem alterações na interface proteína superfície-água e/ou compressão local da estrutura proteica em uma escala de tempo rápida (em relação à escala de tempo NMR)11. Falas cruzadas que apresentam grandes mudanças químicas não lineares a dependência da pressão pode indicar a presença de estados conformacionais de maior energia12,13. Por outro lado, as mudanças de intensidade de pico apontam para grandes transições conformais em uma escala de tempo lenta, como mudanças nas populações estaduais dobradas/desdobradas. A presença de intermediários dobráveis ou estados de maior energia pode ser detectada a partir de grandes variações na magnitude da variação de volume ao desdobrar medido para diferentes resíduos de uma determinada proteína14,15,16,17. Com base em nossa experiência, mesmo pequenas proteínas que são tipicamente classificadas como pastas de dois estados exibem respostas não uniformes à pressão, o que fornece informações úteis sobre sua estabilidade de dobramento local. Descrito aqui é um protocolo para aquisição e análise da intensidade de pico de amida e 1H de mudanças químicas de dependência de pressão, utilizando como proteína modelo o motivo isolado de reconhecimento de RNA 2 (RRM2) da heterogênea ribonucleoproteína nuclear A1 (hnRNPA1).
Access restricted. Please log in or start a trial to view this content.
Protocolo
NOTA: O protocolo descrito aqui requer (i) uma bomba de alta pressão e célula com um tubo de zircônia endurecido em alumíniode 2,5kbar 18 , (ii) o software SPARKY19 para análise do espectro NMR, e (iii) um software de montagem de curva.
1. Preparação da amostra, montagem da célula de alta pressão e configuração dos experimentos.
- Escolha do tampão: Use mistura igual de tampões aniônicos e cátônicos, como fosfato e Tris20,21.
NOTA: A pKa de tampões aniônicos como fosfato e MES está associada a um volume de reação substancial (ou seja, a diferença nos volumes parciais molais do ácido e dos produtos ionizados). O pH desses buffers pode, portanto, ser significativamente afetado por uma mudança de pressão (~0,25-0,5 pH unidade/kbar). - Certifique-se de que o volume amostral necessário seja semelhante ao de um tubo NMR de diâmetro padrão de 3 mm (~300μL).
- Introduza a amostra de 15N com uma pipeta de vidro no tubo de zircônia. Certifique-se de que a amostra se senta na parte inferior do tubo. Completo com 200 μL de óleo mineral para evitar que a amostra se misture com o líquido de transmissão (por exemplo, água). Encha o resto do tubo com líquido de transmissão.
- Coloque um anel O de uso único em cima do tubo de zircônia e deslize o tubo para dentro da base (Figura 1A, B). Em seguida, conecte o tubo à linha de corda de alta pressão e aperte a base à célula primeiro manualmente. Em seguida, aplique 14,7 Nm de torque para evitar vazamentos com menor pressão(Figura 1C,D).
- Para verificar a integridade do conjunto da célula de pressão, pressurize o tubo até 300 barras fora do espectrômetro usando suporte celular e recipiente de contenção. Aguarde 15 minutos, reinicie a pressão para 1 barra e verifique se há vazamentos com uma limpeza limpa sem fiapos.
- Insira o tubo nãopressurizado no espectrômetro, guiando cuidadosamente a linha da corda. Deslize o tubo no espectrômetro até atingir a posição de sessão da amostra(Figura 1E).
- Travar, shim, combinar e ajustar os canais 1H e 15N como de costume.
NOTA: Os calços para tubos de zircônia de alta pressão são muito diferentes dos tubos NMR padrão. Recomenda-se economizar os calços otimizados para uso futuro. - Configure um 1H-15N-HSQC ou TROSY-HSQC e registou um experimento de referência em condições atmosféricas (1bar).
2. Registrando experimentos de RMN de alta pressão
- Aumente gradualmente a pressão de 1 bar para 2,5 kbar com incrementos de 500 barras para testar a estabilidade geral da proteína. Defina a velocidade da bomba de pressão por padrão em ~18 barras/s. Se as taxas precisas de dobramento/desdobramento não forem conhecidas, deixe que a amostra equilibre 15-20 minutos após cada incremento de 500 barras. Registo de um espectro a 2,5 kbar.
- Diminua gradualmente a pressão de volta para 1 barra com 500 passos de barra para testar a reversibilidade da perturbação da pressão. Registo de outro espectro em condições atmosféricas e compare as mudanças químicas e intensidades de pico com o do espectro de referência anteriormente registrado nas mesmas condições.
NOTA: Se os cruzamentos nativos forem mais intensos após a corrida de pressão, pode indicar que pequenos agregados presentes na solução à pressão atmosférica podem ter dissociado e redobrado adequadamente. Por outro lado, uma perda na intensidade ou mudanças químicas significativas sugerem que a proteína pode experimentar um erro não reversível em condições de alta pressão. - Grave uma série de experimentos 2D de 1 bar a 2,5 kbar a cada 500 bar. Recomenda-se registrar experimentos adicionais próximos ao ponto de inflexão da transição dobrável/desdobramento para melhorar a precisão do ajuste.
3. Analisando mudanças de intensidade de pico
- Processe todos os espectros e transfira a atribuição backbone do espectro de referência em 1 bar para o espectro registrado em 500 bar, e depois transfira a atribuição de 500 bar para 1 kbar e assim por diante.
NOTA: Como a pressão induz uma mudança não uniforme de mudanças químicas de 1H e 15N, não copie simplesmente a atribuição da espinha dorsal de um espectro para o outro. Ajuste manualmente. - No menu Sparky, clique em Peak > Peak List (lt). Na janela Lista de picos, clique em Opções e selecione a opção para exibir ambas as frequências (ppm) e altura de dados. Salve a lista obtida para cada espectro.
- Em um software de montagem de curva, copie a identidade de crosspeak e os valores de intensidade máxima para ter os valores de pressão (na barra) como a variável eixo X e a intensidade como variável do eixo Y.
- Se for observado um desdobramento completo ou quase completo (>80%), encaixe os perfis individuais de intensidade de pico para extrair a energia livre e a mudança de volume após o desdobramento, respectivamente, utilizando um modelo simples de dois estados:
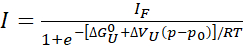 Eq. 1
Eq. 1
Onde, "Eu" é a intensidade observada de um crosspeak a uma determinada pressão p e IF é a intensidade do mesmo cruzamento em um estado totalmente dobrado. R é a constante do gás, T é a temperatura absoluta, ΔGU0 a diferença de energia padrão gibbs livre entre os estados desdobrados e dobrados na pressão atmosférica p0 (1 barra), e ΔVu a mudança de volume ao se desdobrar. Com a pressão p na barra e temperatura T em kelvin, R = 1,987 cal/K, ΔGU0 é em cal/mol, e ΔVU está em cal/mol/bar. Multiplique os valores ΔVU obtidos do ajuste por 41,84 para converter em mL/mol. Os valores ΔVU estão tipicamente na faixa de -50 a -150 mL/mol para proteínas globulares22. Aqui, todos os parâmetros são expressos em relação à reação desdobrada, mas podem ser facilmente convertidos em parâmetros de reação dobráveis (ΔGU0 = -ΔGF0 e ΔVU = -ΔVF).
4. Analisar mudanças de mudança química
- Organize as colunas no software para ter pontos de pressão como variável e as mudanças químicas de 1H extraídas das listas Sparky como o eixo Y.
- Ajuste a dependência de pressão das mudanças químicas de 1H para uma equação quadrática simples:
δ(p) = δ0(p0) + B1(p-p0) + B2 (p-p0)2 Eq. 2
Onde, δ(p)é a mudança química de 1H medida de uma fala cruzada a uma determinada pressão p e δ0(p0) as mudanças químicas de 1H do mesmo cruzamento no espectro de referência registrado em 1 bar. B1 e B2 representam os parâmetros de primeira e segunda ordem expressos em ppm/bar e ppm/barra2, respectivamente.
Access restricted. Please log in or start a trial to view this content.
Resultados
O protocolo descrito aqui foi usado para sondar a dependência de pressão do RRM2, o segundo motivo de reconhecimento de RNA do hnRNPA1 (resíduos 95-106), que é quase completamente desdobrado dentro da faixa de 2,5 kbar (>90%). 1 H-15N spectra foram coletados em 1 bar, 500 bar, 750 bar, 1 kbar, 1,5 kbar, 2 kbar e 2,5 kbar(Figura 2). Como nenhum dos cruzamentos nativos era visível acima do nível de ruído em 2,5 kbar, todos os resíduos correspondentes foram atribu?...
Access restricted. Please log in or start a trial to view this content.
Discussão
Este estudo detalha um protocolo implementado para sondar respostas estruturais e termodinâmicas proteicas à perturbação da pressão. Os experimentos de alta pressão registrados aqui no RRM2 demonstram que grandes variações nos valores ΔVU, indicativos de desdobramentos não totalmente cooperativos, podem ser encontradas em uma proteína de domínio único relativamente pequena. Um quadro semelhante emerge da análise de mudanças químicas de 1H sob pressão. Deve-se notar que Kalbitzer e c...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Todos os autores leram e aprovaram o manuscrito. Eles não declaram conflitos de interesse.
Agradecimentos
Este trabalho foi apoiado por fundos do Roy J. Carver Charitable Trust para Julien Roche. Agradecemos a J. D. Levengood e B. S. Tolbert por fornecerem gentilmente a amostra RRM2.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Bruker Nmr Cell 2.5 Kbar | Daedalus Innovations LLC | NMRCELL-B | |
| Sparky3 | University of California San Francisco, CA | N/A | |
| Xtreme-60 Syringe pump | Daedalus Innovations LLC | XTREME-60 |
Referências
- Alderson, R. T., Kay, L. E. Unveiling invisible protein states with NMR spectroscopy. Current Opinion in Structural Biology. 60, 39-49 (2020).
- Korzhnev, D. M., Kay, L. E. probing invisible, low-populated states of protein molecules by relaxation dispersion NMR spectroscopy: An application to protein folding. Accounts of Chemical Research. 41, 442-451 (2008).
- Loria, P. J., Berlow, R. B., Watt, E. D. Characterization of enzyme motions by solution NMR relaxation dispersion. Accounts of Chemical Research. 41, 214-221 (2008).
- Ishima, R. CPMG relaxation dispersion. Methods in Molecular Biology. 1084, 29-49 (2014).
- Longo, D. L., et al. Chemical exchange saturation transfer (CEST): an efficient tool for detecting molecular information on proteins' behaviour. Analyst. 39, 2687-2690 (2014).
- Fawzi, N. L., Ying, J., Torchia, D. A., Clore, M. G. Probing exchange kinetics and atomic resolution dynamics in high-molecular-weight complexes using dark-state exchange saturation transfer NMR spectroscopy. Nature Protocols. 7, 1523-1533 (2012).
- Anthis, N. J., Clore, M. G. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48, 35-116 (2015).
- Roche, J., et al. Cavities determine the pressure unfolding of proteins. Proceedings of the National Academy of Sciences of the United States of America. 109, 6945-6950 (2012).
- Chen, C. R., Makhatadze, G. I. Molecular determinant of the effects of hydrostatic pressure on protein folding stability. Nature Communications. 8, 14561(2017).
- Roche, J., Royer, C. A. Lessons from pressure denaturation of proteins. Journal of the Royal Society Interface. 15, 20180244(2018).
- Xu, X., Gagné, D., Aramini, J. M., Gardner, K. H. Volume and compressibility differences between protein conformations revealed by high-pressure NMR. Biophysical Journal. 120, 924-935 (2021).
- Akasaka, K., Li, H. Low-lying excited states of proteins revealed from non-linear pressure shifts in 1H and 15N NMR. Biochemistry. 40, 8665-8671 (2001).
- Akasaka, K. Probing conformational fluctuation of proteins by pressure perturbation. Chemical Reviews. 106, 1814-1835 (2006).
- Kitahara, R., Yokoyama, S., Akasaka, K. NMR snapshots of a fluctuating protein structure: ubiquitin at 30 bar-3 kbar. Journal of Molecular Biology. 347, 277-285 (2005).
- Roche, J., et al. remodeling of the folding free energy landscape of Staphylococcal nuclease by cavity-creating mutations. Biochemistry. 51, 9535-9546 (2012).
- Nucci, N. V., Fuglestad, B., Athanasoula, E. A., Wand, J. A. Role of cavities and hydration in the pressure unfolding of T4 lysozyme. Proceedings of the National Academy of Sciences of the United States of America. 111, 13846-13851 (2014).
- Maeno, A., et al. Cavity as a source of conformational fluctuation and high-energy state: High-pressure NMR study of a cavity-enlarged mutant of T4 lysozyme. Biophysical Journal. 108, 133-145 (2015).
- Peterson, R. W., Wand, J. A. Self-contained high-pressure cell, apparatus, and procedure for the preparation of encapsulated proteins dissolved in low viscosity fluids for nuclear magnetic resonance spectroscopy. Review of Scientific Instruments. 76, 094101(2005).
- Goddard, T. D., Kneller, D. G. Sparky 3. , University of California San Francisco. San Francisco, CA. (2010).
- Caro, J. A., Wand, J. A. Practical aspects of high-pressure NMR spectroscopy and its applications in protein biophysics and structural biology. Methods. 148, 67-80 (2018).
- Kitamura, T., Itoh, J. Reaction volume of protonic ionization for buffering agents. Prediction of pressure dependence of pH and pOH. Journal of Solution Chemistry. 16, 715-725 (1987).
- Royer, C. A. Revisiting volume changes in pressure-induced protein unfolding. Biochimica et Biophysica Acta. 1595, 201-209 (2002).
- Erlach, M. B., et al. Relationship between nonliner pressure-induced chemical shift changes and thermodynamic parameters. Journal of Physical Chemistry B. 118, 5681-5690 (2014).
- de Oliveira, G. A. P., Silva, J. L. A hypothesis to reconcile the physical and chemical unfolding of proteins. Proceedings of the National Academy of Sciences of The United States of America. 112, 2775-2784 (2015).
- Nguyen, L. M., Roche, J. High-pressure NMR techniques for the study of protein dynamics, folding and aggregation. Journal of Magnetic Resonance. 277, 179-185 (2017).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados