Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Expériences de RMN à haute pression pour détecter les états conformationnels bas des protéines
Dans cet article
Résumé
Nous fournissons une description détaillée des étapes nécessaires à l’assemblage d’une cellule haute pression, à la mise en place et à l’enregistrement d’expériences de RMN à haute pression, et enfin à l’analyse des changements d’intensité maximale et de décalage chimique sous pression. Ces expériences peuvent fournir des informations précieuses sur les voies de repliement et la stabilité structurelle des protéines.
Résumé
La haute pression est une méthode de perturbation bien connue qui peut être utilisée pour déstabiliser les protéines globulaires et dissocier les complexes protéiques de manière réversible. La pression hydrostatique entraîne des équilibres thermodynamiques vers le(s) état(s) avec le volume molaire inférieur. L’augmentation de la pression offre donc la possibilité d’ajuster finement la stabilité des protéines globulaires et les équilibres d’oligomérisation des complexes protéiques. Les expériences de RMN à haute pression permettent une caractérisation détaillée des facteurs régissant la stabilité des protéines globulaires, leurs mécanismes de repliement et leurs mécanismes d’oligomérisation en combinant la capacité de réglage fin de la stabilité de la perturbation de la pression et la résolution du site offerte par la spectroscopie RMN en solution. Nous présentons ici un protocole pour sonder la stabilité de repliement local d’une protéine via un ensemble d’expériences 2D 1H-15N enregistrées de 1 bar à 2,5 kbar. Les étapes requises pour l’acquisition et l’analyse de telles expériences sont illustrées par les données acquises sur le domaine RRM2 de hnRNPA1.
Introduction
Il est reconnu depuis longtemps que les états conformationnels de protéines et de complexes protéiques à haute énergie et peu peuplés jouent un rôle clé dans de nombreuses voies biologiques1,2,3. Grâce à des expériences basées sur carr-Purcell-Meiboom-Gill (CPMG)4,Chemical Exchange Saturation Transfer (CEST)5et DEST(SDET)6 séquences d’impulsions (entre autres), la spectroscopie RMN en solution est apparue comme une méthode de choix pour caractériser les états conformationnels transitoires7. Parallèlement à ces expériences, des perturbations telles que la température, le pH ou les dénaturants chimiques peuvent être introduites pour augmenter la population relative de sous-états conformationnels d’énergie plus élevée. De même, les équilibres protéiques peuvent également être perturbés par l’application d’une pression hydrostatique élevée. Selon l’ampleur du changement de volume associé aux changements conformationnels correspondants, une augmentation de pression de quelques centaines à quelques milliers de bars peut stabiliser de manière significative un état d’énergie plus élevé ou provoquer le déploiement complet d’une protéine8,9,10. Les spectres rmN des protéines présentent généralement deux types de changements avec la pression hydrostatique: (i) les changements de décalage chimique et (ii) les changements d’intensité de pointe. Les changements de décalage chimique reflètent les changements à l’interface protéine-eau de surface et/ou la compression locale de la structure protéique sur une échelle de temps rapide (par rapport à l’échelle de temps RMN)11. Les croisements présentant une grande dépendance de pression de décalage chimique non linéaire peuvent indiquer la présence d’états conformationnels d’énergie plus élevés12,13. D’autre part, les changements d’intensité maximale indiquent des transitions conformationnelles majeures sur une échelle de temps lente, telles que des changements dans les populations d’États repliés / dépliés. La présence d’intermédiaires de repliement ou d’états d’énergie plus élevés peut être détectée à partir de grandes variations de l’ampleur du changement de volume lors du dépliage mesurée pour différents résidusd’uneprotéine donnée14,15,16,17. D’après notre expérience, même les petites protéines qui sont généralement classées comme des dossiers à deux états présentent des réponses non uniformes à la pression, ce qui fournit des informations utiles sur leur stabilité de repliement local. Décrit ici est un protocole pour l’acquisition et l’analyse de l’intensité maximale de l’amideet de la dépendance à la pression des changements chimiques 1 H, en utilisant comme protéine modèle le motif de reconnaissance d’ARN isolé 2 (RRM2) de la ribonucléoprotéine nucléaire hétérogène A1 (hnRNPA1).
Access restricted. Please log in or start a trial to view this content.
Protocole
REMARQUE: Le protocole décrit ici nécessite (i) une pompe et une cellule haute pression avec un tube en zircone trempé en aluminium de 2,5 kbar18,(ii) le logiciel SPARKY19 pour l’analyse des spectres RMN, et (iii) un logiciel de raccord de courbe.
1. Préparation de l’échantillon, assemblage de la cellule haute pression et mise en place des expériences.
- Choix du tampon: Utilisez un mélange égal de tampons anioniques et cationiques, tels que le phosphate et Tris20,21.
REMARQUE: Le pKa des tampons anioniques tels que le phosphate et le MES est associé à un volume de réaction important (c.-à-d. la différence entre les volumes molaux partiels de l’acide et des produits ionisés). Le pH de ces tampons peut donc être significativement affecté par un changement de pression (~0,25-0,5 unité de pH/kbar). - Assurez-vous que le volume d’échantillon requis est similaire à celui d’un tube RMN standard de 3 mm de diamètre (~300 μL).
- Introduisez l’échantillon marqué 15N avec une pipette en verre dans le tube en zircone. Assurez-vous que l’échantillon s’esso bien au bas du tube. Complétez avec 200 μL d’huile minérale pour empêcher l’échantillon de se mélanger au liquide de transmission (p. ex. eau). Remplissez le reste du tube avec du liquide de transmission.
- Placez un joint torique à usage unique sur le dessus du tube en zircone et faites glisser le tube dans la base (Figure 1A, B). Ensuite, connectez le tube à la ligne d’attache haute pression et serrez la base à la cellule d’abord à la main. Ensuite, appliquez 14,7 Nm de couple pour éviter les fuites à basse pression (Figure 1C, D).
- Pour vérifier l’intégrité de l’ensemble de la cellule de pression, pressuriser le tube jusqu’à 300 bars à l’extérieur du spectromètre à l’aide du support de cellule et du récipient de confinement. Attendez 15 minutes, réinitialisez la pression à 1 bar et vérifiez les fuites avec une lingette propre non pelucheuse.
- Insérez le tube non pressurisé dans le spectromètre en guidant soigneusement la ligne d’attache. Faites glisser le tube dans le spectromètre jusqu’à atteindre la position assise de l’échantillon (Figure 1E).
- Verrouillez, calez, assortissez et réglez les canaux 1H et 15N comme d’habitude.
REMARQUE: Les cales pour tubes en zircone haute pression sont très différentes des tubes RMN standard. Il est recommandé de sauvegarder les cales optimisées pour une utilisation ultérieure. - Mettez en place un 1H-15N-HSQC ou TROSY-HSQC et enregistrez une expérience de référence dans des conditions atmosphériques (1 bar).
2. Enregistrement d’expériences de RMN à haute pression
- Augmentez progressivement la pression de 1 bar à 2,5 kbar avec des incréments de 500 bar pour tester la stabilité globale de la protéine. Réglez la vitesse de la pompe à pression par défaut à ~18 bar/s. Si les taux de pliage/dépliage précis ne sont pas connus, laissez l’échantillon s’équilibrer 15 à 20 minutes après chaque incrément de 500 bars. Enregistrez un spectre à 2,5 kbar.
- Diminuez progressivement la pression à 1 bar avec des pas de 500 bar pour tester la réversibilité de la perturbation de pression. Enregistrez un autre spectre aux conditions atmosphériques et comparez les décalages chimiques et les intensités de crête avec ceux du spectre de référence précédemment enregistrés dans les mêmes conditions.
REMARQUE: Si les croisements natifs sont plus intenses après l’exécution de la pression, cela peut indiquer que de petits agrégats présents en solution à la pression atmosphérique peuvent s’être dissociés et correctement repliés. D’autre part, une perte d’intensité ou des changements de changement chimique importants suggèrent que la protéine peut subir un mauvais repliement non réversible dans des conditions de haute pression. - Enregistrez une série d’expériences 2D de 1 bar à 2,5 kbar tous les 500 bar. Il est recommandé d’enregistrer des expériences supplémentaires près du point d’inflexion de la transition pliage/dépliage pour améliorer la précision de l’ajustement.
3. Analyse des changements d’intensité maximale
- Traiter tous les spectres et transférer l’affectation de l’épine dorsale du spectre de référence à 1 bar au spectre enregistré à 500 bar, puis transférer l’affectation de 500 bar à 1 kbar et ainsi de suite.
REMARQUE: Étant donné que la pression induit un décalage non uniforme de 1H et 15N décalages chimiques, ne copiez pas simplement l’affectation de l’épine dorsale d’un spectre à l’autre. Ajustez-le manuellement. - Dans le menu Sparky, cliquez sur Peak > Peak List (lt). Dans la fenêtre Liste des pics, cliquez sur Options et sélectionnez l’option pour afficher à la fois fréquences (ppm) et hauteur des données. Enregistrez la liste obtenue pour chaque spectre.
- Dans un logiciel d’ajustement de courbe, copiez les valeurs d’identité de croisement et d’intensité maximale pour avoir les valeurs de pression (en bar) comme variable de l’axe X et l’intensité comme variable de l’axe Y.
- Si un dépliage complet ou presque complet (>80%) est observé, adaptez les profils d’intensité de crête individuels pour extraire l’énergie libre et le changement de volume lors du dépliage, respectivement, à l’aide d’un modèle simple à deux états:
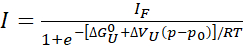 Eq. 1
Eq. 1
Où, « I » est l’intensité observée d’un crosspeak à une pression donnée p et IF est l’intensité du même crosspeak dans un état complètement plié. R est la constante de gaz, T est la température absolue, ΔGU0 la différence d’énergie libre de Gibbs standard entre les états déplié et plié à la pression atmosphérique p0 (1 bar), et ΔVu le changement de volume lors du dépliage. Avec la pression p en bar et la température T en kelvin, R = 1,987 cal/K, ΔGU0 est en cal/mol et ΔVU est en cal/mol/bar. Multipliez les valeurs ΔVU obtenues à partir de l’ajustement par 41,84 pour les convertir en mL/mol. Les valeurs ΔVU sont généralement comprises entre -50 et -150 mL/mol pour les protéines globulaires22. Ici, tous les paramètres sont exprimés en ce qui concerne la réaction qui se déroule mais peuvent facilement être convertis en paramètres de réaction de pliage (ΔGU0 = -ΔGF0 et ΔVU = -ΔVF).
4. Analyse des changements de changement chimique
- Organisez les colonnes dans le logiciel afin d’avoir des points de pression variables et les décalages chimiques 1H extraits des listes Sparky comme l’axe Y.
- Adapter la dépendance à la pression des changements chimiques 1H à une équation quadratique simple:
δ(p) = δ0(p0) + B1(p-p0) + B2 (p-p0)2 Eq. 2
Où, δ(p) est le décalage chimique mesuré de 1H d’un crosspeak à une pression donnée p et δ0(p0) les décalages chimiques 1H du même crosspeak dans le spectre de référence enregistré à 1 bar. B1 et B2 représentent les paramètres de premier et de deuxième ordre exprimés en ppm/bar et ppm/bar2, respectivement.
Access restricted. Please log in or start a trial to view this content.
Résultats
Le protocole décrit ici a été utilisé pour sonder la dépendance à la pression de RRM2, le deuxième motif de reconnaissance d’ARN de hnRNPA1 (résidus 95-106), qui est presque complètement déplié dans la plage de 2,5 kbar (>90%). 1 Les spectresH-15N ont été collectés à 1 bar, 500 bar, 750 bar, 1 kbar, 1,5 kbar, 2 kbar et 2,5 kbar(Figure 2). Étant donné qu’aucun des croisements natifs n’était visible au-dessus du niveau de bruit à 2,5 kbar, tous le...
Access restricted. Please log in or start a trial to view this content.
Discussion
Cette étude détaille un protocole mis en œuvre pour sonder les réponses structurelles et thermodynamiques des protéines à la perturbation de la pression. Les expériences à haute pression enregistrées ici sur RRM2 démontrent que de grandes variations dans les valeurs ΔVU, révélatrices d’un déploiement non entièrement coopératif, peuvent être trouvées dans une protéine relativement petite à domaine unique. Une image similaire se dégage de l’analyse des changements de décalage chimique <...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Tous les auteurs ont lu et approuvé le manuscrit. Ils ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par des fonds du Roy J. Carver Charitable Trust à Julien Roche. Nous remercions J. D. Levengood et B. S. Tolbert d’avoir aimablement fourni l’échantillon RRM2.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Bruker Nmr Cell 2.5 Kbar | Daedalus Innovations LLC | NMRCELL-B | |
| Sparky3 | University of California San Francisco, CA | N/A | |
| Xtreme-60 Syringe pump | Daedalus Innovations LLC | XTREME-60 |
Références
- Alderson, R. T., Kay, L. E. Unveiling invisible protein states with NMR spectroscopy. Current Opinion in Structural Biology. 60, 39-49 (2020).
- Korzhnev, D. M., Kay, L. E. probing invisible, low-populated states of protein molecules by relaxation dispersion NMR spectroscopy: An application to protein folding. Accounts of Chemical Research. 41, 442-451 (2008).
- Loria, P. J., Berlow, R. B., Watt, E. D. Characterization of enzyme motions by solution NMR relaxation dispersion. Accounts of Chemical Research. 41, 214-221 (2008).
- Ishima, R. CPMG relaxation dispersion. Methods in Molecular Biology. 1084, 29-49 (2014).
- Longo, D. L., et al. Chemical exchange saturation transfer (CEST): an efficient tool for detecting molecular information on proteins' behaviour. Analyst. 39, 2687-2690 (2014).
- Fawzi, N. L., Ying, J., Torchia, D. A., Clore, M. G. Probing exchange kinetics and atomic resolution dynamics in high-molecular-weight complexes using dark-state exchange saturation transfer NMR spectroscopy. Nature Protocols. 7, 1523-1533 (2012).
- Anthis, N. J., Clore, M. G. Visualizing transient dark states by NMR spectroscopy. Quarterly Reviews of Biophysics. 48, 35-116 (2015).
- Roche, J., et al. Cavities determine the pressure unfolding of proteins. Proceedings of the National Academy of Sciences of the United States of America. 109, 6945-6950 (2012).
- Chen, C. R., Makhatadze, G. I. Molecular determinant of the effects of hydrostatic pressure on protein folding stability. Nature Communications. 8, 14561(2017).
- Roche, J., Royer, C. A. Lessons from pressure denaturation of proteins. Journal of the Royal Society Interface. 15, 20180244(2018).
- Xu, X., Gagné, D., Aramini, J. M., Gardner, K. H. Volume and compressibility differences between protein conformations revealed by high-pressure NMR. Biophysical Journal. 120, 924-935 (2021).
- Akasaka, K., Li, H. Low-lying excited states of proteins revealed from non-linear pressure shifts in 1H and 15N NMR. Biochemistry. 40, 8665-8671 (2001).
- Akasaka, K. Probing conformational fluctuation of proteins by pressure perturbation. Chemical Reviews. 106, 1814-1835 (2006).
- Kitahara, R., Yokoyama, S., Akasaka, K. NMR snapshots of a fluctuating protein structure: ubiquitin at 30 bar-3 kbar. Journal of Molecular Biology. 347, 277-285 (2005).
- Roche, J., et al. remodeling of the folding free energy landscape of Staphylococcal nuclease by cavity-creating mutations. Biochemistry. 51, 9535-9546 (2012).
- Nucci, N. V., Fuglestad, B., Athanasoula, E. A., Wand, J. A. Role of cavities and hydration in the pressure unfolding of T4 lysozyme. Proceedings of the National Academy of Sciences of the United States of America. 111, 13846-13851 (2014).
- Maeno, A., et al. Cavity as a source of conformational fluctuation and high-energy state: High-pressure NMR study of a cavity-enlarged mutant of T4 lysozyme. Biophysical Journal. 108, 133-145 (2015).
- Peterson, R. W., Wand, J. A. Self-contained high-pressure cell, apparatus, and procedure for the preparation of encapsulated proteins dissolved in low viscosity fluids for nuclear magnetic resonance spectroscopy. Review of Scientific Instruments. 76, 094101(2005).
- Goddard, T. D., Kneller, D. G. Sparky 3. , University of California San Francisco. San Francisco, CA. (2010).
- Caro, J. A., Wand, J. A. Practical aspects of high-pressure NMR spectroscopy and its applications in protein biophysics and structural biology. Methods. 148, 67-80 (2018).
- Kitamura, T., Itoh, J. Reaction volume of protonic ionization for buffering agents. Prediction of pressure dependence of pH and pOH. Journal of Solution Chemistry. 16, 715-725 (1987).
- Royer, C. A. Revisiting volume changes in pressure-induced protein unfolding. Biochimica et Biophysica Acta. 1595, 201-209 (2002).
- Erlach, M. B., et al. Relationship between nonliner pressure-induced chemical shift changes and thermodynamic parameters. Journal of Physical Chemistry B. 118, 5681-5690 (2014).
- de Oliveira, G. A. P., Silva, J. L. A hypothesis to reconcile the physical and chemical unfolding of proteins. Proceedings of the National Academy of Sciences of The United States of America. 112, 2775-2784 (2015).
- Nguyen, L. M., Roche, J. High-pressure NMR techniques for the study of protein dynamics, folding and aggregation. Journal of Magnetic Resonance. 277, 179-185 (2017).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon