Method Article
Optogenética bioluminiscente 2.0: Aprovechamiento de la bioluminiscencia para activar proteínas fotosensoriales in vitro e in vivo
En este artículo
Resumen
La luz de bioluminiscencia emitida por una enzima luciferasa que oxida un sustrato de molécula pequeña, una luciferina, se puede aprovechar para activar proteínas fotosensoriales, agregando así otra dimensión a la estimulación de la luz y permitiendo la manipulación de una multitud de funciones mediadas por la luz en las células a través de escalas temporales y espaciales.
Resumen
La bioluminiscencia, la luz emitida por una enzima luciferasa que oxida un sustrato de molécula pequeña, una luciferina, se ha utilizado in vitro e in vivo para activar los canales iónicos dependientes de la luz y las bombas en las neuronas. Si bien este enfoque de optogenética bioluminiscente (BL-OG) confiere un componente quimiogenético a las herramientas optogenéticas, no se limita a su uso en neurociencia. Más bien, la bioluminiscencia se puede aprovechar para activar cualquier proteína fotosensorial, lo que permite la manipulación de una multitud de funciones mediadas por la luz en las células. Una variedad de pares luciferasa-luciferina se pueden combinar con proteínas fotosensoriales que requieren diferentes longitudes de onda de luz e intensidades de luz.
Dependiendo de la aplicación específica, se puede lograr una entrega de luz eficiente mediante el uso de proteínas de fusión luciferasa-fotorreceptor o mediante una simple co-transfección. Las proteínas fotosensoriales basadas en la dimerización dependiente de la luz o los cambios conformacionales pueden ser impulsadas por la bioluminiscencia para afectar los procesos celulares desde la localización de proteínas, la regulación de las vías de señalización intracelular hasta la transcripción. El siguiente protocolo detalla la ejecución experimental de la activación de la bioluminiscencia en células y organismos y describe los resultados utilizando recombinasas impulsadas por bioluminiscencia y factores de transcripción. El protocolo proporciona a los investigadores los procedimientos básicos para llevar a cabo la optogenética bioluminiscente in vitro e in vivo. Los enfoques descritos pueden ampliarse e individualizarse a una multitud de paradigmas experimentales diferentes.
Introducción
Las proteínas fotosensoriales pueden ser activadas por la luz de una fuente de luz física o de una enzima luciferasa en presencia de su sustrato, la luciferina, para generar bioluminiscencia. Para aplicaciones que requieren escalas de tiempo de mililitros o incluso femtosegundos y/o resolución espacial de una sola célula, las fuentes de luz físicas (láseres y diodos emisores de luz (LED)) son las únicas que se pueden ajustar a estas escalas. Ejemplos de ello son la restricción espacial de la luz utilizada para estimular polos opuestos en el desarrollo de larvas de Drosophila con control temporal de milisegundos1 o la estimulación precisa de estructuras subcelulares individuales como los túbulos mitocondriales2. Sin embargo, muchas otras aplicaciones para interruptores ópticos tienen diferentes prioridades, incluido el control espacial extendido y la aplicación repetida de forma no invasiva y sin daños por luz, pero con un control temporal definido en escalas de tiempo diminutas e intensidades sintonizables. Aquí, el uso de luciferasas como una fuente de luz alternativa para activar los dominios de detección de luz tiene varias ventajas. En contraste con la activación de la luz de la fibra óptica, la bioluminiscencia alcanza todos los dominios de detección de luz expresados en la población celular objetivo a medida que la fuente de luz está codificada genéticamente. El uso de la bioluminiscencia alivia las preocupaciones sobre el daño tisular y celular por la fibra óptica y la exposición física prolongada a la luz. La luz se enciende con la aplicación del sustrato de luciferasa. El inicio es inmediato in vitro e in vivo dependiendo de la vía de administración y dura ~15-30 min; la presencia prolongada o la estimulación fásica de la luz se puede lograr con diferentes luciferinas y con aplicaciones adicionales o repetidas de sustrato3. Por último, la emisión de bioluminiscencia se puede ajustar variando la concentración de luciferina.
Se ha demostrado ampliamente el uso de la bioluminiscencia para activar fotorreceptores en movimiento iónico, es decir, elementos optogenéticos, como las canalrodopsinas o las bombas4,5,6,7,8. Este enfoque de OptoGenética BioLuminiscente (BL-OG) se ha empleado en experimentos in vivo en ratones y ratas5,6,7,9,10,11,12. Se encontró que la activación BL-OG de opsinas requiere una cantidad de bioluminiscencia de al menos ~ 33 μW / mm2, con la eficiencia de activación aumentando con una mayor emisión de luz6,9. Los fotorreceptores sensoriales que se mueven por iones son un subgrupo del gran contingente de fotorreceptores sensoriales que se encuentran en la naturaleza y que no se mueven iones13,14. La extensión de la bioluminiscencia a la activación de fotorreceptores en movimiento no iónicos, como los dominios fotosensibilizantes de plantas o bacterias, es alentada por los informes15,16 de que los fotosensores en movimiento sin iones son significativamente más sensibles a la luz que las canalrodopsinas, lo que garantiza una unidad aún mejor de los sensores de luz con bioluminiscencia que la ya obtenida con elementos optogenéticos que se mueven por iones. Recientemente, varias publicaciones informaron sobre el uso de la bioluminiscencia como fuente de luz para la activación de una variedad de fotorreceptores, incluidos los dominios de detección de voltaje de luz-oxígeno (LOV), los dominios de luz azul que usan flavina (BLUF) y los criptocromos (CRY)3,17,18,19,20,21,22 (Tabla 1 ). Las aplicaciones para la activación impulsada por bioluminiscencia de interruptores ópticos se dirigieron a procesos intracelulares que van desde la muerte celular inducida por especies reactivas de oxígeno, la síntesis de cAMP, el reclutamiento y disociación de proteínas hasta la recombinación genómica y la inducción de la transcripción.
Este protocolo describe el diseño general de herramientas optogenéticas impulsadas por bioluminiscencia y detalla los procedimientos para la ejecución experimental de la activación de la bioluminiscencia en células y organismos. Incluye descripciones sobre cómo configurar una habitación, una campana de cultivo de tejidos y una incubadora, y un microscopio para trabajar con bioluminiscencia, así como los pasos desde la preparación de la luciferina hasta su aplicación. Este protocolo proporciona a los investigadores los procedimientos básicos para llevar a cabo la OptoGenética Bioluminiscente (BL-OG) in vitro e in vivo. Los enfoques descritos pueden ampliarse e individualizarse a diferentes paradigmas experimentales. Anticipamos que este protocolo facilitará la adopción del uso de la bioluminiscencia en estudios biológicos optogenéticos.
Protocolo
Todos los procedimientos en el estudio actual se realizaron utilizando protocolos aprobados por el Comité Institucional de Cuidado y Uso de Animales (IACUC) para el manejo de animales en la Universidad Central de Michigan, MI.
1. Activación de la bioluminiscencia de proteínas fotosensoriales in vitro
- Construcciones
- Seleccione una secuencia de luciferasa o una secuencia de fusión de proteínas luciferasa-fluorescentes que resulte en la expresión de un emisor de luz que produzca luz de una longitud de onda que coincida con el fotorreceptor que se activará.
NOTA: Por ejemplo, las luciferasas emisoras de luz azul, como las variantes de gaussia luciferasa o NanoLuc, se pueden emparejar con fotorreceptores sensibles a la luz azul como CRY / Ca2 + y proteína de unión a integrina (CIB), LOV o Vivid (VVD). - Si aún no está disponible en otros investigadores o depósitos de plásmidos, utilice técnicas estándar de biología molecular para clonar el ADN en un plásmido de expresión de mamíferos.
NOTA: La elección de los promotores está dictada por la necesidad de proporcionar una expresión fuerte y constitutiva del módulo emisor de luz, como la proporcionada por los promotores CAG y CMV. - Para los estudios iniciales, use plásmidos separados para la co-transfección del emisor de luz y el sensor de luz. Generar proteínas de fusión de las dos partes según sea necesario y para estudios posteriores.
- Obtenga ADN de plásmidos de alta calidad utilizando kits mini, midi o maxiprep de acuerdo con los protocolos del fabricante.
- Seleccione una secuencia de luciferasa o una secuencia de fusión de proteínas luciferasa-fluorescentes que resulte en la expresión de un emisor de luz que produzca luz de una longitud de onda que coincida con el fotorreceptor que se activará.
- Cultivo celular y transfección
NOTA: Las células HeLa y las células HEK293 se utilizan como ejemplos en este protocolo.- Celdas de placa en formatos y números según el uso final deseado.
NOTA: En la Tabla 2 se dan ejemplos específicos. La densidad celular en el momento del recubrimiento determinará qué tan pronto se pueden transfectar las células.- Para evaluar la transcripción activada por bioluminiscencia mediante microscopía de fluorescencia, placa HEK293 células en poli-D-lisina (PDL) recubiertas de 12 mm con fundas colocadas en platos de 24 pocillos.
- Para evaluar la transcripción activada por bioluminiscencia midiendo la emisión de luz de una luciferasa reportera ortogonal en un luminómetro, bloquee las células HeLa inicialmente en platos de 6 o 12 pocillos para la transfección, pero vuelva a placarlos después de la transfección (consulte el paso 4).
- Si se va a llevar a cabo una estimulación de bioluminiscencia repetida en cámaras de imágenes de células vivas, seleccione los cobertores del tamaño apropiado y colóquelos en placas de múltiples pocillos del tamaño apropiado (placas de 24 pocillos para latas de 12 mm; placas de 12 pocillos para latas de 15 mm y 18 mm). Sembrar las celdas en la parte superior de los coverslips usando los números de celda especificados en la Tabla 2. Si el tipo de célula seleccionado no se adhiere bien a la superficie de cultivo, coloque las células en platos recubiertos de PDL.
- Realizar la transfección por lipofección según la recomendación del fabricante o utilizar cualquier método de transfección adecuado para el tipo celular seleccionado.
NOTA: La Tabla 3 detalla los experimentos de transfección para dos fotorreceptores diferentes, EL222 y CRY2/CIB, y sus respectivos plásmidos reporteros, además de diferentes proteínas emisoras de luz. Las proporciones de los diversos plásmidos funcionan bien para los ejemplos seleccionados, pero deberán optimizarse para cada par de emisor de luz / sensor de luz. - Después de la transfección, coloque las células en una incubadora que esté completamente sellada a la luz (Figura 1).
- Dependiendo del uso final deseado, use las células para la estimulación de la bioluminiscencia al día siguiente en sus pocillos / platos originales, o vuelva a colocarlas 3-4 h después de la lipofección. Para leer la transcripción de un gen reportero de luciferasa luciérnaga en un luminómetro, vuelva a colocar las células en placas blancas de 96 pocillos.
NOTA: Realice todas las manipulaciones en una habitación hermética a la luz en una campana de flujo laminar iluminada por luz roja (Figura 2).- Lave las células transfectadas una vez con el medio Eagle modificado (DMEM) de Dulbecco o la solución salina tamponada con fosfato (PBS).
- Añadir el volumen mínimo de un reactivo tripsinizante a los pocillos (24 pocillos: 100 μL; 12 pocillos: 150 μL; 6 pocillos: 300 μL) e incubar las células durante 3 min a 37 °C.
- Agregue un medio de cultivo para lograr una concentración celular que produzca la densidad celular adecuada para el siguiente paso de recubrimiento (por ejemplo, resuspend células en un pocillo 24 en un volumen final de 1.2 mL para el recubrimiento en 10 pocillos de una placa de 96 pocillos; resuspend células en un pocillo 12 en un volumen final de 2.4 mL para el recubrimiento en 20 pocillos de una placa de 96 pocillos). Agrupe las células transfectadas de varios pozos dependiendo del número de pozos necesarios al final.
- Coloque las células transfectadas en su formato final y devuelva las placas a la incubadora protegida contra la luz.
- Celdas de placa en formatos y números según el uso final deseado.
- Activación de la bioluminiscencia in vitro
- Preparar el sustrato de luciferasa (luciferina).
- Preparar existencias de 50 mM disolviendo 5 mg de celetenterazina liofilizada (CTZ) en 250 μL de su disolvente específico. Asegúrese de que toda la CTZ a lo largo de las paredes del vial se disuelva mediante pipeteo o vórtice. Proteja el vial de la luz directa.
- Preparar alícuotas de 50 μL en tubos de microcentrífuga negra de 0,5 ml y almacenar a -80 °C para su uso futuro.
NOTA: La CTZ disuelta en disolvente no se congela a -80 °C. Las alícuotas se pueden quitar y devolver al congelador varias veces para hacer soluciones de trabajo, siempre y cuando la exposición a la luz y la temperatura ambiente se mantenga al mínimo.
- Estimulación luminiscencia de luz de bioluminiscencia única
NOTA: Todas las manipulaciones se llevan a cabo en una habitación hermética a la luz en una campana de flujo laminar iluminada por luz roja (Figura 2).- Preparar una solución de trabajo de luciferina en medio de cultivo celular (DMEM o NeuroBasal). Ajuste la concentración de la luciferina de tal manera que la concentración final sea de 100 μM. Prepare todas las diluciones de CTZ en medio poco antes de agregarlas a las células, ya que CTZ se oxida con el tiempo.
NOTA: Si se reemplaza todo el volumen del medio, la solución de trabajo será de 100 μM. Si se agrega un medio que contiene luciferina a las células, la concentración será mayor por el factor de dilución (por ejemplo, agregar 50 μL de medio que contiene 300 μM de luciferina a 100 μL de medio en el pozo dará como resultado una dilución de 1: 3 y, por lo tanto, una concentración final de luciferina de 100 μM). - Agregue un medio que contenga luciferina a las células e incube durante la duración deseada de la estimulación de la luz.
NOTA: Esto puede ser tan corto como 1 minuto o tan largo como 15 minutos y puede ser incluso más corto o más largo. El tiempo para dejar el medio que contiene luciferina en las células depende de la vida media y la cinética de la combinación seleccionada de luciferasa-luciferina. - Monitoree la emisión de luz a una concentración final de luciferina de 100 μM por el ojo después de apagar la luz roja; espere unos segundos hasta que los ojos se hayan ajustado a la oscuridad completa. Documente la emisión de luz tomando una fotografía (incluso con un teléfono celular).
- Termine la estimulación de la luz eliminando el medio que contiene luciferina y reemplazándolo con el medio de cultivo. Dependiendo de la sensibilidad de los experimentos, lave las células con medio de cultivo una o dos veces después de eliminar el medio que contiene luciferina para eliminar toda la luciferina. Si las células no se adhieren bien a la superficie de cultivo, colóquelas en platos recubiertos de PDL para evitar perder las células durante los lavados.
- Devuelva las células a la incubadora protegida contra la luz durante 16-24 h.
- Preparar una solución de trabajo de luciferina en medio de cultivo celular (DMEM o NeuroBasal). Ajuste la concentración de la luciferina de tal manera que la concentración final sea de 100 μM. Prepare todas las diluciones de CTZ en medio poco antes de agregarlas a las células, ya que CTZ se oxida con el tiempo.
- Estimulación luminiscencia de luz de bioluminiscencia repetida
NOTA: Todas las manipulaciones se llevan a cabo en una habitación que se puede hacer hermética a la luz y ser iluminada por luz roja.- Configure la cámara de imágenes de células vivas. Cree un compartimento hermético a la luz alrededor del microscopio de imágenes de células vivas utilizando una caja y láminas de plástico negras o cortinas negras (Figura 3). Cubra todas las fuentes de luz presentes dentro del compartimento hermético a la luz y la habitación (por ejemplo, indicadores LED en el microscopio o los instrumentos).
- Configure el sistema de perfusión con la solución deseada para la admisión y el puerto de salida de la cámara que conduce a un contenedor de residuos.
NOTA: Por ejemplo, la solución de imagen puede ser solución de Tyrode (cloruro de sodio (124 mM), cloruro de potasio (3 mM), HEPES (10 mM), cloruro de calcio dihidratado (2 mM), cloruro de magnesio hexahidratado (1 mM), D-glucosa (20 mM)). - Prepare una solución de trabajo de luciferina en la solución de imágenes. Alícuota en tantos tubos de microcentrífuga como el número de estimulaciones repetidas. Ajuste la concentración de la luciferina de tal manera que la concentración final en la cámara de imágenes sea de 100 μM.
- Coloque una cubierta con células transfectadas en la cámara.
- Mientras mantiene la bomba en funcionamiento, retire el tubo de entrada de la bomba del vaso de precipitados de admisión y sumérjalo rápidamente en la solución de luciferina, manteniendo el tiempo de transición lo más corto posible para evitar cualquier vacío de aire en el tubo.
- Tan pronto como se haya tomado la solución de luciferina, coloque el tubo de entrada de nuevo en el vaso de precipitados de admisión. Repita este proceso tantas veces como sea necesario y a intervalos de varios minutos a horas, dependiendo del patrón fisiológico al que se supone que las células están expuestas.
- Devolver las células a la incubadora protegida contra la luz durante 16-24 h para la transcripción, o durante el tiempo que se debe evaluar el efecto de la estimulación de la luz.
- Preparar el sustrato de luciferasa (luciferina).
2. Activación de la bioluminiscencia de proteínas fotosensoriales in vivo
- Construcciones
- Seleccione una secuencia de luciferasa o una secuencia de fusión de proteínas luciferasa-fluorescentes que resulte en la expresión de un emisor de luz que produzca luz de una longitud de onda que coincida con el fotorreceptor que se activará.
- Utilice técnicas estándar de biología molecular para clonar el ADN en un plásmido pAAV, si aún no está disponible en otros investigadores o depósitos de plásmidos.
- Elija promotores fuertes para la expresión de los módulos emisores de luz, como CAG o CMV.
- Utilice enfoques estándar para preparar existencias virales de alto título6 o prepare comercialmente vectores virales.
- Para los estudios iniciales, utilice vectores virales separados para la cotransducción del emisor de luz y el sensor de luz para permitir el ajuste de las proporciones de los diferentes componentes si es necesario.
- Transducción AAV
- Inyectar el órgano diana del animal de experimentación con vectores virales del emisor de luz, sensor de luz y informador análogos a las relaciones de concentración utilizadas para las transfecciones in vitro (Tabla 3).
- Devolver a los animales a sus jaulas de origen durante al menos 2 semanas para permitir la máxima expresión de todos los componentes.
NOTA: Si el órgano objetivo está dentro del cuerpo y protegido de la luz ambiental, los animales pueden ser alojados en condiciones normales de luz.
- Activación de la bioluminiscencia in vivo
- Preparar el sustrato de luciferasa (luciferina).
- Saque un vial de CTZ soluble en agua del congelador de -80 °C y déjelo calentar a temperatura ambiente. Manténgalo protegido de la luz.
- Por vial de 500 μg, agregue 250 μL de agua estéril, usando una jeringa o abriendo el vial y agregando agua con una pipeta, luego colocando el tapón de goma nuevamente en el vial de vidrio.
- Incubar el vial de vidrio reconstituido en un baño de agua a 55 °C durante unos minutos para disolver completamente el polvo.
- Transfiera la solución a un tubo de microcentrífuga negro. Enjuague las paredes del vial de vidrio para recuperar todo el CTZ.
- Retire la cantidad de solución necesaria para el día. Guarde la solución restante a 4 °C para usarla al día siguiente. ¡No te congeles!
- Realizar los mismos pasos (2.3.1.1.-2.3.1.5) para un vial de vehículo.
- Estimulación luminisciosa de bioluminiscencia
- Retire el volumen de luciferina/vehículo necesario para el tamaño del animal y la ruta de aplicación elegida (Tabla 4).
- Inyecte a los animales con luciferina o vehículo. Repita la estimulación de luz de bioluminiscencia según el diseño experimental. Por ejemplo, si se desea la activación de una recombinasa durante un paradigma de comportamiento específico, inyecte a los animales justo antes de la prueba de comportamiento. Si el objetivo es la transcripción fásica de una molécula, inyecte a los animales repetidamente durante días.
- Recopilar datos de los animales estimulados por bioluminiscencia según lo diseñado.
- Preparar el sustrato de luciferasa (luciferina).
Resultados
Existen numerosos eventos intracelulares que pueden ser manipulados con actuadores que responden a la luz, y que son susceptibles de activación bimodal con fuentes de luz físicas y biológicas. A continuación se presentan ejemplos que emplean un integrador de calcio fotosensible (Ca2 +), translocación de proteínas inducida por la luz, un factor de transcripción sensible a la luz y una recombinasa fotosensible. Los ejemplos ilustran la viabilidad de utilizar la bioluminiscencia para activar varios tipos de fotorreceptores. Los experimentos presentados no se optimizaron específicamente con respecto a la aplicación de diodos emisores de luz (LED), la luciferasa elegida, o con respecto a las concentraciones y el momento de la aplicación de luciferina.
La expresión rápida regulada por la luz y la actividad (FLARE) es un sistema optogenético que permite la transcripción de un gen reportero con la co-incidencia de aumento intracelular de Ca2+ y luz23 (Figura 4A). Se requiere la presencia de Ca2 + para acercar la proteasa al sitio de escisión de la proteasa accesible solo con estimulación de luz, lo que resulta en la liberación del factor de transcripción. Las células HEK293 fueron co-transfectadas con los componentes originales de FLARE, una construcción dual firefly (FLuc)-dTomato reporter, y una variante de Gaussia luciferasa anclada a la membrana sbGLuc6. En presencia de un aumento del Ca2+ intracelular a través de la exposición de las células a 2 μM de ionomicina y 5 mM de cloruro de calcio (CaCl2), la aplicación de LED azul condujo a una expresión robusta del informador de fluorescencia en comparación con las células dejadas en la oscuridad, así como a la expresión de FLuc determinada midiendo la luminiscencia al agregar el sustrato FLuc, D-luciferina. Se alcanzaron niveles similares de expresión de FLuc con la bioluminiscencia emitida por sbGLuc tras la aplicación del sustrato sbGLuc (CTZ) junto con ionomicina y CaCl2. Tenga en cuenta que las luciferasas utilizadas para la activación de la luz (sbGLuc) y para informar el efecto de la activación de la luz (transcripción de FLuc) solo producen luz con sus respectivas luciferinas (CTZ vs. D-luciferina) y no reaccionan de forma cruzada.
Se combinaron diferentes componentes para generar un sistema de transcripción inducido por la luz basado en la heterodimerización de criptocromos23,24 (Figura 4B). CRY2 se fusionó con una proteasa, mientras que el CIB unido a la membrana se fusionó con el sitio de escisión de la proteasa y el factor de transcripción. La translocación de proteínas inducida por la luz liberó el factor de transcripción, lo que llevó a la expresión de FLuc y dTomato, como se muestra en la Figura 4A. Si bien la presencia del componente del factor de transcripción por sí sola dio lugar a una señal de fondo considerable posiblemente debido a la proteólisis espontánea, tanto la luz física (LED) como la bioluminiscencia (CTZ) aumentaron considerablemente la expresión de FLuc medida en un sistema de imágenes in vivo (IVIS).
En otro conjunto de experimentos, se empleó NanoLuc (luciferina: furimazina o hCTZ) para la regulación optogenética de la transcripción a través de la dimerización de CRY/CIB y el factor de transcripción fotosensible, EL22225,26,27. La Figura 5A, B muestra los esquemas de los diferentes componentes en los estados oscuro y claro y la luciferasa co-transfectada o fusionada al sensor de luz. En la Figura 5C se muestran varias comparaciones. La bioluminiscencia, inducida por la adición de hCTZ a las células HEK293 que expresan las construcciones y la eliminan después de 15 minutos, fue más eficiente en la conducción de la transcripción del reportero que 20 minutos de exposición a la luz LED tanto para CRY / CIB como para EL222. Para CRY/CIB, una hora de exposición al LED fue suficiente para alcanzar un nivel de transcripción comparable a 15 min de bioluminiscencia. En contraste, para EL222, incluso 60 minutos de LED fueron apenas la mitad de efectivos que una breve exposición a la bioluminiscencia. No hubo diferencias significativas en la eficacia de la transcripción entre los dos sistemas cuando se co-transfectaron, aunque las proteínas de fusión de CRY/CIB fueron más eficientes que las de EL222. Para ambos sistemas, las proteínas de fusión condujeron a niveles de transcripción significativamente más altos que los componentes co-transfectados. CRY/CIB mostró niveles de fondo consistentemente más altos con la aplicación del vehículo en comparación con EL222, que tenía una transcripción de fondo insignificante. El aumento de las concentraciones de hCTZ por sí solo no tuvo ningún efecto sobre la transcripción del gen reportero.
Las recombinasas fotoactivables proporcionan una herramienta versátil para las manipulaciones optogenómicas. Probamos la activación de la bioluminiscencia de una Cre dividida fotosensible recombinasa basada en la proteína Vivid LOV, iCreV28. La Figura 6A muestra un esquema de los diferentes componentes, sbGLuc, iCreV y un reportero de fluorescencia lox-stop-lox (tdTomato) antes y después de la aplicación de CTZ. Los resultados de la aplicación de CTZ en relación con los controles (sin CTZ o LED) se muestran en la Figura 6B. Hay alguna expresión de fondo incluso en la oscuridad (sin CTZ); sin embargo, en presencia de CTZ, la expresión se incrementa robustamente sobre el fondo y es similar a la inducida con la aplicación de LED.

Figura 1: Incubadora sellada a la luz. Solapa de caja de cartón que cubre la luz del panel de control iluminado (flecha superior). Cubierta impermeable a la luz sobre la puerta de vidrio de la incubadora (flecha inferior) para proteger las células de la exposición a la luz. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Campana de flujo laminar iluminada por luz roja. Configuración que muestra una campana de cultivo de tejido de flujo laminar estándar iluminada por luz roja. La flecha indica una lámpara de escritorio estándar con una bombilla roja. Todas las manipulaciones bajo luz roja se llevan a cabo en una habitación oscura y hermética a la luz. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Compartimentos herméticos a la luz alrededor de los microscopios de imágenes de células vivas. Dos ejemplos de configuraciones de microscopio de imágenes de células vivas que muestran el uso de una caja sólida con cortinas de plástico solo en la parte frontal (paneles izquierdos: superior e inferior) o cortinas negras alrededor de la configuración de imágenes (paneles derechos: superior e inferior). Los lados frontales en ambos ejemplos permanecen abiertos y enrollados cuando no están en uso (paneles superiores: izquierda y derecha). Las cortinas negras frontales se enrollan hacia abajo para evitar que la luz en la habitación (por ejemplo, pantallas de computadora) ingrese al área de imágenes cuando se realiza estimulación de bioluminiscencia de células vivas y / o imágenes (paneles inferiores: izquierda y derecha). Haga clic aquí para ver una versión más grande de esta figura.
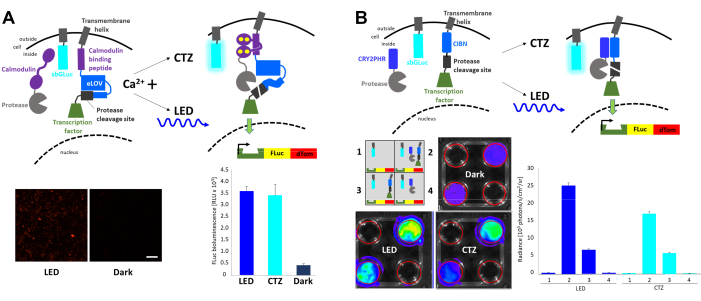
Figura 4: Bioluminiscencia para la integración de eventos de señalización intracelular. (A) Esquemas de los componentes FLARE co-transfectados con sbGLuc. En presencia de Ca2+ y la proximidad resultante de la proteasa al sitio de escisión de la proteasa, ya sea bioluminiscencia o LED conducirá al despliegue de LOV, la exposición del sitio de escisión y la liberación del factor de transcripción. Las células fueron expuestas a LED (ciclo de trabajo 33%, 2 s encendido/4 s apagado durante 40 min; potencia de luz de 3,5 mW, irradiancia de 4,72 mW/cm2 ) o a bioluminiscencia (concentración final de CTZ de 100 μM durante 15 min) o dejadas en la oscuridad. Imágenes microscópicas de células HEK293 que expresan los componentes anteriores después del tratamiento para aumentar los niveles de Ca2 + y la exposición al LED (izquierda). Luminiscencia FLuc medida en un luminómetro comparando la exposición a LED, bioluminiscencia (CTZ) o izquierda en la oscuridad (derecha). (B) Esquemas de un sistema de transcripción no dependiente de Ca2+ co-transfectado con sbGLuc. Las células HEK293 en placas de 4 pocillos se transfectaron con cuatro arreglos diferentes de componentes como se muestra en el esquema. Las placas se expusieron a LED (ciclo de trabajo 33%, 2 s encendido / 4 s apagado durante 40 min; potencia de luz de 3,5 mW, irradiancia de 4,72 mW / cm2 ) o bioluminiscencia (concentración final de CTZ de 100 μM) agregando CTZ y dejándolo encendido durante 15 min; las placas de control se dejaron en la oscuridad. La transcripción del reportero de FLuc se midió en un IVIS. Las imágenes IVIS de platos representativos se muestran a la izquierda; las mediciones de luminosidad de varias réplicas con referencia a los controles oscuros se muestran a la derecha. Barra de escala = 100 μm. Abreviaturas: FLARE = Expresión rápida regulada por la luz y la actividad; LOV = luz-oxígeno-voltaje-detección; LED = diodo emisor de luz; CTZ = coelenterazina; FLuc = luciferasa luciérnaga; dTom = dTomato; CRY2 = criptocromo 2; CRY2PHR = región de homología de la fotoliasa CRY2; CIB1 = Ca2+- y proteína de unión a integrina 1; CIBN = N-terminal de CIB1; IVIS = sistema de imágenes in vivo . Haga clic aquí para ver una versión más grande de esta figura.
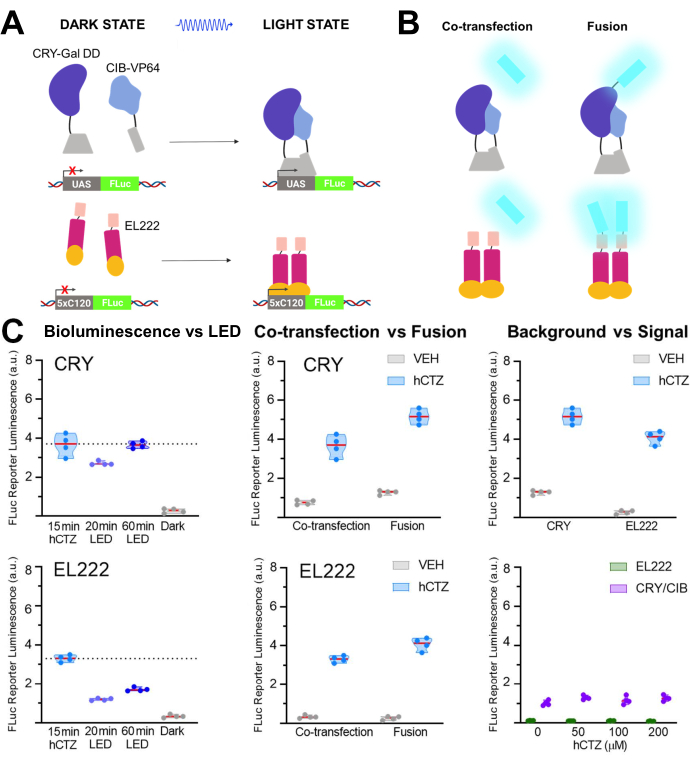
Figura 5: Bioluminiscencia para la conducción de la transcripción. (A) Esquemas de dos sistemas de transcripción fotoactivables en sus estados oscuro y claro. (B) NanoLuc fue co-transfectado o fusionado a las partes sensibles a la luz como se muestra (N-NanoLuc-CRY-GalDD-C; N-NanoLuc-VP16-EL222-C). (C) Comparaciones utilizando ambos sistemas con respecto a las fuentes de luz, el diseño de construcción y la señal al ruido. Las células fueron expuestas a LED (ciclo de trabajo 33%, 2 s encendido/4 s apagado durante 40 min; potencia ligera de 3,5 mW, irradiancia de 4,72 mW/cm2 ) o a bioluminiscencia durante 15 min (concentración final de hCTZ de 100 μM; excepto cuando se observen concentraciones diferentes). Oscuras, las placas se dejaron intactas en la incubadora entre la transformación inicial de los plásmidos y la medición de FLuc; VEH, las placas se manejaron igual que las que recibieron hCTZ, pero recibieron el vehículo en su lugar. Diferencias en los niveles de transcripción: hCTZ, CRY co-transfectado vs. EL222 - no significativo; hCTZ, luciferasa - fusión de fotoproteínas CRY vs. EL222 - p < 0,005; hCTZ, CRY co-transfección vs. fusión - p < 0,005; hCTZ, EL222 co-transfección vs. fusión - p < 0.01; vehículo, CRY vs. EL222 - p < 0.05. Abreviaturas: UAS = secuencia de activación ascendente; LED = diodo emisor de luz; CTZ = coelenterazina; FLuc = luciferasa luciérnaga; CRY = criptocromo; CIB = Ca2+- y proteína de unión a integrina; VEH = vehículo. Haga clic aquí para ver una versión más grande de esta figura.
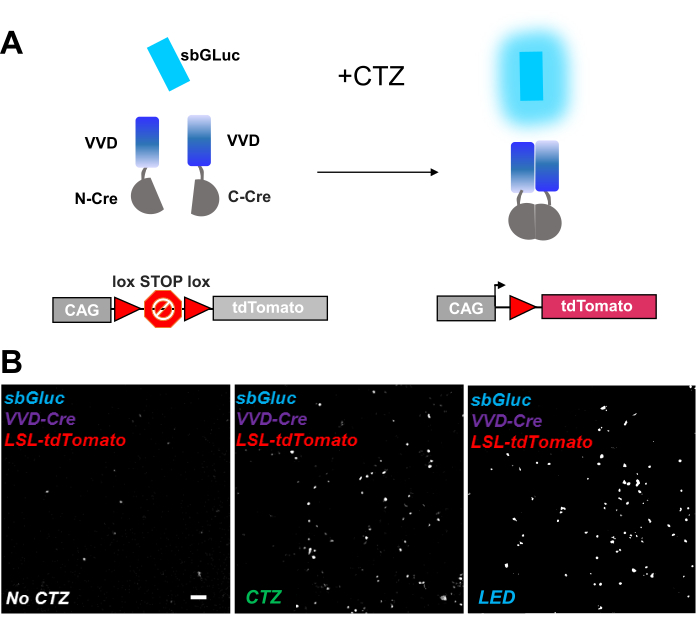
Figura 6: Bioluminiscencia para la manipulación optogenómica. (A) Esquemas de manipulación optogenómica impulsada por bioluminiscencia utilizando sbGLuc, los componentes divididos de iCreV y un casete reportero LSL, antes y después de la aplicación de luz. (B) Las células HEK293 fueron lipofectadas con plásmidos, luego mantenidas en la oscuridad. Veinticuatro horas después, las células fueron tratadas durante 30 min con solo medio (sin CTZ) o con CTZ (concentración final de 100 μM) o con LED (ciclo de trabajo 25%, 5 s encendido / 15 s apagado durante 5 min; potencia ligera de 14,81 mW, irradiancia de 20 mW / cm2 ) como control positivo. Imágenes microscópicas de fluorescencia de tdTomato utilizando las condiciones indicadas. Barra de escala = 100 μm. Abreviaturas: LSL = lox-stop-lox; CTZ = coelenterazina; LED = diodo emisor de luz; VVD = Vívido. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Activación de la bioluminiscencia de los fotorreceptores. Haga clic aquí para descargar esta tabla.
Tabla 2: Directrices para el recubrimiento y transfección de celdas en diferentes formatos. Haga clic aquí para descargar esta tabla.
Tabla 3: Proporciones de varios plásmidos para la transfección. Haga clic aquí para descargar esta tabla.
Tabla 4: Vías de inyección, volúmenes y concentraciones de luciferina para aplicaciones in vivo (ratón de 25 g). Haga clic aquí para descargar esta tabla.
Discusión
Existe una gama de luciferasas y luciferinas con longitudes de onda de emisión de luz que coinciden con los espectros de activación de las proteínas fotosensoriales de la luz azul a la roja14,29. Aparte de alinear las longitudes de onda de emisión y excitación, no hay una forma confiable de determinar a priori qué emparejamiento funcionará mejor. Por lo tanto, la necesidad de determinar experimentalmente cómo funcionan los pares luciferina-luciferasa en células y organismos en la conducción de sistemas fotosensoriales.
Los protocolos descritos en esta presentación describen cómo preparar la luciferina y cómo aplicarla in vitro e in vivo, junto con pautas sobre la configuración de salas, campanas de cultivo de tejidos, incubadoras y microscopios para experimentos que utilizan bioluminiscencia. En los experimentos representativos, se utilizaron diferentes luciferasas (NanoLuc, Gaussia luciferasa) con varias proteínas fotosensoriales (CRY/CIB, EL222, VVD, LOV), demostrando los efectos de la bioluminiscencia frente a la luz física, la coinfección frente a las proteínas de fusión, las comparaciones señal-ruido y diferentes ensayos de lectura. Se describen más aplicaciones de proteínas fotosensoriales activadoras de la bioluminiscencia en publicaciones de varios grupos, dirigidas a la inducción de la muerte celular, la síntesis de cAMP y el movimiento de proteínas, además de la transcripción (Tabla 1).
Simplemente la coinfectación de componentes emisores de luz y detección de luz es un buen comienzo. Las variables son las relaciones molares del emisor y el sensor; Las incógnitas son los niveles de fondo de la actividad del sensor en la oscuridad, la actividad del sensor en relación con la intensidad y la duración de la luz, y la eficiencia de la activación del sensor comparando la luz física y biológica. Si bien las construcciones de fusión tienen la ventaja de mantener la relación molar del emisor y el sensor en 1: 1 y acercar el emisor de luz al dominio de detección de luz, entran en juego otras consideraciones, como dónde atar (N- o C-terminal) y cómo vincular (longitud y composición del enlazador) sin afectar el rendimiento del actuador fotosensorial.
Para experimentos tanto in vitro como in vivo, existen múltiples opciones para ajustar la emisión de luz bioluminiscente, ya sea variando la concentración de la luciferina y/o variando el tiempo en que la luciferina se pone a disposición del sensor respectivo. La cantidad mínima y el tiempo están determinados por la presencia o ausencia del efecto esperado con la activación de la luz. En contraste, los máximos respectivos están determinados principalmente por la tolerancia de las células a altas concentraciones de luciferina durante tiempos prolongados. La concentración de CTZ elegida en los ejemplos anteriores, 100 μM, está cerca del límite superior para varios tipos de células, desde células HEK293 hasta neuronas. El objetivo es utilizar una concentración lo más baja posible durante el menor tiempo posible para lograr la activación del dominio de fotosensibilidad objetivo. Esto se logrará más fácilmente utilizando luciferasas con alta emisión de luz y fotorreceptores con alta sensibilidad a la luz.
La bioluminiscencia para conducir fotorreceptores se ha utilizado en roedores (ratones, ratas) con proteínas fotoséreas expresadas en el hígado, el músculo, la médula espinal y el cerebro, así como a través de células que expresan fotorreceptores trasplantadas por vía subcutánea o intraperitoneal. En principio, no hay límites que impidan que el enfoque se aplique a diferentes especies, desde primates no humanos hasta peces o moscas. Dependiendo de la permeabilidad del organismo para la luciferina, la aplicación puede ser tan fácil como aplicar la luciferina al agua circundante (por ejemplo, en larvas de peces30). Antes de usar BL-OG en cualquier organismo nuevo, se deben realizar experimentos piloto para garantizar que la luciferina alcance sus objetivos a través de la ruta de aplicación elegida.
Los aspectos críticos del diseño experimental son los diversos controles que son importantes para la interpretación de los resultados. Las células que expresan un reportero impulsado por una luciferasa que actúa sobre una proteína fotosensorial deben compararse con las células que carecen de la luciferasa o que carecen de la proteína fotosensorial. Además, se deben hacer comparaciones entre las células expuestas a la luciferina, el vehículo o mantenidas en la oscuridad. También es importante darse cuenta de las limitaciones de los diferentes ensayos para evaluar los efectos de la activación de fotorreceptores impulsada por bioluminiscencia. Por ejemplo, la eficacia de la transcripción activada por bioluminiscencia se puede probar de diferentes maneras, dependiendo de si el gen reportero es una luciferasa ortogonal (luminómetro, IVIS) o una proteína fluorescente (clasificación celular activada por fluorescencia, análisis de imágenes microscopía). Si bien los efectos básicos deben ser reproducibles en todas las plataformas de prueba, los aspectos cuantitativos de los efectos pueden variar considerablemente.
La activación de la bioluminiscencia de los fotorreceptores se ha demostrado hasta ahora para un número limitado de luciferasas y proteínas fotosensoriales, respectivamente, tanto in vitro como in vivo. Se puede extender a la gran clase de fotorreceptores para activar muchos más procesos biológicos. Tal expansión del enfoque se promueve aún más por el desarrollo continuo de nuevas luciferasas y pares de proteínas luciferasa-fluorescencia con una emisión de luz mucho mayor que las luciferasas naturales y con características cinéticas sintonizables para diferentes aplicaciones. Estos avances son paralelos a la generación de nuevas luciferinas, lo que aumenta aún más el brillo y las paletas de colores29. Esta plataforma de herramientas ofrece aplicaciones para manipular e investigar la dinámica intracelular y las interacciones celulares dentro de células, tejidos y organismos vivos.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Agradecemos a nuestros colegas por las construcciones, específicamente a A. Ting para la proteasa Ca-FLARE, el factor de transcripción y el reportero (Addgene # 92214, 92213, 92202), H. Kwon para TM-CIBN-BLITz1-TetR-VP16 y NES-CRY2PHR-TevC (Addgene # 89878, 89877), C. Tucker para CRY-GalΔDD (B1013) y CIB-VP64 (B1016) (Addgene # 92035, 92037), M. Walsh por pGL2-GAL4-UAS-Luc (Addgene # 33020), K. Gardner por VP-EL222 y C120-Fluc, y A. Cetin y H. Zeng por hacer que iCreV esté disponible antes de la publicación. Este trabajo fue apoyado por subvenciones de NSF (NeuroNex 1707352), NIH (U01NS099709), la Fundación W.M. Keck y el Consejo Sueco de Investigación a A.B. (2016-06760).
Materiales
| Name | Company | Catalog Number | Comments |
| ABI 25W Deep Red 660 nm LED Light Bulb | Amazon | to be used with any lamp stand | |
| Black Microcentrifuge Tubes, 0.5 mL, Argos Technologies | Fisher Scientific | 03-391-166 | |
| Black Microcentrifuge Tubes, 1.5 mL, Argos Technologies | Fisher Scientific | 03-391-161 | |
| Black Nylon, Polyurethane-Coated Fabric (1.5 m x 2.7 m) x 0.12 mm (thick) | THOR LABS | BK-5 | |
| C120-Fluc | K. Gardner | ||
| CaCl2 | Sigma | C8106; CAS: 10035-04-8 | |
| Ca-FLARE protease, transcription factor and reporter | Addgene # 92214, 92213, 92202 | A. Ting | |
| CIB-VP64 (B1016) | Addgene # 92037 | C. Tucker | |
| CRY-GalΔDD (B1013) | Addgene # 92035 | C. Tucker | |
| CTZ | Prolume Inc. (NanoLight) | 303 | formulation for in vitro applications with Gaussia luciferases |
| CTZ (Water soluble native coelenterazine) | Prolume Inc. (NanoLight) | 3031 | formulation for in vivo applications with Gaussia luciferases |
| D-(+)-Glucose | Sigma | G8270; CAS: 50-99-7 | |
| D-Luciferin, Potassium Salt | Gold Biotechnology | LUCK | |
| DMEM | Thermo Fisher | 11960044 | |
| D-PBS, no calcium, no magnesium | Thermo Fisher | 14190144 | |
| hCTZ | Prolume Inc. (NanoLight) | 301 | formulation for in vitro applications with Oplophorus luciferases |
| HEK293 | ATCC | CRL-1573 | |
| HeLa | ATCC | CCL-2 | |
| HEPES | Sigma | H3375; CAS: 7365-45-9 | |
| iCreV | A. Cetin and H. Zeng | ||
| In Vivo Imaging System (IVIS) | Perkin-Elmer | Lumina LT | |
| KCl | Sigma | P5405; CAS: 7447-40-7 | |
| LED Array Driver | Amuza | LAD-1 | |
| LED Array for Multiwell Plates | Amuza | LEDA-x | |
| Lipofectamine 2000 Reagent | Invitrogen | 11668-019 | Transfection reagent |
| Luminometer | Molecular Devices | SpectraMax L | |
| MgCl2 Hexahydrate | Sigma | M2670; CAS: 7791-18-6 | |
| NaCl | Sigma | S7653; CAS: 7647-14-5 | |
| NanoFuel Solvent | Prolume Inc. (NanoLight) | 399 | for dissolving CTZ preparations for in vitro use |
| NaOH | Sigma | 221465; CAS: 1310-73-2 | |
| NES-CRY2PHR-TevC | Addgene # 89877 | H. Kwon | |
| Opti-MEM | Thermo Fisher | 11058021 | transfection medium |
| PDL coated coverslips (12 mm, 15 mm, 18 mm) | Neuvitro Corporation | GG-12-PDL, GG-15-PDL , GG-18-PDL | |
| pGL2-GAL4-UAS-Luc | Addgene #33020 | M. Walsh | |
| Prizmatix USB Pulser TTL Generator for Optogenetics | Goldstone Scientific | ||
| TM-CIBN-BLITz1-TetR-VP16 | Addgene # 89878 | H. Kwon | |
| TrypLE Express | Gibco | 12604-013 | |
| Vehicle (Water-soluble carrier without CTZ) | Prolume Inc. (NanoLight) | 3031C | control for in vivo applications with CTZ |
| VP-EL222 | K. Gardner |
Referencias
- Johnson, H. E., et al. The spatiotemporal limits of developmental Erk signaling. Developmental Cell. 40 (2), 185-192 (2017).
- Wang, Y., et al. Photostimulation by femtosecond laser triggers restorable fragmentation in single mitochondrion. Journal of Biophotonics. 10 (2), 286-293 (2017).
- Li, T., et al. A synthetic BRET-based optogenetic device for pulsatile transgene expression enabling glucose homeostasis in mice. Nature Communications. 12 (1), 615(2021).
- Berglund, K., Birkner, E., Augustine, G. J., Hochgeschwender, U. Light-emitting channelrhodopsins for combined optogenetic and chemical-genetic control of neurons. PLoS One. 8 (3), 59759(2013).
- Tung, J. K., Gutekunst, C. -A., Gross, R. E. Inhibitory luminopsins: genetically-encoded bioluminescent opsins for versatile, scalable, and hardware-independent optogenetic inhibition. Scientific Reports. 5, 14366(2015).
- Berglund, K., et al. Luminopsins integrate opto- and chemogenetics by using physical and biological light sources for opsin activation. Proceedings of the National Academy of Sciences of the United States of America. 113 (3), 358-367 (2016).
- Gomez-Ramirez, M., More, A. I., Friedman, N. G., Hochgeschwender, U., Moore, C. I. The BioLuminescent-OptoGenetic in vivo response to coelenterazine is proportional, sensitive and specific in neocortex. Journal of Neuroscience Research. 98 (3), 471-480 (2020).
- Moore, C. I., Berglund, K. BL-OG: BioLuminescent-OptoGenetics. Journal of Neuroscience Research. 98 (3), 469-470 (2020).
- Park, S. Y., et al. Novel luciferase-opsin combinations for improved luminopsins. Journal of Neuroscience Research. 98 (3), 410-421 (2020).
- Jaiswal, P. B., Tung, J. K., Gross, R. E., English, A. W. Motoneuron activity is required for enhancements in functional recovery after peripheral nerve injury in exercised female mice. Journal of Neuroscience Research. 98 (3), 448-457 (2020).
- Zenchak, J. R., et al. Bioluminescence-driven optogenetic activation of transplanted neural precursor cells improves motor deficits in a Parkinson's disease mouse model. Journal of Neuroscience Research. 98 (3), 458-468 (2020).
- Tung, J. K., Shiu, F. H., Ding, K., Gross, R. E. Chemically activated luminopsins allow optogenetic inhibition of distributed nodes in an epileptic network for non-invasive and multi-site suppression of seizure activity. Neurobiology of Disease. 109, Pt A 1-10 (2018).
- Hegemann, P. Algal sensory photoreceptors. Annual Review of Plant Biology. 59, 167-189 (2008).
- Losi, A., Gardner, K. H., Moglich, A. Blue-light receptors for optogenetics. Chemical Reviews. 118 (21), 10659-10709 (2018).
- Proshkina, G. M., Shramova, E. I., Shilova, O. N., Ryabova, A. V., Deyev, S. M. Phototoxicity of flavoprotein miniSOG induced by bioluminescence resonance energy transfer in genetically encoded system NanoLuc-miniSOG is comparable with its LED-excited phototoxicity. Journal of Photochemistry and Photobiology B: Biology. 188, 107-115 (2018).
- Kawano, F., Okazaki, R., Yazawa, M., Sato, M. A photoactivatable Cre-loxP recombination system for optogenetic genome engineering. Nature Chemical Biology. 12 (12), 1059-1064 (2016).
- Shramova, E. I., Proshkina, G. M., Chumakov, S. P., Khodarovich, Y. M., Deyev, S. M. Flavoprotein miniSOG cytotoxisity can be induced by bioluminescence resonance energy transfer. Acta Naturae. 8 (4), 118-123 (2016).
- Naim, N., et al. Luminescence-activated nucleotide cyclase regulates spatial and temporal cAMP synthesis. Journal of Biological Chemistry. 294 (4), 1095-1103 (2019).
- Kim, C. K., Cho, K. F., Kim, M. W., Ting, A. Y. Luciferase-LOV BRET enables versatile and specific transcriptional readout of cellular protein-protein interactions. Elife. 8, 43826(2019).
- Parag-Sharma, K., et al. Engineered BRET-based biologic light sources enable spatiotemporal control over diverse optogenetic systems. ACS Synthetic Biology. 9 (1), 1-9 (2020).
- Kim, E. H., et al. Self-luminescent photodynamic therapy using breast cancer targeted proteins. Science Advances. 6 (37), (2020).
- Kim, C. K., et al. A Molecular calcium integrator reveals a striatal cell type driving aversion. Cell. 183 (7), 2003-2019 (2020).
- Wang, W., et al. A light- and calcium-gated transcription factor for imaging and manipulating activated neurons. Nature Biotechnology. 35 (9), 864-871 (2017).
- Lee, D., Hyun, J. H., Jung, K., Hannan, P., Kwon, H. -B. A calcium- and light-gated switch to induce gene expression in activated neurons. Nature Biotechnology. 35 (9), 858-863 (2017).
- Pathak, G. P., et al. Bidirectional approaches for optogenetic regulation of gene expression in mammalian cells using Arabidopsis cryptochrome 2. Nucleic Acids Research. 45 (20), 167(2017).
- Nishio, H., Walsh, M. J. CCAAT displacement protein/cut homolog recruits G9a histone lysine methyltransferase to repress transcription. Proceedings of the National Academy of Sciences of the United States of America. 101 (31), 11257-11262 (2004).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Yao, S., et al. RecV recombinase system for in vivo targeted optogenomic modifications of single cells or cell populations. Nature Methods. 17 (4), 422-429 (2020).
- Love, A. C., Prescher, J. A. Seeing (and using) the light: Recent developments in bioluminescence technology. Cell Chemical Biology. 27 (8), 904-920 (2020).
- Naumann, E. A., Kampff, A. R., Prober, D. A., Schier, A. F., Engert, F. Monitoring neural activity with bioluminescence during natural behavior. Nature Neuroscience. 13 (4), 513-520 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados