Method Article
Caracterización de modificaciones de ARN en neuronas individuales mediante espectrometría de masas
En este artículo
Resumen
Las modificaciones post-transcripcionales del ARN representan una capa poco estudiada de regulación de la traducción que recientemente se ha relacionado con la plasticidad del sistema nervioso central. Aquí, se describe la preparación de muestras y el enfoque de cromatografía líquida-espectrometría de masas en tándem para la caracterización simultánea de numerosas modificaciones de ARN en neuronas individuales.
Resumen
Las modificaciones post-transcripcionales (PTM) del ARN representan un mecanismo poco estudiado involucrado en la regulación de la traducción en el sistema nervioso central (SNC). La evidencia reciente ha relacionado modificaciones específicas del ARN neuronal con paradigmas de aprendizaje y memoria. Desafortunadamente, los métodos convencionales para la detección de estas características epitranscriptómicas solo son capaces de caracterizar modificaciones de ARN altamente abundantes en tejidos a granel, lo que impide la evaluación de perfiles únicos de PTM que pueden existir para neuronas individuales dentro de los circuitos conductuales activados. En este protocolo, se describe un enfoque, el análisis de modificación de ARN de una sola neurona por espectrometría de masas (SNRMA-MS), para detectar y cuantificar simultáneamente numerosos ribonucleósidos modificados en neuronas individuales. El enfoque se valida utilizando neuronas individuales del molusco marino, Aplysia californica, comenzando con el aislamiento quirúrgico y el tratamiento enzimático de los principales ganglios del SNC para exponer los cuerpos celulares de las neuronas, seguido por el aislamiento manual de una sola neurona utilizando agujas afiladas y una micropipeta. A continuación, se realiza un tratamiento mecánico y térmico de la muestra en un pequeño volumen de tampón para liberar ARN de una célula individual para la posterior digestión del ARN. Los nucleósidos modificados se identifican y cuantifican utilizando un método optimizado de cromatografía líquida-espectrometría de masas. SNRMA-MS se emplea para establecer patrones de modificación de ARN para neuronas individuales e identificadas de A. californica que tienen morfologías y funciones conocidas. Se presentan ejemplos de SNRMA-MS cualitativos y cuantitativos que destacan la distribución heterogénea de las modificaciones de ARN a través de neuronas individuales en redes neuronales.
Introducción
Las modificaciones en los nucleósidos canónicos del ARN han sido cada vez más reconocidas por sus innumerables funciones en la regulación de la traducción de proteínas. Hasta la fecha se han reportado más de 150 modificaciones únicas de ARN que varían en complejidad desde la metilación, la adición de heteroátomos hasta la conjugación con metabolitos celulares 1,2. Este alfabeto de ARN expandido, también conocido como epitranscriptome, es generado por escritores enzimáticos y es responsable de alterar la estabilidad3, el plegado4 y la eficiencia de traducción 5,6 de los ARN celulares. Las modificaciones seleccionadas de ARN también pueden revertirse a través de borradores enzimáticos 7,8, mientras que otras se agregan a los ARN subestoquiométricamente 9,10, lo que representa un panorama complejo de secuencias de ARN modificadas y no modificadas en sistemas biológicos.
La importancia de las modificaciones del ARN en la función biológica está indicada por la distribución desigual de las modificaciones entre los diferentes órganos y tejidos, incluidas las subregiones del sistema nervioso central (SNC)11. Esta heterogeneidad química se ha relacionado con el desarrollo del SNC12, la respuesta al estrés13 y la plasticidad dependiente de la actividad14. Las subregiones del SNC comprenden además poblaciones heterogéneas de células en las que las células individuales exhiben perfiles químicos distintos 15,16,17,18. Incluso las células individuales del mismo tipo pueden mostrar transcriptomas únicos, en parte debido a los microambientes tisulares19 y la expresión génica estocástica20. Sin embargo, aunque la caracterización del transcriptoma unicelular es algo rutinaria, no existen métodos análogos para establecer epitranscriptomas unicelulares para múltiples modificaciones de ARN. Se necesitan nuevos enfoques que sean capaces de perfilar la distribución de las modificaciones del ARN en células individuales para un análisis exhaustivo de la heterogeneidad celular y la influencia reguladora de las modificaciones post-transcripcionales (PTM) en el SNC y otros sistemas biológicos.
La medición simultánea de numerosas modificaciones de ARN en células / tejidos a granel se logra fácilmente utilizando técnicas de cromatografía líquida-espectrometría de masas en tándem (LC-MS / MS). Para el análisis LC-MS/MS de ribonucleósidos modificados, el ARN se extrae de las células (típicamente 103-10 6 células), se purifica por precipitación y resuspensión, y posteriormente se digiere en nucleósidos. La mezcla de muestra que consiste en nucleósidos canónicos y modificados se inyecta en el sistema LC-MS para la separación y detección de analitos, lo que lleva a la determinación del complemento completo de modificaciones de ARN en un organismo 21,22,23. LC-MS/MS se utilizó recientemente para determinar 26 modificaciones de ARN en el SNC del modelo neurobiológico, Aplysia californica (A. californica). Las abundancias de algunas de estas marcas epitranscriptómicas exhibieron dinámicas dependientes del tiempo y la región que se correlacionaron con cambios de comportamiento en el animal24. Sin embargo, solo fue posible detectar modificaciones de ARN en muestras a granel que contenían >103 células debido a la sensibilidad limitada del método. Estas muestras más grandes probablemente ocultaron perfiles de modificación de ARN únicos y funcionalmente importantes de células individuales con promedios poblacionales. Aunque el control cuidadoso de las condiciones de preparación de muestras ha mejorado los límites de detección de modificaciones de ARN en muestras de pequeño volumen 25,26,27,28, sigue siendo necesario métodos analíticos que puedan detectar y cuantificar múltiples ribonucleósidos modificados en células individuales.
Este protocolo introduce el análisis de modificación de ARN de una sola neurona por espectrometría de masas (SNRMA-MS), que permite la detección de más de una docena de modificaciones de ARN en neuronas individuales del SNC de A. californica29. El enfoque consiste en el aislamiento quirúrgico de células individuales identificadas de los principales ganglios del SNC, seguido de un flujo de trabajo optimizado de preparación de muestras que involucra lisis celular mecánica, desnaturalización de ARN e hidrólisis enzimática en un tampón compatible con EM. La identificación y cuantificación de nucleósidos modificados post-transcripcionalmente se logra utilizando LC-MS / MS. SNRMA-MS satisface una necesidad insatisfecha en el campo del análisis de modificación de ARN al facilitar la adquisición de perfiles de modificación de ARN post-transcripcionales para neuronas individuales y tiene potencial para su aplicación futura a otros tipos de células.
Protocolo
1. Preparación de materiales y soluciones
- Preparar agua de mar artificial (ASW) con 460 mM NaCl, 10 mM KCl, 10 mM CaCl2, 22 mM MgCl2, 26 mM MgSO4 y 10 mM HEPES en agua obtenida de un estricto sistema de purificación. Ajuste el pH a 7.8 usando 1 M NaOH o HCl. Por lo general, prepare 1 L de ASW y guárdelo a 14 ° C hasta su uso.
- Preparar una solución de antibióticos ASW con 10.000 U/ml de penicilina G, 10 μg/μL de estreptomicina y 10 μg/μL de gentamicina y conservar a -20 °C. Inmediatamente antes del experimento, descongele y diluya la solución madre de antibióticos congelados 1:100 en 20-40 ml de ASW. La concentración final de antibióticos en la solución de ASW de trabajo es de 100 U/mL de penicilina G, 100 μg/mL de estreptomicina y 100 μg/mL de gentamicina.
- Preparar un tampón de digestión de ARN combinando 1 μL de 10 μg/μL de albúmina sérica bovina, 0,5 μL de 0,5 μg/μL de pentostatina, 0,495 μL de fosfatasa alcalina U/μL, 1 μL de fosfodiesterasa I (en 10 mM MgCl2) y 0,38 μL de endonucleasa de Serratia marcescens (25 U) por muestra. Si analiza varias muestras, prepare una mezcla maestra en un tubo separado que contenga el volumen de cada reactivo multiplicado por el número de muestras a analizar, más una más para tener en cuenta la pérdida incidental de reactivos de los pasos de transferencia de pipetas.
- Prepare varias agujas afiladas (ya sea de vidrio o metal) para el aislamiento manual de las neuronas. Haga agujas de metal en casa mediante grabado electroquímico de alambre de tungsteno como en30 o cómprelos. Prepare agujas de vidrio a partir de capilares de vidrio de borosilicato de pared gruesa o estándar (diámetro exterior de 1 mm) utilizando un extractor de micropipetas. Para el presente método, mantenga el diámetro de la punta de la aguja entre 1-5 μm, con una longitud de cuello de 100-150 μm.
NOTA: La fabricación de agujas de metal y vidrio se puede optimizar para producir herramientas que se adapten a las necesidades individuales del investigador y al modelo biológico específico que se está investigando.
2. Aislamiento de una sola neurona
- Enfriar una solución de MgCl2 de 0,33 M a 14 °C. Usando una jeringa de 50 ml, anestesia A. californica (150-250 g) inyectando la solución de MgCl2 en la cavidad corporal del animal. Los mejores resultados se obtienen con una relación de 1:3 entre el volumen de solución (ml) y la masa corporal animal (g). Espere aproximadamente 3 minutos para que el animal se relaje, asegurándose de que no muestre contracciones corporales en respuesta a la estimulación táctil.
- Coloque el lado ventral del animal (lado del pie) hacia arriba en una bandeja de disección. Diseccionar al animal usando tijeras quirúrgicas con una punta roma colocada hacia el animal, haciendo cuidadosamente un corte longitudinal a través del pie.
- Fije los lados rostral, caudal y lateral del cuerpo del animal para exponer los órganos internos y los ganglios del SNC ubicados en la cavidad corporal. Aislar los ganglios principales del SNC del animal cortando quirúrgicamente los nervios y algunos conectivos que se originan en los ganglios.
- Sumergir los ganglios en una solución de proteasa tipo XIV de Streptomyces griseus (10 mg/ml en solución de antibióticos ASW) e incubar a 34 °C durante 30 min o 1 h (para ganglio cerebral).
NOTA: La duración de la incubación depende de la temporada, el tamaño y la condición del animal, así como de las neuronas objetivo. El aislamiento de algunas neuronas requiere una incubación más larga que otras y debe determinarse experimentalmente. - Enjuague los ganglios 6x con solución de antibióticos ASW y transfiera todos los ganglios en un plato recubierto de polímero de silicona lleno de solución de antibióticos ASW con una pipeta de transferencia de polipropileno que se ha cortado a una abertura de ~ 5 mm. Trate la pipeta con 1 mg/ml de albúmina sérica bovina en ASW para minimizar la adherencia de los ganglios a la pipeta (opcional). Mantenga los ganglios sumergidos en ASW en todo momento.
NOTA: El tratamiento enzimático reduce la estabilidad mecánica de las neuronas y el tejido conectivo circundante y, como resultado, las membranas neuronales pueden dañarse debido a la exposición al aire durante la transferencia de ganglios. - Fije los ganglios y use micro tijeras y fórceps finos para eliminar las vainas ganglionares. Con un tratamiento enzimático suficientemente fuerte, use agujas de vidrio o metal para desenvainar.
- Identificar visualmente las neuronas de interés de A. californica . En este trabajo se estudiaron las siguientes células: R2 en el ganglio abdominal, LPl1 en el ganglio pleural, células metacerebrales (CCM) en el ganglio cerebral y células B2 en el ganglio bucal. Tome imágenes ópticas de todas las preparaciones neuronales y ganglionares utilizando un microscopio calibrado a un aumento total de 20x para determinar los tamaños y volúmenes de cada tipo de célula.
- Usando un capilar de vidrio tirado o agujas de tungsteno afiladas, aísle cuidadosamente la célula identificada del ganglio a granel.
- Extraiga una pequeña cantidad (1 μL) de ASW en una micropipeta de plástico y luego transfiera la célula aislada a un tubo de muestra de PCR que contenga 4 μL de acetato de amonio de 0.365 M (pH 9.2). Para mediciones en blanco, recolecte 5 μL de alícuotas de la solución de antibióticos ASW del plato que contiene el ganglio y mezcle con el tampón de digestión (descrito a continuación).
3. Lisis celular y digestión de ARN para SNRMA-MS
- Lisar las neuronas aisladas mediante aspiración repetida y prescindir de una micropipeta (~100 μm de diámetro interior) en 0,365 M de acetato de amonio. Algunas células más pequeñas pueden no romperse inmediatamente; para lisarlos, aplique presión a través del diámetro de la celda con un capilar de vidrio tirado.
- Utilice un termociclador para calentar las muestras con el siguiente programa de temperatura: 95 °C durante 3 min, 10 °C durante 3 min, mantener a 10 °C. Retire el tubo de muestra del termociclador.
- Añadir 3.375 μL de tampón de digestión de ARN para cada muestra y mezclar la solución con una micropipeta retirando y dispensando la solución varias veces. Use una centrífuga de sobremesa en miniatura a 2700 x g durante 30 s para hacer girar cualquier gota líquida que se adhiera a las paredes del tubo de PCR.
- Incubar las muestras en el termociclador a 37 °C durante 3 h, seguido de una retención a 10 °C (tapa calentada ajustada a ON). Inmediatamente después de que la muestra se haya enfriado a 10 °C, transfiera 7 μL de la solución a un vial de automuestreador equipado con un inserto de 250 μL, teniendo cuidado de evitar la formación de burbujas en el tubo de automuestreador.
4. Cromatografía líquida-espectrometría de masas en tándem
NOTA: Analice los resúmenes de una sola neurona y los estándares de nucleósidos modificados auténticos utilizando un sistema LC-MS / MS equipado con una fuente de ionización de electropulverización y una válvula desviadora de seis puertos.
- Para preparar el sistema LC para la separación de nucleósidos canónicos y modificados, equilibre una columna C18 (150 mm x 2,1 mm, tamaño de partícula de 2,2 μm, diámetro de poro de 120 Å) con 99% de fase A móvil (5 mM de acetato de amonio, pH 5,6) y 1% de fase B móvil (fase móvil 60/40 A/acetonitrilo (ACN)) a un caudal de 0,2 mL/min durante 12 min a 36 °C. Utilice disolventes de grado LC-MS para la preparación de fases móviles.
- Mientras el LC equilibra, calibre el espectrómetro de masas introduciendo una solución de 1 mM de acetato de sodio en 50/50 ACN/agua al espectrómetro de masas a través de una bomba de jeringa con un caudal de 5 μL/min. Después de la calibración, vuelva a conectar el flujo LC al espectrómetro de masas.
- Programe los siguientes parámetros de gradiente lineal: 1% B durante 0-5 min, 5% B a 9 min, 7% B a 11 min, 10% B a 13 min, 15% B a 32 min, 40% B a 38 min, 50% B a 43 min, 100% B a 50 min, 100% B a 60 min, 1% B a 61 min, y un reequilibrio de 12 minutos al 1% B antes de la siguiente inyección.
- Opere el instrumento MS en modo positivo con los siguientes parámetros: voltaje capilar ajustado a 4.500 V, temperatura de secado 275 °C, gas de secado N2 5 L/min y gas nebulizante 1 bar. Ajuste la válvula de desvío a residuos durante los primeros 2 minutos de análisis y a la fuente durante el resto de la ejecución.
- Recopile espectros de masas en un rango m /z de 110-600. Seleccione iones para la disociación inducida por colisión a 35-40 eV durante un tiempo de ciclo de 3 s utilizando una lista de masas preferida construida utilizando la base de datos2 y una ventana de aislamiento de ± 0.5. Utilice la exclusión activa para excluir los iones de la fragmentación después de tres espectros.
- Establezca la adquisición dinámica de espectros MS/MS para iones con intensidades por encima y por debajo de 50.000 recuentos a 4 Hz y 1 Hz, respectivamente, y un umbral mínimo para la selección de iones a 1.990 recuentos.
- Para SNRMA-MS cuantitativo, construya curvas de calibración utilizando áreas pico de cromatograma iónico (EIC) extraídas obtenidas para estándares de nucleósidos modificados a un mínimo de cinco concentraciones para permitir la interpolación de concentraciones de analitos endógenos desconocidos.
NOTA: Las neuronas obtenidas de animales con masas corporales de 150-250 g generalmente requieren curvas de calibración para nucleósidos modificados que van desde 0.02 pmol a 2 pmol, pero estos valores pueden variar dependiendo de la sensibilidad del instrumento.
5. Análisis de datos
- Generar EIC para nucleósidos modificados (m/z a partir de los valores de la base de datos2). Verificar las identidades de los nucleósidos modificados putativos comparando sus espectros MS2 y características de retención LC con los valores de la base de datos2. Consulte la Tabla 1 para obtener una lista de las modificaciones típicas del ARN detectadas en neuronas individuales de A. californica.
- Integre manualmente los picos que corresponden a nucleósidos modificados y canónicos y registre estos valores en una hoja de cálculo. Normalice el área de pico para cada nucleósido modificado con la suma de las áreas de pico para citidina canónica, uridina y guanosina detectadas en la muestra.
NOTA: La adenosina no está incluida en la normalización debido a su papel en el SNC como neuromodulador dinámico31. - Construir una matriz de datos que consista en cada muestra de una sola neurona y las correspondientes áreas de pico normalizadas para nucleósidos modificados que exhibieron relaciones señal-ruido >10. Realice el análisis de componentes principales (PCA) y muestre los dos primeros componentes principales en el gráfico de puntuación. Construir curvas de calibración lineales a partir de las áreas de pico EIC obtenidas de la dilución en serie de los estándares de nucleósidos y calcular las concentraciones de analitos detectados.
Resultados
SNRMA-MS implica el aislamiento manual de neuronas identificadas en pequeños volúmenes de muestra para lisis, digestión y análisis LC-MS / MS (Figura 1A). Este flujo de trabajo detectó rutinariamente más de una docena de modificaciones de ARN en neuronas individuales del SNC de A. californica (Figura 1B), lo que representa una cobertura de casi la mitad del epitranscriptoma conocido de este animal24 en una sola célula. Por ejemplo, someter la neurona LPl1 (~500 μm de diámetro) a SNRMA-MS resultó en la detección de 15 ± 1 modificaciones de ARN (n = 3). Los nucleósidos modificados se identificaron positivamente sobre la base de tres atributos: propiedades de retención de LC, masa exacta y perfiles de fragmentación MS2 en comparación con los valores proporcionados en la base de datos2. La Tabla 1 muestra una lista de todas las modificaciones de ARN detectadas en la neurona LPl1. El espectrómetro de masas de tiempo de vuelo cuadrupolo de alta resolución utilizado para estos experimentos permitió una precisión de masa de 4 ppm, así como la detección del ion fragmento MS2 característico en m/z 164 para el nucleósido modificado N6,6-dimetiladenosina (m66A, Figura 1B). Combinado con los datos de separación de LC, que concuerdan con los hallazgos depositados en la base de datos (m66A elutes después de N6-metiladenosina (m6A)), el enfoque SNRMA-MS demostró la asignación correcta de identidades de nucleósidos modificadas.
La plataforma SNRMA-MS se puede aprovechar para establecer perfiles de modificación de ARN de neuronas individuales e investigar su relación con los tejidos a granel. Las neuronas R2 son células colinérgicas grandes (~500 μm de diámetro) que residen en el ganglio abdominal. SNRMA-MS se utilizó para analizar las modificaciones de ARN en las neuronas R2, así como el ganglio abdominal masivo circundante (Figura 2A). Las áreas pico normalizadas para las modificaciones de ARN en cada muestra de neurona/ganglio (n = 7) se utilizaron como entradas para PCA, revelando que las neuronas R2 exhiben perfiles de modificación de ARN distintos en comparación con los ganglios en los que residen (Figura 2B). Esto se evidencia en los puntos de datos para las neuronas R2 y los ganglios abdominales que ocupan diferentes regiones de la gráfica de puntuación PCA. Se obtuvo un apoyo adicional para patrones únicos de nucleósidos modificados de una cohorte separada de animales (n = 7) en la que se realizaron comparaciones por pares para 13 modificaciones de ARN que se detectaron comúnmente tanto en neuronas individuales como en tejido a granel (Figura 2C). Dos nucleósidos modificados, pseudouridina (Ψ) y 2'-O-metilguanosina (Gm), tuvieron una abundancia significativamente mayor en los ganglios abdominales en comparación con las neuronas R2. Al ver un subconjunto de las modificaciones de ARN con pares de neuronas r-ganglios indicados, todos los ganglios abdominales exhibieron niveles más altos de Ψ y Gm, y todas menos una de las neuronas R2 tenían mayores abundancias de 2'-O-metiladenosina (Am) que su ganglio correspondiente (Figura 2D). En general, los resultados de SNRMA-MS revelan por primera vez que los perfiles de modificación de ARN de células individuales pueden divergir de las células a granel en el mismo tejido.
El uso de SNRMA-MS para investigar el animal modelo A. californica proporciona una oportunidad única para caracterizar los perfiles de modificación de ARN en neuronas identificadas y funcionalmente distintas. Las modificaciones del ARN fueron evaluadas por SNRMA-MS en cuatro células identificadas: R2 y LPl1 (células colinérgicas homólogas implicadas en la liberación mucosa defensiva)32, MCCs (neuronas moduladoras serotoninérgicas implicadas en la alimentación)33 y células B2 (neuronas peptónrgicas implicadas en la motilidad intestinal)34. El PCA de seis modificaciones de ARN en estas neuronas identificadas, ya sea aislado inmediatamente después del tratamiento enzimático o cultivado en una preparación ganglionar durante 48 h, demostró la estabilidad y la dinámica de los epitrancrisomas unicelulares. Las modificaciones de ARN en células funcionalmente diferentes formaron grupos únicos en la gráfica de puntuación, mientras que las neuronas R2 / LPl1 homólogas se agruparon (Figura 3A). La gráfica de carga muestra que las diferencias fueron impulsadas principalmente por la abundancia de isómeros posicionales de metiladenosina, incluyendo 2'-O-metiladenosina (Am) y N1-metiladenosina (m1A) (Figura 3B). En el mismo análisis, se realizó una comparación de células recién aisladas y células cultivadas in situ (es decir, en sus respectivos ganglios) durante 48 h. Como se muestra en la gráfica de puntuación de PCA, las células funcionalmente diferentes permanecieron distinguibles por sus perfiles de modificación de ARN.
El SNRMA-MS cuantitativo se puede utilizar para determinar cantidades absolutas de modificaciones de ARN seleccionadas para las que se dispone de estándares auténticos. Se generaron curvas de calibración externas para m1A, Ψ, 2'-O-metilcitidina (Cm), Am y m6A, y la cantidad de cada nucleósido modificado en los pares de células MCC y R2/LPl1 se determinó por interpolación (Figura 4A-E). Las cantidades intracelulares de m1A y Ψ en dos pares de MCC simétricos parecieron ser similares, mientras que se observaron mayores diferencias en las cantidades de estas modificaciones en tres pares de células R2/LPl1. Para tener en cuenta las diferencias debidas al tamaño físico de las células estudiadas, las cantidades de modificación de ARN se normalizaron mediante volúmenes celulares calculados a partir de la medición óptica de los diámetros celulares para producir concentraciones intracelulares de nucleósidos modificados. Se observaron diferencias significativas en las concentraciones intracelulares de Cm y Am entre los MCC y las neuronas R2/LPl1. En general, SNRMA-MS permite el perfil cualitativo y cuantitativo de las modificaciones de ARN en neuronas individuales.
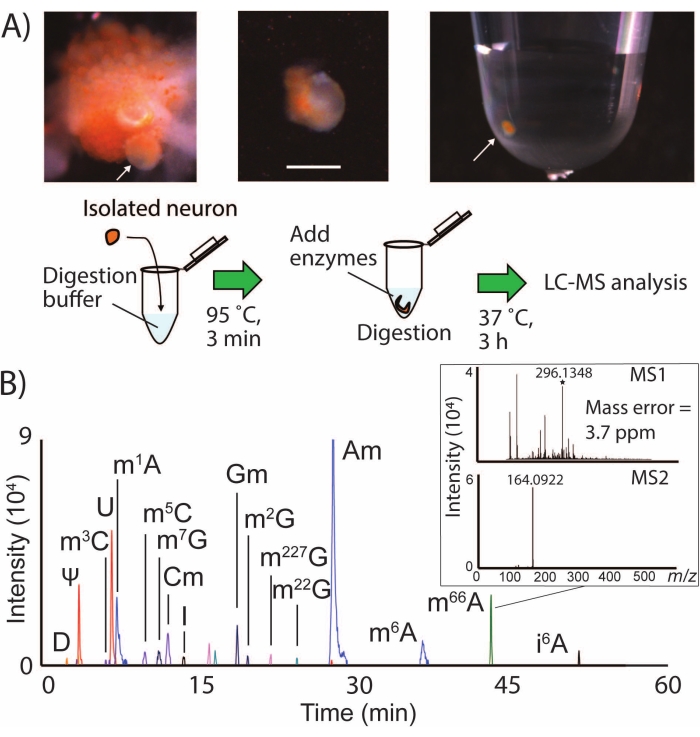
Figura 1: Flujo de trabajo de SNRMA-MS y detección de múltiples modificaciones de ARN en neuronas individuales por LC-MS / MS. (A) Fotografías del ganglio bucal desenvainado y el aislamiento de una sola neurona en un tubo de muestra. Barra de escala = 200 μm, las flechas indican la célula B1 identificada. También se muestra un diagrama del procedimiento de preparación de la muestra para el análisis LC-MS/MS. (B) EIC superpuestos para modificaciones de ARN en una sola neurona LPl1, con un recuadro que muestra los espectros MS1 y MS2 para N6, N6-dimetiladenosina (m66A). Consulte la Tabla 1 para los valores m/z utilizados para generar EIC para nucleósidos modificados. Esta cifra se ha modificado de29. Haga clic aquí para ver una versión más grande de esta figura.
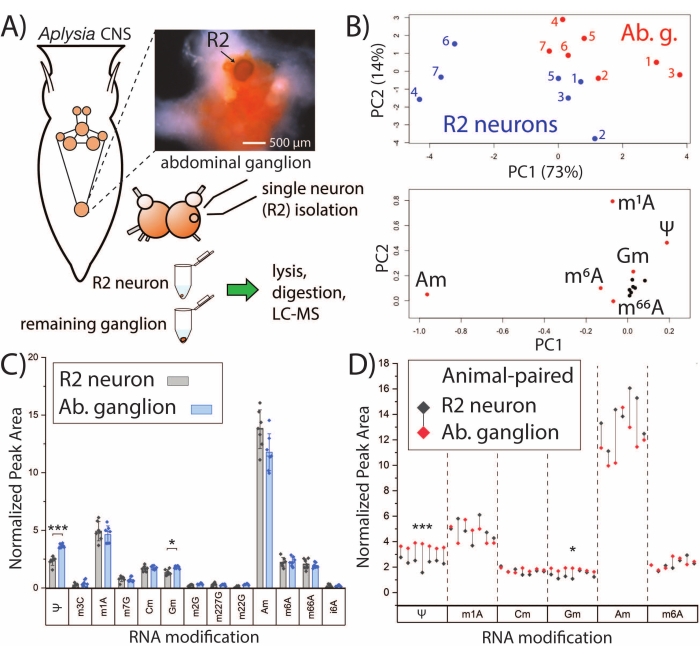
Figura 2: SNRMA-MS distingue los perfiles de modificación de ARN de neuronas individuales y tejido a granel. (A) Esquema de A. californica CNS y flujo de trabajo para analizar neuronas R2 y el ganglio abdominal circundante. La fotografía muestra el ganglio abdominal y la neurona R2 (inyectada con tinte Fast Green para mayor visibilidad). (B) Se utilizaron áreas pico relativas de 13 modificaciones de ARN para generar las gráficas de puntuación PCA (arriba) y carga (abajo). (C) Comparación por pares de modificaciones de ARN en el ganglio abdominal y la neurona R2. Las barras de error representan ±1 desviación estándar (DE), *p < 0,05, ***p < 5 x 10−4, prueba t emparejada con corrección de Bonferroni−Holm. (D) Comparación de modificaciones seleccionadas de ARN del panel C en el que se muestran pares de ganglios R2-abdominales para cada animal con líneas de gota. Esta cifra se ha modificado de29. Haga clic aquí para ver una versión más grande de esta figura.
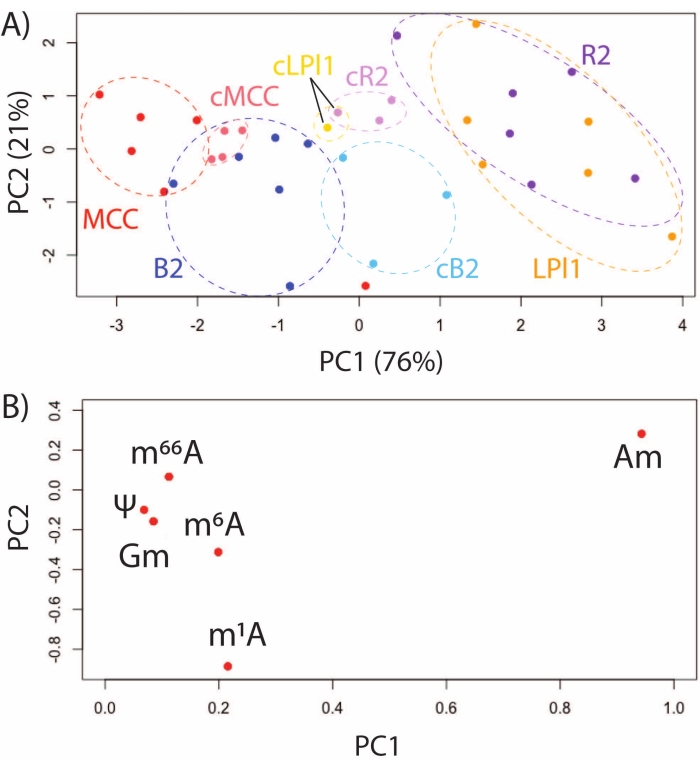
Figura 3: Perfil de modificaciones de ARN en neuronas identificadas y funcionalmente diferentes del SNC de A. californica ( A) Gráfico de puntuación PCA para MCC y células B2, R2 y LPl1 que fueron recién aisladas o aisladas después del cultivo in situ durante 48 h (denotado por cMCC, cB2, cR2, cLPl1), y (B) parcelas de carga para seis modificaciones de ARN comúnmente detectadas en estas células. Esta cifra se ha modificado de29. Haga clic aquí para ver una versión más grande de esta figura.
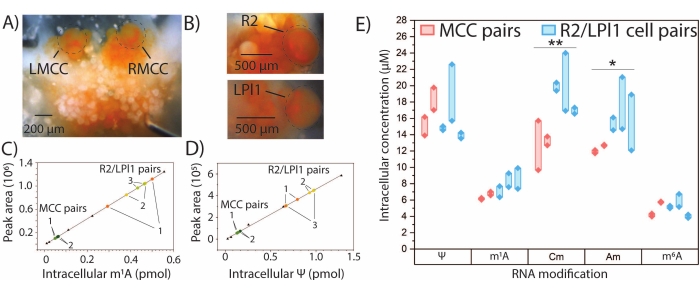
Figura 4: SNRMA-MS cuantitativa en neuronas A. californica . La SNRMA-EM cuantitativa proporciona cantidades absolutas y concentraciones intracelulares para varios nucleósidos modificados en neuronas A. californica únicas identificadas. Fotografías de (A) MCC izquierdo y derecho (LMCC y RMCC, respectivamente) en el ganglio cerebral y (B) R2 en el ganglio abdominal y LPl1 en el ganglio pleural. Las celdas están rodeadas de círculos para mejorar la visibilidad. Gráficos de calibración lineal para (C) m1A y (D) Ψ (triángulos) utilizados para la interpolación de cantidades de nucleósidos modificados en celdas individuales (puntos de color). Los pares de células de cada animal están etiquetados del 1 al 3. (E) Concentraciones intracelulares de cinco nucleósidos modificados en pares de células MCC y R2/LPl1. Las líneas gruesas conectan pares de celdas. *p < 0,05, **p < 0,005, prueba t pareada con corrección de Bonferroni−Holm, n = 2 animales (cuatro células en total) para MCC y n = 3 animales (seis células en total) para R2/LPl1. Esta cifra se ha modificado de29. Haga clic aquí para ver una versión más grande de esta figura.
| Modificación de ARN | Abreviatura | Orden de elución (C18) | m/z para EIC | m/z para MS2 |
| dihidrouridina | D | 1 | 247.09 | 115 |
| pseudouridina | Y | 2 | 245.08 | 209/179/155 |
| 3-metilcitidina | m3C | 3 | 258.11 | 126 |
| N1-metiladenosina | m1A | 4 | 282.12 | 150 |
| 5-metilcitidina | m5C | 5 | 258.11 | 126 |
| N7-metilguanosina | m7G | 6 | 298.12 | 166 |
| 2'-O-metilcitidina | Cm | 7 | 258.11 | 112 |
| Inosina | I6A | 8 | 269.09 | 137 |
| 2'-O-metilguanosina | Gm | 9 | 298.12 | 152 |
| N2-metilguanosina | m2G | 10 | 298.12 | 166 |
| N2,N2,N7-trimetilguanosina | m227G | 11 | 326.15 | 194 |
| N2,N2,-dimetilguanosina | m22G | 12 | 312.13 | 180 |
| 2'-O-metiladenosina | AM | 13 | 282.12 | 136 |
| N6-metiladenosina | m6A | 14 | 282.12 | 150 |
| N6,N6-dimetiladenosina | m66A | 15 | 296.14 | 164 |
| N6-isopenteniladenosina | i6A | 16 | 336.17 | 204/136/148 |
Tabla 1: Modificaciones de ARN detectadas en neuronas individuales de A. californica. Se proporcionan atributos para la caracterización de nucleósidos modificados, incluido el orden de retención LC, m / z para generar EIC y los fragmentos CID correspondientes.
Discusión
SNRMA-MS aprovecha un enfoque optimizado de preparación de muestras, lo que resulta en un volumen de muestra pequeño y compatible con MS que se puede entregar a la plataforma LC-MS. El pretratamiento enzimático inicial de los ganglios del SNC dicta tanto la facilidad con la que se pueden desenvainar como la durabilidad de las neuronas individuales durante el aislamiento. El ganglio cerebral a menudo requiere un tratamiento enzimático prolongado debido a su vaina relativamente gruesa en comparación con los ganglios bucales, pleurales y abdominales. Los investigadores individuales que realizan los aislamientos de una sola neurona pueden tener diferentes preferencias sobre qué tan duradera debe ser la vaina cuando se usan microescisores y fórceps finos. Sin embargo, es importante que los ganglios no se digieran en exceso, ya que esto puede conducir a una pérdida en su integridad estructural, pérdida de información posicional que es crítica para identificar las células diana y / o lisis celular. Después del aislamiento del ganglio, es importante asegurarse de que se aspire un volumen mínimo de medio de aislamiento al transferir la neurona al tubo de muestra. El medio ASW contiene una alta concentración de sales que pueden interferir con las enzimas digestivas durante la hidrólisis de ARN y también diluirá la muestra.
Durante la etapa de lisis mecánica, es común que las neuronas grandes (>250 μm de diámetro) se rompan al pasar repetidamente a través de una micropipeta. Las neuronas más pequeñas pueden requerir atención adicional para garantizar la lisis celular, que generalmente implica presionar un capilar de vidrio en la célula. En este caso, es posible que un volumen parcial del tampón de la muestra se extraiga hacia el capilar debido a las fuerzas capilares. Este volumen se puede devolver al tubo de muestra aplicando presión en el extremo del capilar de vidrio para garantizar que no se pierda ninguna muestra.
Los mejores resultados se obtienen incluyendo un paso de calentamiento antes de agregar enzimas para la digestión del ARN. Esto es probable porque el calentamiento a 95 °C desnaturaliza las estructuras secundarias de ARN que pueden impedir la actividad de las RNasas35 y disminuir la cantidad de nucleósidos liberados de los biopolímeros de ARN. Los experimentos de control utilizando muestras con picos de metionina marcada con isótopos estables se realizaron previamente para investigar si los artefactos de modificación de ARN inducidos por calor fueron la causa del aumento de las áreas de pico observadas para las modificaciones de ARN en relación con un protocolo SNRMA-MS sin calor29. No se observó tal etiquetado, lo que indica que las señales mejoradas para las modificaciones de ARN utilizando el método optimizado SNRMA-MS se debieron a una digestión superior del ARN.
Los métodos convencionales para aislar el ARN total de las células implican la extracción líquido-líquido (LLE) con fenol-cloroformo y la posterior precipitación, lavado y resuspensión de ARN. Estos enfoques han demostrado ser útiles para experimentos de reacción en cadena de la polimerasa con transcripción inversa donde la expresión de genes seleccionados puede ser fácilmente monitoreada en neuronas A. californica identificadas 36,37. Sin embargo, los métodos LLE no pueden recuperar suficiente ARN para la detección de ribonucleósidos modificados por LC-MS, mientras que el método SNRMA-MS descrito aquí permite la detección de numerosas modificaciones de ARN29. Con el fin de evaluar los perfiles de modificación de tipos específicos de ARN (por ejemplo, ARNr, ARNt, ARNm) en muestras de tejido/célula a granel, se han aplicado24 la extracción en fase sólida de intercambio aniónico, el enriquecimiento basado en sonda de hibridación38 y el fraccionamiento cromatográfico39, pero aún no se dispone de métodos similares para la purificación del ARN unicelular. El desarrollo de enfoques de fraccionamiento de ARN que son capaces de aislar tipos específicos de ARN de células individuales proporcionaría información adicional sobre la función de las modificaciones de ARN.
SNRMA-MS reveló heterogeneidad previamente no caracterizada en el panorama de modificación de ARN de neuronas individuales en A. californica y es concebible que existan perfiles ptM igualmente distintos en las células de mamíferos. Debido a que las células de mamíferos son relativamente pequeñas en comparación con las grandes neuronas de A. californica analizadas en este protocolo, se necesitan mejoras en el manejo de pequeños volúmenes de muestra para facilitar los límites de detección más bajos. Aunque actualmente SNRMA-MS está limitado a volúmenes de ~ 5 μL, se espera que se puedan lograr mejoras sustanciales mediante la incorporación de dispositivos de manejo de líquidos microfluídicos en el flujo de trabajo. Además, la implementación de aislamientos celulares automatizados o semiautomatizados aumentaría el rendimiento de la muestra y permitiría el análisis de modificación de ARN unicelular de poblaciones celulares más grandes. Al interactuar con las separaciones LC de nanoflujo27, se podrá lograr la caracterización de las marcas epitranscriptómicas en células aún más pequeñas.
Divulgaciones
Los autores no declaran intereses financieros contrapuestos.
Agradecimientos
Este trabajo fue financiado por el Instituto Nacional sobre el Abuso de Drogas bajo el Premio No. P30DA018310 y el Instituto Nacional de Investigación del Genoma Humano bajo el Premio no. RM1HG010023. K.D.C. reconoce el apoyo de una beca postdoctoral del Instituto Beckman. El contenido es responsabilidad exclusiva de los autores y no representa necesariamente las opiniones oficiales de las agencias de financiación.
Materiales
| Name | Company | Catalog Number | Comments |
| Animals | |||
| Aplysia californica | National Resource for Aplysia (Miami, FL) | 150–250 g (adult) | |
| Benchtop equipment | |||
| Glass capillary puller | Sutter | P-97 | |
| Milli-Q water purification system | Millipore | ||
| Minicentrifuge for PCR tubes | LW Scientific | ZS-1 | |
| Optical Microscope | Zeiss | Stemi 2000C | |
| Thermocycler | Techne | EW-93945-01 | |
| HPLC column and consumables | |||
| Acclaim RSLC 120 C18 column | Thermo Scientific | 71399 | |
| Autosampler vials | Thermo Scientific | C4011-13 | |
| LC and MS Instrumentation and Software | |||
| DataAnalysis 4.4 software | Bruker | ||
| Dionex Ultimate 3000 nanoLC | Thermo Scientific | Equipped with online degasser, autosampler, and thermostatted column compartment | |
| Impact HD UHR QqTOF mass spectrometer | Bruker | Equipped with ESI source | |
| RStudio | RStudio | ||
| Microdissection tools | |||
| Microscissors extra fine vannas 3.5” | Roboz | RS-5640 | |
| Tungsten needles | Roboz | RS-6065 | |
| Reagents/Materials | |||
| 4-(2-hydroxyethyl)-1-piperazineethane-sulfonic acid (HEPES) | Fisher Scientific | H3375 | |
| Alkaline phosphatase | Worthington Biochemical Corp. | LS004081 | |
| Ammonium acetate | Honeywell | 17836 | |
| Benzonase (endonuclease from S. marcescens) | EMD Millipore | 70746-4 | |
| Bovine serum albumin | Sigma-Aldrich | A2153 | |
| Calcium chloride | Sigma-Aldrich | C4901 | |
| Gentamycin sulfate | Fisher Scientific | G1264 | |
| Magnesium chloride | Sigma-Aldrich | M9272 | |
| Magnesium sulfate | Sigma-Aldrich | 208094 | |
| Nucleosides test mix | Sigma-Aldrich | 47310-U | |
| Penicillin G | Sigma-Aldrich | P7794 | |
| Pentostatin | Sigma-Aldrich | SML0508 | |
| Phosphodiesterase I | Worthington Biochemical Corp. | LS003926 | |
| Protease type XIV from Streptomyces griseus | Sigma-Aldrich | P5147 | |
| Sodium chloride | Sigma-Aldrich | S9888 | |
| Standard glass capillaries | A-M Systems | 626000 | 1 mm o.d., 0.5 mm i.d., 4 in |
| Streptomycin sulfate | Sigma-Aldrich | S9137 |
Referencias
- Cantara, W. A., et al. The RNA modification database, RNAMDB: 2011 update. Nucleic Acids Research. 39, 195-201 (2011).
- Boccaletto, P., et al. MODOMICS: a database of RNA modification pathways. 2017 update. Nucleic Acids Research. 46, 303-307 (2017).
- Kimura, S., Waldor, M. K. The RNA degradosome promotes tRNA quality control through clearance of hypomodified tRNA. Proceedings of the National Academy of Sciences of the United States of America. 116 (4), 1394-1403 (2019).
- Helm, M. Post-transcriptional nucleotide modification and alternative folding of RNA. Nucleic Acids Research. 34 (2), 721-733 (2006).
- Rezgui, V. A. N., et al. tRNA tKUUU, tQUUG, and tEUUC wobble position modifications fine-tune protein translation by promoting ribosome A-site binding. Proceedings of the National Academy of Sciences of the United States of America. 110 (30), 12289-12294 (2013).
- Shanmugam, R., et al. Cytosine methylation of tRNA-Asp by DNMT2 has a role in translation of proteins containing poly-Asp sequences. Cell Discovery. 1 (1), 1-10 (2015).
- Jia, G., et al. N 6-Methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO. Nature Chemical Biology. 7 (12), 885-887 (2011).
- Li, X., et al. Transcriptome-wide mapping reveals reversible and dynamic N 1 -methyladenosine methylome. Nature Chemical Biology. 12 (5), 311-316 (2016).
- Krogh, N., et al. Profiling of 2′-O-Me in human rRNA reveals a subset of fractionally modified positions and provides evidence for ribosome heterogeneity. Nucleic Acids Research. 44 (16), 7884-7895 (2016).
- Babaian, A., et al. Loss of m1acp3Ψ Ribosomal RNA Modification Is a Major Feature of Cancer. Cell Reports. 31 (5), 107611 (2020).
- Chang, M., et al. Region-specific RNA m6A methylation represents a new layer of control in the gene regulatory network in the mouse brain. Open Biology. 7 (9), 170166 (2017).
- Wang, C. -. X., et al. METTL3-mediated m6A modification is required for cerebellar development. PLOS Biology. 16 (6), 2004880 (2018).
- Engel, M., et al. The role of m6A/m-RNA methylation in stress response regulation. Neuron. 99 (2), 389-403 (2018).
- Widagdo, J., et al. Experience-dependent accumulation of N6-Methyladenosine in the prefrontal cortex is associated with memory processes in mice. Journal of Neuroscience. 36 (25), 6771-6777 (2016).
- Eberwine, J., et al. Analysis of gene expression in single live neurons. Proceedings of the National Academy of Sciences of the United States of America. 89 (7), 3010-3014 (1992).
- Cong, Y., et al. Ultrasensitive single-cell proteomics workflow identifies >1000 protein groups per mammalian cell. Chemical Science. 12 (3), 1001-1006 (2021).
- Rubakhin, S. S., Romanova, E. V., Nemes, P., Sweedler, J. V. Profiling metabolites and peptides in single cells. Nature Methods. 8 (4), 20-29 (2011).
- Nemes, P., Knolhoff, A. M., Rubakhin, S. S., Sweedler, J. V. Metabolic differentiation of neuronal phenotypes by single-cell capillary electrophoresis-electrospray ionization-mass spectrometry. Analytical Chemistry. 83 (17), 6810-6817 (2011).
- Lovatt, D., et al. Transcriptome in vivo analysis (TIVA) of spatially defined single cells in live tissue. Nature Methods. 11 (2), 190-196 (2014).
- Kærn, M., Elston, T. C., Blake, W. J., Collins, J. J. Stochasticity in gene expression: from theories to phenotypes. Nature Reviews Genetics. 6 (6), 451-464 (2005).
- Chan, C. T. Y., et al. A quantitative systems approach reveals dynamic control of tRNA modifications during cellular stress. PLOS Genetics. 6 (12), 1001247 (2010).
- Sun, C., Jora, M., Solivio, B., Limbach, P. A., Addepalli, B. The effects of ultraviolet radiation on nucleoside modifications in RNA. ACS Chemical Biology. 13 (3), 567-572 (2018).
- Heiss, M., Hagelskamp, F., Marchand, V., Motorin, Y., Kellner, S. Cell culture NAIL-MS allows insight into human tRNA and rRNA modification dynamics in vivo. Nature Communications. 12 (1), 389 (2021).
- Clark, K. D., Lee, C., Gillette, R., Sweedler, J. V. Characterization of neuronal RNA modifications during non-associative learning in aplysia reveals key roles for tRNAs in behavioral sensitization. ACS Central Science. 7 (7), 1183-1190 (2021).
- Basanta-Sanchez, M., Temple, S., Ansari, S. A., D'Amico, A., Agris, P. F. Attomole quantification and global profile of RNA modifications: Epitranscriptome of human neural stem cells. Nucleic Acids Research. 44 (3), 26 (2016).
- Huang, W., et al. Determination of DNA and RNA methylation in circulating tumor cells by mass spectrometry. Analytical Chemistry. 88 (2), 1378-1384 (2016).
- Sarin, L. P., et al. Nano LC-MS using capillary columns enables accurate quantification of modified ribonucleosides at low femtomol levels. RNA. 24 (10), 1403-1417 (2018).
- Clark, K. D., Philip, M. C., Tan, Y., Sweedler, J. V. Biphasic liquid microjunction extraction for profiling neuronal RNA modifications by liquid chromatography-tandem mass spectrometry. Analytical Chemistry. 92 (18), 12647-12655 (2020).
- Clark, K. D., Rubakhin, S. S., Sweedler, J. V. Single-neuron RNA modification analysis by mass spectrometry: Characterizing RNA modification patterns and dynamics with single-cell resolution. Analytical Chemistry. 93 (43), 14537-14544 (2021).
- Guise, O. L., Ahner, J. W., Jung, M. -. C., Goughnour, P. C., Yates, J. T. Reproducible electrochemical etching of tungsten probe tips. Nano Letters. 2 (3), 191-193 (2002).
- Peng, W., Wu, Z., Song, K., Zhang, S., Li, Y., Xu, M. Regulation of sleep homeostasis mediator adenosine by basal forebrain glutamatergic neurons. Science. 369 (6508), (2020).
- Rayport, S. G., Ambron, R. T., Babiarz, J. Identified cholinergic neurons R2 and LPl1 control mucus release in Aplysia. Journal of Neurophysiology. 49 (4), 864-876 (1983).
- Rosen, S. C., Weiss, K. R., Goldstein, R. S., Kupfermann, I. The role of a modulatory neuron in feeding and satiation in Aplysia: effects of lesioning of the serotonergic metacerebral cells. Journal of Neuroscience. 9 (5), 1562-1578 (1989).
- Lloyd, P. E., Kupfermann, I., Weiss, K. R. Central peptidergic neurons regulate gut motility in Aplysia. Journal of Neurophysiology. 59 (5), 1613-1626 (1988).
- Crain, P. F. Preparation and enzymatic hydrolysis of DNA and RNA for mass spectrometry. Methods in Enzymology. 193, 782-790 (1990).
- Kadakkuzha, B. M., et al. Age-associated bidirectional modulation of gene expression in single identified R15 neuron of Aplysia. BMC Genomics. 14 (1), 880 (2013).
- Akhmedov, K., Kadakkuzha, B. M., Puthanveettil, S. V. Aplysia ganglia preparation for electrophysiological and molecular analyses of single neurons. Journal of Visualized Experiments: JoVE. (83), e51075 (2014).
- Tardu, M., Jones, J. D., Kennedy, R. T., Lin, Q., Koutmou, K. S. Identification and quantification of modified nucleosides in saccharomyces cerevisiae mRNAs. ACS Chemical Biology. 14 (7), 1403-1409 (2019).
- Heiss, M., Reichle, V. F., Kellner, S. Observing the fate of tRNA and its modifications by nucleic acid isotope labeling mass spectrometry: NAIL-MS. RNA Biology. 14 (9), 1260-1268 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados