Method Article
Caratterizzazione delle modifiche dell'RNA in singoli neuroni utilizzando la spettrometria di massa
In questo articolo
Riepilogo
Le modificazioni post-trascrizionali dell'RNA rappresentano uno strato poco studiato di regolazione della traduzione che è stato recentemente collegato alla plasticità del sistema nervoso centrale. Qui, viene descritta la preparazione del campione e l'approccio di cromatografia liquida-spettrometria di massa tandem per la caratterizzazione simultanea di numerose modifiche dell'RNA in singoli neuroni.
Abstract
Le modificazioni post-trascrizionali (PTM) dell'RNA rappresentano un meccanismo poco studiato coinvolto nella regolazione della traduzione nel sistema nervoso centrale (SNC). Prove recenti hanno collegato specifiche modifiche dell'RNA neuronale ai paradigmi di apprendimento e memoria. Sfortunatamente, i metodi convenzionali per il rilevamento di queste caratteristiche epitrascrittomiche sono in grado di caratterizzare solo modifiche dell'RNA molto abbondanti nei tessuti di massa, precludendo la valutazione di profili PTM unici che possono esistere per i singoli neuroni all'interno dei circuiti comportamentali attivati. In questo protocollo, viene descritto un approccio – analisi di modifica dell'RNA a singolo neurone mediante spettrometria di massa (SNRMA-MS) – per rilevare e quantificare simultaneamente numerosi ribonucleosidi modificati in singoli neuroni. L'approccio è convalidato utilizzando singoli neuroni del mollusco marino, Aplysia californica, a partire dall'isolamento chirurgico e dal trattamento enzimatico dei principali gangli del SNC per esporre i corpi cellulari dei neuroni, seguito dall'isolamento manuale a singolo neurone utilizzando aghi affilati e una micropipetta. Successivamente, il trattamento meccanico e termico del campione in un piccolo volume di tampone viene effettuato per liberare l'RNA da una singola cellula per la successiva digestione dell'RNA. I nucleosidi modificati vengono quindi identificati e quantificati utilizzando un metodo ottimizzato di cromatografia liquida-spettrometria di massa. SNRMA-MS è impiegato per stabilire modelli di modifica dell'RNA per singoli neuroni identificati da A. californica che hanno morfologie e funzioni note. Vengono presentati esempi di SNRMA-MS qualitativi e quantitativi che evidenziano la distribuzione eterogenea delle modifiche dell'RNA tra i singoli neuroni nelle reti neuronali.
Introduzione
Le modifiche nei nucleosidi canonici dell'RNA sono state sempre più riconosciute per la loro miriade di ruoli nella regolazione della traduzione proteica. Ad oggi sono state riportate oltre 150 modificazioni uniche dell'RNA che variano in complessità dalla metilazione, all'aggiunta di eteroatomi, alla coniugazione con metaboliti cellulari 1,2. Questo alfabeto a RNA espanso, noto anche come epitrastoma, è generato da scrittori enzimatici ed è responsabile dell'alterazione della stabilità3, del ripiegamento4 e dell'efficienza di traduzione 5,6 degli RNA cellulari. Le modifiche selezionate dell'RNA possono anche essere invertite tramite gomme enzimatiche 7,8, mentre altre sono aggiunte agli RNA substoichiometricamente 9,10, rendendo un panorama complesso di sequenze di RNA modificate e non modificate nei sistemi biologici.
L'importanza delle modificazioni dell'RNA nella funzione biologica è indicata dalla distribuzione ineguale delle modificazioni tra diversi organi e tessuti, comprese le sottoregioni del sistema nervoso centrale (SNC)11. Questa eterogeneità chimica è stata collegata allo sviluppo del SNC12, alla risposta allo stress13 e alla plasticità dipendente dall'attività14. Le sottoregioni del SNC comprendono inoltre popolazioni eterogenee di cellule in cui le singole cellule presentano profili chimici distinti 15,16,17,18. Anche singole cellule dello stesso tipo possono mostrare trascrittomi unici, in parte a causa dei microambienti tissutali19 e dell'espressione genica stocastica20. Tuttavia, mentre la caratterizzazione del trascrittoma a singola cellula è in qualche modo di routine, non esistono metodi analoghi per stabilire epitrascrittomi a singola cellula per modifiche multiple dell'RNA. Nuovi approcci in grado di profilare la distribuzione delle modificazioni dell'RNA nelle singole cellule sono necessari per un'analisi completa dell'eterogeneità cellulare e dell'influenza regolatoria delle modificazioni post-trascrizionali (PTM) nel SNC e in altri sistemi biologici.
La misurazione simultanea di numerose modifiche dell'RNA in cellule / tessuti di massa è facilmente realizzabile utilizzando tecniche di cromatografia liquida-spettrometria di massa tandem (LC-MS / MS). Per l'analisi LC-MS/MS di ribonucleosidi modificati, l'RNA viene estratto dalle cellule (tipicamente 10 3-106 cellule), purificato per precipitazione e risospensione e successivamente digerito in nucleosidi. La miscela campione costituita da nucleosidi canonici e modificati viene quindi iniettata nel sistema LC-MS per la separazione e il rilevamento degli analiti, portando alla determinazione dell'intero complemento di modificazioni dell'RNA in un organismo 21,22,23. LC-MS/MS è stato recentemente utilizzato per determinare 26 modificazioni dell'RNA nel SNC del modello neurobiologico, Aplysia californica (A. californica). L'abbondanza di alcuni di questi segni epitrascrittomici ha mostrato dinamiche dipendenti dal tempo e dalla regione che si correlavano con i cambiamenti comportamentali nell'animale24. Tuttavia, è stato possibile rilevare modifiche dell'RNA solo in campioni di massa contenenti >103 cellule a causa della limitata sensibilità del metodo. Questi campioni più grandi probabilmente nascondevano profili di modifica dell'RNA unici e funzionalmente importanti di singole cellule con medie di popolazione. Sebbene un attento controllo delle condizioni di preparazione del campione abbia migliorato i limiti di rilevamento per le modifiche dell'RNA in campioni di piccolo volume 25,26,27,28, rimane la necessità di metodi analitici in grado di rilevare e quantificare più ribonucleosidi modificati in singole cellule.
Questo protocollo introduce l'analisi di modificazione dell'RNA a singolo neurone mediante spettrometria di massa (SNRMA-MS), che consente il rilevamento di oltre una dozzina di modifiche dell'RNA in singoli neuroni dal SNC di A. californica29. L'approccio consiste nell'isolamento chirurgico di singole cellule identificate dai principali gangli del SNC seguito da un flusso di lavoro di preparazione del campione ottimizzato che coinvolge la lisi cellulare meccanica, la denaturazione dell'RNA e l'idrolisi enzimatica in un tampone compatibile con la SM. L'identificazione e la quantificazione dei nucleosidi post-trascrizionalmente modificati viene quindi effettuata utilizzando LC-MS/ MS. SNRMA-MS soddisfa un'esigenza insoddisfatta nel campo dell'analisi della modifica dell'RNA facilitando l'acquisizione di profili di modifica dell'RNA post-trascrizionale per singoli neuroni e ha il potenziale per future applicazioni ad altri tipi di cellule.
Protocollo
1. Preparazione di materiali e soluzioni
- Preparare acqua di mare artificiale (ASW) con 460 mM NaCl, 10 mM KCl, 10 mM CaCl2, 22 mM MgCl2, 26 mM MgSO4 e 10 mM HEPES in acqua ottenuta da un rigoroso sistema di purificazione. Regolare il pH a 7,8 utilizzando 1 M NaOH o HCl. In genere, preparare 1 L di ASW e conservare a 14 °C fino all'uso.
- Preparare una soluzione di antibiotici ASW con 10.000 U/mL di penicillina G, 10 μg/μL di streptomicina e 10 μg/μL di gentamicina e conservare a -20 °C. Immediatamente prima dell'esperimento, scongelare e diluire la soluzione madre di antibiotici congelati 1:100 in 20-40 ml di ASW. La concentrazione finale di antibiotici nella soluzione ASW funzionante è di 100 U/mL di penicillina G, 100 μg/mL di streptomicina e 100 μg/mL di gentamicina.
- Preparare un tampone di digestione dell'RNA combinando 1 μL di 10 μg/μL di albumina sierica bovina, 0,5 μL di 0,5 μg/μL di pentostatina, 0,495 μL di 2 U/μL di fosfatasi alcalina, 1 μL di 0,1 U fosfodiesterasi I (in 10 mM MgCl2) e 0,38 μL di endonucleasi da Serratia marcescens (25 U) per campione. Se si analizzano più campioni, preparare una miscela master in un tubo separato contenente il volume di ciascun reagente moltiplicato per il numero di campioni da analizzare, più un altro per tenere conto della perdita accidentale di reagente dalle fasi di trasferimento del pipet.
- Preparare diversi aghi affilati (di vetro o metallo) per l'isolamento manuale dei neuroni. Realizza aghi metallici in casa mediante incisione elettrochimica del filo di tungsteno come in30 o acquistali. Preparare aghi di vetro da capillari di vetro borosilicato a parete spessa o standard (diametro esterno di 1 mm) utilizzando un estrattore di micropipette. Per il presente metodo, mantenere il diametro della punta dell'ago tra 1-5 μm, con una lunghezza del collo di 100-150 μm.
NOTA: La fabbricazione di aghi metallici e di vetro può essere ottimizzata per produrre strumenti che soddisfino le esigenze individuali del ricercatore e il modello biologico specifico studiato.
2. Isolamento del singolo neurone
- Raffreddare una soluzione di MgCl2 da 0,33 M a 14 °C. Utilizzando una siringa da 50 ml, anestetizzare A. californica (150-250 g) iniettando la soluzione di MgCl2 nella cavità corporea dell'animale. I migliori risultati si ottengono con un rapporto 1:3 tra il volume della soluzione (mL) e la massa corporea animale (g). Attendere circa 3 minuti affinché l'animale si rilassi, assicurandosi che non mostri contrazioni corporee in risposta alla stimolazione tattile.
- Posizionare il lato ventrale dell'animale (lato del piede) in un vassoio di dissezione. Sezionare l'animale usando forbici chirurgiche con una punta smussata posizionata verso l'animale, facendo con cura un taglio longitudinale attraverso il piede.
- Fissare i lati rostrale, caudale e laterale del corpo animale per esporre gli organi interni e i gangli del SNC situati nella cavità corporea. Isolare i principali gangli del SNC dall'animale tagliando chirurgicamente i nervi e alcuni connettivi provenienti dai gangli.
- Immergere i gangli in una soluzione di proteasi di tipo XIV da Streptomyces griseus (10 mg/mL in soluzione di antibiotici ASW) e incubare a 34 °C per 30 minuti o 1 ora (per il ganglio cerebrale).
NOTA: La durata dell'incubazione dipende dalla stagione, dalle dimensioni e dalle condizioni dell'animale, nonché dai neuroni mirati. L'isolamento di alcuni neuroni richiede un'incubazione più lunga di altri e dovrebbe essere determinato sperimentalmente. - Risciacquare i gangli 6x con una soluzione di antibiotici ASW e trasferire tutti i gangli in un piatto rivestito di polimeri siliconici riempito con soluzione di antibiotici ASW utilizzando un pipetto di trasferimento in polipropilene che è stato tagliato ad un'apertura di ~ 5 mm. Trattare il pipetto con 1 mg/mL di albumina sierica bovina in ASW per ridurre al minimo l'attaccamento dei gangli al pipetto (opzionale). Mantieni i gangli immersi in ASW in ogni momento.
NOTA: Il trattamento enzimatico riduce la stabilità meccanica dei neuroni e del tessuto connettivo circostante e, di conseguenza, le membrane neuronali possono essere danneggiate a causa dell'esposizione all'aria durante il trasferimento dei gangli. - Fissare i gangli e utilizzare micro forbici e pinze sottili per rimuovere le guaine gangliari. Con un trattamento enzimatico sufficientemente forte, utilizzare aghi di vetro o metallo per il deguarigimento.
- Identificare visivamente i neuroni di interesse di A. californica . In questo lavoro, sono state studiate le seguenti cellule: R2 nel ganglio addominale, LPl1 nel ganglio pleurico, cellule metacerebrali (MCC) nel ganglio cerebrale e cellule B2 nel ganglio buccale. Scatta immagini ottiche di tutti i preparati neurali e gangliari utilizzando un microscopio calibrato con ingrandimento totale 20x per determinare le dimensioni e i volumi di ciascun tipo di cellula.
- Utilizzando un capillare di vetro tirato o aghi di tungsteno affilati, isolare accuratamente la cellula identificata dal ganglio sfuso.
- Prelevare una piccola quantità (1 μL) di ASW in una micropipetta di plastica, quindi trasferire la cella isolata in una provetta PCR contenente 4 μL di acetato di ammonio 0,365 M (pH 9,2). Per le misurazioni in bianco, raccogliere aliquote di 5 μL della soluzione di antibiotici ASW dal piatto contenente il ganglio e mescolare con tampone digestivo (descritto di seguito).
3. Lisi cellulare e digestione dell'RNA per SNRMA-MS
- Lisare i neuroni isolati mediante aspirazione ripetuta e rinunciando a una micropipetta (~ 100 μm di diametro interno) in 0,365 M acetato di ammonio. Alcune cellule più piccole potrebbero non rompersi immediatamente; per lisarli, applicare una pressione sul diametro della cella con un capillare di vetro tirato.
- Utilizzare un termociclatore per riscaldare i campioni con il seguente programma di temperatura: 95 °C per 3 min, 10 °C per 3 min, tenere a 10 °C. Rimuovere la provetta del campione dal termociclatore.
- Aggiungere 3,375 μL di tampone di digestione dell'RNA per ogni campione e mescolare la soluzione usando una micropipetta prelevando ed erogando la soluzione più volte. Utilizzare una centrifuga da banco in miniatura a 2700 x g per 30 s per far girare le goccioline liquide aggrappate alle pareti del tubo PCR.
- Incubare i campioni nel termociclatore a 37 °C per 3 ore, seguito da un fermo a 10 °C (coperchio riscaldato impostato su ON). Subito dopo che il campione si è raffreddato a 10 °C, trasferire 7 μL della soluzione in un flaconcino per autocampionatori dotato di un inserto da 250 μL, avendo cura di evitare la formazione di bolle nel tubo dell'autocampionatore.
4. Cromatografia liquida-spettrometria di massa tandem
NOTA: Analizzare i digest a neurone singolo e gli autentici standard nucleosidici modificati utilizzando un sistema LC-MS/MS dotato di sorgente di ionizzazione elettrospray e valvola deviatrice a sei porte.
- Per preparare il sistema LC alla separazione dei nucleosidi canonici e modificati, equilibrare una colonna C18 (150 mm x 2,1 mm, dimensione delle particelle 2,2 μm, diametro dei pori 120 Å) con il 99% di fase mobile A (5 mM di acetato di ammonio, pH 5,6) e l'1% di fase mobile B (60/40 di fase mobile A/acetonitrile (ACN)) ad una portata di 0,2 mL/min per 12 min a 36 °C. Utilizzare solventi di grado LC-MS per la preparazione di fasi mobili.
- Mentre l'LC si equilibra, calibrare lo spettrometro di massa introducendo una soluzione da 1 mM di acetato di sodio in 50/50 ACN/acqua allo spettrometro di massa tramite pompa a siringa con una portata di 5 μL/min. Dopo la calibrazione, ricollegare il flusso LC allo spettrometro di massa.
- Programmare i seguenti parametri di gradiente lineare: 1% B per 0-5 min, 5% B a 9 min, 7% B a 11 min, 10% B a 13 min, 15% B a 32 min, 40% B a 38 min, 50% B a 43 min, 100% B a 50 min, 100% B a 60 min, 1% B a 61 min, e un riequilibrio di 12 minuti all'1% B prima dell'iniezione successiva.
- Azionare lo strumento MS in modalità positiva con i seguenti parametri: tensione capillare impostata su 4.500 V, temperatura di essiccazione 275 °C, N2 gas di essiccazione 5 L/min e gas di nebulizzazione 1 bar. Impostare la valvola deviatrice sui rifiuti per i primi 2 minuti di analisi e sull'origine per il resto della corsa.
- Raccogliere spettri di massa su un intervallo m/z di 110-600. Selezionare gli ioni per la dissociazione indotta dalla collisione a 35-40 eV su un tempo di ciclo di 3 s utilizzando un elenco di massa preferito costruito utilizzando il database2 e una finestra di isolamento di ± 0,5. Utilizzare l'esclusione attiva per escludere gli ioni dalla frammentazione dopo tre spettri.
- Impostare l'acquisizione dinamica degli spettri MS/MS per ioni con intensità superiori e inferiori a 50.000 conteggi a 4 Hz e 1 Hz, rispettivamente, e una soglia minima per la selezione degli ioni a 1.990 conteggi.
- Per SNRMA-MS quantitativo, costruire curve di calibrazione utilizzando aree di picco del cromatogramma ionico estratto (EIC) ottenute per standard nucleosidici modificati ad un minimo di cinque concentrazioni per consentire l'interpolazione di concentrazioni di analiti endogeni sconosciuti.
NOTA: I neuroni ottenuti da animali con masse corporee di 150-250 g richiedono tipicamente curve di calibrazione per nucleosidi modificati che vanno da 0,02 pmol a 2 pmol, ma questi valori possono variare a seconda della sensibilità dello strumento.
5. Analisi dei dati
- Generare EIC per nucleosidi modificati (m/z dai valori del database2). Verificare le identità dei nucleosidi modificati putativi confrontando i loro spettri MS2 e le caratteristiche di ritenzione LC con i valori del database2. Vedere la Tabella 1 per un elenco delle tipiche modifiche dell'RNA rilevate in singoli neuroni da A. californica.
- Integrare manualmente i picchi che corrispondono ai nucleosidi modificati e canonici e registrare questi valori in un foglio di calcolo. Normalizzare l'area di picco per ogni nucleoside modificato con la somma delle aree di picco per citidina canonica, uridina e guanosina rilevate nel campione.
NOTA: L'adenosina non è inclusa nella normalizzazione a causa del suo ruolo nel SNC come neuromodulatore dinamico31. - Costruisci una matrice di dati composta da ciascun campione di neuroni singoli e dalle corrispondenti aree di picco normalizzate per nucleosidi modificati che hanno mostrato rapporti segnale-rumore >10. Eseguire l'analisi dei componenti principali (PCA) e visualizzare i primi due componenti principali nel grafico del punteggio. Costruisci curve di calibrazione lineare dalle aree di picco EIC ottenute dalla diluizione seriale di standard nucleosidici e calcola le concentrazioni di analiti rilevati.
Risultati
SNRMA-MS comporta l'isolamento manuale dei neuroni identificati in piccoli volumi di campioni per la lisi, la digestione e l'analisi LC-MS/MS (Figura 1A). Questo flusso di lavoro ha rilevato di routine oltre una dozzina di modifiche dell'RNA in singoli neuroni dal SNC di A. californica (Figura 1B), che rappresentano una copertura di quasi la metà dell'epitrastoma noto di questo animale24 in una singola cellula. Ad esempio, l'assoggettamento del neurone LPl1 (~ 500 μm di diametro) a SNRMA-MS ha comportato il rilevamento di 15 modifiche dell'RNA ± 1 (n = 3). I nucleosidi modificati sono stati identificati positivamente sulla base di tre attributi: proprietà di ritenzione LC, massa esatta e profili di frammentazione MS2 rispetto ai valori forniti nel database2. La Tabella 1 mostra un elenco di tutte le modifiche dell'RNA rilevate nel neurone LPl1. Lo spettrometro di massa a tempo di volo a quadrupolo ad alta risoluzione utilizzato per questi esperimenti ha consentito una precisione di massa di 4 ppm e il rilevamento del caratteristico ione frammento MS2 a m/z 164 per il nucleoside modificato N6,6-dimetildidennosina (m66A, Figura 1B). In combinazione con i dati di separazione LC, che concordano con i risultati depositati nel database (m66A elutes dopo N6-metildidenosina (m6A)), l'approccio SNRMA-MS ha dimostrato la corretta assegnazione delle identità nucleosidiche modificate.
La piattaforma SNRMA-MS può essere sfruttata per stabilire profili di modifica dell'RNA di singoli neuroni e studiare la loro relazione con i tessuti di massa. I neuroni R2 sono grandi (~ 500 μm di diametro) cellule colinergiche che risiedono nel ganglio addominale. SNRMA-MS è stato utilizzato per analizzare le modifiche dell'RNA nei neuroni R2 e nel ganglio addominale di massa circostante (Figura 2A). Le aree di picco normalizzate per le modifiche dell'RNA in ciascun campione neurone/ganglio (n = 7) sono state utilizzate come input per la PCA, rivelando che i neuroni R2 presentano profili di modifica dell'RNA distinti rispetto ai gangli in cui risiedono (Figura 2B). Ciò è evidenziato dai punti dati per i neuroni R2 e i gangli addominali che occupano diverse regioni del grafico del punteggio PCA. Un ulteriore supporto per modelli nucleosidici modificati unici è stato ottenuto da una coorte separata di animali (n = 7) in cui sono stati eseguiti confronti a coppie per 13 modifiche dell'RNA che sono state comunemente rilevate sia nei singoli neuroni che nel tessuto di massa (Figura 2C). Due nucleosidi modificati, pseudouridina (Ψ) e 2'-O-metilguanosina (Gm), avevano un'abbondanza significativamente più elevata nei gangli addominali rispetto ai neuroni R2. Osservando un sottoinsieme delle modifiche dell'RNA con coppie neurone-ganglio R2 indicate, tutti i gangli addominali mostravano livelli più elevati di Ψ e Gm, e tutti tranne uno dei neuroni R2 avevano abbondanze più elevate di 2'-O-metildenosina (Am) rispetto al loro ganglio corrispondente (Figura 2D). Nel complesso, i risultati di SNRMA-MS rivelano per la prima volta che i profili di modifica dell'RNA delle singole cellule possono divergere dalle cellule di massa nello stesso tessuto.
L'utilizzo di SNRMA-MS per studiare l'animale modello A. californica offre un'opportunità unica per caratterizzare i profili di modifica dell'RNA in neuroni identificati e funzionalmente distinti. Le modificazioni dell'RNA sono state valutate da SNRMA-MS in quattro cellule identificate: R2 e LPl1 (cellule omologhe, colinergiche coinvolte nel rilascio mucoso difensivo)32, MCC (neuroni modulatori serotoninergici coinvolti nell'alimentazione)33 e cellule B2 (neuroni peptidergici coinvolti nella motilità intestinale)34. La PCA di sei modificazioni dell'RNA in questi neuroni identificati, isolati immediatamente dopo il trattamento enzimatico o coltivati in una preparazione gangliare per 48 ore, ha dimostrato la stabilità e la dinamica degli epitrascrittomi a singola cellula. Le modifiche dell'RNA in cellule funzionalmente diverse hanno formato cluster unici nel grafico del punteggio mentre i neuroni Omologhi R2 / LPl1 co-raggruppati (Figura 3A). Il diagramma di carico mostra che le differenze sono state principalmente guidate dall'abbondanza di isomeri posizionali di metildonosina, tra cui 2'-O-metildidenosina (Am) e N1-metildenosina (m1A) (Figura 3B). Nella stessa analisi, è stato eseguito un confronto tra cellule appena isolate e cellule coltivate in situ (cioè nei rispettivi gangli) per 48 ore. Come mostrato nel grafico del punteggio PCA, cellule funzionalmente diverse sono rimaste distinguibili dai loro profili di modifica dell'RNA.
SNRMA-MS quantitativo può essere utilizzato per determinare quantità assolute di modifiche selezionate dell'RNA per le quali sono disponibili standard autentici. Sono state generate curve di calibrazione esterne per m1A, Ψ, 2'-O-metilcitidina (Cm), Am e m6A, e la quantità di ciascun nucleoside modificato nelle coppie di cellule MCC e R2/LPl1 è stata determinata per interpolazione (Figura 4A-E). Le quantità intracellulari di m1A e Ψ in due coppie di MCC simmetrici sembravano essere simili, mentre maggiori differenze nella quantità di queste modificazioni sono state osservate in tre coppie di cellule R2 / LPl1. Al fine di tenere conto delle differenze dovute alle dimensioni fisiche delle cellule studiate, le quantità di modifica dell'RNA sono state normalizzate dai volumi cellulari calcolati dalla misurazione ottica dei diametri cellulari per produrre concentrazioni intracellulari di nucleosidi modificati. Differenze significative nelle concentrazioni intracellulari di Cm e Am sono state osservate tra MCC e neuroni R2/LPl1. Nel complesso, SNRMA-MS consente la profilazione sia qualitativa che quantitativa delle modifiche dell'RNA in singoli neuroni.
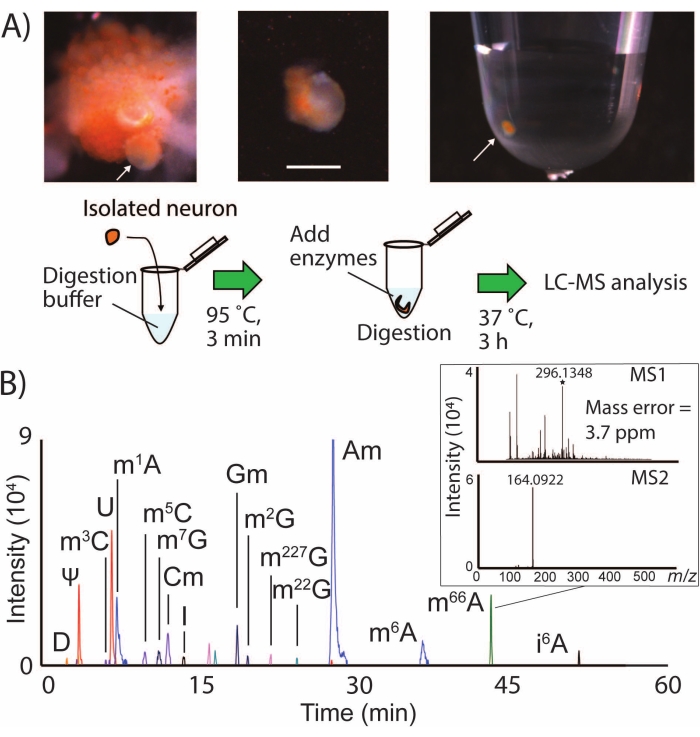
Figura 1: Flusso di lavoro SNRMA-MS e rilevamento di più modifiche dell'RNA in singoli neuroni da parte di LC-MS/MS. (A) Fotografie del ganglio buccale denutrito e dell'isolamento di un singolo neurone in una provetta campione. Barra della scala = 200 μm, le frecce indicano la cella B1 identificata. Viene inoltre mostrato un diagramma della procedura di preparazione del campione per l'analisi LC-MS/MS. (B) EIC sovrapposti per modifiche dell'RNA in un singolo neurone LPl1, con inserto che mostra spettri MS1 e MS2 per N6, N6-dimetildilamnosina (m66A). Vedere tabella 1 per i valori m/z utilizzati per generare EIC per nucleosidi modificati. Questa cifra è stata modificata da29. Fare clic qui per visualizzare una versione più grande di questa figura.
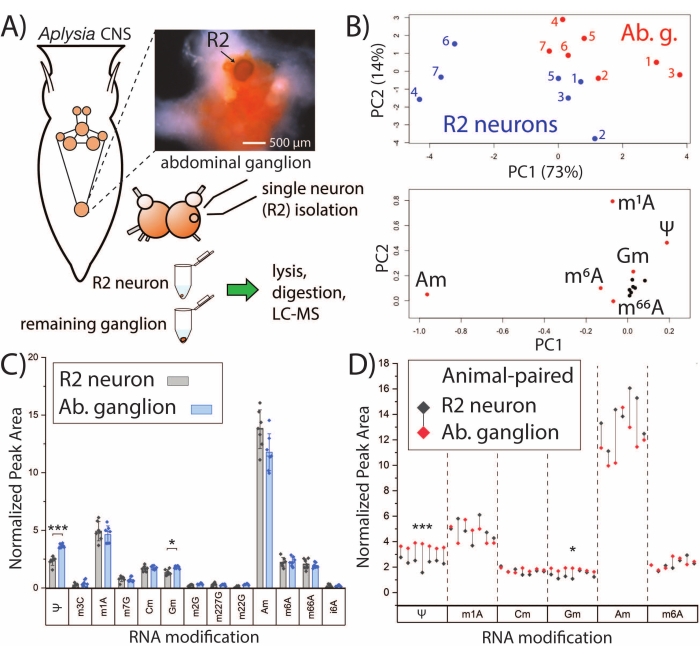
Figura 2: SNRMA-MS distingue i profili di modifica dell'RNA di singoli neuroni e tessuto di massa. (A) Schema del SNC di A. californica e flusso di lavoro per l'analisi dei neuroni R2 e del ganglio addominale circostante. La fotografia mostra il ganglio addominale e il neurone R2 (iniettato con colorante Fast Green per la visibilità). (B) Le aree di picco relative da 13 modifiche dell'RNA sono state utilizzate per generare i grafici del punteggio PCA (in alto) e di carico (in basso). (C) Confronto a coppie delle modificazioni dell'RNA nel ganglio addominale e nel neurone R2. Le barre di errore rappresentano ±1 deviazione standard (SD), *p < 0,05, ***p < 5 x 10−4, t-test accoppiato con correzione Bonferroni−Holm. (D) Confronto di alcune modificazioni dell'RNA dal pannello C in cui le coppie di ganglio R2-addominale per ciascun animale sono mostrate con linee di goccia. Questa cifra è stata modificata da29. Fare clic qui per visualizzare una versione più grande di questa figura.
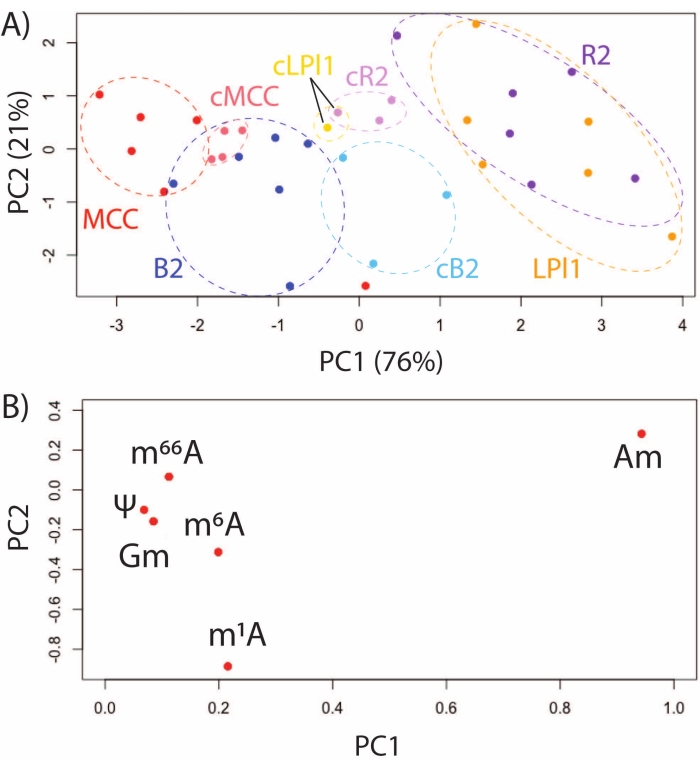
Figura 3: Profilazione delle modifiche dell'RNA in neuroni identificati, funzionalmente diversi dal SNC A. californica . (A) Grafico del punteggio PCA per MCC e cellule B2, R2 e LPl1 che sono state appena isolate o isolate dopo coltura in situ per 48 ore (indicata da cMCC, cB2, cR2, cLPl1) e (B) grafici di carico per sei modifiche dell'RNA comunemente rilevate in queste cellule. Questa cifra è stata modificata da29. Fare clic qui per visualizzare una versione più grande di questa figura.
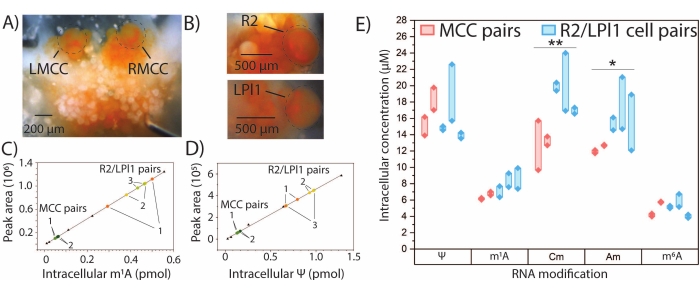
Figura 4: SNRMA-MS quantitativo nei neuroni di A. californica . Quantitative SNRMA-MS fornisce quantità assolute e concentrazioni intracellulari per diversi nucleosidi modificati in singoli neuroni A. californica identificati. Fotografie di (A) MCC sinistro e destro (LMCC e RMCC, rispettivamente) nel ganglio cerebrale e (B) R2 nel ganglio addominale e LPl1 nel ganglio pleurico. Le celle sono cerchiate per migliorare la visibilità. Grafici di calibrazione lineare per (C) m1A e (D) Ψ (triangoli) utilizzati per l'interpolazione di quantità nucleosidiche modificate in singole celle (punti colorati). Le coppie di cellule di ciascun animale sono etichettate 1-3. (E) Concentrazioni intracellulari di cinque nucleosidi modificati in coppie di cellule MCC e R2/LPl1. Linee spesse collegano coppie di celle. *p < 0,05, **p < 0,005, t-test accoppiato con correzione di Bonferroni-Holm, n = 2 animali (quattro cellule totali) per MCC e n = 3 animali (sei cellule totali) per R2/LPl1. Questa cifra è stata modificata da29. Fare clic qui per visualizzare una versione più grande di questa figura.
| Modifica dell'RNA | Abbreviazione | Ordine di eluizione (C18) | m/z per EIC | m/z per MS2 |
| diidrouridina | D | 1 | 247.09 | 115 |
| pseudouridina | Y | 2 | 245.08 | 209/179/155 |
| 3-metilcitidina | m3C · | 3 | 258.11 | 126 |
| N1-metildenosina | m1A · | 4 | 282.12 | 150 |
| 5-metilcitidina | m5C · | 5 | 258.11 | 126 |
| N7-metilguanosina | m7G · | 6 | 298.12 | 166 |
| 2'-O-metilcitidina | Centimetro | 7 | 258.11 | 112 |
| inosina | I6A · | 8 | 269.09 | 137 |
| 2'-O-metilguanosina | Gm | 9 | 298.12 | 152 |
| N2-metilguanosina | m2G · | 10 | 298.12 | 166 |
| N2,N2,N7-trimetilfguanosina | m227G | 11 | 326.15 | 194 |
| N2,N2,-dimetilguanosina | m22G | 12 | 312.13 | 180 |
| 2'-O-metildenosina | Sono | 13 | 282.12 | 136 |
| N6-metildenosina | m6A · | 14 | 282.12 | 150 |
| N6,N6-dimetildidenosina | m66A · | 15 | 296.14 | 164 |
| N6-isopentenyladenosina | i6A · | 16 | 336.17 | 204/136/148 |
Tabella 1: Modifiche dell'RNA rilevate in singoli neuroni di A. californica. Vengono forniti attributi per la caratterizzazione dei nucleosidi modificati, tra cui l'ordine di ritenzione LC, m/z per la generazione di EIC e i corrispondenti frammenti CID.
Discussione
SNRMA-MS sfrutta un approccio ottimizzato alla preparazione dei campioni, risultando in un piccolo volume di campioni compatibile con MS che può essere consegnato alla piattaforma LC-MS. Il pretrattamento enzimatico iniziale dei gangli del SNC determina sia la facilità con cui possono essere denutriti sia la durata dei singoli neuroni durante l'isolamento. Il ganglio cerebrale richiede spesso un trattamento enzimatico esteso a causa della sua guaina relativamente spessa rispetto ai gangli buccali, pleurici e addominali. I singoli ricercatori che eseguono gli isolamenti del singolo neurone possono avere preferenze diverse su quanto dovrebbe essere duratura la guaina quando si usano microscissori e pinze fini. Tuttavia, è importante che i gangli non siano eccessivamente digeriti, in quanto ciò può portare a una perdita della loro integrità strutturale, perdita di informazioni posizionali che sono fondamentali per identificare le cellule bersaglio e / o lisi cellulare. Dopo l'isolamento dal ganglio, è importante assicurarsi che venga aspirato un volume minimo di mezzo di isolamento durante il trasferimento del neurone alla provetta del campione. Il mezzo ASW contiene un'alta concentrazione di sali che possono interferire con gli enzimi digestivi durante l'idrolisi dell'RNA e diluiranno anche il campione.
Durante la fase di lisi meccanica, è comune che i neuroni di grandi dimensioni (>250 μm di diametro) si rompano al passaggio ripetuto attraverso una micropipetta. I neuroni più piccoli possono richiedere ulteriore attenzione per garantire la lisi cellulare, che in genere comporta la pressione di un capillare di vetro sulla cellula. In questo caso, è possibile che un volume parziale del tampone del campione venga aspirato nel capillare a causa delle forze capillari. Questo volume può essere reimmesso nella provetta del campione applicando una pressione all'estremità del capillare di vetro per garantire che nessun campione venga perso.
I migliori risultati si ottengono includendo una fase di riscaldamento prima di aggiungere enzimi per la digestione dell'RNA. Ciò è probabilmente dovuto al fatto che il riscaldamento a 95 °C denatura le strutture secondarie dell'RNA che possono impedire l'attività della RNasi35 e diminuire la quantità di nucleosidi rilasciati dai biopolimeri dell'RNA. Esperimenti di controllo utilizzando campioni con spike con metionina marcata con isotopi stabili sono stati precedentemente eseguiti per indagare se gli artefatti di modifica dell'RNA indotti dal calore fossero la causa dell'aumento delle aree di picco osservate per le modifiche dell'RNA rispetto a un protocollo SNRMA-MS senza calore29. Tale etichettatura non è stata osservata, indicando che i segnali migliorati per le modifiche dell'RNA utilizzando il metodo SNRMA-MS ottimizzato erano dovuti a una digestione superiore dell'RNA.
I metodi convenzionali per isolare l'RNA totale dalle cellule comportano l'estrazione liquido-liquido (LLE) con fenolo-cloroformio e la successiva precipitazione, lavaggio e risospensione dell'RNA. Questi approcci si sono dimostrati utili per esperimenti di reazione a catena della polimerasi a trascrizione inversa in cui l'espressione di geni selezionati può essere facilmente monitorata nei neuroni A. californica identificati 36,37. Tuttavia, i metodi LLE non possono recuperare RNA sufficiente per il rilevamento di ribonucleosidi modificati da LC-MS, mentre il metodo SNRMA-MS qui descritto consente il rilevamento di numerose modifiche dell'RNA29. Al fine di valutare i profili di modifica di specifici tipi di RNA (ad esempio, rRNA, tRNA, mRNA) in campioni di tessuto / cellule alla rinfusa, l'estrazione in fase solida a scambio anionico24, l'arricchimento basato su sonda di ibridazione38 e il frazionamento cromatografico39 approcci sono stati applicati, ma metodi simili non sono ancora disponibili per la purificazione dell'RNA a cellula singola. Lo sviluppo di approcci di frazionamento dell'RNA in grado di isolare specifici tipi di RNA da singole cellule fornirebbe ulteriori informazioni sulla funzione delle modifiche dell'RNA.
SNRMA-MS ha rivelato un'eterogeneità precedentemente non caratterizzata nel panorama di modificazione dell'RNA di singoli neuroni in A. californica ed è concepibile che esistano profili PTM altrettanto distinti tra le cellule di mammifero. Poiché le cellule di mammifero sono relativamente piccole rispetto ai grandi neuroni di A. californica analizzati in questo protocollo, sono necessari miglioramenti nella gestione di piccoli volumi di campioni per facilitare limiti di rilevamento più bassi. Sebbene attualmente SNRMA-MS sia limitato a volumi di ~ 5 μL, si prevede che sia possibile ottenere miglioramenti sostanziali incorporando dispositivi di gestione dei liquidi microfluidici nel flusso di lavoro. Inoltre, l'implementazione di isolamenti cellulari automatizzati o semi-automatizzati aumenterebbe la produttività del campione e consentirebbe l'analisi della modifica dell'RNA a cellula singola di popolazioni cellulari più grandi. Interfacciandosi con le separazioni LCnanoflow 27, sarà possibile ottenere la caratterizzazione dei segni epitrascrittomici in cellule ancora più piccole.
Divulgazioni
Gli autori non dichiarano interessi finanziari concorrenti.
Riconoscimenti
Questo lavoro è stato finanziato dal National Institute on Drug Abuse sotto il premio no. P30DA018310 e il National Human Genome Research Institute sotto il premio n. RM1HG010023. K.D.C. riconosce il sostegno di una borsa di studio post-dottorato del Beckman Institute. Il contenuto è di esclusiva responsabilità degli autori e non rappresenta necessariamente le opinioni ufficiali delle agenzie di finanziamento.
Materiali
| Name | Company | Catalog Number | Comments |
| Animals | |||
| Aplysia californica | National Resource for Aplysia (Miami, FL) | 150–250 g (adult) | |
| Benchtop equipment | |||
| Glass capillary puller | Sutter | P-97 | |
| Milli-Q water purification system | Millipore | ||
| Minicentrifuge for PCR tubes | LW Scientific | ZS-1 | |
| Optical Microscope | Zeiss | Stemi 2000C | |
| Thermocycler | Techne | EW-93945-01 | |
| HPLC column and consumables | |||
| Acclaim RSLC 120 C18 column | Thermo Scientific | 71399 | |
| Autosampler vials | Thermo Scientific | C4011-13 | |
| LC and MS Instrumentation and Software | |||
| DataAnalysis 4.4 software | Bruker | ||
| Dionex Ultimate 3000 nanoLC | Thermo Scientific | Equipped with online degasser, autosampler, and thermostatted column compartment | |
| Impact HD UHR QqTOF mass spectrometer | Bruker | Equipped with ESI source | |
| RStudio | RStudio | ||
| Microdissection tools | |||
| Microscissors extra fine vannas 3.5” | Roboz | RS-5640 | |
| Tungsten needles | Roboz | RS-6065 | |
| Reagents/Materials | |||
| 4-(2-hydroxyethyl)-1-piperazineethane-sulfonic acid (HEPES) | Fisher Scientific | H3375 | |
| Alkaline phosphatase | Worthington Biochemical Corp. | LS004081 | |
| Ammonium acetate | Honeywell | 17836 | |
| Benzonase (endonuclease from S. marcescens) | EMD Millipore | 70746-4 | |
| Bovine serum albumin | Sigma-Aldrich | A2153 | |
| Calcium chloride | Sigma-Aldrich | C4901 | |
| Gentamycin sulfate | Fisher Scientific | G1264 | |
| Magnesium chloride | Sigma-Aldrich | M9272 | |
| Magnesium sulfate | Sigma-Aldrich | 208094 | |
| Nucleosides test mix | Sigma-Aldrich | 47310-U | |
| Penicillin G | Sigma-Aldrich | P7794 | |
| Pentostatin | Sigma-Aldrich | SML0508 | |
| Phosphodiesterase I | Worthington Biochemical Corp. | LS003926 | |
| Protease type XIV from Streptomyces griseus | Sigma-Aldrich | P5147 | |
| Sodium chloride | Sigma-Aldrich | S9888 | |
| Standard glass capillaries | A-M Systems | 626000 | 1 mm o.d., 0.5 mm i.d., 4 in |
| Streptomycin sulfate | Sigma-Aldrich | S9137 |
Riferimenti
- Cantara, W. A., et al. The RNA modification database, RNAMDB: 2011 update. Nucleic Acids Research. 39, 195-201 (2011).
- Boccaletto, P., et al. MODOMICS: a database of RNA modification pathways. 2017 update. Nucleic Acids Research. 46, 303-307 (2017).
- Kimura, S., Waldor, M. K. The RNA degradosome promotes tRNA quality control through clearance of hypomodified tRNA. Proceedings of the National Academy of Sciences of the United States of America. 116 (4), 1394-1403 (2019).
- Helm, M. Post-transcriptional nucleotide modification and alternative folding of RNA. Nucleic Acids Research. 34 (2), 721-733 (2006).
- Rezgui, V. A. N., et al. tRNA tKUUU, tQUUG, and tEUUC wobble position modifications fine-tune protein translation by promoting ribosome A-site binding. Proceedings of the National Academy of Sciences of the United States of America. 110 (30), 12289-12294 (2013).
- Shanmugam, R., et al. Cytosine methylation of tRNA-Asp by DNMT2 has a role in translation of proteins containing poly-Asp sequences. Cell Discovery. 1 (1), 1-10 (2015).
- Jia, G., et al. N 6-Methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO. Nature Chemical Biology. 7 (12), 885-887 (2011).
- Li, X., et al. Transcriptome-wide mapping reveals reversible and dynamic N 1 -methyladenosine methylome. Nature Chemical Biology. 12 (5), 311-316 (2016).
- Krogh, N., et al. Profiling of 2′-O-Me in human rRNA reveals a subset of fractionally modified positions and provides evidence for ribosome heterogeneity. Nucleic Acids Research. 44 (16), 7884-7895 (2016).
- Babaian, A., et al. Loss of m1acp3Ψ Ribosomal RNA Modification Is a Major Feature of Cancer. Cell Reports. 31 (5), 107611 (2020).
- Chang, M., et al. Region-specific RNA m6A methylation represents a new layer of control in the gene regulatory network in the mouse brain. Open Biology. 7 (9), 170166 (2017).
- Wang, C. -. X., et al. METTL3-mediated m6A modification is required for cerebellar development. PLOS Biology. 16 (6), 2004880 (2018).
- Engel, M., et al. The role of m6A/m-RNA methylation in stress response regulation. Neuron. 99 (2), 389-403 (2018).
- Widagdo, J., et al. Experience-dependent accumulation of N6-Methyladenosine in the prefrontal cortex is associated with memory processes in mice. Journal of Neuroscience. 36 (25), 6771-6777 (2016).
- Eberwine, J., et al. Analysis of gene expression in single live neurons. Proceedings of the National Academy of Sciences of the United States of America. 89 (7), 3010-3014 (1992).
- Cong, Y., et al. Ultrasensitive single-cell proteomics workflow identifies >1000 protein groups per mammalian cell. Chemical Science. 12 (3), 1001-1006 (2021).
- Rubakhin, S. S., Romanova, E. V., Nemes, P., Sweedler, J. V. Profiling metabolites and peptides in single cells. Nature Methods. 8 (4), 20-29 (2011).
- Nemes, P., Knolhoff, A. M., Rubakhin, S. S., Sweedler, J. V. Metabolic differentiation of neuronal phenotypes by single-cell capillary electrophoresis-electrospray ionization-mass spectrometry. Analytical Chemistry. 83 (17), 6810-6817 (2011).
- Lovatt, D., et al. Transcriptome in vivo analysis (TIVA) of spatially defined single cells in live tissue. Nature Methods. 11 (2), 190-196 (2014).
- Kærn, M., Elston, T. C., Blake, W. J., Collins, J. J. Stochasticity in gene expression: from theories to phenotypes. Nature Reviews Genetics. 6 (6), 451-464 (2005).
- Chan, C. T. Y., et al. A quantitative systems approach reveals dynamic control of tRNA modifications during cellular stress. PLOS Genetics. 6 (12), 1001247 (2010).
- Sun, C., Jora, M., Solivio, B., Limbach, P. A., Addepalli, B. The effects of ultraviolet radiation on nucleoside modifications in RNA. ACS Chemical Biology. 13 (3), 567-572 (2018).
- Heiss, M., Hagelskamp, F., Marchand, V., Motorin, Y., Kellner, S. Cell culture NAIL-MS allows insight into human tRNA and rRNA modification dynamics in vivo. Nature Communications. 12 (1), 389 (2021).
- Clark, K. D., Lee, C., Gillette, R., Sweedler, J. V. Characterization of neuronal RNA modifications during non-associative learning in aplysia reveals key roles for tRNAs in behavioral sensitization. ACS Central Science. 7 (7), 1183-1190 (2021).
- Basanta-Sanchez, M., Temple, S., Ansari, S. A., D'Amico, A., Agris, P. F. Attomole quantification and global profile of RNA modifications: Epitranscriptome of human neural stem cells. Nucleic Acids Research. 44 (3), 26 (2016).
- Huang, W., et al. Determination of DNA and RNA methylation in circulating tumor cells by mass spectrometry. Analytical Chemistry. 88 (2), 1378-1384 (2016).
- Sarin, L. P., et al. Nano LC-MS using capillary columns enables accurate quantification of modified ribonucleosides at low femtomol levels. RNA. 24 (10), 1403-1417 (2018).
- Clark, K. D., Philip, M. C., Tan, Y., Sweedler, J. V. Biphasic liquid microjunction extraction for profiling neuronal RNA modifications by liquid chromatography-tandem mass spectrometry. Analytical Chemistry. 92 (18), 12647-12655 (2020).
- Clark, K. D., Rubakhin, S. S., Sweedler, J. V. Single-neuron RNA modification analysis by mass spectrometry: Characterizing RNA modification patterns and dynamics with single-cell resolution. Analytical Chemistry. 93 (43), 14537-14544 (2021).
- Guise, O. L., Ahner, J. W., Jung, M. -. C., Goughnour, P. C., Yates, J. T. Reproducible electrochemical etching of tungsten probe tips. Nano Letters. 2 (3), 191-193 (2002).
- Peng, W., Wu, Z., Song, K., Zhang, S., Li, Y., Xu, M. Regulation of sleep homeostasis mediator adenosine by basal forebrain glutamatergic neurons. Science. 369 (6508), (2020).
- Rayport, S. G., Ambron, R. T., Babiarz, J. Identified cholinergic neurons R2 and LPl1 control mucus release in Aplysia. Journal of Neurophysiology. 49 (4), 864-876 (1983).
- Rosen, S. C., Weiss, K. R., Goldstein, R. S., Kupfermann, I. The role of a modulatory neuron in feeding and satiation in Aplysia: effects of lesioning of the serotonergic metacerebral cells. Journal of Neuroscience. 9 (5), 1562-1578 (1989).
- Lloyd, P. E., Kupfermann, I., Weiss, K. R. Central peptidergic neurons regulate gut motility in Aplysia. Journal of Neurophysiology. 59 (5), 1613-1626 (1988).
- Crain, P. F. Preparation and enzymatic hydrolysis of DNA and RNA for mass spectrometry. Methods in Enzymology. 193, 782-790 (1990).
- Kadakkuzha, B. M., et al. Age-associated bidirectional modulation of gene expression in single identified R15 neuron of Aplysia. BMC Genomics. 14 (1), 880 (2013).
- Akhmedov, K., Kadakkuzha, B. M., Puthanveettil, S. V. Aplysia ganglia preparation for electrophysiological and molecular analyses of single neurons. Journal of Visualized Experiments: JoVE. (83), e51075 (2014).
- Tardu, M., Jones, J. D., Kennedy, R. T., Lin, Q., Koutmou, K. S. Identification and quantification of modified nucleosides in saccharomyces cerevisiae mRNAs. ACS Chemical Biology. 14 (7), 1403-1409 (2019).
- Heiss, M., Reichle, V. F., Kellner, S. Observing the fate of tRNA and its modifications by nucleic acid isotope labeling mass spectrometry: NAIL-MS. RNA Biology. 14 (9), 1260-1268 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon