Method Article
Характеристика модификаций РНК в одиночных нейронах с помощью масс-спектрометрии
В этой статье
Резюме
Посттранскрипционные модификации РНК представляют собой недостаточно изученный слой регуляции трансляции, который недавно был связан с пластичностью центральной нервной системы. Здесь описан подход пробоподготовки и жидкостной хроматографии-тандемной масс-спектрометрии для одновременной характеристики многочисленных модификаций РНК в отдельных нейронах.
Аннотация
Посттранскрипционные модификации (ПТМ) РНК представляют собой недостаточно изученный механизм, участвующий в регуляции трансляции в центральной нервной системе (ЦНС). Недавние данные связывают специфические модификации нейронной РНК с парадигмами обучения и памяти. К сожалению, традиционные методы обнаружения этих эпитранскриптомных особенностей способны характеризовать только высокообильные модификации РНК в объемных тканях, исключая оценку уникальных профилей PTM, которые могут существовать для отдельных нейронов в активированных поведенческих цепях. В этом протоколе описан подход — анализ модификации РНК с одним нейроном методом масс-спектрометрии (SNRMA-MS) — для одновременного обнаружения и количественной оценки многочисленных модифицированных рибонуклеозидов в отдельных нейронах. Подход подтвержден с использованием отдельных нейронов морского моллюска, Aplysia californica, начиная с хирургической изоляции и ферментативного лечения основных ганглиев ЦНС для выявления тел нейронных клеток, с последующей ручной изоляцией одного нейрона с использованием острых игл и микропипетки. Далее проводится механическая и термическая обработка образца в небольшом объеме буфера для высвобождения РНК из отдельной клетки для последующего переваривания РНК. Модифицированные нуклеозиды затем идентифицируются и количественно оцениваются с использованием оптимизированного метода жидкостной хроматографии-масс-спектрометрии. SNRMA-MS используется для установления паттернов модификации РНК для одиночных, идентифицированных нейронов из A. californica , которые имеют известные морфологии и функции. Приведены примеры качественной и количественной SNRMA-MS, которые подчеркивают гетерогенное распределение модификаций РНК по отдельным нейронам в нейронных сетях.
Введение
Модификации канонических нуклеозидов РНК все чаще признаются за их мириады ролей в регуляции трансляции белка. На сегодняшний день зарегистрировано более 150 уникальных модификаций РНК, которые варьируются по сложности от метилирования, добавления гетероатома, до конъюгации с клеточными метаболитами 1,2. Этот расширенный алфавит РНК, также известный как эпитранскриптом, генерируется ферментативными писателями и отвечает за изменение стабильности3, сворачивания4 и эффективности трансляции 5,6 клеточных РНК. Некоторые модификации РНК также могут быть обращены вспять с помощью ферментативных ластиков 7,8, тогда как другие добавляются к РНК субстехиометрически 9,10, что создает сложный ландшафт модифицированных и немодифицированных последовательностей РНК в биологических системах.
О важности модификаций РНК в биологической функции свидетельствует неравномерное распределение модификаций по различным органам и тканям, включая субрегионы центральной нервной системы (ЦНС)11. Эта химическая гетерогенность была связана с развитием ЦНС12, реакцией на стресс13 и пластичностью, зависящей от активности14. Субрегионы ЦНС также включают гетерогенные популяции клеток, в которых отдельные клетки демонстрируют различные химические профили 15,16,17,18. Даже отдельные клетки одного типа могут демонстрировать уникальные транскриптомы, отчасти из-за микроокружениятканей 19 и стохастической экспрессии генов20. Однако, хотя характеристика одноклеточного транскриптома является несколько рутинной, не существует аналогичных методов установления одноклеточных эпитранскриптомов для множественных модификаций РНК. Новые подходы, способные профилировать распределение модификаций РНК в отдельных клетках, необходимы для комплексного анализа клеточной гетерогенности и регуляторного влияния посттранскрипционных модификаций (ПТМ) в ЦНС и других биологических системах.
Одновременное измерение многочисленных модификаций РНК в объемных клетках / тканях легко осуществляется с использованием методов жидкостной хроматографии-тандемной масс-спектрометрии (LC-MS / MS). Для анализа LC-MS/MS модифицированных рибонуклеозидов РНК извлекают из клеток (обычно 10 3-10 6 клеток), очищают осаждением и реуспензией, а затем переваривают в нуклеозиды. Затем образец смеси, состоящей из канонических и модифицированных нуклеозидов, вводят в систему LC-MS для разделения и обнаружения аналитов, что приводит к определению полного комплемента модификаций РНК в организме 21,22,23. LC-MS/MS недавно использовался для определения 26 модификаций РНК в ЦНС нейробиологической модели Aplysia californica (A. californica). Обилие некоторых из этих эпитранскриптомных меток продемонстрировало зависящую от времени и региона динамику, которая коррелировала с поведенческими изменениями у животного24. Однако было возможно обнаружить модификации РНК только в объемных образцах, содержащих >103 клеток из-за ограниченной чувствительности метода. Эти более крупные образцы, вероятно, скрывали уникальные и функционально важные профили модификации РНК отдельных клеток со средними популяционными показателями. Хотя тщательный контроль условий подготовки образцов улучшил пределы обнаружения модификаций РНК в малообъемных образцах 25,26,27,28, по-прежнему существует потребность в аналитических методах, которые могут обнаруживать и количественно оценивать множественные модифицированные рибонуклеозиды в отдельных клетках.
Этот протокол вводит анализ модификации РНК с одним нейроном с помощью масс-спектрометрии (SNRMA-MS), который позволяет обнаружить более десятка модификаций РНК в отдельных нейронах из ЦНС A. californica29. Подход состоит из хирургической изоляции одиночных идентифицированных клеток из основных ганглиев ЦНС с последующим оптимизированным рабочим процессом подготовки образцов, включающим механический лизис клеток, денатурацию РНК и ферментативный гидролиз в MS-совместимом буфере. Идентификация и количественная оценка посттранскрипционно модифицированных нуклеозидов затем осуществляется с использованием LC-MS / MS. SNRMA-MS удовлетворяет неудовлетворенную потребность в области анализа модификации РНК, облегчая получение профилей модификации посттранскрипционной РНК для отдельных нейронов и имеет потенциал для будущего применения к другим типам клеток.
протокол
1. Подготовка материалов и растворов
- Подготовьте искусственную морскую воду (ASW) с 460 мМ NaCl, 10 мМ KCl, 10 мМ CaCl2, 22 мМ MgCl2, 26 мМ MgSO4 и 10 мМ HEPES в воде, полученной из системы строгой очистки. Отрегулируйте pH до 7,8, используя 1 M NaOH или HCl. Обычно готовят 1 л ASW и хранят при 14 °C до использования.
- Готовят раствор ASW-антибиотиков с 10 000 Ед/мл пенициллина G, 10 мкг/мкл стрептомицина и 10 мкг/мкл гентамицина и хранят при -20 °C. Непосредственно перед экспериментом разморозьте и разбавьте замороженный раствор антибиотиков 1:100 в 20-40 мл ПРОТИВОЛОД. Конечная концентрация антибиотиков в рабочем растворе ASW составляет 100 Ед/мл пенициллина G, 100 мкг/мл стрептомицина и 100 мкг/мл гентамицина.
- Получают буфер переваривания РНК, комбинируя 1 мкл 10 мкг/мкл бычьего сывороточного альбумина, 0,5 мкл 0,5 мкг/мкл пентостатина, 0,495 мкл 2 Ед/мкл щелочной фосфатазы, 1 мкл 0,1 Ед фосфодиэстеразы I (в 10 мМ MgCl2) и 0,38 мкл эндонуклеазы из Serratia marcescens (25 ЕД) на образец. При анализе нескольких образцов подготовьте мастер-смесь в отдельной пробирке, содержащей объем каждого реагента, умноженный на количество анализируемых образцов, плюс еще один для учета случайных потерь реагента от этапов переноса пипетки.
- Подготовьте несколько острых игл (стеклянных или металлических) для ручной изоляции нейронов. Изготовьте металлические иглы в домашних условиях путем электрохимического травления вольфрамовой проволоки как в30 или приобретите их. Подготовьте стеклянные иглы из толстых или стандартных настенных боросиликатных стеклянных капилляров (наружный диаметр 1 мм) с помощью съемника микропипетки. Для настоящего способа держите диаметр наконечника иглы в пределах 1-5 мкм, с длиной горлышка 100-150 мкм.
ПРИМЕЧАНИЕ: Изготовление как металлических, так и стеклянных игл может быть оптимизировано для производства инструментов, которые соответствуют индивидуальным потребностям исследователя и конкретной исследуемой биологической модели.
2. Выделение одного нейрона
- Охладить раствор 0,33 MMgCl2 до 14 °C. Используя шприц объемом 50 мл, обезболивают A. californica (150-250 г) путем введения раствора MgCl2 в полость тела животного. Наилучшие результаты получены при соотношении объема раствора (мл) к массе тела животного (г) 1:3. Подождите примерно 3 минуты, пока животное расслабится, гарантируя, что оно не проявляет сокращений тела в ответ на тактильную стимуляцию.
- Поместите брюшную сторону животного (со стороны ноги) вверх в рассекающий лоток. Рассекните животное хирургическими ножницами одним тупым кончиком, расположенным в сторону животного, аккуратно сделав продольный разрез через ногу.
- Прикрепите ростральную, каудальную и боковую стороны тела животного, чтобы обнажить внутренние органы и ганглии ЦНС, расположенные в полости тела. Изолируйте крупные ганглии ЦНС от животного путем хирургического разрыва нервов и некоторых соединительных соединений, происходящих из ганглиев.
- Погрузить ганглии в раствор протеазы XIV типа из Streptomyces griseus (10 мг/мл в растворе ASW-антибиотиков) и инкубировать при 34 °C в течение 30 мин или 1 ч (для церебрального ганглия).
ПРИМЕЧАНИЕ: Продолжительность инкубации зависит от сезона, размера и состояния животного, а также целевых нейронов. Выделение одних нейронов требует более длительной инкубации, чем других, и должно определяться экспериментально. - Промыть ганглии 6x раствором ASW-антибиотиков и перенести все ганглии в силиконовую форму с полимерным покрытием, заполненную раствором ASW-антибиотиков, используя полипропиленовую трансферную пипетку, которая была разрезана до отверстия ~ 5 мм. Обработайте пипетку 1 мг/мл бычьего сывороточного альбумина в ASW, чтобы свести к минимуму прилипание ганглия к пипетке (необязательно). Держите ганглии погруженными в противолодочную оборону в любое время.
ПРИМЕЧАНИЕ: Ферментативное лечение снижает механическую стабильность нейронов и окружающей соединительной ткани и, как следствие, нейронные мембраны могут быть повреждены из-за воздействия воздуха во время переноса ганглиев. - Зажмите ганглии и используйте микроножницы и тонкие щипцы, чтобы удалить ганглиозные оболочки. При достаточно сильной ферментативной обработке для снятия оболочки используют стеклянные или металлические иглы.
- Визуально идентифицируют интересующие нейроны A. californica . В этой работе были изучены следующие клетки: R2 в брюшном ганглии, LPl1 в плевральном ганглии, метацеребральные клетки (MCC) в мозговом ганглии и клетки B2 в щечном ганглии. Сделайте оптические изображения всех нейронных и ганглионных препаратов с помощью калиброванного микроскопа с общим увеличением в 20 раз, чтобы определить размеры и объемы каждого типа клеток.
- Используя натянутый стеклянный капилляр или острые вольфрамовые иглы, тщательно изолируйте идентифицированную клетку от объемного ганглия.
- Вытяните небольшое количество (1 мкл) ПРОТИВОЛО в пластиковую микропипетку, а затем перенесите изолированную клетку в пробирку для ПЦР, содержащую 4 мкл 0,365 М ацетата аммония (рН 9,2). Для пустых измерений собирают 5 мкл аликвот раствора ASW-антибиотиков из чашки, содержащей ганглий, и смешивают с буфером пищеварения (описано ниже).
3. Лизис клеток и сбраживание РНК для SNRMA-MS
- Лизируют изолированные нейроны путем повторной аспирации и обхода микропипеткой (~100 мкм внутреннего диаметра) в 0,365 М ацетата аммония. Некоторые более мелкие клетки могут не сразу разорваться; чтобы лизировать их, прикладывайте давление по всему диаметру клетки с помощью натянутого стеклянного капилляра.
- Используйте термоциклер для нагрева образцов со следующей температурной программой: 95 °C в течение 3 мин, 10 °C в течение 3 мин, держите при 10 °C. Извлеките пробку из термоциклера.
- Добавьте 3,375 мкл буфера для переваривания РНК для каждого образца и перемешайте раствор с помощью микропипетки, выведя и дозируя раствор несколько раз. Используйте миниатюрную настольную центрифугу при 2700 х г в течение 30 с, чтобы раскрутить любые капли жидкости, цепляющиеся за стенки трубки ПЦР.
- Инкубируйте образцы в термоциклере при 37 °C в течение 3 ч, а затем держите при 10 °C (нагретая крышка установлена на ON). Сразу после того, как образец остынет до 10 °C, переместите 7 мкл раствора во флакон автосамплера, оснащенный вставкой на 250 мкл, стараясь избежать образования пузырьков в трубке автосамплера.
4. Жидкостная хроматография-тандемная масс-спектрометрия
ПРИМЕЧАНИЕ: Анализ однонейронных дайджестов и аутентичных модифицированных нуклеозидных стандартов с использованием системы LC-MS/MS, оснащенной электрораспылительным источником ионизации и шестипортовым отводным клапаном.
- Для подготовки системы LC к разделению канонических и модифицированных нуклеозидов уравновешивают колонну C18 (150 мм x 2,1 мм, размер частиц 2,2 мкм, диаметр пор 120 Å) с 99% подвижной фазой A (5 мМ ацетата аммония, рН 5,6) и 1% подвижной фазой B (60/40 подвижной фазы A/ацетонитрила (ACN)) со скоростью потока 0,2 мл/мин в течение 12 мин при 36 °C. Используйте растворители класса LC-MS для приготовления подвижных фаз.
- Пока LC уравновешивается, калибруйте масс-спектрометр, вводя 1 мМ раствор ацетата натрия в 50/50 ACN/воду в масс-спектрометр через шприцевой насос со скоростью потока 5 мкл/мин. После калибровки повторно подключите поток LC к масс-спектрометру.
- Программируют следующие параметры линейного градиента: 1% B в течение 0-5 мин, 5% B в течение 9 мин, 7% B в 11 мин, 10% B в 13 мин, 15% B в течение 32 мин, 40% B в 38 мин, 50% B в 43 мин, 100% B в 50 мин, 100% B в 60 мин, 1% B в 61 мин, и 12-минутное повторное уравновешивание при 1% В перед следующей инъекцией.
- Работа прибора МС в положительном режиме со следующими параметрами: капиллярное напряжение установлено на уровне 4 500 В, температура сушки 275 °C,N2 сушильного газа 5 л/мин и распылительный газ 1 бар. Установите отводной клапан на отходы в течение первых 2 минут анализа и на источник в течение оставшейся части пробега.
- Собирайте масс-спектры в диапазоне м /з 110-600. Выбор ионов для диссоциации, вызванной столкновением, при 35-40 эВ в течение цикла 3 с использованием предпочтительного списка масс, построенного с использованием базы данных2 и окна изоляции ± 0,5. Используйте активное исключение, чтобы исключить ионы из фрагментации после трех спектров.
- Установите динамическое получение спектров MS/MS для ионов с интенсивностью выше и ниже 50 000 при 4 Гц и 1 Гц соответственно и минимальное пороговое значение для выбора ионов при 1 990 отсчетах.
- Для количественных SNRMA-MS построить калибровочные кривые с использованием пиковых областей экстрагированной ионной хроматограммы (EIC), полученных для модифицированных нуклеозидных эталонов при минимум пяти концентрациях, чтобы обеспечить интерполяцию неизвестных эндогенных концентраций анализируемого вещества.
НЕЙРОНЫ, полученные от животных с массой тела 150-250 г, обычно требуют калибровочных кривых для модифицированных нуклеозидов в диапазоне от 0,02 пмоль до 2 пмоль, но эти значения могут варьироваться в зависимости от чувствительности прибора.
5. Анализ данных
- Генерация EIC для модифицированных нуклеозидов (m/z из значений базы данных2). Проверьте идентичность предполагаемых модифицированных нуклеозидов, сравнив их спектры MS2 и характеристики удержания LC со значениями базы данных2. См. Таблицу 1 для списка типичных модификаций РНК, обнаруженных в отдельных нейронах из A. californica.
- Вручную интегрируйте пики, соответствующие модифицированным и каноническим нуклеозидам, и записывайте эти значения в электронную таблицу. Нормализуйте пиковую область для каждого модифицированного нуклеозида суммой пиковых областей для канонических цитидина, уридина и гуанозина, обнаруженных в образце.
ПРИМЕЧАНИЕ: Аденозин не включен в нормализацию из-за его роли в ЦНС в качестве динамического нейромодулятора31. - Построить матрицу данных, состоящую из каждого образца одного нейрона и соответствующих нормализованных пиковых областей для модифицированных нуклеозидов, которые демонстрировали отношение сигнал/шум >10. Выполните анализ главных компонентов (PCA) и отобразите первые два основных компонента на графике оценки. Построение линейных калибровочных кривых из пиковых областей KIK, полученных из последовательного разбавления нуклеозидных эталонов, и расчет концентраций обнаруженных аналитов.
Результаты
SNRMA-MS включает ручную изоляцию идентифицированных нейронов в небольшие объемы образцов для лизиса, пищеварения и анализа LC-MS / MS (рисунок 1A). Этот рабочий процесс регулярно обнаруживал более десятка модификаций РНК в отдельных нейронах из ЦНС A. californica (рисунок 1B), представляя собой покрытие почти половины известного эпитранскриптома этого животного24 в одной клетке. Например, подвергание нейрона LPl1 (диаметр ~500 мкм) SNRMA-MS привело к обнаружению 15 ± 1 модификации РНК (n = 3). Модифицированные нуклеозиды были положительно идентифицированы на основе трех признаков: свойств удержания LC, точной массы и профилей фрагментации MS2 по сравнению со значениями, представленными в базе данных2. В таблице 1 приведен список всех модификаций РНК, обнаруженных в нейроне LPl1. Использованный для этих экспериментов квадрупольный масс-спектрометр высокого разрешения позволил получить точность массы 4 ppm, а также обнаружить характерный ион фрагмента MS2 при m/z 164 для модифицированного нуклеозида N6,6-диметиладенозина (m66A, рисунок 1B). В сочетании с данными разделения LC, которые согласуются с результатами, депонированными в базе данных (m66A элюируется после N6-метиладенозина (m6A)), подход SNRMA-MS продемонстрировал правильное присвоение модифицированных нуклеозидных идентичностей.
Платформа SNRMA-MS может быть использована для создания профилей модификации РНК отдельных нейронов и исследования их связи с объемными тканями. Нейроны R2 представляют собой большие (~ 500 мкм в диаметре) холинергические клетки, которые находятся в брюшном ганглии. SNRMA-MS использовался для анализа модификаций РНК в нейронах R2, а также в окружающих объемных брюшных ганглиях (рисунок 2A). Нормализованные пиковые области для модификаций РНК в каждом образце нейрона/ганглия (n = 7) использовались в качестве входных данных для PCA, показывая, что нейроны R2 демонстрируют различные профили модификации РНК по сравнению с ганглиями, в которых они находятся (рисунок 2B). Об этом свидетельствуют данные о нейронах R2 и брюшных ганглиях, занимающих различные области графика оценки PCA. Дальнейшая поддержка уникальных модифицированных нуклеозидных паттернов была получена из отдельной когорты животных (n = 7), в которой были выполнены парные сравнения для 13 модификаций РНК, которые обычно обнаруживались как в отдельных нейронах, так и в объемной ткани (рисунок 2C). Два модифицированных нуклеозида, псевдоуридин (Ψ) и 2'-O-метилгуанозин (Gm), были значительно выше в абдоминальных ганглиях по сравнению с нейронами R2. При просмотре подмножества модификаций РНК с указанными парами нейрон-ганглий R2 все брюшные ганглии демонстрировали более высокие уровни Ψ и Gm, и все, кроме одного из нейронов R2, имели более высокое содержание 2'-O-метиладенозина (Am), чем их соответствующий ганглий (рисунок 2D). В целом, результаты SNRMA-MS впервые показывают, что профили модификации РНК отдельных клеток могут отличаться от объемных клеток в одной и той же ткани.
Использование SNRMA-MS для исследования модели животного A. californica дает уникальную возможность охарактеризовать профили модификации РНК в идентифицированных, функционально отличных нейронах. Модификации РНК оценивали с помощью SNRMA-MS в четырех идентифицированных клетках: R2 и LPl1 (гомологичные холинергические клетки, участвующие в защитном высвобождении слизистой)32, MCC (серотонинергические модулирующие нейроны, участвующие в питании)33 и клетки B2 (пептидергические нейроны, участвующие в подвижности кишечника)34. PCA шести модификаций РНК в этих идентифицированных нейронах, либо выделенных сразу после ферментативного лечения, либо культивированных в ганглиозном препарате в течение 48 ч, продемонстрировали стабильность и динамику одноклеточных эпитранскриптомов. Модификации РНК в функционально различных клетках образовывали уникальные кластеры на оценочном графике, в то время как гомологичные нейроны R2/LPl1 совместно группировались (рисунок 3A). График загрузки показывает, что различия были обусловлены главным образом обилием позиционных изомеров метиладенозина, включая 2'-O-метиладенозин (Am) и N1-метиладенозин (m1A) (рисунок 3B). В том же анализе проводилось сравнение свежеизолированных клеток и клеток, культивируемых in situ (т.е. в соответствующих ганглиях) в течение 48 ч. Как показано на графике оценки PCA, функционально разные клетки оставались различимыми по их профилям модификации РНК.
Количественная SNRMA-MS может быть использована для определения абсолютных количеств избранных модификаций РНК, для которых доступны аутентичные стандарты. Внешние калибровочные кривые были сгенерированы для m1A, Ψ, 2'-O-метилцитидина (Cm), Am иm6A, а количество каждого модифицированного нуклеозида в парах клеток MCC и R2/LPl1 определяли путем интерполяции (рисунок 4A-E). Внутриклеточные величины m1A и Ψ в двух парах симметричных MCC оказались похожими, в то время как большие различия в количествах этих модификаций наблюдались в трех парах клеток R2 / LPl1. Чтобы учесть различия из-за физического размера изученных клеток, величины модификации РНК были нормализованы клеточными объемами, рассчитанными из оптического измерения диаметров клеток, чтобы получить внутриклеточные концентрации модифицированных нуклеозидов. Значительные различия во внутриклеточных концентрациях Cm и Am наблюдались между MCC и нейронами R2/LPl1. В целом, SNRMA-MS обеспечивает как качественное, так и количественное профилирование модификаций РНК в отдельных нейронах.
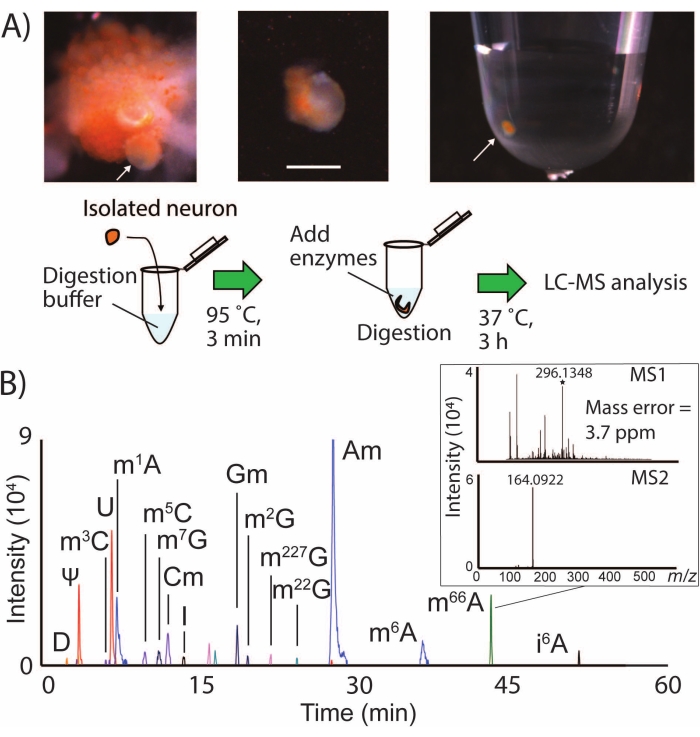
Рисунок 1: Рабочий процесс SNRMA-MS и обнаружение множественных модификаций РНК в отдельных нейронах с помощью LC-MS/MS. (A) Фотографии дешифрованного буккального ганглия и выделения одного нейрона в пробку. Шкала = 200 мкм, стрелки указывают на идентифицированную ячейку B1. Также показана схема процедуры пробоподготовки для анализа LC-MS/MS. (B) Наложенные EIC для модификаций РНК в одном нейроне LPl1 со вставкой, показывающей спектры MS1 и MS2 для N6, N6-диметиладенозина (m66A). В таблице 1 приведены значения m/z , используемые для генерации ОИК для модифицированных нуклеозидов. Эта цифра была изменена с29. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
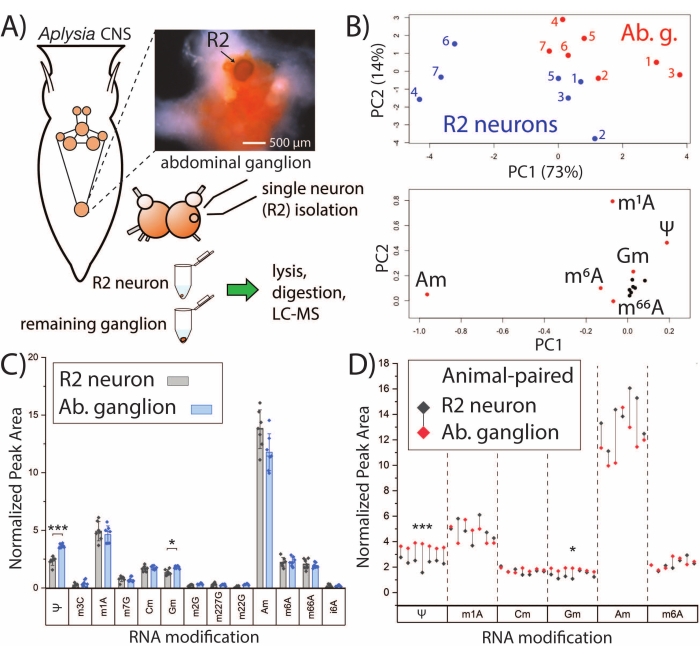
Рисунок 2: SNRMA-MS различает профили модификации РНК отдельных нейронов и объемной ткани. (A) Схема A. californica CNS и рабочий процесс для анализа нейронов R2 и окружающего брюшного ганглия. На фотографии изображен брюшной ганглий и нейрон R2 (вводится краситель Fast Green для видимости). (B) Относительные пиковые области из 13 модификаций РНК использовались для генерации графиков оценки PCA (вверху) и загрузки (внизу). (C) Попарное сравнение модификаций РНК в брюшном ганглии и нейроне R2. Полосы погрешностей представляют собой стандартное отклонение ±1 (SD), *p < 0,05, ***p < 5 x 10−4, парный t-тест с поправкой Бонферрони−Холма. (D) Сравнение отдельных модификаций РНК из панели C, в которой R2-абдоминальные ганглиозные пары для каждого животного показаны с помощью капельных линий. Эта цифра была изменена с29. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
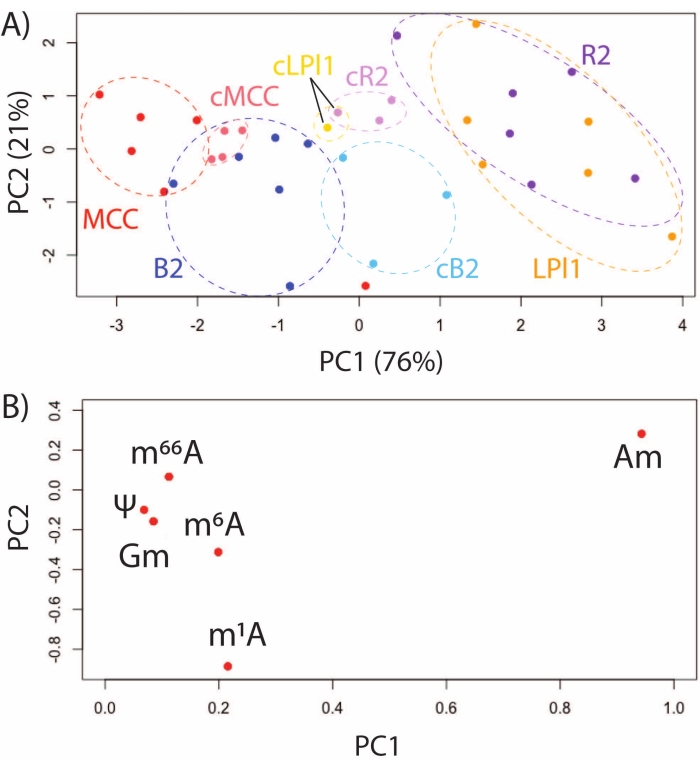
Рисунок 3: Профилирование модификаций РНК в идентифицированных, функционально отличающихся нейронах из графика оценки A. californica CNS. (A) PCA для MCC и клеток B2, R2 и LPl1, которые были либо недавно выделены, либо изолированы после культуры in situ в течение 48 ч (обозначенных cMCC, cB2, cR2, cLPl1) и (B) графиков загрузки для шести модификаций РНК, обычно обнаруживаемых в этих клетках. Эта цифра была изменена с29. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
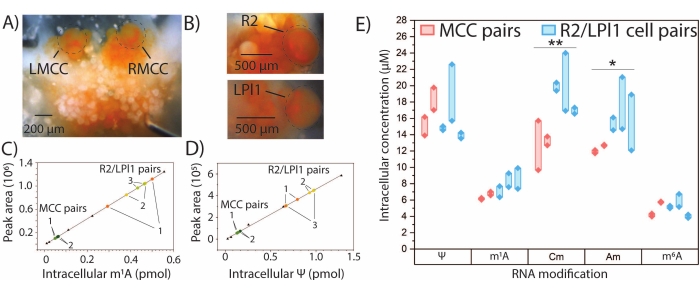
Рисунок 4: Количественная SNRMA-MS в нейронах A. californica . Количественный SNRMA-MS обеспечивает абсолютные количества и внутриклеточные концентрации для нескольких модифицированных нуклеозидов в одиночных, идентифицированных нейронах A. californica . Фотографии (A) левого и правого MCC (LMCC и RMCC соответственно) в мозговом ганглии и (B) R2 в брюшном ганглии и LPl1 в плевральном ганглии. Ячейки обведены для улучшения видимости. Линейные калибровочные графики для (C)m 1A и (D) Ψ (треугольники), используемые для интерполяции модифицированных нуклеозидных количеств в отдельных ячейках (цветные точки). Пары клеток от каждого животного маркируются 1-3. (E) Внутриклеточные концентрации пяти модифицированных нуклеозидов в клеточных парах MCC и R2/LPl1. Толстые линии соединяют пары ячеек. *p < 0,05, **p < 0,005, парный t-тест с коррекцией Бонферрони−Холма, n = 2 животных (всего четыре клетки) для MCC и n = 3 животных (всего шесть клеток) для R2/LPl1. Эта цифра была изменена с29. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
| Модификация РНК | Сокращение | Порядок элюирования (C18) | м/з для KIK | м/з для MS2 |
| дигидроуридин | D | 1 | 247.09 | 115 |
| псевдоуридин | Y | 2 | 245.08 | 209/179/155 |
| 3-метилцитидин | м3С | 3 | 258.11 | 126 |
| N1-метиладенозин | м1А | 4 | 282.12 | 150 |
| 5-метилцитидин | м5С | 5 | 258.11 | 126 |
| N7-метилгуанозин | м7Г | 6 | 298.12 | 166 |
| 2'-O-метилцитидин | См | 7 | 258.11 | 112 |
| инозин | И6А | 8 | 269.09 | 137 |
| 2'-O-метилгуанозин | Гм | 9 | 298.12 | 152 |
| N2-метилгуанозин | м2Г | 10 | 298.12 | 166 |
| N2,N2,N7-триметилгуанозин | м227Г | 11 | 326.15 | 194 |
| N2,N2,-диметилгуанозин | м22Г | 12 | 312.13 | 180 |
| 2'-O-метиладенозин | Есмь | 13 | 282.12 | 136 |
| N6-метиладенозин | м6А | 14 | 282.12 | 150 |
| N6,N6-диметиладенозин | м66А | 15 | 296.14 | 164 |
| N6-изопентениладенозин | и6А | 16 | 336.17 | 204/136/148 |
Таблица 1: Модификации РНК, обнаруженные в одиночных нейронах из A. californica. Приведены атрибуты для характеристики модифицированных нуклеозидов, включая порядок удержания LC, m/z для генерации EIC и соответствующие фрагменты CID.
Обсуждение
SNRMA-MS использует оптимизированный подход к подготовке образцов, в результате чего получается небольшой, совместимый с MS объем образцов, который может быть доставлен на платформу LC-MS. Первоначальная ферментативная предварительная обработка ганглиев ЦНС диктует как легкость, с которой они могут быть удалены, так и долговечность отдельных нейронов во время изоляции. Церебральный ганглий часто требует длительного ферментативного лечения из-за его относительно толстой оболочки по сравнению с щечными, плевральными и брюшными ганглиями. Отдельные исследователи, выполняющие изоляцию отдельных нейронов, могут иметь разные предпочтения в отношении того, насколько прочной должна быть оболочка при использовании микросублиц и тонких щипцов. Однако важно, чтобы ганглии не переваривались, так как это может привести к потере их структурной целостности, потере позиционной информации, которая имеет решающее значение для идентификации клеток-мишеней, и / или лизису клеток. После выделения из ганглия важно обеспечить аспирацию минимального объема изоляционной среды при переносе нейрона в пробоотборную трубку. Противолодочная среда содержит высокую концентрацию солей, которые могут мешать пищеварительным ферментам во время гидролиза РНК, а также разбавляют образец.
Во время этапа механического лизиса крупные нейроны (диаметром >250 мкм) разрываются при многократном прохождении через микропипетку. Меньшие нейроны могут потребовать дополнительного внимания для обеспечения лизиса клеток, который обычно включает в себя надавливание стеклянного капилляра на клетку. В этом случае возможно, что частичный объем буфера образца будет втянут в капилляр из-за капиллярных сил. Этот объем может быть доставлен обратно в пробирку, применяя давление к концу стеклянного капилляра, чтобы гарантировать, что образец не будет потерян.
Наилучшие результаты получаются при включении стадии нагрева перед добавлением ферментов для переваривания РНК. Вероятно, это связано с тем, что нагревание при 95 °C денатурирует вторичные структуры РНК, которые могут препятствовать активностиRNases 35 и уменьшать количество нуклеозидов, высвобождаемых из биополимеров РНК. Контрольные эксперименты с использованием образцов, наполненных метионином, меченым стабильными изотопами, ранее проводились для изучения того, были ли артефакты модификации РНК, индуцированные теплом, причиной увеличения пиковых областей, наблюдаемых для модификаций РНК по сравнению с безтепловым протоколом SNRMA-MS29. Такой маркировки не наблюдалось, что указывает на то, что улучшенные сигналы для модификаций РНК с использованием оптимизированного метода SNRMA-MS были обусловлены превосходным перевариванием РНК.
Традиционные методы выделения общей РНК из клеток включают жидко-жидкостную экстракцию (LLE) фенол-хлороформом и последующее осаждение РНК, промывку и повторное суспендирование. Эти подходы оказались полезными для экспериментов с полимеразной цепной реакцией с обратной транскрипцией, где экспрессия избранных генов может быть легко контролироваться в идентифицированных нейронах A. californica 36,37. Однако методы LLE не могут восстановить достаточное количество РНК для обнаружения модифицированных рибонуклеозидов LC-MS, тогда как метод SNRMA-MS, описанный в настоящем описании, позволяет обнаружить многочисленные модификации РНК29. Для оценки профилей модификации конкретных типов РНК (например, рРНК, тРНК, мРНК) в объемных образцах тканей/клеток были применены подходы анионообменной твердой фазы24, обогащения38 на основе зонда гибридизации и хроматографического фракционирования39, но аналогичные методы еще не доступны для очистки одноклеточной РНК. Разработка подходов к фракционированию РНК, которые способны выделять определенные типы РНК из отдельных клеток, даст дополнительное представление о функции модификаций РНК.
SNRMA-MS выявила ранее неохарактерную гетерогенность в ландшафте модификации РНК отдельных нейронов в A. californica , и вполне возможно, что аналогичные различные профили PTM существуют в клетках млекопитающих. Поскольку клетки млекопитающих относительно малы по сравнению с большими нейронами A. californica , проанализированными в этом протоколе, необходимы улучшения в обработке небольших объемов образцов для облегчения более низких пределов обнаружения. Хотя в настоящее время SNRMA-MS ограничена объемами ~ 5 мкл, ожидается, что существенные улучшения могут быть достигнуты путем включения микрофлюидных устройств обработки жидкостей в рабочий процесс. Кроме того, внедрение автоматизированной или полуавтоматизированной изоляции клеток увеличит пропускную способность образца и позволит проводить анализ модификации одноклеточной РНК более крупных клеточных популяций. При сопряжении с нанопотоковыми LC-разделениями27 характеристика эпитранскриптомных меток в еще меньших клетках будет достижима.
Раскрытие информации
Авторы заявляют об отсутствии конкурирующих финансовых интересов.
Благодарности
Эта работа финансировалась Национальным институтом по борьбе со злоупотреблением наркотиками в рамках премии No. P30DA018310 и Национальный научно-исследовательский институт генома человека под номером РМ1ХГ010023. K.D.C. признает поддержку со стороны постдокторской стипендии Института Бекмана. Содержание является исключительной ответственностью авторов и не обязательно отражает официальную точку зрения финансирующих учреждений.
Материалы
| Name | Company | Catalog Number | Comments |
| Animals | |||
| Aplysia californica | National Resource for Aplysia (Miami, FL) | 150–250 g (adult) | |
| Benchtop equipment | |||
| Glass capillary puller | Sutter | P-97 | |
| Milli-Q water purification system | Millipore | ||
| Minicentrifuge for PCR tubes | LW Scientific | ZS-1 | |
| Optical Microscope | Zeiss | Stemi 2000C | |
| Thermocycler | Techne | EW-93945-01 | |
| HPLC column and consumables | |||
| Acclaim RSLC 120 C18 column | Thermo Scientific | 71399 | |
| Autosampler vials | Thermo Scientific | C4011-13 | |
| LC and MS Instrumentation and Software | |||
| DataAnalysis 4.4 software | Bruker | ||
| Dionex Ultimate 3000 nanoLC | Thermo Scientific | Equipped with online degasser, autosampler, and thermostatted column compartment | |
| Impact HD UHR QqTOF mass spectrometer | Bruker | Equipped with ESI source | |
| RStudio | RStudio | ||
| Microdissection tools | |||
| Microscissors extra fine vannas 3.5” | Roboz | RS-5640 | |
| Tungsten needles | Roboz | RS-6065 | |
| Reagents/Materials | |||
| 4-(2-hydroxyethyl)-1-piperazineethane-sulfonic acid (HEPES) | Fisher Scientific | H3375 | |
| Alkaline phosphatase | Worthington Biochemical Corp. | LS004081 | |
| Ammonium acetate | Honeywell | 17836 | |
| Benzonase (endonuclease from S. marcescens) | EMD Millipore | 70746-4 | |
| Bovine serum albumin | Sigma-Aldrich | A2153 | |
| Calcium chloride | Sigma-Aldrich | C4901 | |
| Gentamycin sulfate | Fisher Scientific | G1264 | |
| Magnesium chloride | Sigma-Aldrich | M9272 | |
| Magnesium sulfate | Sigma-Aldrich | 208094 | |
| Nucleosides test mix | Sigma-Aldrich | 47310-U | |
| Penicillin G | Sigma-Aldrich | P7794 | |
| Pentostatin | Sigma-Aldrich | SML0508 | |
| Phosphodiesterase I | Worthington Biochemical Corp. | LS003926 | |
| Protease type XIV from Streptomyces griseus | Sigma-Aldrich | P5147 | |
| Sodium chloride | Sigma-Aldrich | S9888 | |
| Standard glass capillaries | A-M Systems | 626000 | 1 mm o.d., 0.5 mm i.d., 4 in |
| Streptomycin sulfate | Sigma-Aldrich | S9137 |
Ссылки
- Cantara, W. A., et al. The RNA modification database, RNAMDB: 2011 update. Nucleic Acids Research. 39, 195-201 (2011).
- Boccaletto, P., et al. MODOMICS: a database of RNA modification pathways. 2017 update. Nucleic Acids Research. 46, 303-307 (2017).
- Kimura, S., Waldor, M. K. The RNA degradosome promotes tRNA quality control through clearance of hypomodified tRNA. Proceedings of the National Academy of Sciences of the United States of America. 116 (4), 1394-1403 (2019).
- Helm, M. Post-transcriptional nucleotide modification and alternative folding of RNA. Nucleic Acids Research. 34 (2), 721-733 (2006).
- Rezgui, V. A. N., et al. tRNA tKUUU, tQUUG, and tEUUC wobble position modifications fine-tune protein translation by promoting ribosome A-site binding. Proceedings of the National Academy of Sciences of the United States of America. 110 (30), 12289-12294 (2013).
- Shanmugam, R., et al. Cytosine methylation of tRNA-Asp by DNMT2 has a role in translation of proteins containing poly-Asp sequences. Cell Discovery. 1 (1), 1-10 (2015).
- Jia, G., et al. N 6-Methyladenosine in nuclear RNA is a major substrate of the obesity-associated FTO. Nature Chemical Biology. 7 (12), 885-887 (2011).
- Li, X., et al. Transcriptome-wide mapping reveals reversible and dynamic N 1 -methyladenosine methylome. Nature Chemical Biology. 12 (5), 311-316 (2016).
- Krogh, N., et al. Profiling of 2′-O-Me in human rRNA reveals a subset of fractionally modified positions and provides evidence for ribosome heterogeneity. Nucleic Acids Research. 44 (16), 7884-7895 (2016).
- Babaian, A., et al. Loss of m1acp3Ψ Ribosomal RNA Modification Is a Major Feature of Cancer. Cell Reports. 31 (5), 107611 (2020).
- Chang, M., et al. Region-specific RNA m6A methylation represents a new layer of control in the gene regulatory network in the mouse brain. Open Biology. 7 (9), 170166 (2017).
- Wang, C. -. X., et al. METTL3-mediated m6A modification is required for cerebellar development. PLOS Biology. 16 (6), 2004880 (2018).
- Engel, M., et al. The role of m6A/m-RNA methylation in stress response regulation. Neuron. 99 (2), 389-403 (2018).
- Widagdo, J., et al. Experience-dependent accumulation of N6-Methyladenosine in the prefrontal cortex is associated with memory processes in mice. Journal of Neuroscience. 36 (25), 6771-6777 (2016).
- Eberwine, J., et al. Analysis of gene expression in single live neurons. Proceedings of the National Academy of Sciences of the United States of America. 89 (7), 3010-3014 (1992).
- Cong, Y., et al. Ultrasensitive single-cell proteomics workflow identifies >1000 protein groups per mammalian cell. Chemical Science. 12 (3), 1001-1006 (2021).
- Rubakhin, S. S., Romanova, E. V., Nemes, P., Sweedler, J. V. Profiling metabolites and peptides in single cells. Nature Methods. 8 (4), 20-29 (2011).
- Nemes, P., Knolhoff, A. M., Rubakhin, S. S., Sweedler, J. V. Metabolic differentiation of neuronal phenotypes by single-cell capillary electrophoresis-electrospray ionization-mass spectrometry. Analytical Chemistry. 83 (17), 6810-6817 (2011).
- Lovatt, D., et al. Transcriptome in vivo analysis (TIVA) of spatially defined single cells in live tissue. Nature Methods. 11 (2), 190-196 (2014).
- Kærn, M., Elston, T. C., Blake, W. J., Collins, J. J. Stochasticity in gene expression: from theories to phenotypes. Nature Reviews Genetics. 6 (6), 451-464 (2005).
- Chan, C. T. Y., et al. A quantitative systems approach reveals dynamic control of tRNA modifications during cellular stress. PLOS Genetics. 6 (12), 1001247 (2010).
- Sun, C., Jora, M., Solivio, B., Limbach, P. A., Addepalli, B. The effects of ultraviolet radiation on nucleoside modifications in RNA. ACS Chemical Biology. 13 (3), 567-572 (2018).
- Heiss, M., Hagelskamp, F., Marchand, V., Motorin, Y., Kellner, S. Cell culture NAIL-MS allows insight into human tRNA and rRNA modification dynamics in vivo. Nature Communications. 12 (1), 389 (2021).
- Clark, K. D., Lee, C., Gillette, R., Sweedler, J. V. Characterization of neuronal RNA modifications during non-associative learning in aplysia reveals key roles for tRNAs in behavioral sensitization. ACS Central Science. 7 (7), 1183-1190 (2021).
- Basanta-Sanchez, M., Temple, S., Ansari, S. A., D'Amico, A., Agris, P. F. Attomole quantification and global profile of RNA modifications: Epitranscriptome of human neural stem cells. Nucleic Acids Research. 44 (3), 26 (2016).
- Huang, W., et al. Determination of DNA and RNA methylation in circulating tumor cells by mass spectrometry. Analytical Chemistry. 88 (2), 1378-1384 (2016).
- Sarin, L. P., et al. Nano LC-MS using capillary columns enables accurate quantification of modified ribonucleosides at low femtomol levels. RNA. 24 (10), 1403-1417 (2018).
- Clark, K. D., Philip, M. C., Tan, Y., Sweedler, J. V. Biphasic liquid microjunction extraction for profiling neuronal RNA modifications by liquid chromatography-tandem mass spectrometry. Analytical Chemistry. 92 (18), 12647-12655 (2020).
- Clark, K. D., Rubakhin, S. S., Sweedler, J. V. Single-neuron RNA modification analysis by mass spectrometry: Characterizing RNA modification patterns and dynamics with single-cell resolution. Analytical Chemistry. 93 (43), 14537-14544 (2021).
- Guise, O. L., Ahner, J. W., Jung, M. -. C., Goughnour, P. C., Yates, J. T. Reproducible electrochemical etching of tungsten probe tips. Nano Letters. 2 (3), 191-193 (2002).
- Peng, W., Wu, Z., Song, K., Zhang, S., Li, Y., Xu, M. Regulation of sleep homeostasis mediator adenosine by basal forebrain glutamatergic neurons. Science. 369 (6508), (2020).
- Rayport, S. G., Ambron, R. T., Babiarz, J. Identified cholinergic neurons R2 and LPl1 control mucus release in Aplysia. Journal of Neurophysiology. 49 (4), 864-876 (1983).
- Rosen, S. C., Weiss, K. R., Goldstein, R. S., Kupfermann, I. The role of a modulatory neuron in feeding and satiation in Aplysia: effects of lesioning of the serotonergic metacerebral cells. Journal of Neuroscience. 9 (5), 1562-1578 (1989).
- Lloyd, P. E., Kupfermann, I., Weiss, K. R. Central peptidergic neurons regulate gut motility in Aplysia. Journal of Neurophysiology. 59 (5), 1613-1626 (1988).
- Crain, P. F. Preparation and enzymatic hydrolysis of DNA and RNA for mass spectrometry. Methods in Enzymology. 193, 782-790 (1990).
- Kadakkuzha, B. M., et al. Age-associated bidirectional modulation of gene expression in single identified R15 neuron of Aplysia. BMC Genomics. 14 (1), 880 (2013).
- Akhmedov, K., Kadakkuzha, B. M., Puthanveettil, S. V. Aplysia ganglia preparation for electrophysiological and molecular analyses of single neurons. Journal of Visualized Experiments: JoVE. (83), e51075 (2014).
- Tardu, M., Jones, J. D., Kennedy, R. T., Lin, Q., Koutmou, K. S. Identification and quantification of modified nucleosides in saccharomyces cerevisiae mRNAs. ACS Chemical Biology. 14 (7), 1403-1409 (2019).
- Heiss, M., Reichle, V. F., Kellner, S. Observing the fate of tRNA and its modifications by nucleic acid isotope labeling mass spectrometry: NAIL-MS. RNA Biology. 14 (9), 1260-1268 (2017).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены