Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Evaluación de la cicatrización de fracturas óseas mediante microtomografía computarizada
* Estos autores han contribuido por igual
En este artículo
Resumen
La microtomografía computarizada (μCT) es una herramienta de imagen no destructiva que es fundamental para evaluar la estructura ósea en estudios preclínicos, sin embargo, existe una falta de consenso sobre los procedimientos de μCT para analizar el callo de cicatrización ósea. Este estudio proporciona un protocolo de μCT paso a paso que permite el seguimiento de la cicatrización de fracturas.
Resumen
La microtomografía computarizada (μCT) es la modalidad de imagen más común para caracterizar la morfología tridimensional (3D) del hueso y el hueso recién formado durante la cicatrización de fracturas en investigaciones científicas traslacionales. Los estudios de la cicatrización de fracturas de huesos largos en roedores suelen implicar la curación secundaria y la formación de un callo mineralizado. La forma del callo formado y la densidad del hueso recién formado pueden variar sustancialmente entre los puntos de tiempo y los tratamientos. Mientras que las metodologías estándar para cuantificar los parámetros del hueso cortical y trabecular intacto se utilizan ampliamente y están integradas en el software disponible comercialmente, existe una falta de consenso sobre los procedimientos para analizar el callo en proceso de curación. El propósito de este trabajo es describir un protocolo estandarizado que cuantifique la fracción de volumen óseo y la densidad mineral del callo en la cicatrización. El protocolo describe diferentes parámetros que deben tenerse en cuenta durante la obtención de imágenes y el análisis, incluida la alineación de la muestra durante la obtención de imágenes, el tamaño del volumen de interés y el número de cortes que se contornean para definir el callo.
Introducción
Las imágenes de microtomografía computarizada (μCT) se han utilizado ampliamente en la investigación ósea preclínica, proporcionando imágenes no invasivas de alta resolución para evaluar la microestructura de los huesos 1,2,3,4,5. La μCT implica un gran número de imágenes de rayos X, obtenidas a partir de una muestra giratoria o mediante el uso de una fuente de rayos X giratoria y un detector. Los algoritmos se utilizan para reconstruir datos volumétricos 3D en forma de una pila de cortes de imágenes. La TC clínica es el estándar de oro para la obtención de imágenes en 3D de huesos humanos, y la μCT es una técnica comúnmente utilizada para evaluar la eficiencia de la cicatrización ósea en animales de experimentación 1,2,3,4,6,7. El hueso mineralizado tiene un excelente contraste con los rayos X, mientras que los tejidos blandos tienen un contraste relativamente pobre a menos que se use un agente de contraste. En la evaluación de la cicatrización de fracturas, la μCT genera imágenes que proporcionan información detallada sobre la estructura 3D y la densidad del callo mineralizado. La exploración in vivo con μCT también se puede utilizar para la evaluación longitudinal y temporal de la cicatrización de fracturas.
La cuantificación del hueso cortical y trabecular intacto mediante μCT está generalmente bien establecida y estandarizada8. Aunque los estudios preclínicos utilizan una variedad de metodologías de cuantificación para analizar la cicatrización de fracturas 9,10,11, aún no se ha publicado un protocolo detallado de análisis de imágenes μCT para la cuantificación de callos. Por lo tanto, el objetivo de este estudio es proporcionar un protocolo detallado paso a paso para la obtención de imágenes por μCT y el análisis de la cicatriz ósea del callo.
Protocolo
Se desarrolló el siguiente protocolo para caracterizar el callo curativo de huesos largos extraído de ratones sacrificados. Sin embargo, la mayoría de los pasos se pueden aplicar a ratas y también se pueden utilizar para el escaneo in vivo de huesos fracturados. El protocolo describe un sistema μCT en particular y un software específico de procesamiento, análisis y visualización de imágenes (consulte la Tabla de materiales), sin embargo, la metodología es generalmente aplicable a otros escáneres y software. El protocolo fue aprobado por el Comité Institucional de Cuidado y Uso de Animales de la Facultad de Medicina de la Universidad Estatal de Pensilvania. Los ratones utilizados en este estudio fueron ratones machos C57BL/6J de 16 semanas de edad (peso promedio de 31,45 ± 3,2 g).
1. Recolección y conservación de tejidos
NOTA: Utilice un modelo de fractura murina adecuado. Para este estudio, se utilizó el modelo de fractura tibial abierta diafisaria media de acuerdo con el protocolo estándar descrito en12,13.
- Al final del experimento del modelo de fractura, eutanasia del ratón mediante la administración de una inyección intraperitoneal de ketamina o xilacina (500 mg/kg o 50 mg/kg, respectivamente).
- Con unas tijeras, extraiga el hueso fracturado desde la mitad del fémur hasta la articulación tibioastragalina sin alterar el sitio de la fractura. Extirpe los músculos que rodean el hueso, dejando solo el tejido blando que está en contacto directo con el hueso para soportar el sitio de la fractura durante los pasos de procesamiento posteriores. Retire el clavo intramedular con pinzas hemostáticas rectas para micromosquitos.
- Conservar las muestras en formol a 4 °C o en solución salina a -20 °C. La elección del vehículo de conservación depende de las aplicaciones previstas aguas abajo hasta μCT. En este estudio, las muestras se conservaron en solución salina a -20 °C.
2. Escaneo μCT
- Preparación de la muestra
- Para el escaneo simultáneo de múltiples muestras, coloque hasta seis muestras en un dispositivo de escaneo impreso en 3D desarrollado a medida (Figura 1 A, B) o similar. El escaneo simultáneo reduce el tiempo y el costo del escaneo. El accesorio personalizado utilizado en este estudio contiene seis ranuras para contener las muestras de huesos largos y un orificio central para un maniquí de hidroxiapatita (HA) (Figura 1A, B; Tabla de Materiales).
NOTA: El maniquí de HA servirá como estándar en el paso 4.2 (ver más abajo) para convertir las unidades μCT (típicamente Hounsfield) a densidad de HA (mgHA/ccm). - Coloque el dispositivo preparado en una jeringa o en un tubo cónico que sea similar al diámetro del campo de visión (FOV; Figura 1C). En este estudio, se utilizó una jeringa de 20 mm para igualar el campo de visión de 21,5 mm.
- Para evitar que las muestras se sequen durante el proceso de escaneo, llene la jeringa o el tubo canónico con el conservante utilizado en el paso 1.3 (en este estudio se utilizó solución salina).
- Para el escaneo simultáneo de múltiples muestras, coloque hasta seis muestras en un dispositivo de escaneo impreso en 3D desarrollado a medida (Figura 1 A, B) o similar. El escaneo simultáneo reduce el tiempo y el costo del escaneo. El accesorio personalizado utilizado en este estudio contiene seis ranuras para contener las muestras de huesos largos y un orificio central para un maniquí de hidroxiapatita (HA) (Figura 1A, B; Tabla de Materiales).
- Escaneo
- Antes de escanear, confirme que la máquina μCT esté calibrada de la siguiente manera: coloque un maniquí de alta disponibilidad en la línea central del campo de visión de μCT, escanee el maniquí y mida la densidad de ácido hialurónico. Asegúrese de que la densidad medida sea consistente con la densidad proporcionada por el fabricante.
- Alinee la línea central del dispositivo de muestra con la línea central aproximada del campo de visión μCT. Esto asegura que las muestras estén dentro del campo de visión y que sus ejes largos tengan una orientación que coincida aproximadamente con la dirección axial de las imágenes resultantes.
NOTA: Esta orientación estandarizada puede ayudar posteriormente a que el procedimiento de análisis sea menos propenso a variabilidades, como en la cantidad de tejido considerado dentro del volumen de interés. - Ajuste los parámetros de escaneo del sistema μCT (Tabla de materiales). Los parámetros utilizados en este estudio son 10,5 μm (tamaño de vóxel isotrópico), 55 kVp (energía/intensidad), 145 μA (corriente) y 300 ms (tiempo de integración). Determine el tamaño del vóxel en función del grosor aproximado de las trabéculas del ratón (20-60 μm)8. Inspeccione visualmente el escaneo en diferentes vistas para asegurarse de que cubre todo el volumen de todas las muestras de callos.

Figura 1: Estructura del dispositivo de escaneo personalizado. (A) Imágenes del dispositivo de escaneo (arriba), que muestran las seis ranuras de muestra, y el maniquí de alta disponibilidad (abajo). (B) Imágenes que muestren la muestra de hueso largo (arriba) y el maniquí de AH (abajo) colocados en las ranuras dedicadas. (C) Imágenes que muestren el dispositivo de escaneo colocado en una jeringa de 20 mm. Haga clic aquí para ver una versión más grande de esta figura.
3. Segmentación de imágenes
NOTA: Las imágenes RAW se reconstruyen automáticamente en datos de secuencia de imágenes.
- Conversión de imágenes: convierta los datos de la secuencia de imágenes reconstruida en secuencias de imágenes DICOM utilizando un software de procesamiento de imágenes (consulte la Tabla de materiales). Importe secuencias de imágenes DICOM en el software (consulte la Tabla de materiales) para el procesamiento, análisis y visualización de imágenes (Figura 2A).
- Recorte de imágenes: una muestra a la vez, recorte cada pila de imágenes y asegúrese de que toda la muestra esté incluida en el volumen recortado (Figura 2B). Guarde la imagen recortada de la siguiente manera: haga clic en la pestaña Archivo en la parte superior izquierda de la pantalla, seleccione Guardar proyecto y, a continuación, seleccione Minimizar tamaño del proyecto en las opciones que aparecen en la pantalla. El archivo se guardará en el formato de software comercial.
- Eliminación de ruido de la imagen: utilice un método de filtrado para reducir el nivel de ruido y evitar el desenfoque de la siguiente manera.
- Haga clic en la pestaña Archivo y elija la imagen que se procesará con Open Data. La imagen abierta aparecerá en la ventana de vista del proyecto en la esquina superior izquierda de la pantalla.
- Haga clic con el botón derecho para seleccionar Procesamiento de imágenes y, a continuación, Filtrar espacio aislado. Haga clic en Crear.
- Realice lo siguiente en la ventana Propiedades (en la esquina inferior izquierda de la pantalla): elija Datos como tipo de vista previa; seleccione el tipo de filtro en el menú desplegable junto a Filtro; elegir 3D para la interpretación; seleccione Separable en el menú desplegable junto a tipo de kernel; rellene los valores que se utilizarán para la desviación estándar y el factor de tamaño del kernel en el cuadro vacío disponible junto a cada uno; seleccione Igual que la entrada en el menú desplegable junto a la salida; haga clic en Aplicar.
NOTA: La elección del tipo de filtro (las opciones disponibles son bilateral, caja, gaussiana, mediana, exponencial recursiva, delineada, difusión anisotrópica, medias no locales, enmascaramiento de enfoque y filtro FFT) y los parámetros depende del nivel de ruido y del tamaño del vóxel de las imágenes escaneadas. Para el filtro gaussiano, 3 x 3 x 3 y 5 x 5 x 5 son valores comúnmente utilizados para el factor de tamaño del kernel, y 0.5-2.0 se usa comúnmente para la desviación estándar8. En este estudio, se aplicó un filtro gaussiano y se utilizaron 5 x 5 x 5 y 0.8 para el factor de tamaño de kernel y la desviación estándar, respectivamente.
- Realineación de imágenes
NOTA: Este es un paso opcional. Cuando se produce una desalineación de las muestras de huesos largos en relación con los ejes de coordenadas del sistema de imágenes durante el proceso de exploración, se puede aplicar un método de alineación digital para corregir la desalineación (Figura 2C).- Cree una imagen renderizada en 3D de la muestra de la siguiente manera. En la ventana de vista del proyecto, seleccione la imagen filtrada y recortada (creada en el paso 3.3). Haga clic con el botón derecho para seleccionar Pantalla y, a continuación, Procesamiento de volumen en el menú desplegable y, a continuación, haga clic en Crear. Compruebe visualmente la imagen renderizada en 3D en los planos sagital y frontal.
- Gire manualmente el volumen renderizado para obtener una buena alineación en el eje longitudinal. Aplique la transformación a las imágenes giradas de la siguiente manera: en la ventana de propiedades, haga clic en el Editor de transformación, luego vaya al editor-manipulador de transformación y seleccione Transformador en el menú desplegable. Ahora la muestra se puede rotar y realinear. Una vez finalizado el proceso de realineación, vuelva a hacer clic en el Editor de transformación para bloquear la imagen.
- Vuelva a muestrear la imagen filtrada (creada en el paso 3.3) para crear nuevos cortes de imagen de plano transversal (axial) de la siguiente manera: En la ventana Vista de proyecto, seleccione la imagen del paso 3.4.2. Haga clic con el botón derecho para seleccionar Transformación de geometría y, a continuación, Remuestrear imagen transformada en el menú desplegable y haga clic en Crear. En la ventana de propiedades, vaya a Datos y realice lo siguiente: para la interpolación, seleccione Estándar en el menú desplegable; en mode, elija Extended; en conservar, elija Tamaño de vóxel; En Padding Value, escriba cero en el cuadro en blanco disponible. A continuación, haga clic en Aplicar.
- Definición del volumen de interés (VOI)
- Revisa los cortes transversales de la imagen e identifica el plano central del callo de la fractura. Definir el VOI en función de los extremos proximal y distal del callo. En los casos en que los extremos del callo sean difíciles de definir, defina el VOI en función de una distancia estandarizada del plano central del callo (Figura 2D).
NOTA: Durante las fases de cicatrización que preceden a la remodelación ósea, es fácil definir los bordes del callo mineralizado porque la estructura trabecular del hueso tejido recién formado es distinta de la estructura cortical del hueso original. Sin embargo, cuando se produce la fase de remodelación, el hueso recién formado adquiere la estructura cortical gradualmente; Por lo tanto, definir los bordes del callo se vuelve cada vez más difícil.
- Revisa los cortes transversales de la imagen e identifica el plano central del callo de la fractura. Definir el VOI en función de los extremos proximal y distal del callo. En los casos en que los extremos del callo sean difíciles de definir, defina el VOI en función de una distancia estandarizada del plano central del callo (Figura 2D).

Figura 2: Segmentación de imágenes. (A) Una imagen que muestra seis muestras dentro de un escaneo. (B) Recorte de imágenes para aislar muestras individuales. (C) Alineación digital para corregir un eje longitudinal desalineado (línea punteada amarilla). (D) Definición del VOI y del plano central del callo. Haga clic aquí para ver una versión más grande de esta figura.
4. Análisis de imágenes
- Segmentación del callo y del hueso cortical
- Contornee el contorno exterior del callo de forma semiautomática utilizando la herramienta Lazo de segmentación con las opciones de trazado automático y trazado de bordes (Figura 3A) de la siguiente manera:
- Después de volver a ensamblar las imágenes transformadas (paso 3.4.3), haga clic en la pestaña Segmentación en la segunda fila de pestañas de la parte superior de la pantalla. En la ventana del editor de segmentación, seleccione una imagen transformada (creada en el paso 3.4.3) en el menú desplegable junto a la imagen.
- En la ventana MATERIALES, haga doble clic en Agregar; Al hacerlo, aparecerán dos pestañas llamadas Material3 y Material4. Haga clic con el botón derecho para cambiar el nombre del material3 a callo y el material4 a hueso cortical.
- En la ventana SELECCIÓN, haga clic en el icono del lazo; en las opciones que aparecen, seleccione A mano alzada para el modo 2D, Interior para el modo 3D y Trazado automático y Trazar bordes para las opciones. Utilice el lazo para marcar las regiones de interés.
- Repita este paso de contorneado con cortes muestreados a lo largo del VOI (Figura 3B). Las rebanadas contorneadas se pueden espaciar (por ejemplo, separadas por 20 rebanadas).
NOTA: En regiones con estructuras de callos complejas, el usuario puede considerar la posibilidad de reducir el espaciado entre las rebanadas contorneadas para capturar más fragmentos (Figura 3A, B). - Interpolar a través de los contornos de callos contorneados para crear una etiqueta de callo completa (Figura 3C,D) de la siguiente manera: en la ventana MATERIALES, elija el archivo Callo (creado en el paso 4.1.1.2.), haga clic en la pestaña Selección en la parte superior de la pantalla y seleccione Interpolar en el menú desplegable. En la ventana SELECCIÓN, haga clic en el signo más.
- Abra el archivo Cortical Bone creado en el paso 4.1.1.2. Segmente el hueso cortical, incluida la cavidad medular, como se describe para el callo en los pasos 4.1.1 y 4.1.2. (Figura 4A,B). Interpolar la corteza perióstica contorneada para crear una etiqueta ósea cortical como se describe para el callo en el paso 4.1.3 (Figura 4C, D).
- Calcule el volumen contorneado y el valor medio de gris del callo de la siguiente manera: haga clic en la pestaña Segmentación en la fila superior de la pantalla y seleccione Estadísticas de materiales en el menú desplegable. Esto generará una tabla que contiene todos los valores calculados. Los valores del hueso cortical y el callo (después de restar el hueso cortical) se proporcionan por separado. Una vez generada la tabla, haga clic en Exportar al espacio de trabajo para guardar los datos.
- Contornee el contorno exterior del callo de forma semiautomática utilizando la herramienta Lazo de segmentación con las opciones de trazado automático y trazado de bordes (Figura 3A) de la siguiente manera:
- Conversión de unidades de escala de grises a densidad mineral ósea
- Recorte la imagen 3D del maniquí de alta disponibilidad de 4,5 mm (Figura 2B) de toda la imagen y haga clic en Segmentación. La resina del maniquí de HA contiene cinco pequeños cilindros de HA (Figura 1A). Para el cilindro de alta disponibilidad que tiene la densidad más alta, defina la primera y la última rebanada mediante inspección visual.
- Dibuje dos círculos en la primera y la última división (evitando los bordes) con la herramienta Pincel (Figura 5A) de la siguiente manera: en la ventana MATERIALES, haga clic en Agregar cuatro veces. Haga clic con el botón derecho para cambiar el nombre de material3, material4, material5 y material6 a fantasma1, fantasma2, fantasma3 y fantasma4, respectivamente. Seleccione Phantom1, haga clic en el icono del pincel en la ventana SELECCIÓN y use el control deslizante para ajustar el tamaño del pincel (trazado circular) en función del tamaño del fantasma (el tamaño del círculo debe ser menor que el del fantasma).
- Aplique interpolación entre los dos círculos para crear un volumen para cada cilindro de alta disponibilidad (Figura 5B) de la siguiente manera: en la ventana MATERIALES, seleccione Phantom1, haga clic en la pestaña Selección en la fila superior de la pantalla y seleccione Interpolar en el menú desplegable. En la ventana SELECCIÓN, haga clic en el signo más.
- Repita el proceso de segmentación con tres de los cilindros de HA restantes, comenzando con la segunda densidad de HA más alta y terminando con la segunda densidad de HA más baja (Figura 5B). El cilindro con la densidad de HA más baja se puede excluir porque a menudo es difícil de segmentar.
- Utilice las etiquetas 3D generadas para calcular los valores medios de gris de los cuatro cilindros de alta disponibilidad analizados. Utilizando una hoja de cálculo (ver Tabla de Materiales) o similar, represente los valores medios de gris y los valores correspondientes de densidad mineral ósea (DMO) proporcionados por el fabricante del maniquí. Genere una ecuación de correlación entre la DMO y los valores de gris mediante regresión lineal.
- Segmentación del callo mineralizado y cálculo de la DMO
- Con base en la ecuación de correlación generada en el paso 4.2.5 y el umbral elegido que diferencia el callo mineralizado y no mineralizado, determine el umbral de valor de gris correspondiente. En consecuencia, etiquete el área del callo con valores de gris mayores que el umbral como callo mineralizado y etiquete el resto como no mineralizado (Figura 6A, B). En este estudio, se utilizaron 250 mgHA/ccm como umbral para el callo mineralizado14,15.
- Calcule los volúmenes totales de callos y callos mineralizados. A partir de estos valores, calcule la fracción de volumen óseo (volumen de callo mineralizado normalizado al volumen total de callo = BV/TV). Utilícese el valor medio de gris medido para el callo total para calcular la DMO del callo utilizando la ecuación de correlación generada en 4.2.5.
NOTA: Con base en el objetivo del estudio y el software utilizado para el análisis, se pueden calcular otros parámetros como el SMI (índice del modelo de estructura), el espesor trabecular y el grado de anisotropía.

Figura 3: Segmentación del límite exterior del callo. (A) Contorno del límite exterior del callo (línea roja). (B) Contornos en cortes muestreados a lo largo del VOI (cortes rojos). (C) Una etiqueta de callo 3D creada por interpolación (volumen rojo). (D) Una sección transversal de la etiqueta del callo que se muestra en C (incluido el hueso cortical). Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Segmentación del hueso cortical. (A) Contorno de la superficie perióstica de la corteza (línea verde). (B) Contornos en cortes muestreados a lo largo del VOI (cortes verdes). (C) Una etiqueta 3D del hueso cortical (que contiene la cavidad medular; verde) y el callo (rojo) creado a partir de etiquetas interpoladas de la corteza perióstica y el callo. (D) Una sección transversal del callo (rojo) y el hueso cortical (que contiene la cavidad intramedular; verde). Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Conversión de unidades de escala de grises a DMO. (A) Contornos del cilindro de HA en la primera y la última rebanada (círculos rojos). (B) Cilindros de HA interpolados en 3D (izquierda) y secciones transversales (derecha). Marrón: mayor densidad de AH; azul: segunda densidad más alta de AH; violeta: tercera densidad más alta de AH; verde: cuarta densidad más alta de HA. Haga clic aquí para ver una versión más grande de esta figura.
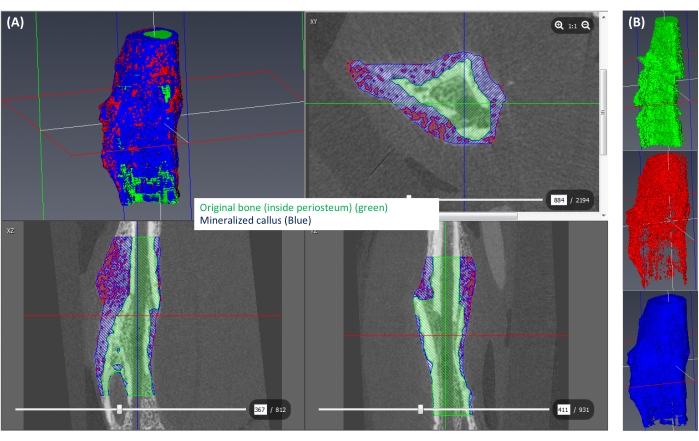
Figura 6: Segmentación del callo mineralizado. (A) El callo mineralizado (≥250 mgHA/ccm) se muestra en azul, el resto del callo (<250 mgHA/ccm) se muestra en rojo y el espacio correspondiente al hueso original se muestra en verde. (B) Una vista 3D de cada etiqueta aislada. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Para monitorizar la formación ósea durante la cicatrización de la fractura, se indujo una fractura tibial abierta a mitad diafisaria en ratones machos adultos C75BL/6J. La fractura se estabilizó con un clavo intramedular, modelo establecido de cicatrización secundaria13. Los tejidos callosos se recolectaron a los días 14, 21 y 28 después de la fractura12. Estos puntos de tiempo representan diferentes fases de curación. La formación de hueso endocondral durante la c...
Discusión
El propósito de este estudio es describir un protocolo detallado para el análisis de μCT con el objetivo de cuantificar con precisión la estructura del callo mineralizado en 3D, que a menudo es fundamental en los estudios de cicatrización de huesos y fracturas. El protocolo utiliza una plataforma de software de análisis de imágenes 3D de última generación de uso general que facilita la visualización de imágenes, la segmentación / etiquetado y las mediciones que van desde simples hasta complejas.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Este trabajo fue apoyado por los Institutos Nacionales de Salud (NIH, por sus siglas en inglés) R01 DK121327 a R.A.E y R01 AR071968 a F.K.
Materiales
| Name | Company | Catalog Number | Comments |
| 10% neutral buffered formalin | Fisher chemical | SF100-20 | Used for bone tissue fixation |
| Avizo | Thermo Scientific | Image processing and analysis software | |
| Hydroxyapatite phantom | Micro-CT HA D4.5, QRM | QRM-70128 | |
| Image Processing Language | Scanco | Used to convert raw images to DICOM images | |
| Micro-Mosquito Straight Hemostatic Forceps | Medline | Used to remove the intramedullary pin | |
| Microsoft Excel | Microsoft | Spreadsheet software | |
| Scanco mCT system (vivaCT 40) | Scanco | Used for µCT imaging |
Referencias
- Rüegsegger, P., Koller, B., Müller, R. A microtomographic system for the nondestructive evaluation of bone architecture. Calcified Tissue International. 58 (1), 24-29 (1996).
- Müller, R., et al. Morphometric analysis of human bone biopsies: a quantitative structural comparison of histological sections and micro-computed tomography. Bone. 23 (1), 59-66 (1998).
- Waarsing, J. H., et al. Detecting and tracking local changes in the tibiae of individual rats: a novel method to analyse longitudinal in vivo micro-CT data. Bone. 34 (1), 163-169 (2004).
- Boyd, S. K., Davison, P., Müller, R., Gasser, J. A. Monitoring individual morphological changes over time in ovariectomized rats by in vivo micro-computed tomography. Bone. 39 (4), 854-862 (2006).
- Christiansen, B. A. Effect of micro-computed tomography voxel size and segmentation method on trabecular bone microstructure measures in mice. Bone Reports. 5, 136-140 (2016).
- Holdsworth, D. W., Thornton, M. M. Micro-CT in small animal and specimen imaging. Trends in Biotechnology. 20 (8), 34-39 (2002).
- Schambach, S. J., Bag, S., Schilling, L., Groden, C., Brockmann, M. A. Application of micro-CT in small animal imaging. Methods. 50 (1), 2-13 (2010).
- Bouxsein, M. L., et al. Guidelines for assessment of bone microstructure in rodents using micro-computed tomography. Journal of Bone and Mineral Research. 25 (7), 1468-1486 (2010).
- Morgan, E. F., et al. Micro-computed tomography assessment of fracture healing: Relationships among callus structure, composition, and mechanical function. Bone. 44 (2), 335-344 (2009).
- O'Neill, K. R., et al. Micro-computed tomography assessment of the progression of fracture healing in mice. Bone. 50 (6), 1357-1367 (2012).
- Bissinger, O., et al. Fully automated segmentation of callus by micro-CT compared to biomechanics. Journal of Orthopaedic Surgery and Research. 12 (1), 108 (2017).
- Brown, M. L., et al. Delayed fracture healing and increased callus adiposity in a C57BL/6J murine model of obesity-associated type 2 diabetes mellitus. PLOS One. 9 (6), 99656 (2014).
- Khajuria, D. K., et al. Aberrant structure of fibrillar collagen and elevated levels of advanced glycation end products typify delayed fracture healing in the diet-induced obesity mouse model. Bone. 137, 115436 (2020).
- Sigurdsen, U., Reikeras, O., Hoiseth, A., Utvag, S. E. Correlations between strength and quantitative computed tomography measurement of callus mineralization in experimental tibial fractures. Clinical Biomechanics. 26 (1), 95-100 (2011).
- Duvall, C. L., Taylor, W. R., Weiss, D., Wojtowicz, A. M., Guldberg, R. E. Impaired angiogenesis, early callus formation, and late stage remodeling in fracture healing of osteopontin-deficient mice. Journal of Bone and Mineral Research. 22 (2), 286-297 (2007).
- Gerstenfeld, L. C., et al. Comparison of effects of the bisphosphonate alendronate versus the RANKL inhibitor denosumab on murine fracture healing. Journal of Bone and Mineral Research. 24 (2), 196-208 (2009).
- Alentado, V. J., et al. Validation of the modified radiographic union score for tibia fractures (mRUST) in murine femoral fractures. Frontiers in Endocrinology. 13, 911058 (2022).
- Yu, K. E., et al. Enhancement of impaired MRSA-infected fracture healing by combinatorial antibiotics and modulation of sustained inflammation. Journal of Bone and Mineral Research. 37 (1), 1352-1365 (2022).
- Nyman, J. S., et al. Quantitative measures of femoral fracture repair in rats derived by micro-computed tomography. Journal of Biomechanics. 42 (7), 891-897 (2009).
- Fiset, S., et al. Experimental validation of the radiographic union score for tibial fractures (RUST) using micro-computed tomography scanning and biomechanical testing in an in-vivo rat model. The Journal of Bone and Joint Surgery. 100 (21), 1871-1878 (2018).
- Shefelbine, S. J., et al. Prediction of fracture callus mechanical properties using micro-CT images and voxel-based finite element analysis. Bone. 36 (3), 480-488 (2005).
- Liu, Y., et al. Glucocorticoid-induced delayed fracture healing and impaired bone biomechanical properties in mice. Clinical Interventions in Aging. 13, 1465-1474 (2018).
- Watson, P. J., Fitton, L. C., Meloro, C., Fagan, M. J., Gröning, F. Mechanical adaptation of trabecular bone morphology in the mammalian mandible. Scientific Reports. 8 (1), 7277 (2018).
- Nie, C., Wang, Z., Liu, X. The effect of depression on fracture healing and osteoblast differentiation in rats. Neuropsychiatric Disease and Treatment. 14, 1705-1713 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados