Method Article
Focalización génica altamente eficiente mediada por CRISPR/Cas9 en células madre embrionarias para desarrollar modelos de ratón manipulados genéticamente
En este artículo
Resumen
Aquí presentamos un protocolo para desarrollar modelos de ratón modificados genéticamente utilizando células madre embrionarias, especialmente para ADN grande knock-in (KI). Este protocolo se ajusta utilizando la edición del genoma CRISPR / Cas9, lo que resulta en una eficiencia de KI significativamente mejorada en comparación con el método convencional de orientación de ADN linealizado mediado por recombinación homóloga.
Resumen
El sistema CRISPR/Cas9 ha permitido desarrollar ratones modificados genéticamente mediante edición directa del genoma utilizando cigotos fertilizados. Sin embargo, aunque la eficiencia en el desarrollo de ratones knockout de genes mediante la inducción de una pequeña mutación indel sería suficiente, la eficiencia de la edición del genoma embrionario para hacer knock-in de ADN de gran tamaño (KI) sigue siendo baja. Por lo tanto, en contraste con el método KI directo en embriones, la orientación génica utilizando células madre embrionarias (ESC) seguida de inyección de embriones para desarrollar ratones quimera todavía tiene varias ventajas (por ejemplo, la orientación de alto rendimiento in vitro, la manipulación multialelo y la manipulación de genes Cre y flox se puede llevar a cabo en un período corto). Además, las cepas con embriones in vitro difíciles de manejar, como BALB/c, también se pueden utilizar para la orientación de ESC. Este protocolo describe el método optimizado para KI de ADN de gran tamaño (varios kb) en ESCs mediante la aplicación de la edición del genoma mediada por CRISPR / Cas9 seguida de la producción de ratones quimeras para desarrollar modelos de ratón manipulados genéticamente.
Introducción
La producción de ratones modificados genéticamente y el análisis de su fenotipo nos permite comprender las funciones genéticas específicas en detalle, in vivo. Se han descubierto numerosos hallazgos importantes utilizando modelos animales modificados genéticamente en el campo de las ciencias de la vida. Además, desde el informe de la tecnología de edición del genoma utilizando CRISPR/Cas91, la investigación con ratones modificados genéticamente se ha extendido rápidamente a muchos laboratorios 2,3. La edición del genoma de cigotos de ratón por CRISPR/Cas9 ha logrado una eficiencia aceptable para desarrollar modificaciones cortas del ADN, como la eliminación 4 del gen orientado a la mutación indel, el reemplazo de nucleótido único o la inserción de etiquetas peptídicas cortas utilizando oligonucleótidos monocatenarios (ssODN) como donantes knock-in (KI)5. Por otro lado, el KI de grandes fragmentos de ADN en cigotos por edición del genoma permanece en una baja eficiencia en comparación con la modificación del ADN de tamaño corto 6,7. Además, es difícil utilizar cepas de ratón como BALB/c, que es una cepa importante para áreas de investigación específicas como la inmunología, para la edición del genoma basada en cigotos porque sus embriones preimplantacionales son susceptibles a la manipulación in vitro.
Otra forma de desarrollar modelos de ratón modificados genéticamente es utilizar la técnica de orientación de células madre embrionarias (ESC) seguida de la inyección de ESC en el embrión preimplantacional para producir quimeras 8,9,10, que todavía se usa rutinariamente como método convencional. Aunque la tasa de adquisición para obtener clones precisos de KI-ESC no es muy alta en los métodos convencionales de focalización de ESC, la focalización de ESC ofrece algunas ventajas en comparación con la edición del genoma del cigoto, especialmente para KI de ADN largo. Por ejemplo, la eficiencia KI de fragmentos largos de ADN (> varios kb) en el genoma del cigoto es menos evidente6,7, y se necesitan muchos cigotos para desarrollar incluso una línea de ratón KI, lo cual es indeseable en la perspectiva actual de los experimentos con animales. En contraste con la edición del genoma del cigoto, el ADN largo dirigido a las ESC seguido de la producción de quimeras necesita significativamente menos embriones que la edición del genoma del cigoto. Además, aunque los embriones preimplantacionales de BALB/c son susceptibles de manipulación in vitro, sus ESCs pueden mantenerse y manipularse in vitro 11 como otros ESC competentes 129 o F1 de fondo, por lo tanto, aplicables a las producciones de quimeras. Sin embargo, aunque un vector objetivo contiene 5' y 3' brazos homólogos y cassettes de genes de resistencia a fármacos para la selección positiva o negativa, la eficiencia convencional de KI de las ESC es generalmente insuficiente, debido a la alta frecuencia de integración genómica aleatoria 8,10, por lo tanto, se requiere un método mejorado con una eficiencia precisa de focalización de ESC. Recientemente, informamos sobre un método ESC KI ajustado utilizando la edición del genoma basada en CRISPR / Cas9 para lograr una mayor eficiencia de KI que los métodos convencionales de focalización11. El método que describimos aquí se basa en este procedimiento que permite KI de ADN largo (> varios a 10 kb) a ESC con una eficiencia aceptable para trabajos rutinarios sin selección de fármacos; Por lo tanto, el procedimiento de construcción del vector sería mucho más fácil y necesitaría un período más corto, o el período de cultivo celular también sería significativamente más corto.
Protocolo
Todos los experimentos con ratones fueron aprobados por el Comité Institucional de Cuidado y Uso de Animales de la Universidad de Tokio (número de aprobación PA17-63) y la Universidad de Osaka (número de aprobación Biken-AP-H30-01) y se realizaron de acuerdo con sus directrices, así como las directrices ARRIVE (https://arriveguidelines.org).
1. Construcción de vectores de focalización
- Amplificar un casete KI (CreERT aquí, el ADN de plantilla para la PCR es originalmente de una compañía comercial de síntesis de genes) y un fragmento aproximado de 1 kb de brazos de homología de 5' o 3' por PCR. Para la clonación de ADN (paso 1.3), agregue secuencias de superposición de 15 mer en el extremo 5' de cada cebador de PCR. Purificar los fragmentos de ADN de PCR mediante electroforesis en gel de agarosa, seguida de la extracción del fragmento de ADN utilizando un kit de purificación de ADN.
- Linealizar el plásmido de la columna vertebral (por ejemplo, pUC19 o pBS) utilizando una enzima de restricción apropiada que digiera de forma única en algún lugar del sitio de clonación múltiple para la clonación. Luego, purifica el plásmido digerido usando un kit de purificación de ADN.
- Clone cada fragmento de PCR (por ejemplo, brazo de homología de 5', brazo de homología de 3') y secuencia de KI simultáneamente en el plásmido digerido utilizando un kit de clonación de ADN de acuerdo con las instrucciones del fabricante.
- Transformar las células competentes utilizando el plásmido construido (paso 1.3) calentando a 42 °C durante 1 minuto en un baño maría, luego sembrarlas en una placa LB que contenga la concentración adecuada de antibióticos como ampicilina o kanamicina. Cultivarlos durante la noche para selecciones de clones resistentes.
- Recoger varias colonias individuales (de cuatro a ocho) utilizando puntas de pipeta de 200 μL y transferir las puntas a 3 ml de LB líquido que contenga antibióticos apropiados (100 μg/ml de ampicilina o 20 μL/ml de kanamicina) y cultivar durante la noche a 37 °C con agitación.
- Purificar el plásmido al día siguiente utilizando un kit de purificación de plásmidos comerciales de grado libre de endotoxinas de acuerdo con las instrucciones del fabricante. Confirmar la secuencia clonada en el plásmido mediante secuenciación de Sanger. Ajustar la concentración de plásmidos a 1 μg de ADN/μL utilizando agua libre de nucleasas. Utilice el plásmido como vector dirigido al gen.
2. Preparación del fibroblasto embrionario de ratón (MEF) como células alimentadoras para ESC
- Sacrificar ratones hembra ICR embarazadas de 8-10 semanas de edad (14,5 días después del coito) por luxación cervical. Limpie bien el abdomen usando etanol al 70% (v / v) para la esterilización, corte el abdomen con tijeras orbitales y fórceps, y recupere el útero que contiene el feto.
- Coloque el útero en una placa de 100 mm que contenga 10 ml de suero bovino de fosfato (PBS) que contenga 100 U/ml de penicilina y 100 μg/ml de estreptomicina. Extraer las placentas y los tejidos extraembrionarios de los fetos usando tijeras orbitarias y fórceps. Picar los fetos con tijeras orbitarias y transferir la solución de PBS que contiene las células fetales picadas a un tubo cónico de 50 ml.
- Centrifugar a 280 x g durante 5 min a 4 °C y desechar el sobrenadante por aspiración. Añadir 10 ml de solución de tripsina-EDTA al 0,25% (p/v), mezclar bien con pipeteo y, a continuación, incubar durante 10 min a 37 °C al baño maría para la digestión de tripsina.
- Añadir 20 ml de medio MEF (DMEM que contiene 10% (v/v) de suero fetal bovino, 100 U/ml de penicilina y 100 μg/ml de estreptomicina) para detener la reacción enzimática, mezclar bien mediante una inversión suave y centrifugar a 280 x g durante 5 min a 4 °C. Deseche el sobrenadante por aspiración, agregue 10 ml de medio MEF y mezcle bien pipeteando.
- Siembre la suspensión celular en varios platos nuevos de 100 mm (prepare el mismo número de platos que el número de fetos con 10 ml de medio MEF para un plato de 100 mm). Cambie el medio de 4 a 5 h después de la siembra para eliminar las células no unidas y dejar que crezca la MEF adjunta. Pasar las células cuando alcanzan ~ 80% de confluencia usando tripsina.
- Coloque las placas de cultivo que contienen la proliferación de MEF en medio MEF, aproximadamente 12-15 días después de la siembra, en un dispositivo irradiador de rayos X. Exponga el MEF con una radiografía de 50 Gy (total) para detener el ciclo celular de acuerdo con las instrucciones del fabricante.
- Tripsinizar la MEF irradiada añadiendo 2 ml de tripsina-EDTA al 0,25% durante una placa de 100 mm durante 5 min a 37 °C, luego añadir 4 ml de medio MEF para detener la digestión de tripsina y recoger la suspensión celular en un nuevo tubo de 50 ml.
- Lavar la MEF con un medio MEF fresco por centrifugación a 280 x g durante 5 min a 4 °C, contar el número de células utilizando un contador de células con tinción de azul de tripano y congelarlas a 1,6 x 106 células/tubo en un medio de congelación celular. Almacenar hasta su uso; Las células se pueden almacenar de manera estable en nitrógeno líquido durante varios años.
3. Preparación de la mezcla Cas9-RNP-DNA
- Disolver el tracrRNA y el crisprRNA en tampón de dilución (cada concentración de ARN a 200 μM) mediante pipeteo. Mezclar la solución de tracrRNA, la solución de crisprRNA y el tampón de dilución (relación de volumen a 2:2:5) golpeando suavemente y recociendo cada ARN utilizando un termociclador a 95 °C durante 10 min seguido de ciclos reductores de 1 °C/min hasta que la temperatura alcance los 4 °C.
NOTA: La secuencia de ARNg se diseñó utilizando CRISPOR, http://crispor.tefor.net. - Incubar los ARN recocidos, 10 μg/μL de nucleasa Cas9, y el tampón de electroporación mezclado en una relación de volumen de 5:3:2 a 37 °C durante 20 min para formar el complejo Cas9-RNP. En el trabajo rutinario, mezcle e incube 1,25 μL de ARN recocido, 0,75 μL de nucleasa Cas9 y 0,5 μL del tampón de electroporación para la edición del genoma de un locus.
- Mezclar los siguientes materiales en un tubo de 1,5 ml: 10 μL de tampón de electroporación, 1 μL de complejo Cas9-RNP (paso 3.2) y 1 μL de vector objetivo circular (1 μg/μL, paso 1.6). La cantidad total de mezcla Cas9-RNP-DNA es de 12 μL. Mantenga la mezcla Cas9-RNP-DNA en hielo hasta la electroporación.
NOTA: Para evitar la re-escisión del ARNg después del KI, diseñe el ARNg en una posición donde la secuencia de reconocimiento de ARNg se divida por la integración del donante de KI. Si el ARNg no puede diseñarse en tal ubicación, varias mutaciones silenciosas de nucleótidos, por las cuales la secuencia de aminoácidos no cambia, se incluyen en el plásmido del donante de KI.
4. Focalización génica de las ESCs
- Para preparar las placas de cultivo celular recubiertas de gelatina, agregue una solución de gelatina al 0,1% (p/v) a 2 ml para un plato de 60 mm, 500 μL para una placa de 24 pocillos, 100 μL para una placa de 96 pocillos e incubar durante 2 h en una incubadora húmeda. Retire la solución de gelatina, lávela dos veces con la misma cantidad de PBS y guárdela a temperatura ambiente hasta que la use.
- Descongele la cepa de MEF congelada mitóticamente inactivada (paso 2.2) utilizando un baño maría a 37 °C durante 1 min, y coloque la MEF en un plato de 60 mm recubierto de gelatina (un tubo de MEF congelado que contenga 1,6 x 106 células para dos placas de 60 mm, paso 2.2) 1 día antes de la siembra ESC.
- Descongele un tubo de ESC congelado (almacenado a 2 x 10 5 ESCs/tubo; el recuento de celdas se realizó utilizando un contador de celdas) utilizando un baño maría a 37 °C durante 1 min, y coloque 1 x 105 ESCs en un plato de 60 mm que contenga MEF presembrado (paso 4.1).
- Cultivo en medio de cultivo ESC de 4 ml (ESCM, medio modificado basado en DMEM con suero fetal bovino al 15% (v/v), sustrato de L-glutamina de 2 mM, penicilina 100 U/mL, estreptomicina 100 μg/mL, 2-mercaptoetanol 0,1 mM, factor inhibidor de leucemia y t2i (0,2 μM PD0325901 y CHIR99021 de 3 μM) que mantiene la pluripotencia de ESC11) a 37 °C con 5% deCO2 hasta que el ESC alcanza una confluencia del 50% al 70%.
NOTA: Utilizamos tres líneas diferentes de ESC: JM8. A3 de B6N, V6.5 de B6-129 F1 y BALB/c ESC desarrollado internamente. Los mismos protocolos KI en este manuscrito se utilizan para todas las líneas ESC. - Lavar el ESC de cultivo con 4 ml de PBS, luego tratar con 800 μL de solución de tripsina-EDTA al 0,25% durante 5 min a 37 °C para la digestión. Añadir 1 ml de ESCM para inactivar la tripsina y disociar las ESCs a células individuales mediante pipeteo.
- Centrifugar la solución que contiene ESC durante 5 min a 280 x g, desechar el sobrenadante, resuspender las ESC en 1 ml fresco de ESCM y contar el número de células utilizando un contador de células con tinción de azul de tripano. Transfiera 1 x 10 5 ESCs a un nuevo tubo de1,5 ml y lávelos tres veces con PBS por centrifugación a 500 x g, 4 °C.
- Resuspender el pellet de ESC en 12 μL de mezcla Cas9-RNP-DNA (paso 3.3) y mezclar bien con un pipeteo suave para evitar el burbujeo. Servir el ESC resuspendido para electroporación. El sistema de electroporación utiliza un solo pulso a 1.400 V y 30 ms para la transducción Cas9-RNP-DNA (Figura 1).
- Cultivar las ESC electroporadas en una placa de 60 mm que contenga 4 mL de ESCM con MEF mitóticamente inactivada (sección 4.2). Cambia el medio todos los días.
- Lavar las ESC con 4 mL de PBS de 3 a 5 días después de la electroporación, luego tratar con 800 μL de tripsina-EDTA al 0,25% y cultivar a 37 °C durante 5 min en una incubadora húmeda para la digestión. Añadir 2 ml de ESCM para detener la digestión de tripsina y centrifugar la mezcla celular a 280 x g durante 5 min.
- Deseche el sobrenadante, resuspenda las ESC en 1 ml de ESCM fresco y cuente la concentración celular utilizando un contador celular con tinción de azul de tripano. Paso de los CES a 1 x 103 ESC por plato de 60 mm que contenga ESCM y MEF de alimentación. Cambie el medio todos los días hasta que la colonia se recupere.
NOTA: La razón por la que el ESC se pasa una vez antes de la recogida fue que la edición del genoma puede continuar ocurriendo después de la división ESC, por lo que la primera colonia no siempre es el clon en muchos casos. Por lo tanto, el primer paso de ESC antes de la recogida de la colonia es un paso esencial para recoger clones individuales.
5. Genotipado por PCR de CES dirigidas
- Aspirar ESCM de la placa de cultivo ESC de 5 a 7 días después del primer paso (paso 4.9) y agregar 4 mL de PBS. Recoger colonias ESC individuales con 5 μL de PBS utilizando una pipeta de 20 μL bajo un microscopio estereoscópico (aumento de 30x a 40x). Coloque las colonias individuales en un pocillo de una placa de fondo redondo de 96 pocillos que contenga 15 μL de solución de tripsina-EDTA al 0,25%.
- Mantenga la placa de 96 pocillos en hielo hasta que se recojan 48 colonias individuales. Incubar la placa de 96 pocillos durante 5 minutos en una incubadora húmeda al 5% deCO2 a 37 °C, luego añadir 80 μL de ESCM para detener la digestión de tripsina y disociar las colonias de ESC en células individuales mediante pipeteo.
NOTA: Dado que la eficiencia de KI de este método suele ser del 10% al 50%, recogemos rutinariamente 48 clones y primero realizamos el genotipado utilizando 24 de ellos. Si no se pueden obtener múltiples clones de KI en este punto, evaluamos KI utilizando los 24 clones restantes. - Transfiera 40 μL de suspensión de ESC (paso 5) a una placa de 96 pocillos recubierta de gelatina que contenga 50 μL de ESCM por pocillo para genotipado por PCR. Transfiera los 60 μL restantes de suspensión de ESC a un pocillo en una placa de 24 pocillos recubierta de gelatina, que contiene 500 μL de ESCM y MEF de alimentación, para hacer el stock de ESC congelado.
- Stock clones individuales de ESC cultivados en la placa de 24 pocillos (paso 5.3) hasta que alcancen una confluencia del 60% al 80%. Recuperar clones individuales de ESC mediante tripsinización como se mencionó anteriormente, recolectar células en un nuevo tubo de 1,5 ml y centrifugar a 280 x g durante 5 min. Desechar el sobrenadante, resuspender las células en 500 μL de medio de congelación celular y congelarlas con un congelador profundo a -80 °C.
- Para el genotipado por PCR, cultive clones de ESC en la placa libre de alimentador de 96 pocillos (paso 5.3) hasta que el ESC alcance más del 90% de confluencia. Retirar la ESCM de cada pocillo por aspiración y lavar dos veces con 100 μL de PBS.
- Aspirar el PBS y añadir 100 μL de tampón de lisis que contenga proteinasa K, mezclar bien y transferir el lisado ESC a un nuevo tubo de 1,5 ml. Calentar el lisado ESC a 65 °C durante al menos 1 h utilizando una cámara de calor. Extraer y purificar el ADN genómico utilizando un método convencional de purificación de ADN fenol-cloroformo seguido de precipitación de etanol.
- Disuelva el ADN precipitado en 20 μL de agua libre de DNasa y determine la pureza y concentración de ADN utilizando un espectrofotómetro. Realice PCR genómica utilizando conjuntos de cebadores específicos del locus para amplificar la región objetivo. Luego, verifique la secuencia y elija los clones ESC con las secuencias KI deseadas.
6. Preparación del embrión en estadio de ocho células y microinyección de ESCs
- Inyecte 5 UI de gonadotropina sérica de yegua gestante (PMSG) por vía intraperitoneal en hembras adultas de ICR. Después de 48 h, inyecte 5 UI de gonadotropina coriónica humana (hCG) en las mismas hembras, luego aparee a las hembras tratadas con hormonas con los machos ICR. Revise el tapón copulatorio en la vagina al día siguiente (día embrionario 0.5, ED0.5).
NOTA: Para evaluar el quimerismo por las proporciones de color del pelaje, use blastocisto de la cepa ICR como receptor para un ESC que tenga un fondo de color de pelaje negro o agutí, o blastocisto de la cepa C57BL / 6 como receptor para un ESC que tenga el fondo albino. - Recoja el oviducto en ED1.5 y colóquelo en gotas de medio tamponado HEPES (FHM). Recupere los embriones en estadio de dos o cuatro células lavando el oviducto con FHM utilizando una aguja de lavado insertada en el infundíbulo.
- Lave los embriones recolectados moviéndolos en varias gotas frescas de FHM con una pipeta bucal. Cultivo de los embriones en 50 μL de gota de KSOM en una placa de cultivo celular de 35 mm cubierta con aceite mineral a 37 °C, incubadora de CO2 al 5% durante 1 día hasta que se alcance la etapa de ocho células o mórula (ED2.5 ).
- Si los embriones no se utilizan el siguiente día de recolección, congelarlos por vitrificación de acuerdo con el protocolo CARD (http://card.medic.kumamoto-u.ac.jp/card/english/sigen/manual/ebvitri.html), y almacenarlos en nitrógeno líquido hasta su uso.
- Preparar pipetas de vidrio para sujetar los embriones (80-100 μm de diámetro exterior) y la inyección de ESC (unos 13 μm de diámetro) utilizando un extractor y microforja.
- Integre una pipeta de sujeción que contenga FHM en un soporte capilar conectado a un microinyector y colocado en el lado izquierdo del micromanipulador. Conecte una pipeta de inyección a otro microinyector y configúrela en el lado derecho. Alinee las dos pipetas rectas en el campo de visión del microscopio.
- Dispense 5 μL de gotas de polivinilpirrolidona (PVP) al 12% (p/v) en medio FHM y gotas de 5 μL de FHM en la misma tapa de un plato de 60 mm una al lado de la otra y cubra las gotas con aceite mineral. Transfiera los embriones en la gota FHM de 5 μL y agregue 1-5 μL de la suspensión ESC a la gota FHM que contiene embriones.
NOTA: Deje la suspensión ESC y MEF durante 30 minutos en la ESM de 4 ml en una placa de cultivo de 60 mm antes de la preparación de la gota. Dado que los MEF se adhieren al fondo más rápido que los ESC, esta incubación de 30 minutos permite la concentración de células madre embrionarias no unidas en el sobrenadante. - Lavar dentro de la pipeta de inyección con PVP, luego pasar a la gota que contiene los embriones y las ESC.
- Recoja tres dobletes ECS individuales que acaban de terminar su división celular (seis células) en la pipeta de inyección. Sujetar un embrión de ocho células o en estadio mórula, que haya completado la compactación, con la pipeta de sujeción por aspiración. Haga un agujero en la zona pelúcida con un pulso piezoeléctrico (intensidad: 3; velocidad: 3). Expulsar el ESC de seis células dentro de la zona pelúcida y sacar la pipeta de los embriones.
- Lave suavemente los embriones inyectados con ESC con varias gotas de KSOM con una pipeta bucal e incubarlos en una nueva gota de KSOM de 50 μL cubierta con aceite mineral a 37 °C, 5% deCO2 hasta que los embriones se desarrollen hasta la etapa de blastocisto.
- Transfiera los 10 blastocistos inyectados con ESC a cada cuerno uterino de ratones hembra pseudopreñados (2,5 días después del coito, 20 blastocistos por cabeza).
- Después de que la madre sustituta haya dado a luz, seleccione al menos dos quimeras masculinas con la proporción de quimerismo más alta (70% o más según lo confirmado por el color del pelaje) y luego aparearlas con hembras de cepa apropiadas para obtener el KI heterocigoto F1.
- Criar ratones KI individuales con socios apropiados para obtener ratones con genotipos deseables o antecedentes genéticos adecuados para la investigación.
Resultados
Nos hemos dirigido a genes específicos en el ESC seguidos de la producción de quimeras para desarrollar la producción de ratones manipulados genéticamente de acuerdo con nuestro manuscrito anterior11. El genotipado de ESC (descrito en la sección 4) se realiza de forma rutinaria mediante PCR utilizando cebadores. Los cebadores están diseñados sobre secuencias genómicas fuera de los brazos de homología y las secuencias específicas en el fragmento de ADN KI (Figura 2A). En ese caso, no se amplifica ningún alelo de tipo salvaje, mientras que un amplicón de PCR de un tamaño específico se detecta solo cuando el ADN exógeno objetivo es KI en el locus objetivo. Los resultados representativos de la PCR de genotipado se muestran en la Figura 2B. Nueve de los 22 clones (40,9%) mostraron una banda específica de KI en este caso. En la Tabla 1 se muestran tres resultados representativos de focalización, incluido el resultado que se muestra en la Figura 2. Estos resultados indican que el método que se muestra aquí es eficiente y reproducible para el gen KI sin ninguna selección de fármacos.
La ESC se recoge en una pipeta de inyección, luego se hace un orificio en la zona pelúcida utilizando un plus piezoeléctrico, y la ESC se libera entre las células embrionarias (Figura 3). Técnicamente, este protocolo para inyectar un ESC en un embrión de ocho células o en etapa mórula es similar al protocolo para la inyección de ESC en un blastocisto comúnmente utilizado en muchas instalaciones con ratones. Los ratones quiméricos representativos se muestran en la Figura 4. Para la evaluación del color del pelaje del quimerismo, el embrión de la cepa ICR (albino, pelo blanco) se utilizó como receptor de B6 o B6-129 F1 ESC, y los embriones B6 se utilizaron como receptores de BALB/c ESCs (Figura 4).
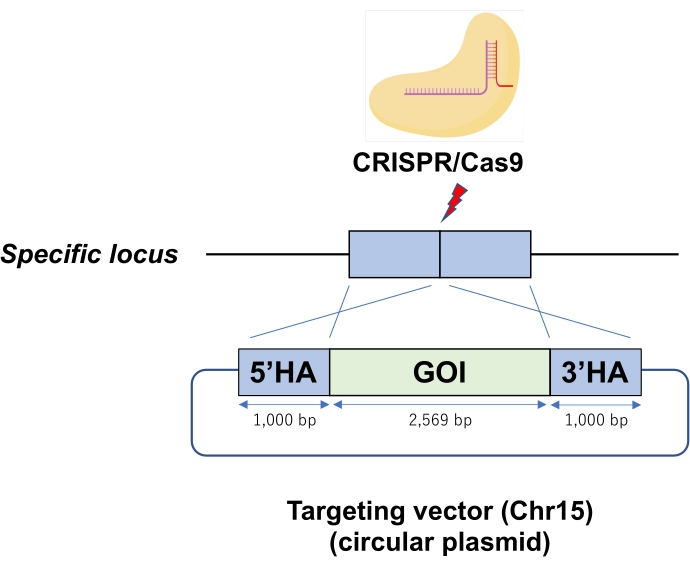
Figura 1: Un esquema de la integración de plásmidos circulares mediados por ribonucleoproteína (RNP) CRISPR/Cas9 en el locus específico de un genoma ESC. La electroporación introduce un plásmido circular como vector de focalización en ESCs con Cas9-RNP. Abreviaturas: GOI = gen de interés, HA = brazo de homología. Haga clic aquí para ver una versión más grande de esta figura.
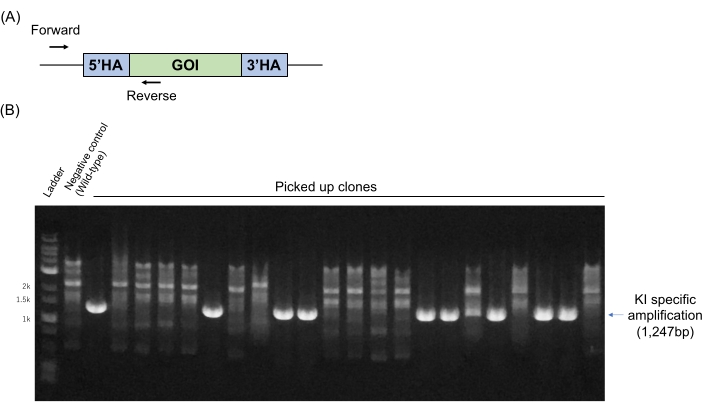
Figura 2: Análisis genómicos de PCR para el cribado de KI de clones ESC dirigidos. (A) Los cebadores de PCR específicos de KI están diseñados en secuencias genómicas fuera de los brazos de homología (hacia adelante) y en las secuencias específicas en el fragmento de ADN de KI (reverso). (B) Resultados representativos de la PCR de genotipado. Se utilizó un genoma de tipo salvaje como control negativo. Abreviaturas: GOI = gen de interés, HA = brazo de homología. Haga clic aquí para ver una versión más grande de esta figura.
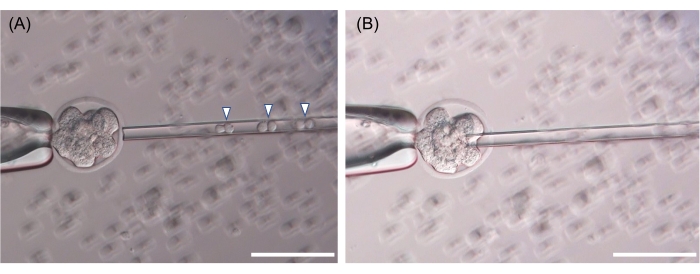
Figura 3: Imágenes representativas de la inyección embrionaria en estadio de ocho células. (A) Para la microinyección, se recogen tres dobletes de ESC (seis células, puntas de flecha). (B) Se hace un agujero en la zona pelúcida con un pulso piezoeléctrico, y las ESC se expulsan entre cada blastómero. La barra de escala que se muestra es de 50 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Imágenes representativas de ratones quimera . (A,B) El ESC derivado de C57BL/6N (JM8. A3, cabello agutí; A) o B6-129 F1 (cabello agutí; B) se inyectan en embriones ICR (albinos, pelo blanco), seguidos de la transferencia de los embriones a la madre sustituta. (C) El ESC derivado de BALB/c (albino, pelo blanco) se inyecta en embriones C57BL/6J (pelo negro), seguido de la transferencia de los embriones a madres sustitutas. Haga clic aquí para ver una versión más grande de esta figura.
| ID del proyecto | Coromosoma | 5'HA longitud (bp) | 3'HA longitud (bp) | Tamaño de inserción (bp) | Número de clones analizados | Número de clones de KI | Eficiencia (%) | Observaciones |
| R26-CC* | Chr6 | 965 | 1006 | 5321 | 23 | 2 | 8.7% | - |
| R4-03* | Chr8 | 1000 | 997 | 3070 | 22 | 6 | 27.3% | - |
| P4-01* | Chr15 | 1000 | 1000 | 2569 | 22 | 9 | 40.9% | Se muestra en la figura 2 |
Tabla 1: Eficiencias de KI en tres loci genómicos independientes en el ESC. *Estos proyectos no han sido publicados hasta ahora. El nombre de estos genes será revelado en manuscritos independientes en el futuro.
Discusión
La orientación génica de las ESC seguida de la producción de quimeras se ha utilizado convencionalmente para el desarrollo de ratones manipulados genéticamente. Sin embargo, la eficiencia de la introducción de genes sigue siendo baja a pesar de que el vector objetivo contiene brazos de homología largos (> varios kb en lo habitual) con casetes de genes de selección de fármacos positivos o negativos. Nuestro protocolo introdujo un método ESC KI ajustado de ADN exógeno largo utilizando un plásmido circular sin ningún casete de selección de fármacos como vector dirigido acompañado de edición del genoma mediada por Cas9-RNP a una eficiencia aceptable para trabajos de rutina. Por lo tanto, este protocolo podría ayudar a reducir sustancialmente la cantidad de tiempo necesario para producir ratones quiméricos modificados genéticamente en comparación con la orientación convencional de ESC.
En este protocolo, utilizamos CRISPR / Cas9-RNP para inducir roturas de doble cadena específicas del sitio en el genoma, y se usó un plásmido circular como vector objetivo en lugar de uno linealizado. Los plásmidos linealizados se utilizan convencionalmente como vector de focalización para el gen KI debido a su mayor eficiencia de integración genómica 8,9,10. Sin embargo, muchas integraciones genómicas son inespecíficas a pesar de que el vector contiene brazos homólogos9. Por otro lado, los plásmidos circulares rara vez se utilizan como vectores de focalización debido a su característica difícil de integrar en el genoma de las CES12 o fibroblastos13. Por lo tanto, sería posible que la aplicación de un plásmido circular como vector objetivo acompañado de edición del genoma mediada por CRISPR / Cas9 minimice la integración aleatoria no específica, pero maximice la integración específica del sitio en el genoma ESC. Sería notable que la eficiencia de inducción de la rotura de doble cadena por CRISPR/Cas9 es bastante importante14, por lo que sería esencial utilizar ARNg que induce una ruptura de doble cadena con alta eficiencia. Se debe considerar rediseñar el ARNg cuando la eficiencia del KI es demasiado baja. En el caso mostrado en la Figura 2, el 40,9% de los clones de ESC mostraron una banda específica de KI. De hecho, como laboratorio central del instituto, realizamos rutinariamente la orientación génica utilizando ESCs por el método descrito aquí y hemos logrado eficiencias de KI del 10% -50% para secuencias de 1-2 kb como Cre, CreERT o reporteros fluorescentes, aunque hay algunas variaciones dependiendo del locus del gen. Las secuencias de ADN más largas que tenemos con éxito KI usando este método es de aproximadamente 11.2 kb en el locus Rosa26, y la eficiencia fue del 12.2% (cinco colonias de KI de 41 colonias analizadas).
También cabe señalar que este protocolo utiliza embriones de ocho células o en estadio mórula como receptores para la microinyección de ESC, pero no embriones en estadio de blastocisto. Aunque no hemos realizado experimentos de comparación en este artículo, varios informes han demostrado que la inyección de ESCs en embriones de ocho células o en etapa mórula mejora significativamente la eficiencia de la contribución de ESC a la descendencia quimérica en comparación con la inyección de ESC en blastocistos15,16,17,18 . De hecho, las inyecciones de ESC de varias líneas de ESC, incluyendo C57BL / 6, B6-129 F1 y BALB / c, comúnmente resultaron en el desarrollo de quimeras de alto color de capa en algunos, pero no todos, los descendientes (ver Figura 4). La limitación del método es que las ESCs inyectadas en el embrión preimplantacional no siempre podían contribuir a las células germinales en una quimera19,20. La transmisión germinal de las ESCs dependería de la calidad de cada clon de ESC19. Por lo tanto, sería ventajoso desarrollar múltiples líneas de clonación ESC como respaldo para la producción estable de ratones quiméricos. En conclusión, los protocolos presentados aquí utilizan vectores de focalización simples que incluyen solo cada brazo de homología y gen de interés para KI sin ningún casete resistente a los medicamentos. Por lo tanto, la construcción del vector sería mucho más fácil, y el período de cultivo podría acortarse en comparación con la técnica convencional de focalización de ESC. Esto ayudaría a producir rápida y facilitativamente varios tipos de ratones modificados genéticamente para futuros análisis en ciencias de la vida.
Divulgaciones
Los autores no tienen intereses contrapuestos que revelar.
Agradecimientos
Agradecemos a Saki Nishioka en la Universidad de Osaka, Biotechnology Research and Development (organización sin fines de lucro), y Mio Kikuchi y Reiko Sakamoto en el Instituto de Ciencias Médicas de la Universidad de Tokio, por su excelente asistencia técnica. Esta labor contó con el apoyo de: las subvenciones KAKENHI del Ministerio de Educación, Cultura, Deportes, Ciencia y Tecnología (MEXT)/Sociedad Japonesa para la Promoción de la Ciencia (JSPS) a MI (JP19H05750, JP21H05033) y MO (20H03162); la subvención de la Agencia de Ciencia y Tecnología de Japón (JST) a MI (JPMJCR21N1); el Instituto Nacional de Salud Infantil y Desarrollo Humano Eunice Kennedy Shriver a MI (R01HD088412); la Fundación Bill & Melinda Gates a MI (subvención de Grand Challenges Explorations INV-001902); y la subvención para el Proyecto de Investigación Conjunta del Instituto de Investigación de Enfermedades Microbianas de la Universidad de Osaka a MI y MO.
Materiales
| Name | Company | Catalog Number | Comments |
| BALB/c ESC | - | - | ESC developed from BALB/c strain |
| Bambanker | Nippon Genetics | CS-02-001 | Cell-freezeing medium. Section 2.6 and elsewhere |
| Cas9 Nuclease V3 | IDT | 1081059 | Section 3.2 and elsewhere. |
| CHIR99021 | FUJIFILM Wako | 038-23101 | Section 4.3 |
| CreERT gene fragment | GeneWiz | Section 1.1. | |
| CRISPR-Cas9 crisprRNA | IDT | crisprRNA. Section 3.1 and elsewhere. | |
| CRISPR-Cas9 tracrRNA | IDT | 1072534 | tracrRNA. Section 3.1 and elsewhere. |
| DMEM | Nacalai | 08458-45 | MEF medium. Section 2.3 and elsewhere |
| Duplex buffer | IDT | 1072534 | RNA dilution buffer. Section 3.1 and elsewhere. |
| FastGene Gel/PCR Extraction Kit | Nippon Genetics | FG-91302 | Section 1.1 and 1.2. |
| GlutaMax | Thermo Fisher | 35-050-061 | L-glutatime substrate |
| hCG | ASKA Animal Health | Section 6.1. | |
| In-Fusion HD Cloning Kit | Clontech | 639648 | DNA cloning kit. Section 1.3 and elsewhere |
| JM8.A3 ESC | EuMMCR | - | ESC developed from C57BL/6N strain |
| Knock-out DMEM | Thermo Fisher | 10829018 | Section 4.3 and elsewhere, DMEM-based modified commercial medium. |
| KSOM | Merck | MR-121-D | Section 6.3 and 6.9. |
| Leukemia inhibitory factor | FUJIFILM Wako | 125-05603 | Section 4.3. No unit concentration data is supplied by the provider. Used 1,000-fold dilution in this protocol. |
| Neon Electroporation system | Thermo Fisher | MPK5000 | Section 3.2, 4.5 and elsewhere. The system containes electroporation buffer as well used in section 3.2. |
| NucleoSpin Plasmid Transfection-grade | Takara | U0490B | Section 1.6. |
| PD0325901 | FUJIFILM Wako | 162-25291 | Section 4.3 |
| PMSG | ASKA Animal Health | Section 6.1. | |
| Tail lysis buffer | Nacalai | 06169-95 | Section 5.5. |
| Trypsin-EDTA | Nacalai | 32777-15 | Section 2.2 and elsewhere |
| V6.5 ESC | - | - | ESC developed from B6J-129 F1 strain |
| X-ray irradiation device | Hitachi | MBR-1618R-BE | Section 2.6. |
Referencias
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Wang, H., et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153 (4), 910-918 (2013).
- Yang, H., et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 154 (6), 1370-1379 (2013).
- Oji, A., et al. CRISPR/Cas9 mediated genome editing in ES cells and its application for chimeric analysis in mice. Scientific Reports. 6, 31666(2016).
- Kaneko, T., Mashimo, T. Simple genome editing of rodent intact embryos by electroporation. PLoS One. 10 (11), 0142755(2015).
- Chen, F., et al. High-frequency genome editing using ssDNA oligonucleotides with zinc-finger nucleases. Nature Methods. 8 (9), 753-755 (2011).
- Hashimoto, M., Yamashita, Y., Takemoto, T. Electroporation of Cas9 protein/sgRNA into early pronuclear zygotes generates non-mosaic mutants in the mouse. Developmental Biology. 418 (1), 1-9 (2016).
- Mansour, S. L., Thomas, K. R., Capecchi, M. R. Disruption of the proto-oncogene int-2 in mouse embryo-derived stem cells: a general strategy for targeting mutations to non-selectable genes. Nature. 336 (6197), 348-352 (1988).
- Johnson, R. S., et al. Targeting of nonexpressed genes in embryonic stem cells via homologous recombination. Science. 245 (4923), 1234-1236 (1989).
- Hasty, P., Ramires-Solis, R., Krumlauf, R., Bradley, A. Introduction of a subtle mutation into the Hox-2.6 locus in embryonic stem cells. Nature. 350 (6315), 243-246 (1991).
- Ozawa, M., Emori, C., Ikawa, M. Gene targeting in mouse embryonic stem cells via CRISPR/Cas9 ribonucleoprotein (RNP) mediated genome editing. Genome Editing in Animals - Methods and Protocols 2nd edition. Hatada, I. , (2022).
- Yagi, M., et al. Derivation of ground-state female ES cells maintaining gamete-derived DNA methylation. Nature. 548 (7666), 224-227 (2017).
- Gassmann, M., Donoho, G., Berg, P. Maintenance of an extrachromosomal plasmid vector in mouse embryonic stem cells. Proceedings of National Academy of Sciences. 92 (5), 1292-1296 (1995).
- Okita, K., Nakagawa, M., Hyenjong, H., Ichisaka, T., Yamanaka, S. Generation of mouse induced pluripotent stem cells without viral vectors. Science. 322 (5903), 949-953 (2008).
- Poueymirou, W. T., et al. F0 generation mice fully derived from gene-targeted embryonic stem cells allowing immediate phenotypic analyses. Nature Biotechnology. 25 (1), 91-99 (2007).
- Hu, M., et al. Efficient production of chimeric mice from embryonic stem cells injected into 4- to 8-cell and blastocyst embryos. Journal of Animal Science and Biotechnology. 4 (1), 1-7 (2013).
- Guo, J., et al. Contribution of Mouse Embryonic Stem Cells and Induced Pluripotent Stem Cells to Chimeras through Injection and Coculture of Embryos. Stem Cells International. 2014, 409021(2014).
- Bodai, Z., Bishop, A. L., Gantz, V. M., Komor, A. C. Targeting double-strand break indel byproducts with secondary guide RNAs improves Cas9 HDR-mediated genome editing efficiencies. Nature Communications. 13 (1), 2351(2022).
- Kobayashi, T., Goto, T., Oikawa, M., Sanbo, M., Yoshida, F., Terada, R., Niizeki, N., Kajitani, N., Kazuki, K., Kazuki, Y., Hochi, S., Nakauchi, H., Surani, A., Hirabayashi, M. Blastocyst complementation using Prdm14-deficient rats enables efficient germline transmission and generation of functional mouse spermatids in rats. Nature Communications. 12 (1), 1328(2021).
- Miura, K., Matoba, S., Hirose, M., Ogura, A. Generation of chimeric mice with spermatozoa fully derived from embryonic stem cells using a triple-target CRISPR method for Nanos3. Biology of Reproduction. 104 (1), 223-233 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados