Method Article
Detección multiparamétrica de fármacos organoides tumorales mediante imágenes de células vivas de campo amplio para análisis a granel y de un solo organoide
En este artículo
Resumen
Este protocolo describe un método semiautomatizado para exámenes de detección de fármacos con organoides de rendimiento medio a alto y software de análisis de imágenes automatizado e independiente del microscopio para cuantificar y visualizar las respuestas multiparamétricas a fármacos de un solo organoide para capturar la heterogeneidad intratumoral.
Resumen
Los organoides tumorales derivados del paciente (PDTO) son muy prometedores para la investigación preclínica y traslacional y para predecir la respuesta terapéutica del paciente a partir de exámenes de detección de fármacos ex vivo . Sin embargo, los ensayos actuales de detección de fármacos basados en trifosfato de adenosina (ATP) no capturan la complejidad de una respuesta al fármaco (citostática o citotóxica) y la heterogeneidad intratumoral que se ha demostrado que se conserva en las PDTO debido a una lectura masiva. Las imágenes de células vivas son una herramienta poderosa para superar este problema y visualizar las respuestas a los medicamentos más en profundidad. Sin embargo, el software de análisis de imágenes a menudo no está adaptado a la tridimensionalidad de los PDTO, requiere tintes de viabilidad fluorescentes o no es compatible con un formato de microplaca de 384 pocillos. Este documento describe una metodología semiautomatizada para sembrar, tratar e obtener imágenes de PDTO en un formato de 384 pocillos de alto rendimiento utilizando sistemas convencionales de imágenes de células vivas de campo amplio. Además, desarrollamos un software de análisis de imágenes sin marcadores de viabilidad para cuantificar las métricas de respuesta a los medicamentos basadas en la tasa de crecimiento que mejoran la reproducibilidad y corrigen las variaciones de la tasa de crecimiento entre diferentes líneas de PDTO. Usando la métrica normalizada de respuesta al fármaco, que califica la respuesta al fármaco en función de la tasa de crecimiento normalizada a una condición de control positiva y negativa, y un colorante de muerte celular fluorescente, las respuestas citotóxicas y citostáticas a los fármacos se pueden distinguir fácilmente, mejorando profundamente la clasificación de respondedores y no respondedores. Además, la heterogeneidad farmacológica de la respuesta puede cuantificarse a partir del análisis de respuesta al fármaco de un solo organoide para identificar clones potenciales y resistentes. En última instancia, este método tiene como objetivo mejorar la predicción de la respuesta a la terapia clínica mediante la captura de una firma de respuesta farmacológica multiparamétrica, que incluye la detención cinética del crecimiento y la cuantificación de la muerte celular.
Introducción
En los últimos años, el descubrimiento de fármacos contra el cáncer in vitro , la detección de fármacos y la investigación fundamental han pasado del uso de modelos tradicionales de cáncer bidimensional (2D) con líneas celulares inmortalizadas a modelos de cáncer tridimensionales (3D) más relevantes fisiológicamente. Esto ha estimulado la adopción de esferoides tumorales con líneas celulares de cáncer establecidas, que recrean interacciones y estructuras más complejas de célula a célula presentes en tumores sólidos. Actualmente, los organoides tumorales derivados de pacientes (PDTO) son el modelo de cáncer 3D más avanzado y fisiológicamente relevante disponible para la investigación in vitro del cáncer, ya que proporcionan ventajas adicionales sobre los esferoides tumorales, a saber, la heterogeneidad encontrada en pacientes con cáncer1. Los PDTO se establecen a partir del tejido tumoral originado en pacientes con cáncer y, por lo tanto, conservan tanto el fenotipo como el genotipo del tumor. Como tales, las PDTO se están volviendo invaluables para la investigación fundamental y traslacional del cáncer y tienen el potencial de mejorar en gran medida la oncología de precisión2.
A pesar de su potencial prometedor, estos sofisticados modelos 3D in vitro de cáncer a menudo están infrautilizados debido a la falta de métodos de análisis avanzados. El ensayo más comúnmente utilizado determina el número de células viables en el PDTO a través de la cuantificación de ATP3 intracelular. Estos ensayos son normalmente análisis masivos de un solo punto de tiempo, por lo que pasan por alto las respuestas críticas dependientes del tiempo y descuidan las respuestas clonales. Específicamente, la capacidad de monitorear el crecimiento de las PDTO (tasa de crecimiento) y su respuesta a terapias específicas es de alto interés 4,5. La respuesta normalizada al fármaco (NDR), que califica la respuesta al fármaco en función de la tasa de crecimiento normalizada a una condición positiva (ctrl +) y control negativo (ctrl-), también se ha informado recientemente que es una métrica crucial para evaluar la sensibilidad a los medicamentos contra el cáncer con la detección basada en células, aunque esto se hizo predominantemente para las líneas celulares 2D6. Por lo tanto, se necesitan métodos de análisis más sofisticados para aprovechar al máximo estos modelos de cáncer 3D clínicamente más representativos y complejos. La microscopía es considerada un enfoque poderoso para estudiar la complejidad de estos modelos de organoides7.
Este artículo describe un método para monitorear las respuestas cinéticas a los medicamentos en modelos 3D de cáncer, utilizando microscopios convencionales de campo amplio y sistemas de imágenes de células vivas. Se realizaron adaptaciones al protocolo descrito por Driehuis et al.4 para que fuera compatible con la automatización utilizando un robot de pipeteo, un dispensador digital de medicamentos y un sistema de imágenes de células vivas para aumentar la reproducibilidad y reducir el número de horas de trabajo "práctico". Este método permite el cribado farmacológico de rendimiento medio a alto tanto de esferoides tumorales con líneas celulares cancerosas establecidas (consulte la Tabla suplementaria S1 para líneas celulares probadas), como de las PDTO, en un formato de microplaca y multiorganoide de 384 pocillos. Mediante el uso de un proceso de aprendizaje automático de red convolucional, la identificación automatizada y el seguimiento de esferoides tumorales individuales o PDTO podrían realizarse únicamente a partir de imágenes de campo claro y sin el uso de colorantes fluorescentes de etiquetado de células vivas8. Esto es muy ventajoso, ya que la mayoría de la identificación con imágenes de campo claro requiere anotación manual (que es laboriosa y requiere mucho tiempo) o requiere la adición de colorantes fluorescentes, que pueden confundir las respuestas farmacológicas relacionadas con el estrés oxidativo inducido por la fotoxicidad9.
El software de análisis de imágenes resultante desarrollado internamente amplía la funcionalidad de los sistemas convencionales de imágenes de células vivas, ya que los módulos de análisis de imágenes 3D no están disponibles, están restringidos por la plataforma o no son compatibles con microplacas de 384 pocillos e imágenes de pozos completos. Además, estos módulos a menudo tienen un precio alto y ofrecen lecturas limitadas de organoides a granel. Por lo tanto, este método es muy relevante para los científicos que tienen acceso a sistemas de imágenes de células vivas ampliamente disponibles y tienen como objetivo extraer más información sobre la respuesta a un fármaco en comparación con el ensayo basado en ATP estándar de oro pero rudimentario. Con la adición de indicadores específicos de muerte celular, las respuestas citostáticas a los fármacos se pueden distinguir de las respuestas citotóxicas, proporcionando así una mayor comprensión de las acciones mecanicistas de los fármacos actualmente inalcanzables a partir del análisis de un solo punto temporal. Finalmente, las imágenes de células vivas permiten el seguimiento de organoides individuales para obtener métricas de respuesta a fármacos de organoides individuales para capturar la heterogeneidad de la respuesta e identificar posibles subclones resistentes.
El objetivo de este método y el software de análisis de imágenes asociado es implementar una automatización de bajo costo en la detección de fármacos organoides para limitar la intervención del usuario y reducir la variabilidad en el manejo, el análisis de imágenes y el análisis de datos. Para que este software esté disponible para los investigadores, es independiente del microscopio y la plataforma, y se pone a disposición una aplicación basada en la nube. Por lo tanto, al admitir sistemas convencionales de imágenes de células vivas, también pretendemos mejorar su funcionalidad para aplicaciones y análisis de cultivo 3D.
Protocolo
Se utilizaron organoides derivados de pacientes con adenocarcinoma ductal pancreático humano (PDAC). Los fragmentos de resección tisular se obtuvieron de pacientes sometidos a cirugía curativa en el Hospital Universitario de Amberes. Se obtuvo el consentimiento informado por escrito de todos los pacientes, y el estudio fue aprobado por el Comité de Ética de la UZA (ref. 14/47/480). Los detalles relacionados con todos los materiales, reactivos, equipos y software utilizados en este protocolo se proporcionan en la Tabla de materiales. En la figura 1 se presenta una descripción general del flujo de trabajo. En el material complementario se proporcionan datos de ejemplo para reproducir el protocolo.
1. Día 0: Preparación de organoides de 2 o 3 días
- Precaliente las microplacas a 37 °C durante la noche y descongele la matriz extracelular (MEC) a 4 °C.
- Preparar el medio de cultivo organoide PDAC completo: suplemento ADF+++ (DMEM/F12 avanzado, suplemento de glutamina al 1%, HEPES al 1% y penicilina/estreptomicina al 1%) con proteína Wnt surrogada-Fc-Fusion al 0,5 nM, medio condicionado de proteína de fusión Noggin-Fc al 4%, medio condicionado de proteína de fusión Rpso3-Fc al 4%, 1x B27, 1 mM N-acetilcisteína (NAC), nicotinamida de 5 mM, 500 nM A83-01, 100 ng/ml FGF10 y 10 nM gastrina).
- Establecer los PDTOs según el método de elección.
NOTA: Driehuis et al. proporcionan un protocolo detallado que describe el método convencional para establecer, cultivar y pasar PDTOs en domos ECM4. - Disociar enzimáticamente los organoides en domos ECM.
- Aspirar el medio y lavar 1x con solución salina tamponada con fosfato (PBS). Agregue la enzima de disociación (por ejemplo, 2 ml en una microplaca de 6 pocillos) y pipete hacia arriba y hacia abajo 10 veces con una pipeta de 1 ml para disociar mecánicamente los organoides y las cúpulas de ECM.
- Incubar durante 10 min a 37 °C, pipetear hacia arriba y hacia abajo y comprobar si los organoides están disociados a células individuales. Repita este paso si es necesario.
- Recoja la suspensión celular en un tubo de 15 ml, agregue ADF+++ a un volumen de 10 ml, centrifugar durante 5 min a 450 × g a temperatura ambiente y aspirar el sobrenadante con una pipeta Pasteur y una bomba de succión.
- Resuspender el pellet en 100-200 μL de medio completo dependiendo del tamaño del pellet y contar el número de células utilizando el método de elección. Por ejemplo: mezclar 10 μL de la suspensión celular + 10 μL de Trypan Blue y contar con un contador de celdas automatizado.
- Placas de celdas individuales en domos ECM.
- Diluir la suspensión celular y añadir 2/3 ECM de acuerdo con la Tabla 1. Pipetear hasta diez gotas de 20 μL por pocillo en una placa precalentada de 6 pocillos. Invertir la placa e incubar durante 30 min a 37 °C.
- Superponer con medio completo suplementado con 10 μM Y-27632 e incubar durante 2-3 días en una incubadora.
NOTA: Diez domos que contienen 75,000 celdas cada uno suelen ser suficientes para llenar una microplaca de 384 pocillos a una concentración de 200 organoides / pocillo, excluyendo los pocillos en el borde.
2. Días 2 - 3: Cosecha y siembra de organoides de 2 o 3 días
- Recoge organoides intactos de las cúpulas ECM.
NOTA: Los organoides tienden a adherirse a superficies plásticas (por ejemplo, tubos, puntas de pipeta). Para evitar esto, los artículos de plástico se pueden enjuagar previamente con una solución de albúmina sérica bovina (BSA) / PBS al 0,1%.- Aspirar el medio y lavar 1x con PBS. Añadir 1-2 ml de solución de recolección de organoides fría (4 °C) a una placa de 6 pocillos dependiendo del número de domos ECM e incubar en hielo en una plataforma de agitación durante 10 min.
- Pipetear hacia arriba y hacia abajo con una pipeta de 1 ml para disociar las cúpulas de ECM, incubar durante 10 minutos adicionales en hielo y verificar visualmente bajo un microscopio si la ECM está disociada.
- Opcional: si se prefiere una distribución de tamaño más uniforme, filtrar la suspensión a través de un filtro celular de 70 μm antes de la centrifugación.
- Recoger los organoides en un tubo de 15 ml prerecubierto con BSA/PBS al 0,1%, añadir ADF+++ hasta 10 ml y centrifugar durante 5 min a 200 × g a 4 °C. Aspirar el sobrenadante y resuspender el pellet en hasta 1.000 μL de medio organoide PDAC completo dependiendo del tamaño del pellet para obtener una concentración de >6.000 organoides/mL.
- Cuente los organoides utilizando cualquier método de conteo de su elección, preferiblemente uno basado en imágenes.
- Siembra los organoides.
NOTA: Consulte la Tabla de materiales para conocer el volumen mínimo/pocillo para dos tipos diferentes de microplacas de 384 pocillos utilizadas en este protocolo.- Preenfríe todos los artículos de plástico a -20 °C o en hielo durante al menos 20 minutos antes de usarlos para evitar la solidificación de la ECM.
- Preparar la solución de siembra a partir de 1 ml de solución madre de organoides (paso 2.1.6) utilizando medio completo para sembrar ~200 organoides por pocillo en 50 μL, el volumen mínimo utilizado para llenar un pocillo. Utilice el Archivo suplementario 1 para calcular la cantidad de solución de siembra de organoides. Añadir un volumen residual de 1.500 μL cuando se utilice un depósito de 25 ml y una pipeta multicanal o un robot de pipeteo.
- Siembra los organoides usando un robot pipeteador.
NOTA: Tanto la solución de siembra como la microplaca deben enfriarse a 4 °C durante el pipeteo para evitar la solidificación de la ECM. Por lo tanto, un depósito de 25 ml y un soporte de microplaca se imprimieron en 3D para ser utilizados en combinación con el robot de pipeteo que puede contener los elementos de enfriamiento enumerados en la Tabla de materiales. Se proporcionan archivos STL para imprimir en 3D el material de laboratorio personalizado (Archivo complementario 2 y Archivo complementario 3) y archivos JSON de material de laboratorio personalizado para el robot de pipeteo (Archivo complementario 4 y Archivo complementario 5).- Diseñe el protocolo de dispensación utilizando la herramienta de diseño de protocolos en línea. Se proporciona un archivo JSON de ejemplo (archivo complementario 6), en el que el material de laboratorio personalizado ya está cargado y se utiliza una pipeta p300 (Gen2) de ocho canales con las puntas de pipeta correspondientes.
- Abra la aplicación de control del robot de pipeteo, seleccione protocolos, haga clic en Importar y arrastre y suelte el Archivo complementario 6 en el campo designado.
- Seleccione el protocolo importado y coloque todo el material de laboratorio, incluidos los elementos de refrigeración y los artículos de plástico, en las cubiertas de acuerdo con el diseño que se muestra en el campo Configuración de la plataforma . Utilice la ranura izquierda para el depósito de 25 ml y el elemento refrigerante, como se muestra en el archivo complementario 7.
- Haga clic en Ejecutar protocolo y proceda a la configuración. Abra la pestaña Configuración de Labware, haga clic en Run Labware Position Check y siga las instrucciones para calibrar el robot de pipeteo al nuevo hardware.
NOTA: Los datos de desplazamiento de material de laboratorio se pueden almacenar para más adelante, pero se recomienda ejecutar la comprobación de posición del material de laboratorio antes de cada ejecución. - Llene el depósito de 25 ml (colocado encima del elemento refrigerante) con la solución de siembra de organoides enfriada y haga clic en Start Run (Iniciar ejecución).
NOTA: Los pocillos superior e inferior también se llenan con solución de suspensión organoide debido al uso de la pipeta de ocho canales. - Centrifugar la microplaca durante 1 min a 100 × g a 4 °C.
- Incubar a 37 °C durante al menos 30 min.
- Llene los pocillos exteriores en blanco con al menos 50 μL deH2Opara evitar la evaporación.
- Incubar a 37 °C durante la noche para eliminar cualquier burbuja en el pozo que pueda interferir con el análisis de la imagen.
3. Día 4: Tratamiento farmacológico y dispensación de reactivos con dispensador digital de medicamentos
- Cree el protocolo de dispensación de medicamentos utilizando el software de control de dispensadores digitales de medicamentos.
- Desplácese sobre la placa 1 sobre el diseño de la placa, seleccione editar atributos de placa y rellene el tipo de placa: 384 pocillos, volumen adicional (μL): 50 y límite DMSO (%): 1.
- Agregue fluidos haciendo clic en el botón + junto a Fluidos. Haga doble clic en el fluido recién creado y asígnele un nombre; seleccione la clase (basada en DMSO o acuosa + Tween 20) y la concentración.
NOTA: Todos los medicamentos y reactivos deben disolverse en 100% DMSO o 0.3% Tween-20. Se puede utilizar una solución madre de 1-10 mM, teniendo en cuenta una concentración máxima de DMSO del <1%. La Tabla 2 proporciona ejemplos de las diluciones requeridas para reactivos y terapias fluorescentes comunes. - Diseño de la placa
- Para la titulación de medicamentos, seleccione pozos y haga clic en Titulación. Para el líquido, seleccione el fármaco de interés, elija la concentración más alta (por ejemplo, 2,000 nM) y la concentración más baja (por ejemplo, 10 nM); Para las réplicas, elija un mínimo de 2 y elija el patrón de valoración deseado.
NOTA: El patrón de valoración dependerá de muchos factores, incluyendo cuánto compuesto debe caber en una sola placa, si los pocillos deben ser aleatorizados y el número de réplicas y controles. - Para el control positivo, seleccione tres pocillos, haga clic en Establecer valor y complete 2 μM de estaurosporina de 10 mM en DMSO, lo que inducirá la muerte celular máxima.
- Para Cytotox Green, seleccione todos los pocillos utilizados, haga clic en Establecer valor e ingrese 60 nM/pocillo.
NOTA: La tinción fluorescente Cytotox Green indica células que han muerto y, por lo tanto, no interferirán con el monitoreo de la respuesta al medicamento. Aquí, no se requiere un marcador fluorescente para las células vivas. - Para el control negativo y la normalización DMSO, seleccione todos los pozos con cuatro pozos adicionales para el control del vehículo, haga clic con el botón derecho, seleccione normalización, seleccione normalizar la clase de fluido: basada en DMSO y normalizar al volumen de clase más alto para obtener una concentración de DMSO igual en cada pozo.
NOTA: Las concentraciones de DMSO deben ser <1%. Se proporciona un ejemplo de archivo de titulación de medicamentos TDD (Archivo suplementario 8). - Haga clic en la flecha debajo de Ejecutar en la esquina superior izquierda, seleccione Simular siempre y haga clic en Simular para identificar cualquier error y obtener los volúmenes de cada medicamento a preparar.
NOTA: Para superar una advertencia cuando el volumen de dispensación inicial es demasiado bajo, "Se recomienda un pozo de advertencia de dispensación de 30 nL o más para cada fluido en cada placa", seleccione dos pocillos en el borde que estén llenos de agua, seleccione Establecer valor e ingrese 10 μM del medicamento para el cual se produce la advertencia. Esto prepara el cartucho del fármaco con un volumen superior a 30 nL. Estos mismos pocillos se pueden usar para cebar el cartucho DMSO estableciendo una normalización a % del valor de volumen total (por ejemplo, 0.5%).
- Para la titulación de medicamentos, seleccione pozos y haga clic en Titulación. Para el líquido, seleccione el fármaco de interés, elija la concentración más alta (por ejemplo, 2,000 nM) y la concentración más baja (por ejemplo, 10 nM); Para las réplicas, elija un mínimo de 2 y elija el patrón de valoración deseado.
- Desmarque Simular siempre debajo del botón Ejecutar ; haga clic en Ejecutar para iniciar el protocolo de dispensación de medicamentos y siga las instrucciones.
- Aplique la membrana de sellado a la microplaca para evitar la evaporación.
- Incubar el colorante Cytotox Green 1-2 h a 37 °C en la incubadora y continuar con el paso 4.
4. Adquiera imágenes con el generador de imágenes de células vivas
NOTA: Para la tasa de crecimiento y NDR, se debe adquirir una exploración en el punto de tiempo 0 (T0 = iniciar el tratamiento) 1-2 h después de agregar Cytotox Green.
- Abra el software de control del generador de imágenes de celda activa, seleccione Editor de métodos Nuevo, vaya al archivo > importar y seleccione el archivo XML del método de ejemplo (archivo complementario 9). Alternativamente, cree un nuevo archivo y seleccione Placa: (CORE384fb_OpticalImaging) - Corning 384 Flat Black (Corning #4588), Sin tapa y sin casete de humedad; Aplicación: Sólo imágenes; Objetivo: 4x; Patrón: Central; Compruebe los canales Brightfield y Green (intensidad del LED (%) = 40; Tiempo de exposición (ms) = 200).
NOTA: La configuración del canal verde funciona bien para una concentración de 60 nM Cytotox Green. La opción Live viewer se puede utilizar para ajustar el desplazamiento del enfoque y/o la configuración del led en tiempo real. - Haga clic en Inicio para iniciar el escaneo en T0.
- Repita el escaneo cada 24 h durante un máximo de 5 días utilizando el mismo método. Como alternativa, para ejecutar la medición de lapso de tiempo automáticamente, ajuste el método en el software Control del generador de imágenes de células vivas a un experimento cinético haciendo clic y arrastrando la pestaña Bucle cinético al campo del método . Del mismo modo, las pestañas Temperatura y Gas deben arrastrarse al campo de método para ajustar el sistema a 37 °C y 5% deCO2 para garantizar las condiciones correctas dentro del generador de imágenes de células vivas durante el experimento.
5. Análisis de imágenes y datos
- Combinar y comprimir datos
- El software de control de imágenes de células vivas genera una carpeta para cada escaneo en cada punto de tiempo. Cree una nueva carpeta, copie las carpetas de experimento individuales en esta nueva carpeta principal y agregue _0h, _24h, _48h, _72h, _96h y _120h a los nombres de carpeta de experimento correspondientes.
- Prepare un mapa de placas XLSX desde el software de control del dispensador digital de medicamentos haciendo clic derecho en el diseño del mapa de placas del protocolo de dispensación de medicamentos y copie todos los pocillos; pegue los datos en un archivo XLSX. Elimine los datos de Cytotox Green y staurosporine y agregue una matriz para Cell Line y Replicate. Escriba ctrl- y ctrl+. Consulte el Archivo complementario 10 para ver un ejemplo de mapa de placas.
- Abra la herramienta de compresión de datos, haga clic en Examinar, seleccione la carpeta principal y haga clic en Ejecutar para iniciar la compresión de datos de imagen. Todos los archivos de imagen TIFF para los diferentes puntos de tiempo se comprimen en un solo HDF5 para cada pocillo en una nueva carpeta de conjuntos de datos dentro de la carpeta parental.
- Análisis de imágenes
- Vaya a la plataforma de aplicaciones web de análisis de imágenes, inicie sesión y haga clic en Agregar nuevo proyecto en la pestaña Inicio . Introduzca el nombre del proyecto, continúe, seleccione Agregar nuevo experimento y cargue la carpeta de conjuntos de datos que contiene los archivos HDF5.
- Después de cargar, vaya a la carpeta de proyectos y experimentos y haga clic en Cargar mapa de placas para obtener funcionalidades adicionales. Haga clic en Ejecutar análisis, seleccione Análisis multiorganoide, Parámetros predeterminados y haga clic en Analizar para iniciar el análisis de imágenes.
- Haga clic en Descargar resultados para descargar las tablas de datos sin procesar que contienen las mediciones para cada pozo (por ejemplo, área total de campo claro, área verde de fluorescencia total, etc.) y las imágenes / videos segmentados para confirmar la precisión del análisis y el procesamiento posterior de datos.
- Métricas de respuesta a medicamentos basadas en la tasa de crecimiento y respuesta normalizada a los medicamentos
- Seleccione el archivo Raw_NDR.xlsx de la carpeta de resultados (se requiere mapa de placas) (Archivo complementario 11) y cárguelo en el script R Official_NDR_7point (Archivo complementario 12) para generar automáticamente tablas de valores GR (normalizado a ctrl-) y NDR (normalizado a ctrl y ctrl+) (Archivo complementario 13, Archivo complementario 14, Archivo complementario 15 y Archivo complementario 16 ). Los valores GR y NDR se calculan a partir del parámetro como se muestra en la ecuación (1) utilizando el R-script (Archivo Suplementario 12).
Área total de supervivencia = Área total de campo brillante - Área verde total (1)
Donde 0 < NDR <1 = efecto citostático (detención del crecimiento) y NDR < 0 = respuesta citotóxica (muerte celular).
NOTA: El guión R fue adaptado de Gupta et al.6. - De la tabla clonal_data.xlsx, recupere los datos de respuesta de un solo organoide y tráquelos como un gráfico de burbujas.
- Utilice el factor Z10 para evaluar la calidad de la prueba de detección de drogas de una corrida (consulte la ecuación (2)). Descarte un experimento con un factor Z < 0.5.
 (2)
(2)
- Seleccione el archivo Raw_NDR.xlsx de la carpeta de resultados (se requiere mapa de placas) (Archivo complementario 11) y cárguelo en el script R Official_NDR_7point (Archivo complementario 12) para generar automáticamente tablas de valores GR (normalizado a ctrl-) y NDR (normalizado a ctrl y ctrl+) (Archivo complementario 13, Archivo complementario 14, Archivo complementario 15 y Archivo complementario 16 ). Los valores GR y NDR se calculan a partir del parámetro como se muestra en la ecuación (1) utilizando el R-script (Archivo Suplementario 12).
Resultados
El protocolo de pipeteo automatizado garantiza una distribución uniforme de PDAC_060 PDTO en todas las columnas de la microplaca de 384 pocillos (Figura 2A). Como era de esperar, se observó una variación en el número y el área media de PDTO entre los pozos (Figura 2A, B). El área de supervivencia total (área total de campo claro - área verde total) combina la segmentación de organoides sin etiqueta con la señal de muerte celular basada en fluorescencia y es, en nuestra experiencia, el parámetro más robusto para estudiar las respuestas a los fármacos a lo largo del tiempo (Figura 2C)8. Para tener en cuenta las variaciones en la siembra celular y el tamaño de los organoides, se deben usar métricas basadas en la tasa de crecimiento para reducir las variaciones entre réplicas, como se muestra en las barras de error reducidas en la Figura 2D frente a la Figura 2C, y un factor Z más alto que indica una calidad de detección de medicamentos muy mejorada (Figura 2E).
La curva dosis-respuesta NDR (Figura 2G), normalizada a ctrl- y ctrl+, es claramente superior a la curva dosis-respuesta GR (Figura 2F), normalizada a ctrl-, ya que aumenta la separación de las curvas de respuesta al fármaco y representa con mayor precisión las respuestas citotóxicas a los fármacos. En la Figura 3 se muestra un ejemplo de las imágenes asociadas para la PDTO tratada con paclitaxel ctrl, ctrl+ y gemcitabina/80 nM de gemcitabina/80 nM. Una observación interesante es que el efecto citotóxico de la gemcitabina fue dominante en la terapia combinada ya que no se observó ningún valor agregado de paclitaxel.
A continuación, se utilizaron dos líneas PDTO adicionales, PDAC_052 y PDAC_087. Se observó una clara diferencia en la tasa de crecimiento entre estas líneas (Figura 4A), que apoya el uso de métricas de GR. Una vez más, las curvas dosis-respuesta NDR (Figura 4C) dieron lugar a un aumento del rango dinámico y la separación entre los tres pacientes diferentes en comparación con las curvas GR (Figura 4B). Además, el protocolo permite la determinación de NDR a lo largo del tiempo y muestra que PDAC_052 y PDAC_060 tuvieron una respuesta citostática muy similar a una dosis baja de gem-pac (Figura 4D), mientras que se pudo observar una clara respuesta citostática diferencial versus citotóxica para las dosis medias (Figura 4E) y altas (Figura 4F) de gem-pac. Estas respuestas farmacológicas fueron consistentes con las respuestas clínicas observadas en los pacientes (Figura 4G).
Finalmente, un beneficio importante del enfoque y el software es que las respuestas de un solo organoide se pueden cuantificar para estudiar la heterogeneidad de la respuesta e identificar subclones potencialmente resistentes. La Figura 5 proporciona una visión clara de la dinámica clonal de los diferentes pacientes y muestra que PDAC-087 tuvo los subclones más resistentes después del tratamiento, lo que es consistente con la enfermedad agresiva y altamente resistente observada en el paciente. Curiosamente, este paciente también fue el menos sensible a la estaurosporina ctrl +.
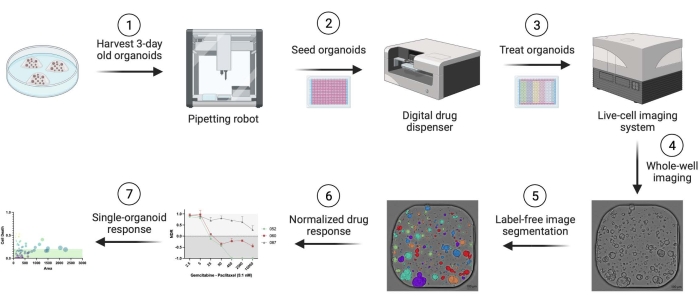
Figura 1: Descripción general del flujo de trabajo. Haga clic aquí para ver una versión más grande de esta figura.
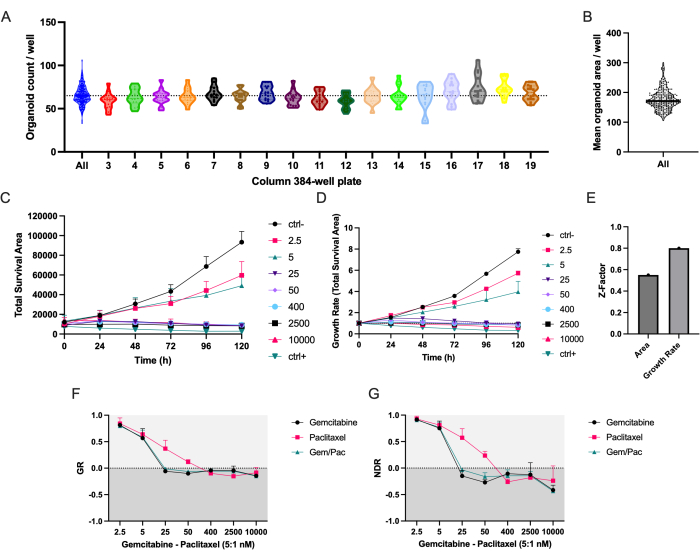
Figura 2: Precisión de siembra y métricas de respuesta a fármacos . (A) Conteos de organoides/pocillo de PDAC_060 PDTO sembrados en una microplaca de 384 pocillos utilizando el robot de pipeteo. Cada punto representa el recuento en un solo pozo y las parcelas están separadas por las columnas de microplacas de 384 pocillos. (B) Área media de PDTO/pozo. (C) área de supervivencia total (área total de campo claro - área verde total) y (D) tasa de crecimiento (área de supervivencia total normalizada a T0 = 1) de PDAC_060 PDTO tratados con una proporción de 5:1 de gemcitabina/paclitaxel. (E) Factor Z como métrica para la calidad del ensayo. (F) Curva de tasa de crecimiento-dosis-respuesta normalizada a ctrl- y (G) curva normalizada de respuesta al fármaco normalizada a ctrl- y ctrl+. Las barras de error indican la media ± DE de dos pocillos. Abreviaturas: PDAC = adenocarcinoma ductal pancreático; PDTO = organoide tumoral derivado del paciente; GR = tasa de crecimiento; NDR = respuesta normalizada al fármaco. Haga clic aquí para ver una versión más grande de esta figura.
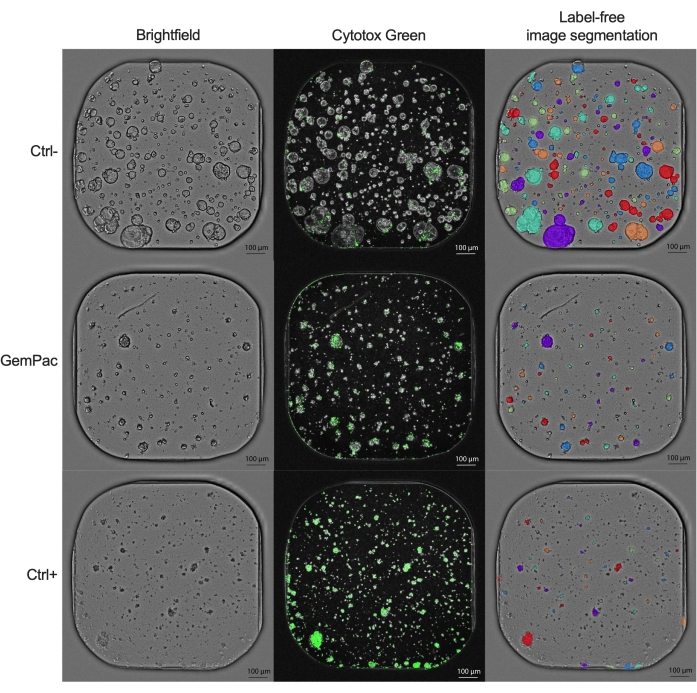
Figura 3: Imágenes de ejemplo. Imágenes representativas de PDAC_060 PDTO tratada con vehículo (ctrl-), gemcitabina de 400 nM/paclitaxel de 80 nM y estaurosporina de 2 μM (ctrl+). La columna izquierda muestra imágenes de campo claro, la columna central muestra la señal fluorescente verde Cytotox y la columna derecha muestra las imágenes de campo claro anotadas sin etiqueta utilizando el módulo de análisis de organoides. Barras de escala = 100 μm. Abreviaturas: PDAC = adenocarcinoma ductal pancreático; PDTO = organoide tumoral derivado del paciente; GemPac = gemcitabina/paclitaxel. Haga clic aquí para ver una versión más grande de esta figura.
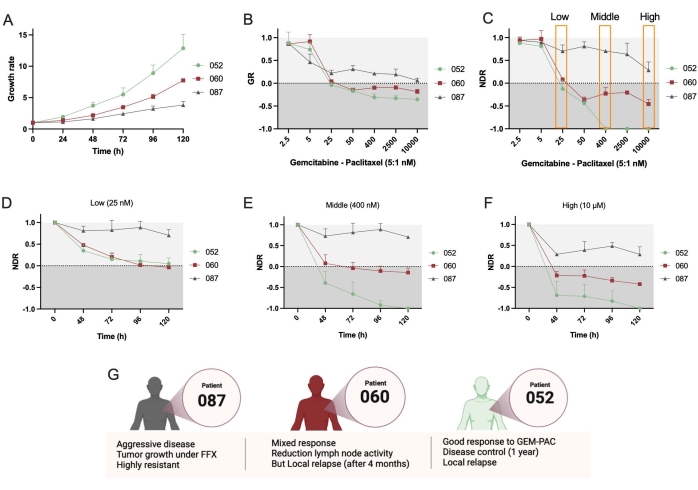
Figura 4: Comparación de la respuesta farmacológica entre pacientes. (A) Comparación de la tasa de crecimiento (basada en el área de supervivencia total) de las líneas PDTO PDAC_052, PDAC_060 y PDAC_087. (B) Curva de tasa de crecimiento-dosis-respuesta normalizada a ctrl- y (C) curva normalizada de respuesta al fármaco normalizada a ctrl- y ctrl+. NDR cinética de una dosis (D) baja, (E) media y (F) alta de gemcitabina/paclitaxel (relación 5:1). (G) Características clínicas de los pacientes con PDAC. Las barras de error indican la media ± DE de dos pocillos. Abreviaturas: PDAC = adenocarcinoma ductal pancreático; PDTO = organoide tumoral derivado del paciente; GR = tasa de crecimiento; NDR = respuesta normalizada al fármaco; FFX = folfirinox. Haga clic aquí para ver una versión más grande de esta figura.
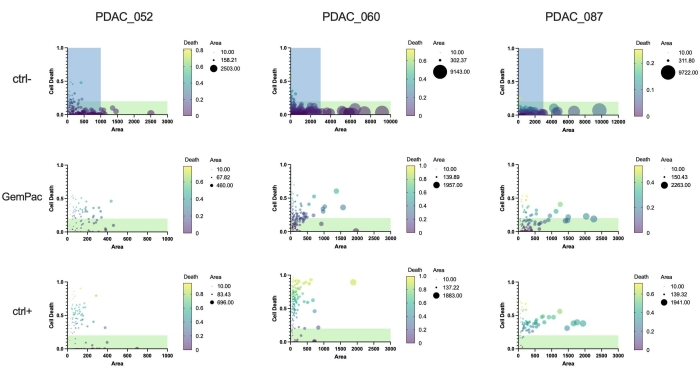
Figura 5: Métricas de organoides individuales. Respuesta a dosis única de organoide basada en la muerte celular (área verde/área de campo claro) y el área (campo claro) de PDAC_052, PDAC_060 y PDAC_087 PDTO tratados con vehículo (ctrl-), gemcitabina de 400 nM/80 nM paclitaxel y estaurosporina 2 μM (ctrl+). Las regiones verdes indican organoides viables; las regiones azules indican el rango x-as de gráficos GemPac y ctrl+. Abreviaturas: PDAC = adenocarcinoma ductal pancreático; PDTO = organoide tumoral derivado del paciente; GemPac = gemcitabina/paclitaxel. Haga clic aquí para ver una versión más grande de esta figura.
| Material de suspensión celular | Células/Gota | # Gotas (20 μL) | Existencias (1/3) | ECM (2/3) |
| 1,13 × 107 células/ml | 75,000 | 10 | 75 uL | 150 μL |
| 1,13 × 107 células/ml | 75,000 | 5 | 40 uL | 80 μL |
Tabla 1: Dilución para recubrimiento en domos ECM. Abreviatura: ECM = matriz extracelular.
| Compuesto | Concentración de existencias | Dilución | Concentración de trabajo | Solvente | Concentración de pozos | Comentarios |
| Cytotox Verde | 1 mM (DMSO) | 1/10 | 10 μM | DMSO | 60 nM | Marcador de muerte celular |
| Cytotox Rojo | 1 mM (DMSO) | 1/10 | 10 μM | DMSO | 250 nM | Marcador de muerte celular |
| Caspasa 3/7 Verde | 5 mM (DMSO) | 1/2 | 2,5 mM | DMSO | 2,5 μM | Marcador apoptótico |
| Hoechst | 20 mM (H2O) | 1/200 | 100 μM | 0,33% Preadolescentes/PBS | 50 nM | Marcador nuclear |
| Estaurosporina | 10 mM (DMSO) | / | 1 - 10 mM | / | 2 – 5 μM | Control positivo |
| Gemcitabina | 10 mM (DMSO) | / | 1 - 10 mM | / | Valoración | Quimioterapia |
| Paclitaxel | 10 mM (DMSO) | / | 1 - 10 mM | / | Valoración | Quimioterapia |
| Cisplatino | 5 mM (0,9% NaCl) | 1/2 | 2,5 mM | 0.6% Tween/PBS | Valoración | Quimioterapia |
Tabla 2: Ejemplos de diluciones de fármacos de uso frecuente y reactivos fluorescentes. Cada compuesto debe disolverse en 100% DMSO o 0.3% Tween/PBS.
Tabla suplementaria S1: Descripción general de las líneas celulares de cáncer compatibles. Estático: los esferoides no son migratorios. Fusionar: los esferoides migran unos hacia otros y se fusionan. Haga clic aquí para descargar este archivo.
Archivo complementario 1: Herramienta de cálculo de solución de siembra de organoides. Haga clic aquí para descargar este archivo.
Archivo complementario 2: archivo STL para impresión 3D de material de laboratorio personalizado 'Microplate Holder'. Haga clic aquí para descargar este archivo.
Archivo complementario 3: Archivo STL para impresión 3D de material de laboratorio personalizado '2 x 25 mL Reservoir Holder'. Haga clic aquí para descargar este archivo.
Archivo complementario 4: Archivo JSON para el robot de pipeteo de material de laboratorio personalizado 'Microplate Holder'. Haga clic aquí para descargar este archivo.
Archivo complementario 5: Archivo JSON para el robot de pipeteo de material de laboratorio personalizado '2 x 25 mL Reservoir Holder_WithCooler'. Haga clic aquí para descargar este archivo.
Archivo complementario 6: Archivo JSON para el protocolo de robot de pipeteo 'Plating_ PDO_384well_Cooled_Row2-23'. Haga clic aquí para descargar este archivo.
Archivo complementario 7: Descripción general de la configuración del escritorio del robot de pipeteo. (A) Elementos de enfriamiento y (B) depósito y microplaca. Haga clic aquí para descargar este archivo.
Archivo complementario 8: Archivo TDD para el protocolo del dispensador digital de medicamentos. Haga clic aquí para descargar este archivo.
Archivo complementario 9: Archivo XML para el protocolo del generador de imágenes de células vivas para imágenes de campo claro y fluorescencia. Haga clic aquí para descargar este archivo.
Archivo complementario 10: Ejemplo de mapa de placas. Haga clic aquí para descargar este archivo.
Archivo complementario 11: Archivo de entrada de ejemplo para el script NDR R. Abreviatura: NDR = respuesta normalizada al fármaco. Haga clic aquí para descargar este archivo.
Archivo complementario 12: Script NDR R de respuesta normalizada al fármaco. Abreviatura: NDR = respuesta normalizada al fármaco. Haga clic aquí para descargar este archivo.
Archivo complementario 13: Archivo de salida de ejemplo de valores GR de script NDR R. Abreviaturas: GR = tasa de crecimiento; NDR = respuesta normalizada al fármaco. Haga clic aquí para descargar este archivo.
Archivo complementario 14: Ejemplo de archivo de salida del script NDR R con valores GR transpuestos. Abreviaturas: GR = tasa de crecimiento; NDR = respuesta normalizada al fármaco. Haga clic aquí para descargar este archivo.
S upplementary File 15: Archivo de salida de ejemplo de valores NDR NDR script NDR. Abreviatura: NDR = respuesta normalizada al fármaco. Haga clic aquí para descargar este archivo.
Archivo complementario 16: Archivo de salida de ejemplo del script NDR R con valores NDR transpuestos. Abreviatura: NDR = respuesta normalizada al fármaco. Haga clic aquí para descargar este archivo.
Discusión
La detección de drogas PDTO de rendimiento medio a alto a menudo se basa en lecturas que solo extraen una fracción de la información que los organoides podrían proporcionar. Se ha vuelto cada vez más claro que, para que la tecnología de organoides en rápida evolución alcance un mayor potencial científico y clínico, se requieren críticamente ensayos 3D más avanzados, lecturas y métodos de análisis. Aquí, se describe una línea de detección avanzada, que no solo aumenta la reproducibilidad, sino que también mejora considerablemente la traducibilidad clínica al incorporar una lectura de imágenes de células vivas impulsada por IA. Además del software de análisis desarrollado internamente, se implementa el uso de la métrica normalizada de respuesta a medicamentos (NDR), que demuestra claramente su capacidad para definir las diferencias específicas del paciente en la respuesta al tratamiento6.
La inclusión de esta métrica de normalización será, sin duda, de gran valor, recordando que numerosos estudios tienen como objetivo delinear las respuestas al tratamiento basadas en diferencias menores en el área bajo la curva (AUC) o la concentración inhibitoria semimáxima (IC50) (ya que la mayoría de las curvas dosis-respuesta se superponen/están ubicadas cerca unas de otras)11,12 . Las métricas de la tasa de crecimiento ya se han implementado en protocolos de detección de fármacos organoides utilizando el ensayo basado en ATP, pero se basan en la normalización de los pocillos de referencia lisados en el punto de tiempo 04. En contraste, este método permite la normalización de la tasa de crecimiento intrapozo, que no solo explica las diferencias entre pacientes en la tasa de crecimiento de PDTO, sino también las diferencias entre pozos resultantes de las variaciones en la densidad de siembra y los efectos dependientes de la ubicación de la placa para aumentar la reproducibilidad. Además, adaptamos la NDR para aumentar aún más la separación de la respuesta de PDTO interpaciente al incluir un control positivo para la normalización 6,8.
Además, el análisis, que es compatible con formatos de alto rendimiento y automatización, puede detectar con precisión las respuestas individuales de los organoides, lo que permite cuantificar la resistencia subclonal, la principal fuerza impulsora de la recaída y progresión tumoral13. Por ejemplo, aunque PDAC052 y PDAC060 mostraron una buena respuesta al tratamiento in vitro (basado en la NDR), el análisis adicional de un solo organoide fue capaz de detectar una población pequeña (población más grande con PDAC060) de subclones que no responden al tratamiento. Curiosamente, esto se correspondió altamente con la observación clínica, dado que PDAC052 y PDAC060 tuvieron una respuesta duradera (no se detectó actividad tumoral), pero finalmente ambos fueron diagnosticados con progresión tumoral local (debido a la presencia de clones resistentes). En comparación con las lecturas 3D convencionales (ensayo basado en ATP y tamaño/números), se espera que esta línea de detección avanzada aumente el rendimiento predictivo al extraer más información clínicamente relevante de estos "pacientes en el laboratorio". Esta hipótesis ahora se está probando mediante el cribado de muestras clínicas de PDTO en el laboratorio de los autores con este método para correlacionar ex vivo con la respuesta in vivo y el resultado clínico.
Para obtener más información sobre los mecanismos de respuesta a un fármaco, los reactivos convencionales de imágenes fluorescentes de células vivas, además de los colorantes de citotoxicidad, son compatibles con este método para estudiar los mecanismos de muerte celular. Hemos demostrado previamente la compatibilidad de este método con el Reactivo Verde Caspasa 3/7 de Sartorius para estudiar la inducción de apoptosis dependiente de caspasa después del tratamiento con cisplatino8. La compatibilidad con otros colorantes para estudiar el estrés oxidativo (reactivos CellROX) o la hipoxia (reactivos de hipoxia Image-iT) aún no se ha probado. Sin embargo, estos reactivos ya han sido utilizados con éxito en modelos 3D in vitro 14,15.
El software de análisis de imágenes también es compatible con otros formatos de placas o métodos de cultivo (por ejemplo, placas de microcavidades, domos ECM) si se pueden capturar imágenes claras y enfocadas de los organoides. Esto es a menudo un desafío para los organoides cultivados en domos, ya que crecen en diferentes planos z, lo que requiere la funcionalidad de apilamiento z del microscopio que no siempre está disponible. Por lo tanto, recomendamos el uso de microplacas ULA de 384 pocillos de fondo plano para garantizar imágenes de calidad suficiente.
Además, el análisis es compatible con otros sistemas de imágenes de células vivas, como se mostró anteriormente para imágenes de contraste de fase capturadas con un sistema IncuCyte ZOOM8. Una limitación del sistema de imágenes de células vivas Spark Cyto que se utilizó en este manuscrito es la capacidad de una placa para mediciones cinéticas. Sin embargo, la expansión Spark Motion aumenta su capacidad hasta 40 microplacas que se pueden cribar a granel. La compatibilidad del software desarrollado internamente se ampliará a estos y otros sistemas para ofrecer una solución independiente de la plataforma, con el objetivo de estandarizar y automatizar las tuberías de análisis de imágenes y datos. La aplicación basada en la web también incluirá herramientas gráficas interactivas y cálculos automatizados de métricas de medicamentos, como se muestra en este documento, para reducir el tiempo de análisis manual.
El algoritmo de segmentación PDTO sin etiquetas fue entrenado y probado en varios modelos de esferoides y PDTO cultivados internamente con distintas diferencias morfológicas (sólido, semisólido, quístico), y en consecuencia puede detectarlos con alta precisión8. Una limitación del modelo es que la inclusión de PDTOs quísticas aumentó la detección no deseada de burbujas presentes en el pozo después de la siembra. Sin embargo, la incubación nocturna fue suficiente para eliminar la mayoría de estas burbujas, lo que permitió un escaneo cualitativo del punto de tiempo 0. La precisión de la segmentación de la imagen organoide y el método deben ser validados por otros usuarios y, en función de sus comentarios, el software puede ser entrenado aún más para obtener un algoritmo de análisis de imágenes robusto y automatizado. Además, nuestro objetivo es obtener más datos clínicos para correlacionar la respuesta farmacológica ex vivo cuantificada por este método con la respuesta clínica en el paciente para identificar los mejores parámetros para predecir la respuesta a la terapia y desarrollar aún más este método para la medicina funcional de precisión del cáncer16.
Divulgaciones
Los autores declaran no tener ningún conflicto de intereses.
Agradecimientos
Parte de esta investigación fue financiada por donaciones de diferentes donantes, incluidos Dedert Schilde vzw y el Sr. Willy Floren. Este trabajo está parcialmente financiado por la Fundación de Investigación Flamenca, 12S9221N (A.L.), G044420N (S.V., A.L., E.G), 1S27021N (M.L), y por el Fondo de Investigación Industrial de la Universidad de Amberes, PS ID 45151 (S.V., A.L., C.D.). La figura 1 se creó con BioRender.com.
Materiales
| Name | Company | Catalog Number | Comments |
| 6-well plate | Greiner | 657160 | |
| 8-Channel p300 (GEN 2) pipette | Opentrons | ||
| 300 µL Tips | Opentrons | ||
| 384-well flat-bottom ULA microplate | Corning | 4588 | minimum volume 50 µL |
| 384-well flat-bottom ULA Phenoplate | Perkin Elmer | 6057802 | minimum volume 75 µL |
| A8301 | Tocris Bioscience | 2939 | |
| ADF+++ | Advanced DMEM/F12, 1% GlutaMAX, 1% HEPES, 1% penicillin/streptomycin | ||
| Advanced DMEM/F-12 | ThermoFisher Scientific | 12634 | |
| B27 | ThermoFisher Scientific | 17504044 | |
| Breathe easy sealing membrane | Sigma-Aldrich | Z380059 | |
| Caspase 3/7 Green | Sartorius | 4440 | |
| Cell Counting Slides for TC10/TC20 | Bio-Rad Laboratories | 1450017 | |
| CellTiter-Glo 3D | Promega | G9681 | ATP-assay |
| Cooler for 25 mL reservoir | VWR (Diversified Biotech) | 490006-908 | |
| Cooling element 12 x 8 x 3 cm | Bol.com | 9200000107744702 | For custom microplate holder OT-2 |
| Cultrex Organoid Harvesting Solution | R&D systems | 3700-100-01 | |
| Cultrex PathClear Reduced Growth Factor BME, Type 2 | R&D systems | 3533-010-02 | extracellular matrix (ECM) |
| Cytotox Green | Sartorius | 4633 | |
| Cytotox Red | Sartorius | 4632 | |
| D300e | Tecan | Digital drug dispenser | |
| D300e Control v3.3.5 | Tecan | Control software D300e | |
| FGF10 | Peprotech | 100-26 | |
| Full Medium | ADF+++ supplemented with 0.5 nM WNT surrogate-Fc-Fusion protein, 4% Noggin-Fc Fusion Protein conditioned medium, 4% Rpso3-Fc Fusion Protein conditioned medium, 1x B27, 1 mM N-acetyl cysteine (NAC), 5 mM nicotinamide, 500 nM A83-01, 100 ng/mL FGF10, and 10 nM Gastrin | ||
| Gastrin | Sigma-Aldrich | G9145 | |
| Gemcitabine | Selleck Chemicals | S1714 | |
| GlutaMAX | ThermoFisher Scientific | 35050 | |
| HEPES | ThermoFisher Scientific | 15630056 | |
| Hoechst 33342 Solution (20 mM) | ThermoFisher Scientific | 62259 | |
| Human pancreatic ductal adenocarcinoma (PDAC) patient-derived organoids | Biobank@uza (Antwerp, Belgium; ID: BE71030031000; Belgian Virtual Tumorbank funded by the National Cancer Plan) | ||
| N-acetyl-cysteine | Sigma-Aldrich | A9165-25G | |
| Nicotinamide | Sigma-Aldrich | N0636-100G | |
| Noggin-Fc Fusion Protein conditioned medium | Immunoprecise | N002 | |
| Opentrons App v6.0.1 | Opentrons | OT-2 control software | |
| Opentrons Protocol Designer Tool | Opentrons | https://designer.opentrons.com/ | |
| Orbits data compression tool | www.orbits-oncology.com or contact corresponding author | ||
| Orbits image analysis webapp | University of Antwerp | www.orbits-oncology.com or contact corresponding author | |
| OT-2 | Opentrons | Pipetting robot | |
| Paclitaxel | Selleck Chemicals | S1150 | |
| Pasteur Pipette 230 mm | Novolab | A33696 | |
| Peniciline-Streptomycin | ThermoFisher Scientific | 15140 | |
| Prism 9 | GraphPad | ||
| Rspo3-Fc Fusion Protein conditioned medium | Immunoprecise | N003 | |
| Spark Cyto 600 | Tecan | Live-cell imaging and multi-mode platereader | |
| SparkControl v3.1 | Tecan | Spark Cyto control software | |
| Staurosporine | Tocris Bioscience | 1285 | |
| Sterile 25 mL reservoir | VWR (Diversified Biotech) | 10141-922 | |
| T8 plus cassette | Tecan | ||
| TC20 | Bio-Rad Laboratories | automated cell counter | |
| TrypLE | ThermoFisher Scientific | 12604-021 | dissociation enzyme |
| Tween-20 | Acros Organics | 233360010 | |
| WNT Surrogate-Fc-Fusion protein | Immunoprecise | N001 | |
| Y-27632 | Selleck Chemicals | S1049 |
Referencias
- Drost, J., Clevers, H. Organoids in cancer research. Nature Reviews Cancer. 18 (7), 407-418 (2018).
- Veninga, V., Voest, E. E. Tumor organoids: Opportunities and challenges to guide precision medicine. Cancer Cell. 39 (9), 1190-1201 (2021).
- Le Compte, M., et al. Patient-derived organoids as individual patient models for chemoradiation response prediction in gastrointestinal malignancies. Critical Reviews in Oncology/Hematology. 157, 103190(2021).
- Driehuis, E., Kretzschmar, K., Clevers, H. Establishment of patient-derived cancer organoids for drug-screening applications. Nature Protocols. 15 (10), 3380-3409 (2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nature Methods. 13 (6), 521-527 (2016).
- Gupta, A., Gautam, P., Wennerberg, K., Aittokallio, T. A normalized drug response metric improves accuracy and consistency of anticancer drug sensitivity quantification in cell-based screening. Communications Biology. 3 (1), 42(2020).
- Rios, A. C., Clevers, H. Imaging organoids: a bright future ahead. Nature Methods. 15 (1), 24-26 (2018).
- Deben, C., Cardenas, E., et al. OrBITS: label-free and time-lapse monitoring of patient derived organoids for advanced drug screening. Cellular Oncology. , (2022).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. Bioessays. 39 (8), (2017).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Engel, R. M., et al. Patient-derived colorectal cancer organoids upregulate revival stem cell marker genes following chemotherapeutic treatment. Journal of Clinical Medicine. 9 (1), 128(2020).
- Hennig, A., et al. Detecting drug resistance in pancreatic cancer organoids guides optimized chemotherapy treatment. The Journal of Pathology. 257 (5), 607-619 (2022).
- D'Alterio, C., Scala, S., Sozzi, G., Roz, L., Bertolini, G. Paradoxical effects of chemotherapy on tumor relapse and metastasis promotion. Seminars in Cancer Biology. 60, 351-361 (2020).
- Njoroge, R. N., et al. Organoids model distinct Vitamin E effects at different stages of prostate cancer evolution. Scientific Reports. 7 (1), 16285(2017).
- Godet, I., et al. Fate-mapping post-hypoxic tumor cells reveals a ROS-resistant phenotype that promotes metastasis. Nature Communications. 10 (1), 4862(2019).
- Letai, A. Functional precision cancer medicine-moving beyond pure genomics. Nature Medicine. 23 (9), 1028-1035 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados