Method Article
Screening multiparametrico di farmaci organoidi tumorali mediante imaging a cellule vive a campo ampio per analisi di massa e organoidi singoli
In questo articolo
Riepilogo
Questo protocollo descrive un metodo semi-automatico per screening di farmaci organoidi a medio-alto rendimento e un software di analisi delle immagini automatizzato indipendente dal microscopio per quantificare e visualizzare risposte multiparametriche a singolo organoide per catturare l'eterogeneità intratumorale.
Abstract
Gli organoidi tumorali derivati dal paziente (PDTO) sono molto promettenti per la ricerca preclinica e traslazionale e per prevedere la risposta terapeutica del paziente dagli screening ex vivo dei farmaci. Tuttavia, gli attuali test di screening dei farmaci basati sull'adenosina trifosfato (ATP) non catturano la complessità di una risposta farmacologica (citostatica o citotossica) e l'eterogeneità intratumorale che ha dimostrato di essere mantenuta nei PDTO a causa di una lettura di massa. L'imaging delle cellule vive è un potente strumento per superare questo problema e visualizzare le risposte ai farmaci in modo più approfondito. Tuttavia, il software di analisi delle immagini spesso non è adatto alla tridimensionalità dei PDTO, richiede coloranti fluorescenti di vitalità o non è compatibile con un formato di micropiastre a 384 pozzetti. Questo documento descrive una metodologia semi-automatizzata per seminare, trattare e visualizzare PDTO in un formato ad alto throughput, 384 pozzetti utilizzando sistemi di imaging convenzionali, a largo campo, a cellule vive. Inoltre, abbiamo sviluppato un software di analisi delle immagini privo di marcatori di vitalità per quantificare le metriche di risposta ai farmaci basate sul tasso di crescita che migliorano la riproducibilità e correggono le variazioni del tasso di crescita tra le diverse linee PDTO. Utilizzando la metrica normalizzata della risposta ai farmaci, che assegna un punteggio alla risposta al farmaco in base al tasso di crescita normalizzato a una condizione di controllo positiva e negativa, e un colorante di morte cellulare fluorescente, è possibile distinguere facilmente le risposte citotossiche e citostatiche ai farmaci, migliorando profondamente la classificazione dei responder e dei non responder. Inoltre, l'eterogeneità della risposta ai farmaci può essere quantificata dall'analisi della risposta farmacologica a singolo organoide per identificare potenziali cloni resistenti. In definitiva, questo metodo mira a migliorare la previsione della risposta clinica alla terapia catturando una firma di risposta farmacologica multiparametrica, che include l'arresto della crescita cinetica e la quantificazione della morte cellulare.
Introduzione
Negli ultimi anni, la scoperta di farmaci antitumorali in vitro , lo screening dei farmaci e la ricerca fondamentale sono passati dall'uso di tradizionali modelli di cancro bidimensionale (2D) con linee cellulari immortalizzate a modelli di cancro tridimensionali (3D) più fisiologicamente rilevanti. Ciò ha stimolato l'adozione di sferoidi tumorali con linee cellulari tumorali stabilite, che ricreano interazioni e strutture cellula-cellula più complesse presenti nei tumori solidi. Attualmente, gli organoidi tumorali derivati dal paziente (PDTO) sono il modello di cancro 3D più avanzato e fisiologicamente rilevante disponibile per la ricerca sul cancro in vitro, in quanto forniscono ulteriori vantaggi rispetto agli sferoidi tumorali, vale a dire l'eterogeneità riscontrata nei pazienti oncologici1. I PDTO sono stabiliti da tessuto tumorale proveniente da pazienti oncologici e quindi mantengono sia il fenotipo tumorale che il genotipo. In quanto tali, i PDTO stanno diventando preziosi per la ricerca fondamentale e traslazionale sul cancro e hanno il potenziale per migliorare notevolmente l'oncologia di precisione2.
Nonostante il loro potenziale promettente, questi sofisticati modelli 3D in vitro di cancro sono spesso sottoutilizzati a causa della mancanza di metodi di analisi avanzati. Il test più comunemente usato determina il numero di cellule vitali nel PDTO attraverso la quantificazione dell'ATP3 intracellulare. Questi test sono normalmente analisi di massa a singolo punto temporale, trascurando così le risposte critiche dipendenti dal tempo e trascurando le risposte clonali. In particolare, la capacità di monitorare la crescita dei PDTO (growth rate) e la loro risposta a specifiche terapie è di grande interesse 4,5. La risposta normalizzata al farmaco (NDR), che assegna un punteggio alla risposta al farmaco in base al tasso di crescita normalizzato a una condizione positiva (ctrl+) e di controllo negativo (ctrl-), è stata recentemente segnalata come una metrica cruciale per valutare la sensibilità ai farmaci antitumorali con lo screening basato sulle cellule, sebbene ciò sia stato fatto prevalentemente per le linee cellulari 2D6. Pertanto, sono necessari metodi di analisi più sofisticati per sfruttare appieno questi modelli di cancro 3D clinicamente più rappresentativi e complessi. La microscopia è considerata un approccio potente per studiare la complessità di questi modelli organoidi7.
Questo articolo descrive un metodo per monitorare le risposte cinetiche ai farmaci in modelli 3D di cancro, utilizzando microscopi convenzionali a campo largo e sistemi di imaging di cellule vive. Sono stati apportati adattamenti al protocollo descritto da Driehuis et al.4 per essere compatibili con l'automazione utilizzando un robot pipettaggio, un distributore digitale di farmaci e un sistema di imaging di cellule vive per aumentare la riproducibilità e ridurre il numero di ore di lavoro "pratiche". Questo metodo consente lo screening farmacologico a medio-alto rendimento sia degli sferoidi tumorali con linee cellulari tumorali consolidate (vedere la Tabella supplementare S1 per le linee cellulari testate), sia dei PDTO, in un formato micropiatto a 384 pozzetti e multiorganoide. Utilizzando un processo di apprendimento automatico di rete convoluzionale, l'identificazione e il tracciamento automatizzati di singoli sferoidi tumorali o PDTO potrebbero essere eseguiti esclusivamente dall'imaging in campo chiaro e senza l'uso di coloranti fluorescenti per l'etichettatura delle cellule vive8. Ciò è molto vantaggioso, poiché la maggior parte dell'identificazione con imaging in campo chiaro richiede l'annotazione manuale (che è laboriosa e richiede tempo) o richiede l'aggiunta di coloranti fluorescenti, che possono confondere le risposte farmacologiche correlate allo stress ossidativo indotto dalla fotoxicità9.
Il software di analisi delle immagini risultante sviluppato internamente estende la funzionalità dei sistemi di imaging a celle vive convenzionali, poiché i moduli di analisi delle immagini 3D non sono disponibili, limitati dalla piattaforma o non sono compatibili con le micropiastre a 384 pozzetti e l'imaging a pozzetto intero. Inoltre, questi moduli sono spesso costosi e offrono letture organoidi di massa limitate. Pertanto, questo metodo è molto rilevante per gli scienziati che hanno accesso a sistemi di imaging di cellule vive ampiamente disponibili e mirano a estrarre più informazioni su una risposta farmacologica rispetto al test basato sull'ATP standard ma rudimentale. Con l'aggiunta di specifici indicatori di morte cellulare, le risposte citostatiche ai farmaci possono essere distinte dalle risposte citotossiche, fornendo così ulteriori informazioni sulle azioni meccanicistiche dei farmaci attualmente irraggiungibili dall'analisi del singolo punto temporale. Infine, l'imaging delle cellule vive consente il tracciamento dei singoli organoidi per ottenere metriche di risposta ai singoli organoidi per catturare l'eterogeneità della risposta e identificare potenziali subcloni resistenti.
L'obiettivo di questo metodo e del software di analisi delle immagini associato è quello di implementare l'automazione a basso costo nello screening dei farmaci organoidi per limitare l'intervento dell'utente e ridurre la variabilità nella gestione, nell'analisi delle immagini e nell'analisi dei dati. Per rendere questo software disponibile ai ricercatori, è indipendente dal microscopio e dalla piattaforma e viene resa disponibile un'applicazione basata su cloud. Pertanto, supportando i sistemi di imaging di cellule vive convenzionali, miriamo anche a migliorare la loro funzionalità per applicazioni e analisi di coltura 3D.
Protocollo
Sono stati utilizzati organoidi derivati dal paziente con adenocarcinoma duttale pancreatico umano (PDAC). I frammenti di resezione tissutale sono stati ottenuti da pazienti sottoposti a chirurgia curativa presso l'ospedale universitario di Anversa. Il consenso informato scritto è stato ottenuto da tutti i pazienti e lo studio è stato approvato dal Comitato Etico UZA (rif. 14/47/480). I dettagli relativi a tutti i materiali, i reagenti, le apparecchiature e il software utilizzati in questo protocollo sono forniti nella tabella dei materiali. Una panoramica del flusso di lavoro è presentata nella Figura 1. Dati di esempio sono forniti nel materiale supplementare per riprodurre il protocollo.
1. Giorno 0: Preparazione di organoidi di 2 o 3 giorni
- Preriscaldare le micropiastre a 37 °C durante la notte e scongelare la matrice extracellulare (ECM) a 4 °C.
- Preparare il terreno di coltura organoide PDAC completo: supplemento ADF + ++ (Advanced DMEM / F12, integratore di glutammina all'1%, HEPES all'1% e penicillina / streptomicina all'1%, con 0,5 nM WNT surrogato-Fc-Fusione proteina, 4% Noggin-Fc Fusion Protein mezzo condizionato, 4% Rpso3-Fc Fusion Protein mezzo condizionato, 1x B27, 1 mM N-acetil cisteina (NAC), 5 mM nicotinamide, 500 nM A83-01, 100 ng / mL FGF10 e 10 nM Gastrina).
- Stabilire i PDTO secondo il metodo di scelta.
NOTA: Un protocollo dettagliato è fornito da Driehuis et al., che descrive il metodo convenzionale per stabilire, coltivare e far passare PDTO nelle cupole ECM4. - Dissociare enzimaticamente gli organoidi nelle cupole ECM.
- Aspirare il terreno e lavare 1 volta con soluzione salina tamponata fosfaticamente (PBS). Aggiungere l'enzima di dissociazione (ad esempio, 2 mL in una micropiastra a 6 pozzetti) e pipettare su e giù 10 volte con una pipetta da 1 mL per dissociare meccanicamente gli organoidi e le cupole ECM.
- Incubare per 10 minuti a 37 °C, pipettare su e giù e controllare se gli organoidi sono dissociati in singole cellule. Se necessario, ripetere questo passaggio.
- Raccogliere la sospensione cellulare in un tubo da 15 ml, aggiungere ADF+++ a un volume di 10 ml, centrifugare per 5 minuti a 450 × g a temperatura ambiente e aspirare il surnatante con una pipetta Pasteur e una pompa di aspirazione.
- Risospendere il pellet in 100-200 μL di mezzo pieno a seconda delle dimensioni del pellet e contare il numero di celle utilizzando il metodo scelto. Ad esempio: mescolare 10 μL della sospensione cellulare + 10 μL di Trypan Blue e contare con un contatore automatico delle cellule.
- Piastra a celle singole nelle cupole ECM.
- Diluire la sospensione cellulare e aggiungere 2/3 ECM secondo la Tabella 1. Pipettare fino a dieci goccioline da 20 μL per pozzetto in una piastra preriscaldata a 6 pozzetti. Capovolgere la piastra e incubare per 30 minuti a 37 °C.
- Sovrapposizione con mezzo pieno integrato con 10 μM Y-27632 e incubare per 2-3 giorni in un incubatore.
NOTA: Dieci cupole contenenti 75.000 celle ciascuna sono di solito sufficienti per riempire una micropiastra da 384 pozzetti ad una concentrazione di 200 organoidi / pozzetto, esclusi i pozzetti sul bordo.
2. Giorni 2 - 3: Raccolta e semina organoidi di 2 o 3 giorni
- Raccogli organoidi intatti dalle cupole ECM.
NOTA: Gli organoidi tendono ad aderire alle superfici di plastica (ad esempio, tubi, punte di pipette). Per evitare ciò, gli articoli in plastica possono essere prerisciacquati con una soluzione di albumina sierica bovina allo 0,1% (BSA)/PBS.- Aspirare il mezzo e lavare 1x con PBS. Aggiungere 1-2 ml di soluzione di raccolta organoide fredda (4 °C) in una piastra a 6 pozzetti a seconda del numero di cupole ECM e incubare su ghiaccio su una piattaforma di agitazione per 10 minuti.
- Pipettare su e giù con una pipetta da 1 mL per dissociare le cupole ECM, incubare per altri 10 minuti sul ghiaccio e controllare visivamente al microscopio se l'ECM è dissociato.
- Opzionale: se si preferisce una distribuzione dimensionale più uniforme, filtrare la sospensione attraverso un filtro cellulare da 70 μm prima della centrifugazione.
- Raccogliere gli organoidi in una provetta da 15 mL prerivestita con BSA/PBS allo 0,1%, aggiungere ADF+++ fino a 10 ml e centrifugare per 5 minuti a 200 × g a 4 °C. Aspirare il surnatante e risospendere il pellet in un massimo di 1.000 μL di mezzo organoide PDAC completo a seconda delle dimensioni del pellet per ottenere una concentrazione di >6.000 organoidi/ml.
- Contare gli organoidi utilizzando qualsiasi metodo di conteggio scelto, preferibilmente basato su immagini.
- Seminare gli organoidi.
NOTA: Vedere la tabella dei materiali per il volume minimo/pozzetto per due diversi tipi di micropiastre a 384 pozzetti utilizzati in questo protocollo.- Preraffreddare tutte le materie plastiche a -20 °C o su ghiaccio per almeno 20 minuti prima dell'uso per evitare la solidificazione dell'ECM.
- Preparare la soluzione di semina da 1 mL di soluzione madre di organoidi (fase 2.1.6) utilizzando terreno pieno per seminare ~200 organoidi per pozzetto in 50 μL, il volume minimo utilizzato per riempire un pozzetto. Utilizzare il file supplementare 1 per calcolare la quantità di soluzione di semina organoide. Aggiungere un volume residuo di 1.500 μL quando si utilizza un serbatoio da 25 ml e una pipetta multicanale o un robot di pipettaggio.
- Seminare gli organoidi utilizzando un robot di pipettaggio.
NOTA: Sia la soluzione di semina che la micropiastra devono essere raffreddate a 4 °C durante il pipettaggio per evitare la solidificazione dell'ECM. Pertanto, un serbatoio da 25 ml e un supporto per micropiastre sono stati stampati in 3D per essere utilizzati in combinazione con il robot di pipettaggio che può contenere elementi di raffreddamento elencati nella tabella dei materiali. Vengono forniti file STL per la stampa 3D del labware personalizzato (Supplementary File 2 e Supplementary File 3) e file JSON labware personalizzati per il robot di pipettaggio (Supplementary File 4 e Supplementary File 5).- Progettare il protocollo di erogazione utilizzando lo strumento di progettazione del protocollo online. Viene fornito un file JSON di esempio (file supplementare 6), in cui il labware personalizzato è già caricato e viene utilizzata una pipetta p300 (Gen2) a otto canali con le punte delle pipette corrispondenti.
- Apri l'app di controllo del robot di pipettaggio, seleziona i protocolli, fai clic su Importa e trascina e rilascia il file supplementare 6 nel campo designato.
- Selezionare il protocollo importato e posizionare tutti gli articoli da laboratorio, inclusi elementi di raffreddamento e oggetti in plastica, nei deck in base al layout mostrato nel campo Deck Setup . Utilizzare lo slot sinistro per il serbatoio da 25 mL e l'elemento di raffreddamento come mostrato nel file supplementare 7.
- Fare clic su Esegui protocollo e procedere all'installazione. Aprire la scheda Labware Setup, fare clic su Run Labware Position Check e seguire le istruzioni per calibrare il robot di pipettaggio sul nuovo hardware.
NOTA: i dati di offset Labware possono essere memorizzati per un secondo momento, ma si consiglia di eseguire il controllo della posizione del labware prima di ogni esecuzione. - Riempire il serbatoio da 25 mL (posto sopra l'elemento di raffreddamento) con la soluzione di semina organoide raffreddata e fare clic su Start Run.
NOTA: Anche i pozzetti superiore e inferiore si riempiono di soluzione di sospensione organoide a causa dell'uso della pipetta a otto canali. - Centrifugare la micropiastra per 1 min a 100 × g a 4 °C.
- Incubare a 37 °C per almeno 30 min.
- Riempire i pozzetti vuoti esterni con almeno 50 μL di H2O per evitare l'evaporazione.
- Incubare a 37 °C durante la notte per rimuovere eventuali bolle nel pozzetto che possono interferire con l'analisi dell'immagine.
3. Giorno 4: trattamento farmacologico e dispensazione di reagenti con distributore digitale di farmaci
- Creare il protocollo di erogazione del farmaco utilizzando il software di controllo del distributore digitale di droga.
- Passare il puntatore del mouse sulla piastra 1 sopra il layout della piastra, selezionare Modifica attributi piastra e compilare il tipo di piastra: pozzetto 384, volume aggiuntivo (μL): 50 e limite DMSO (%): 1.
- Aggiungi fluidi facendo clic sul pulsante + accanto a Fluidi. Fare doppio clic sul fluido appena creato e assegnargli un nome; selezionare la classe (basata su DMSO o acquosa + Tween 20) e concentrazione.
NOTA: Tutti i farmaci e i reagenti devono essere sciolti in DMSO al 100% o Tween-20 allo 0,3%. È possibile utilizzare una soluzione madre 1-10 mM, tenendo conto di una concentrazione massima di DMSO del <1%. La tabella 2 fornisce esempi delle diluizioni necessarie per i comuni reagenti fluorescenti e le terapie. - Layout della piastra
- Per la titolazione del farmaco, selezionare i pozzetti e fare clic su Titolazione. Per il fluido, selezionare il farmaco di interesse, scegliere la concentrazione più alta (ad esempio, 2.000 nM) e la concentrazione più bassa (ad esempio, 10 nM); Per le repliche, scegliete almeno 2 e scegliete il modello di titolazione desiderato.
NOTA: Il modello di titolazione dipenderà da molti fattori, tra cui la quantità di composto da inserire in una singola piastra, se i pozzetti devono essere randomizzati e il numero di repliche e controlli. - Per il controllo positivo, selezionare tre pozzetti, fare clic su Imposta valore e compilare 2 μM di staurosporina da 10 mM di stock in DMSO, che indurrà la massima morte cellulare.
- Per Cytotox Green, selezionare tutti i pozzetti utilizzati, fare clic su Imposta valore e immettere 60 nM/pozzetto.
NOTA: La colorazione fluorescente Cytotox Green indica le cellule che sono morte e quindi non interferirà con il monitoraggio della risposta ai farmaci. Qui, non è richiesto alcun marcatore fluorescente per le cellule vive. - Per il controllo negativo e la normalizzazione DMSO, selezionare tutti i pozzetti con altri quattro pozzetti per il controllo del veicolo, fare clic con il pulsante destro del mouse, selezionare normalizzazione, selezionare normalizzare la classe del fluido: basata su DMSO e normalizzare al volume di classe più alto per ottenere una concentrazione DMSO uguale in ciascun pozzetto.
NOTA: le concentrazioni di DMSO devono essere <1%. Viene fornito un esempio di file di titolazione del farmaco TDD (file supplementare 8). - Fare clic sulla freccia sotto Esegui nell'angolo in alto a sinistra, selezionare Simula sempre e fare clic su Simula per identificare eventuali errori e ottenere i volumi di ciascun farmaco da preparare.
NOTA: per superare un avviso quando il volume di erogazione iniziale è troppo basso, "Si consiglia un pozzetto di avviso di erogazione di 30 nL o superiore per ciascun fluido su ciascuna piastra", selezionare due pozzetti sul bordo riempiti d'acqua, selezionare Imposta valore e immettere 10 μM del farmaco per il quale si verifica l'avviso. Questo innesca la cartuccia del farmaco con un volume superiore a 30 nL. Questi stessi pozzetti possono essere utilizzati per innescare la cartuccia DMSO impostando una normalizzazione a % del valore del volume totale (ad esempio, 0,5%).
- Per la titolazione del farmaco, selezionare i pozzetti e fare clic su Titolazione. Per il fluido, selezionare il farmaco di interesse, scegliere la concentrazione più alta (ad esempio, 2.000 nM) e la concentrazione più bassa (ad esempio, 10 nM); Per le repliche, scegliete almeno 2 e scegliete il modello di titolazione desiderato.
- Deseleziona Simula sempre sotto il pulsante Esegui ; clicca su Esegui per avviare il protocollo di erogazione del farmaco e seguire le istruzioni.
- Applicare la membrana sigillante sulla micropiastra per evitare l'evaporazione.
- Incubare il colorante Cytotox Green 1-2 h a 37 °C nell'incubatore e procedere al punto 4.
4. Acquisisci immagini con l'imager live-cell
NOTA: Per il tasso di crescita e NDR, una scansione al punto temporale 0 (T0 = inizio trattamento) deve essere acquisita 1-2 ore dopo l'aggiunta di Cytotox Green.
- Aprire il software di controllo dell'imager a celle vive, selezionare Method Editor Nuovo, passare a File > importare e selezionare il file XML del metodo di esempio (file supplementare 9). In alternativa, creare un nuovo file e selezionare Piatto: (CORE384fb_OpticalImaging) - Corning 384 Flat Black (Corning #4588), Nessun coperchio e Nessuna cassetta di umidità; Applicazione: Solo immagini; Obiettivo: 4x; Modello: Centrale; Controllare i canali Brightfield e Green (intensità Led (%) = 40; Tempo di esposizione (ms) = 200).
NOTA: Le impostazioni del canale verde funzionano bene per una concentrazione di 60 nM Cytotox Green. L'opzione Live viewer può essere utilizzata per regolare l'offset della messa a fuoco e/o le impostazioni dei led in tempo reale. - Fare clic su Start per avviare la scansione a T0.
- Ripetere la scansione ogni 24 ore per un massimo di 5 giorni utilizzando lo stesso metodo. In alternativa, per eseguire automaticamente la misurazione del timelapse, regolare il metodo nel software di controllo dell'imager a celle vive su un esperimento cinetico facendo clic e trascinando la scheda Loop cinetico nel campo del metodo . Allo stesso modo, le schede Temperatura e Gas devono essere trascinate nel campo del metodo per impostare il sistema a 37 °C e 5% di CO2 per garantire le condizioni corrette all'interno dell'imager di cellule vive durante l'esperimento.
5. Analisi di immagini e dati
- Unione e compressione dei dati
- Il software live-cell imager Control genera una cartella per ogni scansione in ogni punto temporale. Crea una nuova cartella, copia le singole cartelle dell'esperimento in questa nuova cartella principale e aggiungi _0h, _24h, _48h, _72h, _96h e _120h ai nomi delle cartelle dell'esperimento corrispondenti.
- Preparare una mappa delle piastre XLSX dal software di controllo del distributore digitale di farmaci facendo clic con il pulsante destro del mouse sul layout della mappa delle piastre dal protocollo di erogazione del farmaco e copiare tutti i pozzetti; incollare i dati in un file XLSX. Rimuovere i dati di Cytotox Green e staurosporine e aggiungere una matrice per Cell Line e Replicate. Immettere ctrl- e ctrl+. Vedere il file supplementare 10 per un esempio di mappa delle lastre.
- Apri lo strumento di compressione dei dati, fai clic su Sfoglia, seleziona la cartella principale e fai clic su Esegui per avviare la compressione dei dati dell'immagine. Tutti i file di immagine TIFF per i diversi timepoint vengono compressi in un singolo HDF5 per ogni pozzetto in una nuova cartella dataset all'interno della cartella padre.
- Analisi delle immagini
- Vai alla piattaforma webapp di analisi delle immagini, accedi e fai clic su Aggiungi nuovo progetto nella scheda Home . Immettere il nome del progetto, continuare, selezionare Aggiungi nuovo esperimento e caricare la cartella datasets contenente i file HDF5.
- Dopo il caricamento, vai alla cartella del progetto e dell'esperimento e fai clic su Carica Platemap per ulteriori funzionalità. Fare clic su Esegui analisi, selezionare Analisi multiorganoide, Parametri predefiniti e fare clic su Analizza per avviare l'analisi dell'immagine.
- Fare clic su Scarica risultati per scaricare le tabelle dei dati grezzi che contengono le misurazioni per ciascun pozzetto (ad esempio, area totale del campo chiaro, area verde di fluorescenza totale, ecc.) e le immagini / video segmentati per confermare l'accuratezza dell'analisi e l'ulteriore elaborazione dei dati.
- Metriche di risposta ai farmaci basate sul tasso di crescita e risposta normalizzata ai farmaci
- Selezionare il file Raw_NDR.xlsx dalla cartella dei risultati (mappa targhe richiesta) (file supplementare 11) e caricarlo nel Official_NDR_7point R-script (file supplementare 12) per generare automaticamente tabelle dei valori GR (normalizzato a ctrl-) e NDR (normalizzato a ctrl- e ctrl+) (file supplementare 13, file supplementare 14, file supplementare 15 e file supplementare 16 ). I valori GR e NDR vengono calcolati dal parametro come mostrato nell'equazione (1) utilizzando lo script R (file supplementare 12).
Area di sopravvivenza totale = Area totale di campo libero - Area verde totale (1)
Dove 0 < NDR <1 = effetto citostatico (arresto della crescita) e NDR < 0 = risposta citotossica (morte cellulare).
NOTA: Lo script R è stato adattato da Gupta et al.6. - Dalla tabella clonal_data.xlsx, recuperare i dati di risposta a singolo organoide e tracciarli come grafico a bolle.
- Utilizzare il fattore Z10 per valutare la qualità dello schermo farmacologico di una corsa (vedere l'equazione (2)). Scartare un esperimento con un fattore Z < 0,5.
 (2)
(2)
- Selezionare il file Raw_NDR.xlsx dalla cartella dei risultati (mappa targhe richiesta) (file supplementare 11) e caricarlo nel Official_NDR_7point R-script (file supplementare 12) per generare automaticamente tabelle dei valori GR (normalizzato a ctrl-) e NDR (normalizzato a ctrl- e ctrl+) (file supplementare 13, file supplementare 14, file supplementare 15 e file supplementare 16 ). I valori GR e NDR vengono calcolati dal parametro come mostrato nell'equazione (1) utilizzando lo script R (file supplementare 12).
Risultati
Il protocollo di pipettaggio automatizzato garantisce una distribuzione uniforme dei PDTO PDAC_060 in tutte le colonne della micropiastra a 384 pozzetti (Figura 2A). Come previsto, è stata osservata una variazione nel numero e nell'area media dei PDTO tra i pozzi (Figura 2A,B). L'area di sopravvivenza totale (area totale in campo chiaro - area verde totale) combina la segmentazione organoide label-free con il segnale di morte cellulare basato sulla fluorescenza ed è, nella nostra esperienza, il parametro più robusto per studiare le risposte ai farmaci nel tempo (Figura 2C)8. Per tenere conto delle variazioni nella semina cellulare e delle dimensioni degli organoidi, è necessario utilizzare metriche basate sul tasso di crescita per ridurre le variazioni tra le repliche, come mostrato dalle barre di errore ridotte nella Figura 2D rispetto alla Figura 2C, e un fattore Z più alto che indica una qualità dello schermo del farmaco fortemente migliorata (Figura 2E).
La curva dose-risposta NDR (Figura 2G), normalizzata a ctrl- e ctrl+, è chiaramente superiore alla curva dose-risposta GR (Figura 2F), normalizzata a ctrl-, in quanto aumenta la separazione delle curve di risposta al farmaco e rappresenta più accuratamente le risposte citotossiche ai farmaci. Un esempio delle immagini associate per ctrl-, ctrl+ e PDTO trattato con paclitaxel da 400 nM/80 nM trattati con paclitaxel è mostrato nella Figura 3. Un'osservazione interessante è che l'effetto citotossico della gemcitabina è stato dominante nella terapia di associazione in quanto non è stato osservato alcun valore aggiunto del paclitaxel.
Successivamente, sono state utilizzate due linee PDTO aggiuntive, PDAC_052 e PDAC_087. È stata osservata una chiara differenza nel tasso di crescita tra queste linee (Figura 4A), che supporta l'uso di metriche GR. Ancora una volta, le curve dose-risposta NDR (Figura 4C) hanno determinato un aumento della gamma dinamica e della separazione tra i tre diversi pazienti rispetto alle curve GR (Figura 4B). Inoltre, il protocollo consente la determinazione del NDR nel tempo e mostra che PDAC_052 e PDAC_060 hanno avuto una risposta farmacologica citostatica molto simile a una bassa dose di gem-pac (Figura 4D), mentre una chiara risposta citostatica differenziale rispetto a quella citotossica potrebbe essere osservata per le dosi medie (Figura 4E) e alte (Figura 4F) di gem-pac. Queste risposte farmacologiche erano coerenti con le risposte cliniche osservate nei pazienti (Figura 4G).
Infine, uno dei principali vantaggi dell'approccio e del software è che le risposte ai farmaci a singolo organoide possono essere quantificate per studiare l'eterogeneità della risposta e identificare subcloni potenzialmente resistenti. La Figura 5 fornisce una chiara panoramica delle dinamiche clonali dei diversi pazienti e mostra che PDAC-087 aveva i subcloni più resistenti dopo il trattamento, il che è coerente con la malattia aggressiva e altamente resistente osservata nel paziente. È interessante notare che questo paziente era anche il meno sensibile alla staurosporina ctrl +.
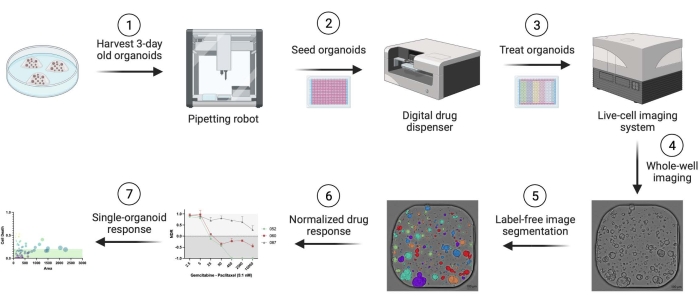
Figura 1: Panoramica del flusso di lavoro. Fare clic qui per visualizzare una versione ingrandita di questa figura.
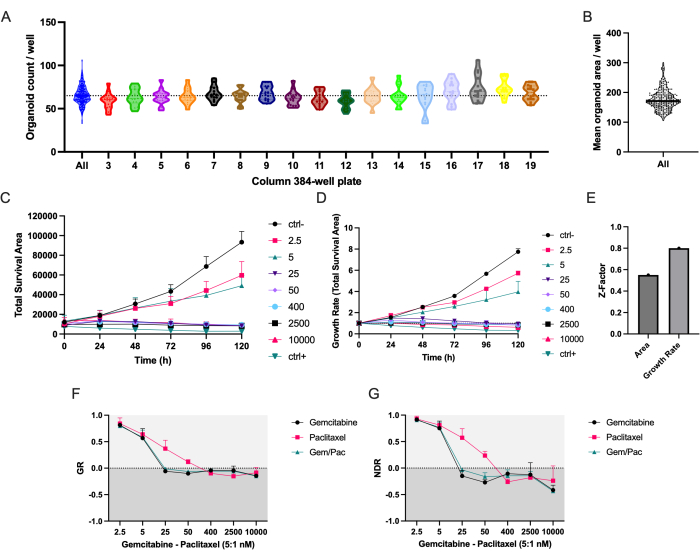
Figura 2: Accuratezza della semina e metriche di risposta ai farmaci . (A) Conta organoide/pozzetto di PDTO PDAC_060 seminati in una micropiastra da 384 pozzetti utilizzando il robot di pipettaggio. Ogni punto rappresenta il conteggio in un singolo pozzo e i grafici sono separati dalle colonne di micropiastre da 384 pozzetti. (B) Area PDTO media/pozzo. (C) Area di sopravvivenza totale (area totale in campo chiaro - area verde totale) e (D) tasso di crescita (area di sopravvivenza totale normalizzata a T0 = 1) di PDAC_060 PDTO trattati con un rapporto 5:1 di gemcitabina/paclitaxel. (E) Fattore Z come metrica per la qualità del test. (F) Curva tasso di crescita-dose risposta normalizzata a ctrl- e (G) curva normalizzata di risposta al farmaco normalizzata a ctrl- e ctrl+. Le barre di errore indicano ± SD media di due pozzi. Abbreviazioni: PDAC = adenocarcinoma duttale pancreatico; PDTO = organoide tumorale derivato dal paziente; GR = tasso di crescita; NDR = risposta normalizzata al farmaco. Fare clic qui per visualizzare una versione ingrandita di questa figura.
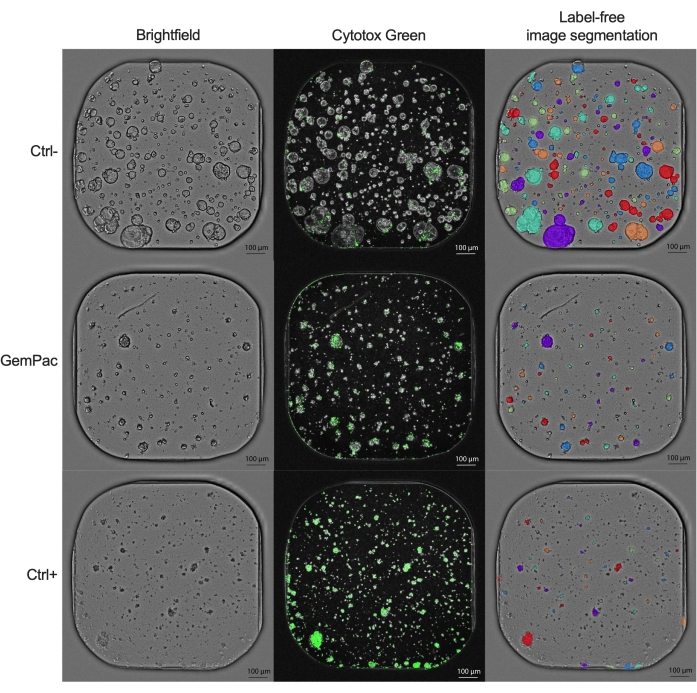
Figura 3: Immagini di esempio. Immagini rappresentative di PDAC_060 PDTO trattati con veicolo (ctrl-), 400 nM gemcitabina/80 nM paclitaxel e 2 μM staurosporina (ctrl+). La colonna di sinistra mostra le immagini in campo chiaro, la colonna centrale mostra il segnale fluorescente verde citotox e la colonna di destra mostra le immagini in campo chiaro annotate senza etichetta utilizzando il modulo di analisi degli organoidi. Barre della scala = 100 μm. Abbreviazioni: PDAC = adenocarcinoma duttale pancreatico; PDTO = organoide tumorale derivato dal paziente; GemPac = gemcitabina/paclitaxel. Fare clic qui per visualizzare una versione ingrandita di questa figura.
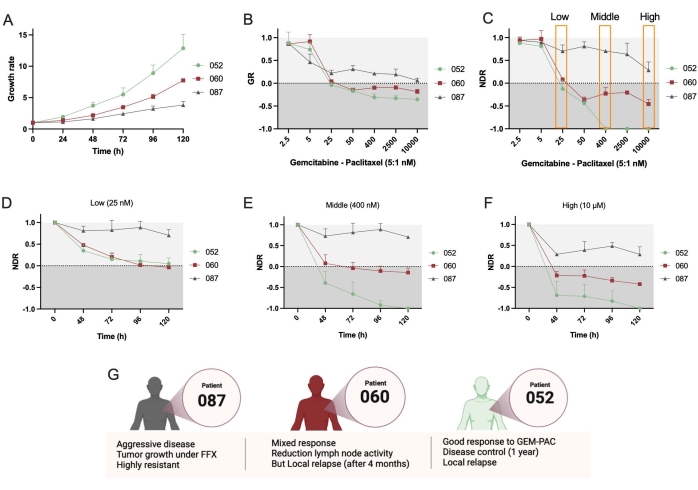
Figura 4: Confronto della risposta interpaziente ai farmaci . (A) Confronto del tasso di crescita (basato sull'area di sopravvivenza totale) delle linee PDTO PDAC_052, PDAC_060 e PDAC_087. (B) Curva tasso di crescita-dose risposta normalizzata a ctrl- e (C) curva di risposta al farmaco normalizzata a ctrl- e ctrl+. NDR cinetico di una dose (D) bassa, (E) media e (F) alta dose di gemcitabina/paclitaxel (rapporto 5:1). (G) Caratteristiche cliniche dei pazienti PDAC. Le barre di errore indicano ± SD media di due pozzi. Abbreviazioni: PDAC = adenocarcinoma duttale pancreatico; PDTO = organoide tumorale derivato dal paziente; GR = tasso di crescita; NDR = risposta normalizzata al farmaco; FFX = folfirinox. Fare clic qui per visualizzare una versione ingrandita di questa figura.
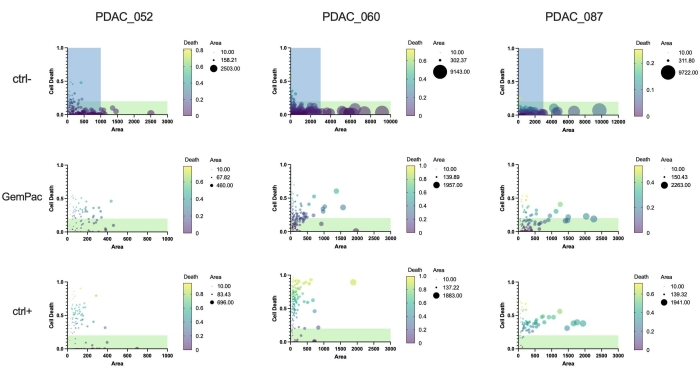
Figura 5: Metriche di organoidi singoli. Risposta alla dose di un singolo organoide basata sulla morte cellulare (area verde/area in campo chiaro) e area (campo chiaro) di PDTO PDAC_052, PDAC_060 e PDAC_087 trattati con veicolo (ctrl-), gemcitabina 400 nM/paclitaxel 80 nM e staurosporina 2 μM (ctrl+). Le regioni verdi indicano organoidi vitali; le regioni blu indicano l'intervallo x-as dei grafici GemPac e ctrl+. Abbreviazioni: PDAC = adenocarcinoma duttale pancreatico; PDTO = organoide tumorale derivato dal paziente; GemPac = gemcitabina/paclitaxel. Fare clic qui per visualizzare una versione ingrandita di questa figura.
| Stock di sospensione cellulare | Celle/Goccia | # Gocce (20 μL) | Stock (1/3) | ECM (2/3) |
| 1,13 × 107 cellule/ml | 75,000 | 10 | 75 uL | 150 μL |
| 1,13 × 107 cellule/ml | 75,000 | 5 | 40 uL | 80 μL |
Tabella 1: Diluizione per placcatura in cupole ECM. Abbreviazione: ECM = matrice extracellulare.
| Composto | Concentrazione delle scorte | Diluizione | Concentrazione di lavoro | Solvente | Concentrazione del pozzo | Commenti |
| Verde Cytotox | 1 mM (DMSO) | 1/10 | 10 μM | DMSO | 60 nM | Marcatore di morte cellulare |
| Cytotox rosso | 1 mM (DMSO) | 1/10 | 10 μM | DMSO | 250 nM | Marcatore di morte cellulare |
| Caspase 3/7 Verde | 5 mM (DMSO) | 1/2 | 2,5 mM | DMSO | 2,5 μM | Marcatore apoptotico |
| Hoechst | 20 mM (H2O) | 1/200 | 100 μM | 0,33% interpolazione/PBS | 50 nM | Marcatore nucleare |
| Staurosporina | 10 mM (DMSO) | / | 1 - 10 mM | / | 2 – 5 μM | Controllo positivo |
| Gemcitabina | 10 mM (DMSO) | / | 1 - 10 mM | / | Titolazione | Chemioterapia |
| Paclitaxel | 10 mM (DMSO) | / | 1 - 10 mM | / | Titolazione | Chemioterapia |
| Cisplatino | 5 mM (0,9% NaCl) | 1/2 | 2,5 mM | 0,6% Tween/PBS | Titolazione | Chemioterapia |
Tabella 2: Esempi di diluizioni di farmaci e reagenti fluorescenti usati frequentemente. Ogni composto deve essere sciolto in DMSO al 100% o Tween/PBS allo 0,3%.
Tabella supplementare S1: Panoramica delle linee cellulari tumorali compatibili. Statico: gli sferoidi non sono migratori. Unisci: gli sferoidi migrano l'uno verso l'altro e si fondono insieme. Clicca qui per scaricare questo file.
File supplementare 1: Strumento di calcolo della soluzione di semina degli organoidi. Clicca qui per scaricare questo file.
File supplementare 2: file STL per la stampa 3D di articoli da laboratorio personalizzati 'Microplate Holder'. Clicca qui per scaricare questo file.
File supplementare 3: file STL per la stampa 3D di articoli da laboratorio personalizzati "2 x 25 mL Reservoir Holder". Clicca qui per scaricare questo file.
File supplementare 4: file JSON per robot di pipettaggio labware personalizzato 'Microplate Holder'. Clicca qui per scaricare questo file.
File supplementare 5: file JSON per robot di pipettaggio labware personalizzato '2 x 25 mL Reservoir Holder_WithCooler'. Clicca qui per scaricare questo file.
File supplementare 6: file JSON per il protocollo robot di pipettaggio 'Plating_ PDO_384well_Cooled_Row2-23'. Clicca qui per scaricare questo file.
File supplementare 7: Panoramica della configurazione della scrivania del robot di pipettaggio. (A) Elementi di raffreddamento e (B) serbatoio e micropiastra. Clicca qui per scaricare questo file.
File supplementare 8: file TDD per il protocollo del distributore digitale di farmaci. Clicca qui per scaricare questo file.
Supplementary File 9: File XML per il protocollo dell'imager a cellule vive per l'imaging in campo chiaro e fluorescenza. Clicca qui per scaricare questo file.
File supplementare 10: Esempio di mappa delle targhe. Clicca qui per scaricare questo file.
File supplementare 11: file di input di esempio per lo script NDR R. Abbreviazione: NDR = risposta normalizzata al farmaco. Clicca qui per scaricare questo file.
File supplementare 12: Script NDR R di risposta ai farmaci normalizzato. Abbreviazione: NDR = risposta normalizzata al farmaco. Clicca qui per scaricare questo file.
File supplementare 13: file di output di esempio dei valori GR dello script NDR R. Abbreviazioni: GR = tasso di crescita; NDR = risposta normalizzata al farmaco. Clicca qui per scaricare questo file.
File supplementare 14: file di output di esempio dello script NDR R con valori GR trasposti. Abbreviazioni: GR = tasso di crescita; NDR = risposta normalizzata al farmaco. Clicca qui per scaricare questo file.
Filecompleto S 15: file di output di esempio di valori NDR di script NDR R. Abbreviazione: NDR = risposta normalizzata al farmaco. Clicca qui per scaricare questo file.
File supplementare 16: file di output di esempio di script NDR R con valori NDR trasposti. Abbreviazione: NDR = risposta normalizzata al farmaco. Clicca qui per scaricare questo file.
Discussione
Lo screening dei farmaci PDTO a medio-alto rendimento spesso si basa su letture che estraggono solo una frazione delle informazioni che gli organoidi potrebbero potenzialmente fornire. È diventato sempre più chiaro che, affinché la tecnologia degli organoidi in rapida evoluzione realizzi un maggiore potenziale scientifico e clinico, sono necessari saggi 3D più avanzati, letture e metodi di analisi. Qui viene descritta una pipeline di screening avanzata, che non solo aumenta la riproducibilità, ma migliora anche considerevolmente la traducibilità clinica incorporando una lettura di imaging di cellule vive guidata dall'intelligenza artificiale. Oltre al software di analisi sviluppato internamente, viene implementato l'uso della metrica di risposta ai farmaci normalizzata (NDR), che dimostra chiaramente la sua capacità di definire le differenze specifiche del paziente nella risposta al trattamento6.
L'inclusione di questa metrica di normalizzazione sarà senza dubbio di enorme valore, ricordando che numerosi studi mirano a delineare le risposte al trattamento sulla base di differenze minori nell'area sotto la curva (AUC) o nella concentrazione inibitoria semi-massima (IC50) (poiché la maggior parte delle curve dose-risposta si sovrappongono/si trovano vicine l'una all'altra)11,12 . Le metriche del tasso di crescita sono già state implementate nei protocolli di screening dei farmaci organoidi utilizzando il test basato su ATP, ma si basano sulla normalizzazione dei pozzi di riferimento lisati al punto temporale 04. Al contrario, questo metodo consente la normalizzazione del tasso di crescita intrawell, che non solo tiene conto delle differenze interpaziente nel tasso di crescita PDTO, ma anche delle differenze interwell derivanti da variazioni nella densità di semina e dagli effetti dipendenti dalla posizione della piastra per aumentare la riproducibilità. Inoltre, abbiamo adattato il rapporto di mancato recapito per aumentare ulteriormente la separazione della risposta PDTO interpaziente includendo un controllo positivo per la normalizzazione 6,8.
Inoltre, l'analisi, compatibile con formati ad alta produttività e automazione, è in grado di rilevare con precisione le risposte organoidi individuali, consentendo la quantificazione della resistenza subclonale, la principale forza trainante della recidiva e della progressione tumorale13. Ad esempio, sebbene PDAC052 e PDAC060 abbiano mostrato una buona risposta al trattamento in vitro (basata sul NDR), l'ulteriore analisi del singolo organoide è stata in grado di rilevare una piccola popolazione (popolazione più grande con PDAC060) di subcloni che non rispondono al trattamento. È interessante notare che ciò corrispondeva molto all'osservazione clinica, dato che PDAC052 e PDAC060 avevano una risposta duratura (nessuna attività tumorale rilevata) ma alla fine sono stati entrambi diagnosticati con progressione tumorale locale (a causa della presenza di cloni resistenti). Rispetto alle letture 3D convenzionali (test basati su ATP e dimensioni/numeri), questa pipeline di screening avanzato dovrebbe aumentare le prestazioni predittive estraendo più informazioni clinicamente rilevanti da questi "pazienti in laboratorio". Questa ipotesi è ora in fase di verifica mediante screening di campioni clinici PDTO nel laboratorio degli autori con questo metodo per correlare ex vivo con la risposta in vivo e l'esito clinico.
Per ottenere maggiori informazioni sui meccanismi di una risposta farmacologica, i reagenti di imaging fluorescenti fluorescenti per l'imaging di cellule vive, oltre ai coloranti di citotossicità, sono compatibili con questo metodo per studiare i meccanismi di morte cellulare. Abbiamo precedentemente dimostrato la compatibilità di questo metodo con il reagente Sartorius Caspase 3/7 Green per studiare l'induzione caspasi-dipendente dell'apoptosi dopo trattamento con cisplatino8. La compatibilità con altri coloranti per studiare lo stress ossidativo (reagenti CellROX) o l'ipossia (reagenti Image-iT Hypoxia) rimane da testare. Tuttavia, questi reagenti sono già stati utilizzati con successo in modelli 3D in vitro 14,15.
Il software di analisi delle immagini è anche compatibile con altri formati di lastre o metodi di coltura (ad esempio, piastre microcavità, cupole ECM) se è possibile acquisire immagini chiare e a fuoco degli organoidi. Questo è spesso difficile per gli organoidi coltivati in cupole poiché crescono in diversi piani z, il che richiede la funzionalità di impilamento z del microscopio che non è sempre disponibile. Pertanto, si consiglia l'uso di micropiastre ULA a fondo piatto a 384 pozzetti per garantire immagini di qualità sufficiente.
Inoltre, l'analisi è compatibile con altri sistemi di imaging di cellule vive, come mostrato in precedenza per le immagini a contrasto di fase acquisite con un sistema IncuCyte ZOOM8. Una limitazione del sistema di imaging delle cellule vive Spark Cyto che è stato utilizzato in questo manoscritto è la capacità di una piastra per le misurazioni cinetiche. Tuttavia, l'espansione Spark Motion aumenta la sua capacità fino a 40 micropiastre che possono essere schermate in blocco. La compatibilità del software sviluppato internamente sarà estesa a questi e ad altri sistemi per offrire una soluzione indipendente dalla piattaforma, con l'obiettivo di standardizzare e automatizzare le pipeline di analisi di immagini e dati. L'applicazione basata sul web includerà anche strumenti grafici interattivi e calcoli metrici automatizzati dei farmaci, come mostrato in questo documento, per ridurre i tempi di analisi manuale.
L'algoritmo di segmentazione PDTO label-free è stato addestrato e testato su vari modelli sferoidi e PDTO cresciuti internamente con differenze morfologiche distinte (solido, semi-solido, cistico) e può di conseguenza rilevarle con elevata precisione8. Una limitazione del modello è che l'inclusione di PDTO cistici ha aumentato il rilevamento indesiderato di bolle presenti nel pozzo dopo la semina. Tuttavia, l'incubazione notturna è stata sufficiente per rimuovere la maggior parte di queste bolle, consentendo una scansione qualitativa del timepoint 0. L'accuratezza della segmentazione dell'immagine organoide e il metodo devono essere convalidati da altri utenti e, in base al loro feedback, il software può essere ulteriormente addestrato per ottenere un algoritmo di analisi delle immagini robusto e automatizzato. Inoltre, miriamo a ottenere più dati clinici per correlare la risposta ex vivo al farmaco quantificata con questo metodo alla risposta clinica nel paziente per identificare i migliori parametri per prevedere la risposta alla terapia e sviluppare ulteriormente questo metodo per la medicina funzionale del cancro di precisione16.
Divulgazioni
Gli autori non dichiarano alcun conflitto di interessi.
Riconoscimenti
Parte di questa ricerca è stata finanziata da donazioni di diversi donatori, tra cui Dedert Schilde vzw e Willy Floren. Questo lavoro è parzialmente finanziato dalla Fondazione fiamminga per la ricerca, 12S9221N (A.L.), G044420N (S.V., A.L., E.G), 1S27021N (M.L), e dal Fondo di ricerca industriale dell'Università di Anversa, PS ID 45151 (S.V., A.L., C.D.). La Figura 1 è stata creata con BioRender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| 6-well plate | Greiner | 657160 | |
| 8-Channel p300 (GEN 2) pipette | Opentrons | ||
| 300 µL Tips | Opentrons | ||
| 384-well flat-bottom ULA microplate | Corning | 4588 | minimum volume 50 µL |
| 384-well flat-bottom ULA Phenoplate | Perkin Elmer | 6057802 | minimum volume 75 µL |
| A8301 | Tocris Bioscience | 2939 | |
| ADF+++ | Advanced DMEM/F12, 1% GlutaMAX, 1% HEPES, 1% penicillin/streptomycin | ||
| Advanced DMEM/F-12 | ThermoFisher Scientific | 12634 | |
| B27 | ThermoFisher Scientific | 17504044 | |
| Breathe easy sealing membrane | Sigma-Aldrich | Z380059 | |
| Caspase 3/7 Green | Sartorius | 4440 | |
| Cell Counting Slides for TC10/TC20 | Bio-Rad Laboratories | 1450017 | |
| CellTiter-Glo 3D | Promega | G9681 | ATP-assay |
| Cooler for 25 mL reservoir | VWR (Diversified Biotech) | 490006-908 | |
| Cooling element 12 x 8 x 3 cm | Bol.com | 9200000107744702 | For custom microplate holder OT-2 |
| Cultrex Organoid Harvesting Solution | R&D systems | 3700-100-01 | |
| Cultrex PathClear Reduced Growth Factor BME, Type 2 | R&D systems | 3533-010-02 | extracellular matrix (ECM) |
| Cytotox Green | Sartorius | 4633 | |
| Cytotox Red | Sartorius | 4632 | |
| D300e | Tecan | Digital drug dispenser | |
| D300e Control v3.3.5 | Tecan | Control software D300e | |
| FGF10 | Peprotech | 100-26 | |
| Full Medium | ADF+++ supplemented with 0.5 nM WNT surrogate-Fc-Fusion protein, 4% Noggin-Fc Fusion Protein conditioned medium, 4% Rpso3-Fc Fusion Protein conditioned medium, 1x B27, 1 mM N-acetyl cysteine (NAC), 5 mM nicotinamide, 500 nM A83-01, 100 ng/mL FGF10, and 10 nM Gastrin | ||
| Gastrin | Sigma-Aldrich | G9145 | |
| Gemcitabine | Selleck Chemicals | S1714 | |
| GlutaMAX | ThermoFisher Scientific | 35050 | |
| HEPES | ThermoFisher Scientific | 15630056 | |
| Hoechst 33342 Solution (20 mM) | ThermoFisher Scientific | 62259 | |
| Human pancreatic ductal adenocarcinoma (PDAC) patient-derived organoids | Biobank@uza (Antwerp, Belgium; ID: BE71030031000; Belgian Virtual Tumorbank funded by the National Cancer Plan) | ||
| N-acetyl-cysteine | Sigma-Aldrich | A9165-25G | |
| Nicotinamide | Sigma-Aldrich | N0636-100G | |
| Noggin-Fc Fusion Protein conditioned medium | Immunoprecise | N002 | |
| Opentrons App v6.0.1 | Opentrons | OT-2 control software | |
| Opentrons Protocol Designer Tool | Opentrons | https://designer.opentrons.com/ | |
| Orbits data compression tool | www.orbits-oncology.com or contact corresponding author | ||
| Orbits image analysis webapp | University of Antwerp | www.orbits-oncology.com or contact corresponding author | |
| OT-2 | Opentrons | Pipetting robot | |
| Paclitaxel | Selleck Chemicals | S1150 | |
| Pasteur Pipette 230 mm | Novolab | A33696 | |
| Peniciline-Streptomycin | ThermoFisher Scientific | 15140 | |
| Prism 9 | GraphPad | ||
| Rspo3-Fc Fusion Protein conditioned medium | Immunoprecise | N003 | |
| Spark Cyto 600 | Tecan | Live-cell imaging and multi-mode platereader | |
| SparkControl v3.1 | Tecan | Spark Cyto control software | |
| Staurosporine | Tocris Bioscience | 1285 | |
| Sterile 25 mL reservoir | VWR (Diversified Biotech) | 10141-922 | |
| T8 plus cassette | Tecan | ||
| TC20 | Bio-Rad Laboratories | automated cell counter | |
| TrypLE | ThermoFisher Scientific | 12604-021 | dissociation enzyme |
| Tween-20 | Acros Organics | 233360010 | |
| WNT Surrogate-Fc-Fusion protein | Immunoprecise | N001 | |
| Y-27632 | Selleck Chemicals | S1049 |
Riferimenti
- Drost, J., Clevers, H. Organoids in cancer research. Nature Reviews Cancer. 18 (7), 407-418 (2018).
- Veninga, V., Voest, E. E. Tumor organoids: Opportunities and challenges to guide precision medicine. Cancer Cell. 39 (9), 1190-1201 (2021).
- Le Compte, M., et al. Patient-derived organoids as individual patient models for chemoradiation response prediction in gastrointestinal malignancies. Critical Reviews in Oncology/Hematology. 157, 103190(2021).
- Driehuis, E., Kretzschmar, K., Clevers, H. Establishment of patient-derived cancer organoids for drug-screening applications. Nature Protocols. 15 (10), 3380-3409 (2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nature Methods. 13 (6), 521-527 (2016).
- Gupta, A., Gautam, P., Wennerberg, K., Aittokallio, T. A normalized drug response metric improves accuracy and consistency of anticancer drug sensitivity quantification in cell-based screening. Communications Biology. 3 (1), 42(2020).
- Rios, A. C., Clevers, H. Imaging organoids: a bright future ahead. Nature Methods. 15 (1), 24-26 (2018).
- Deben, C., Cardenas, E., et al. OrBITS: label-free and time-lapse monitoring of patient derived organoids for advanced drug screening. Cellular Oncology. , (2022).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. Bioessays. 39 (8), (2017).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Engel, R. M., et al. Patient-derived colorectal cancer organoids upregulate revival stem cell marker genes following chemotherapeutic treatment. Journal of Clinical Medicine. 9 (1), 128(2020).
- Hennig, A., et al. Detecting drug resistance in pancreatic cancer organoids guides optimized chemotherapy treatment. The Journal of Pathology. 257 (5), 607-619 (2022).
- D'Alterio, C., Scala, S., Sozzi, G., Roz, L., Bertolini, G. Paradoxical effects of chemotherapy on tumor relapse and metastasis promotion. Seminars in Cancer Biology. 60, 351-361 (2020).
- Njoroge, R. N., et al. Organoids model distinct Vitamin E effects at different stages of prostate cancer evolution. Scientific Reports. 7 (1), 16285(2017).
- Godet, I., et al. Fate-mapping post-hypoxic tumor cells reveals a ROS-resistant phenotype that promotes metastasis. Nature Communications. 10 (1), 4862(2019).
- Letai, A. Functional precision cancer medicine-moving beyond pure genomics. Nature Medicine. 23 (9), 1028-1035 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon