Method Article
バルクおよび単一オルガノイド分析のための広視野生細胞イメージングを用いたマルチパラメトリック腫瘍オルガノイド薬物スクリーニング(英語)
要約
このプロトコルは、中処理から高スループットのオルガノイド薬物スクリーニングのための半自動化された方法と、顕微鏡に依存しない自動画像分析ソフトウェアを説明し、マルチパラメトリックの単一オルガノイド薬物反応を定量化および視覚化して、腫瘍内の不均一性をキャプチャします。
要約
患者由来の腫瘍オルガノイド(PDTO)は、前臨床およびトランスレーショナル研究、および ex vivo 薬物スクリーニングからの患者の治療反応の予測に大きな期待を寄せています。ただし、現在のアデノシン三リン酸(ATP)ベースの薬物スクリーニングアッセイでは、バルク読み出しのためにPDTOに保持されることが示されている薬物反応(細胞増殖抑制または細胞毒性)および腫瘍内の不均一性の複雑さを捉えていません。生細胞イメージングは、この問題を克服し、薬物反応をより詳細に視覚化するための強力なツールです。ただし、画像解析ソフトウェアは、PDTOの3次元性に適合していないか、蛍光生存率色素を必要とするか、384ウェルマイクロプレートフォーマットと互換性がないことがよくあります。この論文では、従来の広視野生細胞イメージングシステムを使用して、ハイスループットの384ウェルフォーマットでPDTOをシード、処理、およびイメージングするための半自動化された方法論について説明します。さらに、生存率マーカーを使用しない画像解析ソフトウェアを開発し、再現性を向上させ、異なるPDTO株間の成長率の変動を修正する成長率ベースの薬物反応指標を定量化しました。陽性および陰性の対照条件に正規化された増殖速度に基づいて薬物応答をスコアリングする正規化薬物応答メトリックと蛍光細胞死色素を使用すると、細胞毒性および細胞増殖抑制薬物応答を簡単に区別でき、応答者と非応答者の分類を大幅に改善します。さらに、薬物応答の不均一性は、単一オルガノイド薬物応答分析から定量化することにより、潜在的な耐性クローンを特定することができます。最終的に、この方法は、速度論的成長停止および細胞死の定量化を含むマルチパラメトリック薬物応答シグネチャを捕捉することによって、臨床治療応答の予測を改善することを目的としている。
概要
近年、 in vitro がんの創薬、薬物スクリーニング、および基礎研究は、不死化細胞株を備えた従来の2次元(2D)がんモデルの使用から、より生理学的に関連する3次元(3D)がんモデルへの移行が進んでいます。これにより、固形腫瘍に存在するより複雑な細胞間相互作用および構造を再現する、確立された癌細胞株を有する腫瘍スフェロイドの採用に拍車がかかる。現在、患者由来の腫瘍オルガノイド(PDTO)は、腫瘍スフェロイドよりも追加の利点、つまりがん患者に見られる不均一性を提供するため、 in vitro がん研究に利用できる最も先進的で生理学的に関連性のある3Dがんモデルです1。PDTOは、がん患者に由来する腫瘍組織から確立されるため、腫瘍表現型と遺伝子型の両方を保持します。このように、PDTOは基礎がんおよびトランスレーショナルがん研究にとって非常に貴重になりつつあり、精密腫瘍学を大幅に改善する可能性を秘めています2。
有望な可能性にもかかわらず、これらの洗練された3Din vitroがんモデルは、高度な分析方法がないため、十分に活用されていないことがよくあります。最も一般的に使用されるアッセイは、細胞内ATP3の定量を介してPDTO中の生存細胞の数を決定する。これらのアッセイは通常、単一時点のバルク分析であるため、重要な時間依存応答を見落とし、クローン応答を無視します。具体的には、PDTOの成長(成長率)および特定の治療法に対するそれらの反応を監視する能力は、非常に興味深い4,5。陽性(ctrl+)および陰性対照(ctrl-)状態に正規化された成長率に基づいて薬物反応をスコアリングする正規化薬物反応(NDR)も、主に2D細胞株に対して行われていましたが、細胞ベースのスクリーニングで抗がん剤感受性を評価するための重要な指標であることが最近報告されています6。したがって、これらのより臨床的に代表的で複雑な3Dがんモデルを十分に活用するには、より高度な分析方法が必要です。顕微鏡検査は、これらのオルガノイドモデルの複雑さを研究するための強力なアプローチと考えられています7。
この論文では、従来の広視野顕微鏡と生細胞イメージングシステムを使用して、3Dがんモデルにおけるキネティックドラッグ応答をモニタリングする方法について説明します。Driehuisら4によって記述されたプロトコルは、ピペッティングロボット、デジタル薬剤ディスペンサー、および生細胞イメージングシステムを使用した自動化と互換性があり、再現性を高め、「実践的」労働時間の数を減らすように適応されました。この方法では、384ウェルマイクロプレートおよびマルチオルガノイドフォーマットで、確立された癌細胞株(試験細胞株については補足表S1を参照)を有する腫瘍スフェロイドとPDTOの両方の中スループットからハイスループットの薬物スクリーニングが可能になります。畳み込みネットワーク機械学習プロセスを使用することにより、蛍光生細胞標識色素を使用せずに、明視野イメージングのみから個々の腫瘍スフェロイドまたはPDTOの自動同定と追跡を実行できます8。明視野イメージングによるほとんどの同定は、手動アノテーションを必要とする(これは面倒で時間がかかる)か、蛍光色素の添加を必要とし、光キシシティ誘発酸化ストレスに関連する薬物応答を混乱させる可能性があるため、これは非常に有利です9。
自社で開発された画像解析ソフトウェアは、3D画像解析モジュールが利用できないか、プラットフォームが制限されているか、384ウェルマイクロプレートおよびホールウェルイメージングと互換性がないため、従来の生細胞イメージングシステムの機能を拡張します。さらに、これらのモジュールは多くの場合、高価格であり、バルクオルガノイドの読み出しが制限されています。したがって、この方法は、広く利用可能な生細胞イメージングシステムにアクセスでき、ゴールドスタンダードであるが初歩的なATPベースのアッセイと比較して、薬物反応に関するより多くの情報を抽出することを目指す科学者にとって非常に関連性があります。特定の細胞死指標を追加することで、細胞増殖抑制薬物応答を細胞傷害反応と区別することができ、単一時点解析では現在達成できない機構的な薬物作用に関するさらなる洞察を提供します。最後に、生細胞イメージングにより、個々のオルガノイド追跡により、単一のオルガノイド薬物応答メトリックを取得し、応答の不均一性をキャプチャし、潜在的な耐性サブクローンを特定できます。
この方法および関連する画像解析ソフトウェアの目標は、オルガノイド薬物スクリーニングにおいて低コストの自動化を実装して、ユーザーの介入を制限し、取り扱い、画像解析、およびデータ解析におけるばらつきを低減することである。このソフトウェアを研究者が利用できるようにするために、顕微鏡やプラットフォームに依存せず、クラウドベースのアプリケーションが利用可能になります。そのため、従来の生細胞イメージングシステムをサポートすることで、3D培養アプリケーションや解析への機能向上も目指しています。
プロトコル
ヒト膵管腺癌(PDAC)患者由来のオルガノイドを使用した。組織切除断片は、アントワープ大学病院で治癒手術を受けている患者から入手した。すべての患者から書面によるインフォームドコンセントが得られ、研究はUZA倫理委員会によって承認されました(参照14/47/480)。このプロトコルで使用されるすべての材料、試薬、機器、およびソフトウェアに関連する詳細は、 材料の表に記載されています。ワークフローの概要を 図 1 に示します。プロトコルを再現するための補足資料には、サンプルデータが記載されています。
1. 0日目:2日または3日齢のオルガノイドの調製
- マイクロプレートを37°Cで一晩予熱し、細胞外マトリックス(ECM)を4°Cで解凍します。
- 完全PDACオルガノイド培養培地を調製します:ADF+++(アドバンストDMEM/F12、1%グルタミンサプリメント、1%HEPES、および1%ペニシリン/ストレプトマイシン)に0.5 nM WNTサロゲート-Fc-融合タンパク質、4%ノギン-Fc融合タンパク質馴化培地、4%Rpso3-Fc融合タンパク質馴化培地、1x B27、1 mM N-アセチルシステイン(NAC)、5 mMニコチンアミド、500 nM A83-01、100 ng/mL FGF10、および10 nMガストリンを補充します。
- 選択した方法に従ってPDTOを確立します。
注:詳細なプロトコルはDriehuisらによって提供されており、ECMドーム4でPDTOを確立、培養、および通過するための従来の方法が説明されています。 - ECMドーム内のオルガノイドを酵素的に解離します。
- 培地を吸引し、リン酸緩衝生理食塩水(PBS)で1回洗浄します。解離酵素(例:6ウェルマイクロプレートに2 mL)を加え、1 mLピペットで10倍にピペットで上下にピペットして、オルガノイドとECMドームを機械的に解離させます。
- 37°Cで10分間インキュベートし、上下にピペットで動かし、オルガノイドが単一細胞に解離しているかどうかを確認します。必要に応じて、この手順を繰り返します。
- 細胞懸濁液を15 mLチューブに回収し、ADF+++を10 mLの容量に加え、室温で450 × g で5分間遠心分離し、パスツールピペットと吸引ポンプで上清を吸引します。
- ペレットのサイズに応じて、ペレットを100〜200 μLのフル培地に再懸濁し、選択した方法を使用して細胞数をカウントします。例:10 μLの細胞懸濁液+ 10 μLのトリパンブルーを混合し、自動セルカウンターでカウントします。
- ECMドーム内のシングルセルをプレート化します。
- 細胞懸濁液を希釈し、 表1に従って2/3 ECMを加える。予熱した6ウェルプレートに、ウェルあたり最大10個の20 μL液滴をピペットで入れます。プレートを反転させ、37°Cで30分間インキュベートします。
- 10 μM Y-27632を添加したフル培地を重ね合わせ、インキュベーター内で2〜3日間インキュベートします。
注:それぞれ75,000個の細胞を含む10個のドームは、通常、エッジのウェルを除いて、200オルガノイド/ウェルの濃度で1つの384ウェルマイクロプレートを満たすのに十分です。
2. 2〜3日目:生後2日または3日齢のオルガノイドを収穫して播種する
- ECMドームからインタクトなオルガノイドを収集します。
注:オルガノイドはプラスチック表面(チューブ、ピペットチップなど)に付着する傾向があります。これを避けるために、プラスチック製品は0.1%ウシ血清アルブミン(BSA)/ PBS溶液ですすぐことができます。- 培地を吸引し、PBSで1回洗浄します。ECMドームの数に応じて、1〜2 mLの冷たい(4°C)オルガノイド採取溶液を6ウェルプレートに加え、氷上で振とうプラットフォームで10分間インキュベートします。
- 1 mLピペットでピペットを上下に動かしてECMドームを解離し、氷上でさらに10分間インキュベートし、ECMが解離しているかどうかを顕微鏡で目視で確認します。
- オプション:より均一なサイズ分布が望ましい場合は、遠心分離前に70 μmのセルストレーナーで懸濁液をろ過します。
- 0.1% BSA/PBSでプレコートした15 mLチューブにオルガノイドを回収し、ADF+++を10 mLまで加え、4°Cで200 × g で5分間遠心分離します。 上清を吸引し、ペレットのサイズに応じて最大1,000 μLのフルPDACオルガノイド培地にペレットを再懸濁して、>6,000オルガノイド/mLの濃度を得ます。
- オルガノイドを計数は、任意の計数方法、好ましくは画像ベースのものを用いて行う。
- オルガノイドを播種します。
注:このプロトコルで使用される2つの異なるタイプの384ウェルマイクロプレートの最小容量/ウェルについては、 材料表 を参照してください。- ECMの固化を防ぐために、使用前にすべてのプラスチック製品を-20°Cまたは氷上で少なくとも20分間予冷します。
- フル培地を使用して1 mLのオルガノイドストック溶液から播種液を調製し(ステップ2.1.6)、ウェルに充填するために使用した最小容量である50 μLでウェルあたり~200個のオルガノイドを播種します。 補足ファイル1 を使用して、オルガノイド播種液の量を計算します。25 mLリザーバーとマルチチャンネルピペットまたはピペッティングロボットを使用する場合は、1,500 μLの残量を追加します。
- ピペッティングロボットを使用してオルガノイドを播種します。
注:ECMの固化を避けるために、ピペッティング中はシード溶液とマイクロプレートの両方を4°Cで冷却する必要があります。そのため、25 mLのリザーバーとマイクロプレートホルダーを3Dプリントして、材料表に記載されている冷却要素を保持できるピペッティングロボットと組み合わせて使用 しました。カスタムラボウェアを3DプリントするためのSTLファイル(補足ファイル2および補足ファイル3)と、ピペッティングロボット用のカスタムラボウェアJSONファイル(補足ファイル4および補足ファイル5)が提供されます。- オンラインのプロトコル設計者ツールを使用して、ディスペンシングプロトコルを設計します。サンプルのJSONファイル(補足ファイル6)では、カスタムラボウェアがすでにロードされており、対応するピペットチップを備えた8チャンネルp300(Gen2)ピペットが使用されています。
- ピペッティングロボット制御アプリを開き、 プロトコルを選択し、 インポートをクリックして、 補足ファイル6 を指定されたフィールドにドラッグアンドドロップします。
- インポートしたプロトコルを選択し、冷却エレメントやプラスチック製品を含むすべてのラボウェアを、[デッキ設定]フィールドに示されているレイアウトに従って デッキ に配置します。補足 ファイル7に示すように、左側のスロットを25 mLリザーバーと冷却エレメントに使用します。
- [プロトコルの実行]をクリックして、セットアップに進みます。[ラボウェアのセットアップ]タブを開き、[ラボウェアの位置チェックを実行]をクリックして、指示に従ってピペッティングロボットを新しいハードウェアに合わせて校正します。
メモ: ラボウェアのオフセットデータは後で使用するために保存できますが、各実行の前にラボウェアの位置チェックを実行することをお勧めします。 - 冷却エレメントの上に置かれた25 mLリザーバーに冷却されたオルガノイド播種溶液を満たし、[ 実行の開始]をクリックします。
注:上部と下部のウェルも、8チャンネルピペットの使用によりオルガノイド懸濁液で満たされます。 - マイクロプレートを4°Cで100 × g で1分間遠心分離します。
- 37°Cで少なくとも30分間インキュベートします。
- 蒸発を避けるために、外側のブランクウェルに少なくとも50 μLのH2Oを充填します。
- 37°Cで一晩インキュベートし、画像解析を妨げる可能性のあるウェル内の気泡を除去します。
3. 4日目:デジタルドラッグディスペンサーによる薬物治療と試薬調剤
- デジタル薬剤ディスペンサー制御ソフトウェアを使用して、薬剤調剤プロトコルを作成します。
- プレートレイアウトの上にあるプレート1にカーソルを合わせ、[プレート属性の編集]を選択して、プレートタイプ:384ウェル、追加容量(μL):50、DMSO制限(%):1を入力します。
- 流体を追加するには、[流体]の横にある[+]ボタンをクリックします。新しく作成した流体をダブルクリックして名前を付けます。クラス(DMSOベースまたは水系+トゥイーン20)と濃度を選択します。
注:すべての薬剤と試薬は、100%DMSOまたは0.3%Tween-20に溶解する必要があります。最大DMSO濃度<1%を考慮して、1〜10 mMのストック溶液を使用できます。 表2 は、一般的な蛍光試薬および治療法に必要な希釈の例を示しています。 - プレートレイアウト
- 薬物滴定の場合は、ウェルを選択し、滴 定をクリックします。 液体の場合、 目的の薬物を選択し、 最高 濃度(例:2,000 nM)と 最低濃度 (例:10 nM)を選択します。 反復の場合は、 最小2を選択し、目的の 滴定パターンを選択します。
注:滴定パターンは、1つのプレートに収まる化合物の量、ウェルをランダム化するかどうか、反復とコントロールの数など、多くの要因によって異なります。 - ポジティブコントロールの場合、3つのウェルを選択し、[値の設定]をクリックして、DMSOの10 mMストックから2 μMのスタウロスポリンを充填すると、最大の細胞死が誘導されます。
- サイトトックスグリーンの場合は、使用済みのウェルをすべて選択し、 値の設定をクリックして、60 nM/ウェルと入力します。
注:Cytotox Green蛍光染色は、死滅した細胞を示すため、薬物反応モニタリングを妨げません。ここでは、生細胞用の蛍光マーカーは必要ありません。 - ネガティブコントロールとDMSO正規化では、ビヒクルコントロール用にさらに4つのウェルを含むすべてのウェルを選択し、右クリックして 正規化を選択し、流体クラスの正規化:DMSOベースを選択し、最高クラスの体積に正規化して、各ウェルで等しい DMSO濃度を取得します。
注:DMSO濃度は<1%である必要があります。TDD薬物滴定ファイル(補足ファイル8)の例が提供されています。 - 左上隅の[実行]の下の矢印をクリックし、[常にシミュレート]を選択し、[シミュレート]をクリックしてエラーを特定し、準備する各薬剤の量を取得します。
注意: 最初の分注量が少なすぎる場合の警告を克服するには、「各プレートの各液に30 nL以上の分注警告ウェルをお勧めします」、水で満たされている端の2つのウェルを選択し、[ 値の設定]を選択して、警告が発生する薬剤 の10μM を入力します。これにより、30 nLを超える容量の薬剤カートリッジがプライミングされます。これらの同じウェルを使用して、 正規化を総体積値の% に設定することにより、DMSOカートリッジをプライミングできます(たとえば、0.5%)。
- 薬物滴定の場合は、ウェルを選択し、滴 定をクリックします。 液体の場合、 目的の薬物を選択し、 最高 濃度(例:2,000 nM)と 最低濃度 (例:10 nM)を選択します。 反復の場合は、 最小2を選択し、目的の 滴定パターンを選択します。
- [実行]ボタンの下にある[常にシミュレート]のチェックを外します。[実行]をクリックして薬剤調剤プロトコルを開始し、指示に従います。
- シーリングメンブレンをマイクロプレートに適用して、蒸発を防ぎます。
- Cytotox Green色素をインキュベーター内で37°Cで1〜2時間インキュベートし、ステップ4に進みます。
4. 生細胞イメージャで画像を取得
注:成長率とNDRについては、サイトトックスグリーンを追加してから1〜2時間後にタイムポイント0(T0 =治療開始)でのスキャンを取得する必要があります。
- ライブセルイメージャー制御ソフトウェアを開き、 メソッドエディターの新規を選択し、 インポート>ファイルに移動して、 サンプルメソッドXMLファイル (補足ファイル9)を選択します。または、新しいファイルを作成して プレート: (CORE384fb_OpticalImaging) - コーニング 384 フラットブラック (コーニング #4588)、蓋なし、湿度カセットなし。アプリケーション:画像のみ。目標: 4x;パターン: 中央;チャネルの明視野と緑を確認します(LED強度(%)= 40;露光時間(ミリ秒)= 200)。
注意: グリーンチャンネル設定は、60 nMのサイトトックスグリーンの濃度に適しています。 ライブビューア オプションを使用して、フォーカスオフセットやLED設定をリアルタイムで調整できます。 - [ 開始 ]をクリックして、T0からスキャンを開始します。
- 同じ方法を使用して、最大24時間ごとに最大5日間スキャンを繰り返します。または、タイムラプス測定を自動的に実行するには、ライブセルイメージャー制御ソフトウェアで、キネティックループタブをクリックしてメソッドフィールドにドラッグすることにより、メソッドをキネティック実験に調整します。同様に、温度タブとガスタブをメソッドフィールドにドラッグして、システムを37°Cと5%CO2に設定し、実験中に生細胞イメージャー内の正しい条件を確保する必要があります。
5.画像とデータの分析
- データのマージと圧縮
- ライブセルイメージャ制御ソフトウェアは、各時点でスキャンごとにフォルダを生成します。新しいフォルダーを作成し、個々の実験フォルダーをこの新しい親フォルダーにコピーして、対応する実験フォルダー名に_0h、_24h、_48h、_72h、_96h、_120hを追加します。
- デジタル薬剤ディスペンサー制御ソフトウェアからXLSXプレートマップを準備し、薬剤調剤プロトコルから プレートマップ レイアウトを右クリックし、すべてのウェルをコピーします。XLSX ファイルにデータを貼り付けます。サイトトックスグリーンとスタウロスポリンのデータを削除し、細胞株と複製のマトリックスを追加します。 ctrl- と ctrl+ を入力します。プレートマップの例については、 補足ファイル10 を参照してください。
- データ圧縮ツールを開き、[参照]をクリックして親フォルダーを選択し、[実行]をクリックして画像データ圧縮を開始します。異なるタイムポイントのすべてのTIFF画像ファイルは、親フォルダ内の新しいデータセットフォルダ内のウェルごとに1つのHDF5に圧縮されます。
- 画像解析
- 画像解析 Web アプリ プラットフォームに移動してログインし、[ホーム] タブの [新しいプロジェクトの追加] をクリックします。プロジェクト名を入力して続行し、 [新しい実験の追加] を選択して、HDF5 ファイルを含むデータセット フォルダーをアップロードします。
- アップロード後、 プロジェクト と 実験のフォルダ に移動し、 Platemapのアップロード をクリックして追加機能を表示します。[分析 の実行]をクリックし、[ マルチオルガノイド分析]、[ デフォルトパラメータ]を選択し、[分析]をクリックして画像 分析 を開始します。
- [ 結果のダウンロード ]をクリックして、各ウェルの測定値(総明視野面積、全蛍光緑域など)とセグメント化された画像/ビデオを含む生データテーブルをダウンロードして、分析の精度とさらなるデータ処理を確認します。
- 成長率ベースの薬物反応指標と正規化された薬物反応
- 結果フォルダ(プレートマップが必要)(補足ファイル11)からRaw_NDR.xlsxファイルを選択し、これをOfficial_NDR_7point Rスクリプト(補足ファイル12)にロードして、GR(ctrl-に正規化)およびNDR(ctrl-およびctrl+に正規化)値テーブル(補足ファイル13、補足ファイル14、補足ファイル15、補足ファイル16)を自動的に生成します。).GR値とNDR値は、式(1)に示すように、Rスクリプト(補足ファイル12)を使用してパラメータから計算されます。
総生存面積=全明視野面積-総緑地面積(1)
ここで、0 < NDR <1 = 細胞増殖抑制効果 (増殖停止)、および NDR < 0 = 細胞傷害応答 (細胞死) です。
注:RスクリプトはGuptaら6から適応されました。 - clonal_data.xlsx表から、単一オルガノイド応答データを取得し、バブルプロットとしてプロットします。
- Zファクター10 を使用して、ランの薬物スクリーニングの品質を評価します(式(2)を参照)。Z 係数が 0.5 <実験を破棄します。
 (2)
(2)
- 結果フォルダ(プレートマップが必要)(補足ファイル11)からRaw_NDR.xlsxファイルを選択し、これをOfficial_NDR_7point Rスクリプト(補足ファイル12)にロードして、GR(ctrl-に正規化)およびNDR(ctrl-およびctrl+に正規化)値テーブル(補足ファイル13、補足ファイル14、補足ファイル15、補足ファイル16)を自動的に生成します。).GR値とNDR値は、式(1)に示すように、Rスクリプト(補足ファイル12)を使用してパラメータから計算されます。
結果
自動ピペッティングプロトコルにより、384ウェルマイクロプレートのすべてのカラムにPDAC_060 PDTOが均一に分布します(図2A)。予想通り、PDTOの数と平均面積にウェル間でばらつきが観察されました(図2A、B)。全生存面積(全明視野面積 - 全緑面積)は、ラベルフリーオルガノイドセグメンテーションと蛍光ベースの細胞死シグナルを組み合わせたもので、私たちの経験では、経時的な薬物反応を研究するための最も堅牢なパラメータです(図2C)8。細胞播種とオルガノイドサイズのばらつきを説明するには、図2Dと図2Cのエラーバーの減少、および薬物スクリーニングの品質が大幅に改善されたことを示すZファクターが高いことからわかるように、増殖速度ベースの測定基準を使用して反復間の変動を減らす必要があります(図2E)。
ctrl-およびctrl+に正規化されたNDR用量反応曲線(図2G)は、薬物反応曲線の分離を増加させ、細胞傷害性薬物反応をより正確に表すため、ctrl-に正規化されたGR用量反応曲線(図2F)よりも明らかに優れています。ctrl-、ctrl+、および400 nMゲムシタビン/80 nMパクリタキセルで処理されたPDTOの関連画像の例を 図3に示します。興味深い観察結果は、パクリタキセルの付加価値が観察されなかったため、ゲムシタビンの細胞毒性効果が併用療法で支配的であったことです。
次に、2つの追加のPDTOライン、PDAC_052とPDAC_087を使用しました。これらの線の間には明らかな成長率の違いが観察され(図4A)、これはGRメトリックの使用を支持しています。繰り返しになりますが、NDR用量反応曲線(図4C)は、GR曲線(図4B)と比較して、3人の異なる患者間のダイナミックレンジと分離の増加をもたらしました。さらに、このプロトコルは、時間の経過に伴うNDRの決定を可能にし、PDAC_052とPDAC_060が低用量のgem-pacに対して非常に類似した細胞増殖抑制薬物反応を示したことを示しています(図4D)、gem-pacの中間用量(図4E)と高用量(図4F)では、細胞増殖抑制反応と細胞毒性反応の明確な差が観察されました。これらの薬物反応は、患者で観察された臨床反応と一致していました(図4G)。
最後に、このアプローチとソフトウェアの主な利点は、単一オルガノイド薬物応答を定量化して、応答の不均一性を研究し、潜在的に耐性のあるサブクローンを特定できることです。 図5 は、異なる患者のクローン動態の明確な概要を提供し、PDAC-087が治療後に最も耐性のあるサブクローンを有することを示しており、これは患者で観察された攻撃的で耐性の高い疾患と一致している。興味深いことに、この患者はctrl +スタウロスポリンに対して最も感受性が低かった。
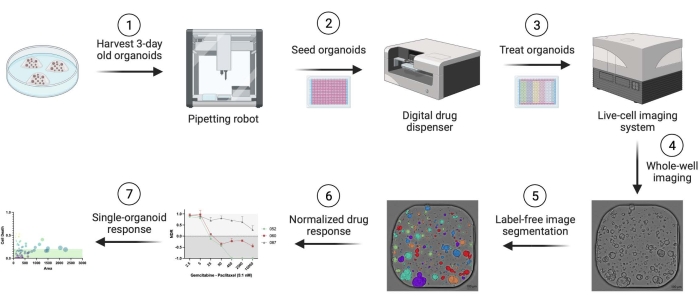
図 1: ワークフローの概要。 この図の拡大版を表示するには、ここをクリックしてください。
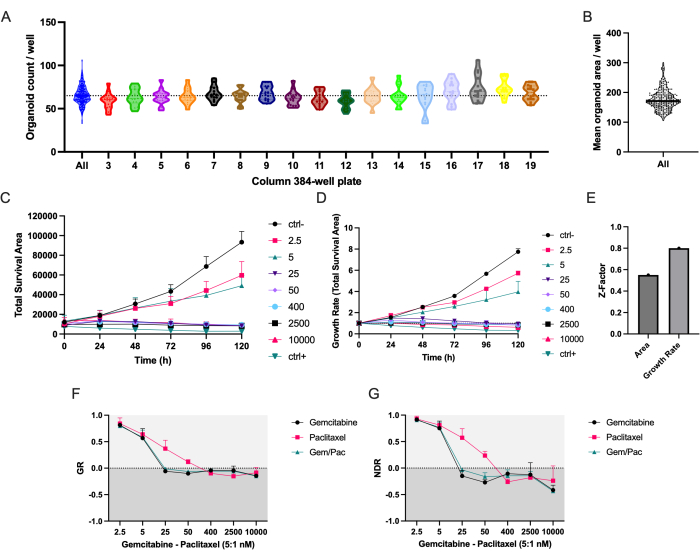
図2:播種精度と薬物反応指標 。 (A)ピペッティングロボットを使用して384ウェルマイクロプレートに播種したPDAC_060 PDTOのオルガノイド数/ウェル。各ドットは単一のウェル内のカウントを表し、プロットは384ウェルマイクロプレートカラムで区切られています。(B)平均PDTO面積/井戸。(C)ゲムシタビン/パクリタキセルの比率が5:1で処理されたPDAC_060 PDTOの総生存面積(全明視野面積-全緑面積)および(D)成長率(T0 = 1に正規化された全生存面積)。(E)アッセイ品質の指標としてのZファクター。(F)ctrl-に正規化された成長率-用量反応曲線、および(G)ctrl-およびctrl+に正規化された正規化された薬物反応曲線。エラーバーは±2つのウェルの平均SDを示します。略語:PDAC =膵管腺癌;PDTO =患者由来の腫瘍オルガノイド;GR =成長率;NDR =正規化された薬物反応。 この図の拡大版を表示するには、ここをクリックしてください。
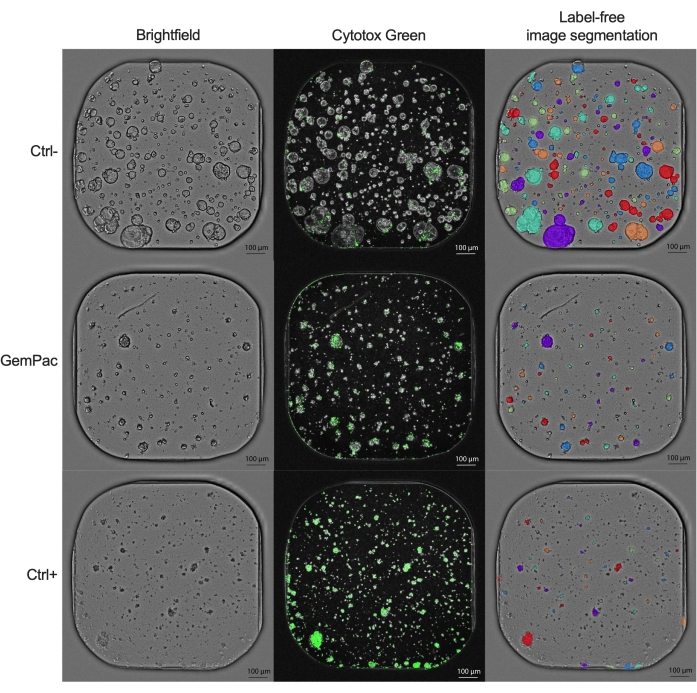
図3:画像の例。 ビヒクル(ctrl-)、400 nMゲムシタビン/80 nMパクリタキセル、および2 μMスタウロスポリン(ctrl+)で処理したPDAC_060 PDTOの代表的な画像。左の列は明視野画像、中央の列はCytotox Green蛍光シグナル、右の列はオルガノイド分析モジュールを使用したラベルフリーの注釈付き明視野画像を示しています。スケールバー= 100μm。略語:PDAC =膵管腺癌;PDTO =患者由来の腫瘍オルガノイド;GemPac = ゲムシタビン/パクリタキセル。 この図の拡大版を表示するには、ここをクリックしてください。
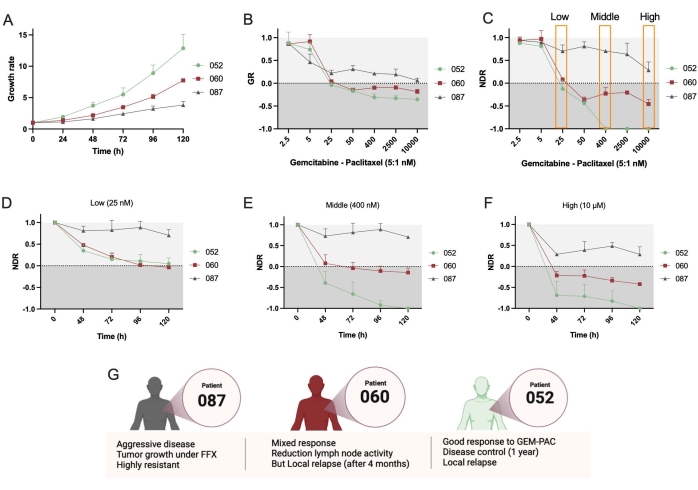
図4:患者間の薬物反応の比較 。 (A)PDAC_052、PDAC_060、およびPDAC_087 PDTO株の成長率(全生存面積に基づく)の比較。(B)ctrl-に正規化された成長率-用量反応曲線、および(C)ctrl-およびctrl+に正規化された正規化された薬物反応曲線。(D)低用量、(E)中用量、および(F)高用量のゲムシタビン/パクリタキセル(5:1比)の速度論的NDR。(G)PDAC患者の臨床的特徴。エラーバーは±2つのウェルの平均SDを示します。略語:PDAC =膵管腺癌;PDTO =患者由来の腫瘍オルガノイド;GR =成長率;NDR =正規化された薬物反応;FFX =フォルフィリノックス。 この図の拡大版を表示するには、ここをクリックしてください。
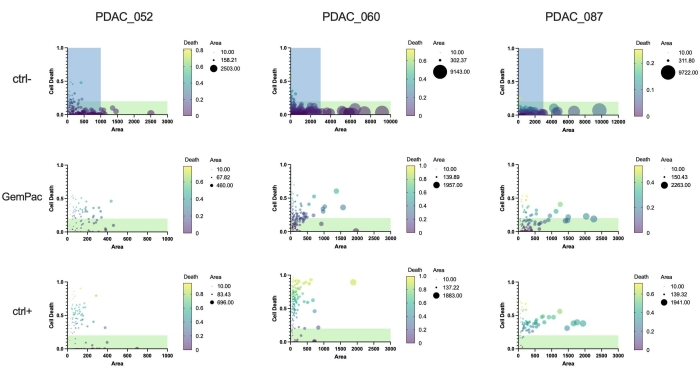
図5:単一オルガノイド測定基準。 ビヒクル(ctrl-)、400 nMゲムシタビン/80 nMパクリタキセル、および2 μMスタウロスポリン(ctrl+)で処理されたPDAC_052、PDAC_060、およびPDAC_087 PDTOの細胞死(緑領域/明視野領域)および面積(明視野)に基づく単一オルガノイド用量反応。緑色の領域は生存可能なオルガノイドを示します。青色の領域は、GemPac および ctrl+ プロットの x-as 範囲を示します。略語:PDAC =膵管腺癌;PDTO =患者由来の腫瘍オルガノイド;GemPac = ゲムシタビン/パクリタキセル。 この図の拡大版を表示するには、ここをクリックしてください。
| セル懸濁ストック | セル/ドロップ | # 滴 (20 μL) | 在庫 (1/3) | ECM (2/3) |
| 1.13 × 107 細胞/mL | 75,000 | 10 | 75 uL | 150 μL |
| 1.13 × 107 細胞/mL | 75,000 | 5 | 40 uL | 80 μL |
表1:ECMドームのめっき用希釈。 略語:ECM =細胞外マトリックス。
| 化合物 | 在庫集中 | 希釈 | 作業濃度 | 溶媒 | 井戸濃度 | コメント |
| サイトトックスグリーン | 1 ミリメートル (DMSO) | 1/10 | 10 μM | ティッカー | 60ナノメートル | 細胞死マーカー |
| サイトトックスレッド | 1 ミリメートル (DMSO) | 1/10 | 10 μM | ティッカー | 250 nM | 細胞死マーカー |
| カスパーゼ3/7グリーン | 5 mM (DMSO) | 1/2 | 2.5 ミリリットル | ティッカー | 2.5 μM | アポトーシスマーカー |
| ヘキスト | 20 mM (H2O) | 1/200 | 100マイクロM | 0.33% トゥイーン/PBS | 50ナノメートル | 核マーカー |
| スタウロスポリン | 10 mM (DMSO) | / | 1 - 10 mM | / | 2 – 5 μM | ポジティブコントロール |
| ゲムシタビン | 10 mM (DMSO) | / | 1 - 10 mM | / | 滴 定 | 化学療法 |
| パクリタキセル | 10 mM (DMSO) | / | 1 - 10 mM | / | 滴 定 | 化学療法 |
| シスプラチン | 5 mM (0.9% NaCl) | 1/2 | 2.5 ミリリットル | 0.6% トゥイーン/PBS | 滴 定 | 化学療法 |
表2:よく使われる薬剤や蛍光試薬の希釈例。 各化合物は、100%DMSOまたは0.3%トゥイーン/PBSのいずれかに溶解する必要があります。
補足表S1:適合がん細胞株の概要。 静的:回転楕円体は渡り鳥ではありません。マージ:回転楕円体は互いに向かって移動し、一緒にマージします。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル1:オルガノイド播種溶液計算ツール。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル2:カスタムラボウェア「マイクロプレートホルダー」を3DプリントするためのSTLファイル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル3:カスタムラボウェア「2 x 25 mLリザーバーホルダー」を3DプリントするためのSTLファイル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル4:カスタムラボウェアピペッティングロボット「マイクロプレートホルダー」のJSONファイル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル5:カスタムラボウェアピペッティングロボット「2 x 25 mLリザーバー Holder_WithCooler」のJSONファイル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル6:ピペッティングロボットプロトコル「Plating_ PDO_384well_Cooled_Row2-23」用のJSONファイル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル7:ピペッティングロボットデスクのセットアップの概要。 (A)冷却エレメント、(B)リザーバーおよびマイクロプレート。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル8:デジタル医薬品ディスペンサーのプロトコル用のTDDファイル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル9:明視野および蛍光イメージング用の生細胞イメージャーのプロトコル用のXMLファイル。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル10:プレートマップの例。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 11: NDR R スクリプトの入力ファイルの例 略語:NDR =正規化された薬物反応。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 12: 正規化された薬物反応 NDR R スクリプト。 略語:NDR =正規化された薬物反応。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 13: NDR R スクリプトの GR 値の出力ファイルの例。 略語:GR =成長率。NDR =正規化された薬物反応。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 14: GR 値が転置された NDR R スクリプトの出力ファイルの例。 略語:GR =成長率。NDR =正規化された薬物反応。 このファイルをダウンロードするには、ここをクリックしてください。
Sアッププレメンタリ ファイル 15: NDR R スクリプトの NDR 値の出力ファイルの例。 略語:NDR =正規化された薬物反応。 このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル 16: NDR 値が転置された NDR R スクリプトの出力ファイルの例。 略語:NDR =正規化された薬物反応。 このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
中スループットからハイスループットのPDTO薬物スクリーニングは、オルガノイドが潜在的に提供できる情報のほんの一部しか抽出しない読み出しに依存することがよくあります。急速に進化するオルガノイド技術がより大きな科学的および臨床的可能性を実現するためには、より高度な3Dアッセイ、読み出し、および分析方法が不可欠であることがますます明らかになっています。ここでは、再現性を高めるだけでなく、AI駆動の生細胞イメージング読み出しを組み込むことで臨床翻訳可能性を大幅に向上させる高度なスクリーニングパイプラインについて説明します。社内で開発された分析ソフトウェアに加えて、正規化された薬物反応指標(NDR)の使用が実装されており、治療反応における患者固有の違いを定義する能力を明確に示しています6。
この正規化指標を含めることは間違いなく非常に価値があり、多くの研究が曲線下面積(AUC)または半最大阻害濃度(IC50)のわずかな違いに基づいて治療反応を描写することを目的としていることを思い出してください(ほとんどの用量反応曲線は互いに重なり合っている/近くに位置しているため)11,12。.成長率の指標は、ATPベースのアッセイを用いたオルガノイド薬物スクリーニングプロトコルですでに実装されていますが、時点0から4で溶解された参照ウェルの正規化に依存しています。対照的に、この方法では、PDTO成長率の患者間差だけでなく、播種密度の変動とプレート位置依存効果に起因するウェル間差も考慮して再現性を高めるウェル内成長率の正規化が可能になります。さらに、正常化のためのポジティブコントロールを含めることにより、患者間PDTO応答の分離をさらに高めるようにNDRを適応させました6,8。
さらに、ハイスループットおよび自動化フォーマットと互換性のあるこの分析は、個々のオルガノイド応答を正確に検出できるため、腫瘍の再発と進行の主要な原動力であるサブクローナル耐性の定量が可能になります13。例えば、PDAC052およびPDAC060は in vitro での治療に対して良好な反応を示したが(NDRに基づく)、追加の単一オルガノイド分析では、治療に反応しないサブクローンの小さな(より大きな集団)集団を検出することができた。興味深いことに、PDAC052とPDAC060は持続的な反応(腫瘍活動は検出されなかった)を示したが、最終的には両方とも局所腫瘍進行と診断された(耐性クローンの存在による)ことを考えると、これは臨床観察と非常に一致した。従来の3D読み出し(ATPベースのアッセイとサイズ/数)と比較して、この高度なスクリーニングパイプラインは、これらの「ラボ内の患者」からより臨床的に関連する情報を抽出することにより、予測パフォーマンスを向上させることが期待されます。この仮説は現在、著者の研究室で臨床PDTOサンプルをスクリーニングすることによってテストされています この方法を使用して、 ex vivoを in vivo 応答および臨床転帰と相関させます。
薬物応答のメカニズムに関するより多くの洞察を得るために、細胞毒性色素に加えて、従来の蛍光生細胞イメージング試薬がこの方法に適合し、細胞死のメカニズムを研究します。シスプラチン処理後のカスパーゼ依存的なアポトーシス誘導を研究するために、この方法とザルトリウスカスパーゼ3/7グリーン試薬との適合性を以前に示しました8。酸化ストレス(CellROX試薬)または低酸素症(Image-iT低酸素試薬)を研究するための他の色素との適合性は、まだテストされていません。しかしながら、これらの試薬は、すでに3D インビトロ モデル14、15において首尾よく使用されている。
画像解析ソフトウェアは、オルガノイドの鮮明で焦点の合った画像をキャプチャできる場合、他のプレートフォーマットまたは培養方法(マイクロキャビティプレート、ECMドームなど)とも互換性があります。ドームで培養されたオルガノイドは異なるz面で成長するため、これはしばしば困難であり、常に利用できるとは限らない顕微鏡のzスタッキング機能が必要です。したがって、十分な品質の画像を確保するために、平底ULA 384ウェルマイクロプレートの使用をお勧めします。
さらに、この分析は、IncuCyte ZOOMシステム8でキャプチャされた位相差画像について前に示したように、他の生細胞イメージングシステムと互換性があります。この原稿で使用されたSpark Cyto生細胞イメージングシステムの制限は、速度論的測定のためのワンプレート容量です。ただし、Spark Motion拡張により、容量は最大40枚のマイクロプレートに増加し、まとめてスクリーニングできます。自社開発のソフトウェアの互換性をこれらのシステムや他のシステムに拡張し、プラットフォームに依存しないソリューションを提供し、画像およびデータ分析パイプラインの標準化と自動化を目標としています。Webベースのアプリケーションには、このホワイトペーパーに示すように、インタラクティブなグラフ作成ツールと自動化された薬物測定計算も含まれ、手動分析時間を短縮します。
ラベルフリーのPDTOセグメンテーションアルゴリズムは、明確な形態学的違い(固体、半固体、嚢胞性)を持つさまざまな社内成長回転楕円体およびPDTOモデルでトレーニングおよびテストされており、その結果、これらを高精度で検出できます8。このモデルの限界は、嚢胞性PDTOを含めると、播種後のウェルに存在する気泡の望ましくない検出が増加したことです。しかし、一晩のインキュベーションは、これらの気泡のほとんどを除去するのに十分であり、定性的なタイムポイント0スキャンを可能にした。オルガノイド画像のセグメンテーションと方法の精度は、他のユーザーによって検証される必要があり、そのフィードバックに基づいて、ソフトウェアをさらにトレーニングして、堅牢で自動化された画像分析アルゴリズムを取得できます。さらに、この方法で定量化された 生体外 薬物反応を患者の臨床反応と相関させるためのより多くの臨床データを取得し、治療反応を予測するための最良のパラメーターを特定し、機能的精密癌医学のためにこの方法をさらに開発することを目指しています16。
開示事項
著者は利益相反を宣言しません。
謝辞
この研究の一部は、Dedert Schilde vzwやWilly Floren氏を含むさまざまなドナーからの寄付によって資金提供されました。この研究の一部は、フランダース研究財団、12S9221N(A.L.)、G044420N(S.V.、A.L.、E.G.)、1S27021N(M.L.)、およびアントワープ大学の産業研究基金、PS ID 45151(S.V.、A.L.、C.D.)によって資金提供されています。 図 1 は BioRender.com で作成されました。
資料
| Name | Company | Catalog Number | Comments |
| 6-well plate | Greiner | 657160 | |
| 8-Channel p300 (GEN 2) pipette | Opentrons | ||
| 300 µL Tips | Opentrons | ||
| 384-well flat-bottom ULA microplate | Corning | 4588 | minimum volume 50 µL |
| 384-well flat-bottom ULA Phenoplate | Perkin Elmer | 6057802 | minimum volume 75 µL |
| A8301 | Tocris Bioscience | 2939 | |
| ADF+++ | Advanced DMEM/F12, 1% GlutaMAX, 1% HEPES, 1% penicillin/streptomycin | ||
| Advanced DMEM/F-12 | ThermoFisher Scientific | 12634 | |
| B27 | ThermoFisher Scientific | 17504044 | |
| Breathe easy sealing membrane | Sigma-Aldrich | Z380059 | |
| Caspase 3/7 Green | Sartorius | 4440 | |
| Cell Counting Slides for TC10/TC20 | Bio-Rad Laboratories | 1450017 | |
| CellTiter-Glo 3D | Promega | G9681 | ATP-assay |
| Cooler for 25 mL reservoir | VWR (Diversified Biotech) | 490006-908 | |
| Cooling element 12 x 8 x 3 cm | Bol.com | 9200000107744702 | For custom microplate holder OT-2 |
| Cultrex Organoid Harvesting Solution | R&D systems | 3700-100-01 | |
| Cultrex PathClear Reduced Growth Factor BME, Type 2 | R&D systems | 3533-010-02 | extracellular matrix (ECM) |
| Cytotox Green | Sartorius | 4633 | |
| Cytotox Red | Sartorius | 4632 | |
| D300e | Tecan | Digital drug dispenser | |
| D300e Control v3.3.5 | Tecan | Control software D300e | |
| FGF10 | Peprotech | 100-26 | |
| Full Medium | ADF+++ supplemented with 0.5 nM WNT surrogate-Fc-Fusion protein, 4% Noggin-Fc Fusion Protein conditioned medium, 4% Rpso3-Fc Fusion Protein conditioned medium, 1x B27, 1 mM N-acetyl cysteine (NAC), 5 mM nicotinamide, 500 nM A83-01, 100 ng/mL FGF10, and 10 nM Gastrin | ||
| Gastrin | Sigma-Aldrich | G9145 | |
| Gemcitabine | Selleck Chemicals | S1714 | |
| GlutaMAX | ThermoFisher Scientific | 35050 | |
| HEPES | ThermoFisher Scientific | 15630056 | |
| Hoechst 33342 Solution (20 mM) | ThermoFisher Scientific | 62259 | |
| Human pancreatic ductal adenocarcinoma (PDAC) patient-derived organoids | Biobank@uza (Antwerp, Belgium; ID: BE71030031000; Belgian Virtual Tumorbank funded by the National Cancer Plan) | ||
| N-acetyl-cysteine | Sigma-Aldrich | A9165-25G | |
| Nicotinamide | Sigma-Aldrich | N0636-100G | |
| Noggin-Fc Fusion Protein conditioned medium | Immunoprecise | N002 | |
| Opentrons App v6.0.1 | Opentrons | OT-2 control software | |
| Opentrons Protocol Designer Tool | Opentrons | https://designer.opentrons.com/ | |
| Orbits data compression tool | www.orbits-oncology.com or contact corresponding author | ||
| Orbits image analysis webapp | University of Antwerp | www.orbits-oncology.com or contact corresponding author | |
| OT-2 | Opentrons | Pipetting robot | |
| Paclitaxel | Selleck Chemicals | S1150 | |
| Pasteur Pipette 230 mm | Novolab | A33696 | |
| Peniciline-Streptomycin | ThermoFisher Scientific | 15140 | |
| Prism 9 | GraphPad | ||
| Rspo3-Fc Fusion Protein conditioned medium | Immunoprecise | N003 | |
| Spark Cyto 600 | Tecan | Live-cell imaging and multi-mode platereader | |
| SparkControl v3.1 | Tecan | Spark Cyto control software | |
| Staurosporine | Tocris Bioscience | 1285 | |
| Sterile 25 mL reservoir | VWR (Diversified Biotech) | 10141-922 | |
| T8 plus cassette | Tecan | ||
| TC20 | Bio-Rad Laboratories | automated cell counter | |
| TrypLE | ThermoFisher Scientific | 12604-021 | dissociation enzyme |
| Tween-20 | Acros Organics | 233360010 | |
| WNT Surrogate-Fc-Fusion protein | Immunoprecise | N001 | |
| Y-27632 | Selleck Chemicals | S1049 |
参考文献
- Drost, J., Clevers, H. Organoids in cancer research. Nature Reviews Cancer. 18 (7), 407-418 (2018).
- Veninga, V., Voest, E. E. Tumor organoids: Opportunities and challenges to guide precision medicine. Cancer Cell. 39 (9), 1190-1201 (2021).
- Le Compte, M., et al. Patient-derived organoids as individual patient models for chemoradiation response prediction in gastrointestinal malignancies. Critical Reviews in Oncology/Hematology. 157, 103190(2021).
- Driehuis, E., Kretzschmar, K., Clevers, H. Establishment of patient-derived cancer organoids for drug-screening applications. Nature Protocols. 15 (10), 3380-3409 (2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nature Methods. 13 (6), 521-527 (2016).
- Gupta, A., Gautam, P., Wennerberg, K., Aittokallio, T. A normalized drug response metric improves accuracy and consistency of anticancer drug sensitivity quantification in cell-based screening. Communications Biology. 3 (1), 42(2020).
- Rios, A. C., Clevers, H. Imaging organoids: a bright future ahead. Nature Methods. 15 (1), 24-26 (2018).
- Deben, C., Cardenas, E., et al. OrBITS: label-free and time-lapse monitoring of patient derived organoids for advanced drug screening. Cellular Oncology. , (2022).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. Bioessays. 39 (8), (2017).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Engel, R. M., et al. Patient-derived colorectal cancer organoids upregulate revival stem cell marker genes following chemotherapeutic treatment. Journal of Clinical Medicine. 9 (1), 128(2020).
- Hennig, A., et al. Detecting drug resistance in pancreatic cancer organoids guides optimized chemotherapy treatment. The Journal of Pathology. 257 (5), 607-619 (2022).
- D'Alterio, C., Scala, S., Sozzi, G., Roz, L., Bertolini, G. Paradoxical effects of chemotherapy on tumor relapse and metastasis promotion. Seminars in Cancer Biology. 60, 351-361 (2020).
- Njoroge, R. N., et al. Organoids model distinct Vitamin E effects at different stages of prostate cancer evolution. Scientific Reports. 7 (1), 16285(2017).
- Godet, I., et al. Fate-mapping post-hypoxic tumor cells reveals a ROS-resistant phenotype that promotes metastasis. Nature Communications. 10 (1), 4862(2019).
- Letai, A. Functional precision cancer medicine-moving beyond pure genomics. Nature Medicine. 23 (9), 1028-1035 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved